Method Article
3 ' نهاية تسلسل إعداد مكتبة مع A-seq2
In This Article
Summary
ويصف هذا البروتوكول طريقة لرسم الخرائط مرناً قبل 3 ' نهاية تجهيز المواقع.
Abstract
وأظهرت الدراسات في العقد الماضي مجموعة متنوعة معقدة ودينامية مرناً قبل الانقسام وردود الفعل بوليادينيليشن. مرناس مع مناطق 3 'أهمل طويلاً (تحيلها) يتم إنشاؤها في الخلايا المتمايزة حين تكاثر الخلايا تفضيلي التعبير عن النصوص مع قصيرة 3' تحيلها. ونحن تصف البروتوكول A seq، الآن في نسخته الثانية، التي وضعت خريطة مواقع بوليادينيليشن على نطاق الجينوم ودراسة تنظيم مرناً قبل 3 ' نهاية المعالجة. كما يستفيد هذا البروتوكول الحالي من بوليادينيلاتي (ذيول poly(A)) التي يتم إضافتها أثناء نشوء حيوي لمعظم الثدييات مرناس لإثراء مرناس المجهزة تماما. محول الحمض النووي مع ديوكسيوراسيل في موقفها الرابعة تسمح معالجة دقيقة مرناً 3 ' نهاية الأجزاء للتسلسل. لا بما في ذلك زراعة الخلايا وليجيشنز بين عشية وضحاها، يتطلب البروتوكول وقت التدريب العملي على ح حوالي 8. جنبا إلى جنب معها، يتم توفير حزمة برامج سهلة الاستخدام لتحليل البيانات المشتقة من التسلسل. A-seq2 والبرامج المرتبطة بها تحليل توفر حلاً فعالة وموثوق بها للتعيين من قبل-مرناً 3 ' ينتهي في طائفة واسعة من الظروف، من 106 أو خلايا أقل.
Introduction
التقاط وتسلسل مرناً 3 ' ينتهي يتيح دراسة تجهيز مرناً والتحديد الكمي للتعبير الجيني. سبب ذيولها poly(A)، مرناس التوكسينات يمكن أن تكون كفاءة تنقيته من ليساتيس خلية الإجمالي مع المعطل تداولها حبة اليغو-ديوكسيثيميديني (oligo(dT)) الجزيئات، التي يمكن أيضا أن رئيس الوزراء كدنا التوليف. بيد أن هذا النهج قد اثنين من العوائق. أولاً، يمكن أن تمتد من أ أن تكون داخلية للنصوص أيضا رئيس الوزراء توليف كدنا، أسفر عن مواقع زائفة poly(A). Poly(A) الثاني، ومتجانسة تمتد تحديات محددة للتسلسل، إلى جانب كونها غير مفيدة لتحديد نص. قد اقترحت نهوج مختلفة للتحايل على هذه القيود، مثل النسخ العكسي عن طريق poly(A) ذيول تليها "ح رناسي" الهضم (ف 3-seq 1)، استخدم من التمهيدي تسلسل مخصص المنتهي في 20 Ts (seq 2 ف- 2)، الاختيار الأولى من الأجزاء رنا مع ذيول poly(A) النيوكليوتيدات ما يزيد على 50 مع تمهيدي45 CU5T متبوعاً بالهضم "ح رناسي" (ما يلي: 3 ' 3)، واستخدام اليغو-dT التمهيدي الذي يحتوي على محول 3' في دبوس (seq أ 4).
الأسلوب A-seq2 5 وضعت مؤخرا يهدف إلى تجاوز التسلسل عن طريق poly(A) وفي نفس الوقت تقليل نسبة dimers التي يتم إنشاؤها بواسطة ربط الذاتي لمحولات، خاصة التي تحدث عند تركيز مولى للمحولات يفوق تركيز إدراج. ويمكن القضاء على هذه المشكلة عندما تكون متصلة كلا محولات لنفس النوع من نهايات بولينوكليوتيدي كما هو الحال في أ-seq2، أين هي متصلة المحولات 3 'إلى 5' نهاية الأجزاء رنا والمحولات 5 'إلى 5' ينتهي كدناس بعد النسخ العكسي. الأسلوب أكثر ملاءمة من أعمالنا المقترح سابقا A-seq-التي كان تسلسلها في 5 '-إلى-3' التحكم في اتجاه مما يتطلب تحديداً الحمض النووي الريبي التجزؤ-، مع الحفاظ على درجة عالية من دقة في تحديد موقع poly(A). حوالي 80% من ما يلي التسلسل في عينات نموذجية خريطة فريدة للجينوم وتؤدي إلى كشف هوية من 20,000 أكثر مجموعات الموقع poly(A)، أكثر من 70% من أي تداخل مع المشروح 3 ' تحيلها.
وباختصار، يبدأ البروتوكول A-seq2 مع تجزئة مرناً وربط محولات عكس-تكملة 3 'إلى 5' نهايات الأجزاء رنا. بولي (A)-تحتوي على الكشف ثم يتم عكس نسخها مع تمهيدي oligo(dT) طويلة النوكليوتيدات 25 (nt) يحتوي النوكليوتيدات مرساة في نهاية 3 '، من دو في موقف 4 وبيوتين في 5' نهاية، يسمح ملزمة لكدنا الخرز streptavidin المغناطيسي. تتم إزالة معظم التمهيدي، بما في ذلك البيوتين، من كدنا قبل الانقسام في دو بمزيج الإنزيم المستخدم، التي تحتوي على "الحمض النووي اليوراسيل" glycosylase (UDG) و "الثامن اندونوكلياسي" جليكوسيلاسي لياز الحمض النووي. رد الفعل هذا يترك نهايات سليمة لربط محول 5 '، واليسار إتس الثلاثة بعد انشقاق تبقى لوضع علامة على موقع الذيل poly(A). لكل 5 '3' المحولات ومرفقه بربط للمستلم 5 ' الغايات، يتم إنشاء لا dimers محول. يسمح قرار الكتلة في تسلسل الدولة من أحدث الصكوك الأربعة النوكليوتيدات عشوائية-أسعار الصرف السوقية قدم في بداية ما يلي: ويمكن أيضا بمثابة المعرف الفريد الجزيئية (أومي) لكشف وإزالة الآثار التضخيم PCR. يمكن زيادة حجم أومي كما فعلت في دراسات أخرى 6. ينشئ البروتوكول بما يلي: أن يتم عكس مكملة مرناً 3 'ينتهي، كل شيء بدءاً من تيترامير معشاة ذات شواهد تليها 3 Ts-تجهيز من القراءات التي لها للملخص التشخيصي 3 في نهاية يبدأ 5' مع تصحيح PCR التضخيم التحف استغلال أميس، إزالة 3 ' تسلسل محول، وعكس التكامل. أيضا تحديد حسابياً القراءات التي قد تكون قد نشأت من فتيلة oligo(dT) الداخلية الغنية بالمواقع والتخلص منها. المواقع الزائفة التي تفتقر عموما إلى واحد من 18 تتسم جيدا وإشارات حفظت poly(A) الذي ينبغي أن يقع النيوكليوتيدات ~ 21 المنبع من موقع الانقسام الظاهر 7.
يتطلب البروتوكول وقت التدريب العملي على ح حوالي 8، لا عد زراعة الخلايا وليجيشنز بين عشية وضحاها. المرتبطة بها قراءة تحليل البرمجيات تمكن من تحديد موقع poly(A) درجة عالية من دقة. من موقع poly(A) إنشاء مجموعات استناداً إلى عينات 4 كذلك والضوء على هذه المخطوطة (اثنان replicates البيولوجية siRNA التحكم وخلايا سي-هنرنبك-تعامل) 84% التداخل مع جينات مشروح، وهذه، 75% من التداخل مع 3 ' UTR، 86% أما مع 3 ' UTR أو إكسون المحطة طرفية. معامل ارتباط Pearson للتعبير عن 3 ' ينتهي في عينات تكرار 0.92، وعادة ما يتم الحصول على قيم من خلال 0.9 مع الأسلوب. وهكذا، A-seq2 هو أسلوب مناسب يعطي نتائج جداً استنساخه.
Protocol
1-"نمو الخلايا" ومرناً العزلة
- تنمو الخلايا طبقاً للتصميم الخاص بك التجريبية في لوحات 6-جيدا إلى ~ 1 × 10 6 خلايا كل بئر في التقاء 80 ٪. إزالة متوسط النمو
- وغسل الخلايا مرة واحدة مع الفوسفات مخزنة المالحة. الخلايا الموجودة على اللوحة مباشرة بإضافة 1 مل من المخزن المؤقت لتحلل من الطقم مرناً والعزل. نقل لزوجة في أنبوب بلاستيكي 15 مل مع تلميح ماصة 1 مل. استخدام ملعقة مطاط لفصل المواد الخلية من سطح اللوحة تماما. القص
- الحمض النووي اللزج الذي يتضمن ليستي مع حقنه 1 مل إرفاقه إبرة حقن ز 23 قبل عدة قوي صعودا وهبوطاً حركات المكبس حتى لم يعد لزج. نقطة إبرة المحقن إلى وسط الجزء السفلي لتجنب إخراج ليساتي خارج الأنبوبة.
- نقل في أنبوب 1.5 مل استخدام المحاقن. تدور 5 دقيقة في 20,000 x ز و 4 درجة مئوية لإزالة الأنقاض. استخدام الحمض النووي قنينات 1.5 مل ربط منخفضة في جميع أنحاء البروتوكول. أغسل
- في حين يعمل الطرد المركزي، 300 ميليلتر من حراكه اليغو (dT) 25 المغناطيسي الخرز على رف مغناطيسي مع 500 ميليلتر من تحلل المخزن المؤقت. مزيج من هذه الأنابيب 2-3 مرات على الرف. إزالة المخزن المؤقت بعد الحل الواضح. جمع المادة طافية واضحة من الخطوة 1، 4 وإضافة إلى الخرز. ريسوسبيند ووضع أنابيب على عجلة دوارة للحد الأدنى 10
- وضع الأنابيب على رف مغناطيسية. إزالة السائل واضحة بعد 2 دقيقة إضافة 0.8 مل المخزن المؤقت A من الطقم مرناً والعزل. تشغيل الأنبوب بدرجة 180 درجة على الرف، 2-3 مرات. كرر هذه الخطوة الغسيل مرة أخرى مع المخزن المؤقت ألف
- يغسل الخرز 2 مرات مع 0.8 مل من المخزن المؤقت ب كما هو موضح في الخطوة 1، 6. إضافة 33 ميليلتر ح 2 س
- إلى الوت مرناً منضم من الخرز، والخرز ريسوسبيند. الحرارة إلى 75 درجة مئوية لمدة 5 دقائق على كتلة ساخنة. تدور فورا أنابيب 1 s ومكان لهم على الرف المغناطيسية. نقل المادة طافية إلى أنبوب جديد. ويمكن تخزين العينات في-80 درجة مئوية حتى استخدامها مرة أخرى. ميليلتر
- 66 إضافة ميليلتر التحلل القلوي المخزن المؤقت إلى 33 مرناً (الخطوة 1.8)، مزيج والحرارة لمدة 5 دقائق بالضبط عند 95 درجة مئوية في كتلة تدفئة. فورا تشيل أنابيب على مدت
- "عزل الحمض النووي الريبي" مع مجموعة مواد تنظيف رنا.
ملاحظة: تأكيد وحدة التخزين؛ ينبغي أن يكون 100 ميليلتر.- إضافة 350 ميليلتر RLT المخزن المؤقت من الطقم و 250 ميليلتر الإيثانول. تحميل على العمود وتدور لمدة 30 ثانية في 8,000 س ز في درجة حرارة الغرفة (RT). يغسل مع 500 ميليلتر RPE العازلة من الطقم. يغسل مع 500 الإيثانول 80% ميليلتر. تدور لمدة 5 دقائق في ز x 20,000 الجافة العمود. إضافة 36 ميليلتر ح 2 س إلى عمود وتدور لتجاهل العمود 1 دقيقة في غ. س 20,000 وحفظ النذرة.
2-5 ' إنهاء الفسفرة والعلاج الدناز
- إضافة 5 ميليلتر بولينوكليوتيدي كيناز المخزن المؤقت، 5 ميليلتر 10 ملم ATP، 1 ميليلتر ribonuclease المانع، 1 ميليلتر الدناز وميليلتر بولينوكليوتيدي كيناز 2 إلى عينات واحتضان في 37 درجة مئوية لإعداد 30 دقيقة اختيارياً يمزج رد الفعل الرئيسي في جميع أنحاء البروتوكول بخلط كميات 1.1 x n (n = عدد العينات) كل مكون من مكونات.
- تغيير المخزن المؤقت وإزالة ATP على عمود دوران لمنع إضافة poly(A) في الخطوة التالية-
أعمدة-سبين
- بريسبين في غ س 735 1 كحد أدنى لنقل الأعمدة الجديدة 1.5 مل القنينات وتحميل ردود فعل كيناز على الأعمدة. زيادة ونقصان الأعمدة 2 دقيقة في 735 x زاي تجاهل الأعمدة ووضع الأنابيب مع ردود الفعل التي تم جمعها على الجليد أو تخزينها في-80 درجة مئوية.
3. حظر 3 ' ينتهي مع ثلاثي كورديسيبين
ملاحظة: من الضروري حظر 3 ' نهايات الأجزاء رنا لتجنب ما كونكاتيميريزيشن في ربط اللاحقة ردود فعل-3 ' نهايات لا يتم حظره بالفعل قبل ( دوري) الفوسفات بعد تعامل التحلل المائي بالإضافة 3 ' النوكليوتيدات فاصل سلسلة داتب (كورديسيبين ثلاثي الفوسفات) مع معونة بوليميراز poly(A). وهنا استخدمت بوليميراز poly(A) الخميرة (يباب)، الذي أعرب عن وتنقيته كما هو موضح في 8 في تركيز من 0.5 ملغ/مل. خميرة أو كولاي PAP كلا لها تقريبا نفس النشاط لإضافة 3 ' داتب ويمكن شراؤها تجارياً (انظر الجدول للمواد)-
المخزن المؤقت لرد فعل بوليميراز- ميليلتر 13.5 إضافة 5 × poly(A) مركزة، 2 ميليلتر من 10 ملم 3 ' داتب ومثبط رناسي ميليلتر 1 1 بوليميراز poly(A) ميليلتر لرد فعل خطوة 2.2.1. ميكس وتدور على إينكوباتي س. 1 في 37 درجة مئوية للحد الأدنى 30 32.5 إضافة ميليلتر ح 2 س لكل رد فعل. تنقية الحمض النووي الريبي كما في الخطوة 1.10.1. الوتي الجيش الملكي النيبالي مع 14 ميليلتر ح 2 أولمبيك
4. ربط 3 عكس ' محولات إلى 5 ' "نهاية الأجزاء رنا"
- بوضع ردود الفعل في مركز فراغ لمدة 10 دقائق إلى تقليص وحدة التخزين بإضافة 3 ميليلتر ميليلتر. 6 10 × الجيش الملكي النيبالي T4 ربط المخزن المؤقت، 3 ATP ملم 10 ميليلتر ، 15 ميليلتر PEG-8000، مثبط رناسي ميليلتر 1، 1 ميليلتر من 0.1 ملم عكس تكمل 3 ' محول " revRA3 " (انظر الجدول للمواد) ومزيج 1 ميليلتر ارتفاع تركيز الحمض النووي الريبي ليجاسى 1،.
- احتضان ردود الفعل عند 24 درجة مئوية ح 16 في خلاط مياه دافئة مع خلط متقطعة 1,000 لفة في الدقيقة. إضافة 70 ميليلتر ح 2 س لكل رد فعل، ومزيج. تنقية الحمض النووي الريبي كما في الخطوة 1.10.1. الوت الجيش الملكي النيبالي مع 14 ميليلتر ح 2 يمكن تخزين العينات O. في-80 درجة مئوية عند هذه النقطة-
5. عكس النسخ (RT)
- مكان أنابيب الواتس في مركز فراغ لمدة 3 دقائق إلى الحد من الحجم لنقل ردود ميليلتر. 11 إلى 200 ميليلتر PCR. أضف 1 ميليلتر 0.05 مم RT التمهيدي " بيو-دو-dT25 ". الحرارة لمدة 5 دقائق في 70 درجة مئوية في cycler بكر وترك على RT للحد الأدنى 5 إضافة
- دنتبس ملم 10 ميليلتر 1 4 5 x ميليلتر المنتسخة العكسية المخزن المؤقت، 1 ميليلتر 0.1 M DTT، 1 ميليلتر رناسي المانع والمنتسخه العكسية 1 ميليلتر. خلط والحرارة ردود الفعل لمدة 10 دقائق إلى 55 درجة مئوية و 10 دقيقة إلى 80 درجة مئوية في cycler PCR. الحفاظ على الجليد أو في-80 درجة مئوية لتخزين أطول.
6. الهضم مع "اليوراسيل DNA Glycosylase ميكس الإنزيم"
- ميليلتر 100 بيبيت الخرز ستريبتافيدين في قنينة 1.5 مل، ريسوسبيند في 800 ميليلتر البيوتين ملزمة المخزن المؤقت ووضع على رف مغناطيسية. عكس أنابيب 2-3 مرات. إزالة المخزن المؤقت عندما واضحة. كرر الخطوة الغسيل. ريسوسبيند الخرز في 200 ميليلتر البيوتين ملزمة العازلة.
- إضافة النسخ العكسي كرد فعل للحل الخرز واحتضان 20 دقيقة في 4 درجات مئوية على عجلة دوارة. أغسل الخرز x 2 مع ربط البيوتين المخزن المؤقت كما في الخطوة 6.1 و 2 x مع المخزن المؤقت عشرة على رف مغناطيسية. ريسوسبيند الخرز في 50 ميليلتر عشرة العازلة، وإضافة 2 ميليلتر اليوراسيل DNA glycosylase إنزيم مزيج، واحتضان ح 1 في 37 درجة مئوية في خلاط مع خلط متقطعة. المخزن المؤقت
- إضافة 50 ميليلتر ح 2 س، 11 ميليلتر من "ح رناسي" وميليلتر 1 ح رناسي إلى ردود الفعل. احتضان في 37 درجة مئوية لأنابيب مكان 20 دقيقة على رف مغناطيسية ونقل السوائل المحتوية على كدنا ملصوق بأنبوب جديد
- تنقية كدنا ملصوق.
- ميليلتر 550 إضافة المخزن المؤقت PB من الطقم تنقية PCR إلى الانقسام وردود الفعل. إضافة 10 ميليلتر من خلات الصوديوم 3 أمتار، pH 5.2 لخفض درجة الحموضة. تحميل ردود الفعل في أعمدة الدوران شطف الحد الأدنى وتدور في 17,000 س ز للحد الأدنى 1 تجاهل
- 750 إضافة ميليلتر المخزن المؤقت PE للأعمدة وتدور في 17,000 س ز للحد الأدنى 1-التدفق عبر. تدور الأعمدة في 17,000 س ز لمدة 1 دقيقة حتى تجف. نقل الأعمدة إلى قنينة 1.5 مل، إضافة 16 ميليلتر ح 2 س وتدور في 17,000 س ز للحد الأدنى 1 وضع ردود فعل في مركز فراغ ل 8 دقيقة التركيز إلى وحدة تخزين ميليلتر 7-
7. ربط 5 ' محولات إلى 5 ' نهايات كدنا
- لكدنا معزولة، إضافة 3 ميليلتر 10 س رنا T4 العازلة ليجاسى 1، 3 ATP ملم 10 ميليلتر، 15 ميليلتر PEG-8000، 1 ميليلتر 50 ميكرون " revDA5 " اليغو ، و 1 ميليلتر ارتفاع تركيز T4 الجيش الملكي النيبالي ليجاسى 1. احتضان عند 24 درجة مئوية ميليلتر 70 إضافة حاء 20 ح 2 س لكل رد فعل. ويمكن تخزين العينات في-20 درجة مئوية في هذه المرحلة-
8. رائد بكر والتضخيم من المكتبات وتحديد حجم
- في رد فعل تجريبية، تحديد العدد الأمثل لبكر دورات للوصول إلى التضخيم المكتبة ضمن المرحلة الأسية.
- 25 بيبيت ميكس بوليميراز الدنا ميليلتر والرد ربط ميليلتر 20، 2 ميليلتر ح 2 س، 1.5 ميليلتر 10 ميكرون إلى الأمام بكر التمهيدي (RP1) و 1.5 ميليلتر 10 ميكرون عكس PCR الفهرس التمهيدي في أنبوب بكر ميليلتر 200- تشغيل
- cycler مع البرنامج التالي: 3 دقيقة 95 درجة مئوية، تليها دورات 20 من 20 s 98 درجة مئوية، 20 ق 67 درجة مئوية وق 72 30 ° 7 جمع جيم ميليلتر مختبرين بعد دورات 6، 8، 10، 12، 14 و 16 و 18 مباشرة من cycler. أضف 1 ميليلتر 10 × تحميل المخزن المؤقت (والغليسيرول 50%، زايلين زيلين نفط 0.05%). ملاحظة: الرجاء اتباع توصيات المورد إذا كان استخدام الإرسال المتعدد عند الجمع بين الرموز الشريطية.
- منتجات منفصلة في فتحات صغيرة في 2% [اغروس] هلام في مخزن x TBE 1 يحتوي على 01:10، 00 تمييع الفلورسنت الصبغة الخضراء.
- مختبرين تحميل على 2% [اغروس] هلام وتشغيل الجل على 100 فولت ل 15 دقيقة تصور الهجرة من منتجات PCR على نظام توثيق جل.
- استخدام عدد الدورات في بداية التضخيم الهائل في رد فعل التجريبية لفعل بكر واسعة النطاق مع ضعف وحدات التخزين المستخدمة لرد الفعل التجريبي ( الشكل 2).
- للتفاعلات بكر واسعة النطاق، التركيز وتحلية ردود الفعل الأولى مع مجموعة تنقية PCR وفصل المنتجات الموجودة في فتحات واسعة على 2% [اغروس] المواد الهلامية في المخزن المؤقت x TBE 1-
- قطع جل الشرائح التي تحتوي على 200-350 nt الحمض النووي المنتجات. تذوب الهلام في المخزن المؤقت تشاوتروبيك في RT لمدة تصل إلى 30 دقيقة. استخراج الحمض النووي من شرائح هلام مع مجموعة أدوات استخراج هلام. لا الحرارة إلى 50 درجة مئوية لمنع التحيز في ربط الغنية بالحمض الخلوي الصبغي 9.
- يقدم لتسلسلها.
ملاحظة: بشكل عام، 50 دورة واحدة للقراءة (SR50) كافية (انظر، على سبيل المثال، https://www.illumina.com/technology/next-generation-sequencing.html).
9. تجهيز البيانات
ملاحظة: تتم معالجة البيانات التسلسل الناتج (في شكل فستق) مع البرمجيات المتاحة في المستودع جيتلاب (https://git.scicore.unibas.ch/zavolan_public/A-seq2-processing). التحليل يتضمن أربع خطوات رئيسية: (1) تحميل بوابة المستودع، (2) تركيب بيئة ظاهرية، (3) وضع معايير محددة في ملف التكوين و (4) الشروع في التحليل من خلال ‘ سناكيماكي ’ 10-تحليل كامل القيام به في الخطوة 4 يتطلب أمر واحد فقط. ويمكن الاطلاع على وصف مفصل خطوة بخطوة للتحليل في الملف التمهيدي في المستودع جيتلاب ووصف قصير المتوفرة أدناه. جميع خطوات المعالجة الفردية هي إنجاز بتنفيذ أدوات متاحة للجمهور، وأما من مصادر خارجية أو أعد داخليا. خط الأنابيب الحسابية تعتمد على بيئة ظاهرية بيثون 3 المستندة إلى أناكوندا 11 تتوفر حزمة 10 سناكيماكي. تشغيله على أجهزة الكمبيوتر مع نظام التشغيل شبيه يونكس وتم اختباره في بيئة لينكس مع تثبيت نظام التشغيل CentOS 6.5 و 40 جيجابايت من ذاكرة الوصول العشوائي المتاحة. تبعيات برنامج يتم التحكم تلقائياً داخل بيئة افتراضية. أدوات البرمجيات المتاحة للعموم التالية هي المطلوبة وتثبيتها وبالتالي جنبا إلى جنب مع البيئة: سناكيماكي (v3.9.1) 10، فاستكس (v0.0.14) مجموعة أدوات 12، ستار (v2.5.2a) 13 ، كوتادابت (v1.12) 14, 14 ، سامتولس (v1.3.1) 15, 16 ، بيدتولس (v2.26.0) 17-
- قبل معالجة البيانات من يقرأ على كدناس
ملاحظة: قد تختلف عمق التسلسل بين يدير، وتبعاً لهذا الصك، يمكن تقسيم البيانات من نموذج واحد على ملفات متعددة في تسلسل. إذا كان هذا هو الحال، سلسلة الملفات التي تتوافق مع عينة واحدة في ملف واحد إدخال الذي يتم استخدامه في الخطوات التالية.- من فستق بتحويل الملف إلى تنسيق fasta.
- استخراج يقرأ مع بنية صحيح (ثيميدينيس 3 في مواقف 5 و 6 و 7 من القراءة)-
ملاحظة: قراءة يتم إعدادها بشكل صحيح وفقا للبروتوكول التجريبي المبين أعلاه ينبغي أن يكون الهيكل (من 5 ' نهاية): عكس الباركود 4-النوكليوتيدات-ثيميدينيس 3-تكملة لنسخة 3 ' نهاية. - تخزين معلومات عن تيترامير البدء في السطر وصف التسلسل.
ملاحظة: تيترامير بمثابة معرف فريد جزيئية (أومي) التي تسهل تصحيح التحف التضخيم لاحقاً في التحليل- - إزالة النيوكليوتيدات السبعة الأولى من القراءة ' s 5 ' نهاية.
- تصحيح لإدراج التحف التضخيم بالاحتفاظ بنسخة واحدة فقط من ما يلي بنفس التسلسل واومي.
- إزالة الجزء 3 ' نهاية تطابق تسلسل محول وتكملة ثم عكس التسلسل. فقط المضي قدما في ما يلي: أن يكون طول الحد أدنى (الافتراضي: 15 nt).
ملاحظة: اعتماداً على طول الجزء مرناً الأصلي وعدد دورات التسلسل، و 3 ' نهاية القراءة قد يحتوي على جزء من 3 ' محول، والتي يتم إزالتها في هذه الخطوة.
- استخراج كل القراءات التي تفي بالمعايير التالية: الحد الأقصى النيوكليوتيدات غير معروف 2 (' N ')، % 80 كحد أقصى كما، والنوكليوتيدات الأخير للقراءة لا ألف وتعتبر هذه القراءات ذات نوعية كافية لاستخدامها في التحليل-
- الخريطة على ما يلي للجينوم مع أداة أن يتعامل مع القراءات المقسمة، ويقوم بإنشاء ملف إخراج في تنسيق أم.
يستخدم
- "ستار إذا"، قم بإنشاء ملف مع الرقم القياسي للجينوم التي ينبغي تعيينها على ما يلي. تتطلب هذه الخطوة للجينوم البشري، 35 غيغابايت من الذاكرة (RAM). خريطة
- على ما يلي للجينوم.
ملاحظة: (الملاحظات الخاصة بنجمة) القطع الناعمة معطل لإرغام الحكومة على تعيين 3 ' نهاية كل قراءة هذا هو النوكليوتيدات المنبع مباشرة من موقع الانقسام.
- تحويل في سوق بكارا للأسلحة إلى ملف سرير. إذا كانت قراءة خرائط لمواقع متعددة، تبقى فقط تلك مع أدنى تحرير مسافة.
ملاحظة: يتم استخدام عدد نسخ قراءة معين في موقع معين كنقاط. يتم عد ما يلي: أن خريطة بمواقع متعددة فركايشنلي في كل موقع بوزن يساوي 1/عدد المواقع التي قراءة الخرائط- - انهيار القراءات التي تختلف بوجود خطأ في تسلسل المحتمل. إذا كانت خريطة بين القراءات المتميزة لنفس الموقع (بداية ونهاية الموقف من التعيينات متطابقة) وحصة أومي نفسه، ينظر لهم كبكر التكرارات، وإبقاء واحد فقط.
- الاستدلال الفردية كل ما قبل-مرناً 3 ' إنهاء تجهيز المواقع.
ملاحظة: لقراءة فردية يقدم الأدلة على 3 ' تنتهي عند ما النيوكليوتيدات الأربعة الأخيرة تم تعيينها إلى الجينوم دون خطأ. الموقف الذي 3 ' نهاية قراءة الخرائط التي يتم تخزينها كموقع الانقسام. - كشف 3 ' وضع حد للمواقع التي يمكن أن تكون قد نشأت من فتيلة الداخلية. تعريف الموقع كقطعة أثرية فتيلة الداخلية عند تلبية nt المصب موقع الانقسام في الجينوم 10 واحد من المعايير التالية: يحتوي على أكثر من ستة كما، ويحتوي على ستة متتالية ك، أو يبدأ بأحد تيتراميرس التالية: AAAA، أغا، آغا، آآج .
- توليد جدول 3 الفردية ' إنهاء تجهيز المواقع في شكل السرير.
- بشكل مستقل ينظم تحديد مجموعات الموقع poly(A).
ملاحظة: اتبع الخطوات الموضحة هنا الإجراء الذي تم تقديمه في منشور مسبق 5.- ابدأ بجمع 3 الفردية ' إنهاء تجهيز المواقع التي تم الحصول عليها في جميع عينات الدراسة.
- تعليم poly(A) معروفة إشارات 7 في منطقة 09:00 ص النيوكليوتيدات حول كل 3 الفردية ' نهاية تجهيز الموقع.
- Poly(A) تحديد المواقع المعرب عنها أعلاه الخلفية في كل عينة على النحو التالي.
- فرز المواقع حسب تعبيرها الخام داخل النموذج الحالي. اجتياز قائمة المواقع من أعلى إلى أسفل، إقران مواقع المرتبة الدنيا موقع مرتبة أعلى إذا كانت تقع على مسافة محددة مسبقاً في الجينوم (الافتراضي: 25 nt حتى-أو المتلقين للمعلومات) من موقع رفيع المستوى-
ملاحظة: جميع الرتب الدنيا المواقع المرتبطة بموقع رفيع المستوى تعريف كتلة التعبير الذي هو عدد القراءات توثيق جميع هذه المواقع. - فرز هذه المجموعات بالتعبير واجتياز قائمة مجموعات من أعلى إلى أدنى التعبير، تحديد عتبة التعبير ج فيه إشارة النسبة المئوية لمجموعات مع poly(A) مشروحة انخفاض أقل عتبة المحددة مسبقاً ( الافتراضي: 90%).
- تجاهل المواقع من أي كتلة أدناه قطع.
- فرز المواقع حسب تعبيرها الخام داخل النموذج الحالي. اجتياز قائمة المواقع من أعلى إلى أسفل، إقران مواقع المرتبة الدنيا موقع مرتبة أعلى إذا كانت تقع على مسافة محددة مسبقاً في الجينوم (الافتراضي: 25 nt حتى-أو المتلقين للمعلومات) من موقع رفيع المستوى-
- عن كثب متباعدة الكتلة 3 ' وضع حد للمواقع التي تم الحصول عليها عبر العينات.
ملاحظة: نوع 3 ' عدد مرات القراءة في نهاية تجهيز المواقع أولاً بالعدد من دعم عينات ومن ثم على مجموع تطبيع (يقرأ كل مليون (لفة في الدقيقة)) عبر العينات. تعبر القائمة من أعلى إلى أسفل، إقران مواقع المرتبة الدنيا مواقع أعلى مرتبة عند هذه المسافة إلى موقع أعلى رتبة ليست أكبر من حد معرفة مسبقاً (الافتراضي: 12 nt). كلما أي من 3 التي تشكل ' الموقع نهاية يتداخل مع إشارة poly(A) مشروح أو بإشارة poly(A) مباشرة المصب، يتم وضع علامة نظام المجموعة المطابق لمواصلة التفتيش للكشف عن فتيلة الداخلية-
موقع - poly(A) دمج مجموعات-
ملاحظة: عند وضع علامة كتلة كمرشح فتيلة داخلية المفترضة، أنه أما دمجها في كتلة المتلقين للمعلومات إذا كانت حصة المجموعتين إشاراتها poly(A) أو الاحتفاظ بها إذا كان الموقع الأكثر المصب في الكتلة إشارة poly(A) الموقع كحد أدنى مسافة المنبع (الافتراضي: 15 nt). وأخيراً، يتم دمج المجموعات المتقاربة إذا: (ط) أنهما يشتركان في نفس signal(s) poly(A)، أو (الثاني) تمتد الكتلة الناتجة لا تزيد كحد أقصى (الافتراضي: 25 nt). - تخزين الكتل في تنسيق ملف سرير مع مجموع تطبيع عدد مرات القراءة من كل 3 ' في نهاية مواقع في كل مجموعة كنقاط.
النتائج
بولي (A)-التي تحتوي على الحمض النووي الريبي قد تم عزلة من الخلايا المستزرعة، مجزأة بواسطة التحلل المائي القلوي، وقدمت كدناس عكس النسخ مع الإشعال oligo(dT). كدنا الناتجة كانت معطلة في الخرز streptavidin، كان المشقوق دو في رد فعل الختان محددة اليوراسيل ومحولات كانت متصلة إلى 5 'وكان التسلسل 3' ينتهي الجزء ملصوق وتدرج. ويصور الشكل 1 مخطط رسومي للتجربة.
لخلايا هيلا و HEK293، كانت 106 خلايا كافية لتحديد مواقع poly(A) للغالبية العظمى من الجينات ترميز البروتين في نهاية الإجراء. ومع ذلك، لأنواع الخلايا أو الأنسجة قد يكون من الضروري لاختبار التشبع في عدد المواقع poly(A) التي تم تحديدها كعدد الخلايا التي استخدمت في التجربة الأخرى زيادات. النتائج ممثلة لبكر التجريبية خطوة وجزء الحمض النووي وتحليل العينة قبل تسلسل ترد في الشكل 2.
ويبين الشكل 3 خطوات ما قبل المعالجة التحليل الحسابي، بدءاً من الملف فستق التي تم الحصول عليها من جهاز التسلسل وانتهاء بما يلي: التحقق من الجودة، وقلص محول التي على استعداد ليتم تعيينها إلى الجينوم. ويبين الشكل 4 تحليل الخطوات التي تبدأ برسم خرائط ليقرأ على الجينوم ونهاية المقابلة مع كتالوج مرناً 3 ' نهاية تجهيز المواقع التي تم تحديدها في نموذج معين. عندما يتم تحليل عينات متعددة، تنفذ خطوات إضافية لمطابقة نهاية 3 ' تجهيز المواقع التي تم العثور عليها في عينات الأفراد وتقديم تقرير وفرة عبر العينات. تظهر هذه الخطوات في الشكل 5.
وهكذا، مرة واحدة وقد تم تعيين تسلسل عينات، تحليل تسلسل الناتجة عن قراءة الملفات (في شكل فستق) عن طريق خط أنابيب المعالجة المتاحة مباشرة. بعد إضافة معلومات حول العينات لملف التكوين، سيؤدي تنفيذ خط الأنابيب في نوعين رئيسيين من ملفات الإخراج: 1) سرير--الملفات مع جميع 3 'إنهاء تجهيز المواقع المحددة في عينات فردية (مثل " sample1.3pSites.noIP.bed.gz ")، و 2) ملف-سرير مع جميع مجموعات موقع poly(A) (clusters.merged.bed) عبر كافة العينات للدراسة. يتضمن الإخراج أيضا إحداثيات الجينوم لكل القراءات من كل عينة فردية (مثل "sample1. STAR_out/Aligned.sortedByCoord.out.bam ") التي يمكن عرضها في وقت لاحق في مستعرض جينوم مثل إيجف16. عموما يوفر الفحص البصري للتشكيلات الجانبية لقراءة لمحة أولى توزيع مواقع poly(A) في الجينوم والتغييرات التي تحدث على الاضطرابات المحددة التي تم الاضطلاع بها في الدراسة. على سبيل المثال، يظهر في الشكل 6 استجابة جين معين لضرب إلى أسفل البروتين هنرنبك.
كما يتم توفير ملخصات لهذه التوزيعات على نطاق الجينوم (الجدول 1). على وجه التحديد، تحتوي ملفات الإخراج في الدليل "التهم/annotation_overlap" كسور مواقع التي تتداخل مع ميزات المشروح محددة (من الملف الفروق المقدمة كمدخل؛ والمشروح هي: 3 ' UTR، إكسون المحطة الطرفية، إكسون، إنترون، إينتيرجينيك). وأخيراً، لكل عينة، يتم أيضا حفظ النتائج من خطوات المعالجة الفردية (على سبيل المثال، "sample1.summary.tsv"). وهذا يشمل الإعداد: الخام يقرأ في كل عينة، ما يلي: أن يكون هيكل المتوقعة لنهاية 5 '، ما يلي: أن تبقى بعد انهيار كامل بكر التكرارات، يقرأ عالية الجودة وفقا للمعايير المحددة في الخطوة 9، 2، يقرأ تلك الخريطة فريد للجينوم (بعد انهيار تلك التي نتجت عن أخطاء التسلسل، راجع الخطوة 9.5)، يقرأ الخرائط المتعددة (بعد انهيار تلك التي نتجت عن أخطاء التسلسل، راجع الخطوة 9.5)، الخام (غير متفاوت المسافات) 3 'نهاية تجهيز مواقع في كل عينة، الخام 3' نهاية تجهيز المواقع دون فتيلة الداخلية المرشحين المحتملين، فريدة من نوعها 3 ' نهاية تجهيز المواقع من جميع العينات دون المرشحين فتيلة الداخلية، ومجموعة نهائية من مجموعات الموقع poly(A).
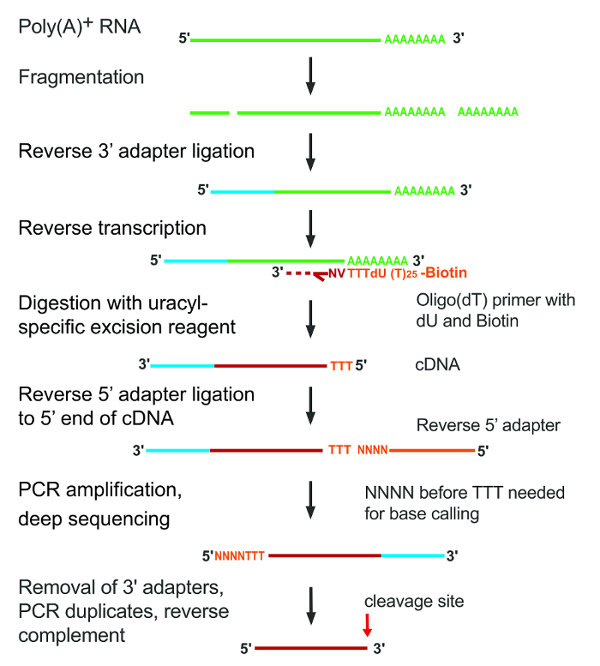
رقم 1: الخطوات الرئيسية للبروتوكول A-seq2- يتم الإشارة إلى الخطوات الفردية على الجانب الأيسر من هذا الرقم. إدراج الأجزاء رنا يصور كخطوط خضراء أن يتحول اللون الأحمر لكدنا بعد النسخ العكسي؛ محولات ملونة بالأزرق الفاتح أو اللون البرتقالي. الرجاء انقر هنا لمشاهدة نسخة أكبر من هذا الرقم-
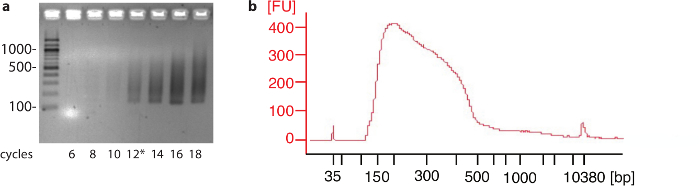
رقم 2: الطيار بكر والمنتج النهائي الشخصية- () مختبرين من رد فعل بكر جمعت في دورات مختلفة وفصلها عن المواد الهلامية [اغروس] 2%. تشير الأرقام إلى اليسار إلى الحجم في النيوكليوتيدات العصابات كل منها في سلم الحمض النووي. في هذه التجربة تم اختيار دورات 12 (*) لتفاعل PCR واسعة النطاق. (ب) مثال على عينة بعد اختيار حجم تشغيل محلل حجم جزء من الكشف عن حجم متوسط من النيوكليوتيدات حوالي 280. تشير الأرقام إلى اليسار [فو] إلى كثافة إشارة النسبي. الرجاء انقر هنا لمشاهدة نسخة أكبر من هذا الرقم-
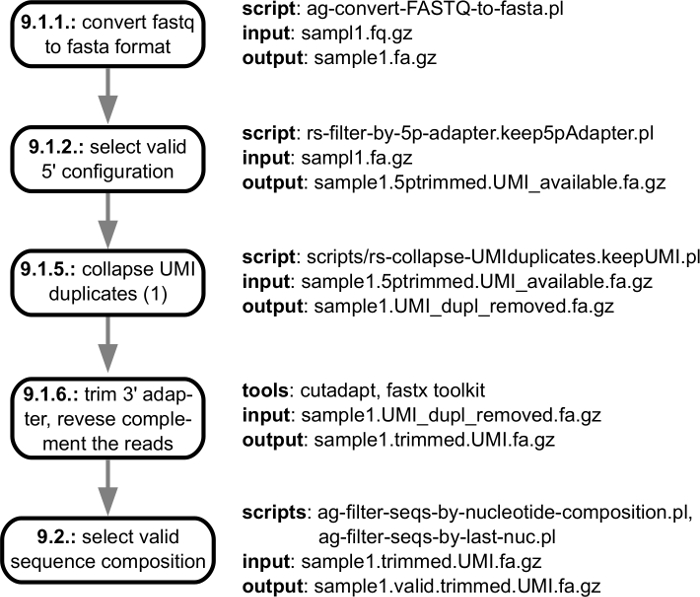
الشكل 3: مخطط لمعالجة ما قبل لتسلسل ما يلي:- تتم معالجة الملفات فستق مع القراءات التي يتم إنشاؤها بواسطة البرامج المرتبطة الصك تسلسل تحديد القراءات عالية الجودة التي سوف يتم تعيينها إلى الجينوم المقابلة. ويوضح الشكل مواصفات المدخلات والمخرجات من الخطوات الفردية في خط الأنابيب، مع وصلات إلى الخطوات الفردية للبروتوكول هو موضح في القسم "معالجة البيانات". الرجاء انقر هنا لمشاهدة نسخة أكبر من هذا الرقم-
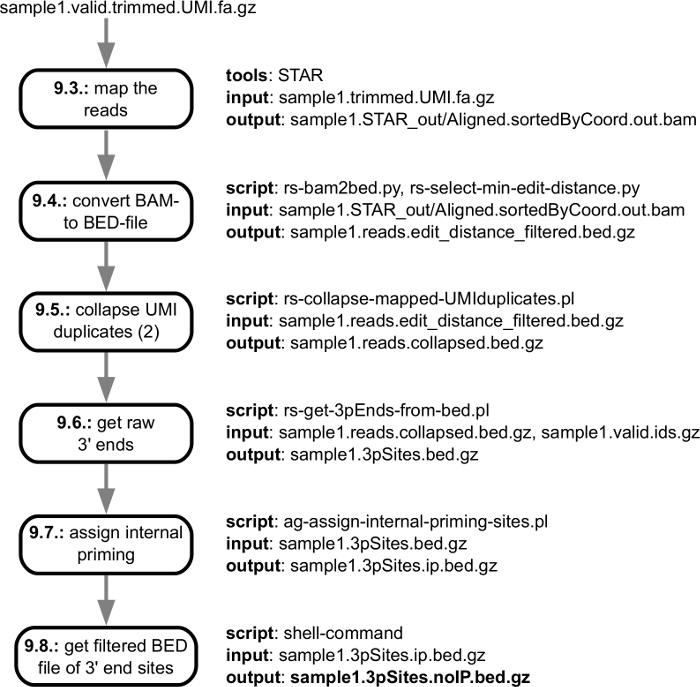
الشكل 4: قراءة الخطوط العريضة لتسلسل المعالجة، من الخطوة لرسم خرائط الجينوم لتوليد الفردية 3 ' نهاية تجهيز المواقع. ويوضح الشكل مواصفات المدخلات والمخرجات من الخطوات الفردية في خط الأنابيب، مع وصلات إلى iنديفيدوال خطوات للبروتوكول هو موضح في القسم "معالجة البيانات". تم وضع علامة ملف الإخراج الرئيسية التي يتم تسليمها إلى المستخدم بخط غامق. الرجاء انقر هنا لمشاهدة نسخة أكبر من هذا الرقم-
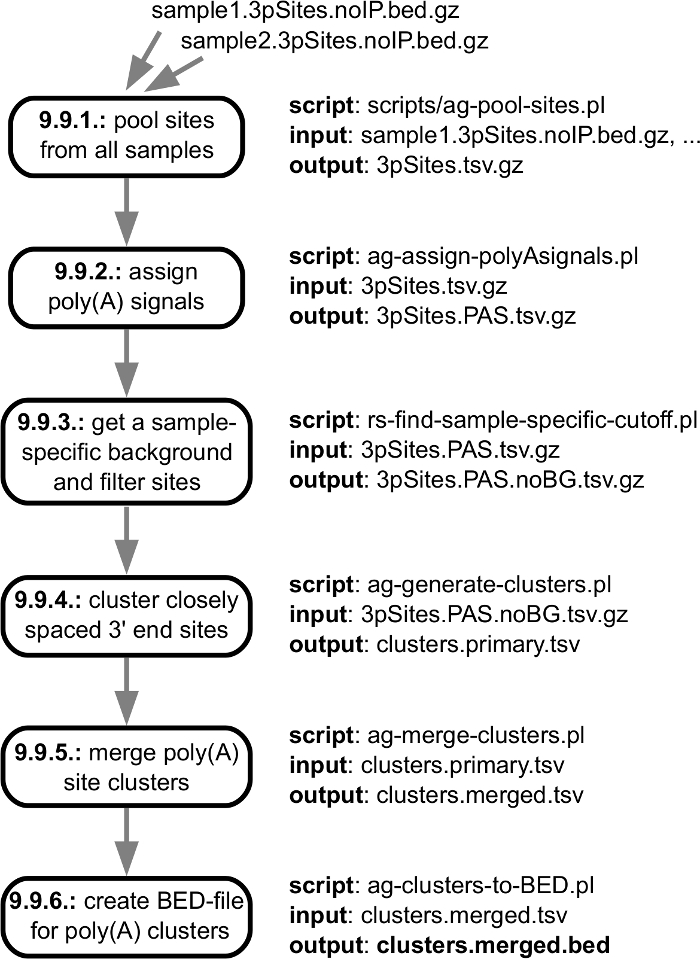
الرقم 5: الخطوط العريضة للخطوات التي يتم اتخاذها لإنشاء مجموعات من التنظيم المشترك 3 ' نهاية تسلسل المواقع. ويوضح الشكل مواصفات المدخلات والمخرجات من الخطوات الفردية في خط الأنابيب، مع وصلات إلى الخطوات الفردية للبروتوكول هو موضح في القسم "معالجة البيانات". تم وضع علامة ملف الإخراج الرئيسي بخط غامق. الرجاء انقر هنا لمشاهدة نسخة أكبر من هذا الرقم-
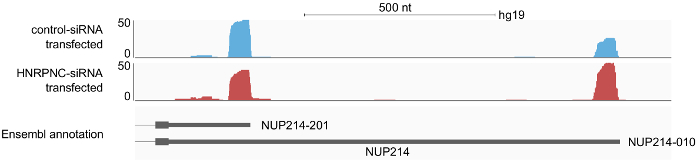
رقم 6: نتائج المثال ملف من 3 ' إنهاء تجهيز ما يلي على طول إكسون المحطة الطرفية من الجينات NUP214، أظهرت في مستعرض الجينوم 16 إيجف- تم إعداد ما يلي: أ-seq2 من عينتين من الخلايا HEK 293، تعامل مع تحكم-siRNA أو siRNA هنرنبك. ما يلي: أن توثيق المواقع poly(A) التي كانت مشروحة بأنابيب تحليل تم حفظها في تنسيق سوق بكارا للأسلحة التي تم استخدامها كمدخلات لمستعرض الجينوم إيجف. 3 'نهايات قمم قراءة خريطة مرناً 3' الغايات التي هي مشروحة في انسيمبل. ملفات التعريف تشير إلى زيادة في استخدام isoform UTR طويلة 3 ' عند هنرنبك تدق لأسفل. الرجاء انقر هنا لمشاهدة نسخة أكبر من هذا الرقم-
| سي-مراقبة النسخ المتماثل 1 | سي-مراقبة النسخ المتماثل 2 | |
| معرف: 29765 | معرف: 32682 | |
| عدد الخام ما يلي: | 44210258 | 68570640 |
| عدد القراءات صالحة بعد التشذيب وتصفية | 14024538 | 21211793 |
| عدد فريد تعيين ما يلي: | 6953674 | 13946436 |
| يقرأ عدد من رسم خرائط لمواقع متعددة | 2040646 | 2925839 |
| عدد فردي 3 ' نهاية تجهيز المواقع | 1107493 | 1710353 |
الجدول 1: إخراج المثال أنابيب التحليل. ملخصات القراءات التي تم الحصول عليها في الخطوات الفردية.
Discussion
العديد من الموارد الأساسية والعوامل المساعدة التي تشارك في تجهيز مرناً قبل 3 ' نهاية تتجلى في المناظر الطبيعية معقدة في المقابل بوليادينيليشن. بالإضافة إلى ذلك، بوليادينيليشن أيضا استجابة للتغيرات في العمليات الأخرى مثل النسخ والربط. 3 'نهاية الانقسام في مرناس قبل هي عادة تحديد مواقع استناداً إلى ذيول poly(A) المميزة التي يتم إضافتها إلى المنتجات انشقاق 5'. استخدام معظم وسائل الإشعال oligo(dT) الأطوال المتغيرة التي تسمح التحويل المحددة من بولي (أ)-تتضمن مرناس إلى كدناس في رد فعل نسخ عكسي. مشكلة شائعة لهذا النهج فتيلة الداخلية إلى تسلسل A الغنية نتج عنه انشقاق artifactual المواقع. وقد اقترحت اثنين من الأساليب التي تهدف إلى التحايل على هذه القطع الأثرية في مرحلة إعداد عينة. في أسلوب seq ف 3- 1، على وجه التحديد هي متصلة محولات لينتهي ذيول poly(A) مع مساعدة من اليغو جبيرة تليها الجزئي T1 رناسي الهضم والنسخ العكسي مع TTP في رد الفعل ديوكسينوكليوتيدي فقط. ثم يتم هضمها هيتيرودوبليكسيس poly(A)-poly(dT) الناتج مع "ح رناسي" والأجزاء رنا المتبقية هي معزولة ومتصلة لمحولات ومتسلسلة. طريقة بسيطة وأنيقة، ف 2-seq، يستخدم تسلسل مخصص التمهيدي تخطي وأبلغت تمتد oligo(dT) المتبقية في رد فعل تسلسل المؤلف نفسه 2. في أسلوب ذات صلة، 3 ' يقرأ 3، التمهيدي طويلاً على نحو غير عادي من 5 لنا و 45 واتس، التي تتضمن أيضا بيوتين هو تعتيق للجيش الملكي النيبالي مجزأة، تليها يغسل صارمة لتحديد لجزيئات الحمض النووي الريبي مع ذيول poly(A) النيوكليوتيدات ما يزيد على 50. على الرغم من أن يقرأ 3 ' تواتر فتيلة الداخلية يخفض بشكل كبير، فإنه لا تماما القضاء عليه 3. كما اقترحت بروتوكولات لتسلسل الحمض النووي الريبي مباشرة، ولكن القراءات الناتجة غير قصيرة ونسبة عالية من الخطأ وهذا النهج لم يكن كذلك نمواً 18،،من1920. بوليا-Seq والبروتوكولات Seq كوانت تجارياً ضم فتيلة oligo(dT) تقوم بخطوة فتيلة عشوائي لكدنا الثانية حبلا توليف 20. استخدام تبديل قالب رد فعل النسخ العكسي مع المنتسخة العكسية فيروس اللوكيميا مولوني موريني (مملف) يؤدي إلى توليد كدناس مع linkers في خطوة واحدة، ومما يمكن أن تظهر لا محول dimers Seq نظام تقييم الأداء وأساليب ساباس 21 , 22.
الأسلوب A-seq2 قدم هنا تبرز في استفادتها النوكليوتيدات كليفابل (dU) ضمن كتاب oligo(dT) بيوتينيلاتيد. يجمع هذا التعديل الأداة المساعدة لإثراء oligo(dT) المهجنة، بوليادينيلاتيد الأهداف مع إزالة معظم تسلسل25 اليغو (dT) من أجزاء معزولة قبل أن يتم إعداد المكتبات والحفاظ على الملخص الثلاثة، التي وتشير إلى وجود الذيل poly(A) السابقة. وفي المقابل، ترك الأساليب التي تستخدم "رناسي ح" لإزالة poly(A) من جزيئات الحمض النووي الريبي عشوائياً عدة كما. منذ في A-seq2، يتم تسلسل من 3 ' نهاية خيوط معنى المضادة، مواقع الانقسام ويتوقع أن تكون موجودة بعد عزر نننتت في بداية ما يلي تسلسل الخام. تيتراميرس معشاة ذات شواهد تخدم ليس فقط للسماح لقاعدة الدعوة بل أيضا في القضاء على بكر التضخيم التحف. كما يمكن أن تستوعب UMIs أطول. إمكانية فتيلة الداخلية لا تزال في seq2 أ ويتم معالجتها حسابياً، أولاً بتجاهل 3 'ينتهي مع تسلسل المتلقين للمعلومات المرمزة جينوميكالي، الغنية بألف ومن ثم طريق تجاهل 3' نهاية الكتل التي يمكن أن تفسر فتيلة الداخلية في إشارة poly(A) الغنية بحد ذاتها. تحليل حديث للمواقع poly(A) الاستدلال فريد من خلال عدد كبير من البروتوكولات يشير إلى أن المواقع التي فريدة من نوعها إلى A-seq2 بتوزيع النوكليوتيدات المتوقعة وموقع داخل الجينات، مماثلة لغيرها 3 ' في نهاية تسلسل البروتوكولات.
هو خطوة حاسمة في seq2 بتحديد بوليادينيلاتيد الجيش الملكي النيبالي وإزالة الريباسي الكشف والكشف الصغيرة المختلفة. يتم ذلك بسهولة بمجموعة مرناً وعزل مع اليغو (dT)25 المغناطيسي الخرز. من حيث المبدأ، يعطي مجموع الجيش الملكي النيبالي معزولة مع الفينول الذي يحتوي على الحلول أيضا الجيش الملكي النيبالي التي يمكن أن تخضع لاختيار المزيد من طقم مرناً والعزل أو اليغو (dT) [اغروس] ذات جودة عالية. خطوة التي يمكن أن تختلف في seq2 أ هو العلاج مع التحلل القلوية التي يمكن اختصارها أو الموسعة للحصول على الأجزاء رنا ذات أحجام مختلفة. الحرجة أيضا إضافة 3 'داتب إلى 3' نهايات الأجزاء رنا بوليميراز poly(A) بكفاءة. في البروتوكول هو موضح هنا، يتم تطبيق هذا العلاج على كافة الأجزاء رنا، لتجنب كونكاتيميريزيشن أثناء عملية التفاعل عملية ربط. وأخيراً، نلاحظ أنه على الرغم من أن الجيش الملكي النيبالي ليجاسى 1 يستخدم عادة ليجاسى الحمض النووي الريبي، فإنه أيضا ليجاتيس الحمض النووي الذين تقطعت بهم السبل كفاءة واحدة، كما فعلنا هنا سد محول إلى 5 ' نهاية الجزيئات كدنا.
وهكذا، A-seq2 فعالة وسهلة لتنفيذ البروتوكول المتعلق بتحديد مرناً قبل 3 ' نهاية تجهيز المواقع. ويمكن أن تشمل التطورات في المستقبل كذلك تقليل تعقيد البروتوكول وكمية المواد المطلوبة. تمكين مجموعة مرتبطة من أدوات تحليل البيانات الحسابية كذلك تجهيز 3 ' نهاية تسلسل ما يلي حصل مع طائفة واسعة من بروتوكولات متجانسة.
Disclosures
الكتاب ليس لها علاقة بالكشف عن.
Acknowledgements
يشكر المؤلفون السيدة بياتريس ديميترياديس للحصول على مساعدة خلية ثقافة. هذا العمل كان تدعمه "مؤسسة العلوم الوطنية السويسرية" منح #31003A_170216 و 51NF40_141735 (الجيش الملكي النيبالي نككر & المرض).
Materials
| Name | Company | Catalog Number | Comments |
| Materials | |||
| Agarose, ultra pure | Invitrogen | 16500-500 | |
| 2100 Bioanalyzer | Agilent | G2940CA | |
| Cordycepin triphosphate (3’ dATP) | SIGMA | C9137 | |
| DNA low bind vials, 1.5 ml | Eppendorf | 22431021 | |
| Dulbecco’s Phosphate Buffered Saline | SIGMA | D8637 | |
| Dynabeads mRNA-DIRECT Kit | Ambion | AM61012 | |
| GR-Green dye | Excellgen | EG-1071 | use 1:10,000 dillution |
| HiSeq 2500 or NextSeq 500 next generation sequencers | Illumina | inquire with supplier | |
| KAPA HiFi Hotstart DNA polymerase mix | KAPA/Roche | KK2602 | |
| Nuclease free water | Ambion | AM9937 | |
| Poly(A) polymerase, yeast | Thermo Fisher Scientific | 74225Z25KU | |
| Poly(A) polymerase, E.coli | New England Biolabs | M0276L | |
| Polynucleotide kinase | Thermo Fisher Scientific | EK0032 | |
| QIAEX II Gel Extraction Kit | Qiagen | 20021 | |
| QIAquick PCR Purification Kit | Qiagen | 28104 | |
| QIAquick Gel Extraction Kit | Qiagen | 28704 | |
| RNA ligase 1, high concentration | New England Biolabs | M0437M | includes PEG-8000 |
| RNeasy MinElute RNA Cleanup kit | Qiagen | 74204 | |
| RNase H | New England Biolabs | M0279 | |
| RNasin Plus, ribonuclease inhibitor | Promega | N2618 | |
| Superscript IV reverse transcriptase | Thermo Fisher Scientiific | 18090050 | |
| Turbo DNase | Ambion | AM2238 | |
| USER enzyme mix | New England Biolabs | M5505 | |
| Dyna-Mag-2 magnetic rack | Thermo Fisher Scientific | 12321D | |
| Thermomixer C | Eppendorf | 5382000015 | Heated mixer with heated lid |
| MicroSpin columns | GE-Healthcare | 27-5325-01 | |
| Name | Company | Catalog Number | Comments |
| Buffers | |||
| Alkaline hydrolysis buffer, 1.5 x | Mix 1 part 0.1 M Na2CO3 and 9 parts 0.1 M NaHCO3. Add EDTA to 1 mM. Adjust pH to 9.2. Store aliquots at -20 °C. | ||
| 5x poly(A) polymerase buffer | Thermo Fisher Scientiific | 100 mM Tris-HCl, pH 7.0, 3 mM MnCl2, 0.1 mM EDTA, 1 mM DTT, 0.5 mg/ml acetylated BSA, 50% glycerol | |
| Biotin binding buffer | 20 mM TrisCl pH 7.5, 2 M NaCl, 0.1% NP40 | ||
| TEN buffer | 10 mM TrisCl, pH 7.5, 1 mM EDTA, 0.02% NP40 | ||
| Name | Company | Catalog Number | Sequence |
| Oligonucleotides according to Illumina TruSeq Small RNA Sample Prep Kits, for GA-IIx and Hiseq2000/2500 sequencers | Microsynth | ||
| revRA3 (RNA) | Microsynth | 5’ amino CCUUGGCACCCGAGAAUUCCA 3’ | |
| revDA5 | Microsynth | 5’ amino GTTCAGAGTTCTACAGTCCGAC GATCNNNN-3’ | |
| Bio-dU-dT25, RT primer | Microsynth | 5' Biotin-TTTTTTTTTTTTTTTTTTTTTTTTTT-dU-TTTVN 3' (V = G, A or C) | |
| PCR primer forward, RP1 | Microsynth | 5' AATGATACGGCGACCACCGAGA TCTACACGTTCAGAGTTCTACAG TCCGA 3' | |
| PCR primer reverse, RPI1, barcode in bold | Microsynth | 5' CAAGCAGAAGACGGCATACGAG ATCGTGATGTGACTGGAGTTCCT TGGCACCCGAGAATTCCA 3' | |
| Name | Company | Catalog Number | Comments |
| Oligonucleotides according to Illumina TruSeq HT-Small RNA Sample Prep Kits, for HiSeq2000/2500 and NextSeq500 sequencers | |||
| HT-rev3A (DNA/RNA) | Microsynth | 5'-amino-GTGACTGGAGTTCAGACGTGTG CTCTTCCrGrAUrC-3' | |
| HT-rev5A | Microsynth | 5' amino-ACACTCTTTCCCTACACGACGCT CTTCCGATCTNNNN 3' | |
| Bio-dU-dT25, RT primer | Microsynth | 5' Biotin-TTTTTTTTTTTTTTTTTTTTTTTTTT-dU-TTTVN 3' | |
| PCR primers forward (D501-506) | Microsynth or Illumina | 5'-AATGATACGGCGACCACCGAGAT CTACAC[i5]ACACTCTTTCCCTACA CGACGCTCTTCCGATCT -3' | |
| PCR primers reverse (D701-D712) | Microsynth or Illumina | 5'-CAAGCAGAAGACGGCATACGAG A[i7]GTGACTGGAGTTCAGACGTG TGCTCTTCCGATC-3' | |
| Documentation for Illumina multiplexing: | Illumina | https://support.illumina.com/content/dam/illumina-support/documents/documentation/chemistry_documentation/experiment-design/illumina-adapter-sequences_1000000002694-01.pdf |
References
- Jan, C. H., Friedman, R. C., Ruby, J. G., Bartel, D. P. Formation, regulation and evolution of Caenorhabditis elegans 3'UTRs. Nature. 469 (7328), 97-101 (2011).
- Spies, N., Burge, C. B., Bartel, D. P. 3' UTR-isoform choice has limited influence on the stability and translational efficiency of most mRNAs in mouse fibroblasts. Genome Res. 23 (12), 2078-2090 (2013).
- Hoque, M., Ji, Z., et al. Analysis of alternative cleavage and polyadenylation by 3' region extraction and deep sequencing. Nat. methods. 10 (2), 133-139 (2013).
- Martin, G., Gruber, A. R., Keller, W., Zavolan, M. Genome-wide analysis of pre-mRNA 3’ end processing reveals a decisive role of human cleavage factor I in the regulation of 3' UTR length. Cell Rep. 1 (6), 753-763 (2012).
- Gruber, A. R., Martin, G., et al. Global 3' UTR shortening has a limited effect on protein abundance in proliferating T cells. Nat. Commun. 5, 5465 (2014).
- Kivioja, T., Vähärautio, A., et al. Counting absolute numbers of molecules using unique molecular identifiers. Nat. methods. 9 (1), 72-74 (2011).
- Gruber, A. J., Schmidt, R., et al. A comprehensive analysis of 3' end sequencing data sets reveals novel polyadenylation signals and the repressive role of heterogeneous ribonucleoprotein C on cleavage and polyadenylation. Genome Res. 26 (8), 1145-1159 (2016).
- Lingner, J., Keller, W. 3'-end labeling of RNA with recombinant yeast poly(A) polymerase. Nucleic Acids Res. 21 (12), 2917-2920 (1993).
- Quail, M. A., Kozarewa, I., et al. A large genome center's improvements to the Illumina sequencing system. Nat. methods. 5 (12), 1005-1010 (2008).
- Rahmann, S. Snakemake--a scalable bioinformatics workflow engine. Bioinformatics. 28 (19), 2520-2522 (2012).
- . Anaconda Software Distribution Available from: https://continuum.io (2016)
- Dobin, A., Davis, C. A., et al. STAR: ultrafast universal RNA-seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- Martin, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet.journal. 17 (1), 10-12 (2011).
- Li, H., Handsaker, B., et al. The Sequence Alignment/Map format and SAMtools. Bioinformatics. 25 (16), 2078-2079 (2009).
- Robinson, J. T., Thorvaldsdóttir, H., et al. Integrative genomics viewer. Nat. Biotechnol. 29 (1), 24-26 (2011).
- Quinlan, A. R., Hall, I. M. BEDTools: a flexible suite of utilities for comparing genomic features. Bioinformatics. 26 (6), 841-842 (2010).
- Ozsolak, F., Platt, A. R., et al. Direct RNA sequencing. Nature. 461 (7265), 814-818 (2009).
- Yao, C., Biesinger, J., et al. Transcriptome-wide analyses of CstF64-RNA interactions in global regulation of mRNA alternative polyadenylation. Proc. Natl. Acad. Sci. U. S. A. 109 (46), 18773-18778 (2012).
- Lin, Y., Li, Z., et al. An in-depth map of polyadenylation sites in cancer. Nucleic Acids Res. 40 (17), 8460-8471 (2012).
- Shepard, P. J., Choi, E. -. A., Lu, J., Flanagan, L. A., Hertel, K. J., Shi, Y. Complex and dynamic landscape of RNA polyadenylation revealed by PAS-Seq. RNA. 17 (4), 761-772 (2011).
- Fu, Y., Sun, Y., et al. Differential genome-wide profiling of tandem 3' UTRs among human breast cancer and normal cells by high-throughput sequencing. Genome Res. 21 (5), 741-747 (2011).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved