Method Article
3' final de preparação da biblioteca com A-seq2 de sequenciamento
Neste Artigo
Resumo
Este protocolo descreve um método para mapeamento pre-mRNA' a extremidade 3 sites de processamento.
Resumo
Estudos na última década têm revelado uma variedade complexa e dinâmica de clivagem pre-mRNA e reações de poliadenilação. mRNAs com tempo de 3' não regiões (UTRs) são gerados em células diferenciadas, Considerando que pilhas proliferating preferencialmente expressam transcrições com curta 3' UTRs. Descrevemos o protocolo A-seq, agora em sua segunda versão, que foi desenvolvido para mapear os sítios de poliadenilação todo o genoma e estudar o Regulamento de pre-mRNA 3' processamento final. Também este protocolo atual aproveita o polyadenylate (poly(A)) caudas que são adicionadas durante a biogênese dos mRNAs mamíferos mais enriquecer para mRNAs totalmente transformados. Um adaptador de DNA com deoxyuracil em sua quarta posição permite o processamento preciso de mRNA 3' final fragmentos para sequenciamento. Não incluindo a cultura de células e a ligadura durante a noite, o protocolo exige cerca de 8 h tempo hands-on. Junto com ele, é fornecido um pacote de software easy-to-use para a análise dos dados de sequenciamento derivada. A-seq2 e o software de análise associado fornecem uma solução eficiente e confiável para o mapeamento de pre-mRNA 3' termina em uma ampla gama de condições, a partir de 106 ou menos células.
Introdução
A captura e sequenciamento de mRNA 3' extremidades permite o estudo do processamento do mRNA e a quantificação da expressão gênica. Devido a suas caudas poli, mRNAs eucarióticos podem ser eficientemente purificados de lisado celular total com grânulo-imobilizado oligo-cromatografia (moléculas oligo(dT)), que também pode aprontar a síntese do cDNA. No entanto, esta abordagem tem dois inconvenientes. Primeiro, trechos de A's que são interno para transcrições também podem prime síntese do cDNA, resultando em sites de poli espúrias. Poli (a) homogênea, segundo trechos representam desafios específicos para sequenciamento, além de não ser informativo para identificação de transcrição. Várias abordagens têm sido propostas para contornar essas limitações, tais como a transcrição reversa através de poli caudas seguidas por digestão RNase H (3P-seq. 1), uso de um primer de sequenciamento personalizado terminando em 20 Ts (2P-seq. 2), pré-selecção de Fragmentos de RNA com caudas poli (a) de mais de 50 nucleótidos com um primer de45 CU5T seguido por digestão RNase H (3' leituras 3) e o uso de um primer de oligo-dT que contém o adaptador 3' em um grampo de cabelo (A-seq. 4).
O recentemente desenvolvido A-seq2 método 5 destina-se a ignorar o sequenciamento através de poli (a) e ao mesmo tempo minimizar a proporção de dímeros são gerados pelo auto ligadura dos adaptadores, ocorrendo especialmente quando a concentração molar de adaptadores supera a concentração de inserção. Este problema pode ser eliminado quando ambos os adaptadores são ligados para o mesmo tipo de polinucleotídicas termina como em A-seq2, onde os 3' adaptadores são ligados à extremidade 5' do RNA fragmentos e os adaptadores de 5' para 5' extremidades dos cDNAs após a transcrição reversa. O método é mais conveniente do que a nossa anteriormente proposto A-seq - no qual sequenciamento foi no 5'-para-3' direção que exige precisamente controlado RNA fragmentação-, mantendo uma alta precisão de identificação de site do poli (a). Cerca de 80% das leituras sequenciais em amostras típicas mapear com exclusividade para o genoma e levar a identificação de mais de 20.000 clusters poli local, mais de 70% das quais se sobrepõem com anotada 3' UTRs.
Em breve, o protocolo A-seq2 começa com fragmentação de mRNA e ligadura dos adaptadores reverso-complemento 3' para as extremidades 5' de fragmentos de RNA. Poli (A)-contendo RNAs são então reversos transcritas com um primer de longa (descolamento) oligo 25 nucleotídeos (nt) que contém um nucleotídeo âncora na extremidade 3', um dU na posição 4 e uma biotina na extremidade 5', que permite a ligação do cDNA de grânulos magnético estreptavidina. A maioria da primeira demão, incluindo a biotina, é removido o cDNA por clivagem em dU por mistura de enzima do usuário, contendo Uracil DNA glycosylase (UDG) e o DNA glycosylase-liase Endonuclease VIII. Esta reação deixa extremidades intactas para a ligadura de um adaptador de 5' e 3 Ts esquerda após clivagem permanecem marcar o local da cauda poli (a). Porque tanto 5' e 3' adaptadores são anexados pela ligadura para destinatário 5' extremidades, dímeros nenhum adaptador são gerados. Quatro nucleótidos aleatório-mers introduzidos no início de leituras permite a resolução de cluster em instrumentos de sequenciamento de estado-da-arte e também pode servir como identificador exclusivo molecular (UMI) para a detecção e remoção de artefatos de amplificação do PCR. O tamanho do UMI pode ser aumentado como feito em outros estudos 6. O protocolo gera leituras que são inverter complementar ao mRNA 3' extremidades, tudo iniciando com um tetrâmero randomizado, seguido por 3 Ts. processamento de leituras que têm os 3 Ts diagnósticos no final inicia sua 5' com a correção de artefatos de amplificação do PCR por explorando os UMIs, remoção de sequências de 3' adaptador e reverter a complementação. Leituras que podem ter se originado de escorva de oligo (descolamento) em sites internos A rica também são identificadas computacionalmente e descartadas. Os sites espúrios geralmente faltam um dos 18 bem caracterizadas e conservada poli sinais que devem ser localizado ~ 21 nucleotídeos upstream da clivagem aparente local 7.
O protocolo exige cerca de 8 h tempo hands-on, sem contar com a cultura de células e a ligadura durante a noite. O associado Leia análise software permite uma identificação de site poli altamente precisos. Do site da poli aglomerados criados com baseados em 4 amostras mais destacadas nesta sobreposição de 84% do manuscrito (duas repetições biológicos de controle siRNA e si-HNRNPC-tratada células) com um gene anotado e destes, 75% de sobreposição com um 3' UTR e 86% com qualquer um 3' UTR ou um terminal exon. O coeficiente de correlação de Pearson de expressão de 3' extremidades nas amostras de replicar é 0,92, e valores de acima de 0,9 normalmente são obtidos com o método. Assim, A-seq2 é um método conveniente que dá resultados muito reprodutíveis.
Protocolo
1. crescimento celular e mRNA isolamento
- desenvolvem-se células de acordo com seu projeto experimental em placas boas 6 ~ 1 x 10 6 células por poço na confluência de 80%.
- Remover o meio de crescimento e lave as células uma vez com tampão fosfato salino. Lyse diretamente as células da placa, adicionando 1 mL de tampão de lise do kit de isolamento do mRNA. Transferir o viscoso lisado em um tubo de 15 mL de plástico com uma ponta de pipeta de 1 mL. Use uma espátula para retirar completamente o material de células da superfície da placa.
- De cisalhamento o lisado contendo DNA viscoso com uma seringa de 1 mL, anexada a uma agulha hipodérmica de 23 G por vários vigorosa e movimentos do êmbolo, descendo até o lisado não é mais viscoso. Aponte a agulha da seringa para o centro da parte inferior para evitar ejetando o lisado fora do tubo.
- Transferir o lisado em um tubo de 1,5 mL, utilizando a seringa. Rotação de 5 min a 20.000 x g e 4 ° C para remover os detritos. Use ampolas de 1,5 mL de baixa ligação DNA em todo o protocolo.
- Enquanto o centrifugador é executado, lavar 300 µ l de ressuspensão oligo (dT) 25 esferas magnéticas em um rack magnético com 500 µ l de tampão de Lise. Misture os tubos de 2 - 3 vezes na prateleira. Remova o buffer depois que a solução é clara. Recolher o sobrenadante claro da etapa 1.4 e adicionar as contas. Resuspenda e coloque os tubos em uma roda giratória de 10 min.
- Coloque os tubos em um rack magnético. Remova o líquido claro depois de 2 min. Adicionar 0,8 mL tampão A partir do kit de isolamento do mRNA. Rode o tubo por graus de 180° na prateleira, 2 - 3 vezes. Repita essa etapa de lavagem mais uma vez com buffer r.
- Lave os grânulos 2 vezes com 0,8 mL de tampão B conforme descrito na etapa 1.6.
- Para Eluir o mRNA limite de grânulos, adicionar 33 µ l H 2 O e ressuspender os grânulos. Aqueça a 75 ° C por 5 min num bloco aquecida. Imediatamente a girar os tubos para 1 s e lugá-los sobre o rack magnético. Transferi o sobrenadante para um tubo novo. As amostras podem ser armazenadas a-80 ° C até utilização posterior.
- Buffer de hidrólise alcalina adicionar 66 µ l para o 33 µ l mRNA (passo 1.8), misture e aqueça por exatamente 5 min a 95 ° C, em um bloco de aquecimento. Relaxa imediatamente os tubos em gelo
- Isolar o RNA com um kit de limpeza de RNA.
Nota: Confirmar o volume; deve ser 100 µ l. Etanol de- buffer de RLT adicionar 350 µ l do kit e 250 µ l. Carga na coluna e rotação por 30 s a 8.000 x g, à temperatura ambiente (RT). Lavar com buffer RPE 500 µ l do kit. Lavar com 500 µ l 80% de etanol. Girar para 5min a 20.000 x g para secar a coluna. Adicionar coluna 36 µ l H 2 O e girar para 1 min no x 20.000 g. descartar a coluna e salvar o eluato.
2. 5 ' acabar a fosforilação e tratamento de DNase
- Adicionar 5 µ l polinucleotídicas quinase reserva, 5 µ l ATP de 10 mM, 1 µ l ribonuclease inibidor, 1 µ l DNase e quinase de polinucleotido 2 µ l de amostras e incubar a 37 ° C para 30 min. opcionalmente preparar misturas de reação mestre em todo o protocolo misturando 1,1 volumes x n (n = número de amostras) de cada componente.
- Mudar o tampão e remover ATP em uma coluna de rotação para evitar a adição de poli na próxima etapa.
- Prespin girar-colunas a 735 x g por 1 min. transferir as colunas para novos frascos de 1,5 mL e carregar as reações da quinase para as colunas. Girar as colunas 2 min em 735 g. x descartar as colunas e coloque os tubos com reações recolhidas no gelo ou armazenam a -80 ° C.
3. Bloqueio 3 ' termina com Cordycepin trifosfato
Nota: é essencial para bloquear a 3 ' extremidades dos fragmentos de RNA para evitar sua concatemerization nas reações subsequentes da ligadura. 3 ' extremidades que já não são bloqueadas por um ( fosfato cíclico) depois de hidrólise são tratados pela adição de um 3 ' dATP (cordycepin trifosfato) nucleotídeos de terminator cadeia com a ajuda da polimerase poli. Aqui, o polymerase de poli (a) de fermento (yPAP), que foi expressa e purificado conforme descrito em 8 foi usado numa concentração de 0,5 mg/mL. Levedura ou Escherichia coli PAP ambos têm quase a mesma actividade para além de 3 ' dATP e podem ser adquiridos comercialmente (consulte a tabela de materiais).
Amortecedor da reação polimerase- Adicionar 13,5 µ l 5 x poli concentrado, 2 µ l de 10mm 3 ' dATP, inibidor de RNase 1 µ l e 1 µ l poli o polymerase para a reação da etapa 2.2.1. Mix e spin de 1 s. Incubar a 37 ° C por 30 min. Adicione 32,5 µ l H 2 O para cada reação. Purifica o RNA como na etapa 1.10.1. Eluir o RNA com 14 µ l H 2 O.
4. Ligadura de reverter 3 ' adaptadores para o 5 ' fim de fragmentos de RNA
- Coloque as reações em um concentrador de vácuo por 10 minutos para reduzir o volume de 6 µ l. adicionar 3 µ l 10 x buffer de ligadura de RNA T4, 3 µ l 10mm ATP , 15 µ l PEG-8000, inibidor de RNase 1 µ l, 1 µ l do complemento reversa de 0,1 mM 3 ' adaptador " revRA3 " (consulte a tabela de materiais) e ligase de alta concentração de RNA 1 µ l 1, misture.
- Incubar as reações a 24 ° C, durante 16 h em um misturador aquecido com mistura intermitente a 1.000 rpm. Adicionar 70 µ l H 2 O para cada reação e misture. Purifica o RNA como na etapa 1.10.1. Eluir o RNA com 14 µ l H 2 o. as amostras podem ser armazenadas a-80 ° C neste momento.
5. Reverter a transcrição (RT)
- lugar os eluídos em um vácuo concentrador por 3 min para reduzir o volume de 11 µ l. reacções de transferência a 200 µ l do PCR tubos. Adicionar 1 µ l 0,05 mM RT da primeira demão " Bio-dU-dT25 ". Aqueça por 5 min a 70 ° C em um cycler PCR e deixe em RT por 5 min.
- Adicionar 1 µ l 10mm dNTPs, 4 µ l 5x buffer de transcriptase reversa, 1 µ l 0,1 M DTT, 1 inibidor de RNase µ l e transcriptase reversa de 1 µ l. Misture e aqueça as reações por 10 min a 55 ° C e a 10 min a 80 ° C em um cycler PCR. Manter em gelo ou a-80 ° C para o armazenamento mais.
6. Digestão com mistura de enzima uracila DNA Glycosylase
- Pipetar 100 µ l Streptavidin-grânulos, dentro de um frasco de 1,5 mL, resuspenda em buffer de vinculação 800 µ l biotina e coloc em uma cremalheira magnética. Inverta os tubos 2 - 3 vezes. Remova o buffer quando clara. Repita a etapa de lavagem. Resuspend os grânulos no buffer de vinculação 200 µ l biotina.
- Adicionar a reação de transcrição reversa para a solução de grânulos e incubar 20 min a 4 ° C em uma roda giratória. Lave os grânulos 2x com ligação biotina buffer como em pisar um rack magnético x 6,1 e 2 com buffer de dez. Resuspend os grânulos no buffer de dez 50 µ l, adicione 2 µ l de DNA uracila glycosylase enzima mistura e incubar 1 h a 37 ° C, em um misturador com mistura intermitente. Buffer de
- Adicionar 50 µ l de H 2 O, 11 µ l de RNase H e 1 µ l RNase H para as reações. Incubar a 37 ° C por 20 min. de tubos de lugar em uma cremalheira magnético e transferir o líquido contendo o cDNA clivado para um novo tubo de
- purificar o cDNA clivado.
- Adicionar 550 µ l de buffer PB do kit de purificação de PCR para as reações de clivagem. Adicione 10 µ l de acetato de sódio de 3 M, pH 5.2 a diminuir o pH. Carregar as reações em colunas de rotação mínima de eluição e girar a 17.000 x g por 1 min.
- Buffer de adicionar 750 µ l PE para colunas e girar a 17.000 x g por 1 min. descartar o escoamento. Gire as colunas a 17.000 x g por 1 min secar. Transferir as colunas para uma ampola de 1,5 mL, adicionar 16 µ l H 2 O e girar a 17.000 x g por 1 min. colocam as reações em um concentrador a vácuo durante 8 min dedicar-se a um volume de 7 µ l.
7. Ligadura dos 5 ' adaptadores para 5 ' extremidades do cDNA
- para o cDNA isolado, adicionar 3 µ l 10 x buffer de RNA T4 ligase 1, 3 µ l 10mm ATP, 15 µ l PEG-8000, 1 µ l 50 µM " revDA5 " oligo e 1 µ l alta concentração do RNA T4 ligase 1. Incube a 24 ° C para 20 h. Adicionar 70 µ l H 2 O para cada reação. As amostras podem ser armazenadas a-20 ° C neste momento.
8. O piloto de PCR, amplificação de bibliotecas e seleção de tamanho
- em uma reação de piloto, determinar o número ideal de PCR ciclos para alcançar a amplificação da biblioteca no âmbito da fase exponencial.
- Pipetar 25 mistura de DNA polimerase µ l, reação de ligadura 20 µ l, 2 µ l H 2 O, 1,5 µ l 10 µM frente PCR primer (RP1) e 1,5 µ l 10 µM reversa PCR índice primário no tubo PCR 200 µ l.
- Executar o reciclador com o seguinte programa: 3 min 95 ° C, seguido de 20 ciclos de 20 s 98 ° C, 20 s 67 ° C e 30 s 72 ° C. coletar 7 µ l partes alíquota após ciclos de 6, 8, 10, 12, 14, 16 e 18, directamente a partir do reciclador. Adicione 1 µ l 10 x carregando buffer (glicerol 50%, 0,05% de xileno cianol). Nota: Por favor, siga as recomendações do fornecedor se usando a multiplexação ao combinar códigos de barras.
- Produtos separados em pequenas vagas em um gel de agarose 2% em 1 tampão de x TBE contendo um 01:10, 00 diluição da tintura verde fluorescente.
- Alíquotas de carga em uma agarose 2% gel e executar o gel em 100 volts durante 15 min. Visualize migração dos produtos PCR em um sistema de documentação do gel.
- Usar o número de ciclos no início da amplificação exponencial na reação de piloto para uma reação de PCR em grande escala com duas vezes os volumes como usado para a piloto reação ( Figura 2).
- Para reações de PCR em grande escala, concentre-se e do desalt as reações primeiro com um kit de purificação de PCR e separar os produtos na vasta slots no gel de agarose 2% em 1 x TBE buffer.
- Cortar fatias de gel contendo 200-350 nt DNA produtos. Derreta o gel no buffer caotrópicas em RT para até 30 min. Extrai o DNA do gel de rodelas com um kit de extração do gel. Não aqueça a 50 ° C para evitar viés na ligação do ADN A rica 9.
- Enviar para sequenciamento.
Nota: Normalmente, 50 ciclos single-leitura (SR50) são suficientes (Veja, por exemplo, https://www.illumina.com/technology/next-generation-sequencing.html).
9. Processamento de dados
Nota: os dados resultantes de sequenciamento (no formato fastq) são processados com o software disponível no repositório do gitlab (https://git.scicore.unibas.ch/zavolan_public/A-seq2-processing). A análise inclui quatro etapas principais: (1) transferindo o repositório git, (2) a instalação de um ambiente virtual, parâmetros específicos (3) configuração no arquivo de configuração e (4) lançando a análise através de ‘ snakemake ’ 10. a análise de toda feita no passo 4 requer apenas um comando. Uma descrição detalhada passo a passo da análise pode ser encontrada no arquivo README no repositório gitlab e uma breve descrição está disponível abaixo. Todas as etapas de processamento individuais são realizadas pela execução de ferramentas à disposição do público, quer a partir de fontes externas ou preparadas em casa. O pipeline computacional depende de um ambiente virtual de python 3 baseado em anaconda 11 com o snakemake pacote disponível 10. Ele é executado em máquinas com sistema operacional Unix-like e foi testado em um ambiente Linux com o sistema operacional CentOS 6.5 instalado e 40 GB de RAM disponível. As dependências do software são controladas automaticamente dentro do ambiente virtual. As seguintes ferramentas de software disponíveis publicamente são necessários e assim instaladas juntamente com o meio ambiente: snakemake (v3.9.1) 10, fastx toolkit (v0.0.14) 12, STAR (v2.5.2a) 13 , cutadapt (v 1.12) 14, samtools (v. 1.3.1) 14 , 15, bedtools (v2.26.0) 16 , 17.
- Pré-processamento de dados de leituras de cDNAs
Nota: A profundidade de sequenciamento pode variar entre as execuções e, dependendo do instrumento, dados de uma amostra podem ser divididos em vários arquivos de sequência. Se este for o caso, concatene os arquivos que correspondem a uma amostra em um único arquivo de entrada que é usado nas etapas a seguir.- Converter o arquivo de fastq para formato fasta.
- Extrato lê com uma estrutura correta (3 thymidines nas posições 5, 6 e 7 da leitura).
Nota: Uma leitura que é preparada corretamente de acordo com o protocolo experimental descrito acima deve ter a estrutura (de 5 a ' final): 4-nucleotide barcode - 3 thymidines - inverter complemento de transcrição 3 ' final. - Armazenar as informações sobre o tetrâmero de partida na linha de descrição da sequência de.
Nota: O tetrâmero serve como um identificador exclusivo molecular (UMI) que facilita a correção de artefatos de amplificação mais tarde na análise. - Remover os sete primeiros nucleotídeos a partir da leitura ' s 5 ' fim.
- Correto para inserir de artefatos de amplificação, mantendo apenas uma cópia do lê com a mesma sequência e UMI.
- Remover a parte do 3 ' corresponde a sequência de adaptador e complemento então inverter a sequência final. Só proceder com leituras que tem um comprimento mínimo (padrão: 15 nt).
Nota: dependendo do comprimento do fragmento de mRNA original e o número de ciclos de sequenciamento, o 3 ' final da leitura pode conter parte do 3 ' adaptador, que é removido nesta etapa.
- Extrair todas as leituras que cumprir os seguintes critérios: máximos 2 desconhecidos nucleotídeos (' N '), máximo 80%, como e o último nucleotídeo da leitura não A. Estas leituras são consideradas de qualidade suficiente para ser usado na análise.
- Mapear as leituras para o genoma com uma ferramenta que manipula leituras emendadas e gera um arquivo de saída no formato BAM.
- Se a estrela é usada, crie um arquivo com o índice do genoma para que as leituras devem ser mapeadas. Para o genoma humano, esta etapa exige 35 GB de memória (RAM).
- Mapear as leituras para o genoma.
Nota: (notas específicas estrela) Soft-clipping é desabilitado para forçar o mapeamento dos 3 ' final de cada ler como este é o nucleotídeo imediatamente a montante do local clivagem.
- Converter o BAM para um arquivo de cama. Se uma leitura de mapas para vários locais, manter somente aqueles com o mais baixo editar distância.
Nota: O número de cópia da leitura mapeado em um local específico é usado como pontuação. Leituras que mapeiam para vários locais são contadas fracionada em cada local com um peso igual a 1/número de locais para que uma leitura de mapas. - Colapso leituras que variam de acordo com um erro provável de sequenciamento. Se duas leituras distintas mapeiam para o mesmo local (posição inicial e final dos mapeamentos são idênticos) e eles compartilham o mesmo UMI, considerá-los como duplicatas PCR e ficar com um só.
- Infer pré-ARNm sempre individual 3 ' acabar com processamento sites.
Nota: Uma leitura individual fornece evidências para um 3 ' acabar quando seus últimos quatro nucleótidos são mapeados para o genoma sem erro. A posição à qual o 3 ' final dos ler mapas é armazenado como clivagem. - Detectar 3 ' acabar com sites que poderiam ter se originado de escorva interna. Definir o local como artefato de escorva interno quando o 10 nt a jusante do site clivagem no genoma satisfazer um dos seguintes critérios: contém mais de seis, como, contém seis anos consecutivos como ou começa com um dos seguintes tetrâmeros: AAAA, AGAA, AAGA AAAG .
- Gerar uma tabela de 3 individuais ' acabar com processamento sites no formato de cama.
- Identificar regulada independentemente clusters de site poli.
Nota: Os passos descritos aqui sigam o procedimento que foi introduzido em uma publicação prévia 5.- Começar por coleta individual 3 ' processamento finais que foram obtidos em todas as amostras do estudo.
- Anotar poli conhecidos sinais 7 na região de 09:00 nucleotídeos em torno de cada individual 3 ' local de processamento final.
- Poli de identificar sites expressados como segue acima o fundo em cada amostra.
- Classificar os sites por sua expressão cru dentro a amostra atual. Percorrer a lista de sites de cima para baixo, associando inferiores sites classificados com um site de classificados mais elevado se eles estão localizados a uma distância predefinida no genoma (padrão: 25 nt - up ou a jusante) do alto escalão site.
Nota: Todos os sites de baixa patente, associados a um site de alto escalão definem um cluster cuja expressão é o número de leituras documentar todos esses sites. - Classificar esses aglomerados por expressão e percorrer a lista de clusters do mais alto de menor expressão, determinando o limiar de expressão c em que a percentagem de clusters com um poli anotado sinal cair abaixo de um limite predefinido ( padrão: 90%).
- Descartar sites de qualquer cluster abaixo do corte.
- Classificar os sites por sua expressão cru dentro a amostra atual. Percorrer a lista de sites de cima para baixo, associando inferiores sites classificados com um site de classificados mais elevado se eles estão localizados a uma distância predefinida no genoma (padrão: 25 nt - up ou a jusante) do alto escalão site.
- Cluster espaçadas 3 ' finais obtidos através de amostras.
Nota: Tipo 3 ' final processamento sites primeiro pelo número de amostras de apoio e, em seguida, pela soma do normalizado ler contagem (lê por milhão (RPM)) através de amostras. Percorrer a lista de cima para baixo, associando inferior do ranking de sites mais bem classificados sites quando sua distância para o local de maior classificação não é maior do que um limite predefinido (padrão: 12 nt). Sempre que qualquer um dos 3 que constituem ' final site sobrepõe-se com um sinal de poli anotado ou tem um sinal de poli (a) diretamente a jusante, o cluster correspondente está marcado para inspeção adicional detectar escorva interna. - Poli de mesclagem site aglomerados.
Nota: Quando um cluster é marcado como um candidato putativo escorva interno, ele é mesclado em um cluster a jusante, se os dois clusters compartilham seus sinais de poli ou mantido, se o site mais a jusante do cluster tem um sinal de poli (a) localizado no mínimo montante da distância (padrão: 15 nt). Finalmente, clusters de espaçadas são mescladas, se: (i) compartilham os mesmos sinais de poli (a), ou (ii) a extensão do cluster resultante não exceda um máximo (padrão: 25 nt). - Armazenar clusters no formato de arquivo de cama com total normalizado lesses a contagem de todos os 3 ' finais em cada cluster como pontuação.
Resultados
Poli (A)-contendo RNA foi isolado de células cultivadas, fragmentadas por hidrólise alcalina e cDNAs foram feitos por reverter a transcrição com primers de oligo (descolamento). O cDNA resultante foi imobilizado em grânulos de estreptavidina, dU foi clivada na reação de excisão específico do uracil, adaptadores estavam ligados a 5' e 3' extremidades do fragmento clivado e as inserções foram sequenciadas. Figura 1 mostra um esquema gráfico do experimento.
Para células HeLa e HEK293, 106 células foram suficientes para identificar sites de poli (a) para a grande maioria dos genes codificantes de proteínas, no final do procedimento. No entanto, para outros tipos de células ou tecidos, pode ser necessário testar a saturação do número de sites identificados poli, como o número de células utilizadas no experimento aumenta. Passo resultados representativos do PCR do piloto e do fragmento de DNA análise da amostra antes de sequenciamento são mostrados na Figura 2.
A Figura 3 mostra as etapas de pré-processamento da análise computacional, a partir do arquivo de fastq obtidos a partir do sequencer e terminando com a qualidade verificada, adaptador-aparado diz que estão pronta a serem mapeados para o genoma. A Figura 4 mostra as etapas de análise que começam com o mapeamento das leituras para o genoma correspondente e o final com o catálogo de mRNA' a extremidade 3 sites que são identificados em uma amostra específica de processamento. Quando múltiplas amostras são analisadas, etapas adicionais são realizadas para coincidir com a extremidade 3' processamento sites que foram encontrados em amostras individuais e relatar sua abundância através de amostras. Estas etapas são mostradas na Figura 5.
Assim, uma vez que as amostras têm sido sequenciadas, a análise da sequência resultante ler arquivos (no formato fastq) por meio do pipeline de processamento disponível é simples. Depois de adicionar as informações sobre as amostras para o arquivo de configuração, a execução do gasoduto irá resultar em dois tipos principais de arquivos de saída: 1) cama-arquivos com todos os 3' acabam o processamento locais identificados em amostras individuais (por exemplo " Sample1.3pSites.noIP.Bed.gz") e 2) um cama-arquivo com clusters de local todos os poli (clusters.merged.bed), através de todas as amostras do estudo. A saída também inclui as coordenadas do genoma para todas as leituras de cada amostra individual (por exemplo, "sample1. STAR_out/aligned.sortedByCoord.out.bam") que mais tarde podem ser visualizados em um navegador de genoma como IGV16. Inspeção visual de FIS a leitura geralmente fornece um primeiro vislumbre da distribuição dos sítios de poli no genoma e as mudanças que ocorrem sobre as perturbações específicas que foram realizadas no estudo. Por exemplo, na Figura 6 é mostrada a resposta de um gene específico para o knock-down da proteína HNRNPC.
Resumos destas distribuições de todo o genoma são também fornecidos (tabela 1). Especificamente, os arquivos de saída no diretório "contagens/annotation_overlap" contêm frações de sites que se sobrepõem com características específicas anotadas (do arquivo gtf fornecido como entrada; anotados são: 3' UTR, terminal exon, exon, intrão, intergênica). Finalmente, para cada amostra, resultados das etapas de processamento individuais também são salvos (por exemplo, "sample1.summary.tsv"). Isso inclui os números de: crus leituras em cada amostra, leituras que têm a estrutura esperada da extremidade 5', leituras que permanecem após o colapso completa duplicatas PCR, alta qualidade lê de acordo com os critérios definidos no passo 9.2, lê o mapa com exclusividade para o genoma (depois de desmaiar aqueles que resultaram dos erros de sequenciamento, consulte a etapa 9.5), mapeamento multi lê (depois de desmaiar aqueles que resultaram dos erros de sequenciamento, consulte a etapa 9.5), sites em cada amostra, cru ' a extremidade 3 sites de processamento de processamento bruto (não-clusterizados) 3' final sem os potenciais candidatos internos de escorva, original 3' terminar processamento sites de todas as amostras sem candidatos de escorva interno e conjunto final de clusters de poli (a) local.
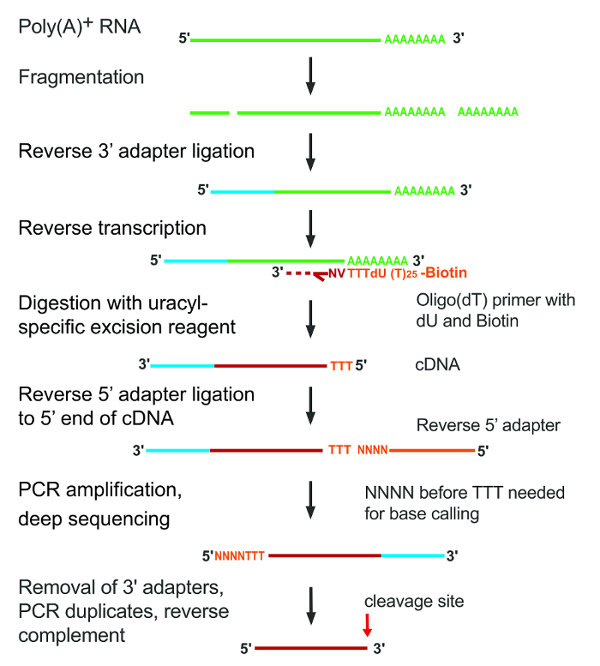
Figura 1: etapas principais do protocolo A-seq2. Etapas individuais são indicadas no lado esquerdo da figura. Inserir fragmentos de RNA são retratados como linhas verdes que acendem uma luz vermelhas para cDNA após a transcrição reversa; adaptadores são coloridos em azul claro ou laranja. Clique aqui para ver uma versão maior desta figura.
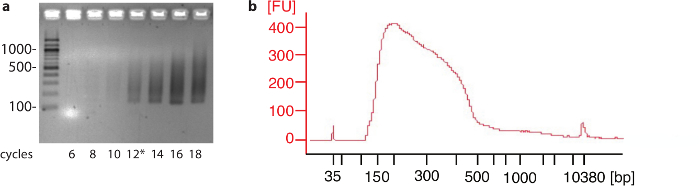
Figura 2: piloto do PCR e perfil do produto final. (um) alíquotas da reação de PCR foram coletadas em ciclos diferentes e separaram em gel de agarose 2%. Os números à esquerda indicam tamanho em nucleotídeos das respectivas bandas na escada de DNA. Neste experimento 12 ciclos (*) foram escolhidos para a reação de PCR de grande escala. (b) exemplo de uma amostra após a seleção de tamanho que executar em um analisador de tamanho de fragmento revelando um tamanho médio de cerca de 280 nucleotídeos. Os números à esquerda [FU] indicam a intensidade de sinal relativo. Clique aqui para ver uma versão maior desta figura.
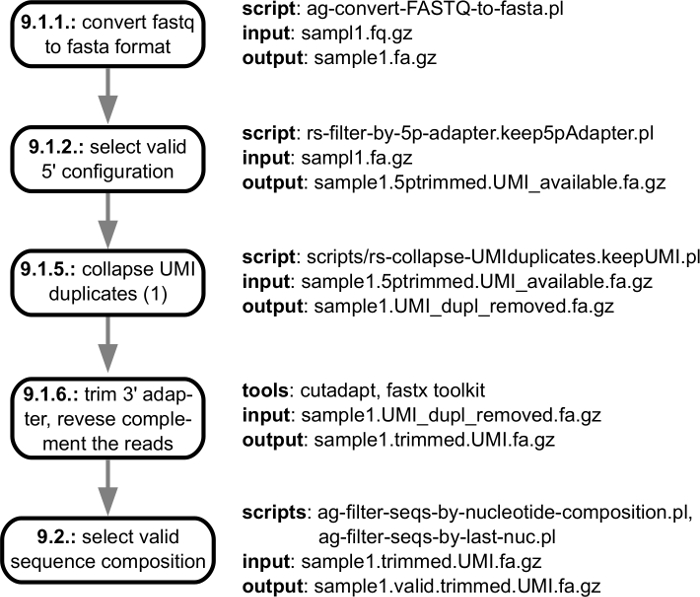
Figura 3: esboço do pré-processamento de sequenciamento leituras. Os arquivos fastq com leituras que são gerados pelo software instrumento associado sequenciamento são processados para identificar leituras de alta qualidade que serão mapeadas para o genoma correspondente. A figura mostra a especificação de entrada/saída de etapas individuais no pipeline, com links para os passos individuais do protocolo descrito na seção "Processamento de dados". Clique aqui para ver uma versão maior desta figura.
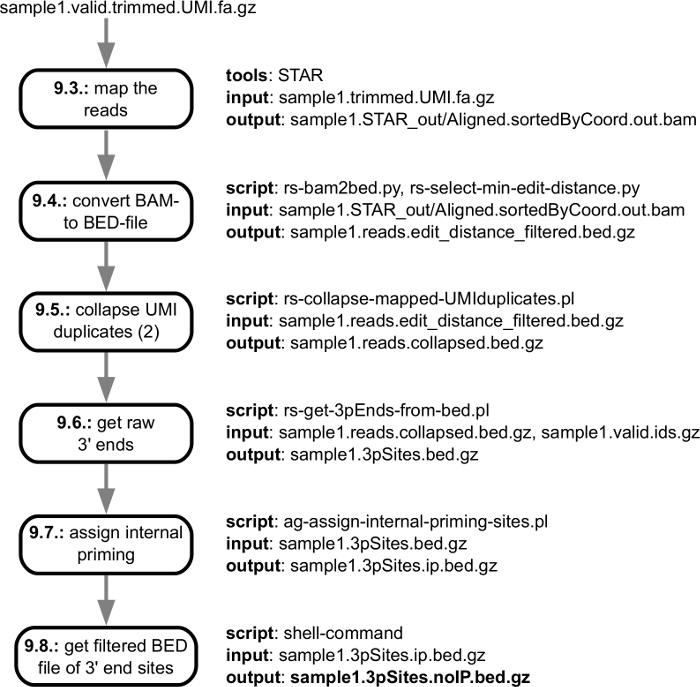
Figura 4: esboço da sequência ler processamento, desde a etapa de mapeamento de genoma para a geração de final individual 3' processamento sites. A figura mostra a especificação de entrada/saída de etapas individuais no pipeline, com links para o isão etapas do protocolo descrito na seção "Processamento de dados". O arquivo de saída principal que é entregue ao usuário é marcado em negrito. Clique aqui para ver uma versão maior desta figura.
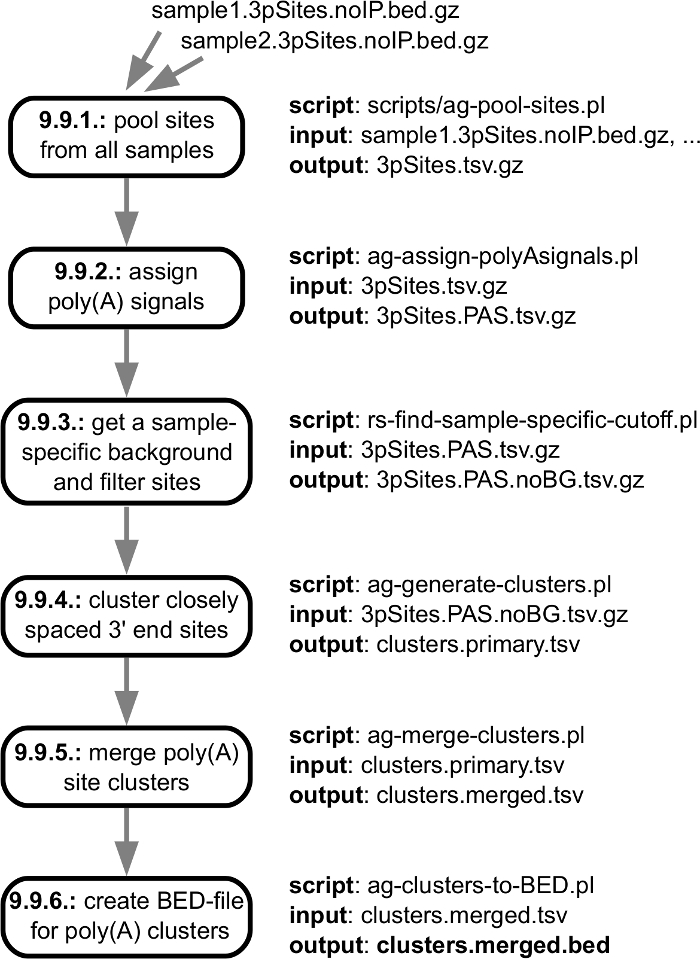
Figura 5: contorno das etapas que forem tomadas para gerar clusters de co-são regulados 3' final sequenciamento sites. A figura mostra a especificação de entrada/saída de etapas individuais no pipeline, com links para os passos individuais do protocolo descrito na seção "Processamento de dados". O arquivo de saída principal é marcado em negrito. Clique aqui para ver uma versão maior desta figura.
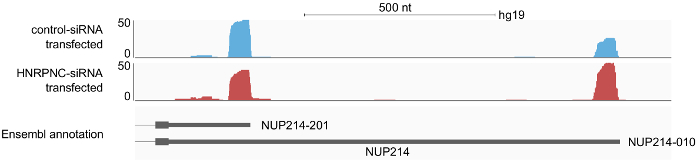
Figura 6: resultados de exemplo do perfil de 3' acabam o processamento leituras ao longo do terminal exon do gene NUP214, mostrado no navegador de genoma de IGV 16 . A-seq2 leituras foram preparadas a partir de duas amostras de células HEK 293, tratadas com um controle-siRNA ou com um HNRNPC siRNA. O lê isso documentado poli sites que foram anotados pelo pipeline análise foram salvos no formato BAM que foi usado como entrada para o navegador do genoma IGV. 3' extremidades de picos de leitura mapeiam de mRNA 3' extremidades que são anotadas em Ensembl. Os perfis indicam um aumento da utilização do longa 3' UTR isoform mediante HNRNPC knock-down. Clique aqui para ver uma versão maior desta figura.
| si-controle replicar 1 | si-controle replicar 2 | |
| identificação: 29765 | identificação: 32682 | |
| número de leituras crus | 44210258 | 68570640 |
| número de leituras válidas após aparar e filtragem | 14024538 | 21211793 |
| número de leituras de mapeamento com exclusividade | 6953674 | 13946436 |
| número de leituras, mapeamento de múltiplos loci | 2040646 | 2925839 |
| número de individual 3' terminar processamento sites | 1107493 | 1710353 |
Tabela 1: exemplo de saída do pipeline de análise. Resumos de leituras que foram obtidos em etapas individuais.
Discussão
A multidão de núcleo e fatores auxiliares que estão envolvidos no processamento de pre-mRNA 3' final é refletida em uma paisagem de poliadenilação correspondentemente complexo. Além disso, poliadenilação também é sensível a mudanças em outros processos como transcrição e emenda. 3' sites de clivagem final em pre-mRNAs são normalmente identificados com base nas caudas poli característicos que são adicionadas aos produtos de clivagem 5'. A maioria dos métodos usar primers oligo (descolamento) de comprimentos variáveis que permitem a conversão específica de poli (A)-contendo mRNAs de cDNAs em uma reação de transcrição reversa. Um problema comum desta abordagem é interna escorva para sequências A rica resultando em sítios de clivagem diferente. Dois métodos que visam contornar este artefato na fase de preparação da amostra têm sido propostos. No método 3P-seq 1, adaptadores são especificamente ligados às extremidades de caudas poli (a) com a ajuda de uma tala oligo seguido por digestão parcial de RNase T1 e transcrição reversa com TTP na reação como a única deoxynucleotide. As resultante poly(A)-poly(dT) heteroduplexes são então digeridos com RNase H e os restantes fragmentos de RNA são isolados, ligados a adaptadores e sequenciados. Um método mais simples e elegante, 2P-seq, que usa uma cartilha de sequenciamento personalizado saltando o trecho restante de oligo (descolamento) na reação de sequenciamento foi relatado pelo mesmo autores 2. Em um método relacionado, 3' lê 3, um primer extraordinariamente longo de 5 nós e 45 Ts, também contendo uma biotina são recozidos para RNA fragmentado, seguido de lavagens rigorosas para selecionar para moléculas de RNA com caudas poli (a) de mais de 50 nucleótidos. Embora 3' leituras reduz drasticamente a frequência de escorva interna, ela não completamente elimina- 3. Protocolos para a sequenciação do ARN direto também têm sido propostos, mas o lê resultante é curto e têm uma alta taxa de erro e essa abordagem não tem sido ainda mais desenvolvidos 18,19,20. A PolyA-Seq e os protocolos de Quant Seq comercializados combinam escorva (descolamento) oligo baseado com um passo de escorva aleatórios para a segunda vertente do cDNA síntese 20. O uso da reação de transcrição reversa do interruptor de modelo com a transcriptase reversa Moloney Murine leucemia vírus (MMLV) leva à geração de cDNAs com linkers em uma única etapa e desse modo não dímeros de adaptador podem aparecer no PAS-Seq e métodos SAPAS 21 , 22.
O método A-seq2 apresentado aqui se destaca em sua utilização de um nucleotídeo cleavable (dU) dentro de uma cartilha de oligo (descolamento) biotinilado. Esta modificação combina o utilitário de enriquecimento de oligo (descolamento) hibridizada, polyadenylated alvos com a remoção da maioria dos25 sequência oligo (dT) dos fragmentos isolados antes de bibliotecas são preparadas e a preservação de três Ts, que indica a presença anterior da cauda poli (a). Em contraste, métodos que utilizam o RNase H para retirar poli (a) as moléculas de RNA aleatoriamente deixam vários como. Desde em A-seq2, sequenciamento é feito da extremidade 3' dos fios antisenso, sítios de clivagem são preditos para ser localizado após o motivo NNNNTTT no início da sequência primas leituras. Os estudo randomizados tetrâmeros servem não só para permitir base chamando mas também na eliminação de artefatos de amplificação do PCR. Mais UMIs também podem ser acomodados. A possibilidade de escorva interna permanece em A-seq2 e é dirigida computacionalmente, primeiro descartando 3' termina com uma sequência de jusante codificado em genomically, A rica e, em seguida, descartando 3' clusters de final que poderiam ser explicadas pela ferragem interna na Sinal de poli A-ricos em si. Uma análise recente de sites de poli inferida com exclusividade por um grande número de protocolos indica que os sites que são exclusivos para A-seq2 têm o nucleotídeo esperada distribuição e localização dentro de genes, semelhantes a outros 3' acabar com protocolos de sequenciamento.
Um passo fundamental na-seq2 é a seleção de polyadenylated RNA e remoção de RNAs ribossomal e vários pequenos RNAs. Isso é feito facilmente por um kit de mRNA-isolamento com grânulos magnético de oligo (dT)25 . Em princípio, o RNA total isolado com fenol contendo soluções também dá alta qualidade do RNA que pode ser ainda mais sujeitos a seleção pelo kit de isolamento do mRNA ou agarose oligo (dT). Uma etapa que pode ser variada em A-seq2 é o tratamento com hidrólise alcalina, que pode ser reduzido ou estendido para obter fragmentos de RNA de tamanhos diferentes. Crítica também é que a adição de dATP 3' para 3' extremidades dos fragmentos de RNA pelo polymerase de poli é eficiente. O protocolo descrito aqui, este tratamento é aplicado para todos os fragmentos de RNA, para evitar concatemerization durante a reação de ligadura. Finalmente, notamos que, embora ligase RNA 1 é normalmente usado como um ligase do RNA, também ligates eficientemente único ADN encalhado, como fizemos aqui para ligar um adaptador à extremidade 5' das moléculas de cDNA.
Assim, A-seq2 é um eficiente e fácil de implementar o protocolo para a identificação de pre-mRNA' a extremidade 3 sites de processamento. Os desenvolvimentos futuros podem incluir reduzindo ainda mais a complexidade do protocolo e a quantidade de material necessário. O conjunto associado de ferramentas de análise de dados computacionais mais habilitar o processamento homogêneo de 3' fim leituras obtidas com uma grande variedade de protocolos de sequenciamento.
Divulgações
Os autores não têm nada para divulgar.
Agradecimentos
Os autores agradecer dona Béatrice Dimitriades ajuda com a cultura de células. Este trabalho foi financiado pela Fundação de ciência nacional Suíça concede #31003A_170216 e 51NF40_141735 (RNA NCCR & doença).
Materiais
| Name | Company | Catalog Number | Comments |
| Materials | |||
| Agarose, ultra pure | Invitrogen | 16500-500 | |
| 2100 Bioanalyzer | Agilent | G2940CA | |
| Cordycepin triphosphate (3’ dATP) | SIGMA | C9137 | |
| DNA low bind vials, 1.5 ml | Eppendorf | 22431021 | |
| Dulbecco’s Phosphate Buffered Saline | SIGMA | D8637 | |
| Dynabeads mRNA-DIRECT Kit | Ambion | AM61012 | |
| GR-Green dye | Excellgen | EG-1071 | use 1:10,000 dillution |
| HiSeq 2500 or NextSeq 500 next generation sequencers | Illumina | inquire with supplier | |
| KAPA HiFi Hotstart DNA polymerase mix | KAPA/Roche | KK2602 | |
| Nuclease free water | Ambion | AM9937 | |
| Poly(A) polymerase, yeast | Thermo Fisher Scientific | 74225Z25KU | |
| Poly(A) polymerase, E.coli | New England Biolabs | M0276L | |
| Polynucleotide kinase | Thermo Fisher Scientific | EK0032 | |
| QIAEX II Gel Extraction Kit | Qiagen | 20021 | |
| QIAquick PCR Purification Kit | Qiagen | 28104 | |
| QIAquick Gel Extraction Kit | Qiagen | 28704 | |
| RNA ligase 1, high concentration | New England Biolabs | M0437M | includes PEG-8000 |
| RNeasy MinElute RNA Cleanup kit | Qiagen | 74204 | |
| RNase H | New England Biolabs | M0279 | |
| RNasin Plus, ribonuclease inhibitor | Promega | N2618 | |
| Superscript IV reverse transcriptase | Thermo Fisher Scientiific | 18090050 | |
| Turbo DNase | Ambion | AM2238 | |
| USER enzyme mix | New England Biolabs | M5505 | |
| Dyna-Mag-2 magnetic rack | Thermo Fisher Scientific | 12321D | |
| Thermomixer C | Eppendorf | 5382000015 | Heated mixer with heated lid |
| MicroSpin columns | GE-Healthcare | 27-5325-01 | |
| Name | Company | Catalog Number | Comments |
| Buffers | |||
| Alkaline hydrolysis buffer, 1.5 x | Mix 1 part 0.1 M Na2CO3 and 9 parts 0.1 M NaHCO3. Add EDTA to 1 mM. Adjust pH to 9.2. Store aliquots at -20 °C. | ||
| 5x poly(A) polymerase buffer | Thermo Fisher Scientiific | 100 mM Tris-HCl, pH 7.0, 3 mM MnCl2, 0.1 mM EDTA, 1 mM DTT, 0.5 mg/ml acetylated BSA, 50% glycerol | |
| Biotin binding buffer | 20 mM TrisCl pH 7.5, 2 M NaCl, 0.1% NP40 | ||
| TEN buffer | 10 mM TrisCl, pH 7.5, 1 mM EDTA, 0.02% NP40 | ||
| Name | Company | Catalog Number | Sequence |
| Oligonucleotides according to Illumina TruSeq Small RNA Sample Prep Kits, for GA-IIx and Hiseq2000/2500 sequencers | Microsynth | ||
| revRA3 (RNA) | Microsynth | 5’ amino CCUUGGCACCCGAGAAUUCCA 3’ | |
| revDA5 | Microsynth | 5’ amino GTTCAGAGTTCTACAGTCCGAC GATCNNNN-3’ | |
| Bio-dU-dT25, RT primer | Microsynth | 5' Biotin-TTTTTTTTTTTTTTTTTTTTTTTTTT-dU-TTTVN 3' (V = G, A or C) | |
| PCR primer forward, RP1 | Microsynth | 5' AATGATACGGCGACCACCGAGA TCTACACGTTCAGAGTTCTACAG TCCGA 3' | |
| PCR primer reverse, RPI1, barcode in bold | Microsynth | 5' CAAGCAGAAGACGGCATACGAG ATCGTGATGTGACTGGAGTTCCT TGGCACCCGAGAATTCCA 3' | |
| Name | Company | Catalog Number | Comments |
| Oligonucleotides according to Illumina TruSeq HT-Small RNA Sample Prep Kits, for HiSeq2000/2500 and NextSeq500 sequencers | |||
| HT-rev3A (DNA/RNA) | Microsynth | 5'-amino-GTGACTGGAGTTCAGACGTGTG CTCTTCCrGrAUrC-3' | |
| HT-rev5A | Microsynth | 5' amino-ACACTCTTTCCCTACACGACGCT CTTCCGATCTNNNN 3' | |
| Bio-dU-dT25, RT primer | Microsynth | 5' Biotin-TTTTTTTTTTTTTTTTTTTTTTTTTT-dU-TTTVN 3' | |
| PCR primers forward (D501-506) | Microsynth or Illumina | 5'-AATGATACGGCGACCACCGAGAT CTACAC[i5]ACACTCTTTCCCTACA CGACGCTCTTCCGATCT -3' | |
| PCR primers reverse (D701-D712) | Microsynth or Illumina | 5'-CAAGCAGAAGACGGCATACGAG A[i7]GTGACTGGAGTTCAGACGTG TGCTCTTCCGATC-3' | |
| Documentation for Illumina multiplexing: | Illumina | https://support.illumina.com/content/dam/illumina-support/documents/documentation/chemistry_documentation/experiment-design/illumina-adapter-sequences_1000000002694-01.pdf |
Referências
- Jan, C. H., Friedman, R. C., Ruby, J. G., Bartel, D. P. Formation, regulation and evolution of Caenorhabditis elegans 3'UTRs. Nature. 469 (7328), 97-101 (2011).
- Spies, N., Burge, C. B., Bartel, D. P. 3' UTR-isoform choice has limited influence on the stability and translational efficiency of most mRNAs in mouse fibroblasts. Genome Res. 23 (12), 2078-2090 (2013).
- Hoque, M., Ji, Z., et al. Analysis of alternative cleavage and polyadenylation by 3' region extraction and deep sequencing. Nat. methods. 10 (2), 133-139 (2013).
- Martin, G., Gruber, A. R., Keller, W., Zavolan, M. Genome-wide analysis of pre-mRNA 3’ end processing reveals a decisive role of human cleavage factor I in the regulation of 3' UTR length. Cell Rep. 1 (6), 753-763 (2012).
- Gruber, A. R., Martin, G., et al. Global 3' UTR shortening has a limited effect on protein abundance in proliferating T cells. Nat. Commun. 5, 5465 (2014).
- Kivioja, T., Vähärautio, A., et al. Counting absolute numbers of molecules using unique molecular identifiers. Nat. methods. 9 (1), 72-74 (2011).
- Gruber, A. J., Schmidt, R., et al. A comprehensive analysis of 3' end sequencing data sets reveals novel polyadenylation signals and the repressive role of heterogeneous ribonucleoprotein C on cleavage and polyadenylation. Genome Res. 26 (8), 1145-1159 (2016).
- Lingner, J., Keller, W. 3'-end labeling of RNA with recombinant yeast poly(A) polymerase. Nucleic Acids Res. 21 (12), 2917-2920 (1993).
- Quail, M. A., Kozarewa, I., et al. A large genome center's improvements to the Illumina sequencing system. Nat. methods. 5 (12), 1005-1010 (2008).
- Rahmann, S. Snakemake--a scalable bioinformatics workflow engine. Bioinformatics. 28 (19), 2520-2522 (2012).
- . Anaconda Software Distribution Available from: https://continuum.io (2016)
- Dobin, A., Davis, C. A., et al. STAR: ultrafast universal RNA-seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- Martin, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet.journal. 17 (1), 10-12 (2011).
- Li, H., Handsaker, B., et al. The Sequence Alignment/Map format and SAMtools. Bioinformatics. 25 (16), 2078-2079 (2009).
- Robinson, J. T., Thorvaldsdóttir, H., et al. Integrative genomics viewer. Nat. Biotechnol. 29 (1), 24-26 (2011).
- Quinlan, A. R., Hall, I. M. BEDTools: a flexible suite of utilities for comparing genomic features. Bioinformatics. 26 (6), 841-842 (2010).
- Ozsolak, F., Platt, A. R., et al. Direct RNA sequencing. Nature. 461 (7265), 814-818 (2009).
- Yao, C., Biesinger, J., et al. Transcriptome-wide analyses of CstF64-RNA interactions in global regulation of mRNA alternative polyadenylation. Proc. Natl. Acad. Sci. U. S. A. 109 (46), 18773-18778 (2012).
- Lin, Y., Li, Z., et al. An in-depth map of polyadenylation sites in cancer. Nucleic Acids Res. 40 (17), 8460-8471 (2012).
- Shepard, P. J., Choi, E. -. A., Lu, J., Flanagan, L. A., Hertel, K. J., Shi, Y. Complex and dynamic landscape of RNA polyadenylation revealed by PAS-Seq. RNA. 17 (4), 761-772 (2011).
- Fu, Y., Sun, Y., et al. Differential genome-wide profiling of tandem 3' UTRs among human breast cancer and normal cells by high-throughput sequencing. Genome Res. 21 (5), 741-747 (2011).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados