Method Article
Extrémité 3' séquençage bibliothèque préparation avec A-seq2
Dans cet article
Résumé
Ce protocole décrit une méthode de cartographie pré-ARNm ' extrémité 3 sites de traitement.
Résumé
Études dans la dernière décennie ont révélé une variété complexe et dynamique de clivage de l’ARN pré-messager et réactions de polyadénylation. ARNm avec longues régions 3' non traduite (RTNS) est générés dans les cellules différenciées tandis que les cellules en prolifération expriment préférentiellement transcrits avec court 3' UTR. Les auteurs décrivent le protocole A-seq, maintenant à sa deuxième version, qui a été développé pour cartographier les sites de polyadénylation génome et étudier la régulation de l’ARN pré-messager 3' fin au traitement. Ce protocole actuel tire également parti de la polyadénylate (poly(A)) de queues qui sont ajoutés au cours de la biogenèse des mammifères plus mRNA d’enrichir à l’ARNm fully-Process. Un adaptateur d’ADN avec deoxyuracil à sa quatrième position permet le traitement précis des ARNm 3' fragments terminaux pour le séquençage. Non compris la culture de cellules et les ligature des trompes au jour le jour, le protocole exige environ 8 h de temps pratique. Avec elle, un logiciel facile à utiliser pour l’analyse des données dérivées de séquençage est fourni. A-seq2 et le logiciel d’analyse associés fournissent une solution efficace et fiable pour le mappage d’un pré-ARNm 3' se termine par un large éventail de conditions, de 106 ou moins de cellules.
Introduction
La capture et le séquençage d’ADN messagère 3' extrémités permet l’étude du traitement de l’ARNm et la quantification de l’expression génique. En raison de leur queue poly (a), ARNm eucaryotes peut être efficacement épuré de lysats cellulaires totales avec perle-immobilisé oligo-deoxythymidine (molécules de oligo(dT)), qui peut également amorcer la synthèse d’ADNc. Toutefois, cette approche présente deux inconvénients. Tout d’abord, s’étend de A qui sont internes aux transcriptions peut également amorcer la synthèse de cDNA, résultant dans des sites parasites poly (a). Deuxièmement, homogène, poly (a) s’étire pose des défis spécifiques pour le séquençage, mis à part ne pas être instructif pour l’identification de la transcription. Différentes approches ont été proposées pour contourner ces limitations, telles que la transcription renversée par le biais de poly (a) queues suivies par digestion RNase H (3P-seq 1), utilisation d’un apprêt de séquençage personnalisé se terminant par 20 Ts (2p-seq 2), présélection des Fragments d’ARN avec queue poly (a) de plus de 50 nucléotides avec un apprêt de45 CU5T suivie de RNase H digestion (lectures de 3' 3) et l’utilisation d’un apprêt d’oligo-dT qui contient l’adaptateur 3' dans une épingle à cheveux (A-seq 4).
L’A-seq2 récemment mis au point la méthode 5 a pour but de contourner la séquence poly (a) et en même temps pour réduire au minimum la proportion de dimères qui sont générés par l’individu-ligature des adaptateurs, survenant en particulier lorsque la concentration molaire d’adaptateurs l’emporte sur la concentration de l’insert. Ce problème peut être éliminé lorsque les deux adaptateurs sont ligaturés au même type de polynucléotide se termine comme dans A-seq2, où les adaptateurs 3' sont ligaturés à l’extrémité 5' de fragments d’ARN et les adaptateurs de 5' à 5' extrémités des ADNc après transcription inverse. La méthode est plus pratique que notre A proposé antérieurement-seq - dont le séquençage a été dans la 5'-3' direction exigeant donc précisément contrôlée RNA fragmentation-, tout en conservant une grande précision de poly (a) identification des sites. Environ 80 % de du est séquencé en échantillons typiques carte unique au génome et conduire à l’identification de plus 20 000 grappes site de poly (a), plus de 70 % de qui recoupait annoté 3' UTR.
En bref, le protocole A-seq2 commence par la fragmentation de l’ARNm et ligature des adaptateurs inverse-complément 3' à l’extrémité 5' des fragments d’ARN. Poly (A)-ARN contenant rétroconcevez puis transcrit avec un apprêt d’oligo (décollement) long de 25 nucléotides (nt) qui contient un nucléotide de l’ancre à l’extrémité 3', un à la position 4 et un biotine à l’extrémité 5', permettant la liaison de l’ARNC aux billes magnétiques streptavidine. La plupart de l’amorce, y compris la biotine, est retiré de l’ADNc par clivage à par la composition de l’enzyme utilisateur contenant l’uracile DNA glycosylase (UDG) et le DNA glycosylase-lyase endonucléase VIII. Cette réaction laisse intacte se termine pour la ligature d’un adaptateur 5' et la gauche Ts trois après clivage reste pour marquer l’emplacement de la queue poly (a). Car les adaptateurs 5' et 3' sont fixées par ligature aux extrémités 5' destinataire, aucune dimères d’adaptateur ne sont générés. Quatre nucléotides aléatoire-mers a présenté au début de lectures permet la résolution de cluster sur les instruments de séquençage de l’état-of-the-art et peuvent aussi servir d’identificateur moléculaire unique (UMI) pour la détection et la suppression des artefacts d’amplification PCR. La taille de l’UMI peut être encore augmentée comme fait dans d’autres études 6. Le protocole génère les lectures qui sont inverser complémentaire de l’ARNm 3' extrémités, tout départ avec un tétramère randomisé suivi 3 Ts. traitement des lectures qui ont les 3 Ts diagnostiques à leur extrémité commence de 5' avec la correction des artefacts d’amplification PCR par exploiter les UMIs, suppression de séquences de 3' adaptateur et inverser la complémentation. Lectures qui peuvent provenir de d’amorçage oligo (décollement) à des sites riches en A internes sont également identifiés par le calcul et mis au rebut. Les fausses sites manquent généralement de l’un des 18 bien caractérisés et signaux conservés poly (a) qui devraient être situé ~ 21 nucléotides en amont du clivage apparent site 7.
Le protocole exige environ 8 h de temps pratique, sans compter la culture cellulaire et les ligature des trompes pendant la nuit. Les associés lire analyse logiciel permet une identification de site de haute précision poly (a). Sur le site de poly (a) grappes créé sur 4 échantillons davantage mis en évidence dans ce manuscrit (deux réplicats biologiques des siARN contrôle et des cellules traitées au TR-HNRNPC) 84 % de chevauchement avec un gène annoté et, sur ce, 75 % de chevauchement avec un 3' UTR et 86 % soit avec un 3' UTR ou un terminal exon. Le coefficient de corrélation de Pearson d’expression des extrémités 3' dans les échantillons est de 0,92 et valeurs de plus de 0,9 sont généralement obtenus par la méthode. A-seq2 est donc une méthode pratique qui donne des résultats très reproductibles.
Protocole
1. ARNm et la croissance cellulaire Isolation
- la croissance de cellules selon votre conception expérimentale en plaques 6 puits à ~ 1 x 10 6 cellules / puits à 80 % confluence.
- Enlever le milieu de croissance et laver les cellules une fois avec un tampon phosphate salin. Lyse directement les cellules sur la plaque en ajoutant 1 mL de tampon de lyse du kit d’isolation des ARNm. Transférer le visqueux lysat dans un tube de 15 mL en plastique avec un embout de pipette de 1 mL. Utilisez une spatule en caoutchouc pour détacher complètement le matériel cellulaire provenant de la surface de la plaque.
- Cisaillement le lysat visqueux contenant l’ADN avec une seringue de 1 mL attachée à une seringue hypodermique 23 G par plusieurs vigoureux et descendre les mouvements du piston jusqu'à ce que le lysat n’est donc plus visqueux. Pointer l’aiguille de la seringue vers le centre du fond pour éviter d’éjecter le lysat hors du tube.
- Transférer le lysat dans un tube de 1,5 mL à l’aide de la seringue. Faire tourner 5 min à 20 000 x g et 4 ° C pour éliminer les débris. Utiliser l’ADN faible liaison 1,5 mL flacons tout au long du protocole.
- Pendant l’exécution de la centrifugeuse, laver 300 µL de suspension oligo (dT) 25 billes magnétiques sur une grille magnétique avec 500 µL de tampon de lyse. Mélanger les tubes 2 - 3 fois sur la grille. Retirez le tampon après que la solution est claire. Recueillir le liquide clair surnageant de l’étape 1.4 et ajouter aux talons. Remettre en suspension et placer les tubes sur une roue en rotation pendant 10 min.
- Placer les tubes sur un support magnétique. Retirer le liquide clair après 2 min. Ajouter 0,8 mL tampon A partir du kit d’isolation des ARNm. Tourner le tube de 180° degrés sur la grille, 2 ou 3 fois. Répétez cette étape de lavage une fois de plus avec le tampon A.
- Laver les billes 2 fois avec 0,8 mL de tampon B comme indiqué au point 1.6.
- Pour éluer les ARNm lié de la perle, ajoutez 33 µL H 2 O et remettre en suspension les perles. Chauffer jusqu'à 75 ° C pendant 5 min sur un bloc chauffant. Tourner immédiatement les tubes pendant 1 s et place sur la grille magnétique. Transférer le surnageant dans un nouveau tube. Échantillons peuvent être conservés à-80 ° C jusqu'à l’utilisation autre.
- Ajouter 66 µL de tampon de l’hydrolyse alcaline à la 33 µL ARNm (étape 1.8), mélanger et chauffer pendant exactement 5 minutes à 95 ° C sur un bloc de chauffage. Réfrigérer immédiatement les tubes sur Ice.
- RNA isoler avec un kit de nettoyage ARN.
NOTE : Confirmer le volume ; Il devrait être de 100 µL. Éthanol de- Ajouter 350 µL de tampon de RLT de la trousse et 250 µL. Charge sur la colonne et un essorage pendant 30 s à 8 000 x g à la température ambiante (RT). Laver avec 500 µL de tampon de RPE de la trousse. Laver avec 500 µL 80 % d’éthanol. Tournez pendant 5 min à 20 000 x g pour sécher la colonne. Ajouter 36 µL H 2 O à colonne et faites tourner pour 1 min à 20 000 x g. jeter la colonne et enregistrer l’éluat.
2. 5 ' mettre fin à la Phosphorylation et la DNase traitement
- Ajouter 5 µL polynucléotide kinase tampon, 5 µL 10 mM ATP, 1 µL ribonucléase inhibiteur, 1 µL DNase et 2 µL polynucléotide kinase à Echantillons et incuber à 37 ° C pendant 30 min. préparer éventuellement réaction maître mélanges tout au long du protocole en mélangeant des 1,1 volumes x n (n = nombre d’échantillons) de chaque composant.
- Changer de tampon et supprimer ATP sur une colonne à centrifuger pour empêcher l’ajout de poly (a) à l’étape suivante.
- Prespin-colonnes à centrifuger à 735 x g pendant 1 min. transférer les colonnes nouveaux flacons de 1,5 mL et chargement les réactions kinase sur les colonnes. Essorage les colonnes 2 min à 735 x g. Jetez les colonnes et placer les tubes avec des réactions recueillies sur la glace ou stockent à -80 ° C.
3. Blocage 3 ' se termine par cordycépine Triphosphate
Remarque : il est essentiel pour bloquer le 3 ' extrémités des fragments d’ARN afin d’éviter leur concatemerization dans les réactions subséquente ligature. 3 ' fins qui ne sont pas déjà bloqués par une ( phosphate cyclique) après hydrolyse sont traités par l’addition d’un 3 ' dATP (cordycépine triphosphate) nucléotides de terminator chaîne à l’aide de poly (a) polymérase. Ici, la polymérase de poly (a) de levure (yPAP), qui a été exprimé et purifiée comme décrit dans 8 a été utilisé à une concentration de 0,5 mg/mL. Levure ou e. coli PAP les deux ont presque la même activité pour ajout de 3 ' dATP et peuvent être achetés dans le commerce (voir la Table des matières).
Tampon de réaction- Ajouter 13,5 µL 5 x concentré poly (a) polymérase, 2 µL de 10 mM 3 ' dATP, inhibiteur de RNase 1 µL et le 1 µL poly (a) polymérase à la réaction de l’étape 2.2.1. Mix et spin pour 1 s. Incuber à 37 ° C pendant 30 min. Ajouter 32,5 µL H 2 O à chaque réaction. Purifier l’ARN comme au point 1.10.1. Éluer l’ARN avec 14 µL H 2 O.
4. Ligature de Reverse 3 ' adaptateurs aux 5 ' fin de Fragments d’ARN
- Placer les réactions dans un concentrateur sous vide pendant 10 min afin de réduire le volume à 6 µL. Ajouter 3 µL 10 x tampon de ligature ARN T4, 3 µL 10 mM ATP , 15 µL PEG-8000, inhibiteur de RNase de 1 µL, 1 µL de complément inverse de 0,1 mM 3 ' adaptateur " revRA3 " (voir la Table des matières) et mélanger de la ligase de forte concentration RNA 1 µL 1,.
- Incuber les réactions à 24 ° C pendant 16 h une table de mixage chauffée avec mélange intermittent à 1 000 tr/min. Ajouter 70 µL H 2 O à chaque réaction et mélanger. Purifier l’ARN comme au point 1.10.1. Éluer l’ARN avec 14 µL H 2 o. échantillons peuvent être conservés à-80 ° C à ce stade.
5. Reverse Transcription (RT)
- Place les éluats dans un concentrateur sous vide pendant 3 min pour réduire le volume à 11 réactions de transfert µL. à 200 µL PCR tubes. Ajouter 1 apprêt de 0,05 mM RT µL " Bio-dU-dT25 ". Chauffer pendant 5 min à 70 ° C dans un cycler PCR et laisser à ta pendant 5 min.
- Ajouter 1 µL 10 mM dNTPs, 4 µL de tampon de la transcriptase inverse x 5, 1 µL 0,1 M TNT, 1 inhibiteur de RNase µL et 1 µL de la transcriptase inverse. Mélanger et chauffer les réactions pendant 10 min à 55 ° C et à 10 min à 80 ° C dans un cycler PCR. Rester sur la glace ou à-80 ° C pour une conservation plus longue.
6. Digestion avec uracile DNA Glycosylase Enzyme Mix
- pipette 100 µL streptavidine-perles dans un flacon de 1,5 mL, resuspendre dans 800 µL de tampon de liaison biotine et placer sur un support magnétique. Inverser les tubes 2 - 3 fois. Retirez le tampon lorsque claire. Répétez l’étape de lavage. Remettre en suspension les perles dans 200 µL de tampon de liaison biotine.
- Ajouter la réaction de transcription inverse à la solution de perles et incuber 20 min à 4 ° C sur une roue en rotation. Laver les billes x 2 avec liaison biotine tampon comme dans l’étape 6.1 et 2 x avec tampon dix sur un support magnétique. Les perles de remettre en suspension dans 50 µL de tampon de dix, ajoutez 2 µL uracile DNA glycosylase enzyme mix et incuber 1 h à 37 ° C dans un mélangeur avec mélange intermittent.
- Ajouter 50 µL H 2 O, 11 µL de RNase H tampon et 1 µL RNase H aux réactions. Incuber à 37 ° C pendant 20 min. tubes de Place sur un support magnétique et transférer le liquide contenant de l’ADNc clivé dans un nouveau tube
- purifier l’ADNc clivée.
- Ajouter 550 µL de tampon de PB de la trousse de purification du PCR pour les réactions de clivage. Ajouter 10 µL d’acétate de sodium 3M, pH 5,2 pour abaisser le pH. Charger les réactions sur les colonnes à centrifuger élution minimal et un essorage à 17 000 x g pendant 1 min.
- Ajouter 750 µL de tampon de PE à colonnes et un essorage à 17 000 x g pendant 1 min. jetez l’accréditif. Faites tourner les colonnes à 17 000 x g pendant 1 min sécher. Transférer les colonnes dans un flacon de 1,5 mL, ajouter 16 µL H 2 O et un essorage à 17 000 x g pendant 1 min. placent les réactions dans un concentrateur sous vide pendant 8 min pour se concentrer sur un volume de 7 µL.
7. Ligature de 5 ' adaptateurs à 5 ' extrémités d’ADNc
- de l’ADNc isolé, ajouter 3 µL 10 x T4 ARN ligase 1 un tampon 3 µL ATP de 10 mM, 15 µL PEG-8000, 1 µL 50 µM " revDA5 " oligo et la ligase d’ARN T4 forte concentration 1 µL 1. Incuber à 24 ° C pour 20 h. Ajouter 70 µL H 2 O à chaque réaction. Échantillons peuvent être conservés à-20 ° C à ce stade.
8. Pilote PCR, Amplification des bibliothèques et sélection de la taille
- dans une réaction du pilote, déterminer le nombre optimal de PCR cycles pour atteindre l’amplification de la bibliothèque au sein de la phase exponentielle.
- Pipette 25 µL ADN polymérase mix, réaction de ligature 20 µL, 2 µL H 2 O, 1,5 µL 10 µM avant amorce PCR (RP1) et 1,5 d’une amorce d’index PCR inverse µL de 10 µM dans tube PCR 200 µL.
- Exécuter le cycler avec le programme suivant : 3 min 95 ° C, suivie de 20 cycles de 20 s 98 ° C, 20 s 67 ° C et 30 s 72 ° C. recueillir 7 µL d’extraits après les cycles de 6, 8, 10, 12, 14, 16 et 18 directement depuis le cycleur. Ajouter 1 µL 10 x chargement tampon (50 % de glycérol, 0,05 % de xylène cyanol). Remarque : Veuillez suivre les recommandations du fournisseur si vous utilisez multiplexage lors de la combinaison des codes à barres.
- Des produits séparés en petites fentes sur un gel d’agarose de 2 % à 1 x TBE tampon contenant un 01:10, 00 dilution du colorant vert fluorescent.
- Aliquotes de charge sur un gel d’agarose 2 % gel et exécutez le gel à 100 volts pendant 15 min. migration de visualiser des produits PCR sur un système de documentation gel.
- Utiliser le nombre de cycles au début d’amplification exponentielle dans la réaction du pilote pour une réaction de PCR à grande échelle avec deux fois les volumes tel qu’utilisé pour la réaction du pilote ( Figure 2).
- Pour des réactions de PCR à grande échelle, de se concentrer et de dessalement les réactions tout d’abord avec un kit de purification de PCR et de séparer les produits sur les fentes larges sur gels d’agarose à 2 % dans un tampon de x TBE 1.
- Découper des tranches de gel contenant 200-350 nt ADN produits. Faire fondre le gel dans le tampon de chaotropes à ta jusqu'à 30 min. Extraire l’ADN des tranches de gel avec un kit d’extraction de gel. Ne pas chauffer à 50 ° C afin d’éviter la partialité dans la liaison d’ADN riches en A 9.
- Soumettre pour l’ordonnancement de.
Remarque : En règle générale, 50 cycles de lecture seule (SR50) sont suffisants (voir, par ex., https://www.illumina.com/technology/next-generation-sequencing.html).
9. Traitement des données
Remarque : les données de séquençage qui en résulte (au format fastq) sont traitées avec le logiciel disponible dans le référentiel de gitlab (https://git.scicore.unibas.ch/zavolan_public/A-seq2-processing). L’analyse comprend quatre grandes étapes : (1) téléchargement le dépôt git, (2) installation d’un environnement virtuel, (3) la définition des paramètres spécifiques dans le fichier de configuration et (4) lancer l’analyse par ‘ snakemake ’ 10. toute l’analyse effectuée à l’étape 4 ne nécessite qu’une seule commande. On trouvera une description détaillée étape par étape de l’analyse dans le fichier README dans le référentiel de gitlab et une brève description est disponible ci-dessous. Toutes les étapes de transformation individuelles sont accomplis par l’exécution d’outils accessibles au public, soit à partir de sources externes ou préparés en interne. Le pipeline calcul dépend un environnement virtuel de python 3 base anaconda 11 avec le paquet de snakemake disponible 10. Il fonctionne sur des machines avec système d’exploitation de type Unix et a été testé dans un environnement Linux CentOS 6.5 le système d’exploitation installé et 40 Go de RAM disponible. Dépendances de logiciel sont contrôlés automatiquement dans l’environnement virtuel. Les outils logiciels disponibles publiquement suivants sont requis et ainsi installés ainsi que de l’environnement : snakemake (v3.9.1) 10, fastx toolkit (v0.0.14) 12, STAR (v2.5.2a) 13 , cutadapt (v1.12) 14, samtools (v1.3.1) 14 , 15, bedtools (v2.26.0) 16 , 17.
- Informatique préalable de lectures d’ADNc
Remarque : la profondeur de séquençage peut varier entre pistes et, selon l’instrument, les données d’un échantillon peuvent être fractionnées sur plusieurs fichiers de séquence. Si c’est le cas, concaténer les fichiers qui correspondent à un échantillon dans un seul fichier d’entrée qui est utilisé dans les étapes suivantes.- Convertir le fichier de fastq au format fasta.
- Extrait de lit avec une structure correcte (3 thymidines aux positions 5, 6 et 7 de la lecture).
Remarque : Une lecture qui est correctement préparée selon le protocole expérimental décrit ci-dessus doit avoir la structure (de 5 ' fin) : 4-nucléotides code à barres - 3 thymidines - inverser complément de transcription 3 ' fin. - Stocker les informations sur le tétramère de départ dans la ligne de description de la séquence.
Remarque : Le tétramère sert d’identificateur moléculaire unique (UMI) qui facilite la correction des artefacts d’amplification plus loin dans l’analyse. - Enlever les sept premiers nucléotides de la lecture ' s 5 ' fin.
- Corriger pour les artefacts d’amplification en gardant qu’une seule copie du se lit avec le même insérer séquence et UMI.
- Supprimer la partie de la 3 ' fin correspondant à la séquence de l’adaptateur et le complément puis inverser la séquence. Seulement procéder à des lectures qui ont une longueur minimale (par défaut : 15 nt).
Remarque : selon la longueur du fragment d’ADN messagère original et le nombre de cycles de séquençage, le 3 ' fin de la lecture peut-être contenir des partie des 3 ' adaptateur, qui est enlevé lors de cette étape.
- Extrait toutes les lectures qui répondent aux critères suivants : maximum 2 nucléotides inconnus (' N '), maximum 80 % comme et dernier nucléotide de la lire, pas de A. Ces lectures sont réputées être de qualité suffisante pour être utilisées dans l’analyse.
- Mapper les lectures sur le génome d’un outil qui gère les lectures épissés et génère un fichier de sortie au format BAM.
- STAR si est utilisé, créez un fichier avec l’index du génome dont les lectures doivent être mappés. Pour le génome humain, cette étape nécessite 35 Go de mémoire (vive RAM).
- Carte les lectures au génome.
NOTE : (notes spécifiques aux étoiles) Soft-découpage est désactivée afin de forcer le mappage des 3 ' fin de chaque lire puisqu’il s’agit le nucléotide immédiatement en amont du site de clivage.
- Convertir le BAM dans un lit-fichier. Si une lecture correspond à plusieurs emplacements, garder seulement ceux qui ont le plus bas edit distance.
Remarque : Le nombre de copies de la lire mappé à un emplacement spécifique est utilisé comme partition. Les lectures qui mappent vers plusieurs emplacements sont comptées très légèrement à chaque emplacement avec un poids égal à 1/nombre d’emplacements auquel correspond une lecture.
Lectures - effondrement qui varient d’une erreur susceptible de séquençage. Si deux lectures distinctes carte au même endroit (la position de début et de fin des mappages sont identiques) et ils partagent le même UMI, les considérer comme des doublons PCR et garder qu’un.
- Déduire tous individuel pré-ARNm 3 ' sites de traitement de fin.
Remarque : Une lecture individuelle constitue une preuve pour un 3 ' fin lorsque ses quatre derniers nucléotides sont mappés au génome sans erreur. La position vers laquelle le 3 ' fin des cartes lecture est stocké dans le site de clivage. - Détecter 3 ' fin des sites qui pourraient provenir de l’amorçage interne. Définir le site artefact d’amorçage interne lorsque les 10 nt en aval du site de clivage dans le génome de satisfaire l’un des critères suivants : contient plus de six comme, contient six consécutives comme ou commence par une des tétramères suivantes : AAAA, AGAA, AAGA ryane .
- Générer une table des 3 individuels ' fin des sites de transformation au format lit.
- Identify réglementés indépendamment les clusters de sites de poly (a).
Remarque : Les étapes décrites ici suivent la procédure qui a été introduite dans une publication antérieure 5.- Commencer par collecter 3 individuels ' fin des sites de traitement qui ont été obtenus dans tous les échantillons de l’étude.
- Annoter connu poly (a) signaux 7 dans la région de 09:00 nucléotides autour de chacune des 3 ' site de traitement fin. Sites
- Identify poly (a) exprimés ainsi le fond de chaque échantillon.
- Trier les sites par leur expression brute au sein de l’échantillon actuel. Parcourir la liste des sites de haut en bas, associant des sites de rang inférieurs avec un site de rang plus élevé si elles sont situées à une distance prédéfinie dans le génome (par défaut : 25 nt up - ou en aval) sur le site de haut rang de.
Remarque : Tous les sites de bas rang associés à un site de haut rang définissent un cluster dont l’expression est le nombre de lectures documenter tous ces sites. - Trier ces grappes par expression et parcourir la liste des grappes du plus élevé au plus faible expression, déterminer le seuil d’expression c au cours de laquelle le pourcentage de bouquets avec un poly (a) annoté du signal baisse sous un seuil prédéfini ( par défaut : 90 %).
- Jeter des sites à partir de n’importe quel cluster au-dessous du seuil de.
- Trier les sites par leur expression brute au sein de l’échantillon actuel. Parcourir la liste des sites de haut en bas, associant des sites de rang inférieurs avec un site de rang plus élevé si elles sont situées à une distance prédéfinie dans le génome (par défaut : 25 nt up - ou en aval) sur le site de haut rang de.
- Cluster rapprochées 3 ' sites obtenus dans les trois échantillons de fin.
NOTE : Tri 3 ' fin traitement sites tout d’abord par le nombre de supporter des échantillons, puis par la somme de la normalisée lu comte (lit par million (tr/min)) dans les trois échantillons. Parcourir la liste de haut en bas, associant sites de rang inférieur avec les sites classés plus élevé lorsque leur distance sur le site de grade supérieur n’est pas supérieure à une limite prédéfinie (par défaut : 12 nt). Chaque fois qu’un des 3 constituant ' fin site chevauche un signal annoté poly (a) ou a un signal de poly (a) directement en aval, le cluster correspondant est marqué pour une inspection plus approfondie détecter l’amorçage interne. - Fusion poly (a) du site grappes.
Remarque : Lorsqu’un cluster est marqué comme un candidat putatif amorçage interne, il est fusionné dans un cluster en aval, si les deux groupes partagent leurs signaux de poly (a) ou conservé si le site plus en aval dans le cluster a un signal de poly (a) situé au minimum distance en amont (par défaut : 15 nt). Enfin, les grappes très rapprochées sont fusionnés si : (i) qu’ils partagent le même signal de poly (a), ou (ii) la durée de la grappe qui en résulte ne dépasse pas un maximum (par défaut : 25 nt). - Stocker les grappes au format de fichier lit avec le total normalisé lues comte de 3 tous les ' fin de sites dans chaque cluster comme score.
Résultats
Poly (A)-contenant ARN a été isolé dans des cellules cultivées, fragmentés par hydrolyse alcaline et d’ADNc ont été faites par reverse transcription avec des amorces oligo (décollement). Le cDNA qui en résulte a été immobilisé sur des billes de streptavidine, dU a été coupée dans la réaction de l’excision spécifique uracile, adaptateurs ont été ligaturés à 5' et 3' extrémités le fragment clivé et les inserts ont été séquencées. La figure 1 illustre un aperçu graphique de l’expérience.
Pour les cellules HeLa et HEK293, 106 cellules étaient suffisantes pour identifier les sites de poly (a) pour la grande majorité des gènes codant pour des protéines à la fin de la procédure. Toutefois, pour d’autres types de cellules ou de tissus, qu'il peut être nécessaire tester la saturation du nombre de sites identifiés poly (a) le nombre de cellules utilisées dans l’expérience augmente. Les résultats représentatifs du pilote PCR étape et le fragment d’ADN analyse de l’échantillon avant de séquençage sont indiquées à la Figure 2.
La figure 3 montre les étapes de pré-traitement de l’analyse computationnelle, à partir du fichier fastq obtenu à partir du séquenceur et se terminant avec les qualité-vérifié, garnis d’adaptateur des lectures qui sont prêts à être mappé au génome. La figure 4 illustre les étapes de l’analyse qui commencent par la cartographie les lectures et le génome correspondant et la fin avec le catalogue de l’extrémité 3' des ARNm sites identifiés dans un échantillon donné de traitement. Si plusieurs échantillons sont analysées, des étapes supplémentaires sont effectués pour correspondre à l’extrémité 3' des sites qui ont été trouvées dans des échantillons individuels de traitement et de signaler leur abondance dans les trois échantillons. Ces étapes sont indiquées à la Figure 5.
Ainsi, une fois les échantillons ont été séquencés, l’analyse de la séquence résultante lire des fichiers (au format fastq) via le pipeline de traitement disponible est simple. Après avoir ajouté les informations sur les exemples pour le fichier de configuration, l’exécution du pipeline se traduira par deux principaux types de fichiers de sortie : 1) lit les fichiers avec toutes les 3' finissent traitement sites identifiés dans des échantillons individuels (par exemple » « « « sample1.3pSites.noIP.Bed.gz ») et 2) lit un fichier avec tous les clusters de sites de poly (clusters.merged.bed) dans l’ensemble de tous les échantillons de l’étude. La sortie inclut également les coordonnées du génome pour toutes les lectures de chaque échantillon individuel (par exemple « sample1. STAR_out/Aligned.sortedByCoord.out.BAM ») qui peuvent plus tard être affichés dans un navigateur de génome comme IGV16. Inspection visuelle des lecture ou les profils fournit généralement un premier aperçu de la répartition des sites de poly (a) dans le génome et les changements qui se produisent lors de perturbations spécifiques qui ont été réalisées dans l’étude. Par exemple, dans la Figure 6 , la réponse d’un gène spécifique à la précipitation de la protéine HNRNPC est montrée.
Synthèses de ces distributions de Génome-large sont également fournis (tableau 1). Spécifiquement, les fichiers de sortie dans le répertoire « comtes/annotation_overlap » contiennent des fractions de sites qui se chevauchent avec caractéristiques annotés (du gtf fichier fourni comme entrée ; annotés sont : 3' UTR, terminal exon, exon, intron, intergénique). Enfin, pour chaque échantillon, les résultats des étapes de transformation individuelles sont également enregistrés (par exemple « sample1.summary.tsv »). Cela inclut les numéros de : lectures brutes dans chaque échantillon, les lectures qui ont la structure attendue de l’extrémité 5', les lectures qui restent après l’effondrement complets doublons PCR, lit de haute qualité selon les critères définis à l’étape 9.2, lit cette carte unique au génome (après l’effondrement de celles qui résultent d’erreurs de séquençage, reportez-vous à l’étape 9,5), multi-cartographie lit (après l’effondrement de celles qui résultent d’erreurs de séquençage, reportez-vous à l’étape 9,5), sites à chaque extrémité de l’échantillon, brut 3' sites de traitement de traitement brut (non ordonné en clusters) 3' fin sans les candidats potentiels internes d’amorçage, unique 3' fin des sites de traitement de tous les échantillons sans candidats internes d’amorçage et ensemble de clusters sites poly (a).
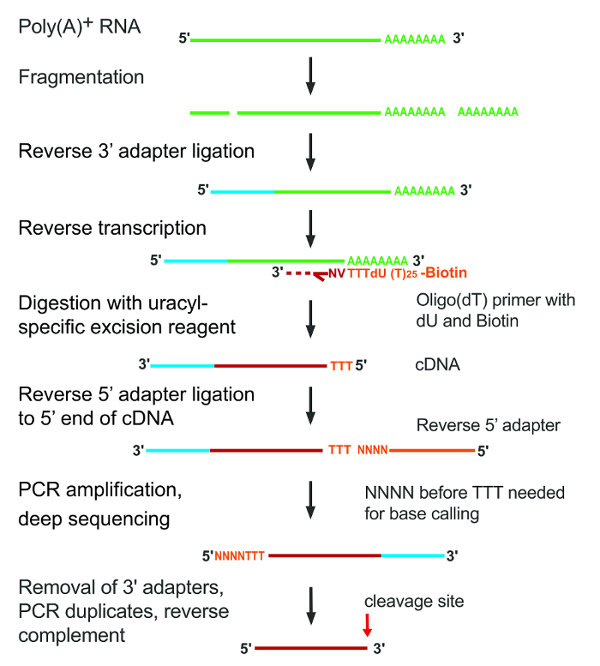
Figure 1 : étapes principales du protocole A-seq2. Différentes étapes sont indiquées sur le côté gauche de la figure. Fragments d’ARN INSERT sont dépeints comme des lignes vertes qui rougissent pour ADNc après transcription inverse ; les adaptateurs sont colorés en bleu ou en orange. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
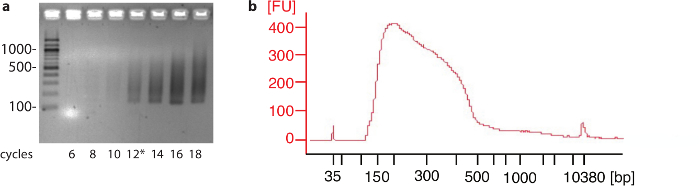
Figure 2 : pilote PCR et profil du produit final. (un) aliquotes de la réaction PCR ont été prélevés à des cycles différents et séparés sur gel d’agarose de 2 %. Les nombres à gauche indiquent taille dans des bandes respectives dans l’échelle de l’ADN, les nucléotides. Dans cette expérience, 12 cycles (*) ont été choisis pour la réaction de PCR à grande échelle. (b) exemple d’un échantillon après sélection de la taille sur un analyseur de taille de fragment révélant une taille moyenne d’environ 280 nucléotides. Les nombres à gauche [FU] indiquent intensité du signal relatif. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
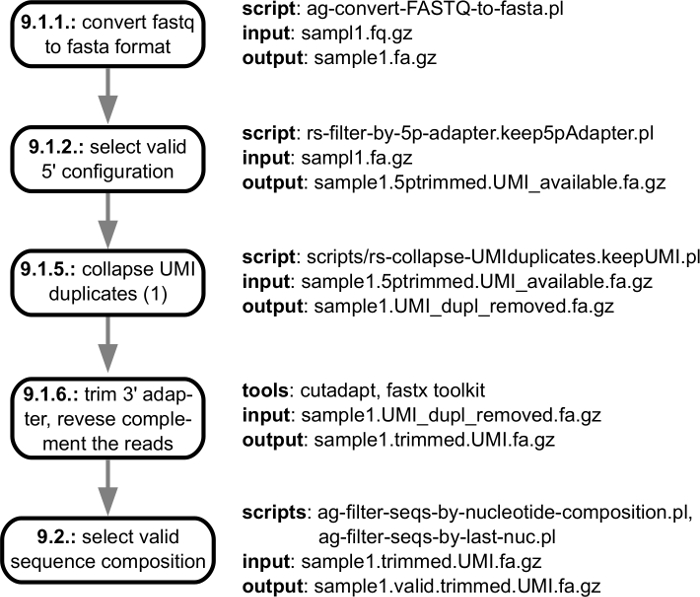
Figure 3 : contour de pré-traitement du séquençage lectures. Les fichiers fastq avec des lectures qui sont générés par le logiciel de séquençage instrument-associés sont traités afin d’identifier des lectures de qualité qui seront mappés au génome correspondant. La figure illustre la spécification d’entrée/sortie des étapes individuelles dans le pipeline, avec des liens vers les différentes étapes du protocole décrit dans la section « Informatique ». S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
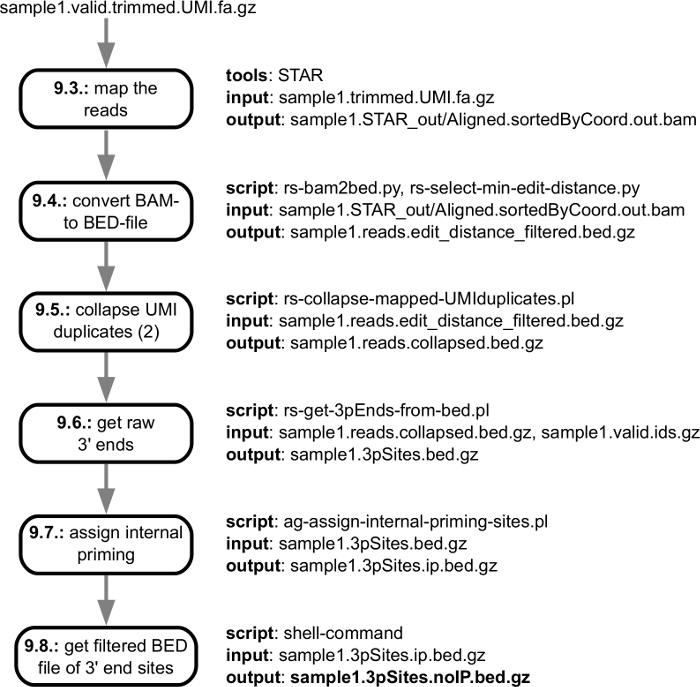
Figure 4 : schéma de séquence lire traitement, de l’étape de la cartographie au génome de la génération de l’extrémité 3' des sites de traitement. La figure illustre la spécification d’entrée/sortie des étapes individuelles dans le pipeline, avec des liens vers le iindividuel des mesures du protocole décrit dans la section « Informatique ». Le fichier de sortie principal qui est remis à l’utilisateur est marqué en gras. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
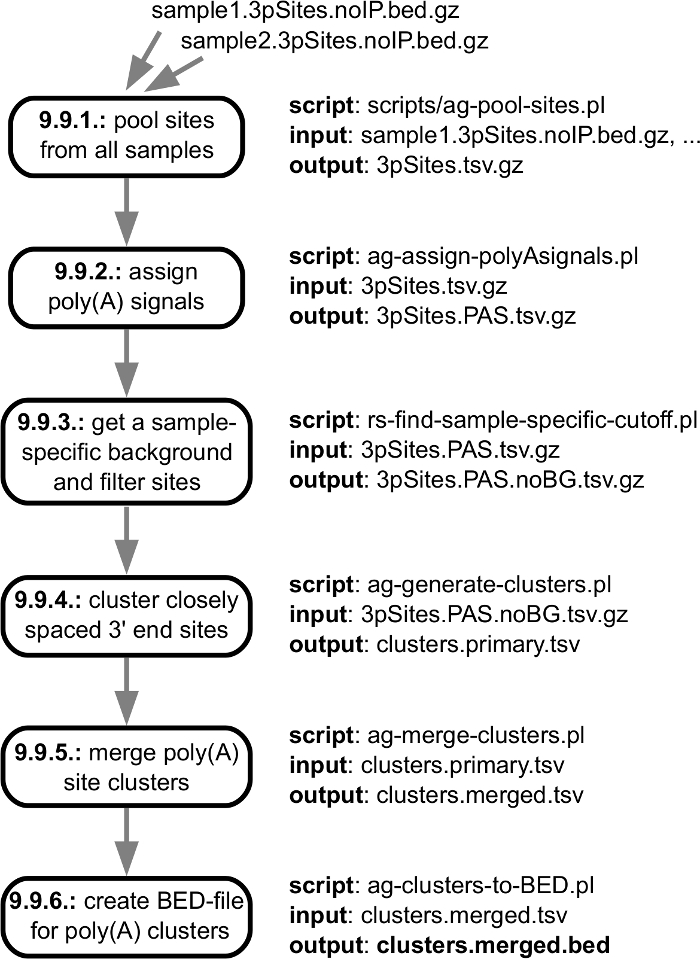
Figure 5 : aperçu des mesures qui sont prises pour produire des grappes de co réglementé 3' fin séquençage sites. La figure illustre la spécification d’entrée/sortie des étapes individuelles dans le pipeline, avec des liens vers les différentes étapes du protocole décrit dans la section « Informatique ». Le fichier de sortie principal est marqué en gras. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
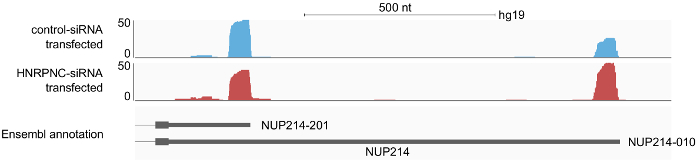
Figure 6 : résultats d’exemple du profil de 3' finissent traitement lectures le long du terminal exon du gène NUP214, affiché dans l’Explorateur de génome IGV 16 . A-seq2 lectures ont été préparés à partir de deux échantillons de cellules HEK 293, traitées avec un siARN contrôle- ou avec un siARN HNRNPC. Le lit que documenté poly (a) les sites qui ont été annotés par le pipeline de l’analyse ont été enregistrés au format BAM qui a été utilisé comme entrée dans le navigateur de génome IGV. L’extrémité 3' des pics lecture carte d’ARNm 3' extrémités qui sont annotées dans Ensembl. Les profils montrent une utilisation accrue de l’isoforme longue de 3' UTR sur HNRNPC précipitation. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
| TR-contrôle répliquer 1 | TR-contrôle répliquer 2 | |
| ID : 29765 | ID : 32682 | |
| nombre de lectures brutes | 44210258 | 68570640 |
| nombre de lectures valides après parage et filtrage | 14024538 | 21211793 |
| nombre de cartographie unique lectures | 6953674 | 13946436 |
| nombre de lectures mappage à plusieurs loci | 2040646 | 2925839 |
| nombre d’extrémité individuel 3' sites de traitement | 1107493 | 1710353 |
Tableau 1 : exemple de sortie du pipeline analyse. Synthèses de lectures qui ont été obtenus à différentes étapes.
Discussion
La multitude de base et des facteurs auxiliaires qui sont impliqués dans le traitement de l’ARN pré-messager 3' extrémité se reflète dans un paysage de polyadénylation proportionnellement complexes. En outre, polyadénylation est également sensible aux changements dans d’autres processus tels que la transcription et épissage. sites de clivage d’extrémité 3' en pré-ARNm sont généralement identifiés basé sur la queue poly (a) typiques qui est ajoutée aux produits de clivage 5'. La plupart des méthodes utilisent des amorces oligo (décollement) de longueurs variables qui permettent la conversion spécifique de poly (A)-contenant des ARNm d’ADNc dans une réaction de transcription inverse. Un problème commun de cette approche est l’amorçage interne aux séquences riches en A aboutissant à des sites de clivage artifactual. Deux méthodes qui visent à contourner cet artefact au stade de la préparation de l’échantillon ont été proposées. Dans la méthode 3P-seq 1, adaptateurs sont spécifiquement ligaturés jusqu’aux extrémités de la queue poly (a) avec l’aide d’un attelle oligo suivi d’une digestion partielle RNase T1 et transcriptase inverse avec TTP dans la réaction comme les désoxyribonucléosides seul. L’hétéroduplex poly(A)-poly(dT) qui en résultent est ensuite digérés avec RNase H et les autres fragments d’ARN sont isolés, ligaturés aux adaptateurs et séquencés. Une méthode plus simple et élégante, 2p-seq, qui utilise un apprêt de séquençage personnalisé sauter le tronçon restant d’oligo (décollement) dans la réaction de séquençage a été rapporté par les mêmes auteurs 2. Dans une méthode associée, 3' lit 3, un apprêt inhabituellement long de 5 45 et nous Ts, contenant également un biotine sont recuits à l’ARN fragmenté, suivie par des lavages strictes pour sélectionner des molécules d’ARN avec queue poly (a) de plus de 50 nucléotides. Bien que 3' lectures réduit considérablement la fréquence de l’amorçage interne, il n’élimine pas complètement il 3. Protocoles pour le séquençage direct de RNA ont également été proposées, mais les lectures qui en résultent sont courtes et ont un taux élevé d’erreur et cette approche n’a pas été davantage développés 18,19,20. La PolyA-Seq et les protocoles Quant Seq commercialisés combinent oligo (décollement) fondé d’amorçage avec une étape d’amorçage aléatoire pour le deuxième brin synthèse de cDNA 20. L’utilisation de la réaction de transcription inverse de commutateur de modèle avec la transcriptase inverse du Virus de la leucémie Murine Moloney (MMLV) conduit à la génération d’ADNc avec linkers en une seule étape et ainsi aucune dimères d’adaptateur ne peuvent apparaître dans le PAS-Seq et méthodes SAPAS 21 , 22.
La méthode A-seq2 présentée ici se distingue par son utilisation d’un nucléotide CLIVABLES (dU) au sein d’une couche d’apprêt biotinylé oligo (décollement). Cette modification combine l’utilité d’enrichir les oligo (décollement) hybridé, polyadénylé cibles avec le retrait de la majeure partie de la séquence de25 oligo (dT) des fragments isolés avant que les bibliothèques sont préparés et la préservation des trois Ts, qui indiquer la présence antérieure de la queue poly (a). En revanche, les méthodes qui utilisent la RNase H pour retirer poly (a) les molécules d’ARN au hasard laissent plusieurs comme. Étant donné que dans A-seq2, séquençage est fait à partir de l’extrémité 3' des brins anti-sens, sites de clivage sont prévus pour être situé après le motif NNNNTTT au début de la séquence brute se lit. Les tétramères aléatoires servent non seulement à permettre à base appelant mais aussi dans l’élimination d’artefacts d’amplification PCR. UMIs plus longues peuvent être hébergés. La possibilité d’amorçage interne reste en A-seq2 et est adressée par le calcul, tout d’abord, en jetant 3' se termine par une séquence en aval génomiquement codé, riches en A et ensuite en jetant des grappes d’extrémité 3' qui pourraient s’expliquer par l’amorçage interne à la Signal de poly (a) riches en A lui-même. Une récente analyse des sites de poly (a) déduit de manière unique par un grand nombre de protocoles indique que les sites qui sont propres à A-seq2 ont la distribution attendue nucléotides et l’emplacement dans les gènes, semblables aux autres 3' fin des protocoles de séquençage.
Une étape cruciale dans l’A-seq2 est la sélection de polyadénylé RNA et la suppression des ARN ribosomiques et divers petits ARN. C’est plus facilement réalisable par un kit d’isolation des ARNm avec oligo (dT)25 billes magnétiques. En principe, les ARN total isolé avec du phénol contenant des solutions aussi donne ARN qui peut être encore soumis à la sélection par le kit d’ARNm-isolation ou d’agarose oligo (dT) de haute qualité. Une étape qui peut être variée en A-seq2 est le traitement par hydrolyse alcaline qui peut être raccourcie ou prolongée pour obtenir des fragments d’ARN de différentes tailles. Critique est aussi que l’ajout de dATP 3' à 3' extrémités des fragments d’ARN par la poly (a) polymérase est efficace. Dans le protocole décrit ici, ce traitement est appliqué à tous les fragments d’ARN, d’éviter les concatemerization au cours de la réaction de ligature. Enfin, nous notons que, bien que ligase RNA 1 est normalement utilisé comme une ligase RNA, il ligates aussi efficacement seul ADN bicaténaire, comme nous l’avons fait ici pour ligaturer un adaptateur à l’extrémité 5' des molécules de cDNA.
A-seq2 est donc un efficace et facile à mettre en œuvre le protocole d’identification des sites de traitement l’extrémité 3' pré-ARNm. Les développements futurs pourraient inclure réduisant davantage la complexité du protocole et de la quantité de matériel requis. Jeu d’outils d’analyse des données informatiques davantage associé permettent le traitement homogène de l’extrémité 3' séquençage lectures obtenues avec un large éventail de protocoles.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Les auteurs remercient Mme Béatrice Dimitriades pour aide à la culture cellulaire. Ce travail a été soutenu par la Swiss National Science Foundation subventions #31003A_170216 et 51NF40_141735 (NCCR RNA & maladie).
matériels
| Name | Company | Catalog Number | Comments |
| Materials | |||
| Agarose, ultra pure | Invitrogen | 16500-500 | |
| 2100 Bioanalyzer | Agilent | G2940CA | |
| Cordycepin triphosphate (3’ dATP) | SIGMA | C9137 | |
| DNA low bind vials, 1.5 ml | Eppendorf | 22431021 | |
| Dulbecco’s Phosphate Buffered Saline | SIGMA | D8637 | |
| Dynabeads mRNA-DIRECT Kit | Ambion | AM61012 | |
| GR-Green dye | Excellgen | EG-1071 | use 1:10,000 dillution |
| HiSeq 2500 or NextSeq 500 next generation sequencers | Illumina | inquire with supplier | |
| KAPA HiFi Hotstart DNA polymerase mix | KAPA/Roche | KK2602 | |
| Nuclease free water | Ambion | AM9937 | |
| Poly(A) polymerase, yeast | Thermo Fisher Scientific | 74225Z25KU | |
| Poly(A) polymerase, E.coli | New England Biolabs | M0276L | |
| Polynucleotide kinase | Thermo Fisher Scientific | EK0032 | |
| QIAEX II Gel Extraction Kit | Qiagen | 20021 | |
| QIAquick PCR Purification Kit | Qiagen | 28104 | |
| QIAquick Gel Extraction Kit | Qiagen | 28704 | |
| RNA ligase 1, high concentration | New England Biolabs | M0437M | includes PEG-8000 |
| RNeasy MinElute RNA Cleanup kit | Qiagen | 74204 | |
| RNase H | New England Biolabs | M0279 | |
| RNasin Plus, ribonuclease inhibitor | Promega | N2618 | |
| Superscript IV reverse transcriptase | Thermo Fisher Scientiific | 18090050 | |
| Turbo DNase | Ambion | AM2238 | |
| USER enzyme mix | New England Biolabs | M5505 | |
| Dyna-Mag-2 magnetic rack | Thermo Fisher Scientific | 12321D | |
| Thermomixer C | Eppendorf | 5382000015 | Heated mixer with heated lid |
| MicroSpin columns | GE-Healthcare | 27-5325-01 | |
| Name | Company | Catalog Number | Comments |
| Buffers | |||
| Alkaline hydrolysis buffer, 1.5 x | Mix 1 part 0.1 M Na2CO3 and 9 parts 0.1 M NaHCO3. Add EDTA to 1 mM. Adjust pH to 9.2. Store aliquots at -20 °C. | ||
| 5x poly(A) polymerase buffer | Thermo Fisher Scientiific | 100 mM Tris-HCl, pH 7.0, 3 mM MnCl2, 0.1 mM EDTA, 1 mM DTT, 0.5 mg/ml acetylated BSA, 50% glycerol | |
| Biotin binding buffer | 20 mM TrisCl pH 7.5, 2 M NaCl, 0.1% NP40 | ||
| TEN buffer | 10 mM TrisCl, pH 7.5, 1 mM EDTA, 0.02% NP40 | ||
| Name | Company | Catalog Number | Sequence |
| Oligonucleotides according to Illumina TruSeq Small RNA Sample Prep Kits, for GA-IIx and Hiseq2000/2500 sequencers | Microsynth | ||
| revRA3 (RNA) | Microsynth | 5’ amino CCUUGGCACCCGAGAAUUCCA 3’ | |
| revDA5 | Microsynth | 5’ amino GTTCAGAGTTCTACAGTCCGAC GATCNNNN-3’ | |
| Bio-dU-dT25, RT primer | Microsynth | 5' Biotin-TTTTTTTTTTTTTTTTTTTTTTTTTT-dU-TTTVN 3' (V = G, A or C) | |
| PCR primer forward, RP1 | Microsynth | 5' AATGATACGGCGACCACCGAGA TCTACACGTTCAGAGTTCTACAG TCCGA 3' | |
| PCR primer reverse, RPI1, barcode in bold | Microsynth | 5' CAAGCAGAAGACGGCATACGAG ATCGTGATGTGACTGGAGTTCCT TGGCACCCGAGAATTCCA 3' | |
| Name | Company | Catalog Number | Comments |
| Oligonucleotides according to Illumina TruSeq HT-Small RNA Sample Prep Kits, for HiSeq2000/2500 and NextSeq500 sequencers | |||
| HT-rev3A (DNA/RNA) | Microsynth | 5'-amino-GTGACTGGAGTTCAGACGTGTG CTCTTCCrGrAUrC-3' | |
| HT-rev5A | Microsynth | 5' amino-ACACTCTTTCCCTACACGACGCT CTTCCGATCTNNNN 3' | |
| Bio-dU-dT25, RT primer | Microsynth | 5' Biotin-TTTTTTTTTTTTTTTTTTTTTTTTTT-dU-TTTVN 3' | |
| PCR primers forward (D501-506) | Microsynth or Illumina | 5'-AATGATACGGCGACCACCGAGAT CTACAC[i5]ACACTCTTTCCCTACA CGACGCTCTTCCGATCT -3' | |
| PCR primers reverse (D701-D712) | Microsynth or Illumina | 5'-CAAGCAGAAGACGGCATACGAG A[i7]GTGACTGGAGTTCAGACGTG TGCTCTTCCGATC-3' | |
| Documentation for Illumina multiplexing: | Illumina | https://support.illumina.com/content/dam/illumina-support/documents/documentation/chemistry_documentation/experiment-design/illumina-adapter-sequences_1000000002694-01.pdf |
Références
- Jan, C. H., Friedman, R. C., Ruby, J. G., Bartel, D. P. Formation, regulation and evolution of Caenorhabditis elegans 3'UTRs. Nature. 469 (7328), 97-101 (2011).
- Spies, N., Burge, C. B., Bartel, D. P. 3' UTR-isoform choice has limited influence on the stability and translational efficiency of most mRNAs in mouse fibroblasts. Genome Res. 23 (12), 2078-2090 (2013).
- Hoque, M., Ji, Z., et al. Analysis of alternative cleavage and polyadenylation by 3' region extraction and deep sequencing. Nat. methods. 10 (2), 133-139 (2013).
- Martin, G., Gruber, A. R., Keller, W., Zavolan, M. Genome-wide analysis of pre-mRNA 3’ end processing reveals a decisive role of human cleavage factor I in the regulation of 3' UTR length. Cell Rep. 1 (6), 753-763 (2012).
- Gruber, A. R., Martin, G., et al. Global 3' UTR shortening has a limited effect on protein abundance in proliferating T cells. Nat. Commun. 5, 5465 (2014).
- Kivioja, T., Vähärautio, A., et al. Counting absolute numbers of molecules using unique molecular identifiers. Nat. methods. 9 (1), 72-74 (2011).
- Gruber, A. J., Schmidt, R., et al. A comprehensive analysis of 3' end sequencing data sets reveals novel polyadenylation signals and the repressive role of heterogeneous ribonucleoprotein C on cleavage and polyadenylation. Genome Res. 26 (8), 1145-1159 (2016).
- Lingner, J., Keller, W. 3'-end labeling of RNA with recombinant yeast poly(A) polymerase. Nucleic Acids Res. 21 (12), 2917-2920 (1993).
- Quail, M. A., Kozarewa, I., et al. A large genome center's improvements to the Illumina sequencing system. Nat. methods. 5 (12), 1005-1010 (2008).
- Rahmann, S. Snakemake--a scalable bioinformatics workflow engine. Bioinformatics. 28 (19), 2520-2522 (2012).
- . Anaconda Software Distribution Available from: https://continuum.io (2016)
- Dobin, A., Davis, C. A., et al. STAR: ultrafast universal RNA-seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- Martin, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet.journal. 17 (1), 10-12 (2011).
- Li, H., Handsaker, B., et al. The Sequence Alignment/Map format and SAMtools. Bioinformatics. 25 (16), 2078-2079 (2009).
- Robinson, J. T., Thorvaldsdóttir, H., et al. Integrative genomics viewer. Nat. Biotechnol. 29 (1), 24-26 (2011).
- Quinlan, A. R., Hall, I. M. BEDTools: a flexible suite of utilities for comparing genomic features. Bioinformatics. 26 (6), 841-842 (2010).
- Ozsolak, F., Platt, A. R., et al. Direct RNA sequencing. Nature. 461 (7265), 814-818 (2009).
- Yao, C., Biesinger, J., et al. Transcriptome-wide analyses of CstF64-RNA interactions in global regulation of mRNA alternative polyadenylation. Proc. Natl. Acad. Sci. U. S. A. 109 (46), 18773-18778 (2012).
- Lin, Y., Li, Z., et al. An in-depth map of polyadenylation sites in cancer. Nucleic Acids Res. 40 (17), 8460-8471 (2012).
- Shepard, P. J., Choi, E. -. A., Lu, J., Flanagan, L. A., Hertel, K. J., Shi, Y. Complex and dynamic landscape of RNA polyadenylation revealed by PAS-Seq. RNA. 17 (4), 761-772 (2011).
- Fu, Y., Sun, Y., et al. Differential genome-wide profiling of tandem 3' UTRs among human breast cancer and normal cells by high-throughput sequencing. Genome Res. 21 (5), 741-747 (2011).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon