A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
تحديد كفاءة المثبط الكيميائي ضد نمو توكسوبلازما غوندي داخل الخلايا باستخدام قياس النمو القائم على لوسيفيراز
In This Article
Summary
عرض هنا هو بروتوكول لتقييم فعالية تثبيط المركبات الكيميائية ضد في المختبر النمو داخل الخلايا من غوندي توكسوبلازما باستخدام اسيه النمو القائم على لوسيفيراز. وتستخدم هذه التقنية لتأكيد التحديد تثبيط عن طريق الحذف الوراثي للجين المستهدف المقابلة. يتم تقييم تثبيط LHVS ضد بروتياز TgCPL كمثال.
Abstract
توكسوبلازما غوندي هو مسببات الأمراض الأولية التي تؤثر على نطاق واسع في البشر. المضادات الحيوية الحالية المستخدمة لعلاج التهاب التوكسوبلازمات السريرية محدودة. وبالإضافة إلى ذلك، فإنها تظهر آثار جانبية سلبية في مجموعات معينة من الناس. لذلك ، فإن اكتشاف علاجات جديدة لـ toxoplasmosis السريري أمر حتمي. الخطوة الأولى من تطوير المضادات الحيوية الجديدة هي تحديد المركبات الكيميائية التي تظهر فعالية عالية في تثبيط نمو الطفيليات باستخدام استراتيجية فحص الإنتاجية العالية. وباعتبارها ممرضاً ملزماً داخل الخلايا، لا يمكن لتوكسوبلازما أن تتكرر إلا داخل الخلايا المضيفة، مما يحظر استخدام قياسات الامتصاص البصري كمؤشر سريع للنمو. عرض هنا هو بروتوكول مفصل لسيفراز القائم على تقييم النمو. على سبيل المثال ، يتم استخدام هذه الطريقة لحساب وقت مضاعفة طفيليات توكسوبلازما البرية وقياس فعالية مورفولاينيوريا - لوسيل - homophenyl - الفينيل السلفون الفينيل (LHVS ، وهو مركب استهداف البروتياز السيستين) فيما يتعلق بتثبيط نمو الطفيليات داخل الخلايا. كما وصفت، هو بروتوكول حذف الجينات CRISPR-Cas9 المستندة في توكسوبلازما باستخدام 50 منطقة متجانسة bp لإعادة تركيب القائم على homology (HDR). من خلال تحديد فعالية تثبيط LHVS في البرية من نوع وTgCPL (توكسوبلازما cathepsin L مثل البروتياز) الطفيليات ناقصة, وتبين أن LHVS يمنع نمو الطفيليات البرية من نوع أكثر كفاءة من نمو الـ Οcpl, مما يشير إلى أن TgCPL هو الهدف الذي يربط LHVS إلى في توكسوبلازما. حساسية عالية وسهولة التشغيل من هذا السهو على أساس التسمم النمو المنقّعة جعلها مناسبة لرصد انتشار توكسوبلازما وتقييم فعالية الدواء بطريقة إنتاجية عالية.
Introduction
Toxoplasma غوندي هو ناجح للغاية ملزمة الطفيلي داخل الخلايا التي تصيب ما يقرب من ثلث السكان البشرية. ويعزى ارتفاع معدل انتقال العدوى في الغالب إلى طرق انتقاله المتنوعة، بما في ذلك استهلاك اللحوم غير المطبوخة جيدا، والتعرض لخزانات الثدييات، وانتقال العدوى الخلقية أثناء الولادة. T. gondii يسبب أساسا العدوى الانتهازية التي يمكن أن تؤدي إلى اعتلال شديد والوفيات في الأفراد المنقوصةالمناعي1,,2,,3,,4,,5,,6. المضادات الحيوية المستخدمة حاليا لعلاج داء التوكسوبلازمات الحادة غير فعالة بشكل خاص في علاج العدوى الخلقية والكامنة وتسبب ردود فعل شديدة في بعض الأفراد3,7,8. وبالتالي ، هناك حاجة ملحة لتحديد العلاجات الجديدة. فهم الاختلافات في العمليات تحت الخلية داخل توكسوبلازما ومضيفها سوف يساعد على تحديد الأهداف المحتملة للأدوية. لذلك ، هناك حاجة إلى تقنيات فعالة ومريحة للتلاعب بالجينوم لدراسة أدوار الجينات الفردية داخل توكسوبلازما. بالإضافة إلى ذلك، Toxoplasma ينتمي إلى Apicomplexa phylum، الذي يتضمن العديد من مسببات الأمراض البشرية الهامة الأخرى، مثل بلازموديوم spp. وCryptosporidium spp. وبالتالي ، يمكن استخدام Toxoplasma ككائن حي نموذجي للمساعدة في دراسة البيولوجيا الأساسية في الطفيليات المعقدة الأخرى.
لتحديد المضادات الحيوية الجديدة ضد مسببات الأمراض الميكروبية ، يتم إجراء فحص عالي الإنتاجية لمكتبة من المركبات الكيميائية في البداية لتحديد فعاليتها في قمع النمو الميكروبي. حتى الآن وقد وضعت العديد من المقاييس النمو على أساس microplate لقياس النمو داخل الخلايا من T. gondii (أي المشعة 3H-uracil أساس القياس الكمي9، الكشف عن الطفيليات المستندة إلى ELISA الكمية باستخدام T. gondii-الأجسام المضادة محددة10،1114، والقياس القائم على البروتين مراسل باستخدام α-galactosidase أو YFP التعبير عن سلالات Toxoplasma 12،13، وطور مؤخرا عالية المحتوى
ولهذه الاستراتيجيات الفردية مزايا فريدة من نوعها؛ ومع ذلك، بعض القيود أيضاً تقييد تطبيقاتها. على سبيل المثال، بما أن توكسوبلازما لا يمكن أن تتكرر إلا داخل الخلايا الحيوانية النواة، فإن الفلور الفلورية والملزمة غير المحددة للأجسام المضادة المضادة لـT. gondii لاستضافة الخلايا تسبب تداخلًا في القياسات القائمة على الفلورسينس. وعلاوة على ذلك، يتطلب استخدام النظائر المشعة امتثالا خاصا للسلامة ومسائل محتملة تتعلق بالسلامة. بعض هذه الفرضيات هي أكثر ملاءمة لتقييم النمو في نقطة زمنية واحدة بدلا من الرصد المستمر للنمو.
عرض هنا هو بروتوكول على أساس luciferase للقياس الكمي لنمو توكسوبلازما داخل الخلايا. في دراسة سابقة، تم استنساخ جين NanoLuc luciferase تحت مروج توبولولين توكسوبلازما، وهذا البناء التعبير luciferase تم نقلها إلى نوع البرية (RHΟku80ku8سلالةhxg) الطفيليات لإنشاء RHАаku80οhxg:: سلالةNLuc (يشار إليها باسم RHΟku80::NLuc فيما يلي)15. هذه السلالة بمثابة سلالة الوالدين لتحديد النمو داخل الخلايا وحذف الجينات في هذه الدراسة. باستخدام RHΟku80:: سلالةNLuc ، تم رصد نمو الطفيليات في الخلايا الليفية القلفة البشرية (HFFs) على مدى فترة 96 ساعة بعد العدوى لحساب وقت مضاعفة الطفيليات.
بالإضافة إلى ذلك ، يمكن تحديد فعالية تثبيط LHVS ضد نمو الطفيليات عن طريق رسم معدلات نمو توكسوبلازما مقابل تركيزات LHVS التسلسلية لتحديد قيمة IC50. وقد ذكرت الأدبيات السابقة أن TgCPL هو هدف رئيسي من LHVS في الطفيليات وأن العلاج مع LHVS يقلل من تطور التهابات توكسوبلازما الحادة والمزمنة16,17,18,19. بالإضافة إلى ذلك، تم استخدام RHΟku80::NLuc كسلالة أبوية لتعديل الجينوم لتوليد سلالة ناقصة TgCPL(RHΟku80cpl::NLuc)،وتم قياس تثبيط LHVS مقابل هذا المتحول. من خلال مراقبة تحول أعلى من قيم IC50 لLHVS في TgCPL-الطفيليات الناقصة مقارنة بسلالة WT ، تم التحقق من أن TgCPL مستهدف من قبل LHVS في الجسم الحي.
في هذا البروتوكول، RHΟku80:: يستخدمNLuc كسلالة الوالدين، والتي تفتقر إلى كفاءة غير متجانسة نهاية الانضمام المسار (NHEJ)، وبالتالي تسهيل المزدوج كروس homology تعتمد على إعادة الجمع (HDR)20،21. بالإضافة إلى ذلك، 50 منطقة متجانسة bp محاطة في كلا طرفي كاسيت مقاومة الأدوية بواسطة PCR. يعمل منتج PCR كقالب إصلاح لإزالة موضع الجينات بأكملها عبر HDR باستخدام أدوات تحرير الجينوم المستندة إلى CRISPR-Cas9. ويمكن دمج هذه المناطق القصيرة المتجانسة بسهولة في الالتمهيديات، مما يوفر استراتيجية مريحة لإنتاج قالب الإصلاح. يمكن تعديل هذا البروتوكول لتنفيذ حذف الجينات العالمية ووضع علامات الجينات الذاتية.
على سبيل المثال ، في أحدث منشور لدينا ، تم تغريب ثلاثة جينات بروتياز ، TgCPL، TgCPB (Toxoplasma cathepsin B-like protease) ، وTgSUB1 (Toxoplasma subtilisin like protease 1) ، تم تغريبها وراثيًا في TgCRT (Toxoplasma chloroquine-resistance الناقل) الطفيليات الناقصة باستخدام هذه الطريقة15. بالإضافة إلى ذلك، TgAMN (a الأمينية المفترضة N [TgAMN, TGGT1_221310]) كان الموسومة داخليا15. وأفاد مختبر لورديو أيضا باستخدام مناطق متجانسة قصيرة في حدود 40-43 نقطة با لإدخال طفرة جينية موجهة من الموقع ووضع علامات الجينات الذاتية في الجينوم توكسوبلازما باستخدام طريقة مماثلة22. تشير هذه التعديلات الناجحة في الجينوم إلى أن منطقة متجانسة 40-50 bp كافية لإعادة تركيب الحمض النووي بكفاءة في سلالة TgKU80-ناقصة ، والتي تبسط إلى حد كبير التلاعب الجينوم في Gondii Toxoplasma.
Protocol
يتم تصنيف توكسوبلازما غوندي في مجموعة المخاطر 2 ويجب التعامل معها على مستوى السلامة البيولوجية 2 (BSL-2). وقد استعرضت اللجنة المؤسسية للسلامة البيولوجية في جامعة كليمسون البروتوكول ووافقت عليه.
1. لوسيفيراز القائم على Toxoplasma النمو القول
- الخلايا الليفية القلفة البشرية البذور (HFFs) 1 أسبوع قبل تلقيح الطفيليات لضمان أن الخلايا المضيفة هي confluent تماما. إجراء عملية تقييم وهمية في لوحة شفافة لضمان بقاء الطفيليات داخل الخلايا طوال فترة التقييم.
ملاحظة: هنا، يتم إجراء عملية التّسّازي في 96 لوحة صغيرة. وفقا للاحتياجات التجريبية، يمكن توسيع نطاقها تصل إلى 384 أو 1536 ميكروسيل جيدا. - تمرير طفيليات توكسوبلازما إلى HFFs ملتوية قبل يومين من الاستخدام عن طريق نقل ~ 0.3-0.4 مل من الطفيليات الملحالة بالكامل إلى قارورة T25. الخلايا المضيفة المصابة في 37 درجة مئوية مع 5٪ CO2 لمدة يومين.
- حقنة 5 مل من الطفيليات الطازجة من خلال إبرة السلامة 21 G 5x لتحرير الطفيليات داخل الخلايا ، ثم تمر عبر فلتر 3 ميكرومتر لإزالة حطام الخلايا المضيفة. شطف الطفيليات المتبقية من قارورة باستخدام 7 مل من متوسط D10 الخالية من الفينول الأحمر، ثم تمر من خلال مرشح مرة أخرى.
- طفيليات الطرد المركزي عند 1000 × غرام لمدة 10 دقيقة في درجة حرارة الغرفة (RT). صب قبالة supernatant وإعادة تعليق بيليه في 10 مل من الفينول الأحمر خالية من D10 وسائل الإعلام.
- عد الطفيليات باستخدام مقياس الهيموكيتومتر لتحديد التركيز.
- تمييع الطفيليات إلى 1 × 104 طفيليات / مل لسلالة البرية من النوع (WT). بالنسبة لسلالات الطفيليات التي تعاني من نقص النمو ، قم بزيادة التركيز وفقًا لذلك لمراقبة زيادة كبيرة في إشارات لوسيفيراز.
- يُعصر وسائل الإعلام بعناية من 96 لوحة صغيرة جيدة مبذرة مسبقًا بHFFs وتطعيم 150 ميكرولتر من إعادة تعليق الطفيليات إلى آبار في شكل ثلاثة أعمدة وخمسة صفوف ، وهو ما يمثل ثلاث نسخ فنية وخمس نقاط زمنية.
- احتضان microplate في 37 درجة مئوية و 5٪ CO2 لمدة 4 ساعة.
- استنشق الوسائط بعناية من الآبار لإزالة الطفيليات غير الغازية ، ثم قم بملء الآبار بوسائط RT phenol الخالية من اللون الأحمر في كل صف (باستثناء الصف الأول).
- مزيج أحجام متساوية من PBS و2x luciferase عازلة ويخفف من الركيزة لوسيفيراز إلى 12.5 ميكرومتر.
- إضافة 100 ميكرولتر من الركيزة لوسيفراز المخفف في كل بئر من الصف العلوي. احتضان microplates في RT لمدة 10 دقيقة للسماح للخلايا لlyse تماما.
- قياس نشاط luciferase باستخدام قارئ microplate. يتم سرد إعدادات قارئ اللوحة في الجدول 1. تمثل كل قراءة العدد الأولي للطفيليات الغازية في 4 ساعة بعد العدوى.
- كرر الخطوات 1.9-1.12 لكل صف كل 24 ساعة لمدة 4 أيام دون تغيير الوسط. تعكس هذه القراءات العدد الإجمالي للطفيليات المنسوخة في 24 ساعة و48 ساعة و72 ساعة و96 ساعة بعد العدوى.
- حساب متوسط القراءات في كل نقطة زمنية وتقسيمها على متوسط القراءات في 4 ساعة لتحديد التغيرات أضعاف في نمو الطفيليات مع مرور الوقت.
- رسم البيانات باستخدام برنامج الرسوم البيانية. وترد جدول قراءة النمو التمثيلي والمؤامرات من RHΟku80:: الطفيلياتNLuc في الشكل 1A, B.
- لحساب مضاعفة الوقت، رسم قيم log2 من التغييرات أضعاف في النقاط الزمنية الفردية خلال وقت الحضانة. استخدم دالة انحدار خطي لحساب الميل، والتي تمثل وقت مضاعفة كل سلالة(الشكل 1A، C).
2. تقييم فعالية تثبيط المركبات الكيميائية ضد نمو توكسوبلازما
ملاحظة: هنا، يتم تقديم تقييم تثبيط LHVS في نمو توكسوبلازما كمثال. يتم اختبار ثمانية تركيزات مختلفة من LHVS، ويتم تنفيذ ثلاثة يكرر التقنية لكل من النسخ البيولوجية الثلاث لكل منRHΟku80::NLuc و RHА80Cpl:: سلالاتNLuc. ku80
- قبل الإصابة بالطفيليات ، وHFFs البذور إلى 96 ميكروصف جيدا في شكل ثلاثة صفوف وتسعة أعمدة لتكرار البيولوجية واحدة لكل مركب لكل سلالة. سيسمح للخلايا المضيفة بالنمو لمدة 7 أيام على الأقل قبل الاستخدام.
- تمرير RHΟku80::NLuc و RHΟku80CPL:: طفيلياتNLuc لمدة يومين قبل الاستخدام. اتبع الخطوات 1.2-1.6 لتنقية الطفيليات وقياسها الكمي. إعادة تعليق الطفيليات في الوسائط الخالية من الفينول الحمراء عند 1 × 104 طفيليات /مل.
- استنشق الوسائط من لوحة تحتوي على HFFs ملتوية وتطعيم كل بشكل جيد مع 150 ميكرولتر من إعادة تعليق الطفيليات. احتضان microplate في 37 درجة مئوية و 5٪ CO2 لمدة 4 ساعة.
- إعداد LHVS في ثمانية تركيزات مختلفة في خزان بئر 12 عن طريق التخفيف المتسلسل. عموما، يتم تقليل التركيزات بمقدار ثلاثة أضعاف بطريقة تخفيف المسلسل.
ملاحظة: يتم تقليل أدنى تركيز بمقدار 6,561 أضعاف بالنسبة إلى أعلى تركيز. يمكن تعديل تغيير أضعاف التخفيف وفقًا لذلك استنادًا إلى خصائص مختلفة للمركبات الفردية. - في 4 ساعة بعد العدوى، يستنشق وسائل الإعلام لإزالة الطفيليات غير الغازية وملء كل جيدا من الأعمدة 2-9 مع 150 ميكرولتر من وسائل الإعلام المكملة مع LHVS بتركيزات مختلفة. اترك العمود الأول مملوءًا بالوسائط العادية ليكون بمثابة عنصر تحكم غير معالج.
- احتضان microplate في 37 درجة مئوية و 5٪ CO2 ل96 ساعة إضافية.
- تنفيذ الخطوات 1.9-1.11 وقياس نشاط luciferase من الآبار الفردية.
- متوسط أنشطة لوسيفيراز من ثلاثة يكرر التقنية من الآبار من كل تركيز LHVS الفردية.
- تقسيم متوسط نشاط لوسيفيراز لكل تركيز LHVS على متوسط نشاط لوسيفراز المشتق من الطفيليات غير المعالجة لحساب نشاط لوسيفيراز العادي كنسبة مئوية.
- مؤامرة الأنشطة luciferase تطبيع ضد تركيزات LHVS الفردية باستخدام برامج الرسوم البيانية(الشكل 2). كما يتم قياس تثبيط البيريميثامين ضد نمو الطفيليات كعنصر تحكم. البيريميثامين هو مضاد حيوي سريري يستخدم لعلاج التوكسوبلازموس الحاد عن طريق تثبيط استقلاب حمض الفوليك في توكسوبلازما.
- حساب قيم IC50 للمركبات الفردية باستخدام الأسلوب المضمن في برنامج الرسوم البيانية ، والاستجابة العادية مقابل [مثبط] ، في إطار برنامج الانحدار "جرعة الاستجابة - تثبيط". يتم حساب IC50 باستخدام الصيغة التالية:
Y = 100/(1 + X/IC50)
حيث: Y يمثل أنشطة اللوسيفراز العادية للخلايا المصابة تحت تركيزات مختلفة من المثبط، وX يمثل تركيزات فردية من المثبط.
3- الحذف الجيني المستند إلى CRISPR-Cas9 في طفيليات توكسوبلازما
- جيل من بناء plasmid التعبير عن دليل الجيش الملكي النيبالي (sgRNA) وCas9 لحذف جين من الفائدة
- انتقل إلى www.ToxoDB.org واسترداد تسلسل ترميز الجينات بالكامل، بما في ذلك الإنترونات والمواد الطاردة، إلى جانب 1.5 كيلوبايت 5'-UTRs و3'-UTRs (المناطق غير المترجمة).
ملاحظة: هنا، يتم استهداف TgCPL (TGGT1_321530) كمثال تمثيلي. - نسخ تسلسل TgCPL المستردة في برنامج تحليل التسلسل (راجع جدول المواد للاسم والإصدار) وتسمية المناطق 5 '- و 3'-UTR.
- حدد رمز الأدوات في شريط القائمة العلوي، ثم حدد الاستنساخ | العثور على مواقع CRISPR.
- اختر 3'(Cas9)' لموقع موقع PAM وحدد المجلد الذي يحتوي على تسلسل جينوم توكسوبلازما في قسم تسجيل التحديد. اترك بقية الإعدادات كإعدادات افتراضية.
- اختيار sgRNA مع المعيارين التاليين: 1) تظهر درجة خصوصية عالية، عموماً > 98٪، و 2) تفتقر إلى G التالية NGG، وهو ترتيب عزر البروتوسبل المجاورة (PAM). يقع عادة sgRNA المختار ة في مواقع قريبة من البداية ووقف codons من الجينات ذات الاهتمام.
- نسخ تسلسل sgRNA المحدد ولصقه في قالب التمهيدي التالي.

يمثل الجزء باللون الأحمر تسلسل SgCPL sgRNA المحدد. ويمكن استبداله مع sgRNAs مختلفة لمختلف الجينات ذات الاهتمام.
ملاحظة: إذا لم يبدأ sgRNA المحدد مع G، إضافة G في بداية sgRNA للمساعدة في تحسين التعبير الخاصة به. - إجراء تفاعل PCR لتعديل البلازميد الموجود مسبقًا المعبر عن sgRNA(الشكل 3A)الذي يستهدف توكسوبلازما uracil phosphoribosyltransferase(TgUPRT)جين23 باستخدام مخلط اتّصال PCR مع الإعدادات المتوفرة في الجدول 2.
- تشغيل منتج PCR على هلام agarose لتأكيد التضخيم الناجح. ومن المتوقع أن يتم تضخيم منتج PCR 10 كيلوبايت(الشكل 3B).
- استخراج منتج PCR باستخدام مجموعة استخراج هلام الحمض النووي والتعميم باستخدام مجموعة الطفرات الموجهة من الموقع. راجع الجدول 3 للحصول على الوصفة. احتضان رد الفعل لمدة 10-20 دقيقة في RT.
- تحويل منتج PCR دائريإلى E. القولونية واختيار 10 استنساخ لمزيد من التحقق من دمج sgRNA المصممة.
- تنمو اثنين من استنساخ واستخراج plasmids. قطع البلازميدات النقية مع BamHI وEcoRV. سوف تسفر البلازميدات المرشح اثنين من العصابات في 2.4 كيلوبايت و 7.2 كيلوبايت(الشكل 3C).
- إرسال plasmids لتسلسل Sanger باستخدام التمهيديات عكس M13 لتأكيد استبدال ناجحة من SgUPRT sgRNA مع sgRNA المصممة(الشكل 3D).
- انتقل إلى www.ToxoDB.org واسترداد تسلسل ترميز الجينات بالكامل، بما في ذلك الإنترونات والمواد الطاردة، إلى جانب 1.5 كيلوبايت 5'-UTRs و3'-UTRs (المناطق غير المترجمة).
- إنشاء قالب إصلاح لحذف الجينات عبر آلية HDR
- وفقًا لمواقع الاستهداف الخاصة بـ sgRNA المختار ، حدد 50 نقطة أساس من 5'UTRs أو 3'UTRs من الجين المستهدف لإعادة التركيب المعتمد على homology (HDR ، انظر قسم المناقشة). ويتبع اختيار المناطق المعايير المذكورة أدناه، تبعاً للموقع الذي تستهدفه الوكالة.
- إذا كان موقع الانقسام بواسطة Cas9 يقع في المنبع من codon البداية ، فحدد ما يلي: تسلسل الحمض النووي 50 bp المنبع من موقع الانقسام كمنطقة HDR اليسرى ، وتسلسل الحمض النووي 50 bp المصب من codon التوقف كمنطقة HDR اليمنى.
- إذا كان موقع الانقسام بواسطة Cas9 بين codons البداية والتوقف ، فحدد ما يلي: تسلسل 50 bp DNA المنبع من codon بداية كمنطقة HDR اليسرى ، وتسلسل الحمض النووي 50 bp المصب من codon التوقف كمنطقة HDR اليمنى.
- إذا كان موقع الانقسام بواسطة Cas9 يقع في المصب من codon التوقف ، حدد ما يلي: تسلسل الحمض النووي 50 bp المنبع من codon البداية كمنطقة HDR اليسرى ، وتسلسل الحمض النووي 50 bp المصب من موقع الانقسام كمنطقة HDR اليمنى.
ملاحظة: بالنسبة لمجين TgCPL، يقع موقع الانقسام بين codons البداية والتوقف. وهكذا ، تم تصميم التمهيديات التالية لتضخيم قالب الإصلاح باستخدام pMDC64 كقالب ، والذي يقوم بترميز كاسيت مقاومة البيريميثامين. التسلسلات في آنال أسود إلى pMDC64 plasmid لتضخيم PCR. المناطق المسماة باللون الأحمر هي تسلسلات محددة TgCPLلإعادة التركيب المتجانسة.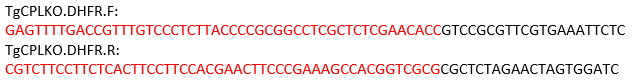
- تنفيذ PCR باستخدام premix PCR في ظل ظروف PCR الموضحة في الجدول 4.
- تشغيل منتج PCR على هلام agarose(الشكل 3E)،تليها استخراج هلام وإجراءات القياس الكمي الحمض النووي القياسية.
ملاحظة: إذا تعذر تكبير النطاق المتوقع بنجاح، قم بتحسين شروط PCR و/أو تبديل أزواج التمهيدي.
- وفقًا لمواقع الاستهداف الخاصة بـ sgRNA المختار ، حدد 50 نقطة أساس من 5'UTRs أو 3'UTRs من الجين المستهدف لإعادة التركيب المعتمد على homology (HDR ، انظر قسم المناقشة). ويتبع اختيار المناطق المعايير المذكورة أدناه، تبعاً للموقع الذي تستهدفه الوكالة.
- التَرَفَط التوكسوبلازما
- تمرير RHΟku80:: طفيلياتNLuc لمدة يومين في قارورة T25 تحتوي على HFFs ملتوية. قارورة T25 من الطفيليات المنكلة بالكامل كافية لحالتين إلى ثلاث حالات نقل.
- المحاقن وتنقية الفلتر الطفيليات كما هو موضح في الخطوة 1.2. إعادة تعليق الطفيليات في عازل السيتوميكس وتدور عند 1000 × ز لمدة 10 دقيقة في RT.
- غسل الطفيليات بيليه مع 10 مل من العازلة سيتوميكس وتدور أسفل الطفيليات في 1000 × ز لمدة 10 دقيقة في RT.
- صب بعناية قبالة supernatant وإعادة تعليق الطفيليات في نفس الحاجز بتركيز 1 × 108 الطفيليات / مل.
- مزيج 2 ميكروغرام من الحمض النووي قالب إصلاح مع 20 ميكروغرام من plasmids التعبير sgRNA/Cas9 (نسبة الكتلة = 1:5، أي ما يعادل نسبة 1:3 المولي). إذا كانت غلة التضخيم لقالب الإصلاح منخفضة، فقلّل من إدخال كل من قطع الحمض النووي وفقًا لذلك. يمكن استخدام ما لا يقل عن 0.5 ميكروغرام من قالب الإصلاح.
- مزيج 400 ميكرولتر من إعادة تعليق الطفيليات، والحمض النووي، و 5 ميكرولتر من 200 mM ATP/500 mM تقليل الجلوتاثيون (GSH) في أنبوب الطرد المركزي 1.5 مل. جلب الحجم الإجمالي إلى 500 ميكرولتر مع المخزن المؤقت سيتوميكس، إذا لزم الأمر.
- نقل خليط من الطفيليات والحمض النووي إلى كوفيت الكهربية (4 ملم عرض الفجوة) وإجراء الكهربية (2 كيلو فولت الجهد، 50 درجة المقاومة) باستخدام جهاز الكهربية.
- نقل الطفيليات الكهربائية إلى قارورة T25 تحتوي على HFFs متجانسة في المتوسط D10 الطازجة. تطبيق المضادات الحيوية المناسبة لاختيار المخدرات بعد 24 ساعة.
- الحفاظ على ضغط المخدرات انتقائية حتى نمو الطفيليات المعدلة وراثيا مستقرة.
- تنقية الحمض النووي الجينومي من السكان خروج المغلوب والتحقق من أجل دمج كاسيت مقاومة البيريميثامين في مكان TgCPL بواسطة PCR. بعد التحقق، انتقل إلى القسم 3.4. إذا لم يكن كذلك ، قم بإجراء جولة أخرى من التطفل الطفيلي واختيار الدواء. عدم القدرة على الكشف عن التكامل الصحيح لكاسيت مقاومة الدواء يشير عادة إلى أن الجين المستهدف ضروري أو أن موضع الجين غير قابل للوصول.
- استنساخ الطفيليات بالضربة القاضية
- البذور اثنين من لوحات صغيرة 96 جيدا مع خلايا HFF واحتضان في 37 درجة مئوية و 5٪ CO2 لمدة أسبوع واحد قبل استنساخ الطفيليات.
- تمرير ~ 0.3-0.4 مل من السكان من الطفيليات المعدلة وراثيا في قارورة T25 التي تحتوي على HFFs ملتوية وتنمو لهم لمدة 2 أيام. النظر في تمرير المزيد من الطفيليات إذا كان متحولة يظهر عيوب النمو.
ملاحظة: لتحقيق أفضل عائد وقابلية البقاء ، يتم إصابة الخلايا المضيفة بشدة من الطفيليات ، ويتم الاحتفاظ بمعظم الطفيليات في المرحلة داخل الخلايا. - الخلايا المضيفة المصابة بالمحاقن والطفيليات الطازجة المطهرة للفلتر كما هو مذكور في الخطوة 1.3. إعادة تعليق الطفيليات في وسط D10 وتدور عليها في 1000 × ز لمدة 10 دقيقة في RT.
- إعادة تعليق الطفيليات بيليه في 10 مل من متوسط D10.
- عد الطفيليات باستخدام مقياس الهيموكيتومتر لتحديد تركيز الطفيليات.
- إجراء تخفيف من خطوتين لتحقيق التركيز إلى 10 طفيليات / مل في متوسط D10 مكمّلبالمضاد الحيوي المناسب. عادة ، يتم تخفيف إعادة تعليق الطفيليات الأولية بمقدار 1000 مرة ، تليها تخفيف ثان ٍ إلى 10 طفيليات / مل.
- يُسْتَرّس ُ الوسائط من 96 لوحة صغيرة جيداً تحتوي على مركبات HFFs مُنْسِبة ومقوّنة 150 ميكرولتر من الطفيليات المخففة في كل بئر.
- ألواح الإنكوبات عند 37 درجة مئوية مع 5% CO2 لمدة 7 أيام دون إزعاج للسماح بتشكيل البلاك. يمكن أن تكون فترة الحضانة أطول إذا أظهرت الطفيليات المعدلة وراثيا عيوب النمو.
- فحص لوحات باستخدام مجهر المرحلة التباين ووضع علامة فقط على الآبار التي تحتوي على لوحة واحدة.
- تنفيذ PCR مستعمرة لتحديد المستنسخين الصحيح.
- استخدام نصائح ماصة لكشط الجزء السفلي من كل بئر لرفع monolayers HFF المصابة.
- Pipet 75 μL من إعادة تعليق الخلية من كل ملحوظ جيدا في 1.5 مل أنابيب الطرد المركزي الدقيقة.
- أنابيب الطرد المركزي لمدة 10 دقيقة بأقصى سرعة في RT. يستنشق بعناية supernatant وإعادة تعليق بيليه في 10.25 ميكرولتر من محلول العازلة التي تحتوي على عازلة التخفيف والحمض النووي المضافة المنصوص عليها في عدة(جدول المواد).
- احتضان العينات لمدة 4 دقيقة في RT، ثم 2 دقيقة في 98 درجة مئوية. بعد ذلك ، يمكن استخدام العينات لPCR أو تخزينها عند -20 درجة مئوية حتى الاستخدام. وتستخدم ثلاث مجموعات من ردود الفعل PCR لاختبار لتكامل شريط مقاومة المخدرات وفقدان الجينات ذات الفائدة(الشكل 4A). راجع الجدول 5 لإعداد تفاعل PCR والجدول 6 لإعدادات thermocycler.
- تحديد المستنسخات الصحيحة ونقل أربع مستنسخات إلى قوارير T25 تحتوي على HFFs ملتوية.
- بعد استنساخ الفردية lyse الخلايا المضيفة، وتنقية الحمض النووي الجينوم لمزيد من التحقق PCR.
- إذا كان الجسم المضاد الذي يعترف بالبروتين المهم متاحًا ، فاتبع إجراءً قياسيًا للتنبال مناعي للتحقق من فقدان البروتين المستهدف في ضربات توكسوبلازما الصحيحة. وتظهر الصور التمثيلية لفحص TgCPL-حذف متحولة في الشكل 4B, C.
النتائج
ويمثل الشكل 1 مثالاً لمنحنى النمو لسلالة RHΟku80::سلالة NLuc والحساب المشتق لمضاعفة وقتها. عموما، يتم تنفيذ القول في ثلاثة يكرر التقنية لكل من النسخ البيولوجية الثلاث لتكرار لحساب الاختلافات من قراءات نشاط لوسيفيراز. من أجل حساب التغير العادي في نمو ...
Discussion
++يصف هذا البروتوكول بروتوكولًا قائمًا على اللوكفراز لتقييم نمو توكسوبلازما داخل الخلايا وتقييم فعالية تثبيط المركبات الكيميائية ضد نمو الطفيليات. بالمقارنة مع الاستراتيجيات الحالية المتاحة لقياس نمو توكسوبلازما داخل الخلايا، هذه الطريقة تظهر حساسية عالية وخصوصية عالية. أثن?...
Disclosures
وليس لدى أصحاب البلاغ ما يكشفون عنه.
Acknowledgements
يود المؤلفان أن يشكرا الدكتورين سيبلي وكاروثرز على مشاركة pSAG1-Cas9-sgRNA-TgUPRT plasmid وAnti-TgCPL وTgActin. تم دعم هذا العمل من قبل صندوق كليمسون بدء التشغيل (إلى Z.D.) ، فرسان فرسان الطب المنجل مؤسسة طب الأطفال طب العيون الوظيفي مبتدئ منحة البحوث (إلى Z.D.) ، منحة تجريبية من منحة NIH COBRE P20GM109094 (إلى Z.D.) ، وNIH R01AI143707 (إلى Z.D.). لم يكن للممولين أي دور في تصميم الدراسة أو جمع البيانات وتحليلها أو قرار نشر المخطوطة أو إعدادها.
Materials
| Name | Company | Catalog Number | Comments |
| Agarose gel extraction kit | New England BioLabs | T1020L | |
| BamHI | New England BioLabs | R0316S | |
| Biotek Synergy H1 Hybrid Multi-Mode Microplate Reader | BioTek Instuments | ||
| BTX Gemini Twin Waveform Electroporation System | Harvard Apparatus | ||
| Chemically competent E. coli cells | New England BioLabs | C29871 | |
| CloneAmp HiFi PCR premix | Takara Bio | 639298 | |
| Coelenterazine h | Prolume | 301-10 hCTZ | |
| EcoRV | New England BioLabs | R3195S | |
| Phire Tissue Direct PCR Master Mix | Thermo Scientific | F170L | |
| Plasmid miniprep kit | Zymo Research | D4054 | |
| Q5 Site-Directed Mutagenesis kit | New England BioLabs | E0554S | |
| Software | |||
| Geneious software for sgRNA design (version: R11) | |||
| GraphPad Prism software (8th version) | |||
| SnapGene for molecular cloning (version: 4.2.11) |
References
- Blader, I. J., Coleman, B. I., Chen, C. T., Gubbels, M. J. Lytic Cycle of Toxoplasma gondii: 15 Years Later. Annual Review of Microbiology. 69 (1), 1-23 (2014).
- Jones, J. L., Kruszon-Moran, D., Rivera, H., Price, C., Wilkins, P. P. Toxoplasma gondii Seroprevalence in the United States 2009-2010 and Comparison with the Past Two Decades. The American Journal of Tropical Medicine and Hygiene. 90 (6), (2014).
- Kieffer, F., Wallon, M. Congenital toxoplasmosis. Handbook of Clinical Neurology. 112, 1099-1101 (2013).
- Hoffmann, S., Batz, M. B., Morris, G. J. Annual cost of illness and quality-adjusted life year losses in the United States due to 14 foodborne pathogens. Journal of Food Protection. 75 (7), 1292-1302 (2012).
- Dubey, J. Toxoplasmosis. Journal of the American Veterinary Medical Association. 205 (11), 1593-1598 (1994).
- Lindsay, D., Dubey, J. Toxoplasma gondii: the changing paradigm of congenital toxoplasmosis. Parasitology. 138 (14), 1-3 (2011).
- Deng, Y., Wu, T., Zhai, S., Li, C. Recent progress on anti-Toxoplasma drugs discovery: Design, synthesis and screening. European Journal of Medicinal Chemistry. 183, 111711 (2019).
- Butler, N. J., Furtado, J. M., Winthrop, K. L., Smith, J. R. Ocular toxoplasmosis II: clinical features, pathology and management. Clinical & Experimental Ophthalmology. 41 (1), 95-108 (2013).
- Pfefferko, E., Pfefferko, L. C. Specific Labeling of Intracellular Toxoplasma gondii with Uracil. Journal of Eukaryotic Microbiology. 24 (3), 449-453 (1977).
- Merli, A., Canessa, A., Melioli, G. Enzyme immunoassay for evaluation of Toxoplasma gondii growth in tissue culture. Journal of Clinical Microbiology. 21 (1), 88-91 (1985).
- Derouin, F., Chastang, C. Enzyme immunoassay to assess effect of antimicrobial agents on Toxoplasma gondii in tissue culture. Antimicrobial Agents and Chemotherapy. 32 (3), 303-307 (1988).
- McFadden, D., Seeber, F., Boothroyd, J. Use of Toxoplasma gondii expressing beta-galactosidase for colorimetric assessment of drug activity in vitro. Antimicrobial Agents and Chemotherapy. 41 (9), 1849-1853 (1997).
- Gubbels, M. J., Li, C., Striepen, B. High-Throughput Growth Assay for Toxoplasma gondii Using Yellow Fluorescent Protein. Antimicrobial Agents and Chemotherapy. 47 (1), 309-316 (2003).
- Touquet, B., et al. High-content imaging assay to evaluate Toxoplasma gondii infection and proliferation: A multiparametric assay to screen new compounds. PLoS ONE. 13 (8), e0201678 (2018).
- Thornton, L. B., et al. An ortholog of Plasmodium falciparum chloroquine resistance transporter (PfCRT) plays a key role in maintaining the integrity of the endolysosomal system in Toxoplasma gondii to facilitate host invasion. PLOS Pathogens. 15 (6), e1007775 (2019).
- Larson, E. T., et al. Toxoplasma gondii cathepsin L is the primary target of the invasion-inhibitory compound morpholinurea-leucyl-homophenyl-vinyl sulfone phenyl. The Journal of Biological Chemistry. 284 (39), 26839-26850 (2009).
- Dou, Z., McGovern, O. L., Cristina, M., Carruthers, V. B. Toxoplasma gondii Ingests and Digests Host Cytosolic Proteins. mBio. 5 (4), e01188-14 (2014).
- Cristina, M., et al. Toxoplasma depends on lysosomal consumption of autophagosomes for persistent infection. Nature Microbiology. 2, 17096 (2017).
- Parussini, F., Coppens, I., Shah, P. P., Diamond, S. L., Carruthers, V. B. Cathepsin L occupies a vacuolar compartment and is a protein maturase within the endo/exocytic system of Toxoplasma gondii. Molecular Microbiology. 76 (6), 1340-1357 (2010).
- Huynh, M. H., Carruthers, V. B. Tagging of endogenous genes in a Toxoplasma gondii strain lacking Ku80. Eukaryotic cell. 8 (4), 530-539 (2009).
- Fox, B. A., Ristuccia, J. G., Gigley, J. P., Bzik, D. J. Efficient gene replacements in Toxoplasma gondii strains deficient for nonhomologous end joining. Eukaryotic Cell. 8 (4), 520-529 (2009).
- Sidik, S. M., Hackett, C. G., Tran, F., Westwood, N. J., Lourido, S. Efficient Genome Engineering of Toxoplasma gondii Using CRISPR/Cas9. PLoS ONE. 9 (6), e100450 (2014).
- Shen, B., Brown, K. M., Lee, T. D., Sibley, D. L. Efficient Gene Disruption in Diverse Strains of Toxoplasma gondii Using CRISPR/CAS9. mBio. 5 (3), e01114-14 (2014).
- Radke, J. R., et al. Defining the cell cycle for the tachyzoite stage of Toxoplasma gondii. Molecular and Biochemical Parasitology. 115 (2), 165-175 (2001).
- Ran, A. F., et al. Genome engineering using the CRISPR-Cas9 system. Nature Protocols. 8 (11), 2281-2308 (2013).
- Labun, K., Montague, T. G., Gagnon, J. A., Thyme, S. B., Valen, E. CHOPCHOP v2: a web tool for the next generation of CRISPR genome engineering. Nucleic Acids Research. 44 (W1), W272-W276 (2016).
- Heigwer, F., Kerr, G., Boutros, M. E-CRISP: fast CRISPR target site identification. Nature Methods. 11 (2), 2812 (2014).
- Peng, D., Tarleton, R. EuPaGDT: a web tool tailored to design CRISPR guide RNAs for eukaryotic pathogens. Microbial Genomics. 1 (4), e000033 (2015).
- Doench, J. G., et al. Rational design of highly active sgRNAs for CRISPR-Cas9-mediated gene inactivation. Nature Biotechnology. 32 (12), 1262-1267 (2014).
- Sidik, S. M., et al. A Genome-wide CRISPR Screen in Toxoplasma Identifies Essential Apicomplexan Genes. Cell. 166 (6), 1423-1435 (2016).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved