Method Article
نمذجة موقع نشط إنزيم باستخدام التصور الجزيئي مجانية
In This Article
Summary
مهارة رئيسية في النمذجة الجزيئية الحيوية هو عرض والتعليق على المواقع النشطة في البروتينات. وقد أثبتت هذه التقنية باستخدام أربعة برامج شعبية مجانية للتصور الجزيئي الكلي: iCn3D، Jmol، PyMOL، وUCSF ChimeraX.
Abstract
مهارات التصور الجزيئي الحيوي هي ذات أهمية قصوى لفهم المفاهيم الرئيسية في العلوم البيولوجية، مثل العلاقات بين الهيكل والوظيفة والتفاعلات الجزيئية. تسمح البرامج المختلفة للمتعلم بمعالجة الهياكل ثلاثية الأبعاد ، وتعزز النمذجة الجزيئية الحيوية التعلم النشط ، وتبني المهارات الحسابية ، وتسد الفجوة بين صور الكتب المدرسية ثنائية الأبعاد والأبعاد الثلاثة للحياة. مهارة حاسمة في هذا المجال هو نموذج موقع البروتين النشط، وعرض أجزاء من الجزيئات الكبيرة التي يمكن أن تتفاعل مع جزيء صغير، أو ليغاند، بطريقة تظهر التفاعلات ملزمة. في هذا البروتوكول، نقوم بوصف هذه العملية باستخدام أربعة برامج نمذجة جزيئية حرة متوفرة: iCn3D، Jmol/JSmol، PyMOL، وUCSF ChimeraX. هذا الدليل موجه للطلاب الذين يسعون إلى تعلم أساسيات برنامج معين، وكذلك المعلمين الذين يدمجون النمذجة الجزيئية الحيوية في مناهجهم الدراسية. يمكن البروتوكول المستخدم من تصميم موقع نشط باستخدام برنامج مرئيات محدد، أو أخذ عينات من العديد من البرامج المجانية المتوفرة. النموذج الذي تم اختياره لهذا البروتوكول هو الجلوكوكيناز البشري ، وهو شكل متساوي من إنزيم hexokinase ، الذي يحفز الخطوة الأولى من انحلال الجليكوليسيس. يرتبط الإنزيم بأحد ركائزه ، بالإضافة إلى تناظر ركيزة غير تفاعلي ، والذي يسمح للمستخدم بتحليل التفاعلات في المجمع الحفاز.
Introduction
فهم تمثيلات العالم الجزيئي أمر بالغ الأهمية لتصبح خبيرا في العلوم الجزيئية الحيوية1، لأن تفسير مثل هذه الصور هو المفتاح لفهم الوظيفة البيولوجية2. عادة ما تأتي مقدمة المتعلم للجزيئات الكبيرة في شكل صور مدرسية ثنائية الأبعاد لأغشية الخلايا ، العضيات ، الجزيئات الكبيرة ، وما إلى ذلك ، ولكن الواقع البيولوجي هو أن هذه هي هياكل ثلاثية الأبعاد ، وفهم خصائصها يتطلب طرقا لتصور واستخراج المعنى من النماذج ثلاثية الأبعاد.
وبناء على ذلك، اكتسب تطوير محو الأمية البصرية الجزيئية الحيوية في دورات علوم الحياة الجزيئية في القسم العلوي اهتماما، حيث قدم عدد من المقالات تقارير عن أهمية وصعوبات تدريس وتقييم مهارات التصور1و3و4و5و6و7و8و9 . وكانت الاستجابة لهذه المواد زيادة في عدد التدخلات الصفية، عادة في غضون فصل دراسي في مؤسسة واحدة، حيث يتم استخدام برامج التصور الجزيئي والنماذج لاستهداف المفاهيم الصعبة2و10و11و12و13و14و15 . بالإضافة إلى ذلك، سعى الباحثون إلى توصيف كيفية استخدام الطلاب لبرامج التصور الجزيئي الحيوي و / أو نماذج للتعامل مع موضوع معين16،17،18،19. وقد وصفت مجموعتنا الخاصة ، BioMolViz ، إطارا ينقسم إلى مواضيع شاملة في محو الأمية البصرية إلى أهداف وأهداف تعليمية لتوجيه مثل هذه التدخلات20،21، ونحن نقود ورش عمل تدرب أعضاء هيئة التدريس على استخدام الإطار في التصميم المتخلف للتقييمات لقياس مهارات محو الأمية البصرية22.
في مركز كل هذا العمل هو مهارة حاسمة : القدرة على التعامل مع هياكل الجزيئات الكبيرة باستخدام برامج التصور الجزيئي الحيوي. وقد تم تطوير هذه الأدوات بشكل مستقل باستخدام مجموعة متنوعة من المنصات؛ لذلك، فإنها يمكن أن تكون فريدة من نوعها بدلا في تشغيلها واستخدامها. وهذا يتطلب تعليمات خاصة بالبرنامج، وتحديد البرنامج الذي يرتاح إليه المستخدم مهم لتسهيل التنفيذ المستمر.
وراء أساسيات التلاعب في هياكل 3D (الدورية ، واختيار ، وتغيير النموذج) ، والهدف الرئيسي هو نموذج للموقع النشط للبروتين. هذه العملية تسمح للمتعلم لتطوير فهمهم في ثلاثة مواضيع شاملة وصفها إطار BioMolViz : التفاعلات الجزيئية ، ligands / التعديلات ، والعلاقات هيكل وظيفة20،21.
أربعة خيارات شعبية من برامج التصور الجزيئي الحيوي وتشمل: Jmol / JSmol23، iCn3D24، PyMOL25، وUCSF Chimera26،27. ونحن نشجع أولئك الجدد على Chimera لاستخدام UCSF ChimeraX ، الجيل القادم من برنامج التصور الجزيئي Chimera ، وهو الإصدار المدعوم حاليا من البرنامج.
في هذا البروتوكول، ونحن نوضح كيفية استخدام كل من هذه البرامج الأربعة لنموذج موقع نشط من glucokinase الإنسان مع مجمع التناظرية الركيزة ملزمة (PDB ID: 3FGU)، وعرض القياسات لتوضيح التفاعلات ملزمة محددة28. النموذج يمثل مجمع الحفاز من الانزيم. لالتقاط الموقع النشط في حالة ما قبل الحفز ، كان من المحتم أن يكون تناظريا غير قابل للتحلل من ATP إلى موقع glucokinase النشط. يحتوي هذا الفوسفوامينوففوسفونيك حمض أدينيلات إستر (ANP) على رابطة الفوسفور والنيتروجين بدلا من الربط المعتاد بين الفوسفور والأوكسجين في هذا الموقف. يحتوي الموقع النشط أيضا على الجلوكوز (BCG المشار إليه في النموذج) والمغنيسيوم (المشار إليه ب MG). بالإضافة إلى ذلك ، هناك أيون البوتاسيوم (K) في الهيكل ، الناتج عن كلوريد البوتاسيوم المستخدم في مذيب التبلور. هذا الأيون ليس حاسما بالنسبة للوظيفة البيولوجية ويقع خارج الموقع النشط.
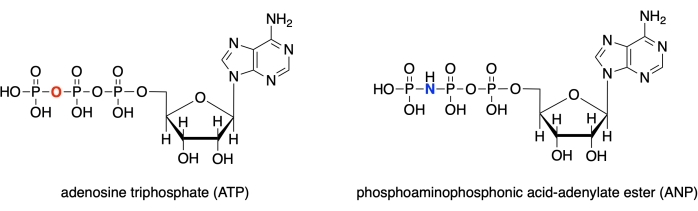
الشكل 1: هياكل ATP / ANP. أدينوسين ثلاثي الفوسفات (ATP) بنية مقارنة مع فوسفوامينوفوسفونيك حمض أدينيلات إستر (ANP). يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
ويوضح البروتوكول اختيار الليجاندات المقيدة للمجمع التناظري الركيزي وتحديد بقايا الموقع النشط ضمن 5 Å من المجمع المقيد ، الذي يلتقط الأحماض الأمينية وجزيئات الماء القادرة على إجراء التفاعلات الجزيئية ذات الصلة ، بما في ذلك التفاعلات الكارهة للماء وفان دير وال.
يتم التلاعب في البداية العرض لإظهار غالبية البروتين في تمثيل الرسوم المتحركة، مع بقايا الأحماض الأمينية الموقع النشط في تمثيل عصا لإظهار الذرات ذات الصلة من البروتين وتسليط الضوء على التفاعلات الجزيئية. بعد الخطوة 3 من البروتوكول لكل برنامج، تم تطبيق هذه التمثيلات ورؤية البروتين متشابهة عبر البرامج(الشكل 2). في نهاية البروتوكول ، يتم إخفاء الرسوم المتحركة البروتين لتبسيط العرض ، والتركيز على الموقع النشط.
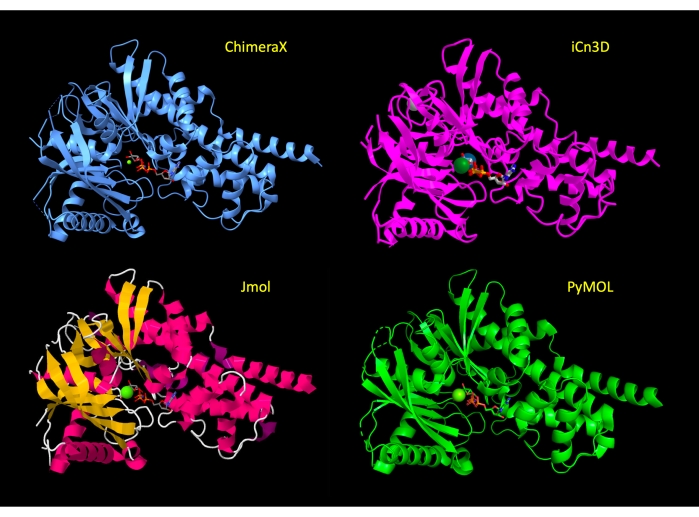
الشكل 2: مقارنة البنية عبر البرامج. مقارنة بنية 3FGU في كل برنامج بعد الخطوة ضبط التمثيل (الخطوة 2 أو 3 من كل بروتوكول). يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
يتم تطبيق التلوين CPK إلى موقع نشط الأحماض الأمينية وليجاندسملزمة 29،30. يميز نظام التلوين هذا ذرات العناصر الكيميائية المختلفة في النماذج الجزيئية الموضحة في الخط والعصا والكرة والعصا وتمثيلات ملء الفضاء. الهيدروجين أبيض، النيتروجين أزرق، الأكسجين أحمر، الكبريت أصفر، والفوسفور برتقالي في نظام تلوين CPK. تقليديا، يستخدم الأسود للكربون، على الرغم من أن في الاستخدام الحديث، قد يختلف تلوين الكربون.
ذرات الهيدروجين غير مرئية في الهياكل البلورية، على الرغم من أن كل من هذه البرامج قادرة على التنبؤ بموقعها. إضافة ذرات الهيدروجين إلى بنية كبيرة الجزيئية الكبيرة يمكن أن تحجب وجهة النظر، وبالتالي لا يتم عرضها في هذا البروتوكول. وبناء على ذلك، ستظهر روابط الهيدروجين من خلال القياس من مركز اثنين من الهترواتوم (على سبيل المثال، الأكسجين إلى الأكسجين، الأكسجين إلى النيتروجين) في هذه الهياكل.
نظرة عامة على البرنامج
واجهات المستخدم الرسومية القابلة للتنزيل (GUIs): PyMOL (الإصدار 2.4.1) ، ChimeraX (الإصدار 1.2.5) ، وJmol (الإصدار 1.8.0_301) هي أدوات النمذجة الجزيئية المستندة إلى واجهة المستخدم الرسومية. هذه الواجهات الثلاثة ميزة خطوط الأوامر لإدخال رمز مكتوب; العديد من القدرات نفسها متوفرة من خلال القوائم والأزرار في واجهة المستخدم الرسومية. ميزة شائعة في سطر الأوامر من هذه البرامج هو أن المستخدم قد تحميل وإعادة تنفيذ الأوامر السابقة باستخدام مفاتيح الأسهم لأعلى ولأسفل على لوحة المفاتيح.
معرفات المستخدمين على الويب: iCn3D (I-see-in-3D) هو عارض يستند إلى WebGL للعرض التفاعلي للهياكل الجزيئية الكلية ثلاثية الأبعاد والمواد الكيميائية على الويب، دون الحاجة إلى تثبيت تطبيق منفصل. لا يستخدم سطر أوامر، على الرغم من أن إصدار الويب الكامل يحتوي على سجل أوامر قابل للتحرير. JSmol هو إصدار جافا سكريبت أو HTML5 من Jmol للاستخدام على موقع ويب أو في نافذة متصفح الويب ، وهو مشابه جدا في التشغيل ل Jmol. يمكن استخدام JSmol لإنشاء دروس عبر الإنترنت ، بما في ذلك الرسوم المتحركة.
بروتوبيديا31،32، FirstGlance في Jmol33، وواجهة الويب JSmol (JUDE) في كلية ميلووكي للهندسة مركز النمذجة الجزيئية الحيوية هي أمثلة على مثل هذه البيئات تصميم على الانترنت Jmol القائم34. ويكي Proteopedia هو أداة تعليمية تسمح للمستخدم بنمذجة بنية الجزيئات الكبيرة وإنشاء صفحات تضم هذه النماذج داخل الموقع35. أداة تأليف المشهد بروتوبيديا، التي بنيت باستخدام JSmol، يدمج واجهة المستخدم الرسومية مع ميزات إضافية غير متوفرة في واجهة المستخدم الرسومية Jmol.
وتستند Jmol و iCn3D على لغة البرمجة جافا؛ JSmol يستخدم إما جافا أو HTML5 ، وتستند PyMOL وChiraX على لغة البرمجة بيثون. كل من هذه البرامج تحميل ملفات بنك بيانات البروتين، والتي يمكن تحميلها من بنك بيانات البروتين RCSB تحت 4 أرقام رقمية PDB معرف36،37. أنواع الملفات الأكثر شيوعا هي ملفات بنك بيانات البروتين (PDB) التي تحتوي على ملحق .pdb وملف المعلومات البلورية (CIF أو mmCIF) التي تحتوي على ملحق .cif. حلت CIF محل PDB كنوع الملف الافتراضي لبنك بيانات البروتين، ولكن تنسيقي الملف يعملان في هذه البرامج. يمكن أن يكون هناك اختلافات طفيفة في الطريقة التي يتم عرض التسلسل / بنية عند استخدام CIF بدلا من ملفات PDB; ومع ذلك، الملفات تعمل بشكل مماثل والاختلافات لن يتم تناولها بالتفصيل هنا. قاعدة بيانات النمذجة الجزيئية (MMDB)، وهي نتاج المركز الوطني لمعلومات التكنولوجيا الحيوية (NCBI)، هي مجموعة فرعية من هياكل PDB التي ارتبطت بها المعلومات القاطعة (على سبيل المثال، السمات البيولوجية، مجالات البروتين المحفوظة)38. iCn3D، وهو منتج من NCBI، قادر على تحميل ملفات PDB التي تحتوي على بيانات MMDB.
لعرض نموذج، يمكن للمستخدم تحميل الملف المطلوب من صفحة "بنك بيانات البروتين" المخصصة للبنية (على سبيل المثال، https://www.rcsb.org/structure/3FGU)،ثم استخدام القائمة المنسدلة "ملف" للبرنامج لفتح الهيكل. جميع البرامج أيضا قادرة على تحميل ملف بنية مباشرة من خلال واجهة، وهذا الأسلوب مفصلة داخل البروتوكولات.
تحتوي كل من ChimeraX وJmol وPyMOL GUIs على إطار واحد أو أكثر من وحدة التحكم التي قد يتم إعادة حجمها عن طريق سحب الزاوية. iCn3D وJSmol موجودة تماما في متصفح الويب. عند استخدام iCn3D، قد يحتاج المستخدم إلى التمرير داخل الإطارات المنبثقة للكشف عن كافة عناصر القائمة، اعتمادا على حجم الشاشة ودقة.
توفر البروتوكولات المفصلة هنا طريقة بسيطة لعرض الموقع النشط للانزيم باستخدام كل برنامج. وتجدر الإشارة إلى أن هناك طرق متعددة لتنفيذ الخطوات في كل برنامج. على سبيل المثال، في ChimeraX، قد يتم تنفيذ نفس المهمة باستخدام القوائم المنسدلة أو شريط الأدوات في الأعلى أو سطر الأوامر. يتم تشجيع المستخدمين المهتمين في تعلم برنامج معين بالتفصيل لاستكشاف الدروس على الانترنت، والكتيبات، والويكي المتاحة لهذه البرامج39،40،41،42،43،44،45،46.
الكتيبات والبرامج التعليمية الموجودة لهذه البرامج تقديم العناصر في هذا البروتوكول كمهام منفصلة. لعرض موقع نشط، يجب على المستخدم تجميع العمليات المطلوبة من الأدلة والبرامج التعليمية المختلفة. تزيد هذه المخطوطة من الدروس الموجودة المتاحة من خلال تقديم بروتوكول خطي لنمذجة موقع نشط يحمل علامة مع التفاعلات الجزيئية ، مما يوفر للمستخدم منطقا لنمذجة الموقع النشطة التي يمكن تطبيقها على نماذج وبرامج أخرى.
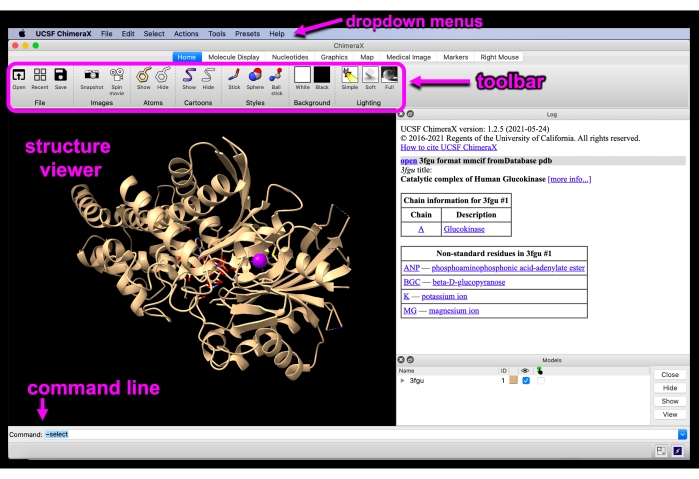
الشكل 3: تشيميراإكس GUI. واجهة واجهة المستخدم الرسومية ChimeraX مع القوائم المنسدلة وشريط الأدوات، عارض البنية، وخط الأوامر المسمى. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
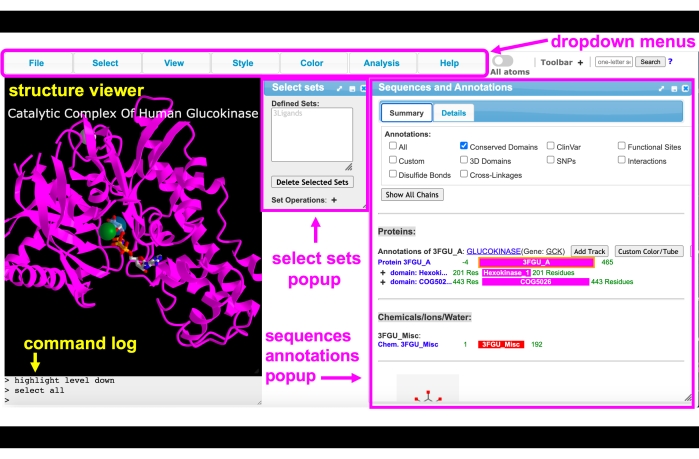
الشكل 4: واجهة المستخدم الرسومية iCn3D. واجهة المستخدم الرسومية iCn3D مع القوائم المنسدلة، شريط الأدوات، عارض الهيكل، سجل الأوامر، حدد مجموعات المنبثقة، وتسلسل والتعليقات التوضيحية القوائم المنبثقة المسمى. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
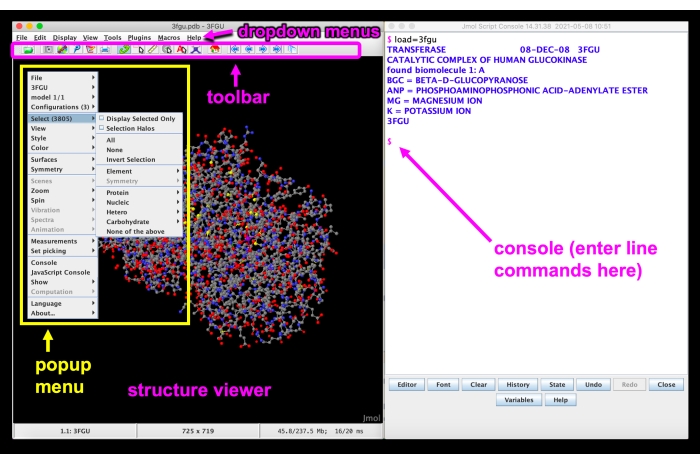
الشكل 5: Jmol GUI. واجهة المستخدم الرسومية Jmol مع القوائم المنسدلة وشريط الأدوات، عارض الهيكل، القائمة المنبثقة، وحدة التحكم / سطر الأوامر المسمى. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
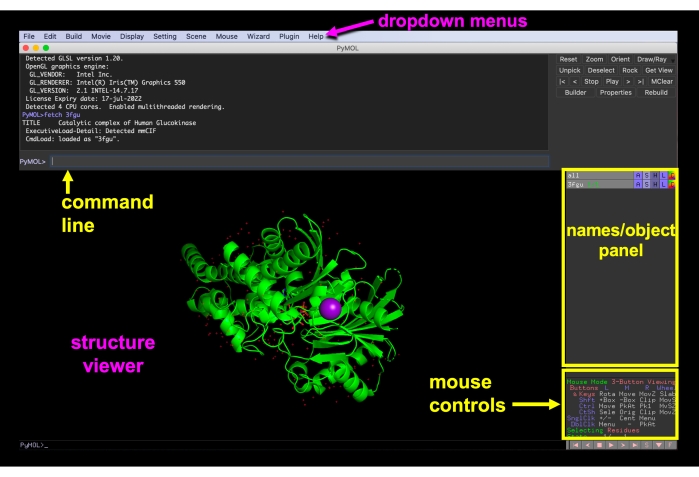
الشكل 6: PyMOL GUI. واجهة المستخدم الرسومية PyMOL مع القوائم المنسدلة، عارض البنية، الأسماء/ لوحة الكائن، قائمة عناصر تحكم الماوس، وخط الأوامر المسمى. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
Protocol
ملاحظة: يتم تحديد البروتوكول لكل برنامج في عشر خطوات شاملة، (1) تحميل الهيكل في البرنامج، (2) تحديد ليغاندس في الموقع النشط، (3) ضبط التمثيل، (4) اختيار المخلفات داخل 5 ألف لتحديد موقع نشط، (5) إظهار تفاعلات الإنزيم مع ligands الموقع النشط، (6) عرض السلاسل الجانبية والعصي وإظهار / ضبط جزيئات الماء بالموقع النشط، (7) تبسيط الهيكل ، (8) وضع العلامات على السلاسل الجانبية والسلاسل الجانبية المستعبدين الهيدروجين ، (9) حفظ التقديم في أي نقطة للعودة إلى العمل عليه أو مشاركته مع الآخرين ، (10) حفظ صورة للتضمين أو الطباعة. الخطوات 1 و 4 و 7-10 متطابقة لكل بروتوكول; ومع ذلك، بسبب تشغيل فريدة من كل برنامج، يتم تنفيذ بعض البروتوكولات بشكل أكثر كفاءة عند تبادل الخطوتين 2/3 و 5/6.
1. بروتوكول UCSF تشيميراإكس
ملاحظة: لوحة التتبع و عناصر تحكم الماوس. لتدوير، انقر واسحب أو استخدم سحب بإصبعين (الماوس: انقر فوق الأيسر واسحب). للتكبير، قرصة وانتشار (ماك) أو التحكم + حركة إصبعين (PC) (الماوس: عجلة التمرير). لترجمة (أي، نقل الهيكل بأكمله) اضغط على الخيار + انقر واسحب (ماك) أو التحول + انقر واسحب (PC) (الماوس: انقر بزر الماوس الأيمن واسحب). لإعادة التوسيط، استخدم القوائم المنسدلة في أعلى الواجهة للنقر على الإجراءات > عرض.
- تحميل البنية في ChimeraX: في سطر الأوامر الموجود في الجزء السفلي من واجهة المستخدم الرسومية مسبوقة ب "الأمر:" ، اكتب:
فتح 3fgu
ملاحظة: بعد إدخال أي أمر خط مكتوب، اضغط على العودة على لوحة المفاتيح لتنفيذه. - تحديد ligands في الموقع النشط: تأكد من وجود تمثيلين، شريط الكرتون والعصي. باستخدام الماوس، تدوير / تكبير البروتين لتصور أفضل ليغاندس عرض بالقرب من مركز البروتين، والتي تظهر على شكل العصي. تحوم فوق ليغاند لإظهار اسمها.
- ضبط التمثيل: استخدم الأوامر في الخطوات الفرعية أدناه لإعادة تلوين البروتين والليغاندس، وتطبيق تلوين CPK على الذرات غير الكربونية، ثم إلغاء تحديد التحديد. تصبح أجزاء مختارة من الجزيء مميزة باللون الأخضر.
- استخدام القوائم المنسدلة في الجزء العلوي من واجهة لتغيير التلوين: انقر على الإجراءات > اللون > الذرة الأزرق. ثم انقر على تحديد > هيكل > ليغاند. لتحديد اللون، انقر على الإجراءات > اللون > رمادي. لتطبيق التلوين CPK، انقر على تحديد > الكل،ثم انقر على الإجراءات > اللون > بواسطة Heteroatom. وأخيرا، قم بإلغاء تحديد من خلال النقر على تحديد > مسح.
ملاحظة: قد يتم أيضا مسح التحديد عن طريق الضغط على عنصر التحكم والنقر فوق الخلفية السوداء لمشاهد البنية أو في سطر الأوامر عن طريق الكتابة: ~select. افتراضيا، بالنسبة لمعظم الهياكل التي تحتوي على سلاسل 1-4، سوف تظهر ChimeraX تلقائيا جزيئات الماء وبقايا الأحماض الأمينية داخل 3.6 Å من الليغند والأيونات. - استخدم القائمة المنسدلة لإخفاء الذرات المعروضة حاليا بالنقر على الإجراءات > Atoms/Bonds > إخفاء.
- استخدم القائمة المنسدلة لإظهار ligands و Mg ion في الموقع النشط من خلال النقر على تحديد هيكل > > Ligand. ثم انقر على الإجراءات > الذرات / السندات > إظهار. بعد ذلك، انقر على تحديد > المخلفات > MG،ومن ثم على الإجراءات > الذرات / السندات > إظهار. لمسح التحديد، انقر على تحديد > مسح.
ملاحظة: بعد إجراء التحديد مع القائمة المنسدلة، الخطوة 1.3.3 قد يتم تنفيذها بالنقر فوق الزرين إخفاء وإظهار في شريط أدوات Atoms.
- استخدام القوائم المنسدلة في الجزء العلوي من واجهة لتغيير التلوين: انقر على الإجراءات > اللون > الذرة الأزرق. ثم انقر على تحديد > هيكل > ليغاند. لتحديد اللون، انقر على الإجراءات > اللون > رمادي. لتطبيق التلوين CPK، انقر على تحديد > الكل،ثم انقر على الإجراءات > اللون > بواسطة Heteroatom. وأخيرا، قم بإلغاء تحديد من خلال النقر على تحديد > مسح.
- اختيار المخلفات داخل 5 Å لتحديد موقع نشط: في المشاهد هيكل، لتحديد التحكم الصحافة ligands + التحول وأداء النقر بالماوس على أي ذرة واحدة أو السندات في كل من ليغندس الثلاثة، أي، BCG، ANP، ومغ.
- بعد ذلك، اضغط على مفتاح السهم لأعلى على لوحة المفاتيح حتى يتم تمييز جميع ذرات الليجاند الثلاثة بتوهج أخضر. حدد هذا التحديد للاستخدام المستقبلي بالنقر فوق في القائمة المنسدلة حدد > تحديد محدد. في القائمة المنبثقة، اكتب:
ligands لتسمية التحديد الحالي، ثم انقر فوق موافق.
ملاحظة: النقر فوق السهم لأعلى مرات عديدة جدا في الخطوة 1.4.1 سيتم تحديد البروتين بأكمله. في هذه الحالة، انقر فوق زر السهم لأسفل حتى يتم تحديد ذرات الليجاند الثلاثة فقط. - استخدم القائمة المنسدلة لتحديد المخلفات داخل 5 Å من الليجاند: انقر على تحديد منطقة >. في النافذة المنبثقة التي تظهر، قم بتبديل القائمة المنسدلة تحديد إلى Residues، وتأكد من أن المربع العلوي محدد (تحقق من المسافة الأقل من (<) وتعيينه إلى 5.0 Å). ثم انقر فوق موافق. سيتم تسليط الضوء على المخلفات التي هي أقل من 5Å بعيدا.
ملاحظة: يمكن تبسيط الخطوات 1.4-1.4.2 بشكل مكثف باستخدام سطر الأوامر، بكتابة:
اسم ليغاندس المجمدة :BGC:MG:ANP
حدد منطقة ليغندس 5 تمديد المخلفات الحقيقية صحيح
- بعد ذلك، اضغط على مفتاح السهم لأعلى على لوحة المفاتيح حتى يتم تمييز جميع ذرات الليجاند الثلاثة بتوهج أخضر. حدد هذا التحديد للاستخدام المستقبلي بالنقر فوق في القائمة المنسدلة حدد > تحديد محدد. في القائمة المنبثقة، اكتب:
- عرض السلاسل الجانبية والعصي وعرض / ضبط جزيئات الماء بالموقع النشط: استخدم القائمة المنسدلة لعرض التحديد والوسط والتكبير على التحديد عن طريق النقر على الإجراءات > Atoms / Bonds > إظهارها لإظهارها. لتوسيط التحديد، انقر على الإجراءات > عرض. ثم، لمسح التحديد، انقر على تحديد > مسح أو انقر فوق أي مكان في المساحة الفارغة .
- إظهار تفاعلات الإنزيم مع ligands الموقع النشط: استخدم القوائم المنسدلة وانقر على تحديد > محددات معرفة من قبل المستخدم > Ligands. ثم انقر على أدوات > تحليل الهيكل > H-السندات. في النافذة المنبثقة، تأكد من تحديد الحد حسب التحديد، ويتم تعيين القائمة المنسدلة إلى مع تحديد طرف واحد على الأقل، ويتم تحديد ذرات التحديد، ثم انقر فوق موافق. لمسح التحديد، انقر على تحديد > مسح.
ملاحظة: تحقق من المربع تسمية المسافة لرؤية أطوال السندات في Å; ومع ذلك، يجعل هذا العرض مشغول جدا. وأخيرا، يمكنك تغيير لون السندات H بالنقر فوق المربع اللون وتحديد لون جديد في الإطار المنبثق. - تبسيط الهيكل: استخدام شريط أدوات الرسوم المتحركة العلوي لإخفاء الرسوم المتحركة أو النقر على القائمة المنسدلة: الإجراءات > الكرتون > إخفاء.
- وضع العلامات على السلاسل الجانبية المرتبطة بالهيدروجين: استخدم الماوس لتحديد المخلفات التي يتم ربط الهيدروجين بها (متصلة بالخطوط المتقطعة)، كما هو الحال في الخطوة 1.4. ثم، في القوائم المنسدلة، انقر على الإجراءات > تسمية > بقايا > اسم التحرير والسرد. بعد ذلك، انقر على تحديد > المحددات المعرفة من قبل المستخدم > Ligands. ثم انقر على الإجراءات > التسمية > بقايا > إيقاف. وأخيرا، قم بإلغاء تحديد، بالنقر على تحديد > مسح.
- حفظ التقديم في أي نقطة للعودة إلى العمل عليه أو مشاركته مع الآخرين: في القائمة المنسدلة انقر على ملف > حفظ. حدد موقعا، وأدخل اسم ملف، وانقر على حفظ.
ملاحظة: تأكد من تعيين التنسيق إلى: جلسة عمل ChimeraX *.cxs. - حفظ صورة للدمج أو الطباعة: استخدم الماوس أولا لتوجيه الجزيء حسب الرغبة. تغيير لون الخلفية إلى الأبيض بكتابة في سطر الأوامر:
تعيين bgColor الأبيض
وأخيرا، انقر على أيقونة اللقطة في شريط الأدوات. سيتم حفظ الصورة على سطح المكتب.
ملاحظة: يتوفر لون الخلفية أيضا في القائمة المنسدلة; على ماك، انقر على تفضيلات > UCSF ChimeraX؛ على جهاز كمبيوتر، انقر على إعدادات > المفضلة > الخلفية.
2. بروتوكول iCn3D
ملاحظة: لوحة التتبع والتحكم بالماوس:لتدوير، انقر واسحب (الماوس: انقر فوق الأيسر واسحب). للتكبير والتصغير والقرص والانتشار (الماوس: تدوير عجلة التمرير). للترجمة (أي تحريك البنية بأكملها) انقر واسحب بإصبعين (الماوس: انقر بزر الماوس الأيمن واسحب). لإعادة التوسيط، مرر الماوس فوق عرض في القوائم المنسدلة العلوية، ثم انقر فوق تحديد المركز.
- تحميل البنية في iCn3D: انتقل إلى عارض البنية ثلاثية الأبعاد المستند إلى الويب iCn3D واكتب 3FGU في مربع معرف الإدخال MMDB أو PDB لتحميل الملف.
- تحديد ligands في الموقع النشط: تحوم فوق التحليل في القائمة المنسدلة، ومن ثم انقر على Seq. والتعليقات التوضيحية. تظهر التسلسلات، في هذه الحالة البروتينات والكيميائية/الأيونات/المياه، في جدول مكدس. انتقل لأسفل لرؤية الموقع النشط ligands ANP، BGC، وMg المدرجة. في المشاهد هيكل، تحوم فوق ligands في الموقع النشط (كما هو مبين العصي في وسط الرسوم المتحركة البروتين) لعرض أسمائهم.
- ضبط التمثيل: لا يلزم إجراء تعديلات أولية لهذا البروتوكول.
- اختيار المخلفات داخل 5 Å لتحديد موقع نشط: لتحديد ligands، استخدم القائمة المنسدلة حدد وانقر على حدد على 3D. تأكد من فحص المخلفات.
- لتحديد ligands، اضغط باستمرار على زر ALT على جهاز كمبيوتر أو زر الخيار على ماك، وانقر على ليغاند الأولى (على سبيل المثال، BCG)باستخدام الماوس أو لوحة التتبع. ثم اضغط على التحكم وانقر على ANP وMG ligands لإضافتها إلى الاختيار.
ملاحظة: سيتم تمييز ligands باللون الأصفر كما يتم تحديدها. - حفظ هذا التحديد باستخدام القائمة المنسدلة: انقر على تحديد > حفظ التحديد واستخدام لوحة المفاتيح لإدخال اسم في النافذة المنبثقة (على سبيل المثال، 3Ligands)،ثم انقر على حفظ. ستظهر الآن النافذة المنبثقة Select Sets .
ملاحظة: إذا كان التحديد غير صحيح، انقر فوق تحديد > تحديد واضح. - حدد المخلفات داخل 5 Å من ligands: في القائمة المنسدلة، انقر على حدد > عن طريق المسافة. في القائمة المنبثقة التي تظهر، قم بتغيير العنصر الثاني (Sphere مع نصف قطر)، إلى 5 Å عن طريق الكتابة في الكتلة. انقر على الكلمة محاصر عرض، ثم أغلق النافذة بالنقر على علامة الصليب في الزاوية اليمنى العليا.
ملاحظة: في القائمة المنبثقة التي تظهر في الخطوة 2.4.3، اترك المجموعة الأولى مع الإدخال "المحدد" و المجموعة الثانية ك "غير محدد". لاحظ أن الذرات/ الهياكل داخل 5 Å تصبح مميزة بتوهج أصفر عند النقر فوق العرض. - حفظ موقع 5 Å النشط باستخدام القائمة المنسدلة: مرر الماوس فوق تحديد وانقر على حفظ التحديد، أدخل اسما في النافذة المنبثقة باستخدام لوحة المفاتيح (على سبيل المثال ، 5Ang)، وانقر على حفظ.
- إنشاء التالي تحديد جديد يجمع بين مجموعتين (5Ang و 3Ligands): في القائمة المنبثقة تحديد مجموعات، ctrl-انقر فوق (PC) أو الأمر انقر فوق (ماك) 5Ang و 3Ligands. انقر على تحديد > حفظ التحديد، استخدم لوحة المفاتيح لكتابة اسم (على سبيل المثال ، 5AFull)، ثم انقر فوق حفظ.
- لتحديد ligands، اضغط باستمرار على زر ALT على جهاز كمبيوتر أو زر الخيار على ماك، وانقر على ليغاند الأولى (على سبيل المثال، BCG)باستخدام الماوس أو لوحة التتبع. ثم اضغط على التحكم وانقر على ANP وMG ligands لإضافتها إلى الاختيار.
- إظهار تفاعلات الإنزيم مع ليغاندس الموقع النشط مثل روابط الهيدروجين: تحوم فوق التحليل في القائمة المنسدلة وانقر على التفاعلات. ستظهر قائمة منبثقة شاملة لجميع التفاعلات غير التكافؤية.
- قم بإلغاء تحديد كل شيء باستثناء مربعات الاختيار "روابط الهيدروجين" و "جسر الملح / الأيونية". انقر على 3Ligands لتحديد المجموعة الأولى و5Ang عن المجموعة الثانية. انقر على النص محاصر يقرأ تفاعلات العرض ثلاثي الأبعاد. أغلق النافذة بالنقر على علامة الصليب في الزاوية اليمنى العليا.
ملاحظة: من المفترض أن يمثل الاتصال/التفاعلات تفاعل ثنائي القطب الناجم عن ثنائي القطب، مما يجعل الشاشة مشغولة في كثير من الأحيان. إذا رغبت في ذلك، تغيير المسافة لأي نوع من التفاعل. - لإظهار روابط الهيدروجين فقط، انقر على 5Afull في الإطار المنبثق مجموعات مختارة. ثم مرر الماوس فوق التحليل في القائمة المنسدلة، ثم انقر على Chem. الربط > إظهار.
- قم بإلغاء تحديد كل شيء باستثناء مربعات الاختيار "روابط الهيدروجين" و "جسر الملح / الأيونية". انقر على 3Ligands لتحديد المجموعة الأولى و5Ang عن المجموعة الثانية. انقر على النص محاصر يقرأ تفاعلات العرض ثلاثي الأبعاد. أغلق النافذة بالنقر على علامة الصليب في الزاوية اليمنى العليا.
- عرض السلاسل الجانبية والعصي وعرض / ضبط جزيئات الماء بالموقع النشط: استخدم قائمة منبثقة مجموعات مختارة وانقر على 5AFull. ثم، في القوائم المنسدلة، انقر على نمط > السلاسل الجانبية > عصا. لتطبيق CPK تلوين انقر على اللون > الذرة. وأخيرا، انقر على نمط > المياه > المجالات (إذا كنت تفضل جزيئات الماء أكبر).
- تبسيط الهيكل: في القائمة المنبثقة مجموعات حدد، انقر على 5AFull. ثم، في القوائم المنسدلة، انقر على عرض > عرض التحديد (لمجرد رؤية موقع الربط 5AFull). بعد ذلك ، انقر على Style > البروتينات > Stick (لإظهار سلسلة البروتين كعصا بدلا من الشريط).
- لتلوين ذرات الكربون في الليجاند بلون متناقض، انقر على المواد الكيميائية في النافذة المنبثقة Select Sets. ثم، في القائمة المنسدلة، انقر على عرض > عرض التحديد. انقر بعد ذلك على حدد > حدد على 3D (تأكد من التحقق من "ذرة"). باستخدام عناصر التحكم الموضحة في الخطوة 2.4.1، استخدم الماوس ولوحة المفاتيح لتحديد كافة ذرات الكربون في BGC و ANP. ثم، في القائمة المنسدلة انقر على اللون > يونيكولور > سماوي > سماوي.
- لإعادة عرض الموقع النشط بأكمله، استخدم الإطار المنبثق Select Sets للنقر على 5AFull. ثم، في القائمة المنسدلة، انقر على عرض > عرض التحديد.
- وضع العلامات ligands والسلاسل الجانبية المستعبدين الهيدروجين: استخدام تحديد مجموعات الإطار المنبثق لتحديد Interface_all،ومن ثم، في القائمة المنسدلة، انقر على تحليل > التسمية > لكل بقايا ورقم.
ملاحظة: يجب عليك إعادة تحديد كل Residue & Number في كل مرة ترغب فيها في إضافة تسمية، على الرغم من أن عنصر القائمة سيتم التحقق منه بالفعل من تسمية سابقة. - حفظ التقديم في أي نقطة للعودة إلى العمل عليه أو مشاركته مع الآخرين: في القائمة المنسدلة، انقر على ملف > مشاركة الارتباط. نسخ عنوان URL القصير (على سبيل المثال: https://structure.ncbi.nlm.nih.gov/icn3d/share.html?r83NqCz41bu7cmcs8) ولصقه على متصفح.
- حفظ صورة لتضمينها أو طباعتها: في القائمة المنسدلة، انقر على تحديد > تبديل تمييز. ثم انقر على نمط > الخلفية > الأبيض. وأخيرا، انقر على ملف > حفظ الملفات > صورة PNG iCn3D واختيار الحجم المطلوب.
3. بروتوكول جومول
ملاحظة: لوحة التتبع والتحكم بالماوس: لتدوير، انقر واسحب (الماوس: انقر فوق الأيسر واسحب). للتكبير: مرر عموديا باستخدام إصبعين (الماوس: تحول + النقر الأيسر + اسحب عموديا). لترجمة (أي، نقل الهيكل بأكمله) التحكم + البديل + انقر والسحب (PC)، والتحكم + الخيار + انقر واسحب (ماك). لإعادة التوسيط: قم بإزاحة + انقر نقرا مزدوجا في المساحة الفارغة من إطار عارض البنية.
- تحميل البنية في Jmol: استخدم القائمة المنسدلة في أعلى واجهة المستخدم الرسومية لإعداد مساحة العمل مع الهيكل بالنقر فوق File > Console. ثم انقر على ملف > الحصول على PDB. في النافذة المنبثقة، اكتب: 3fgu
ثم انقر فوق موافق.
ملاحظة: بالتناوب، استخدم وحدة التحكم Jmol لتحميل البنية، بكتابة: تحميل = 3fgu
ملاحظة: بعد إدخال أي أمر خط مكتوب، اضغط على العودة على لوحة المفاتيح لتنفيذه. - ضبط التمثيل: افتح القائمة المنبثقة بالنقر بزر الماوس الأيمن (أو التحكم + النقر) في أي مكان في إطار عارض البنية.
- لتغيير البروتين إلى تمثيل الرسوم المتحركة، في القائمة المنبثقة، انقر على تحديد > اختيار هالات. بعد ذلك، انقر على حدد > بروتين > الكل. وأخيرا، انقر على نمط > مخطط > الكرتون.
ملاحظة: هالات التحديد يضع مخطط أصفر (توهج) حول كافة الذرات المحددة. - استخدم القائمة المنسدلة العلوية لإخفاء المياه بالنقر على عرض > حدد > الماء. بعد ذلك، انقر على عرض > Atom > None، وأخيرا انقر على عرض > حدد > None.
- لتغيير البروتين إلى تمثيل الرسوم المتحركة، في القائمة المنبثقة، انقر على تحديد > اختيار هالات. بعد ذلك، انقر على حدد > بروتين > الكل. وأخيرا، انقر على نمط > مخطط > الكرتون.
- تحديد ligands في الموقع النشط: استخدام الماوس للتكبير على الموقع النشط، ومن ثم استخدام الأوامر في الخطوات الفرعية لعرض ligands والعصي.
ملاحظة: تظهر أسماء Ligand في وحدة تحكم Jmol عند تحميل الملف. يمكنك أيضا عرض أسماء ligand ملزمة باستخدام القائمة المنبثقة، من خلال النقر على تحديد > hetero > من قبل HETATM.- تحوم فوق ligands مع الماوس لعرض أسمائهم. الموقع النشط هو بالقرب من مركز الهيكل. ligands MG، BGC، و ANP تقع في الموقع النشط.
- حدد ليغاندس BCG و ANP: باستخدام وحدة التحكم Jmol، اكتب:
اختر BGC, ANP - لعرض ligands BCG و ANP والعصي، واستخدام القائمة المنبثقة وانقر على نمط > مخطط > العصي.
- اختيار المخلفات داخل 5 Å لتحديد موقع نشط: في وحدة التحكم Jmol، اكتب الأمر التالي لتحديد الذرات ضمن 5 Å من الليجانات الثلاثة:
حدد داخل (5، (bgc، anp، ملغ))- لتحديد بقايا الأحماض الأمينية الكاملة، اكتب ما يلي في وحدة التحكم واضغط على Enter
تحديد داخل (مجموعة، محددة)
ملاحظة: وحدة Jmol هي أفضل طريقة لتحديد المخلفات داخل 5 Å.
- لتحديد بقايا الأحماض الأمينية الكاملة، اكتب ما يلي في وحدة التحكم واضغط على Enter
- عرض السلاسل الجانبية والعصي وعرض / ضبط جزيئات الماء بالموقع النشط: انقر بزر الماوس الأيمن لطرح القائمة المنبثقة، وتحوم فوق نمط > مخطط > العصي.
ملاحظة: الخطوة 3.5 يظهر السلاسل الجانبية الموقع النشط في تمثيل العصا. سيظل هناك بعض الهالات الفارغة في الهيكل التي تمثل جزيئات الماء في الموقع النشط.- في وحدة تحكم Jmol إعادة تنفيذ الأمر التالي:
حدد داخل (5، (bgc، anp، ملغ))
ملاحظة: لإعادة تنفيذ أمر، انقر داخل وحدة التحكم، ثم استخدم مفاتيح الأسهم على لوحة المفاتيح حتى يظهر هذا الأمر، وانقر فوق إدخال لإعادة تنفيذه. - لعرض ذرات جزيء الماء، قم بإزالة الليجاند والبروتين من التحديد بكتابة الأمرين التاليين:
حدد إزالة البروتين الجماعي
حدد إزالة مجموعة hetero وليس الماء - لعرض جزيئات الماء، انقر على القائمة المنسدلة عرض. تحوم فوق أتوم وانقر على 20٪ فان دير وال . سوف الأيونات المغنيسيوم الأخضر لا تزال تظهر والعصي. عرض أيون المغنيسيوم في تمثيل المجال الأكثر شيوعا عن طريق كتابة الأوامر التالية في وحدة التحكم Jmol:
حدد ملغ
ملء الفراغ 50٪ - إعادة تلوين ليغاندس لتمييزها عن البروتين: في وحدة التحكم Jmol اكتب ما يلي لتنفيذ أمر يعيد تلوين الليجانات في نظام ألوان أخف:
حدد (bgc, anp) والكربون; اللون [211,211,211]
حدد (bgc, anp) والأوكسجين; اللون [255,185,185]
حدد (bgc, anp) والنيتروجين; اللون [150,210,255]
حدد (bgc, anp) والفوسفور; اللون [255,165,75]
حدد ملغ; لون باليغرين
- في وحدة تحكم Jmol إعادة تنفيذ الأمر التالي:
- إظهار تفاعلات الإنزيم مع ligands الموقع النشط: باستخدام وحدة التحكم Jmol تنفيذ كل سطر من الأمر التالي:
تعريف ليغبيند (ANP، BGC، MG)
حدد داخل (5، (bgc، anp، ملغ))
حدد إزالة مجموعة hetero وليس الماء- لإظهار خطوط لتوضيح روابط الهيدروجين، اكتب هذا الأمر في وحدة التحكم Jmol:
ربط 3.3 (ligbind و (الأكسجين أو النيتروجين)) (مختارة و (الأكسجين أو النيتروجين)) تبختر الأصفر
ثم، تعديل سمك الأسطر بكتابة الأمر التالي في وحدة التحكم:
حدد الكل؛ الدعامة 0.1; تحديد لا شيء
- لإظهار خطوط لتوضيح روابط الهيدروجين، اكتب هذا الأمر في وحدة التحكم Jmol:
- تبسيط الهيكل: لإخفاء الرسوم المتحركة للبروتين ومسح الاختيار، في وحدة التحكم Jmol، اكتب:
حدد الكل؛ الرسوم المتحركة قبالة؛ تحديد لا شيء - وضع العلامات ligands والسلاسل الجانبية المستعبدين الهيدروجين: في النافذة المنبثقة، انقر على تعيين التقاط > حدد الذرة. انقر على ذرة في واحدة من بقايا الهيدروجين المستعبدين. تظهر الأحماض الأمينية وأرقام البقايا في وحدة التحكم. ثم استخدم وحدة التحكم لكتابة تسمية، على سبيل المثال:
تسمية غلو-256 - حفظ التقديم في أي نقطة للعودة إلى العمل عليه أو مشاركته مع الآخرين: في القائمة العلوية، انقر على أيقونة الكاميرا. اكتب اسم ملف وحدد موقعا لحفظه.
ملاحظة: يحتوي ملف JPEG المصدر (.jpg) على معلومات لكل من صورة كما تظهر في إطار العرض في وقت التصدير، بالإضافة إلى الحالة الحالية للطراز. لإعادة تحميل النموذج، افتح Jmol واسحب ملف JPEG المحفوظ إلى نافذة عرض Jmol. - حفظ صورة لتضمينها أو طباعتها: في وحدة تحكم Jmol، يمكنك إعادة تلوين الخلفية إلى اللون الأبيض، وذلك بكتابة:
خلفية بيضاء
كما هو الحال في الخطوة 3.9، انقر على أيقونة الكاميرا وحفظ الملف.
4. بروتوكول PyMOL
ملاحظة: لوحة التتبع والتحكم بالماوس: لتدوير، انقر واسحب (الماوس: انقر فوق الأيسر واسحب). للتكبير، قرصة وانتشار (الماوس: انقر بزر الماوس الأيمن واسحب). لترجمة (أي، نقل الهيكل بأكمله)، والتحكم + انقر واسحب (الماوس: الأمر + انقر فوق اليسار والسحب). لإعادة التوسيط انتقل إلى لوحة الكائن الأيمن وانقر على A > الشرق أو المركز.
- تحميل البنية في PyMOL: في سطر الأوامر بالقرب من أعلى واجهة المستخدم الرسومية (مسبوقة ب "PyMOL>")، اكتب:
جلب 3FGU
ملاحظة: بعد إدخال أي أمر خط مكتوب، اضغط على العودة على لوحة المفاتيح لتنفيذه. - ضبط التمثيل: في لوحة الأسماء/الكائنات على الجانب الأيمن من نافذة PyMOL، إلى يمين "3FGU" انقر على H > Waters.
- تحديد السلاسل في الموقع النشط: قم بتشغيل عارض التسلسل أولا بالنقر على القائمة المنسدلة العلوية: عرض تسلسل >.
- مرر الشريط الرمادي إلى اليمين حتى تجد أسماء ligand (BCG، ANP، MG، K).
ملاحظة: هناك تمثيلين، شريط الكرتون والعصي. يتم عرض ليغاندس والعصي. تأكد من تعيين وضع التحديد في عناصر تحكم الماوس على اللوحة اليمنى السفلية إلى وضع Residue و3-Button Viewing بالنقر فوق هذه الأسماء للتبديل عبر الخيارات. - باستخدام الماوس، تدوير والتكبير لجعل ligands مرئية.
- مرر الشريط الرمادي إلى اليمين حتى تجد أسماء ligand (BCG، ANP، MG، K).
- اختيار المخلفات داخل 5Å لتحديد موقع نشط: لتحديد الليجاند في الموقع النشط، انقر على كل واحد(BCG، ANP، MG)في عارض الهيكل. تحديد جديد ينبثق في لوحة الأسماء/الكائن; إلى يمين هذا الكائن الجديد المسمى "سيل"، انقر على الزر A، ثم انقر فوق إعادة تسمية في القائمة المنبثقة.
ملاحظة: لمسح تحديد غير مرغوب فيه، انقر فوق المساحة الفارغة في عارض البنية لإلغاء التحديد.- باستخدام لوحة المفاتيح، احذف الحروف "sele" التي تظهر في الجانب الأيسر العلوي من إطار عارض البنية، وفي مكانها، اكتب:
يغاندس
ملاحظة: يمكن القيام بالخطوات 4.4-4.4.1 باستخدام سطر الأوامر; نوع:
سيل ليغاندس، ريسن BGC + ANP + MG - استخدم هذا التحديد لتحديد المنطقة المحيطة ب ligands عن طريق تكراره أولا ، انقر على ligands > A > مكررة. ثم انقر على sel01 > إعادة تسمية >
باستخدام لوحة المفاتيح، حذف الحروف "se101" واكتب:
نشط - تعديل هذا التحديد لإظهار المخلفات داخل 5 Å: في لوحة الأسماء / الكائن، انقر على > النشطة > تعديل > توسيع > من قبل 5 A، بقايا. ثم، لإظهار هذه المخلفات والعصي، انقر على نشط > S > عرق السوس > العصي. وأخيرا، انقر فوق في المساحة الفارغة في "عارض البنية" لمسح التحديد.
ملاحظة: الخطوة 4.4.3 يمكن القيام به باستخدام سطر الأوامر، اكتب:
sele نشطة، byres جميع ضمن 5 من ligands
إظهار العصي، نشط
- باستخدام لوحة المفاتيح، احذف الحروف "sele" التي تظهر في الجانب الأيسر العلوي من إطار عارض البنية، وفي مكانها، اكتب:
- عرض السلاسل الجانبية والعصي وعرض / ضبط جزيئات الماء بالموقع النشط: في لوحة الأسماء / وجوه انقر على ligands > مكررة >. لإعادة تسمية التحديد، انقر فوق Sel02 > تحديد إعادة تسمية >. حذف الحروف في القائمة إعادة تسمية التي تظهر في أعلى يمين عارض البنية، واكتب:
active_water- لضبط التحديد الجديد لاحتواء جزيئات الماء بالموقع النشط ، انقر على active_water > A > تعديل > حول > الذرات داخل 4 Angstroms. لتعديل هذا الحد وإلى جزيئات الماء، انقر على active_water > تعديل > > تقييد > إلى المذيبات. وأخيرا، انقر على active_water > > مسبقا > الكرة والعصا.
ملاحظة: واجهة المستخدم الرسومية يسمح الاختيار داخل 4 Å; أوامر خط تسمح باختيار مسافة أكثر ملاءمة من 3.3 ألف لجزيئات الماء الترابط الهيدروجين. فان دير وال شعاعي من المجالات لا يمكن تعيينها في واجهة المستخدم الرسومية، ولكن اختيار "الكرة والعصا" هو ما يقرب من 0.5 Å.
ملاحظة: قد يتم تنفيذ الخطوات 4.5-4.5.1 باستخدام سطر الأوامر بكتابة كل سطر من التعليمات البرمجية التالية:
حدد active_water و ((ligands)حول 3.3) و (resn HOH)
إظهار المجالات، active_water
تغيير active_water، vdw =0.5
اعاده
- لضبط التحديد الجديد لاحتواء جزيئات الماء بالموقع النشط ، انقر على active_water > A > تعديل > حول > الذرات داخل 4 Angstroms. لتعديل هذا الحد وإلى جزيئات الماء، انقر على active_water > تعديل > > تقييد > إلى المذيبات. وأخيرا، انقر على active_water > > مسبقا > الكرة والعصا.
- إظهار تفاعلات الإنزيم مع ليغاندس الموقع النشط. تكبير على الموقع النشط من خلال النقر على نشط > تكبير >. للعثور على الاتصالات القطبية بين ligands والموقع النشط، انقر على ligands > > البحث عن > الاتصالات القطبية > إلى أي ذرات. إظهار المسافات كعلامات بالنقر على تسميات > S ligands_polar_contacts >.
- تبسيط الهيكل: إخفاء الرسوم المتحركة للبروتين ، الذي يخفي الجزء من البروتين غير الموجود في الموقع النشط ، من خلال النقر على 3FGU > H > Cartoon في لوحة الأسماء / الكائن. بعد ذلك، قم بإخفاء تسميات طول رابطة الهيدروجين بالنقر على ligands_polar_contacts > H > Labels في لوحة الأسماء/الكائنات.
- لتلوين ليغاندس لتمييزها عن البروتين، انقر على ليغندس > C > حسب العنصر > CHNOS وحدد الخيار حيث "C" هو سماوي (أزرق فاتح).
ملاحظة: الخطوة 4.7.1 قد يتم تنفيذ باستخدام سطر الأوامر. نوع:
لون سماوي، ليغندس
اللون الذري، ليجاندس و!elem C
- لتلوين ليغاندس لتمييزها عن البروتين، انقر على ليغندس > C > حسب العنصر > CHNOS وحدد الخيار حيث "C" هو سماوي (أزرق فاتح).
- وضع العلامات ligands والسلاسل الجانبية المستعبدين الهيدروجين: في لوحة الأسماء / الكائن، على أزرار إلى يمين أي اسم الكائن، انقر على > النشط L > المخلفات.
- حفظ التقديم في أي نقطة للعودة إلى العمل عليه أو مشاركته مع الآخرين: في القائمة المنسدلة انقر على ملف > حفظ الجلسة باسم. ثم حدد موقعا في النافذة المنبثقة واكتب اسم ملف وانقر فوق حفظ.
- حفظ صورة لتضمينها أو طباعتها: أولا، قم بتغيير الخلفية إلى اللون الأبيض في القائمة المنسدلة بالنقر على عرض > الخلفية > الأبيض. تصدير الصورة كملف جديد، عن طريق النقر على ملف > تصدير صورة كما > PNG.
النتائج
بروتوكول نفذت بنجاح لكل من البرامج سيؤدي إلى نموذج الجزيئية التكبير في الموقع النشط، مع بقايا الموقع النشط و ligands تظهر العصي، والكرتون البروتين المخفية، و ligands عرض مع نظام الألوان المتناقضة. وينبغي تصنيف بقايا الأحماض الأمينية المتفاعلة مع معرفاتها، والترابط الهيدروجيني والتفاعلات الأيونية المعروضة مع الخطوط. يمكن تحديد وجود هذه الميزات من خلال الفحص البصري للنموذج.
لتسهيل هذا الفحص وتمكين المستخدم من تحديد ما إذا كان قد قام بتنفيذ خطوات البروتوكول بشكل صحيح، قدمنا أرقاما متحركة تقدم صورة للبنية التالية لكل خطوة. بالنسبة إلى ChimeraX و iCn3D و Jmol و PyMOL ، فإن هذا موضح في الأشكال 7-10، على التوالي.
الشكل 7: إخراج بروتوكول ChimeraX. رسم متحرك يوضح الخطوات 1.1-1.8 من بروتوكول ChimeraX. يرجى الضغط هنا لتحميل هذا الرقم.
الشكل 8: إخراج بروتوكول iCn3D. رسم متحرك يوضح الخطوات 2.1-2.8 من بروتوكول iCn3D. يرجى الضغط هنا لتحميل هذا الرقم.
الشكل 9: إخراج بروتوكول Jmol. رسم متحرك يوضح الخطوات 3.1-3.8 من بروتوكول Jmol. يرجى الضغط هنا لتحميل هذا الرقم.
الشكل 10: إخراج بروتوكول PyMOL. رسم متحرك يوضح الخطوات 4.1-4.8 من بروتوكول PyMOL. يرجى الضغط هنا لتحميل هذا الرقم.
الخطأ الأكثر شيوعا التي يمكن أن تؤثر على نتائج هذه البروتوكولات هو اختيار خاطئ، مما أدى إلى جزء من البنية التي يتم عرضها في تقديم غير مرغوب فيه. هذا عادة نتيجة للخطأ بالنقر فوق، إما على البنية نفسها، أو في أحد أزرار القائمة العرض. ومن الأمثلة على النتيجة دون المستوى الأمثل نموذج يحتوي على بقايا خارج الموقع النشط المعروض كعصي. يمكن للمستخدم البدء في تحليل ما إذا كان هذا الخطأ قد حدث عن طريق فحص بصريا المخلفات المعروضة والعصي وضمان أنها على مقربة من ligands الموقع النشط. طريقة متقدمة لتقييم ما إذا كانت المخلفات المعروضة ضمن 5Å من ligands الموقع النشط هو استخدام أدوات القياس المضمنة في كل برنامج لقياس المسافة بين ligand القريبة وبقايا الموقع النشط. أدوات القياس خارج نطاق هذه المخطوطة؛ ومع ذلك، فإننا نشجع المستخدمين المهتمين لاستكشاف العديد من الدروس على الانترنت بالتفصيل هذا النوع من التحليل.
نقدم مثالا محددا لتنفيذ دون المستوى الأمثل لهذا البروتوكول ، الناتج عن سوء النقر على لوحة الأسماء / الكائنات في PyMOL. يعرض هذا الخطأ البروتين بأكمله ك عصي، بدلا من إظهار الموقع النشط فقط باستخدام هذا التمثيل، كما هو موضح في الشكل 11.
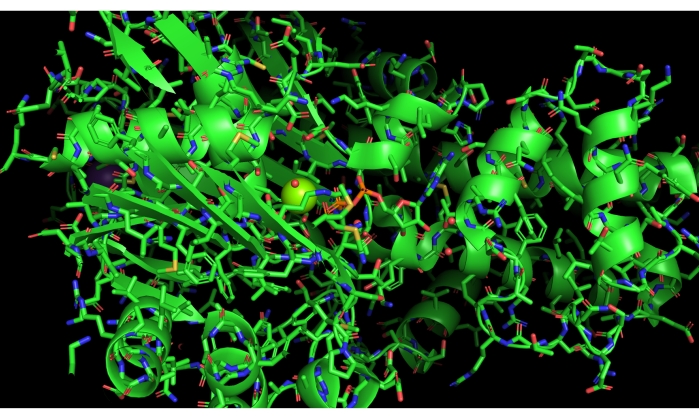
الشكل 11: النتيجة السلبية. مثال على نتيجة سالبة. سوء اختيار الرسوم المتحركة الكاملة في PyMOL وعرض العصي. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
لاستكشاف الأخطاء وإصلاحها، سيحتاج المستخدم إلى إخفاء العصي للطراز بأكمله (المسمى 3FGU في لوحة الأسماء/الكائنات)، ثم إظهار تمثيل العصا فقط التحديد المسمى "نشط"، باستخدام أزرار/أوامر الإخفاء وإظهارها في PyMOL. استرداد النموذج من هذا النوع من الخطأ واضحة نسبيا بمجرد أن يكون المستخدم قادرا على إنشاء التحديدات المناسبة لأجزاء مختلفة من النموذج وعرضها وإخفائها بشكل فعال. ومن المغري لإعادة تشغيل البروتوكول والعمل من خلال الخطوات في وقت آخر; ومع ذلك، فإننا نشجع المستخدم على عدم الخوف من الذهاب "خارج البرنامج النصي" وتجربة النموذج. من خلال تجربتنا، العمل من خلال أخطاء العرض يسهل التقدم في فهم برنامج النمذجة.
يتم عرض جنبا إلى جنب من الإخراج النهائي من بروتوكول تنفيذها بنجاح لكل برنامج في الشكل 12. وتوجه وجهات النظر بالمثل للسماح للمستخدم لمقارنة مظهر النماذج التي تم إنشاؤها في برامج مختلفة.
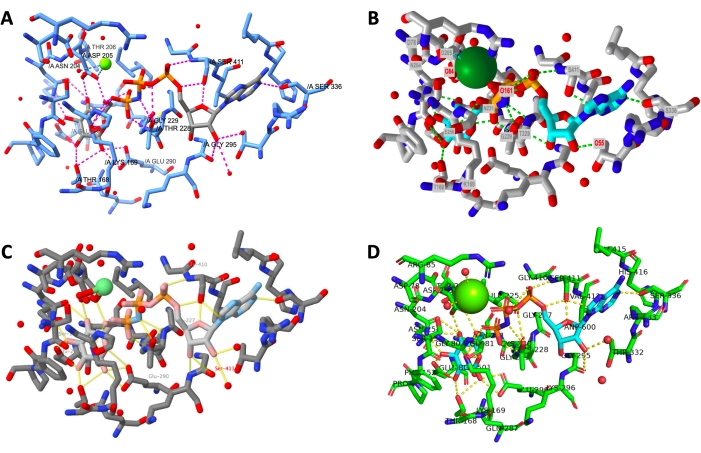
الشكل 12: مقارنة الهيكل النهائي عبر البرامج. مقارنة بنية كل عرض موقع نشط في نهاية البروتوكول. ج: كيميراإكس، ب: iCn3D، C: Jmol، D: PyMOL. تتضمن تسمية الموقع النشط PyMOL جميع مخلفات الموقع النشطة والليغاند. أما المخرجات الأخرى فلم تحمل سوى سلاسل جانبية مرتبطة بالهيدروجين. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
Discussion
يحدد هذا البروتوكول عملية من عشر خطوات لنمذجة موقع نشط إنزيم ، وتطبيقها على أربعة برامج شعبية للنمذجة الجزيئية الحيوية. الخطوات الحاسمة للبروتوكول هي: تحديد الليجاند في الموقع النشط ، واختيار المخلفات داخل 5 Å لتحديد موقع نشط ، وإظهار تفاعلات الإنزيم مع ligands الموقع النشط. التمييز بين ليغاندس ذات الصلة إلى وظيفة البيولوجية أمر بالغ الأهمية، وهذا يسمح للمستخدم لتحديد بقايا الأحماض الأمينية داخل 5 Å التي يمكن أن تلعب دورا في ربط ليغاندس. وأخيرا، فإن استخدام البرنامج لعرض التفاعلات الجزيئية يسمح للمستخدم بتطوير المهارات اللازمة لفهم التفاعلات الجزيئية التي تعزز الربط.
وهناك قيود على بروتوكولات النمذجة الجزيئية المستندة إلى الكمبيوتر هو الاعتماد على أوامر محددة وبناء الجملة. وفي حين أن البروتوكولات الكيميائية الحيوية قد تكون متسامحة مع التغيرات الطفيفة في الإجراءات، فإن التحقيقات القائمة على الحاسوب قد تسفر عن منتجات نهائية مختلفة إلى حد كبير إذا لم يتم الالتزام بهذا الإجراء عن كثب. وهذا مهم بشكل خاص عند استخدام واجهات سطر الأوامر حيث بناء الجملة الخاصة بالبرنامج مطلوب لتحقيق إخراج معين، ويمكن أن يؤدي تغيير ضئيلة ظاهريا في علامات الترقيم أو الأحرف الاستهلالية أمر إلى فشل. هناك العديد من ويكي وأدلة لكل برنامج، حيث يمكن للمستخدم العثور على وإصلاحها إدخال سطر الأوامر؛ يجب على المستخدم الانتباه بعناية إلى تفاصيل بناء جملة الأمر. على الرغم من أن معظم برامج التصور الجزيئي تتضمن أوامر التراجع ، نظرا لتعقيد الواجهات ، إلا أن الأمر التراجع لا يعكس دائما بأمانة الخطوة الأخيرة المنفذة. لذلك، يتم تشجيع حفظ حالة العمل الحالية غالبا، خاصة للمستخدمين الجدد.
ويمكن أن تنشأ قيود أخرى من البيانات المستخدمة لإنشاء النموذج نفسه. في حين أن المعايير الكامنة في بنك بيانات البروتين تضمن مستوى معين من الاتساق ، فإن مستخدمي برامج التصور الجزيئي غالبا ما يواجهون تأثيرات غير متوقعة في تقديم البروتين. أولا، يتم تحديد معظم الهياكل باستخدام التصوير البلوري بالأشعة السينية، والذي يوفر نموذجا واحدا للبروتين. ومع ذلك، غالبا ما تتكون هياكل NMR من نماذج متعددة يمكن تصورها واحدة في كل مرة. ثانيا، قد تحتوي الهياكل التي يتم تحديدها من البلورات أو تجارب المجهر الإلكتروني المبرد على ذرات لا يمكن توضيح موضعها وتظهر كثغرات في تمثيلات معينة للبروتين. قد تحتوي هياكل البروتين على تشكيلات بديلة من السلاسل الجانبية ، والتي تظهر ، عند عرضها في تقديم العصا ، كاثنين من المجموعات البارزة من نفس العمود الفقري للأحماض الأمينية. حتى الأقسام القصيرة من العمود الفقري قد يكون لها مثل هذه الحسابات البديلة ، وفي بعض الأحيان يتم فرض الليجاند في الموقع النشط في أكثر من تشكيل ملزم واحد.
بالنسبة للهيكل البلوري، تتضمن الإحداثيات ثلاثية الأبعاد المودعة جميع مكونات الوحدة غير المتماثلة، والتي توفر معلومات كافية لإعادة إنتاج الوحدة المكررة لبلورة البروتين. في بعض الأحيان، سوف يحتوي هذا الهيكل على سلاسل بروتين إضافية مقارنة بالشكل النشط بيولوجيا للبروتين (على سبيل المثال، متحولة الهيموغلوبين الجنينية، PDB ID: 4MQK). وعلى العكس من ذلك، قد لا تقوم بعض البرامج بتحميل جميع سلاسل الوحدة النشطة بيولوجيا تلقائيا. على سبيل المثال، يقوم البروتيز الرئيسي سارس-CoV2 (PDB ID: 6Y2E) بتحميل نصف جهاز التعتيم النشط بيولوجيا (المؤلف من سلسلتين بروتينيتين) عند جلبه باستخدام الأوامر الموضحة في هذا البروتوكول في ChimeraX و PyMOL و Jmol. على الرغم من أن التعديل الطفيف للأمر سيتم تحميل dimer النشط بيولوجيا، قد لا يكون هذا الاعتبار مباشرة لمستخدم برنامج النمذجة المبتدئ. وهناك مسألة مختلفة يمكن أن تنشأ في تحديد الموقع النشط أو الركيزة نفسها. يتم إجراء التجارب البلورية باستخدام مجموعة متنوعة من الجزيئات ، والتي يمكن أن تكون على غرار الهيكل النهائي. فعلى سبيل المثال، قد تربط جزيئات الكبريتات مواقع ربط الفوسفات في الموقع النشط، أو قد تربط مناطق أخرى غير ذات صلة بالآلية. قد تحجب هذه الجزيئات التحديد الصحيح للموقع النشط نفسه وقد تشير حتى إلى الطالب بأنها جزء من الآلية.
من المفترض أن المستخدم يرغب في تطبيق هذا الإجراء على مواقع نشطة/ربط أخرى. لتطبيق هذا البروتوكول في العمل المستقبلي الذي ينطوي على تحليل المواقع النشطة البروتين الجديد، سوف يحتاج المستخدم لتحديد أي من ligands ملزمة ذات الصلة لوظيفة. لا ترتبط بعض الليجاندات بوظيفة البروتين ، وبدلا من ذلك هي نتيجة لظروف المذيبات أو التبلور المستخدمة لإجراء التجربة (على سبيل المثال ، أيون البوتاسيوم الموجود في نموذج 3FGU). يجب تحديد الخطوط الرئيسية من خلال استشارة المخطوطة الأصلية. مع الممارسة ، وعند الاقتضاء ، فهم بناء الجملة الأمر الخط ، فإن المستخدم تكون قادرة على تطبيق بروتوكول لبرنامج النمذجة المطلوبة إلى أي موقع نشط انزيم ، ونموذج الجزيئات الكبيرة الأخرى من اختيارهم.
تحديد وتحليل الركائز ملزمة و ligands أمر أساسي لتوضيح الآليات الجزيئية والجهود القائمة على هيكل تصميم الأدوية، والتي أدت مباشرة إلى تحسينات في علاجات المرض، بما في ذلك متلازمة نقص المناعة المكتسبة (الإيدز) و COVID-1947،48،49،50،51،52 . في حين أن برامج التصور الجزيئي الفردية تقدم واجهات وتجارب مستخدم مختلفة ، فإن معظمها يوفر ميزات قابلة للمقارنة. من المهم لتطوير محو الأمية التصور الجزيئي الحيوي أن طلاب الكيمياء الحيوية المستوى العلوي تصبح مألوفة مع التصور هيكل والأدوات اللازمة لتوليد مثل هذه الصور4،20،53. وهذا يسمح للطلاب بتجاوز تفسير الصور ثنائية الأبعاد في الكتب المدرسية والمقالات الصحفية وتطوير فرضياتهم الخاصة بسهولة أكبر من البيانات الهيكلية 54 ، والتي ستعدالعلماء الناميينلمعالجة قضايا الصحة العامة المستقبلية وتحسين فهم العمليات الكيميائية الحيوية.
باختصار، يفصل هذا البروتوكول نمذجة الموقع النشط باستخدام أربعة برامج نمذجة حرة حرة رائدة. مجتمعنا ، BioMolViz ، تتبنى نهجا غير البرمجيات محددة للنمذجة الجزيئية الحيوية. تجنبنا على وجه التحديد نقد أو مقارنة ميزات البرنامج ، على الرغم من أن المستخدم أخذ عينات من كل برنامج من المرجح أن تجد أنها تفضل جوانب معينة من النمذجة الجزيئية الكلية في برنامج واحد مقابل آخر. ندعو القراء إلى الاستفادة من إطار BioMolViz ، الذي يفصل أهداف التعلم الجزيئي الحيوي والأهداف المستهدفة في هذا البروتوكول ، واستكشاف الموارد للتعليم والتعلم التصور الجزيئي الحيوي من خلال موقع BioMolViz المجتمعي في http://biomolviz.org.
Disclosures
ويعلن المؤلفان أنه ليس لديهما مصالح مالية ذات صلة أو مادية تتعلق بالبحوث الموصوفة في هذه الورقة.
Acknowledgements
وقد وفرت المؤسسة الوطنية للعلوم التمويل لهذا العمل:
تحسين منحة تعليم العلوم والتكنولوجيا والهندسة والرياضيات الجامعية (جائزة #1712268)
شبكات تنسيق البحوث في المرحلة الجامعية في التعليم الجامعي في علم الأحياء (جائزة # 1920270)
نحن ممتنون لكاستن ثيس، دكتوراه، جامعة ويستفيلد، لمناقشات مفيدة حول Jmol.
Materials
| Name | Company | Catalog Number | Comments |
| ChimeraX (Version 1.2.5) https://www.rbvi.ucsf.edu/chimerax/ | |||
| Computer | Any | ||
| iCn3D (web-based only: https://www.ncbi.nlm.nih.gov/Structure/icn3d/full.html) | |||
| Java (for Jmol) https://java.com/en/download/ | |||
| Jmol (Version 1.8.0_301) http://jmol.sourceforge.net/ | |||
| Mouse (optional) | Any | ||
| PyMOL (Version 2.4.1 - educational): https://pymol.org/2 educational use only version: https://pymol.org/edu/?q=educational |
References
- Loertscher, J., Green, D., Lewis, J. E., Lin, S., Minderhout, V. Identification of threshold concepts for biochemistry. CBE Life Sciences Education. 13 (3), 516-528 (2014).
- Jaswal, S. S., O’Hara, P. B., Williamson, P. L., Springer, A. L. Teaching structure: Student use of software tools for understanding macromolecular structure in an undergraduate biochemistry course: Teaching structure in undergraduate biochemistry. Biochemistry and Molecular Biology Education. 41 (5), 351-359 (2013).
- Tibell, L. A. E., Rundgren, C. -J. Educational challenges of molecular life science: Characteristics and implications for education and research. CBE Life Sciences Education. 9 (1), 25-33 (2010).
- Schönborn, K. J., Anderson, T. R. The importance of visual literacy in the education of biochemists. Biochemistry and Molecular Biology Education. 34 (2), 94-102 (2006).
- Anderson, T. R. Bridging the educational research-teaching practice gap: The importance of bridging the gap between science education research and its application in biochemistry teaching and learning: Barriers and strategies. Biochemistry and Molecular Biology Education. 35 (6), 465-470 (2007).
- Schönborn, K. J., Anderson, T. R. Bridging the educational research-teaching practice gap: Foundations for assessing and developing biochemistry students’ visual literacy. Biochemistry and Molecular Biology Education. 38 (5), 347-354 (2010).
- Bateman, R. C., Craig, P. A. Education corner: A proficiency rubric for biomacromolecular 3D literacy. PDB Newsletter. 45, 5-7 (2010).
- Mnguni, L., Schönborn, K., Anderson, T. Assessment of visualization skills in biochemistry students. South African Journal of Science. 112, 1-8 (2016).
- Craig, P. A., Michel, L. V., Bateman, R. C. A survey of educational uses of molecular visualization freeware. Biochemistry and Molecular Biology Education. 41 (3), 193-205 (2013).
- Loertscher, J., Villafañe, S. M., Lewis, J. E., Minderhout, V. Probing and improving student’s understanding of protein α-Helix structure using targeted assessment and classroom interventions in collaboration with a faculty community of practice. Biochemistry and Molecular Biology Education. 42 (3), 213-223 (2014).
- Abualia, M., et al. Connecting protein structure to intermolecular interactions: A computer modeling laboratory. Journal of Chemical Education. 93 (8), 1353-1363 (2016).
- Carvalho, I., Borges, A. D. L., Bernardes, L. S. C. Medicinal chemistry and molecular modeling: An integration to teach drug structure–activity relationship and the molecular basis of drug action. Journal of Chemical Education. 82 (4), 588(2005).
- Forbes-Lorman, R. M., et al. Physical models have gender-specific effects on student understanding of protein structure-function relationships. Biochemistry and Molecular Biology Education. 44 (4), 326-335 (2016).
- Terrell, C. R., Listenberger, L. L. Using molecular visualization to explore protein structure and function and enhance student facility with computational tools. Biochemistry and Molecular Biology Education. 45 (4), 318-328 (2017).
- Zhang, S., et al. Structure-based drug design of an inhibitor of the SARS-CoV-2 (COVID-19) main protease using free software: A tutorial for students and scientists. European Journal of Medicinal Chemistry. 113390, (2021).
- Roberts, J. R., Hagedorn, E., Dillenburg, P., Patrick, M., Herman, T. Physical models enhance molecular three-dimensional literacy in an introductory biochemistry course. Biochemistry and Molecular Biology Education. 33 (2), 105-110 (2005).
- Jenkinson, J., McGill, G. Visualizing protein interactions and dynamics: Evolving a visual language for molecular animation. CBE Life Sciences Education. 11 (1), 103-110 (2012).
- Bussey, T. J., Orgill, M. What do biochemistry students pay attention to in external representations of protein translation? The case of the Shine–Dalgarno sequence. Chemistry Education Research and Practice. 16 (4), 714-730 (2015).
- Harle, M., Towns, M. H. Students’ understanding of primary and secondary protein structure: Drawing secondary protein structure reveals student understanding better than simple recognition of structures. Biochemistry and Molecular Biology Education. 41 (6), 369-376 (2013).
- Dries, D. R., et al. An expanded framework for biomolecular visualization in the classroom: Learning goals and competencies. Biochemistry and Molecular Biology Education. 45 (1), 69-75 (2017).
- The BioMolViz Framework. BioMolViz. , Available from: http://biomolviz.org/framework (2021).
- Procko, K., et al. Meeting report: BioMolViz workshops for developing assessments of biomolecular visual literacy. Biochemistry and Molecular Biology Education. 49 (2), 278-286 (2021).
- Jmol: an open-source Java viewer for chemical structures. , Available from: http://www.jmol.org/ (2021).
- Wang, J., et al. iCn3D, a web-based 3D viewer for sharing 1D/2D/3D representations of biomolecular structures. Bioinformatics. 36 (1), 131-135 (2020).
- PyMOL . The PyMOL Molecular Graphics System. Version 2.0. , Schrödinger, LLC. (2021).
- Goddard, T. D., et al. UCSF ChimeraX: Meeting modern challenges in visualization and analysis. Protein Science. 27 (1), 14-25 (2018).
- Pettersen, E. F., et al. UCSF ChimeraX: Structure visualization for researchers, educators, and developers. Protein Science. 30 (1), 70-82 (2021).
- Petit, P., et al. The active conformation of human glucokinase is not altered by allosteric activators. Acta Crystallographica. Section D. 67 (11), 929-935 (2011).
- Corey, R. B., Pauling, L. Molecular models of amino acids, peptides and proteins. Review of Scientific Instruments. 24, 621-627 (1953).
- Koltun, W. L. Precision space-filling atomic models. Biopolymers. 3 (6), 665-679 (1965).
- Hodis, E., et al. Proteopedia - a scientific 'wiki' bridging the rift between three-dimensional structure and function of biomacromolecules. Genome Biology. 9 (8), 1-10 (2008).
- Prilusky, J., et al. Proteopedia: A status report on the collaborative, 3D web-encyclopedia of proteins and other biomolecules. Journal of Structural Biology. 175 (2), 244-252 (2011).
- Martz, E. FirstGlance in Jmol. , Available from: https://www.bioinformatics.org/firstglance/fgij/ (2021).
- Jmol User Design Environment (JUDE). MSOE Centerfor BioMolecular Modeling. , Available from: https://cbm.msoe.edu/modelingResources/jmolUserDesignEnvironment/#forward (2021).
- Castro, C. R., et al. A practical guide to teaching with Proteopedia. Biochemistry and Molecular Biology Education. 49 (5), 707-719 (2021).
- Berman, H. M., et al. The protein data bank. Nucleic Acids Research. 28, 235-242 (2000).
- The Protein Data Bank. , Available from: https://www.rcsb.org/ (2021).
- Wang, Y., et al. MMDB: 3D structure data in Entrez. Nucleic Acids Research. 28 (1), 243-245 (2000).
- iCn3D Help Page. , Available from: https://www.ncbi.nlm.nih.gov/Structure/icn3d/docs/icn3d_help.html (2021).
- MSOE Center for BioMolecular Modeling Jmol Training Guide. , Available from: https://cbm.msoe.edu/modelingResources/jmolTrainingGuide/started.html (2021).
- The Online Macromolecular Museum. , Available from: http://earth.callutheran.edu/Academic_Programs/Departments/BioDev/omm/exhibits.htm (2021).
- Jmol/JSmol Interactive Scripting Documentation. , Available from: https://chemapps.stolaf.edu/jmol/docs/ (2021).
- PyMOL Wiki. , Available from: https://pymolwiki.org/index.php/Main_Page (2021).
- PyMOL Advanced Scripting Workshop by Schrödinger. , Available from: https://pymol.org/tutorials/scripting/index.html (2021).
- UCSF ChimeraX User Guide. , Available from: https://www.cgl.ucsf.edu/chimerax/docs/user/index.html (2021).
- UCSF ChimeraX Tutorials. , Available from: https://www.rbvi.ucsf.edu/chimerax/tutorials.html (2021).
- Kuntz, I. D. Structure-based strategies for drug design and discovery. Science. 257 (5073), 1078-1082 (1992).
- Structure-based drug discovery: an overview. Hubbard, R. E. , (2006).
- Patrick, G. L. An introduction to medicinal chemistry, 6th ed. , Oxford University Press. (2017).
- Van Montfort, R. L., Workman, P. Structure-based drug design: aiming for a perfect fit. Essays in Biochemistry. 61 (5), 431-437 (2017).
- Holdgate, G. A., Meek, T. D., Grimley, R. L. Mechanistic enzymology in drug discovery: a fresh perspective. Nature Reviews. Drug Discovery. 17 (2), 115-132 (2018).
- Wang, M. Y., et al. SARS-CoV-2: structure, biology, and structure-based therapeutics development. Frontiers in Cellular and Infection Microbiology. 10, (2020).
- White, B., Kim, S., Sherman, K., Weber, N. Evaluation of molecular visualization software for teaching protein structure differing outcomes from lecture and lab: Differing outcomes from lecture and lab. Biochemistry and Molecular Biology Education. 30 (2), 130-136 (2002).
- Canning, D. R., Cox, J. R. Teaching the structural nature of biological molecules: Molecular visualization in the classroom and in the hands of students. Chemistry Education Research and Practice. 2 (2), 109-122 (2001).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved