需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
如何从MR图像测量皮质折叠:一个循序渐进的教程,以计算本地Gyrification指数
摘要
(皮质折叠)在任何年龄的测量gyrification代表到早期的大脑发育的窗口。因此,我们以前开发了一种算法来衡量当地gyrification千点以上的半球 1。在本文中,我们详细介绍这个地方gyrification指数的计算。
摘要
皮质折叠(gyrification)确定在生命的最初几个月,因此,在此期间发生的不良事件留下的痕迹,将在任何年龄识别。最近审查Mangin和同事2,存在的几种方法,以量化gyrification的不同特点。例如,可用于脑沟形态学如半球间的深度,长度或指数来衡量的形状描述不对称 3 。这些几何特性的优点是易于理解。然而,脑沟形态学紧紧依靠准确识别一个给定的沟,因此提供了一个支离破碎的gyrification描述。基于曲率的测量,其中平滑的绝对平均曲率通常是在皮质表面4千点计算,可以实现更细粒度的gyrification量化。曲率,但不straightforward来理解,因为它仍不清楚,如果curvedness一个生物学意义的关联,如皮质体积或表面之间有任何直接关系。为了解决测量皮质折叠提出的各种问题,我们以前开发了一种算法,量化当地gyrification一个精致的空间分辨率和简单的解释。我们的方法是启发Gyrification指数5,原本比较神经解剖学方法,用于评估跨物种的皮质折叠差异。在我们的实现,我们名升 ocal Gyrification指数(GI 1 升 ),我们衡量皮质金额埋脑沟皱褶内可见皮质的圆形区域的利益金额相比。由于皮层的增长主要是通过径向膨胀 6,我们的方法是专门设计,以确定皮质发育的早期缺陷。
在日是一篇文章中,我们详细介绍当地Gyrification指数的计算,这是目前作为 FreeSurfer软件的一部分( 自由分布http://surfer.nmr.mgh.harvard.edu/,Martinos生物医学成像中心,马萨诸塞州总医院) 。FreeSurfer提供了一套大脑的皮质表面结构MRI数据自动重建工具。在本土与亚毫米级精度的图像空间中提取的皮质表面,然后再用于创造的外表面,这将作为一个为 l GI计算的基础上。利益的一个圆形区域,然后划定的外表面,并使用验证研究 1所述的匹配算法确定其相应区域的皮质表面上的兴趣。这个过程反复迭代很大程度上是重叠的地区利益,gyrification皮质地图ř随后的统计比较(图1)。值得注意的是,另一个地方gyrification的测量提出了类似的灵感托罗和他的同事 7,皮质区的光盘领域的划分与同一个领域中的比例计算,在每个点的折叠指数半径。这两种实现方式的不同,一个由红牛等。是基于欧几里德距离,因此认为皮质区的不连续的补丁,而我们采用了严格的测地线算法和只包括皮质区开放利益的一个圆形区域的脑表面连续补丁。
研究方案
1。重建3D皮质表面
这个协议的第一部分使用的标准 FreeSurfer管道,在Wiki (http://surfer.nmr.mgh.harvard.edu/fswiki )。请注意,在这里详细的命令说明实现皮层表面重建的方法之一,但也可以使用等效命令。
- 导入FreeSurfer原始MRI DICOM和验证的图像质量(例如,方向是正确的,足够的对比度和图像不移动) 。此过程使用下面的命令(替换与一个特定的实例值适当<...>(含)之间的文本,“#”表示注释):
mksubjdirs #创建的文件夹架构FreeSurfer使用
CD / MRI#去到你的主题MRI文件夹
mri_convert -厘米 001.mgz##FreeSurfer格式转换原料的MRI
tkmedit 001.mgz#可视化转换的卷 - 创建的三维皮质网状模型8,9。为了配合埋沟的问题中 ,FreeSurfer首先创建一个单一的白质量,这是用来作为最初的灰白色表面的起点。这表面上看是根据当地的强度梯度优化,并进一步扩大到灰色- CSF的接口。
重建所有- S #启动皮质表面重建
在重建过程结束时,你会得到两个约15万点,每个半球组成的网格模型:一个白色(灰色白色接口)和软脑膜(灰CSF接口)表面。重要的是要注意,所有表面和卷留在本机的空间,让如测量体积,表面积,厚度或gyrification指数来衡量不变形。 - 这些重建的表面精度的检查:
tkmedit T1.mgz?h.pial#白色表面覆盖在绿色和红色软脑膜表面
其中H表示半球:左半球和右半球rh.pial lh.pial。图2(2个版本:动画GIF图像,包括在电影和一个静态的网站)“BERT”主题分布与FreeSurfer包沿正确的白和软脑膜表面重建的一个例子。如果你必须手动正确的重建过程中的结果,你会发现一个FreeSurfer维基教程 (http://surfer.nmr.mgh.harvard.edu/fswiki/FsTutorial/WhiteMatterEdits
2。计算本地Gyrification指数
当你满意你的表面,计算的本地Gyrification指数(L GI)的,使用命令:
重建所有的LGI - S
此命令通常运行3个小时左右两半球的一个研究参与者,根据工作站的电源。 升胃肠过程的不同步骤overviewed图。 1。计算开始建立与使用形态闭运算的外表面。此外表面,记?h.pial_outer_smoothed,进一步说明图。 3。然后,利益重叠的圆形区域约800创建的外表面。对于这些地区的每个人,有兴趣的相应区域上定义软脑膜表面。整个计算结束了与创建个人地图包含一个 L GI皮质表面的每个点的价值(即〜15%半球值)。
3。检查每个半球升 GI计算的结果
tksurfer · H软脑膜叠加 /冲浪h.pial_lgi - fthresh 1 /?
升 GI值覆盖在皮质表面。为正确升 GI值通常是由1和5之间,设置为1的最低阈值(选项fthresh),允许一个快速检查:你不应该看到任何灰色的皮质区。如图正确的个别结果的一个例子。 4。
4。统计组比较
其目的是量化皮质表面的群体效应,在每个顶点,而性别的影响,控制和年龄。您将需要遵循相同的过程,如果你想比较在每个顶点皮质厚度,但
- 第一个选项组间比较升胃肠结果使用下面列出的命令;进一步细节可在获得https://surfer.nmr.mgh.harvard.edu/fswiki/FsTutorial/GroupAnalysis 。
- 首先,你需要创建一个研究特定的模板,让您在输入题目:
make_average_subject -科目 ... ...
上面的命令将创建一个名为“平均”的主题。或者,您可以使用的主题是“FSAVE风靡一时的“分布式FreeSurfer分布。 - 然后,创建一个文本文件,其中包含在您的研究中涉及的科目(“FreeSurfer组描述符文件”)的说明。你FSGD.txt看起来应该像这样:
GroupDescriptorFile 1
集团Control_Male
集团Control_Female
集团Patient_Male
集团Patient_Female
变量年龄
输入 Patient_Male 20
输入 Control_Female 23
<...> - 重新取样升胃肠数据的平均受使用以下命令为每个半球的空间:
mris_preproc - fsgd FSGD.txt -平均目标-半h - MEAS pial_lgi - ?出h.lgi.mgh - 光滑的皮质表面上的数据,以减少噪音信号:
mri_surf2surf - ?半小时-平均- ?SVAL h.lgi.mgh小号- FWHM 10 - ?tval h.10.lgi.mgh - 计算每个顶点的水平组比较。对于您将需要在以上所述的FSGD.txt情况下创建一个对比的文本文件(例如,“contrast.txt”将包含值“1 1 -1 -1 0”计算控制和之间的差异患者同时控制年龄和性别)。最后运行的比较:
mri_glmfit - Ÿ h.10.lgi.mgh - fsgd FSGD.txt斯- ?glmdir h.lgi.glmdir -冲浪平均H -摄氏度contrast.txt - 可视化的结果在你的平均使用tksurfer的主题:
tksurfer平均· H虚增
然后覆盖sig.mgh文件位于该文件夹中加载?h.lgi.glmdir / contrast.txt / sig.mgh。使用选项“配置覆盖”,您可以进一步修改以及正确使用虚假的发现率 10多个比较 p门槛。
- 首先,你需要创建一个研究特定的模板,让您在输入题目:
- 组COM的替代方案坯是用Qdec,图形用户界面在FreeSurfer实施。与当地的Gyrification指数使用的Qdec意味着前顺利升胃肠道的数据:
重建,qcache措施pial_lgi平均 - S
随着Qdec,FreeSurfer组描述符文件是更换一个稍微不同的版本,数据表(qdec.table.dat),其中包括如年龄,不同的团体和其他混杂变量的描述。在http://surfer.nmr.mgh.harvard.edu/fswiki/FsTutorial/QdecGroupAnalysis提供一个详细的说明Qdec利用。
值得注意的是, 如果 L GI不Qdec因变量列表,您必须位于你的主目录。Qdecrc文件中添加以下行:
MEASURE1 = pial_lgi
5。分析
另外,统计分析,最终可能会被计算在 11 FreeSurfer综合皮质parcellation的水平。为此,平均升 GI值可以提取每个半球脑回的利息为34地区,而这些测量可以进一步比较不同群体之间的。这个包裹明智的分析(而不是顶点明智的分析,以上所述)可以有吸引力,因为它限制量统计比较。然而,在每个点上升胃肠量化gyrification,在周围的圆形区域,以便在利益的脑回区域的平均升胃肠也反映了在一定程度上gyrification在邻近地区的利益。
最后,虽然最重要的问题进行了描述在这个协议中, 神父期间可能遇到的其他问题的解决方案eeSurfer 或 L胃肠道加工中可以找到FreeSurfer邮件列表(档案http://surfer.nmr.mgh.harvard.edu/fswiki/FreeSurferSupport )。
6。代表性的成果
作为在该协议的部分1C的描述,你应该总是仔细检查皮质表面重建的准确性之前升胃肠计算。虽然滚动之间的额叶和枕叶,要特别注意,不包括在软脑膜表面船只和膜。白色表面准确如下灰白色界面检查。正确重建的一个例子是在图2(见整个卷的动画GIF图) 。
在年底的 L GI计算,你也将检查每个科目的两个半球的结果。不应该有任何一个 L GI结果小于1的皮层区域。第3条的协议和图4显示如何检查正确的 L GI计算的输出,如果是正确的。
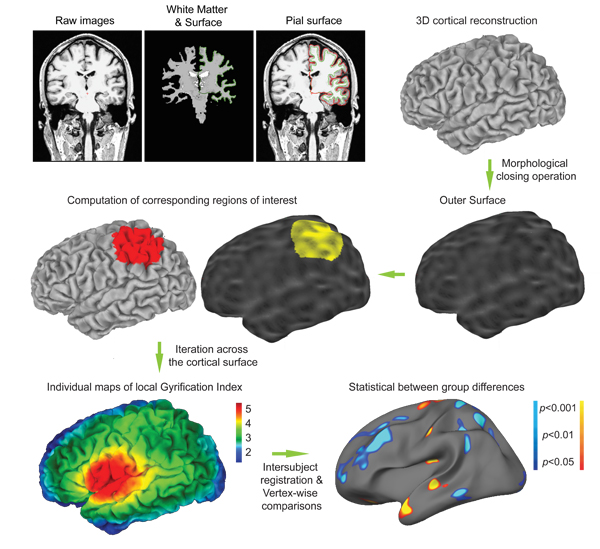
图1 升胃肠计算概述:第一,立体皮质网格模型重建原始图像使用标准的FreeSurfer管道。这些重建算法使用二进制白质量作为出发点,以克服埋沟的问题。皮质网状模型通常包括约15万的顶点和古典用于计算每个点的皮质厚度。同样,当地Gyrification指数(L GI)的将被计算在每个顶点。为此,创建一个外表面。然后确定相应的圆形区域的利益外一个ð皮质表面使用匹配算法。经过约产生重叠地区的利益,创造升胃肠道的个人地图的过程结果800 。这些地图可以很容易地解释:指数5表示有更多的皮质表面内陷内沟周边地区的5个时间,1意味着指数,皮质是平坦的,在周边地区,可见皮质表面的数额; 。最后,统计组比较,计算每个顶点的水平,同样皮质的厚度比较。
这个小电影图1b。个人升胃肠道的皮质地图 。个人皮质LGI在图所示的地图显示了一个360度的旋转。 1。令人吃惊的注意,具有较高的升 GI值的皮层区域对应在子宫内的生活将在创建的第一折:裂裂,颞上沟,并在traparietal沟上的大脑侧视,大脑内侧查看parieto枕沟。 查看电影
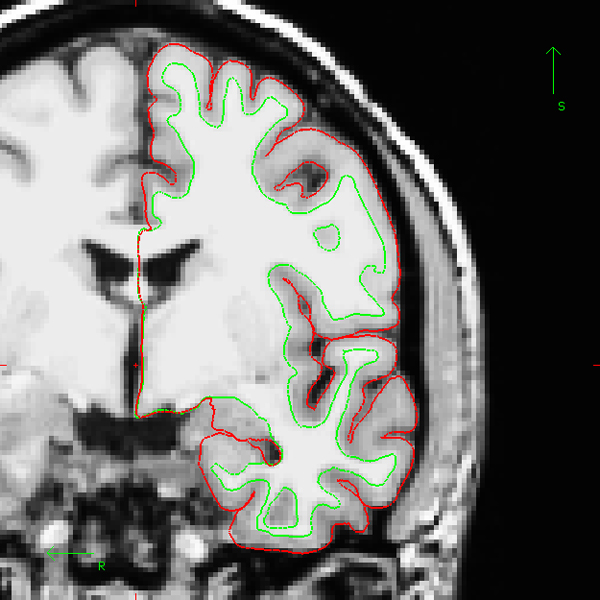
图2。范例足够的皮质表面的重建(一冠状断面)重建过程结束后,皮质表面应准确核实整个脑体积。内皮层表面(表示绿色,白色表面图像)正是应遵循灰白色的接口。外层的皮质表面(即灰色- CSF的接口,表示软脑膜表面,在这里红色),不应包括任何船只或膜的一块。值得注意的是,这里介绍的例子使用“伯特”的主题分布与FreeSurfer包。
图2b。足够的皮质表面reconstru的范例ction(最大音量)。,这个动画的GIF图像显示“BERT”冠状断面上每个主题的左半球皮层表面,滚动,从最正面与FreeSurfer最枕部的冠状切片。电影

图3。 升胃肠道的过程(一冠状断面)部分计算的外表面 的例子,在L胃肠计算的第一步是建立一个半球的表面外层包围。这面(记?在FreeSurfer h.pial_outer_smoothed)可以检查使用tkmedit。在这里,“伯特”的主题分布与FreeSurfer,是作为一个例子。
图3B。这个动画GIF图像的例子外表面计算的 L GI过程(最大音量)的一部分 。观看电影显示冠状断面上每个左半球的外表面,从最正面最枕部的冠状tkmedit节在FreeSurfer滚动。
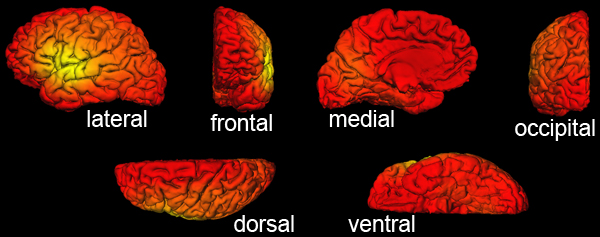
图4。正确的升 GI输出的例子,与FreeSurfer看。“BERT”升胃肠道题目的皮质表面不同方向的值覆盖。颜色代码是默认的“热”叠加与tksurfer在FreeSurfer。使用1的最低门槛,所有的顶点必须是彩色的,没有皮质区应AP梨灰色。值得注意的是,颜色叠加使用tksurfer,也可以检查的最小值和最大值,以及升胃肠道的整体分布直方图的“配置覆盖”选项,可以修改。
讨论
协议上面介绍了如何衡量当地Gyrification指数基于对脑T1加权MRI和进行统计组比较。我们的方法已被专门设计的本地化皮质膨胀过程中的早期中断,因此许多神经发育或精神状况特别感兴趣。在临床样本组比较的例子,可以发现在出版物, 由本集团1,12 或由他人13-16。这个过程是完全自动化的,只需要命令要执行,虽然两个参数可以修改。
第一次修改的参数是
披露声明
作者什么都没有透露。
致谢
这项研究得到了能力的国家研究中心(NCCR)“SYNAPSY - 精神疾病的突触基地”由瑞士国家科学基金会(N ° 51AU40_125759)的资助。从瑞士国家研究基金玛丽Schaer(323500-111165)博士和博士史蒂芬Eliez(3200-063135.00 / / 1,1,3232-063134.00 PP0033 - 102864和32473B的赠款支持发展本地Gyrification指数-121996)和中心在日内瓦的洛桑大学的生物医学成像(CIBM),洛桑联邦理工学院,以及基础Leenaards和Louis - Jeantet。 FreeSurfer软件的发展提供了支持部分由国家研究资源中心(P41 - RR14075,并BIRN002 NCRR BIRN形态项目,U24 RR021382),国家生物医学成像和生物工程研究所(R01 EB001550 ,R01EB006758)美国国家神经紊乱和中风研究所(R01 ñS052585 - 01)以及精神疾病和神经科学发现(心)研究所,是全国医学影像计算联盟(NAMIC)由国立卫生研究院的资助,通过国立卫生研究院医学研究路线图,格兰特U54,的一部分EB005149。埃利森医学基金会资助自闭症及读写障碍项目提供额外的支持。
材料
材质:与UNIX或Mac工作站的2GHz或更快的处理器和4GB的RAM最低FreeSurfer安装( http://surfer.nmr.mgh.harvard.edu/fswiki ,最好是最新版本,但年龄不比版本4.0.3)。 MATLAB是为了计算的本地Gyrification指数,还需要( http://www.mathworks.com/ )随着图像处理工具箱。
数据:质量好的样品(高清晰度,高对比度)脑MRI T1加权数据集。您的受试者必须是最好的年龄和性别相匹配。由于个体间正常的脑形态的变化,各组的科目数应足以识别现有的组间差异(更 - 更好)。一个合理的最低样本大小将大约20名受试者每个组(虽然你也许可以少走,如果强度变化大,如果你的团体紧密性别和年龄相匹配)。
| Name | Company | Catalog Number | Comments |
| 设备名称 | 公司 | 目录编号 | 评论 |
| FreeSurfer | 麻省总医院生物医学成像,Martinos中心 | 比4.0.3版本更新 | |
| MATLAB | Mathworks公司 | 图像处理工具箱 |
参考文献
- Schaer, M. A surface-based approach to quantify local cortical gyrification. IEEE. Trans. Med. Imaging. 27, 161-170 (2008).
- Mangin, J. F., Jouvent, E., Cachia, A. In-vivo measurement of cortical morphology: means and meanings. Curr. Opin. Neurol. 23, 359-367 (2010).
- Mangin, J. F. A framework to study the cortical folding patterns. Neuroimage. 23, S129-S138 (2004).
- Luders, E. A curvature-based approach to estimate local gyrification on the cortical surface. Neuroimage. 29, 1224-1230 (2006).
- Zilles, K., Armstrong, E., Schleicher, A., Kretschmann, H. J. The human pattern of gyrification in the cerebral cortex. Anat. Embryol. (Berl). 179, 173-179 (1988).
- Rakic, P. Specification of cerebral cortical areas. Science. 241, 170-176 (1988).
- Toro, R. Brain size and folding of the human cerebral cortex. Cereb. Cortex. 18, 2352-2357 (2008).
- Fischl, B., Sereno, M. I., Dale, A. M. Cortical surface-based analysis. II: Inflation, flattening, and a surface-based coordinate system. Neuroimage. 9, 195-207 (1999).
- Dale, A. M., Fischl, B., Sereno, M. I. Cortical surface-based analysis. I. Segmentation and surface reconstruction. Neuroimage. 9, 179-194 (1999).
- Genovese, C. R., Lazar, N. A., Nichols, T. Thresholding of statistical maps in functional neuroimaging using the false discovery rate. Neuroimage. 15, 870-878 (2002).
- Desikan, R. S. An automated labeling system for subdividing the human cerebral cortex on MRI scans into gyral based regions of interest. Neuroimage. 31, 968-980 (2006).
- Schaer, M. Congenital heart disease affects local gyrification in 22q11.2 deletion syndrome. Dev. Med. Child. Neurol. 51, 746-753 (2009).
- Palaniyappan, L., Mallikarjun, P., Joseph, V., White, T. P., Liddle, P. F. Folding of the Prefrontal Cortex in Schizophrenia: Regional Differences in Gyrification. Biol. Psychiatry. , (2011).
- Zhang, Y. Decreased gyrification in major depressive disorder. Neuroreport. 20, 378-380 (2009).
- Juranek, J., Salman, M. S. Anomalous development of brain structure and function in spina bifida myelomeningocele. Dev. Disabil. Res. Rev. 16, 23-30 (2010).
- Zhang, Y. Reduced cortical folding in mental retardation. AJNR. Am. J. Neuroradiol. 31, 1063-1067 (2010).
- Kuperberg, G. R. Regionally localized thinning of the cerebral cortex in schizophrenia. Archives of general psychiatry. 60, 878-888 (2003).
- Milad, M. R. Thickness of ventromedial prefrontal cortex in humans is correlated with extinction memory. Proc. Natl. Acad. Sci. U. S. A. 102, 10706-10711 (2005).
- Rauch, S. L. A magnetic resonance imaging study of cortical thickness in animal phobia. Biol. Psychiatry. 55, 946-952 (2004).
- Fjell, A. M. Selective increase of cortical thickness in high-performing elderly--structural indices of optimal cognitive aging. Neuroimage. 29, 984-994 (2006).
- Walhovd, K. B. Regional cortical thickness matters in recall after months more than minutes. Neuroimage. 31, 1343-1351 (2006).
- Gold, B. T. Differing neuropsychological and neuroanatomical correlates of abnormal reading in early-stage semantic dementia and dementia of the Alzheimer type. Neuropsychologia. 43, 833-846 (2005).
- Salat, D. H. Thinning of the cerebral cortex in aging. Cereb. Cortex. 14, 721-730 (2004).
- Schaer, M., Eliez, S. Contribution of structural brain imaging to our understanding of cortical development process. European Psychiatry Reviews. 2, 13-16 (2009).
- Shaw, P. Neurodevelopmental trajectories of the human cerebral cortex. J. Neurosci. 28, 3586-3594 (2008).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。