このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
ローカルGyrificationのインデックスを計算するステップバイステップのチュートリアル:MR画像からの皮質折りたたみを測定する方法
要約
どの年齢でも測定gyrification(皮質フォールディング)は初期の脳の発達にウィンドウを表します。したがって、我々は以前に半球上の点の数千にローカルgyrificationを測定するアルゴリズムを開発 1。本稿では、詳細は、このローカルgyrificationインデックスの計算を。
要約
皮質フォールディング(gyrification)はこの期間中に発生した有害事象はどの年齢でも識別可能となる痕跡を残すように、人生の最初の数ヶ月の間に決定されます。最近マンジャンと同僚2でレビューとして、いくつかの方法がgyrificationの異なる特性を定量化するために存在している。例えば、sulcal形態計測は、3非対称性などの深さ、長さまたはinter -半球の指標として形状記述子を測定するために使用することができます。これらの幾何学的特性は、解釈が容易であるという利点を持っている。しかし、sulcal形態計測はしっかりと溝の特定のセットの正確な識別に依存して、それゆえgyrificationの断片化された説明を提供します。 gyrificationのよりきめ細かな定量化は、平滑化、絶対平均曲率は、通常、皮質表面4上の点の数千で計算される曲率ベースの測定、を達成することができます。曲率は、しかしstraightforwarではない曲がった状態や生物学的に意味のあるような皮質体積や表面のように相関するとの間に直接的な関係があるかどうかが不明確のままであるdは、理解する。皮質フォールディングの測定で発生した様々な問題に対処するため、我々は以前に絶妙な空間分解能で、単純な解釈のローカルgyrificationを定量化するアルゴリズムを開発した。我々の方法は、Gyrificationのインデックス5、元々の種を超え皮質折りたたみの差異を評価するために比較神経解剖学で使用されているメソッドのインスピレーションを得ています。興味の円形領域に表示される皮質の量と比較して我々 名 l OCAL Gyrification指数(L GI 1)、我々はsulcal襞に埋もれ皮質の量を測定する私たちの実装で、で。皮質は、径方向の膨張6まで主に成長することを考えると、私たちの方法は、特に皮質発達の初期の欠陥を識別するように設計されました。
番目の記事では、我々が詳細に今自由にFreeSurferソフトウェアの一部(として配布されているローカルGyrification指数の計算、、ですhttp://surfer.nmr.mgh.harvard.edu/ 、医学、マサチューセッツ総合病院のためのMartinosセンター) 。FreeSurferは、構造的MRIデータから脳の皮質表面の自動復元ツールのセットが用意されています。サブミリメートル精度での画像のネイティブ空間で抽出された皮質表面をさらにリットル GIの計算の基礎となる外表面、の作成 に使用されます。関心の円形領域は、外表面に線引きされており、皮質表面上の利益のそれに対応する領域は、当社のバリデーションスタディ1で説明したようにマッチングアルゴリズムを使用して識別されます。このプロセスは、繰り返しのためにgyrificationの皮質マップで、その結果、利益の大部分は重複領域で反復されるRその後の統計的比較(図1)。注目すべきは、同じようなインスピレーションとローカルgyrificationの別の測定は、各点でのフォールディングインデックスが同じでディスクの面積で割った球に含まれる皮質面積の比率として計算されますトロと仲間7によって提案された半径。二つの実装では、トロらずつという点で異なります。ユークリッド距離をベースにしている皮質領域の不連続なパッチを考慮し、我々のは厳密な測地線のアルゴリズムを使用し、関心の円形領域における脳の表面の皮質領域の開口部の唯一の連続的なパッチを含むのに対しています。
プロトコル
1。 3D皮質表面を再構築
Wikiの(で説明したように、プロトコルのこの最初の部分は、標準FreeSurferのパイプラインを使用していますhttp://surfer.nmr.mgh.harvard.edu/fswiki )。ここで詳述するコマンドが皮質表面再構成を達成するための一つの方法を説明することに注意してください、しかし、同等のコマンドを使用することもできます。
- FreeSurferに生のMRI DICOMをインポートし、画像の品質を検証する(向きが正しいことを、例えば、十分なコントラストとイメージは移動しません)。このプロセスは、次のコマンドを(コメントを示す"#"特定のインスタンスに適切な値に<...>の間にテキストを(含む)に置き換え、および)を使用します:
mksubjdirs #FreeSurferで使用されているフォルダのアーキテクチャを作成する
CD / MRIは#あなたの主題のMRIフォルダに移動します
mri_convert - CM 001.mgzは##FreeSurfer形式で生のMRIを変換する
tkmedit 001.mgzは#変換後のボリュームを可視化 - 三次元皮質メッシュモデル8,9を作成します。埋め込 み溝の問題に対処するためには、FreeSurfer最初の初期灰白色の表面の出発点として使用されている単一の白質のボリュームを、作成します。この面は、強度の局所勾配に応じて最適化し、さらにグレー- CSFのインタフェースに拡張されます。
偵察-すべての- S #皮質表面再構成を開始する
ホワイト(グレーホワイトインタフェース)と軟膜(グレー- CSFインタフェース)表面:再構成プロセスの最後に、2つのメッシュの各半球の約15万点で構成されるモデルを取得します。すべてのサーフェスとボリュームができ、ネイティブ領域に残っていることに注意することは重要ですこのような体積、表面積、変形することなく測定する厚さやgyrification指標として測定。 - これらの再建された曲面の精度を確認します。
tkmedit T1.mgz?h.pial#白い表面が緑色に重ね合わせていると赤の軟膜表面
ここで、hは半球を示す:右半球、左半球とrh.pial用lh.pial。図2(2つのバージョン:Webサイトのための映画と静的ページに含まれるアニメーションGIF画像 ) は FreeSurferパッケージとともに配布"BERT"主題の正しい白と軟膜表面再構成の例を示しています。手動で再構築プロセスの結果を修正する必要がある場合、あなたは(FreeSurfer Wikiのチュートリアルが用意されてhttp://surfer.nmr.mgh.harvard.edu/fswiki/FsTutorial/WhiteMatterEdits 、
2。ローカルGyrificationインデックスを計算する
あなたがあなたの表面に問題がなければ、コマンドを使用してローカルGyrification指数(L GI)を計算する:
偵察-すべての- LGI - S
このコマンドは、通常、ワークステーションのパワーにもよりますが、一つの研究の参加者の両半球のために約3時間かけて実行される。 リットル GIプロセスの異なるステップを図で概説されています。 1。コンピューティングは、形態学的な閉動作を使用して外表面の作成から始まります。この外面、示された?h.pial_outer_smoothedは、さらに図5に示す。 3。その後、関心の円形領域をオーバーラップ約800の外側表面上に作成されます。これらの領域のそれぞれについて、関心の対応する領域は、上で定義され軟膜表面。全体の計算は、皮質表面の各点に対して1 リットル GI値(半球あたりすなわち〜150,000の値)を含む個々のマップの作成 で終わります。
3。各半球のためのl GIの計算結果を確認してください
tksurfer ?H軟膜-オーバーレイ /サーフィン/?h.pial_lgi - fthresh 1
リットル GI値は皮質表面上にオーバーレイされます。正しいLの GI値は通常、1と5の間に含まれているとして、1(オプションfthresh付き)で、最小しきい値を設定すると、迅速なチェックを可能にする:何か灰色の皮質領域を見ることはできないはず。正しい個々の結果の例を図に示します。 4。
4。統計的グループの比較
目的は、性別の効果を制御しながら、皮質表面上の各頂点でのグループの効果を定量化することであると年齢。あなたが各頂点に皮質の厚さを比較したいが
- グループ間リットル GIの結果を比較する最初のオプションは、以下のコマンドを使用して、さらに詳細はから入手できますhttps://surfer.nmr.mgh.harvard.edu/fswiki/FsTutorial/GroupAnalysis 。
- まず、入力のすべての科目を与える研究、特定のテンプレートを作成する必要があります。
make_average_subject -被験者 ...
コマンドは、上記の"平均的"という名前のテーマが作成されます。代わりに、件名"FSAVEを使用することができますFreeSurferディストリビューションの一部として配布激怒"。 - その後、あなたの研究に関与する科目("FreeSurferグループ記述子ファイル ")の記述を含むテキストファイルを作成します。あなたのFSGD.txtは次のようになります。
GroupDescriptorFile 1
グループControl_Male
グループControl_Female
グループPatient_Male
グループPatient_Female
変数年齢
入力 Patient_Male 20
入力 Control_Female 23
<...> - 各半球ごとに次のコマンドを使用して、平均的な被験者の空間のL GIのデータを再サンプリング:
mris_preproc - fsgd FSGD.txt -ターゲットの平均-半H - ?測定pial_lgi - ?出h.lgi.mgh - ノイズに信号を減らすために皮質表面上にデータを平滑化。
mri_surf2surf -半H - ?の平均- ?SVAL h.lgi.mgh - FWHM 10 - ?tval h.10.lgi.mgh - 各頂点のレベルのグループの比較を計算する。そのためにあなたがFSGD.txt上記の場合にはコントラストのテキストファイル(例:を作成する必要があります、"contrast.txtは、"コントロールとの違いを計算する値は"1 1 -1 -1 0"が表示されます患者の年齢と性別をコントロールしつつ)。最後に、比較を実行します。
mri_glmfit - ?Y h.10.lgi.mgh - fsgd FSGD.txt DOSS - glmdir h.lgi.glmdir - ?サーフ平均H - C contrast.txt - tksurferを使用して、平均的な主題の結果を可視化します。
tksurfer平均?hが水増し
その後、オーバーレイとしてフォルダにあるsig.mghファイルをロードする?h.lgi.glmdir / contrast.txt / sig.mgh。オプションを使用して、"オーバーレイを設定する"には、さらにpのしきい値を変更するだけでなく、偽発見率10を用いて多重比較のために正しいこともできます。
- まず、入力のすべての科目を与える研究、特定のテンプレートを作成する必要があります。
- グループCOMの代替オプションパリソンをQdec、FreeSurferで実装グラフィカルユーザインタフェースを使用することです。ローカルGyrificationインデックスとQdecの使用は、事前にスムーズリットル GIのデータに意味:
偵察-すべての- qcache -測定pial_lgi平均 - S
Qdecで、FreeSurferグループ記述子ファイルは、わずかに異なるバージョン、年齢などのさまざまなグループやその他の交絡変数の記述を含むデータテーブル(qdec.table.dat)に置き換えられます。 Qdecの利用の詳細な説明はで提供されていますhttp://surfer.nmr.mgh.harvard.edu/fswiki/FsTutorial/QdecGroupAnalysis 。
。注目すべきは、L GIはQdecの従属変数のリストで利用できない場合、あなたのホームディレクトリにあるQdecrcファイルに次の行を追加する必要があります。
MEASURE1 = pial_lgi
5。分析
また、統計解析は最終的にFreeSurfer 11に統合された皮質parcellationのレベルで計算することができます。その目的のために、平均リットル GI値は、それぞれの半球の興味の34 gyral地域を抽出することができ、これらの測定値は、さらに別の群間で比較することができます。それは統計的比較の量を制限するとして、この小包ワイズ解析では、(上述のように頂点単位の分析とは対照的に)魅力的かもしれない。ただし、各点のL GIは、関心のgyral領域での平均lの GIにもある程度関心の近隣地域でgyrificationが反映されるように、周囲の円形の領域にgyrificationを定量化する。
最後に、最も重要な問題は、このプロトコルでは神父の間に発生する可能性のある他の問題に対する解決策を説明していたものの、eeSurfer または l GI処理はFreeSurferメーリングリスト (のアーカイブで見つけることができますhttp://surfer.nmr.mgh.harvard.edu/fswiki/FreeSurferSupport )。
6。代表的な結果
プロトコルのセクション1cで説明したように、常に慎重に前リットル GIの計算に皮質表面の復元の精度を確認する必要があります。前頭葉と後頭葉の間にスクロールしながら、血管や細胞膜を軟膜表面に含まれていないことに特に注意を払う。白い面が正確に灰白色のインタフェースを以下のことも確認してください。正しい再建の例は、( 全体のボリュームのアニメーションGIFの図を参照 )図2に提供されています。
リットル GI計算の最後に、また各被験者の両半球の結果を確認する必要があります。1より小さいリットル GIの結果を持つ任意の皮質領域は存在しないはずです。プロトコルと図4のセクション3 は 、l GI計算の出力が正しいかどうか、正しいチェックする方法を示します。
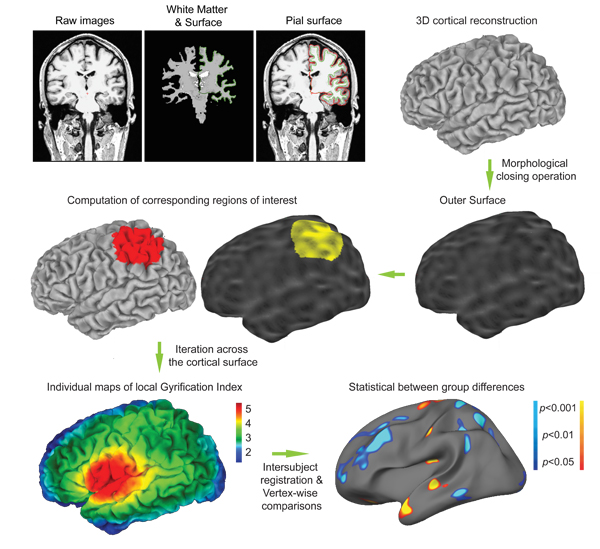
図1 リットル GI計算の概要はまず 、三次元皮質メッシュモデルは、標準FreeSurferのパイプラインを使用してRAW画像から再構成されています。これらの再構成アルゴリズムは埋め込み溝の問題を克服するための出発点としてバイナリ白質のボリュームを使用してください。皮質メッシュモデルは、一般的に約15万の頂点を含んでおり、古典的に各ポイントで皮質の厚さを計算するために使用されています。同様に、ローカルGyrification指数(L GI)は、各頂点で計算されます。その目的のために、外側の表面が作成されます。その後、関心の対応する円形領域は外側で識別されますマッチングアルゴリズムを使用してdを皮質表面。興味の重なり合う領域、L GIの個々のマップの作成 のプロセスの結果を生成する約800の後。これらのマップは簡単に解釈することができます:5の手段のインデックスをより多くの皮質表面が周囲に溝内に陥入5時間があることをその目に見える皮質表面の量、皮質が周辺地域に平坦であることを意味のインデックス。最後に、統計的グループの比較は、皮質の厚さの比較と同様に、各頂点のレベルで計算されます。
図1B。 リットル GIの個々の皮質マップは、この小さな映画は、図1に示すように、個々の皮質LGIマップの360度の回転を示しています。 1。それは、より高いリットル GI値を持つ皮質領域は、子宮内の生活の中で中に作成される最初の倍に相当することに注意することは顕著である:シルビウス裂、上側頭溝とのtraparietalの脳の側面図で溝、および脳の内側のビュー上頭頂後頭溝が。 映画を見る
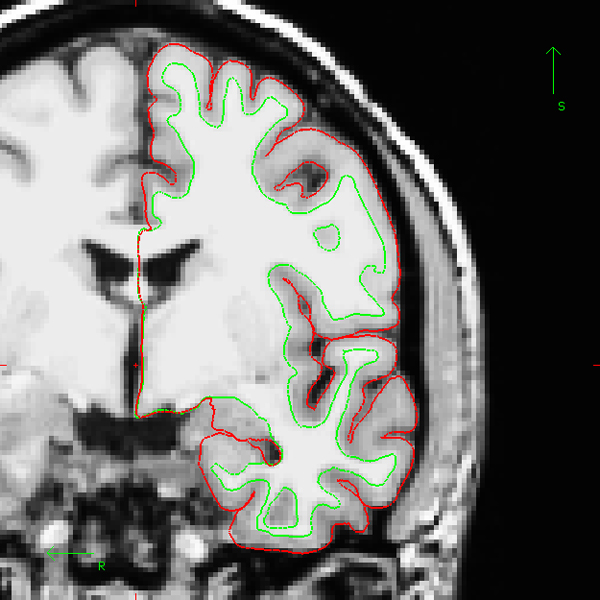
図2。十分な皮質表面再構成(一冠状断面)の例。復興プロセスの終了後、皮質表面は正確に全体脳容積にわたって検証する必要があります。内側の皮質表面には、(画像上に緑色で、白い面を示す)正確に灰白色のインターフェースに従ってください。外側の皮質表面には(すなわちグレー- CSFインタフェース、ここで赤で、軟膜表面を示す)容器や膜のどの部分を含めないでください。注目すべきは、ここで紹介する例では、FreeSurferパッケージとともに配布"BERT"テーマを使用しています。
図2B。十分な皮質表面reconstruの例ction(フルボリューム)が、このアニメーションGIFイメージは、ほとんど正面からFreeSurferで最も頭冠状セクションにスクロールして見られる各冠状セクションの"BERT"主題、の左半球の皮質表面を示しています。 表示ムービーを

図3。外面の例リットル GIプロセス(一冠状断面)の一部として計算。 リットル GI計算の最初のステップは、半球を包む外側の面の作成 です。この表面は、(示さ?FreeSurferでh.pial_outer_smoothed)tkmeditを使って確認することができます。ここでは、FreeSurferと共に配布"BERT"主題を例として使用されます。
図3B。 リットル GIプロセス(フルボリューム)の一部として計算外面の例。このアニメーションGIFの画像として最も正面からFreeSurferでtkmeditで最も頭冠状セクションにスクロールして見られる各冠状断面で左半球の外側面を示しています。 ビューの映画は、
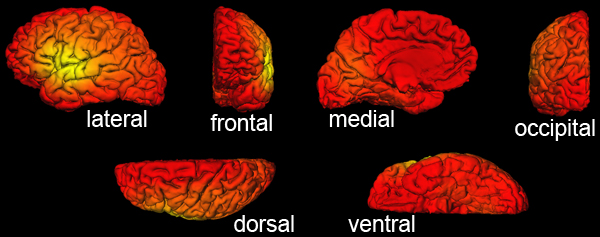
図4。 FreeSurferで見たときに正しいリットル GIの出力例を示します。重ねリットル GI値を持つ"BERT"主題の皮質表面の異なる方向に。カラーコードは、FreeSurferでtksurferで見られるデフォルトの"熱"オーバーレイです。 1の最小しきい値を使用して、すべての頂点が着色する必要があり、皮質領域はapすべきグレーの梨。注目すべきは、色のオーバーレイは、最小値と最大値、同様にL GIの全体的な分布のヒストグラムもチェックできるtksurfer、で"オーバーレイを設定"オプションを使用して変更できます。
ディスカッション
上記のプロトコルは、脳のT1強調MRIに基づくローカルGyrification指数を測定し、統計的グループの比較を実施する方法について説明します。私たちの方法は、特に皮質膨張過程で早期に混乱をローカライズし、多くの神経発達や精神状態に特に関心などであるように設計されています。臨床サンプルのグループの比較の例は私たちのグループ1,12、あるいは他の人13〜16によって?...
開示事項
著者らは、開示することは何もない。
謝辞
スイス国立科学財団(N · 51AU40_125759)によって賄わ - この研究は、研究の能力のナショナルセンター(NCCR)"精神疾患のシナプス拠点SYNAPSY"によってサポートされていました。ローカルGyrification指数の開発は、スイス国立研究基金から博士マリーSchaer(323500〜111165)へと博士ステファンEliez(3200〜063135.00 / 1、3232から063134.00 / 1、PP0033 - 102864および32473Bへの補助金によって支えられて-121996)とジュネーブ - ローザンヌ大学とローザンヌ連邦工科大学の生物医学イメージング(CIBM)だけでなく、基礎のLeenaardsとルイJeantetセンターによる。 FreeSurferソフトウェアの開発のための支援は、研究資源のための国立センター(P41 - RR14075、およびNCRR BIRN形態計測プロジェクトBIRN002、U24 RR021382)、生物医学イメージングと生体工学研究所(R01 EB001550、R01EB006758)、によって部分的に提供されていました国立神経疾患研究所およびストローク(R01 NS052585 - 01)だけでなく、精神障害と神経科学の発見(MIND)研究所とは、医学研究のためのNIHロードマップ、グラントU54を通じて国立衛生研究所によって資金を供給医用画像のコンピューティングのための国民の同盟(NAMIC)、の一部です。 EB005149。追加のサポートは、エリソン医学財団によって資金を供給自閉症&ディスレクシアプロジェクトによって提供されていました。
資料
材質:2GHz以上のプロセッサと4GBのRAMの最小値とUnixやMacワークステーション、FreeSurferがインストールされています(とhttp://surfer.nmr.mgh.harvard.edu/fswiki 、好ましくは最新のバージョンが、ないより古いバージョン4.0.3より)。ローカルGyrificationインデックスを計算するために、MATLABは、(要求されるhttp://www.mathworks.com/ Image Processing Toolboxのと一緒に)。
データ:良質のサンプル(高解像度、高コントラスト)脳MRI T1強調データセット。被験者のあなたのグループは、好ましくは、年齢や性別は整合させる必要があります。脳形態で通常の個体間のばらつきを考えると、各群の被験者数は、既存のグループの違いを( - よりよいより多くの)識別するのに十分なはずです。妥当な最小のサンプルサイズは約20人の被験者になるグループごとに(あなたのグループが緊密に性別と年齢をマッチされている場合、変更の強度が大きく、場合は、おそらく少ないために行くことができますが)。
| Name | Company | Catalog Number | Comments |
| 機器の名前 | 会社 | カタログ番号 | コメント |
| FreeSurfer | 生物医学イメージング、MGHのためのMartinosセンター | 4.0.3より新しいバージョン | |
| MathWorks社のMATLAB | Mathworks社 | 画像処理ツールボックス |
参考文献
- Schaer, M. A surface-based approach to quantify local cortical gyrification. IEEE. Trans. Med. Imaging. 27, 161-170 (2008).
- Mangin, J. F., Jouvent, E., Cachia, A. In-vivo measurement of cortical morphology: means and meanings. Curr. Opin. Neurol. 23, 359-367 (2010).
- Mangin, J. F. A framework to study the cortical folding patterns. Neuroimage. 23, S129-S138 (2004).
- Luders, E. A curvature-based approach to estimate local gyrification on the cortical surface. Neuroimage. 29, 1224-1230 (2006).
- Zilles, K., Armstrong, E., Schleicher, A., Kretschmann, H. J. The human pattern of gyrification in the cerebral cortex. Anat. Embryol. (Berl). 179, 173-179 (1988).
- Rakic, P. Specification of cerebral cortical areas. Science. 241, 170-176 (1988).
- Toro, R. Brain size and folding of the human cerebral cortex. Cereb. Cortex. 18, 2352-2357 (2008).
- Fischl, B., Sereno, M. I., Dale, A. M. Cortical surface-based analysis. II: Inflation, flattening, and a surface-based coordinate system. Neuroimage. 9, 195-207 (1999).
- Dale, A. M., Fischl, B., Sereno, M. I. Cortical surface-based analysis. I. Segmentation and surface reconstruction. Neuroimage. 9, 179-194 (1999).
- Genovese, C. R., Lazar, N. A., Nichols, T. Thresholding of statistical maps in functional neuroimaging using the false discovery rate. Neuroimage. 15, 870-878 (2002).
- Desikan, R. S. An automated labeling system for subdividing the human cerebral cortex on MRI scans into gyral based regions of interest. Neuroimage. 31, 968-980 (2006).
- Schaer, M. Congenital heart disease affects local gyrification in 22q11.2 deletion syndrome. Dev. Med. Child. Neurol. 51, 746-753 (2009).
- Palaniyappan, L., Mallikarjun, P., Joseph, V., White, T. P., Liddle, P. F. Folding of the Prefrontal Cortex in Schizophrenia: Regional Differences in Gyrification. Biol. Psychiatry. , (2011).
- Zhang, Y. Decreased gyrification in major depressive disorder. Neuroreport. 20, 378-380 (2009).
- Juranek, J., Salman, M. S. Anomalous development of brain structure and function in spina bifida myelomeningocele. Dev. Disabil. Res. Rev. 16, 23-30 (2010).
- Zhang, Y. Reduced cortical folding in mental retardation. AJNR. Am. J. Neuroradiol. 31, 1063-1067 (2010).
- Kuperberg, G. R. Regionally localized thinning of the cerebral cortex in schizophrenia. Archives of general psychiatry. 60, 878-888 (2003).
- Milad, M. R. Thickness of ventromedial prefrontal cortex in humans is correlated with extinction memory. Proc. Natl. Acad. Sci. U. S. A. 102, 10706-10711 (2005).
- Rauch, S. L. A magnetic resonance imaging study of cortical thickness in animal phobia. Biol. Psychiatry. 55, 946-952 (2004).
- Fjell, A. M. Selective increase of cortical thickness in high-performing elderly--structural indices of optimal cognitive aging. Neuroimage. 29, 984-994 (2006).
- Walhovd, K. B. Regional cortical thickness matters in recall after months more than minutes. Neuroimage. 31, 1343-1351 (2006).
- Gold, B. T. Differing neuropsychological and neuroanatomical correlates of abnormal reading in early-stage semantic dementia and dementia of the Alzheimer type. Neuropsychologia. 43, 833-846 (2005).
- Salat, D. H. Thinning of the cerebral cortex in aging. Cereb. Cortex. 14, 721-730 (2004).
- Schaer, M., Eliez, S. Contribution of structural brain imaging to our understanding of cortical development process. European Psychiatry Reviews. 2, 13-16 (2009).
- Shaw, P. Neurodevelopmental trajectories of the human cerebral cortex. J. Neurosci. 28, 3586-3594 (2008).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved