Method Article
人线粒体 DNA 中核苷酸的同时定位与定量
摘要
在这里, 我们描述一个方法可以同时定量和基因组范围内的地图核苷酸在高度完整的 dna 在核苷酸决议, 结合酶裂解的基因组 dna 与其碱性水解和随后5´端序.
摘要
建立的方法来估计核苷酸目前在一个基因组的数量是有限的定量的合并核苷酸使用短合成 DNA 片段或质粒作为模板, 然后推断结果的整体基因.或者, 在基因组中存在的核苷酸的数量可以用碱性凝胶或南方印迹来估计。最近的在体内方法采用下一代测序, 允许核苷酸的全基因组映射, 提供嵌入核苷酸的位置和身份。然而, 他们不允许定量的核苷酸的数量被纳入到一个基因组。在这里, 我们描述了如何同时映射和定量的数量核苷酸被纳入人类线粒体 DNA在体内的下一代测序。我们使用高度完整的 DNA 和引进序列特定的双链断裂通过消化它与切, 随后水解合并核苷酸与碱。生成的端与适配器相连, 这些端点在下一代测序机上进行排序。核苷酸的绝对数量可以计算为在识别站点的每个平均读取次数以外的读取次数, 为序列特定的切。这个协议也可以用来映射和定量自由的 dna 的缺口, 并允许适应地图其他 dna 病变, 可以处理 5´-OH 结束或5´磷酸盐结束。此外, 这种方法可以适用于任何生物体, 鉴于适当的参考基因组是可用的。因此, 该协议为研究 dna 复制、5´端处理、dna 损伤和 dna 修复提供了重要的工具。
引言
在真核细胞中, 核苷酸 (rNTPs) 的浓度比脱氧核糖 (dNTPs)1的浓度高得多。dna 聚合歧视核苷酸, 但这种歧视是不完美的, 因此, 核苷酸而不是脱氧核糖可以纳入基因组在 dna 复制。核苷酸可能是最常见的非核苷酸纳入基因组2。大多数这些核苷酸在冈片段成熟过程中被核糖核酸 H2 启动核苷酸切除修复 (中) 或拓扑 1 (在参考3中进行了审查)。无法删除的核苷酸在 DNA2、4中稳定地合并在一起, 并可能以有害和有益的方式影响它 (在评审的5中进行了审阅)。除能作为正面信号以外, 例如在交配类型开关在粟殖6 和标记新生的脱氧核糖核酸子线在不匹配修理期间 (MMR)7,8, 核苷酸影响结构9和周围 DNA 的稳定性由于其核糖的 2´-羟基组的10, 导致复制应力和基因组不稳定性11。核苷酸在基因组 DNA (gDNA) 中的丰度及其在复制和修复机制中的相关性, 以及对基因组稳定性的影响, 使人们有理由以全基因组的方式研究其精确发生和频率。
核糖核酸 H2 活性在人类线粒体中没有发现, 因此在线粒体 DNA 中核苷酸没有有效的去除。有几个途径涉及的核苷酸供应人的线粒体和调查是否干扰线粒体核苷酸池导致高数量的核苷酸在人类线粒体 dna, 我们制定了一个协议的地图和定量这些核苷酸在人类线粒体中分离出成纤维细胞、HeLa 细胞和患者细胞系12。
大多数体外方法 (在复查的13中进行了审查) 以确定 DNA 聚合对 rNTPs 的选择性是基于单核苷酸插入或引物扩展实验, 其中包括在反应中的竞争 rNTPs混合, 允许识别或相对定量的核苷酸纳入短 DNA 模板。对短序列的定量方法可能不反映细胞浓度的 dNTP 和 rNTP 池, 因此提供了对聚合酶选择性的洞察, 但对于整个基因组来说意义有限。研究表明, 在复制较长的 dna 模板 (如质粒) 的过程中, 核苷酸的相对数量可以在使用标记 dNTPs 的测序凝胶上进行可视化, 并在碱性环境中水解 dna (14。此外, gDNA 在碱性水解后对南方印迹进行了分析, 从而使核苷酸的绝对速率的探测和测定在体内在活体中15。这些方法允许对合并频率进行相对比较, 但不提供对合并的核苷酸的位置或身份的洞察力。更近的方法来分析 gDNA中的核苷酸内容, 如海顿-seq16、核糖-seq17、Pu seq18或 emRiboSeq19, 利用嵌入的核苷酸 "对碱性或核糖核酸 H2 治疗的敏感性, 并采用下一代测序方法确定核苷酸基因组范围。这些方法不提供对检测的核苷酸的绝对并网频率的洞察。通过在海顿-seq 协议中加入序列特定的酶解的步骤, 我们在这里描述的方法可以方便地扩展从排序方法获得的信息, 允许同时映射和定量的嵌入核苷酸12。这种方法适用于几乎任何生物体, 因为可以生成高度完整的 DNA 提取物, 并提供合适的参考基因组。该方法可以适应定量和确定任何病变的位置, 可以消化的核酸和叶子5´磷酸盐或 5´-OH 结束。
为了映射和定量核苷酸在基因组 DNA 中, 该方法结合了一个序列特定的切和碱性水解产生 5´-磷酸盐结束在特定的识别序列为切位于和5´的地方OH 结束在位置核苷酸位于。由于生成的自由端随后会与适配器相连, 并使用下一代测序进行测序, 因此在 dna 提取和文库制备过程中, 使用高度完整的 dna 并避免随机碎片是非常重要的。将这些读数归一化到切劈裂点的读数, 可以同时定量和映射检测到的核苷酸。在控制实验中检测到游离 5´, 在碱性水解 DNA 的情况下用氯化钾进行处理。所获得的数据提供了洞察核苷酸的位置和数量, 并允许分析关于核苷酸的内容和注册频率。
研究方案
本协议概述在 图 1 中, 包括 gDNA 的隔离, 用限制性酶消化, 可以定量核苷酸的数量, 用碱水解处理磷酸的核苷酸结合到 gDNA, 磷酸化的自由5和 #180;-OH 结束, ssDNA 结扎的适配器, 第二链合成, PCR 扩增前排序.
1. 适配器和索引引物
- 获取 ARC49、ARC140 寡核苷酸、ARC76/77、适配器和 ARC78-ARC107 索引底漆 (请参见 表 1 )。 注: 寡核苷酸应进行高效液相色谱纯化。ARC76/77 被订购为双面打印器.
- 准备100和 #181; 在三 EDTA (TE) 缓冲区中的每个寡核苷酸的库存解决方案 (请参见 材料表 ) 并存储在-20 和 #176; C.
- 准备10和 #181; m ARC67/77 和2和 #181 的解决方案; ARC49 和指数引物稀释洗脱缓冲液 (EB; 请参见 材料表 )。存储在-20 和 #176; C.
2。细胞的生长和收获
- 在70毫升 Dulbecco 和 #39 中生长 HeLa 细胞, 在37和 #176 的250毫升旋转瓶中补充10% 胎牛血清 (DMEM); C.
- 计数单元格数, 并收集 5x10 6 50 毫升管中的单元格, 离心5分钟, 200 x g, 并丢弃上清液.
- 20 毫升 1x PBS 清洗细胞, 离心5分钟, 在 200 x g, 并丢弃上清液.
- 将小球冷冻在-20 和 #176; C 或继续进行 DNA 纯化.
3。DNA 纯化和定量
- 使用苯酚-氯仿萃取法纯化 gDNA, 如下所述.
- 重2毫升裂解缓冲液中的单元格 (参见 材料表 ) 并在42和 #176 中孵育30分钟; C 在加热块上.
警告: 裂解缓冲液含有有害成分。SDS 溶液是刺激性的, 蛋白酶 K 是敏感的, 刺激性的, 有毒的。穿戴防护服和手套. - 将样品分成两个2毫升的管子, 并加入1卷 (V) 的苯酚-氯仿-异戊醇 (25:24:1).
注意: 苯酚-氯仿-异戊醇是有毒、诱变、腐蚀性的, 对水生环境有害。在油烟机中使用, 穿戴防护服和手套, 并在特殊的苯酚氯仿废物中丢弃. - 混合 30-60 s 和离心5分钟在室温下 1.5万 x g.
注意: 不要涡旋 DNA 以避免引入随机链断裂, 这会扭曲结果. - 将上部、水相转移至新的2毫升管, 并加入 1 V 的苯酚-氯仿-异戊醇 (25:24:1).
- 在 1.5万 x g 处以反转和离心机混合5分钟, 在4和 #176; C.
- 将上部、水相转移到新的2毫升管上, 并加入20和 #181; (5 米) 和 1 V 的冷异丙醇.
注意: 异丙醇是易燃、刺激性和有毒的。把它存放在通风柜里, 穿上防护服和手套, 并远离火焰. - 混合通过反转和孵化至少1小时, 在-20 和 #176; C.
- 离心20分钟, 在 1.5万 x g, 4 和 #176; C 和丢弃上清.
- 用200和 #181 清洗 DNA 颗粒; L 冷70% 乙醇, 离心20分钟, 1.5万 x g 在4和 #176; C 和丢弃上清.
注意: 70% 乙醇是易燃和刺激性。在-20 和 #176 保持工作解决方案, 否则在通风柜内存放, 穿戴防护服和手套, 并使其远离火焰. - 室温下干燥 DNA 颗粒20-25 分钟.
- 在100和 #181 中溶解 DNA 颗粒; 用一根管子将样品放入缓冲池中.
- 重2毫升裂解缓冲液中的单元格 (参见 材料表 ) 并在42和 #176 中孵育30分钟; C 在加热块上.
- 根据制造商和 #39 的规格, 使用 dsDNA 定量试剂定量 DNA 浓度 (请参见 "材料 表 ).
注: 使用 dsDNA 定量试剂, 因为分光光度法可以影响残留苯酚的 DNA 定量. - 将 DNA 存储在-20 和 #176; C 或继续 HincII 治疗.
4。HincII 处理和碱性水解
- 摘要1和 #181; 在含有5和 #181 的反应混合物中的 DNA g; l 10x 缓冲器3.1、1和 #181; l (10 U) HincII 和无核酸 H 2 O 到最终的50和 #181 的容量; l.
注: 为达到最佳结扎条件, 第二链合成和 PCR 扩增, 如果预期 dna 中含有极少的核苷酸, 可能需要增加输入 dna 的数量。同样, 如果核苷酸的数量非常高, 可能有必要减少输入 DNA. - 在37和 #176 孵育30分钟; C.
- 用顺磁珠净化 HincII 处理的 DNA.
注意: 在下列步骤中保持管盖打开, 以避免打开管子来扰乱小球。- 将 1.8 V 的顺磁性珠子加到每个样品中, 小心地由移混合, 在室温下孵育10分钟.
- 使用磁性机架将珠子颗粒5分钟, 然后除去并丢弃上清.
- 用150和 #181 清洗颗粒; 70% 乙醇 (室温), 大约三十年代, 然后除去和丢弃上清液.
- 用200和 #181 清洗颗粒; 70% 乙醇 (室温), 大约三十年代, 然后除去和丢弃上清液.
注: 剩余乙醇可以用10和 #181 去除; L 吸管。水滴可以预先被简单地纺下来. - 在室温下干燥样品15-20 分钟左右.
注: 确切的时间取决于珠子的体积和颗粒的形状, 因此, 球团应该在视觉上进行检查. - 在45和 #181 中从磁性机架和洗颗粒中取出管; L EB, 小心移混合.
- 孵育5分钟, 然后在磁性机架上将珠子颗粒化, 并使用45和 #181; 在步骤4.4 中纯化的 DNA.
- 添加5和 #181; KOH (3 米) 或氯化钾 (3 米) 的 l, 生成总体积为50和 #181 的 DNA; l.
警告: 3 米 KOH 溶液具有腐蚀性。穿戴防护服和手套. - 在55和 #176 孵育2小时; C 在杂交烤箱中, 在冰上5分钟.
注意: 建议在烤箱中进行 KOH 处理, 而不是加热块来保持管子的均匀加热, 防止盖子上的冷凝. - 通过加入10和 #181 沉淀 DNA; 醋酸钠 (3 米, pH 值 = 5.2) 和125和 #181; l 冷100% 乙醇。在冰上孵育5分钟.
注意: 100% 乙醇是易燃和刺激性。存放在通风柜内, 穿戴防护服和手套, 远离火焰. - 颗粒 gDNA 离心 2.1万 x g, 4 和 #176; C 5 分钟, 并丢弃上清.
- 用250和 #181 清洗 DNA 颗粒; L 冷70% 乙醇, 离心机在 2.1万 x g, 4 和 #176; C 为5分钟, 并丢弃上清.
注: 为了除去液滴, 管子可以再一次地被纺下来并且上清可以用10和 #181 去除; l 吸管. - 让颗粒在开管中干燥约5-10 分钟, 直到任何可见液体蒸发.
- 让 DNA 颗粒在20和 #181 中溶解; 在室温下, L EB 30 分钟.
5. 5 和 #180; 结束磷酸化
- 为每个样本预先准备好反应混合物isting 2.5 和 #181; l 10x T4 核苷酸激酶反应缓冲器, 1 和 #181; l (10 U) 3 和 #180;-磷酸酶-减去 T4 核苷酸激酶, 2.5 和 #181; l ATP (10 mM).
- 将19和 #181; 将每个 DNA 样本的 l 转换为新的200和 #181; l 管和变性为3分钟, 在85和 #176; C 在热循环.
- 在冰上冷却 DNA 样品, 并增加6和 #181; 每个样品的反应混合 L.
- 孵育反应混合在37和 #176; c 为30分钟和停止反应通过孵化样品在65和 #176; c 为 20 min.
- 纯化 DNA 如4.3 所述, 使用 1.8 V 的顺磁性珠, 但洗在14和 #181; L EB.
6. ssDNA 结扎
- 预先为每个样品准备反应混合物, 由0.5 和 #181 组成; l ATP (2 毫米), 5 和 #181; l 10x T4 RNA 连接反应缓冲器, 5 和 #181; l CoCl 3 (NH 3 ) 6 (10 mm),0.5、#181; l ARC140 (100 #181; M), 25 和 #181; 50% PEG 8000。混合移.
警告: CoCl 3 (NH 3 ) 6 对水生环境具有致癌、敏感和危险。穿戴防护服和手套. - 转移13和 #181; 从步骤5.5 到新的200和 #181 的纯化 DNA l 管和变性为3分钟, 在85和 #176; C 在热循环.
- 在冰上冷却 DNA, 并增加36和 #181; 每个样品的反应混合, 移混合, 然后简单地旋转.
- 添加1和 #181; L (10 U) T4 RNA 连接到每个反应, 混合移, 并简要地向下旋转.
- 在夜间在室温下孵育样品.
7。第二条线合成
- 纯化结扎的 DNA, 如4.3 所述, 但使用 0.8 V 的顺磁性珠子, 颗粒的珠子为10分钟和洗在20和 #181; L EB.
注: 由于结扎反应混合物的粘度较高, 第一粒化步骤延长. - 将20和 #181 的 DNA 样本转移到一个新的200和 #181, l PCR 管。在14和 #181 中, 按照制造商和 #39 的规格和洗, 重复使用0.8 伏顺磁性珠的净化步骤; L EB.
- 提前准备每个样品的反应组合, 包括2和 #181; 10x T7 DNA 聚合酶反应缓冲液, 2 和 #181; l ARC76/77 (2 #181; M), 2 和 #181; l dNTPs (2 毫米), 0.8 和 #181; l BSA (1 毫克/毫升).
- 传输12.8 和 #181; 将 DNA 纯化为新的200和 #181; l 管, 变性为3分钟, 在85和 #176; C 在热循环.
- 在冰上冷却 DNA 并增加6.8 和 #181; 每个样品的反应混合, 移, 短暂旋转, 室温下孵育5分钟.
- 添加0.4 和 #181; L (4 U) T7 DNA 聚合酶对每个反应和孵化5分钟的室温.
- 纯化 DNA 如4.3 所述, 使用 0.8 V 的顺磁性珠和洗在11和 #181; L EB.
8。PCR 扩增与文库定量
- 在新的200和 #181 中为每个样本准备反应组合; l 管预先由7.5 和 #181 组成; l ARC49 (2 和 #181; m), 7.5 和 #181; l 索引底漆 (2 和 #181; m, 每个样本都是唯一的), 以及25和 #181; l 2x 热启动准备混合.
- 添加10和 #181; 每个反应的 DNA 样本 L。使用以下条件放大库: 变性95和 #176; c 为四十五年代, 其次18周期98和 #176; c 为十五年代, 65 和 #176; c 为三十年代, 72 和 #176; c 为三十年代, 结束以最后伸长在72和 #176; c 为 2 min. 在4和 #176 举行样品;放大后的 C.
- 净化库, 如4.3 所述, 使用 0.8 V 的顺磁性珠子, 并在20和 #181 中洗; 我是缓冲器.
- 定量库使用 dsDNA 定量试剂, 根据制造商和 #39; s 规范 (请参阅 材料表 ).
- 将示例存储在-20 和 #176; C 或继续进行库分析.
9。库分析和池
- 确定每个库的质量, 并使用数字电泳系统估计平均碎片大小.
注意: 平均碎片大小是通过估计电泳曲线下的面积减半, 不理会标记的峰值。在 图 2A 中给出了在 KOH 或氯化钾处理后合适的库剖面的代表性结果. - 计算库的浓度 (nM) 为:
(c/10 3 )/(p * 650)] * 10 9
, 其中 c 是 #181 库的集中位置; L 和 p 是 bp 中的平均碎片大小, 估计为 9.1. - 池相等的摩尔量多达24库, 用不同的索引引物放大以进行排序。将 TE 缓冲区添加到25和 #181 的最后一卷; L 和浓度 10 nM.
注: 根据要汇集的库的数量, 每个库中的 DNA 数量将被调整。如果在步骤9.1 中检测到底漆聚是大约 130 bp 的一个显著峰值, 库池的最终容量可以超过25和 #181; l, 因为纯化步骤是重复的描述, 在 4.3, 使用 0.8 V 的顺磁性珠, 和 DNA 是洗在25和 #181; 我 buffer.- 根据制造商和 #39 的规格和上面所述的平均峰值大小, 使用 dsDNA 定量试剂确定新的库池浓度。进行排序和数据分析 (10 和11节).
10。对
- 在池库上执行75基配对结束顺序排序 12 .
11. 数据分析
- 修剪所有读取以删除适配器序列, 筛选质量和读取长度.
注意: 这可以通过使用 cutadapt 1.2.1 20 来完成, 并带有命令 "cutadapt-f fastq-匹配-读-通配符-安静-m 15-q 10-NNNNNNN 和 #60; 文件和 #62", 其中 NNNNNNN 被实际的适配器序列和 #60 替换; 文件和 #62;替换为 fastq 文件名. - 删除在上一步中使用自定义脚本丢弃的读取的伙伴.
- 将剩余对的配对1与包含库准备中使用的所有寡核苷酸序列的索引 ( 如 使用领结 0.12.8 21 和命令行选项-m1-v2) 对齐。放弃成功对齐的所有对.
- 使用领结与命令行选项-v2-X10000--best, 将剩余对与生物体参考基因组对齐.
- Map 通过将所有齐对的配对 (使用领结与命令行选项-v2) 对齐, 在线粒体分子的起始和结束之间进行跨距的读取.
- 确定5和 #180 的计数;-结束所有单个和配对的端对齐。将这些位置由一个基向上游转移到水解核苷酸的位置.
- 使用自定义脚本将数据从领结文件格式导出到 bedgraph 文件格式, 以便在常见的基因组浏览器中进行可视化。将每个子链的读取正常化为每百万读取.
- 使用 bedgraph 文件中的位置和计数, 参照生物体基因组序列确定 incorp 的身份orated 核苷酸.
注: 对于人类线粒体基因组读取的区域16,200-300 和 5,747-5, 847 为每个链应该被排除, 因为这些区域包含许多自由5和 #180;-结束与核苷酸合并的脱氧核糖核酸聚合酶并且 #947;. - 将总读取数 (不包括十一 HincII 站点上的读取) 与每个 HincII 站点的平均读取次数进行划分, 以获取每个单链断裂的核苷酸数 ( 即 每个线粒体分子的核苷酸数).
结果
通过对上述方法的说明, 对 HeLa 细胞线粒体 DNA 的代表性数据进行了分析12。图 2B显示了在氯化钾治疗后 (左面板) 的人类线粒体 dna 的重 (HS) 和光链 (LS) 的所有 HincII 站点的汇总读取。约70% 的所有检测到的5´端点定位到切割网站, 证明了 HincII 消化的高效率。利用 KOH 处理库在嵌入式核苷酸中水解 DNA 减少了 HincII 站点上的读取次数, 使其数量增加到大约 40% (图 2B, 右面板)。这是意料之中的, 因为在核苷酸公司的网站上产生了大量的5´端, 这表明图书馆的质量是足够的。图 2C说明了5´处理后的定位和频率 (绿色), 在 KOH 处理后由海顿-seq (洋红) 生成的读数, 通过碱性水解检测核苷酸的自由5´端和端部。左侧面板中显示了自由的5´端和核苷酸定位到人类线粒体的 HS, 而那些本地化到 LS 的则显示在右侧的面板中。在核苷酸 (图 2D, 上面板) 或 HincII 站点 (下部面板) 上的原始读数的相对数量分别为 hs 和 ls 的14倍或31倍的相对于 hs 的 ls, 而不观察到类似的偏差核 DNA这一股偏倚可以解释为两股的基本组成不同的差异, 并说明了正常化的重要性, 在 HincII 网站读取。
将读取计数正常化为 HincII 给出了每个线粒体基因组 (图 3A) 的核苷酸数量的定量测量。如图 3B所示, 对每个核苷酸的每个子链的序列组成进行了 KOH 处理后的读取, 显示了不同于1的比值, 表明随机分布的读数表明有不同的核苷酸模式和高的图书馆质量。该比值不受先前消化与 HincII 的影响 , 验证酶的特异性。将嵌入核苷酸的站点上的读数正常化到 HincII 裂解点的位置, 以及基因组核苷酸含量, 可生成每1000个互补碱基 (图 3C)。
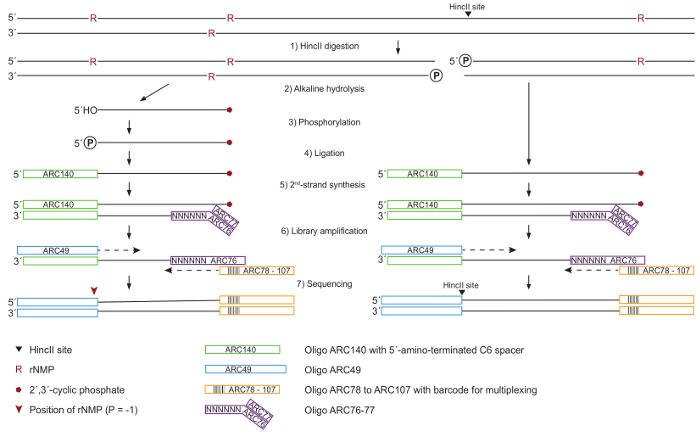
图 1: DNA 处理和库准备的示意图.(1) 整个基因组 DNA 被 HincII 在随后的定量核苷酸中被切割, 在 HincII 点 (黑箭头) 产生钝端。(2) 在核苷酸的地方, DNA 被 KOH 水解, 导致 2´, 3´循环磷酸盐 (红色五边形) 在3´末端和自由 5´-OH 末端。(3) 5´-OH 末端是磷酸化由 T4 核苷酸激酶 3´-磷酸酶-减去。(4) T4 RNA 连接将所有携带磷酸基团的5´端与 ARC140 寡核苷酸结扎。(5) 利用 T7 DNA 聚合酶和含有随机 N6序列的 ARC76-77 寡核苷酸合成第二链。(6) 利用 ARC49 的高保真 DNA 聚合酶和 ARC78 对 ARC107 索引底漆中的一种, 包含了一个用于多路复用的独特条码的库。(7) 5´端是通过配对端测序来定位的。请单击此处查看此图的较大版本.

图 2: 方法验证.(A) 代表 electropherograms 使用自动电泳系统来确定在 KOH 或氯化钾处理下生成的库的质量. (B) 在重 (HS) 和轻链 (LS) 的 HincII 站点上汇总信号人类 dna 在氯化钾 (左面板) 或 KOH (右板) 治疗后。(C) Circos 的自由5´端 (绿色) 和海顿-Seq (自由5´端和核苷酸、洋红) 在 HS (左面板) 和 LS (右面板) 人类线粒体的数字。峰值被规范化到每百万读并且最大峰值被调整到海顿-seq 图书馆的最大读的数量。(D) 在核苷酸 (上部面板) 和 HincII 站点 (下部面板) 中, 在人类线粒体 (图中) 或在核 (校正) 中的反向 (RV) 或正向 (FW) 链中, 对原始读数进行了汇总。dna.图 B、C 和 D 是从引用12改编的。误差线表示平均值的标准误差。请单击此处查看此图的较大版本.

图 3: 代表性结果.(A) 在重 (H) 或轻 (L) 链上, 核苷酸的相对数量被规范化为在 HincII 站点上读取 KOH 处理的库。(B) 核苷酸特征与线粒体 dna 基因组组成的比值 (koh) 和 HincII 与 koh 处理 (HincII + koh) 文库在线粒体 dna 的重 (H) 或光 (L) 链上劈开。(C) 核苷酸频率正常化为1000互补基地的 HincII 和 KOH 处理图书馆的重 (H) 或轻 (L) 链的线粒体。数字是从引用12改编的。误差线表示平均值的标准误差。请单击此处查看此图的较大版本.
| 名称 | 序列 | |||
| ARC49 | AATGATACGGCGACCACCGAGATCTACACTCTTTCCCTACACGACGCTCTTCCGATCT | |||
| ARC76 | GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTNNNN * n | |||
| ARC77 | AGATCGGAAGAGCACACGTCTGAACTCCAGTC * C | |||
| ARC78 | CAAGCAGAAGACGGCATACGAGATCGTGATGTGACTGGAGTTCAGACGTGTGCTCTTCCGATCT | |||
| ARC84 | CAAGCAGAAGACGGCATACGAGATACATCGGTGACTGGAGTTCAGACGTGTGCTCTTCCGATCT | |||
| ARC85 | CAAGCAGAAGACGGCATACGAGATGCCTAAGTGACTGGAGTTCAGACGTGTGCTCTTCCGATCT | |||
| ARC86 | CAAGCAGAAGACGGCATACGAGATTGGTCAGTGACTGGAGTTCAGACGTGTGCTCTTCCGATCT | |||
| ARC87 | CAAGCAGAAGACGGCATACGAGATCACTGTGTGACTGGAGTTCAGACGTGTGCTCTTCCGA | |||
表 1: 寡核苷酸。列出了用于海顿-Seq 的寡核苷酸.粗体面表示索引。* 表示硫键。ARC140 包含一个 5´-氨基组, 而不是一个 5´-OH 组, 结合 C6 链接。这种改变减少结扎过程中 ARC140 concatemers 的形成。
讨论
在这里, 我们提出了一项技术, 同时地图和量化核苷酸在 gDNA, 特别是 dna, 通过简单的介绍在序列特定地点的基因组, 作为一个补充建立的海顿-seq 协议。虽然这项研究的重点是人类的线粒体, 最初的海顿-seq 方法是在酿酒酵母, 说明该方法的翻译到其他生物体12,16。
对于从这种方法获得的可靠结果, 应注意一些关键步骤: (A) 由于顺序适配器结扎到所有可用的5´端, 因此, 使用高度完整的 DNA 是至关重要的。dna 应该被隔离, 图书馆应该在 dna 分离后立即进行, 或者 dna 可以储存在-20 ° c。不建议在冰箱中储存 DNA 很长时间, 或反复冷冻和解冻。(B) 用这种方法生成合适的图书馆, 关键是要在孵化炉中对 DNA 进行 KOH 处理, 而不是加热块, 保证整个样品的均匀加热和定量水解。(C) 此外, 在汇集和排序之前控制图书馆的质量至关重要。dna 应该被量化和分析使用自动电泳系统, 以确保足够数量的图书馆 dna, 确认适当的碎片大小, 并检查底漆聚。
要进行有意义的数据分析, 还必须注意此方法的信息性值依赖于适当的控件来评估背景计数和序列或子线偏差。我们通常在接近70% 的氯化钾样本中实现一个映射效率, 当只消化序列特定的切 (图 2B, 左面板)。此外, 重要的是要确认, 切治疗不影响全面检测合并核苷酸通过比较 HincII 治疗和未经处理的样本 (图 3B).在这些实验中, 我们使用 HincII 来介绍具体的切口, 虽然也可以使用其他高保真的限制酶。
该协议可用于研究其他类型的 DNA 病变, 可以处理 5´-磷酸盐或 5´-OH 结束。结果的准确性取决于处理的特殊性, 需要适当的控制 (例如,野生类型或未经治疗的) 进行验证。此外, 在将此方法与其他应用或与其他生物体的使用相适应时, 应考虑到该方法在其当前的设置中需要大约1µg 的 DNA 被处理到一个库中。由于两端的数量取决于嵌入的核苷酸的数量, 这取决于有机体或突变体, 包括核苷酸的数量较少的样品将需要更多的输入 DNA 来产生足够数量的末端随后的图书馆建设。同样, 如果 dna 样本的核苷酸数量要高得多, 也需要使用较少的输入 dna 来获得最佳的结扎条件、第二链合成和 PCR 扩增。值得注意的是, 本议定书所述的图书馆建设也产生了涵盖核基因组的数据 (如图 2D所示), 只有数据分析侧重于线粒体 dna。这表明, 更大的基因组具有适度较低的核苷酸频率也捕获了这种方法。
在考虑这种方法时, 应该考虑到一些限制: 虽然这种方法在理论上应该适用于任何生物体, 但适当的参考基因组是必要的对齐读。此外, 从我们的协议得到的结果代表了大量细胞的读取。这种方法不能识别单元子集的特定核苷酸合并模式。如果核苷酸被映射在更大的基因组中, 核苷酸的数量非常少, 那么从随机的缺口中辨别核苷酸可能是有挑战性的, 因此需要适当的控制。
我们在这里描述的方法, 扩展了可用的在体内技术, 如海顿-seq16、核糖-seq17、Pu seq18或 emRiboSeq19。这些方法利用了嵌入式核苷酸对碱性或核糖核酸 H2 处理的敏感性, 采用下一代测序来识别核苷酸全基因组, 这使得它们的映射和比较相对合并。通过对 DNA 序列进行裂解, 如上所述, 除了在嵌入核苷酸的碱性水解之外, 核苷酸的读数可以归入这些劈裂点, 不仅允许识别和映射核苷酸, 而且他们的定量为每个脱氧核糖核酸分子。我们的技术在与 dna 复制、dna 修复和 TLS 相关的疾病中的应用, 可以更深入地理解核苷酸在基本分子机制和基因组完整性方面的作用。
披露声明
作者声明他们没有竞争的金融利益。
致谢
这项研究得到了瑞典研究理事会 (www.vr.se) 对 arc (2014-6466 和瑞典战略研究基金会 (www.stratresearch.se) 对 arc (ICA14-0060) 的资助。在这项工作中, MKME 科技大学为其提供了财政支持。资助者在研究设计、数据收集和分析、决定出版或准备原稿方面没有作用。
材料
| Name | Company | Catalog Number | Comments |
| 10x T4 Polynucleotide Kinase Reaction Buffer | New England Biolabs | B0201S | |
| 10x T4 RNA Ligase Reaction Buffer | New England Biolabs | B0216L | |
| 1x PBS | Medicago | 09-9400-100 | dissolve 1 tablet in H2O to a final volume of 1 L |
| 2-Propanol | Sigma-Aldrich | 33539-1L-GL-R | |
| 2100 Bioanalyzer | Agilent Technologies | G2940CA | |
| 50 mL Centrifuge Tube | VWR | 525-0610 | |
| Adenosine 5'-Triphosphate (ATP, 10 mM) | New England Biolabs | P0756S | dilute with EB to 2 mM |
| Agilent DNA 1000 Kit | Agilent Technologies | 5067-1504 | |
| BSA, Molecular Biology Grade (20 mg/mL) | New England Biolabs | B9000S | diltue with nuclease-free H2O to 1 mg/mL |
| Buffer EB | QIAGEN | 19086 | referred to as EB |
| CleanPCR paramagnetic beads | CleanNA | CPCR-0050 | |
| Deoxynucleotide (dNTP) Solution Mix (10 mM each) | New England Biolabs | N0447L | dilute with EB to 2 mM |
| DMEM, high glucose, GlutaMAX Supplement | Gibco | 61965026 | |
| DynaMag 96 Side | Thermo Fisher | 12331D | |
| Ethanol 99.5% analytical grade | Solveco | 1395 | dilute with milliQ water to 70% |
| Ethylenediaminetetraacetic acid solution (EDTA, 0.5 M) | Sigma-Aldrich | 03690-100ML | |
| Fetal bovine serum | Gibco | 10500056 | |
| HEPES buffer pH 8.0 (1 M) sterile BC | AppliChem | A6906,0125 | |
| Hexammine cobalt(III) chloride (CoCl3(NH3)6) | Sigma-Aldrich | H7891-5G | dissolve in nuclease-free H2O for 10 M solution, sterile filter. CAUTION: carcinogenic, sensitizing and hazardous to aquatic environment. |
| HincII | New England Biolabs | R0103S | supplied with NEBuffer 3.1 |
| Hybridiser HB-1D | Techne | FHB4DD | |
| KAPA HiFi HotStart ReadyMix (2X) | Kapa Biosystems | KK2602 | |
| Lysis buffer | 50 mM EDTA, 20 mM HEPES, NaCl 75 mM, Proteinase K (200 µg/mL), 1% SDS | ||
| Micro tube 1.5 mL | Sarstedt | 72.690.001 | |
| Microcentrifuge 5424R | Eppendorf | 5404000014 | |
| Microcentrifuge MiniStar silverline | VWR | 521-2844 | |
| Multiply µStripPro 0.2 mL tube | Sarstedt | 72.991.992 | |
| Nuclease-free water | Ambion | AM9937 | |
| Phenol – chloroform – isoamyl alcohol (25:24:1) | Sigma-Aldrich | 77617-500ML | |
| Potassium chloride (KCl) | VWR | 26764.232 | dissolve in nuclease-free H2O for 3 M solution, sterile filter |
| Potassium hydroxide (KOH) | VWR | 26668.296 | dissolve in nuclease-free H2O for 3 M solution, sterile filter |
| Proteinase K | Ambion | AM2546 | |
| Qubit 3.0 Fluorometer | Invitrogen | Q33216 | |
| Qubit Assay Tubes | Invitrogen | Q32856 | |
| Qubit dsDNA BR Assay Kit | Invitrogen | Q32850 | CAUTION: Contains flammable and toxic components |
| Qubit dsDNA HS Assay Kit | Invitrogen | Q32851 | CAUTION: Contains flammable and toxic components |
| Refrigerated Centrifuge 4K15 | Sigma Laboratory Centrifuges | No. 10740 | |
| SDS Solution, 10% | Invitrogen | 15553-035 | |
| Sodium acetate buffer solution, pH 5.2, 3 M (NaAc) | Sigma-Aldrich | S7899 | |
| Sodium chloride (NaCl) | VWR | 27810.295 | dissolve in nuclease-free H2O for 5 M solution, sterile filter |
| T100 Thermal Cycler | Bio-Rad | 1861096 | |
| T4 Polynucleotide Kinase (3' phosphatase minus) | New England Biolabs | M0236L | |
| T4 RNA Ligase 1 (ssRNA Ligase) | New England Biolabs | M0204L | supplied with PEG 8000 (50%) |
| T7 DNA Polymerase (unmodified) | New England Biolabs | M0274S | supplied with 10x T7 DNA Polymerase Reaction Buffer |
| TE Buffer | Invitrogen | 12090015 | |
| ThermoMixer F2.0 | Eppendorf | 5387000013 |
参考文献
- Traut, T. W. Physiological Concentrations of Purines and Pyrimidines. Mol. Cell. Biochem. 140, 1-22 (1994).
- McElhinny, S. A. N., et al. Abundant ribonucleotide incorporation into DNA by yeast replicative polymerases. Proc. Natl. Acad. Sci. USA. 107, 4949-4954 (2010).
- Williams, J. S., Lujan, S. A., Kunkel, T. A. Processing ribonucleotides incorporated during eukaryotic DNA replication. Nat. Rev. Mol. Cell Biol. 17, 350-363 (2016).
- Clausen, A. R., Zhang, S., Burgers, P. M., Lee, M. Y., Kunkel, T. A. Ribonucleotide incorporation, proofreading and bypass by human DNA polymerase delta. DNA Repair. 12, 121-127 (2013).
- Potenski, C. J., Klein, H. L. How the misincorporation of ribonucleotides into genomic DNA can be both harmful and helpful to cells. Nucleic Acids Res. 42, 10226 (2014).
- Vengrova, S., Dalgaard, J. Z. RNase-sensitive DNA modification(s) initiates S. pombe mating-type switching. Gene. Dev. 18, 794-804 (2004).
- Lujan, S. A., Williams, J. S., Clausen, A. R., Clark, A. B., Kunkel, T. A. Ribonucleotides Are Signals for Mismatch Repair of Leading-Strand Replication Errors. Mol. Cell. 50, 437-443 (2013).
- Ghodgaonkar, M. M., et al. Ribonucleotides Misincorporated into DNA Act as Strand-Discrimination Signals in Eukaryotic Mismatch Repair. Mol. Cell. 50, 323-332 (2013).
- DeRose, E. F., Perera, L., Murray, M. S., Kunkel, T. A., London, R. E. Solution Structure of the Dickerson DNA Dodecamer Containing a Single Ribonucleotide. Biochemistry. 51, 2407-2416 (2012).
- Li, Y. F., Breaker, R. R. Kinetics of RNA degradation by specific base catalysis of transesterification involving the 2 '-hydroxyl group. J. Am. Chem. Soc. 121, 5364-5372 (1999).
- McElhinny, S. A. N., et al. Genome instability due to ribonucleotide incorporation into DNA. Nat. Chem. Biol. 6, 774-781 (2010).
- Berglund, A. K., et al. Nucleotide pools dictate the identity and frequency of ribonucleotide incorporation in mitochondrial DNA. Plos Genet. 13, (2017).
- Brown, J. A., Suo, Z. C. Unlocking the Sugar "Steric Gate" of DNA Polymerases. Biochemistry. 50, 1135-1142 (2011).
- Sparks, J. L., et al. RNase H2-Initiated Ribonucleotide Excision Repair. Mol. Cell. 47, 980-986 (2012).
- Miyabe, I., Kunkel, T. A., Carr, A. M. The Major Roles of DNA Polymerases Epsilon and Delta at the Eukaryotic Replication Fork Are Evolutionarily Conserved. Plos Genet. 7, (2011).
- Clausen, A. R., et al. Tracking replication enzymology in vivo by genome-wide mapping of ribonucleotide incorporation. Nat. Struct. Mol. Biol. 22, 185-191 (2015).
- Koh, K. D., Balachander, S., Hesselberth, J. R., Storici, F. Ribose-seq: global mapping of ribonucleotides embedded in genomic DNA. Nat. Methods. 12, 251 (2015).
- Keszthelyi, A., Daigaku, Y., Ptasinska, K., Miyabe, I., Carr, A. M. Mapping ribonucleotides in genomic DNA and exploring replication dynamics by polymerase usage sequencing (Pu-seq). Nat. Protoc. 10, 1786-1801 (2015).
- Ding, J., Taylor, M. S., Jackson, A. P., Reijns, M. A. M. Genome-wide mapping of embedded ribonucleotides and other noncanonical nucleotides using emRiboSeq and EndoSeq. Nat. Protoc. 10, 1433-1444 (2015).
- Martin, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet.journal. 17, 10-12 (2011).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biol. 10, (2009).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。