Method Article
Mappatura simultanea e quantificazione di ribonucleotidi del DNA mitocondriale umano
In questo articolo
Riepilogo
Qui descriviamo un metodo suscettibile di quantificare contemporaneamente e ribonucleotidi Mappa del genoma nel DNA altamente intatto alla risoluzione del singolo-nucleotide, che unisce la scissione enzimatica del DNA genomico con la sua idrolisi alcalina e successivi 5 ´-fine sequenziamento.
Abstract
Gli approcci di stimare il numero di ribonucleotidi presenti in un genoma stabiliti sono limitati per la quantificazione di ribonucleotidi incorporati utilizzando brevi frammenti sintetici di DNA o plasmidi come modelli e quindi estrapolare i risultati a tutta la genoma. In alternativa, si può stimare il numero di ribonucleotidi presenti in un genoma usando gel alcalino o Southern blot. Approcci più recenti in vivo impiegano sequenziamento di nuova generazione che consente la mappatura del genoma di ribonucleotidi, fornendo la posizione e l'identità di ribonucleotidi incorporati. Tuttavia, non consentono la quantificazione del numero di ribonucleotidi, che sono incorporati in un genoma. Qui descriviamo come contemporaneamente mappare e quantificare il numero di ribonucleotidi, che sono incorporati in umano mitocondriale del DNA in vivo mediante sequenziamento di nuova generazione. Usiamo il DNA altamente intatto e introdurre sequenza specifico doppio filamento interruzioni di digerirlo con un'endonucleasi, successivamente idrolizzazione ribonucleotidi incorporati con alcali. Le estremità generate sono legate con adattatori e queste estremità sono in sequenza su una macchina di sequenziamento di nuova generazione. Il numero assoluto di ribonucleotidi può essere calcolato come il numero di letture all'esterno del sito di riconoscimento per il numero medio di letture presso il sito di riconoscimento per l'endonucleasi specifiche di sequenza. Questo protocollo può anche essere utilizzato per mappare e quantificare gratis nicks nel DNA e permette l'adattamento mappare altri lesioni del DNA che possono essere elaborati a 5 ´-OH estremità o estremità 5 ´-fosfato. Inoltre, questo metodo può essere applicato a qualsiasi organismo, dato che un genoma di riferimento adeguati è disponibile. Questo protocollo fornisce pertanto un importante strumento per studiare la replicazione del DNA, a 5 minuti-fine elaborazione, danno del DNA e riparazione del DNA.
Introduzione
In una cellula eucariotica, la concentrazione di ribonucleotidi (rNTPs) è molto superiore alla concentrazione di deossinucleotidi (dNTPs)1. Polimerasi del DNA di discriminano ribonucleotidi, ma questa discriminazione non è perfetta e, di conseguenza, ribonucleotidi invece di deossinucleotidi possono essere incorporati nel genoma durante la replicazione del DNA. Ribonucleotidi possono essere i più comuni nucleotidi non canonico incorporati nel genoma2. La maggior parte di questi ribonucleotidi vengono rimosse durante la maturazione del frammento di Okazaki di riparazione per escissione iniziati ribonucleotide RNasi H2 (RER) o da 1 topoisomerasi (rivisto in riferimento3). Ribonucleotidi non possono essere rimosso soggiorno stabilmente incorporati nel DNA2,4 e possono influenzare in modi sia nocivi che benefici (rivisto in ha commentato5). Oltre ad essere in grado di agire come segnali positivi, ad esempio nell'accoppiamento tipo passare in Schizosaccharomyces pombe6 e marcatura filo del DNA nascente durante la mancata corrispondenza di riparazione (MMR)7,8, ribonucleotidi influenzano la struttura9 e stabilità del DNA circostante a causa del gruppo di idrossile 2 di loro ribosio10, con conseguente instabilità di stress e genoma replicativa11. L'abbondanza di ribonucleotidi in DNA genomic (gDNA) e la loro rilevanza nella replica e meccanismi di riparazione, nonché le implicazioni per la stabilità del genoma, dare motivo per indagare il loro avvenimento preciso e la frequenza in maniera genoma.
Attività di RNasi H2 non è stato trovato in mitocondri umani e ribonucleotidi pertanto vengono rimossi in modo non efficiente in DNA mitocondriale (mtDNA). Parecchie vie sono coinvolti nella fornitura di nucleotidi di mitocondri umani e per studiare se le dispersioni nella piscina del nucleotide mitocondriale causano un elevato numero di ribonucleotidi nel mtDNA umano, abbiamo sviluppato un protocollo per mappare e quantificare questi ribonucleotidi nel mtDNA umano isolato da fibroblasti, cellule HeLa e paziente cella linee12.
Maggior parte degli approcci in vitro (rivisto in ha commentato13) per determinare la selettività di polimerasi del DNA contro rNTPs sono basata su esperimenti inserimento o primer ribonucleotide singola estensione dove rNTPs concorrenti sono inclusi nella reazione mescolare, consentendo l'identificazione o la quantificazione relativa di incorporazione ribonucleotide in breve modelli di DNA. Approcci quantitativi su brevi sequenze possono non riflettere piscine dNTP e rNTP presso le concentrazioni cellulari e pertanto fornire approfondimenti selettività della polimerasi, ma sono di limitata importanza per quanto riguarda interi genomi. È stato dimostrato che la quantità relativa di ribonucleotidi incorporato durante la replica di un modello di DNA più lungo, ad esempio un plasmide, possa essere fruita su un gel di sequenziamento utilizzando dNTP radioattivo e d'idrolizzazione il DNA in un ambiente alcalino14. Inoltre, è stato analizzato gDNA il Southern blot dopo idrolisi alcalina, consentendo filo specifico sondaggio e determinazione dei tassi di assoluta del ribonucleotide incorporazione in vivo15. Questi approcci consentono un confronto relativo della frequenza di incorporazione ma non consegnare spaccato la posizione o l'identità dei ribonucleotidi incorporati. Approcci più recenti per analizzare il contenuto del ribonucleotide gDNA in vivo, come HydEn-Seq16, ribosio-Seq17, Pu-Seq18o emRiboSeq19, approfitta dei ribonucleotidi incorporati sensibilità a alcalini o trattamento di RNasi H2, rispettivamente e impiegano il sequenziamento di nuova generazione per identificare ribonucleotidi genoma. Questi metodi non forniscono spaccato la frequenza assoluta incorporazione della ribonucleotidi rilevati. Aggiungendo il passo di scissione enzimatica specifica sequenza del protocollo HydEn-seq, il metodo che qui descritto convenientemente estende le informazioni acquisite da un approccio di sequenziamento, consentendo la mappatura simultanea e quantificazione di embedded ribonucleotidi12. Questo metodo è applicabile a qualsiasi organismo dato che estratti di DNA altamente intatti possono essere generati e un genoma di riferimento adeguati è disponibile. Il metodo potrebbe essere adattato per quantificare e determinare la posizione di qualsiasi lesione che può essere digerita da una nucleasi e lascia un 5 ´-fosfato o un fine 5 ´-OH.
Per mappare e quantificare ribonucleotidi in DNA genomic, il metodo combina la scissione di un'endonucleasi specifiche di sequenza e idrolisi alcalina generando 5 ´-fosfato finisce in siti in cui si trova la sequenza di riconoscimento specifico per l'endonucleasi e a 5 minuti- OH finisce alle posizioni dove si trovavano ribonucleotidi. Poiché le estremità libere generate successivamente sono legate con adattatori e sequenziati mediante sequenziamento di nuova generazione, è di importanza utilizzare DNA altamente intatto ed evitare la frammentazione casuale durante la preparazione del DNA estrazione e biblioteca. Valutare queste letture normalizzate per la legge presso i siti di fenditura dell'endonucleasi permette una quantificazione simultanea e la mappatura dei ribonucleotidi rilevati. A 5 minuti-estremità libere vengono rilevate in esperimenti di controllo dove l'idrolisi alcalina del DNA viene sostituito dal trattamento con KCl. I dati acquisiti forniscono approfondimenti ribonucleotide posizione e quantità e permette analisi rispetto alla frequenza di contenuto e incorporazione del ribonucleotide.
Protocollo
questo protocollo è descritto nella Figura 1 e include l'isolamento di gDNA, digestione con enzimi di restrizione per essere in grado di quantificare il numero di ribonucleotidi, trattamento con alcali per idrolizzare fosfodiesterico di ribonucleotidi inglobato il gDNA, la fosforilazione delle estremità gratuito a 5 minuti-OH, ssDNA legatura delle schede, seconda sintesi di filo e l'amplificazione di PCR prima sequenziamento.
1. adattatori e indice primer
- ARC49 ottenere, oligonucleotidi ARC140, ARC76/77, adattatore e ARC78-ARC107 primer di indice (Vedi tabella 1).
Nota: Oligonucleotidi devono essere purificato di HPLC. ARC76/77 sono ordinati come duplex. - Soluzioni di riserva preparare 100 µM di ogni oligonucleotide in tampone Tris-EDTA (TE) (Vedi la Tabella materiali) e conservare a -20 ° C.
- Preparare 10 µM soluzioni di ARC67/77 e 2 µM di primer ARC49 e indice diluendo in tampone di eluizione (EB; si veda la Tabella materiali). Conservare a-20 ° C.
2. Crescita e raccolta di cellule
- cellule HeLa crescere in 70 mL Dulbecco ' s Modified Eagle Medium (DMEM) supplementato con 10% siero bovino fetale in un matraccio di Spinner 250 mL a 37 ° C.
- Contare il numero di cellule e raccogliere 5 x 10 6 cellule in un tubo da 50 mL, centrifugare per 5 minuti a 200 x g e scartare il surnatante.
- Lavare le cellule con 20 mL 1X PBS, centrifugare per 5 minuti a 200 x g e scartare il surnatante.
- Congelare i pellet a-20 ° C o continuare con la purificazione del DNA.
3. Purificazione del DNA e la quantificazione
- purificare gDNA mediante estrazione del fenolo-cloroformio come descritto di seguito.
- Risospendere le cellule in 2 mL di tampone di lisi (Vedi la Tabella materiali) ed incubare per 30 min a 42 ° C su un blocco di riscaldamento.
Attenzione: Il buffer di lisi contiene componenti pericolosi. Soluzione di SDS è irritante, proteinasi K sensibilizzanti, irritanti e tossici. Indossare indumenti protettivi e guanti. - Dividere il campione in due provette da 2 mL e aggiungere 1 volume (V) di fenolo-cloroformio-alcool isoamilico (25:24:1).
Attenzione: Fenolo-cloroformio-alcool isoamilico è tossico, mutageno, corrosivi e pericolosi per gli ambienti acquatici. Utilizzare in una cappa, indossare indumenti protettivi e guanti e gettare nei rifiuti speciali fenolo-cloroformio. - Mix di inversione per 30-60 s e centrifugare per 5 min 15.000 x g a temperatura ambiente.
Nota: Non non vortice DNA per evitare di introdurre le rotture del filo casuale, che falserebbero i risultati. - Trasferire la fase acquosa superiore ad un nuovo tubo di 2 mL ed aggiungere 1 V di fenolo-cloroformio-alcool isoamilico (25:24:1).
- Mix di inversione e centrifugare per 5 min a 15.000 x g a 4 ° C.
- Trasferire la fase acquosa superiore per tubo un nuovo 2 mL e aggiungere 20 µ l NaCl (5 M) e isopropanolo freddo di 1 V.
Attenzione: Isopropanolo è infiammabile, irritante e tossico. Conservarlo in un ventilato, indossare indumenti protettivi e guanti e tenere lontano da fiamme. - Mix di inversione e incubare per almeno 1 h a -20 ° C.
- Centrifugare per 20 min 15.000 x g, 4 ° C e scartare surnatante.
- Pellet di DNA di lavaggio con etanolo 70% freddo 200 µ l, centrifugare per 20 min a 15.000 x g a 4 ° C ed eliminare il surnatante.
Attenzione: etanolo al 70% è infiammabile e irritante. Tenere la soluzione di lavoro a-20 ° C, altrimenti negozio in ventilato, indossare indumenti protettivi e guanti e tenere lontano da fiamme. - Asciugare il pellet di DNA a temperatura ambiente per 20-25 min.
- Pellet di DNA sciogliere in 100 µ l TE buffer e piscina i campioni in uno tubo.
- Risospendere le cellule in 2 mL di tampone di lisi (Vedi la Tabella materiali) ed incubare per 30 min a 42 ° C su un blocco di riscaldamento.
- Concentrazione di DNA quantitate utilizzando un reagente di quantificazione di dsDNA secondo produttore ' s specifiche (Vedi la Tabella materiali).
Nota: Utilizzare un reagente di quantificazione di dsDNA, perché spettrofotometrica quantificazione di DNA può essere influenzata da fenolo residua. - Store DNA a-20 ° C o continuare con il trattamento di HincII.
4. Trattamento HincII e idrolisi alcalina
mix- Digest 1 µ g di DNA in una reazione contenente 5 µ l di tampone 10x 3.1, 1 µ l (10 U) HincII e privo di nucleasi H 2 O ad un volume finale di 50 µ l.
Nota: Per ottenere le condizioni ottimali per la legatura, seconda sintesi di filo e l'amplificazione di PCR, esso può essere necessario aumentare la quantità di DNA input se si prevede che il DNA contiene un numero molto basso di ribonucleotidi. Allo stesso modo, può essere necessario diminuire l'ingresso del DNA se il numero di ribonucleotidi è molto alto. - Incubare per 30 min a 37 ° C.
- HincII purificare trattati DNA con perline paramagnetici.
Nota: Tenere il tubo coperchi aprire nei passaggi seguenti per non disturbare il pellet aprendo i tubi.- Aggiungere 1,8 V di perline paramagnetici a ogni campione, mescolare accuratamente pipettando e incubare a temperatura ambiente per 10 min.
- Utilizzare un rack magnetico per pellet le perline per 5 min, poi rimuovere ed eliminare il surnatante.
- Lavare la pallina con 150 µ l di etanolo al 70% (temperatura ambiente) per circa 30 s quindi rimuovere e scartare il supernatante.
- Lavare la pallina con 200 µ l di etanolo al 70% (temperatura ambiente) per circa 30 s, quindi rimuovere e scartare il surnatante.
Nota: Etanolo residuo può essere rimosso con una pipetta di 10 µ l. Le goccioline possono essere filate giù brevemente in anticipo. - a secco i campioni a temperatura ambiente per circa 15-20 min.
Nota: L'ora esatta dipende dal volume di perline e la forma del pellet, pertanto i pellet devono essere controllati visivamente. - Rimuovere i tubi dal rack magnetico ed eluire pellet in 45 µ l EB, mix pipettando attentamente.
- Incubare per 5 minuti poi a pellet le perline sul rack magnetico e utilizzare 45 µ l di DNA purificato nel passaggio 4.4.
- Aggiungere 5 µ l di KOH (3 M) o KCl (3 M) per il creazione di un volume totale di 50 µ l di DNA.
Attenzione: 3 soluzione di KOH M è corrosivo. Indossare indumenti protettivi e guanti.
Forno - Incubare per 2 ore a 55 ° C in un'ibridazione seguita da 5 min su Ice.
Nota: Si raccomanda di eseguire il trattamento di KOH in un forno piuttosto che un blocco di riscaldamento per mantenere un riscaldamento uniforme del tubo ed evitare la condensa presso il coperchio. - DNA precipitare aggiungendo 10 µ l di sodio acetato (3 M, pH = 5.2) e 125 µ l etanolo 100% freddo. Incubare in ghiaccio per 5 min.
Attenzione: etanolo al 100% è infiammabile e irritante. Archiviare in un ventilato, indossare indumenti protettivi e guanti e tenere lontano da fiamme. - Pellet gDNA mediante centrifugazione a 21.000 x g, 4 ° C per 5 min e scartare il surnatante. Pellet
- Wash DNA con 250 µ l freddo 70% EtOH, centrifugare a 21.000 x g, 4 ° C per 5 min e scartare il surnatante.
Nota: Per rimuovere le goccioline, il tubo può essere filato giù brevemente nuovamente e surnatante può essere rimosso con una pipetta di 10 µ l. - Asciugare il pellet in un tubo aperto per circa 5-10 minuti fino a quando tutto il liquido visibile evaporato.
- Si dissolve pellet di DNA lasciate in 20 µ l EB per 30 min a temperatura ambiente.
5. fosforilazione fine a 5 minuti
- preparare la miscela di reazione per ogni campione in anticipo controesistente di 2,5 µ l 10 x 2,5 µ l ATP (10 mM), 1 della chinasi di polinucleotide T4 di 3 ´-fosfatasi-minus µ l (10 U) e tampone di reazione della chinasi di polinucleotide T4.
- Trasferire 19 µ l di ogni DNA del campione in una nuova provetta da 200 µ l e denaturare per 3 min a 85 ° C in un termo-cycler.
- Cool DNA campioni su ghiaccio e aggiungere 6 µ l della miscela di reazione per ogni campione.
- Miscele di reazione Incubare a 37 ° C per 30 min e fermata la reazione incubando i campioni a 65 ° C per 20 min.
- Purificare il DNA come descritto al punto 4.3, utilizzando 1.8 V di perline paramagnetici ma eluire in 14 µ l EB.
6. ssDNA legatura
- preparare la miscela di reazione per ogni campione in anticipo composto da 0,5 µ l ATP (2 mM), 5 µ l 10 x tampone di reazione ligasi T4 RNA, 5 µ l CoCl 3 (NH 3) 6 (10 mM), 0,5 µ l ARC140 (100 µM) e 25 µ l 50% PEG 8000. Mix ben pipettando.
Attenzione: CoCl 3 (NH 3) 6 è cancerogeno, sensibilizzanti e pericolosi per l'ambiente acquatico. Indossare indumenti protettivi e guanti. - Trasferire 13 µ l di DNA purificato da 5.5 un passo ad un nuovo tubo di 200 µ l e denaturare per 3 min a 85 ° C in un termo-cycler.
- Raffreddare il DNA sul ghiaccio e 36 µ l di miscela di reazione per ogni campione, mescolare pipettando e spin giù brevemente.
- Aggiungere 1 µ l (10 U) di T4 RNA ligasi ad ogni reazione, Miscelare pipettando e spin giù brevemente.
- Incubare i campioni a temperatura ambiente nella notte scura.
7. Sintesi secondo-filo
- purificare legato del DNA come descritto al punto 4.3, ma utilizzare 0,8 V di perline paramagnetici, pellet le perline per 10 min ed eluire in 20 µ l EB.
Nota: A causa della maggiore viscosità della miscela di reazione di legatura, il primo passo d'appallottolamento è prolungato. - Trasferire 20 µ l di campione di DNA in una nuova provetta PCR 200 µ l. Ripetere il passaggio di purificazione mediante 0,8 V di paramagnetici perline seguendo il produttore ' s specifiche ed eluire in 14 µ l EB.
- Preparare il mix di reazione per ogni campione in anticipo che consiste di 2 µ l di tampone di reazione 10 di polimerasi del DNA del x T7, 2 µ l ARC76/77 (2 µM), 2 µ l dNTP (2 mM) e 0,8 µ l BSA (1 mg/mL).
- µ L trasferimento 12,8 purificato del DNA ad un nuovo tubo di 200 µ l, denaturare per 3 min a 85 ° C in un termo-cycler.
- Raffreddare il DNA sul ghiaccio e 6,8 µ l di miscela di reazione per ogni campione, mescolare pipettando, rallentare brevemente e incubare per 5 min a temperatura ambiente.
- 0,4 µ l (4 U) T7 DNA polimerasi in ogni reazione ed incubare per 5 min a temperatura ambiente.
- Purificare il DNA come descritto al punto 4.3, utilizzando 0,8 V di perline paramagnetiche ed eluire in 11 µ l EB.
8. L'amplificazione di PCR e la quantificazione di biblioteca
tubo di- preparare il mix di reazione per ogni campione in un nuovo 200 µ l in anticipo costituito da 7,5 µ l ARC49 (2 µM), 7,5 µ l indice primer (2 µM, unico per ogni campione) e 25 µ l 2 x avviamento a caldo miscela pronta.
- Aggiungere 10 µ l di campione di DNA per ogni reazione. Amplificare la libreria utilizzando le seguenti condizioni: denaturare a 95 ° C per 45 s, seguiti da 18 cicli di 98 ° C per 15 s, 65 ° C per 30 s, 72 ° C per 30 s, terminando con un allungamento finale a 72 ° C per 2 min. Hold campioni al 4 ° C dopo amplificazione.
- Purificare librerie come descritto al punto 4.3, utilizzando 0,8 V di perline paramagnetici ed eluire in 20 µ l di tampone TE.
- Quantitate librerie utilizzando un reagente di quantificazione di dsDNA, secondo il produttore ' s specifiche (Vedi la Tabella materiali).
- Conservare i campioni a-20 ° C o continuare con analisi biblioteca.
9. Analisi della libreria e Pooling
- determinano la qualità di ogni libreria e stimare la dimensione del frammento media utilizzando un sistema di elettroforesi digitale.
Nota: La dimensione del frammento medio è valutato stimando dove l'area sotto la curva dell'elettroferogramma è dimezzato, trascurando le cime da marcatori. Risultati rappresentativi di profili adatti biblioteca dopo trattamento KOH o KCl sono riportati nella Figura 2A. - Calcolare la concentrazione (nM) delle librerie come:
/(p*650) (c/10 3)] * 10 9
dove c è la concentrazione della libreria in ng / µ l e p è la dimensione del frammento medio in bp, come valutato in 9.1. - Piscina molare quantità uguali di fino a 24 biblioteche amplificati con gli iniettori di indice diverso per il sequenziamento. Aggiungere buffer di TE ad un volume finale di 25 µ l e alla concentrazione di 10 nM.
Nota: A seconda del numero di librerie per eseguire il pooling, la quantità di DNA da ogni libreria è regolato. Se sono stati rilevati dimeri dell'iniettore nel passaggio 9.1 come un picco distinto di circa 130 bp, il volume finale della piscina biblioteca può superare 25 µ l, perché la fase di purificazione viene ripetuta come descritto al punto 4.3, utilizzando 0,8 V di perline paramagnetici, e DNA è eluito in 25 µ l TE buf Fer.- Determinare la concentrazione di piscina biblioteca nuova utilizzando un reagente di quantificazione di dsDNA secondo produttore ' s specifiche e la media dimensione di picco come descritto sopra. Procedere al sequenziamento e analisi dei dati (articoli 10 e 11).
10. Sequenziamento
- eseguire sequenziamento accoppiato-fine 75-base su librerie pool 12.
11. analisi dei dati
- Trim tutte le letture per rimuovere sequenze adattatore, filtrare per qualità e leggere lunghezza.
Nota: Questo può essere fatto utilizzando cutadapt 1.2.1 20 con il comando ' cutadapt -f fastq - incontro-lettura-i caratteri jolly - tranquillo -m 15 - q 10 - un NNNNNNN < FILE >', dove NNNNNNN viene sostituito con la sequenza effettiva adattatore e < FILE > è sostituito con il nome del file fastq. - Rimuovere accoppiamenti di letture che sono stati eliminati nel passaggio precedente utilizzando script personalizzati.
- Allineare Mate 1 dei restanti paia ad un indice contenente la sequenza di tutti gli oligonucleotidi utilizzati per la preparazione di biblioteca (ad es., utilizzando Bowtie 0.12.8 21 e la riga di comando Opzioni - m1-v2). Scarta tutte le coppie con successo allineamenti.
- Allineare coppie rimanenti al genoma di riferimento organismo utilizzando Bowtie con la riga di comando Opzioni - v2 -X 10000..--meglio.
- Mappa legge che spaziano tra la molecola mitocondriale inizio e fine allineando Mate 1 di tutte le coppie non allineate (utilizzando Bowtie con la riga di comando Opzioni - v2).
- Determinare il conteggio delle 5 ´-estremità per tutti gli allineamenti di fine singolo o coppia. Spostare la posizione di questi da una base a Monte sulla posizione dove erano l'idrolizzato ribonucleotidi.
- Esportazione dati dal file di bowtie formato in un formato di file di bedgraph utilizzando script personalizzati per la visualizzazione in comune browser genoma. Normalizzare le letture per ogni ciocca per letture al milione.
- Utilizzando i conteggi e la posizione del file di bedgraph, fare riferimento la sequenza del genoma di organismo per determinare l'identità di incorporated ribonucleotidi.
Nota: Per il genoma mitocondriale umano si legge dalle regioni 16.200-300 e 5.747-5.847 per ogni filo dovrebbe essere esclusi dal momento che queste regioni contengono molti 5 ´-estremità libere non correlate all'incorporazione del ribonucleotide di DNA polimerasi γ. - Dividere il totale letture, non tra cui letture a undici siti HincII, con il numero medio di letture al sito HincII per ottenere il numero di ribonucleotidi per rottura del singolo filo, (cioè il numero di ribonucleotidi per molecola mitocondriale).
Risultati
Illustrando la metodologia descritti sopra, rappresentante i dati sono stati generati l'analisi del DNA mitocondriale umano da HeLa cellule12. Figura 2B Mostra il riepilogate letture affatto HincII siti in pesanti (HS) e filo di luce (LS) del mtDNA umano dopo il trattamento di KCl (pannelli a sinistra). Circa 70% di tutti i rilevati 5 ´-End localizzare i siti di taglio, che dimostrano l'alta efficienza della digestione HincII. Trattamento di librerie con KOH per idrolizzare il DNA ribonucleotidi incorporati riduce il numero di letture HincII siti per circa il 40% (Figura 2B, pannelli di destra). Questo è previsto dal momento che un numero elevato di 5 ´-estremità vengono generato presso i siti di incorporazione ribonucleotide ed è indicativo di una sufficiente qualità di biblioteca. Figura 2 illustra la localizzazione e la frequenza di 5 minuti-estremità (verde) dopo letture e trattamento di KCl generati da HydEn-seq (magenta) dopo il trattamento di KOH, rilevazione sia gratuita a 5 minuti-estremità e generato in ribonucleotidi da idrolisi alcalina. A 5 minuti-estremità libere e ribonucleotidi localizzazione al HS del mtDNA umano vengono mostrati nel pannello di sinistra e quelli eseguendo la localizzazione del LS sono mostrati nel pannello di destra. Ai numeri relativi di crude letture presso ribonucleotidi (Figura 2D, pannello superiore) o siti di HincII (pannello inferiore) in HS e LS di mtDNA mostrano, rispettivamente, una copertura più forte 14-fold o 31-fold del LS rispetto il HS, mentre una simile polarizzazione non è stata osservata per DNA nucleare. Questa polarizzazione di filo può essere spiegata con la netta differenza nella composizione di base dei due fili e illustra l'importanza della normalizzazione per letture a HincII siti.
Normalizzando la lettura conta a HincII dà una misura quantitativa del numero di ribonucleotidi al genoma mitocondriale (Figura 3A). Come illustrato in Figura 3B, le letture dopo trattamento di KOH per ogni ribonucleotide normalizzato per la composizione di sequenza di ogni filo Visualizza un rapporto diverso da 1, che indica una distribuzione non casuale di letture suggerendo un ribonucleotide distinti modello e una libreria alta qualità. Tale rapporto viene influenzato dalla digestione precedente con HincII, verificare la specificità di clivaggio dell'enzima. Normalizzare le letture presso i siti di ribonucleotidi incorporati ai siti di clivaggio HincII, come pure ai contenuti del nucleotide del genoma, genera una misura quantitativa di quanti di ogni ribonucleotide sono incorporati per 1.000 basi complementari ( Figura 3).
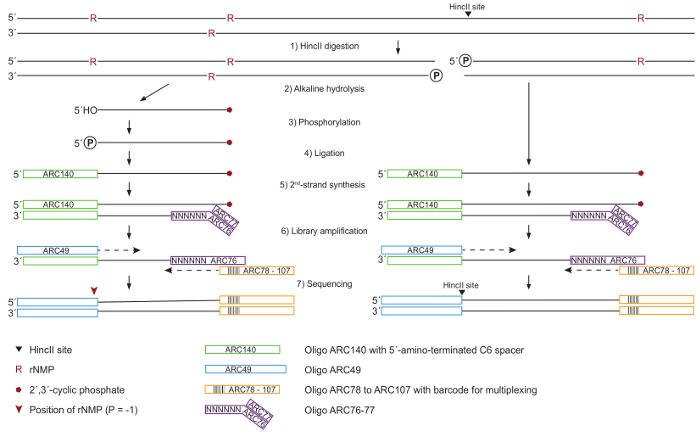
Figura 1: schematico per DNA lavorazione e preparazione di biblioteca. (1) intero genomic DNA viene scisso dalla HincII per la normalizzazione nella successiva quantificazione di ribonucleotidi, generando estremità smussata in siti HincII (freccia nera). (2) il DNA è trattato con KOH per idrolizzare presso siti ribonucleotide, portando a 2, 3 ´-ciclico fosfato (Pentagono rosso) a 3 ´-estremità e gratuito a 5 minuti-OH. (3) a 5 minuti-OH estremità sono fosforilate da T4 polinucleotide chinasi 3 ´-fosfatasi-minus. (4) tutte a 5 minuti-estremità che trasportano un gruppo fosfato sono legate per il oligonucleotide ARC140 di ligasi T4 RNA. (5) il secondo filo è sintetizzato utilizzando T7 polimerasi del DNA e il ARC76-77 oligonucleotidi contenenti sequenze casuali di6 N. (6) la biblioteca è amplificata da una polimerasi di DNA ad alta fedeltà utilizzando ARC49 e uno dei ARC78 di ARC107 primer di indice contenente un codice a barre univoco per multiplexing. (7) a 5 minuti-estremità si trovano tramite sequenziamento accoppiato-fine. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 2: validazione del metodo. (A) rappresentante elettroferogrammi generato utilizzando un sistema di elettroforesi automatizzata per determinare la qualità del generato librerie trattate con segnale KOH o KCl. (B) riassunti in siti HincII pesanti (HS) e filo di luce (LS) mtDNA umano dopo KCl (pannelli a sinistra) o trattamento di KOH (pannelli di destra). Circos (C) figura di 5 ´-estremità libere (verde) e da HydEn-Seq (a 5 minuti-estremità libere e ribonucleotidi, magenta) in HS (pannello di sinistra) e mtDNA umano LS (pannello di destra). Picchi sono normalizzate per milioni letture e il picco massimo è regolato per il numero massimo di letture della biblioteca HydEn-seq. (D) riassunti raw legge alla ribonucleotidi (pannello superiore) e HincII siti (pannello inferiore) in pesante (H) e filo di luce (L) nel mtDNA umano (Mito.) o in senso inverso (RV) o avanti (FW) strand in nucleare (Nuc). DNA. Figure B, C e D sono adattate dal riferimento12. Barre di errore rappresentano l'errore standard della media. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 3: rappresentante risultati. (A), il relativo numero di ribonucleotidi normalizzata a letture HincII siti per le librerie KOH trattati sul pesante (H) o filo di luce (L). (B) rapporto di identità ribonucleotide alla composizione del genoma mtDNA per KOH trattati (KOH) e HincII spaccati con KOH trattati (HincII + KOH) librerie sul pesante (H) o filo di luce (L) del mtDNA. (C) frequenza Ribonucleotide normalizzato a 1.000 basi complementari per HincII e KOH trattati librerie sul pesante (H) o filo di luce (L) del mtDNA. Figure sono adattati dal riferimento12. Barre di errore rappresentano l'errore standard della media. Clicca qui per visualizzare una versione più grande di questa figura.
| Nome | Sequenza | |||
| ARC49 | AATGATACGGCGACCACCGAGATCTACACTCTTTCCCTACACGACGCTCTTCCGATCT | |||
| ARC76 | GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTNNNN * N * N | |||
| ARC77 | AGATCGGAAGAGCACACGTCTGAACTCCAGTC * A * C | |||
| ARC78 | CAAGCAGAAGACGGCATACGAGATCGTGATGTGACTGGAGTTCAGACGTGTGCTCTTCCGATCT | |||
| ARC84 | CAAGCAGAAGACGGCATACGAGATACATCGGTGACTGGAGTTCAGACGTGTGCTCTTCCGATCT | |||
| ARC85 | CAAGCAGAAGACGGCATACGAGATGCCTAAGTGACTGGAGTTCAGACGTGTGCTCTTCCGATCT | |||
| ARC86 | CAAGCAGAAGACGGCATACGAGATTGGTCAGTGACTGGAGTTCAGACGTGTGCTCTTCCGATCT | |||
| ARC87 | CAAGCAGAAGACGGCATACGAGATCACTGTGTGACTGGAGTTCAGACGTGTGCTCTTCCGA | |||
Tabella 1: oligonucleotidi. Sono elencati gli oligonucleotidi utilizzati per HydEn-segg. Grassetto indica l'indicizzazione. * indica un legame fosforotioato. ARC140 contiene un gruppo amminico a 5 minuti invece di un gruppo di 5 ´-OH, in combinazione con un linker di C6. Questa modifica riduce la formazione di ARC140 concatemeri durante la legatura.
Discussione
Qui presentiamo una tecnica per contemporaneamente mappare e quantificare ribonucleotidi in gDNA e mtDNA in particolare, la semplice introduzione di fenditura del DNA in siti specifici di sequenza del genoma come un'aggiunta al protocollo stabilito HydEn-seq. Mentre questo studio si concentra sul mtDNA umano, originariamente il metodo HydEn-seq è stato sviluppato in Saccharomyces cerevisiae, che illustrano la traduzione del metodo a altri organismi12,16.
Per ottenere risultati affidabili ottenuti da questo approccio, si devono sottolineare alcuni passaggi critici: (A) dato che schede di sequenziamento legare a tutti i disponibili a 5 minuti-end, è fondamentale lavorare con DNA altamente intatto. DNA deve essere isolato e biblioteche dovrebbero essere effettuate preferibilmente immediatamente dopo isolamento del DNA o il DNA può essere conservato a-20 ° C. Non si consiglia di conservare il DNA in frigo per un lungo periodo o di congelare e scongelare ripetutamente. (B) per generare librerie adatte con questo metodo, è fondamentale eseguire il trattamento di KOH del DNA in un forno di incubazione, piuttosto che un blocco di riscaldamento, garantendo il riscaldamento omogeneo dell'intero campione e idrolisi quantitativa. (C) Inoltre, è fondamentale per controllare la qualità delle librerie prima di pooling e sequenziamento. Il DNA devono essere quantificati e analizzati utilizzando un sistema di elettroforesi automatizzata per garantire un'adeguata quantità di DNA, delle biblioteche confermare dimensioni frammento appropriato e controllare i dimeri dell'iniettore.
Per un'analisi di dati significativi, è anche importante notare che il valore informativo di questo metodo dipende da controlli appropriati per valutare sfondo conteggi e pregiudizi sequenza o filo. Abbiamo regolarmente raggiungere un'efficienza di mappatura in campioni di KCl di quasi il 70% quando solo digerire con l'endonucleasi specifiche di sequenza (Figura 2B, pannelli a sinistra). Inoltre, è importante confermare che il trattamento di endonucleasi non colpisce il totale rilevamento di ribonucleotidi incorporati confrontando HincII trattati e non trattati di campioni (Figura 3B). In questi esperimenti, abbiamo usato HincII per introdurre tagli specifici del sito, anche se potrebbero essere utilizzati anche altri enzimi di restrizione ad alta fedeltà.
Il protocollo potrebbe essere adattato per studiare altri tipi di lesioni del DNA che possono essere elaborati a 5 ´-fosfato o a 5 minuti-OH finisce. La precisione dei risultati dipende la specificità dell'elaborazione e richiede controlli adeguati (ad es., wild-type o non trattati) per la verifica. Inoltre, quando si adatta questo metodo ad altre applicazioni o per l'utilizzo con altri organismi, si dovrebbe considerare che il metodo nel suo attuale setup richiede circa 1 µ g di DNA che viene elaborato in una raccolta. Poiché il numero delle estremità è dipenda dal numero di ribonucleotidi incorporati, che varia a seconda dell'organismo o mutante, campioni tra cui un numero inferiore di ribonucleotidi richiederebbe più di input del DNA per generare un numero sufficiente di estremità nella costruzione della libreria successivi. Allo stesso modo, se i campioni di DNA hanno un numero molto più elevato di ribonucleotidi, sarebbe anche necessario usando il DNA meno input per ottenere condizioni ottimali per la legatura, seconda sintesi di filo e l'amplificazione di PCR. È interessante nota che la costruzione della libreria come descritto in questo protocollo generato anche dati che riguardano il genoma nucleare (come visualizzato in Figura 2D) e solo l'analisi dei dati si è concentrata sul mtDNA. Ciò illustra che più grandi genomi con frequenze ribonucleotide moderatamente inferiori sono anche catturati da questo metodo.
Quando si considera questo metodo, si devono tener conto di alcune limitazioni: Sebbene questo metodo dovrebbe, in teoria, essere applicabile a qualsiasi organismo, un genoma di riferimento adeguato è necessario per l'allineamento di letture. Inoltre, i risultati ottenuti dal nostro protocollo rappresentano le letture da un gran numero di cellule. Modelli di incorporazione ribonucleotide specifici di un sottoinsieme delle cellule non possono essere identificati da questo approccio. Se ribonucleotidi sono mappati in genomi più grandi con un numero molto basso di ribonucleotidi, e può essere difficile discriminare ribonucleotidi da Nick casuale e pertanto sono necessari controlli appropriati.
Il metodo che descriviamo qui, estende le tecniche disponibili in vivo come HydEn-Seq16, ribosio-Seq17, Pu-Seq18o emRiboSeq19. Questi approcci approfittare della sensibilità dei ribonucleotidi incorporato per alcaline o trattamento di RNasi H2, rispettivamente, che impiegano sequenziamento di nuova generazione per identificare ribonucleotidi genoma, che permette loro mappatura e il confronto delle relativa incorporazione. Fendendo la sequenza di DNA in modo specifico, come descritto in precedenza, oltre a idrolisi alcalina presso ribonucleotidi incorporati, le letture per ribonucleotidi possono essere normalizzate in quei siti di clivaggio, permettendo non solo l'identificazione e la mappatura del ribonucleotidi, ma anche la loro quantificazione per ogni molecola di DNA. L'applicazione della nostra tecnica nel contesto di malattie legate alla replicazione del DNA, riparazione del DNA e TLS potrebbe fornire una più profonda comprensione del ruolo di ribonucleotidi in base dei meccanismi molecolari e l'integrità genomica in generale.
Divulgazioni
Gli autori dichiarano di non avere nessun concorrenti interessi finanziari.
Riconoscimenti
Questo studio è stato sostenuto dalla Swedish Research Council (www.vr.se) concede a ARC (2014-6466 e la Fondazione svedese per la ricerca strategica (www.stratresearch.se) ad arco (ICA14-0060). Chalmers University of Technology ha fornito sostegno finanziario per MKME durante questo lavoro. I finanziatori non avevano alcun ruolo nel disegno dello studio, raccolta dati e analisi, decisione di pubblicare o preparazione del manoscritto.
Materiali
| Name | Company | Catalog Number | Comments |
| 10x T4 Polynucleotide Kinase Reaction Buffer | New England Biolabs | B0201S | |
| 10x T4 RNA Ligase Reaction Buffer | New England Biolabs | B0216L | |
| 1x PBS | Medicago | 09-9400-100 | dissolve 1 tablet in H2O to a final volume of 1 L |
| 2-Propanol | Sigma-Aldrich | 33539-1L-GL-R | |
| 2100 Bioanalyzer | Agilent Technologies | G2940CA | |
| 50 mL Centrifuge Tube | VWR | 525-0610 | |
| Adenosine 5'-Triphosphate (ATP, 10 mM) | New England Biolabs | P0756S | dilute with EB to 2 mM |
| Agilent DNA 1000 Kit | Agilent Technologies | 5067-1504 | |
| BSA, Molecular Biology Grade (20 mg/mL) | New England Biolabs | B9000S | diltue with nuclease-free H2O to 1 mg/mL |
| Buffer EB | QIAGEN | 19086 | referred to as EB |
| CleanPCR paramagnetic beads | CleanNA | CPCR-0050 | |
| Deoxynucleotide (dNTP) Solution Mix (10 mM each) | New England Biolabs | N0447L | dilute with EB to 2 mM |
| DMEM, high glucose, GlutaMAX Supplement | Gibco | 61965026 | |
| DynaMag 96 Side | Thermo Fisher | 12331D | |
| Ethanol 99.5% analytical grade | Solveco | 1395 | dilute with milliQ water to 70% |
| Ethylenediaminetetraacetic acid solution (EDTA, 0.5 M) | Sigma-Aldrich | 03690-100ML | |
| Fetal bovine serum | Gibco | 10500056 | |
| HEPES buffer pH 8.0 (1 M) sterile BC | AppliChem | A6906,0125 | |
| Hexammine cobalt(III) chloride (CoCl3(NH3)6) | Sigma-Aldrich | H7891-5G | dissolve in nuclease-free H2O for 10 M solution, sterile filter. CAUTION: carcinogenic, sensitizing and hazardous to aquatic environment. |
| HincII | New England Biolabs | R0103S | supplied with NEBuffer 3.1 |
| Hybridiser HB-1D | Techne | FHB4DD | |
| KAPA HiFi HotStart ReadyMix (2X) | Kapa Biosystems | KK2602 | |
| Lysis buffer | 50 mM EDTA, 20 mM HEPES, NaCl 75 mM, Proteinase K (200 µg/mL), 1% SDS | ||
| Micro tube 1.5 mL | Sarstedt | 72.690.001 | |
| Microcentrifuge 5424R | Eppendorf | 5404000014 | |
| Microcentrifuge MiniStar silverline | VWR | 521-2844 | |
| Multiply µStripPro 0.2 mL tube | Sarstedt | 72.991.992 | |
| Nuclease-free water | Ambion | AM9937 | |
| Phenol – chloroform – isoamyl alcohol (25:24:1) | Sigma-Aldrich | 77617-500ML | |
| Potassium chloride (KCl) | VWR | 26764.232 | dissolve in nuclease-free H2O for 3 M solution, sterile filter |
| Potassium hydroxide (KOH) | VWR | 26668.296 | dissolve in nuclease-free H2O for 3 M solution, sterile filter |
| Proteinase K | Ambion | AM2546 | |
| Qubit 3.0 Fluorometer | Invitrogen | Q33216 | |
| Qubit Assay Tubes | Invitrogen | Q32856 | |
| Qubit dsDNA BR Assay Kit | Invitrogen | Q32850 | CAUTION: Contains flammable and toxic components |
| Qubit dsDNA HS Assay Kit | Invitrogen | Q32851 | CAUTION: Contains flammable and toxic components |
| Refrigerated Centrifuge 4K15 | Sigma Laboratory Centrifuges | No. 10740 | |
| SDS Solution, 10% | Invitrogen | 15553-035 | |
| Sodium acetate buffer solution, pH 5.2, 3 M (NaAc) | Sigma-Aldrich | S7899 | |
| Sodium chloride (NaCl) | VWR | 27810.295 | dissolve in nuclease-free H2O for 5 M solution, sterile filter |
| T100 Thermal Cycler | Bio-Rad | 1861096 | |
| T4 Polynucleotide Kinase (3' phosphatase minus) | New England Biolabs | M0236L | |
| T4 RNA Ligase 1 (ssRNA Ligase) | New England Biolabs | M0204L | supplied with PEG 8000 (50%) |
| T7 DNA Polymerase (unmodified) | New England Biolabs | M0274S | supplied with 10x T7 DNA Polymerase Reaction Buffer |
| TE Buffer | Invitrogen | 12090015 | |
| ThermoMixer F2.0 | Eppendorf | 5387000013 |
Riferimenti
- Traut, T. W. Physiological Concentrations of Purines and Pyrimidines. Mol. Cell. Biochem. 140, 1-22 (1994).
- McElhinny, S. A. N., et al. Abundant ribonucleotide incorporation into DNA by yeast replicative polymerases. Proc. Natl. Acad. Sci. USA. 107, 4949-4954 (2010).
- Williams, J. S., Lujan, S. A., Kunkel, T. A. Processing ribonucleotides incorporated during eukaryotic DNA replication. Nat. Rev. Mol. Cell Biol. 17, 350-363 (2016).
- Clausen, A. R., Zhang, S., Burgers, P. M., Lee, M. Y., Kunkel, T. A. Ribonucleotide incorporation, proofreading and bypass by human DNA polymerase delta. DNA Repair. 12, 121-127 (2013).
- Potenski, C. J., Klein, H. L. How the misincorporation of ribonucleotides into genomic DNA can be both harmful and helpful to cells. Nucleic Acids Res. 42, 10226 (2014).
- Vengrova, S., Dalgaard, J. Z. RNase-sensitive DNA modification(s) initiates S. pombe mating-type switching. Gene. Dev. 18, 794-804 (2004).
- Lujan, S. A., Williams, J. S., Clausen, A. R., Clark, A. B., Kunkel, T. A. Ribonucleotides Are Signals for Mismatch Repair of Leading-Strand Replication Errors. Mol. Cell. 50, 437-443 (2013).
- Ghodgaonkar, M. M., et al. Ribonucleotides Misincorporated into DNA Act as Strand-Discrimination Signals in Eukaryotic Mismatch Repair. Mol. Cell. 50, 323-332 (2013).
- DeRose, E. F., Perera, L., Murray, M. S., Kunkel, T. A., London, R. E. Solution Structure of the Dickerson DNA Dodecamer Containing a Single Ribonucleotide. Biochemistry. 51, 2407-2416 (2012).
- Li, Y. F., Breaker, R. R. Kinetics of RNA degradation by specific base catalysis of transesterification involving the 2 '-hydroxyl group. J. Am. Chem. Soc. 121, 5364-5372 (1999).
- McElhinny, S. A. N., et al. Genome instability due to ribonucleotide incorporation into DNA. Nat. Chem. Biol. 6, 774-781 (2010).
- Berglund, A. K., et al. Nucleotide pools dictate the identity and frequency of ribonucleotide incorporation in mitochondrial DNA. Plos Genet. 13, (2017).
- Brown, J. A., Suo, Z. C. Unlocking the Sugar "Steric Gate" of DNA Polymerases. Biochemistry. 50, 1135-1142 (2011).
- Sparks, J. L., et al. RNase H2-Initiated Ribonucleotide Excision Repair. Mol. Cell. 47, 980-986 (2012).
- Miyabe, I., Kunkel, T. A., Carr, A. M. The Major Roles of DNA Polymerases Epsilon and Delta at the Eukaryotic Replication Fork Are Evolutionarily Conserved. Plos Genet. 7, (2011).
- Clausen, A. R., et al. Tracking replication enzymology in vivo by genome-wide mapping of ribonucleotide incorporation. Nat. Struct. Mol. Biol. 22, 185-191 (2015).
- Koh, K. D., Balachander, S., Hesselberth, J. R., Storici, F. Ribose-seq: global mapping of ribonucleotides embedded in genomic DNA. Nat. Methods. 12, 251 (2015).
- Keszthelyi, A., Daigaku, Y., Ptasinska, K., Miyabe, I., Carr, A. M. Mapping ribonucleotides in genomic DNA and exploring replication dynamics by polymerase usage sequencing (Pu-seq). Nat. Protoc. 10, 1786-1801 (2015).
- Ding, J., Taylor, M. S., Jackson, A. P., Reijns, M. A. M. Genome-wide mapping of embedded ribonucleotides and other noncanonical nucleotides using emRiboSeq and EndoSeq. Nat. Protoc. 10, 1433-1444 (2015).
- Martin, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet.journal. 17, 10-12 (2011).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biol. 10, (2009).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon