Method Article
crispr 介导的人类真菌病原体白色念珠菌基因组编辑
摘要
白色念珠菌的高效基因组工程是了解治疗方法的发病机制和发展的关键。在这里, 我们描述了一个协议, 以快速和准确地编辑白色念珠菌基因组使用 crispr. 该协议允许调查人员引入各种各样的基因修饰, 包括点突变、插入和删除。
摘要
该方法描述了对二倍体人类真菌病原体白色念珠菌的有效的 crispr 介导的基因组编辑。crispr 介导的白色念珠菌基因组编辑需要 cas9、指导 rna 和修复模板。表达酵母密码子优化 cas9 (casas9) 的质粒已经产生。将直接从 pam 站点 (ngg) 上游的引导序列克隆到 cas9 表达向量中。然后通过引物延伸在体外进行修复模板。修复模板和载体的转化为白色念珠菌导致基因组编辑。根据所使用的修复模板, 调查人员可以引入核苷酸的变化、插入或删除。由于白色念珠菌是二倍体, 突变是在基因的两个等位基因中做出的, 前提是 a 和 b 等位基因不携带干扰指南靶向或修复模板整合的 snps。如果所有家庭成员中都存在适当的保守序列, 则可以并行编辑多基因家族。所描述的白色白色crispr 系统是由 frt 站点和编码翻转。诱导翻转酶后, 抗生素标记 (casas9) 和引导 rna 将从基因组中删除。这使得调查员能够对基因组进行后续编辑。白色念珠菌crispr 是一种强大的真菌基因工程工具, 对所述协议稍作修改, 可以修改其他真菌物种, 包括刺五加、n. castellii 和酿酒酵母。
引言
白色念珠菌是最流行的人类真菌病原体1,2,3。了解白色念珠菌和哺乳动物分子生物学之间的差异对于下一代抗真菌疗法的发展至关重要。这就要求调查人员能够快速、准确地对白色念珠菌进行基因操作。
对白色念珠菌的基因操纵历来具有挑战性。白色念珠菌不维持质粒, 因此所有的结构都必须纳入基因组。此外,白色念珠菌是二倍体;因此, 当淘汰基因或引入突变时, 重要的是要确保这两个副本都已更改 4.此外, 一些白色念珠菌位点是杂合的, 进一步复杂化的基因审讯5。对基因操纵白色念珠菌来说, 通常进行多轮同源重组6。然而, 基因组的二倍体性质和艰苦的结构发展使这一过程成为一个潜在的繁琐过程, 特别是在需要进行多次改变的情况下。这些局限性和白色念珠菌的医学重要性要求开发新技术, 使调查人员能够更容易地操纵白色念珠菌基因组。
聚集定期间隔的短回文重复 (crispr) 介导的基因组编辑是一个强大的工具, 使研究人员能够改变基因组的序列。crispr 需要三个组成部分: 1) 分离目标 dna 的 cas9 核子, 2) 20个以 cas9 为目标的引导 rna, 修复修复裂解部位并纳入预期变化7的模板 dna, 8。一旦指南将 cas9 带到目标基因组序列, cas9 就需要一个基接的相邻主题 (pam) 序列 (ngg) 直接在导引序列的上游来切割 dna 9。对20个基本导轨和 pam 序列的要求提供了高度的目标特异性和限制目标裂解。
crispr 系统的设计是为了编辑各种生物的基因组, 并解决各种各样的问题10。这里描述的是一个灵活, 高效的 crispr 协议编辑一个感兴趣的白色念珠菌基因。该实验将停止密码子引入到一个基因中, 导致翻译终止。根据引入的修复模板, 可以进行其他编辑。一个片段标记为滋养细胞素 (natr) 含有酵母密码优化 cas9 (casas9) 和指南 rna 被纳入白色念珠菌基因组在一个中立的位置。通过同源重组和高效的基因组编辑, 与修复模板编码所需的突变的协变导致裂解修复。下面描述的是 tpk2的编辑, 但所有的白色念珠菌开放阅读框架可以被目标多次由 crispr。crispr 系统是由 frt 位点的侧翼, 可以通过诱导编码在 cas9 表达质粒上的飞酶从白色念珠菌基因组中去除。白色念珠菌crispr 系统使调查人员能够准确、快速地编辑白色念珠菌基因组 11,12。
研究方案
1. 导引 rna 序列的识别和克隆
-
导 rna 序列的识别
- 识别靠近插入停止密码子位置的 5 '-ngg-3 ' pam 序列。(图 1 a)标记的是tpk2的前100个碱基对中的所有 pam 序列 (图 1a)。
请注意:针对每个白色念珠菌开放阅读框架的指南序列可在 http://osf.io/ARDTX 11,12。 - 确定 "转发指南" Primer_3 序列, 它将是 ngg pam 站点的直接上游的20个基础, 并且连续包含的 tt 不会超过5个。左键单击基础直接上游的 ngg, 并拖动光标20个基地, 然后左键单击引物选项卡以添加引物。
- 确定 "反向指南" Primer_3 序列, 它将是 "正向指南" 序列的补充。
请注意:显示的是使用 PAM_3 的指南(图 1b)。 - 右键单击引物, 然后选择 "复制引物数据"。将序列粘贴到文本编辑程序中。
- 识别靠近插入停止密码子位置的 5 '-ngg-3 ' pam 序列。(图 1 a)标记的是tpk2的前100个碱基对中的所有 pam 序列 (图 1a)。
-
将悬垂序列添加到正向和反向指南寡核苷酸中, 以促进克隆 (表 1,图 1 b)。
- 在购买前 Primer_3, 将核苷酸序列 attg 添加到远期指南的 5 ' 结尾。
- 在购买前, 将 g 添加到远期指南的 3 ' 结尾 Primer_3。
- 在购买前, 将核苷酸序列 aaaac 添加到反向指南的 5 ' 结尾 Primer_3。
- 在购买前, 将 c 添加到反向指南的 3 ' 结尾 Primer_3。
-
摘要 cas99 表达向量 pv1524 与 bsmbi。
注: pv1524 含有氨基青霉素 (安培)和滋养氟菊酯 (nat r) 标记.cas9 已经对白色念珠菌进行了编码优化。- 在 1.5 ml 管中加入: 2 微克的 pv1524、5μl 的10倍缓冲器、1μl 的 bsmbi 和 h2o 至 50μl.在55°c 下培养 20分钟 (或者, 在37°c 下, 用 bsmbi 的异构体 esp3i 消化 pv1524 15分钟)。
- 冷却到室温 (rt) 和旋转30秒在 2, 348 x克, 使冷凝管的底部。继续执行步骤1.4 或将消化的质粒存储在-20°c。
-
磷酸盐处理被消化的脊椎。
- 将1μl 的小牛肠磷酸酶 (cip) 加入消化混合物, 在37°c 孵育1小时。
- 使用市售聚合酶链反应 (pcr) 纯化试剂盒 (随试剂盒提供的说明) 纯化消化的质粒, 并将其在30μl 洗脱缓冲液 (eb) 中洗脱。
-
磷化物和退火正向导轨 Primer_3 和反向导轨 Primer_3。
- 在 pcr 管中加入0.5μl 的100μm 正向导轨 Primer_3、0.5μl 的100μm 反向导轨 Primer_3、10x t4 连接酶缓冲液的5μl、t4 多核苷酸激酶的 1μl和43μl 的 h2o。
- 在第二个 pcr 管中加入5μl 的 10倍 t4 连接酶缓冲液、1μl 的 t4 多核苷酸激酶和44μl 的分子生物学级h2o。
请注意:这将作为负控制。 - 在37°c 的温度下将反应混合物培养 30分钟, 然后在95°c 时培养5分钟。
- 将混合物冷却在最慢的坡道速度到 16°c, 退火, 寡核苷酸。然后将退火寡核苷酸混合物置于4°c。
-
将退火寡核苷酸从步骤1.4.3 中加入到消化的 pv1524 中。
- 在 pcr 管中添加以下内容: 10x t4 连接酶缓冲液的1μl、t4 dna 连接酶的0.5μl、退火寡核苷酸混合物的0.5μl、经过 cip 处理的纯化质粒 (20–40 ng) 和 h2o 的10μl 总体积。
- 在 pcr 管中添加以下内容: 10x t4 连接酶缓冲液的1μl、t4 dna 连接酶的0.5μl、经过 cpi 处理的纯化载体 (20–40 ng)、负对照混合物的1μl 和高达10μl 总体积的 h2o.
- 在16°c 的温度下将两个管培养 30分钟, 然后在65°c 下加热 10分钟, 然后冷却至25°c。
- 使用标准的热冲击转化协议, 将结扎混合物5μl 转化为化学上可胜任的大肠杆菌dh5α。在 lb amp/nat 介质上选择 (200μgml 放大器, 50μg/ml nat)。
请注意:如果不能在双选择介质上选择 pv1524 及其衍生物, 就会导致 flp\ frt 在细菌中的引信模块丢失。 - 通过微孔制备四个转化体的质粒, 并使用测序引物对插入序列进行排序 (表 1)。
注意: 在大多数情况下, 测序4转化器足以识别至少1个正确的克隆。 - 保存在-20°c 的 bsmbi 切割位点克隆了一次引导 rna 序列的质粒。
2. 维修模板的设计与生成
- 通过在基因序列中左键单击插入停止密码子, 并添加核苷酸, 对停止密码子和限制消化位点进行编码 (图 1c, 表 1)。
请注意:插入将中断 pam 序列。
请注意:一个限制消化位点将包括在修复模板序列中, 以方便有效筛选克隆 (图 1c)。 - 左键单击10个基地下游的突变将作出突变, 并拖动光标60基地上游。左键单击引物选项卡以添加引物。这将添加 Forward_3 修复模板。左键单击10个基向上的位置, 将突变的位置, 并拖动光标60基地下游。左键单击引物选项卡以添加引物。这将添加修复模板反向3。(图 1c)
-
执行引物扩展以生成修复模板。
- 加入1.2μl 的100μm 修复模板正向底漆, 1.2μl 100 微米修复模板反向底漆, 6μl 脱氧核苷酸三磷酸盐 (dntp) (总浓度为 40 mm), 6 微米缓冲液, 0.6 微米 taq 聚合酶 (3个单位), 每 4个 pcr 中的 45μlh2o 管。
- 通过运行20至30轮 pcr 来执行引物延伸。示例扩展条件: 95°c 时 2分钟, (95°c 时 30秒, 50°c 时 1分钟, 68°c 1分钟) x 34分钟, 68°c 时10分钟。
- 将所有 4个 pcr 管的含量混合在 1.5 ml 管中, 并使用 pcr 纯化试剂盒在 50μl h2o 中纯化产品.
- 通过测定260纳米的吸收率, 对引物延伸产品进行定量, 以确保充足的 dna。
请注意:底漆延伸产品的典型最终浓度为 ~ 200–300 ngμμl。
3.白色念珠菌的修复模板和质粒转化
-
制作醋酸乙酯。
- 混合 10 mm tris-cl、1 mm edta、100 mm 醋酸锂和 h2o (所有库存溶液 ph 7.5), 以实现 50 ml 总体积。
-
制作板材
- 混合 40% peg 3350、100 mm 醋酸锂、10 mm tris-cl、1 mm edta 和h2o(所有库存溶液 ph 值 7.5), 以实现 50 ml 总体积。
-
从步骤1.9 中消化正确克隆的质粒。
- 加入10μg 的质粒, 每10x 缓冲液 4μl, 10mgml 牛血清白蛋白 (bsa) 0.4μl, kpni 0.5 升, saci 0.5 微米, 以及 1.5 ml 管中的h20 至40μl 总体积。在37°c 下隔夜孵化 (图 1d)。
-
在25°c 的酵母肽葡萄糖中培养白色念珠菌sc5314, 野生型原生, 并辅以 0.27 mm 尿素 (ypd + uri), 理想的情况是od 600个以下。
- 颗粒 5 od600单元每个转变通过旋转5分钟在 2 348 x g 和悬挂 5 od 600 颗粒细胞在 100μl ttel 醋酸锂。
- 添加以下 1.5 ml 管的顺序列出: 1) 100μl 细胞从步骤 3.4.1, 2) 40μl 的煮沸和速冷三文鱼精子 dna (10 mg/ml), 3) 10 微克质体消化从步骤 3.3.1, 4) 6 微克纯化修复模板, 和 5) 1 毫升的板。
- 在 1.5 ml 管中按列出的顺序添加以下值: 1) 100μl 细胞, 2) 40μl 的煮三文鱼精子 dna (10mg/ml), 3) h2o 体积等于步骤3.5 中转化 dna 的体积, 4) 1 毫升的 ate。
注意: 这将用作负控件。 - 通过移液轻轻混合转变, 并在25°c 孵育一夜。
- 将细胞放入44°c 的水浴中 25分钟, 热冲击细胞。
- 在台式离心机中旋转 5分钟, 价格为 2, 348 x g, 并取出板份混合物上清液。再次加入1毫升 ypd + uri 和离心机清洗一次, 时间为 5分钟, 2 348 x g。
- 将 ypd + uri 0.1 ml 中的细胞悬浮在滚筒或振动台上, 在25°c 时孵育。
- 在 ypd + uri 上的板, 含有200μml 营养的氟菊酯。殖民地将在2-5天内出现。
4. 单一殖民地的流
-
将100毫米 x 15 毫米 petri 培养皿分成一个季度, 并标记每个象限。
- 用无菌牙签或施药器和条纹穿过象限最长的一侧, 从转化板触摸其中一个菌落。
- 利用无菌技术为单个菌落传播, 使菌落在30°c 下生长2天。
5. 群体 pcr
-
设计了所介绍的约束点上游的正向检查引物 (fcp) ~ 200个基对, 下游设计了反向校核引物 (rcp) ~ 300个基对。
- 加入0.3μl 的 fcp、0.3μl 的 rcp、0.3 微米的热稳定性聚合酶 (extaq 1.5 单位)、3μl 的 dntp (总浓度为 40 mm)、3μl 的 extaq 缓冲区和 23μl的 h2o 到 1.5 ml 管。
请注意:添加 0.5μl/反应二甲基亚硫酸 (dmso) 可提高 pcr 效率。 - 使用 p10 移液头, 注意不要打扰琼脂, 从步骤4.1.2 到从步骤5.1.1 到混合物中, 添加一个单一酵母菌落的0.25μl。
- 通过 pcr 扩增 dna, 并在凝胶上运行5μl 的 pcr, 以确保扩增成功, 注意不要干扰管底部的细胞颗粒。
- 加入0.3μl 的 fcp、0.3μl 的 rcp、0.3 微米的热稳定性聚合酶 (extaq 1.5 单位)、3μl 的 dntp (总浓度为 40 mm)、3μl 的 extaq 缓冲区和 23μl的 h2o 到 1.5 ml 管。
6. 限制消化菌落 pcr
- 添加10μl 的 pcr 产品 (注意不要干扰管底部的细胞颗粒)、3μl 缓冲液、1μl 的限制酶和16μl 的 h2o, 然后按照制造商的指示孵育并在琼脂糖凝胶上进行分解, 以识别皮质基因组编辑。
请注意:这里使用的限制性酶是在 tpk2特定修复模板中编码的站点。
7. 保存应变
- 在30°c 的 ypd + uri 中, 通过限制消化, 从菌落中培养一夜。
- 在适合在-80°c 下储存的试管中加入1毫升的培养物和1毫升的50% 甘油 (使甘油的最终浓度达到 25%)。
- 将正确的克隆存储在-80°c。
请注意:正确的菌株可以在-80°c 下存储多年。
8. 删除 natr 标记
- 流正确的转化物酵母肽麦芽糖 (ypmaltose) (2% 麦芽糖)。
- 从条纹板中选择一个菌落, 在30°c 的液体 ypmaltose 20 g/L 麦芽糖中培养48小时的酵母。
- 在 ypmalose 20 g/L 麦芽糖上贴上200–400个细胞, 在30°c 下孵育24小时。
- 将盘子复制到 ypmaltose 和 ypmalose 200μgml nat 上。
- 在30°c 下孵化24小时。
请注意:不再生长在 ypmalose 200μgml 滋养血菊苷上但在 ypmalose 上生长的菌落已经失去了 natr标记(casas9)和引导 rna。 - 保存丢失 natr 标记 (cascas9)的菌株, 并按照步骤7.1-7.3 中的操作引导 rna。
请注意:类似的质粒, pv1393, 使用sap2 , 而不是m2促进剂。ycb–bsa 将诱导翻转酶和去除 nat r, 如果 pv1393 用于基因编辑。
结果
根据上述建议的指导原则, 设计了针对 c-amp 激酶催化亚基 c. 白色念珠菌tpk2的导 rna 序列和修复模板。序列如下所示 (表 1,图 1)。将导引 rna 克隆到 cacas9 表达向量中, 并与野生白色念珠菌修复模板进行转换。生态ri 限制消化位点和停止修复模板中的密码子会破坏 pam 站点, 并便于筛选正确的突变体 (图 1)。在单个菌落中, 对转化物进行条纹处理, 并通过菌落 pcr 和限制性消化进行筛选, 以纳入修复模板(图 2)。限制消化快速区分野生类型和突变序列。
| 寡核苷酸名称 | 寡核苷酸序列 |
| 前进指南 Primer_3 | atttggggagatatttttccg |
| 反向导轨 Primer_3 | aaaacggaattaagtcacc |
| 修复模板 Forward_3 | tagcaatatcacaatcacaatttacaacgcaccaacttttaagattc ggcga |
| 修复模板 Reverse_3 | attttgccagttttgtggaggggtatttccc gattttta ataaag |
| 正向检查底漆 | 塔亚加阿卡特卡卡卡加格 |
| 反向检查底漆 | 阿克泰塔卡塔塔塔卡特 |
| 测序底漆 | ggacagctgaacttcccc |
表 1: 用于本研究的寡核苷酸清单.为克隆目的添加的网站在指南引物序列中大写和加粗。修复模板中变异基因组 dna 的序列被大写和螺栓化。
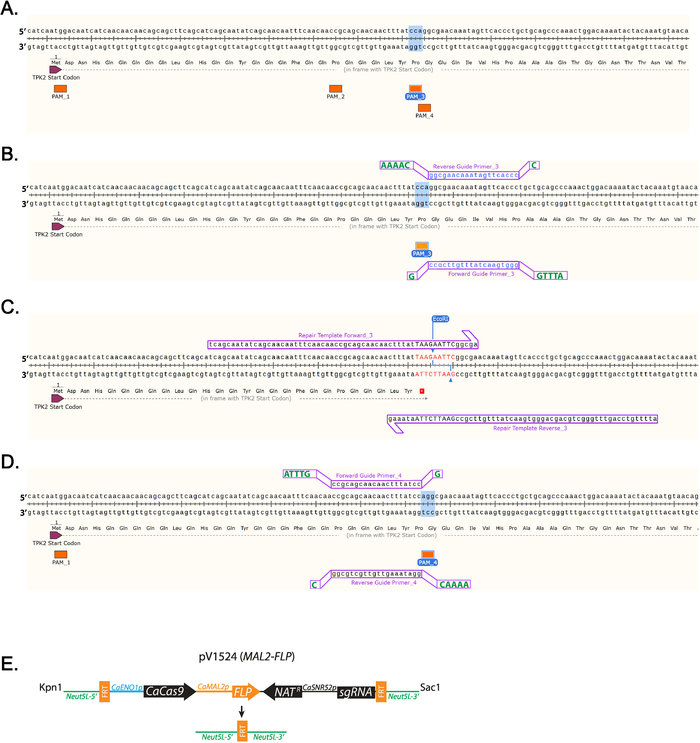
图 1: 导 rna 和修复模板设计示意图.(a)在 tpk2的前100个核苷酸中标记所有 pam 序列。pam 序列 3 (PAM_3) 突出显示, 因为这是本研究中使用的序列。(b) 使用 PAM_3 指导 rna 设计。使用 snapgene 设计的20个基本引物分别是小写和蓝色。克隆所需的其他碱基为大写和绿色显示的偏移量。(c) 将插入 taa 停止密码子和生态ri 站点的修复模板引物插入tpk2阅读框架。与野生类型序列不同的 dna 是红色和大写的。(d) 如何利用 PAM_4 设计 dna 阳性链的指南的例子。(e) pv1524 的原理图后克隆的指南 rna 和消化与 kpni 和 saci。neut5l-5 ' 和 neut5l-3 ' 将载体瞄准白色念珠菌基因组中的 neut5l 位点。caeno1p是驱动酵母优化的cacas9表达式的促进剂。natr是抗营养的抗氟菊酯盒。casnr52p 是促进驱动引导 rna 的表达 (sgrna)。frt 位点通过翻转 (flp) 在翻转表达时去除 crispr 盒式磁带进行切割和重组。vyas等人发表了一个类似于 (e) 的示意图。11.请点击此处查看此图的较大版本.
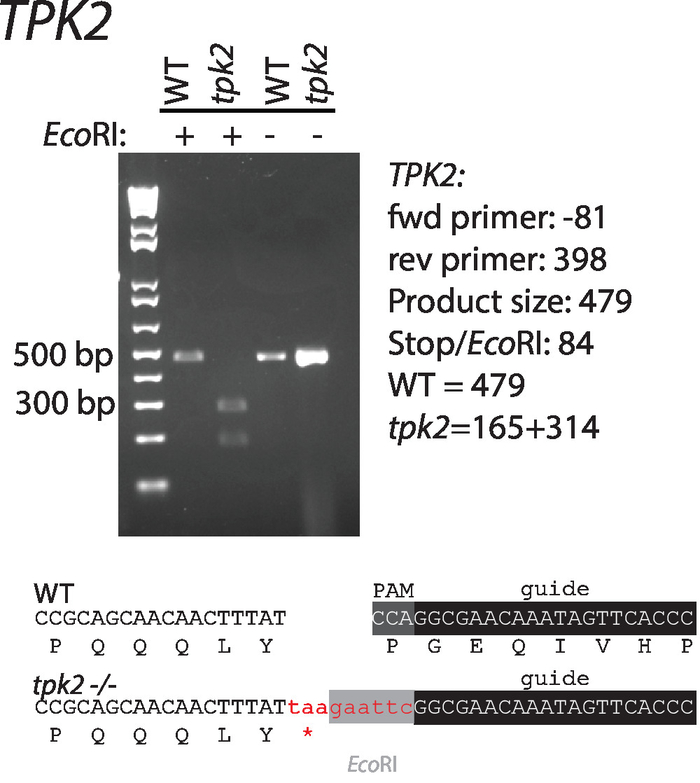
图 2: 介绍和确认一个停止密码子和生态 ri限制网站到 tpk2.表 1列出了用于放大的引物。枯萎病和突变序列显示在凝胶下方。vyas 等人对这一数字作了修改。请点击这里查看此图的较大版本.
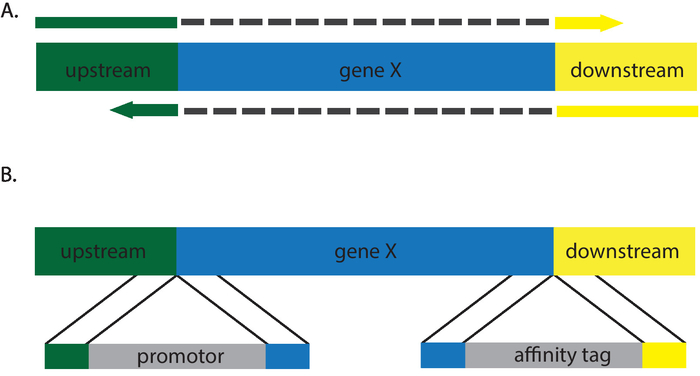
图 3: 将生成 (a) 删除和 (b) 插入的修复模板的卡通说明.(a) 中的灰色破折号表示修复模板引物中不存在的中间序列。请点击这里查看此图的较大版本.
讨论
白色念珠菌crispr 有效地编辑了白色念珠菌基因组。pv1524 对酵母代码优化 cas9 进行编码, 设计使研究人员可以轻松克隆casnr52启动子下游的引导 rna 序列 (图 1)11。必须确保通过测序将指南序列的一个副本克隆到 cass9 表达载体中, 因为额外的副本将阻碍基因组编辑。如果一致地引入了多个导轨副本, 则应降低结扎时使用的退火导轨的浓度。所描述的载体和协议允许针对任何白色念珠菌基因。虽然白色念珠菌是二倍体, 但只需要一个单一的转变就能针对基因的两个等位基因。此外, crispr-cas9 基因组编辑的过程特性使研究人员能够针对基因家族的多个成员。许多基因家族, 如分泌的阿斯巴基蛋白酶 (saps) 和凝集素样序列蛋白 (als) 对白色念珠菌的毒力很重要。crispr 基因组编辑将有助于对这些基因家族的调查。
上面描述的协议将停止密码子引入到开放的读取帧中, 从而导致表型等价于 null (图 2)。通过改变修复模板, 可以进行各种各样的基因交替。通过使用适当的修复模板进行重组, 可以插入无义、误解和无声突变。纳入一个限制站点可以简化变压器筛选, 因为那些没有的变压器必须通过排序12,13进行筛选。此外,白色念珠菌crispr 使研究人员能够生成插入和删除, 使其成为插入关联标记、执行启动式交换和生成敲除的理想系统 (图 3)。筛选这些突变的正确转化器更加费力, 因为有必要对编辑进行排序, 以确认修复模板的正确合并。此外, 南方印迹可能是必要的, 以确保没有在基因组的其他位置插入更多的基因副本。ngg pam 站点的要求对可针对的基因组区域稍有限制。开发使用替代核酸酶 (如 cpf1 或 cas9 系统的变化) 的替代 crispr 系统将缓解其中许多限制14。据调查人员目前所知, 这些系统尚未应用于白色念珠菌。
上述协议中描述的 crispr 系统已经发展到可以应用于各种各样的物种, 包括酿酒酵母、纳穆苏蒂马山和人类病原体念珠菌11.这些酵母的转化和有效编辑需要对描述的协议稍作修改, 但编辑这些交替基因组的框架与白色念珠菌12所述的框架非常相似。此外, 酵母为开发基因组编辑程序提供了一个极好的机制。在酵母中,当 ade2发生突变时, 腺嘌呤生物合成途径的前体积累, 使细胞变红。这种易于观察的表型使研究人员能够识别编辑的细胞, 并快速排除基因组编辑协议的故障。结合广泛的分子生物学工具箱可用于真菌, 编辑许多酵母品种的协议已经开发了15,16。如此广泛地应用基因组编辑技术在真菌中, 有可能对各种科学学科产生重大影响。
crispr 大大提高了白色念珠菌基因组工程的效率, 但迄今为止, crispr 尚未被用于在白色念珠菌中进行基因组宽屏幕。目前的协议需要一个修复模板来引入突变, 因为白色念珠菌的非同源端接途径效率低下 12.为每个基因生成修复模板寡核苷酸是执行全基因组屏幕的一个重要障碍。dna 合成成本的降低与 crispr 技术的进步相结合, 将使删除库的开发更加可行。例如, 来自 casas9 载体的修复模板的表达为针对每个基因11的可持续质粒库的发展铺平了道路。此外, 不需要 casas9 表达载体的瞬态念珠菌 crispr 协议已经开发出 17。此外, 增加的指导表达提高了基因组编辑效率18。这些以及 crispr 技术的其他进步对于在白色念珠菌19、20、21、22 中开发全基因组屏幕至关重要。
白色念珠菌基因组是二倍体, 但 a 和 b 等位基因并不总是相同的5。这种杂合性既带来了挑战, 也带来了机遇。如果我们的目标是同时针对两个等位基因, 则必须使用 pam 站点、引导序列和修复模板, 这些模板将作用于基因的两个副本。然而, 根据基因中存在的单个核苷酸多态性, c.白色 crispr 系统使研究人员能够瞄准单个等位基因。这种精确性有可能使调查人员能够检查等位基因之间的功能差异。由于已观察到等位基因或整个染色体的杂合性 (loh) 丢失, 因此必须仔细确定特定等位基因的目标。在编辑单个白色念珠菌等位基因时, 必须检查相邻的 dna 序列, 以确定克隆是否保持了二倍体 snp 特征。此外,白色念珠菌 crispr 的非目标效应相当低, 但可以考虑关键菌株的全基因组测序。
披露声明
作者没有什么可透露的。
致谢
作者感谢吉尼弗·马格博士对手稿的阅读和有益的评论。这项工作得到了球州立大学实验室启动资金和 nih-1r15a130950-01 至 d. a. b. 的支持。
材料
| Name | Company | Catalog Number | Comments |
| Chemicals | |||
| Agar | BD Bacto | 214010 | |
| agarose | amresco | 0710-500G | |
| Ampicillan | Sigma-Aldrich | A9518 | |
| Bacto Peptone | BD Bacto | 211677 | |
| Bsmb1 | New England Biolabs | R0580L | |
| Calf intestinal phosphatase (CIP) | New England Biolabs | M0290L | |
| Cut Smart Buffer | New England Biolabs | B7204S | |
| Dimethyl Sulfoxide (DMSO) | Sigma-Aldrich | D8418 | |
| dNTPs | New England Biolabs | N0447L | |
| Ethylenediaminetetraacetic acid (EDTA) | Sigma-Aldrich | 3609 | |
| Glacial Acetic Acid | Sigma-Aldrich | 2810000ACS | |
| Glucose | BDH VWR analytical | BDH9230 | |
| Glycerol | Sigma-Aldrich | 49767 | |
| Kpn1 | New England Biolabs | R3142L | |
| LB-Medium | MP | 3002-032 | |
| Lithium Acetate | Sigma-Aldrich | 517992 | |
| L-Tryptophan | Sigma-Aldrich | T0254 | |
| Maltose | Sigma-Aldrich | M5885 | |
| Molecular Biology Water | Sigma-Aldrich | W4502 | |
| NEB3.1 Buffer | New England Biolabs | B7203S | |
| Nourseothricin | Werner Bioagents | 74667 | |
| Poly(ethylene glycol) PEG 3350 | Sigma-Aldrich | P4338 | |
| Sac1 | New England Biolabs | R3156L | |
| Salmon Sperm DNA | Invitrogen | AM9680 | |
| T4 Polynucleotide kinase | New England Biolabs | M0201S | |
| T4 DNA ligase | New England Biolabs | M0202L | |
| Taq polymerase | New England Biolabs | M0267X | |
| Tris HCl | Sigma-Aldrich | T3253 | |
| uridine | Sigma-Aldrich | U3750 | |
| Yeast Extract | BD Bacto | 212750 | |
| Equipment | |||
| Electrophoresis Appartus | |||
| Incubator | |||
| Microcentifuge | |||
| PCR machine | |||
| Replica Plating Apparatus | |||
| Rollerdrum or shaker | |||
| Spectrophotometer | |||
| Waterbath |
参考文献
- Pfaller, M. A., Diekema, D. J. Epidemiology of invasive candidiasis: a persistent public health problem. Clinincal Microbiology Review. 20 (1), 133-163 (2007).
- Magill, S. S., et al. Multistate point-prevalence survey of health care-associated infections. New England Journal of Medicine. 370 (13), 1198-1208 (2014).
- Wisplinghoff, H., et al. Nosocomial bloodstream infections in US hospitals: analysis of 24,179 cases from a prospective nationwide surveillance study. Clinical Infectious Disease. 39 (3), 309-317 (2004).
- Jones, T., et al. The diploid genome sequence of Candida albicans. Proceedings of the National Academy of Science USA. 101 (19), 7329-7334 (2004).
- Muzzey, D., Schwartz, K., Weissman, J. S., Sherlock, G. Assembly of a phased diploid Candida albicans genome facilitates allele-specific measurements and provides a simple model for repeat and indel structure. Genome Biology. 14 (9), (2013).
- Morschhauser, J., Michel, S., Staib, P. Sequential gene disruption in Candida albicans by FLP-mediated site-specific recombination. Molecular Microbiololgy. 32 (3), 547-556 (1999).
- Lau, V., Davie, J. R. The discovery and development of the CRISPR system in applications in genome manipulation. Biochemistry and Cell Biology. 95 (2), 203-210 (2017).
- Doudna, J. A., Charpentier, E. Genome editing. The new frontier of genome engineering with CRISPR-Cas9. Science. 346 (6213), 1258096 (2014).
- Anders, C., Niewoehner, O., Duerst, A., Jinek, M. Structural basis of PAM-dependent target DNA recognition by the Cas9 endonuclease. Nature. 513 (7519), 569-573 (2014).
- Reardon, S. Welcome to the CRISPR zoo. Nature. 531 (7593), 160-163 (2016).
- Vyas, V. K., et al. New CRISPR Mutagenesis Strategies Reveal Variation in Repair Mechanisms among Fungi. mSphere. 3 (2), (2018).
- Vyas, V. K., Barrasa, M. I., Fink, G. R. A Candida albicans CRISPR system permits genetic engineering of essential genes and gene families. Science Advances. 1 (3), e1500248 (2015).
- Evans, B. A., et al. Restriction digest screening facilitates efficient detection of site-directed mutations introduced by CRISPR in C. albicans UME6. PeerJ. 6, e4920 (2018).
- Kim, D., et al. Genome-wide analysis reveals specificities of Cpf1 endonucleases in human cells. Nature Biotechnology. 34 (8), 863-868 (2016).
- Tarasava, K., Oh, E. J., Eckert, C. A., Gill, R. T. CRISPR-enabled tools for engineering microbial genomes and phenotypes. Biotechnology Journal. , e1700586 (2018).
- Raschmanova, H., Weninger, A., Glieder, A., Kovar, K., Vogl, T. Implementing CRISPR-Cas technologies in conventional and non-conventional yeasts: Current state and future prospects. Biotechnology Advances. 36 (3), 641-665 (2018).
- Min, K., Ichikawa, Y., Woolford, C. A., Mitchell, A. P. Candida albicans Gene Deletion with a Transient CRISPR-Cas9 System. mSphere. 1 (3), (2016).
- Ng, H., Dean, N. Dramatic Improvement of CRISPR/Cas9 Editing in Candida albicans by Increased Single Guide RNA Expression. mSphere. 2 (2), (2017).
- Huang, M. Y., Woolford, C. A., Mitchell, A. P. Rapid Gene Concatenation for Genetic Rescue of Multigene Mutants in Candida albicans. mSphere. 3 (2), (2018).
- Shapiro, R. S., et al. A CRISPR-Cas9-based gene drive platform for genetic interaction analysis in Candida albicans. Nature Microbiology. 3 (1), 73-82 (2018).
- Grahl, N., Demers, E. G., Crocker, A. W., Hogan, D. A. Use of RNA-Protein Complexes for Genome Editing in Non-albicans Candida Species. mSphere. 2 (3), (2017).
- Nguyen, N., Quail, M. M. F., Hernday, A. D. An Efficient, Rapid, and Recyclable System for CRISPR-Mediated Genome Editing in Candida albicans. mSphere. 2 (2), (2017).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。