Method Article
CRISPR を介したゲノム人間病原菌カンジダの編集
要約
カンジダ ・ アルビカンスの効率的なゲノム工学は病態解明と治療法の開発を理解する重要です。ここでは、迅速かつ正確に CRISPR を使用してc. アルビカンスゲノムを編集するプロトコルについて述べる。プロトコルでは、研究者の点突然変異、挿入、および削除を含む遺伝の修正のいろいろを紹介することができます。
要約
このメソッドは、二倍体のひと病原菌の効率的な媒介 CRISPR ゲノム編集について説明しますカンジダ ・ アルビカンス。C. アルビカンスに CRISPR を介したゲノム編集 Cas9、必要があります、RNA をガイドし、テンプレートを修復します。酵母のコドンを発現プラスミドに最適化された Cas9 (CaCas9) が生成されています。シーケンスを導く PAM の直接上流サイト (NGG) Cas9 発現ベクターにクローンが作成されます。修理テンプレートがプライマー拡張体外で行われます。C. アルビカンスに修理テンプレートとベクトルの cotransformation はゲノムの編集に します。テンプレートによっては修復を使用、調査官はヌクレオチドの変更、挿入、または削除を導入できます。C. albicansは、二倍体、A と B の対立遺伝子はないテンプレート導入の修理やガイドをターゲットに支障する Snp を抱くことを提供、変異が遺伝子の両方の対立遺伝子で行われます。複数メンバーを持つ遺伝子ファミリーは、家族全員に適した保存されたシーケンスが存在する場合、並列で編集できます。C. アルビカンスCRISPR システム説明は FRT サイトが並ぶ、flippase をエンコードします。Flippase の誘導、耐性マーカー (CaCas9) とガイド RNA がゲノムから削除されます。これをゲノムに以降の編集を実行する使用できます。C. albicans CRISPR は強力な真菌遺伝子工学ツールと記述されていたプロトコルの小変更C. glabrata、N. castellii 、出芽酵母など他の菌種の変更を許可します。
概要
カンジダは、最も一般的な人間の真菌病原体1,2,3です。C. アルビカンスと哺乳類の分子生物学の理解違いは抗真菌治療の次世代の開発に不可欠です。迅速かつ正確に遺伝子操作c.albicansできる捜査官が必要です。
C. albicansの遺伝子操作は歴史的に挑戦されています。C. アルビカンスはプラスミドを保持しない、従ってすべての構成要素は、ゲノムに組み込む必要があります。さらに、 c. アルビカンスは二倍体;したがって、遺伝子をノックアウト、または突然変異を導入すること、いることを確認することが重要だと両方のコピーは変更された4をされています。さらに、いくつかのC. albicansの遺伝子座がヘテロ接合され、さらに複雑な遺伝的尋問5 です。C. アルビカンスを遺伝子操作するには、相同組換え6を複数回実行するが一般的です。ただし、ゲノムと骨の折れる構造開発の二倍体の性質を行った可能性のある退屈なプロセス複数の変更が必要な場合に特に。これらの制限およびC. albicansの医療の重要性は、 C. albicansのゲノムをより簡単に操作する捜査官を可能にする新技術の開発を要求します。
定期的に空間の短い回文繰り返し (CRISPR) をクラスター化-研究者のゲノムの順序を変更することができる強力なツールを介したゲノムの編集です。CRISPR 3 つのコンポーネントが必要です: ターゲット DNA を切断 1)、Cas9 ・ ヌクレアーゼ 2) 20 Cas9 を興味のシーケンスにターゲット ガイド RNA をベースし、3) 胸の谷間サイトを修復し、変更しよう7が組み込まれていますテンプレート DNA を修復 8。Cas9 が protospacer 隣接するモチーフ (PAM) シーケンス (NGG) を必要とするガイドは、ターゲットのゲノム シーケンスに Cas9 をもたらす一度直接9DNA を切断するガイド シーケンスの上流。20 基本ガイドと PAM シーケンスの両方のための要件は、特異性をターゲットの高度を提供する、ターゲットを胸の谷間を制限します。
CRISPR システムは、生物の多様なセットのゲノムを編集し、さまざまな問題10に取り組むために設計されています。ここで説明は、興味のc.albicans遺伝子を編集するための柔軟で効率的な CRISPR プロトコルです。実験は、遺伝子、翻訳が終了されることに終止コドンを紹介します。導入の修理テンプレートによって他の編集が可能です。Nourseothricin (Natr) でマークされたフラグメント酵母を含むコドン最適化 Cas9 (CaCas9) ガイド RNA が中立的なサイトでc. アルビカンスゲノムに組み込まれています。エンコード望ましい突然変異修復テンプレートと cotransformation は、相同組換えと効率的なゲノム編集による開裂の修復に します。下記はTPK2、しかし、すべてC. albicansの開いたリーディング ・ フレームの対象にできる複数回 CRISPR の編集です。CRISPR システムは FRT サイトが並ぶし、CaCas9 発現プラスミドにコードされた flippase の誘導によりc.albicansゲノムから削除することができます。C. albicans CRISPR システムが正確かつ迅速に編集c.albicansゲノム11,12に捜査できます。
プロトコル
1. 身分証明書とガイド RNA シーケンスのクローニング
-
ガイド RNA 配列の同定
- 識別、5'-NGG-3' PAM シーケンス停止コドンが挿入される場所の近くにあります。(図 1 a)というラベルの付いたすべての PAM のシーケンスはTPK2 (図 1 a) の最初 100 塩基対で発見ことがされます。
注:Http://osf.io/ARDTX 11,12ガイド系列各c.albicans開いたリーディング ・ フレームをターゲットを見つけることが。 - 20 となる前方ガイド Primer_3 シーケンスを特定拠点直接上流 NGG PAM のサイト、5 以上を含まない行で Ts。左クリックで直接ベースの上流 NGG のドラッグ カーソル 20 拠点、プライマーを追加するプライマー] タブでをクリックします。
- 前方ガイド シーケンスを補完するものとなるガイド Primer_3 の逆シーケンスを識別します。
注:PAM_3 を使用してガイドを示します (図 1 b)。 - プライマーを右クリックし、「プライマー データをコピー」を選択します。シーケンスをテキスト編集プログラムに貼り付けます。
- 識別、5'-NGG-3' PAM シーケンス停止コドンが挿入される場所の近くにあります。(図 1 a)というラベルの付いたすべての PAM のシーケンスはTPK2 (図 1 a) の最初 100 塩基対で発見ことがされます。
-
(表 1、図 1 b) をクローン作成を容易にする前方および逆引きガイド oligos にオーバー ハングのシーケンスを追加します。
- 購入する前に前方ガイド Primer_3 の 5' 末端に塩基配列 ATTTG を追加します。
- 購入する前に前方ガイド Primer_3 の 3' 末端に G を追加します。
- 購入する前にガイドの逆 Primer_3 の 5' 末端に塩基 AAAAC を追加します。
- 購入する前に、ガイドの逆 Primer_3 の 3' 末端に C を追加します。
-
ダイジェスト CaCas9 式ベクトル pV1524 BsmBI と。
注: pV1524 には、アンピシリン (アンプr) と nourseothricin (Natr) マーカーが含まれています。Cas9 は、 C. albicansのコドン最適化されています。- 追加することによってプラスミッドをダイジェスト: 2 μ g pv1524、バッファー x 10 の 5 μ L、BsmBI、および H の 1 μ L の2O 1.5 mL チューブに 50 μ L に。55 ° C、20 分で孵化させなさい (または、消化 Esp3I、37 ° C での BsmBI、isoschizomer と 15 分のための pv1524)
- 部屋の温度 (RT) と 30 のスピンにクール チューブの底に凝縮させる 2,348 x gで s。1.4 をステップに進むまたは-20 ° C で消化されたプラスミドを格納
-
消化のバックボーンのアルカリホスファターゼ-御馳走。
- 消化の混合物にふくらはぎ腸のアルカリホスファターゼ (CIP) の 1 μ L を追加し、37 ° C 1 時間インキュベートします。
- 市販のポリメラーゼの連鎖反応 (PCR) 精製キット (キットの指示) を使用して消化されたプラスミドを浄化し、30 μ L の溶出バッファー (EB) で溶出します。
-
リン酸化し、アニール ガイド Primer_3 フォワードとリバース ガイド Primer_3。
- 100 μ M 前方の 0.5 μ L ガイド Primer_3、100 μ M の 0.5 μ L を追加 43 μ H2O PCR チューブに T4 ポリヌクレオチド キナーゼの 1 μ L、5 μ L の 10 x T4 リガーゼ バッファー ガイド Primer_3 の逆。
- 第 2 PCR チューブに 10 x T4 リガーゼ バッファーの 5 μ L、T4 ポリヌクレオチド キナーゼの 1 μ L と分子生物学グレード H2O の 44 μ L を追加します。
注:これは負の制御として役立ちます。 - 当時 95 ° C、5 分、30 分の 37 ° C でたちの反応混合物を孵化させなさい。
- アニール 16 ° c 低速ランプに入る率で混合物を冷却、oligos。4 ° C で熱処理したオリゴ混合物を配置します。
-
1.4.3 のステップから消化 pv1524 に焼なまし oligos を縛る。
- PCR チューブに以下を追加します: 10 x T4 リガーゼ バッファー、T4 DNA リガーゼの 0.5 μ L、焼なましオリゴ ミックス 0.5 μ の 1 μ L 消化 CIP 処理精製プラスミド (20-40 ng) と H2O 10 μ L の総ボリュームに。
- PCR チューブに以下を追加します: 10 x T4 リガーゼ バッファー、T4 DNA リガーゼの 0.5 μ L の 1 μ L 消化 CIP 処理精製ベクトル (20-40 ng)、陰性対照の混合物、および H2O 10 μ L の総ボリュームまでの 1 μ L。
- 16 ° C、30 分で、10 分の 65 ° C でたちで両方のチューブをインキュベートし、25 ° C に冷却
- 結紮混合物の 5 μ L を化学的に有能な腸管出血性大腸菌DH5α 標準的な熱衝撃変形のプロトコルを使用してに変換します。LB アンプ/Nat メディア (200 μ g/mL アンプ、50 μ g/mL Nat) を選択します。
注:PV1524 とその誘導体二重選択メディアを選ぶ失敗は細菌で FLP/FRT 切除によって Nat/CaCas9/ガイド モジュールの損失になります。 - Miniprep、によって 4 つの形質転換体からプラスミドを浄化、配列のプライマー (表 1) で挿入の一連のシーケンスです。
注: 4 形質転換体をシーケンス処理時間のほとんどは少なくとも 1 正しいクローンを識別するために十分です。 - -20 ° C にてサイトをカット BsmBI に単一の時間をクローン作成ガイド RNA シーケンスを持つプラスミドを保存します。
2. 設計と修理テンプレートの生成
- 遺伝子の塩基配列で左クリックしてエンコード停止コドンと制限消化サイト (図 1、表 1) ヌクレオチドを追加停止コドンを挿入します。
注:挿入は、PAM のシーケンスが中断されます。
注:制限消化サイトはクローン (図 1) の効率的なスクリーニングを容易にする修理テンプレート順番に表示されます。 - 10 の突然変異の箇所の拠点を下流とドラッグ カーソル 60 塩基上流を左クリックします。プライマーを追加するプライマー] タブをクリックします。これは修理テンプレート Forward_3 を追加します。10 塩基上流の突然変異が行われるとドラッグ カーソル 60 拠点下流を左クリックします。プライマーを追加するプライマー] タブをクリックします。これは修理テンプレートを逆に 3 を追加します。(図 1)
-
修理のテンプレートを生成するプライマー拡張を実行します。
- Deoxynucleotide 三リン酸塩 (dNTPs) の 6 μ L、100 μ m 修理テンプレート逆プライマーの 1.2 μ L 100 μ m 修理テンプレートの前方プライマー 1.2 μ L を追加 (濃度を合計 40 mM)、6 のバッファー、Taq ポリメラーゼ (3 台)、0.6 μ L および H2O 4 PCR のそれぞれに 45 μ μ L チューブ。
- PCR の 20 と 30 でラウンド間実行してプライマー拡張を実行します。延長条件の例: 95 ° C で 2 分 (30 95 ° C、50 ° c、68 ° C で 1 分 1 分で s) x 34、68 ° C で 10 分
- すべて 4 PCR チューブ 1.5 mL チューブの内容を組み合わせて、50 μ L H2o. の製品を浄化するために PCR 精製キットを使用して
- 260 で吸光度を決定することにより十分な DNA を確保するためプライマー拡張製品を量的に表わす nm。
注:プライマー拡張製品の典型的な最終濃度は ~ 200-300 ng/μ L。
3. C. albicans修理テンプレートとプラスミドの変換
-
テ/リチウム アセテートを作る。
- ミックス 10 mM Tris Cl 1 mM EDTA 100 mM リチウム アセテート、H2O (すべて原液 pH 7.5) 50 mL 容量を達成するために。
-
プレートを作る
- ミックス 40% PEG 3350、酢酸リチウム 100 mM、10 mM Tris Cl、1 mM EDTA、H2O (すべて原液 pH 7.5) 50 mL 容量を達成するために。
-
ステップ 1.9 から正しく複製プラスミドを消化します。
- 0.5 μ KpnI、SacI、0.5 μ H20 に 40 μ L の容量 1.5 mL チューブ、10 μ g のプラスミド、10 x バッファーの 4 μ L、10 mg/mL ウシ血清アルブミン (BSA) 0.4 μ L を追加します。37 ° C 一晩 (図 1) で孵化させなさい。
-
C. albicans SC5314、野生型 prototroph、25 ° c 外径600 6 未満に理想的に 0.27 mM ウリジン (YPD + Uri) を添加した酵母ペプトン ブドウ糖での夜行文化を育てます。
- 2,348 x gで 5 分間回して 5 外径600単位変換ごとにセルをペレットし、100 μ L TE/リチウム アセテートの小球形にされたセルの 5 外径600を中断します。
- 次の順序で 1.5 mL チューブに追加: ステップ 3.4.1、ゆで卵、迅速な冷却サケ精子 DNA (10 mg/mL) 3) 10 μ g のプラスミド消化手順 3.3.1, 精製修理テンプレートの 4) 6 μ g からの 2) 40 μ L からの細胞の 1) 100 μ L、5) プレートの 1 mL。
- 次の順序で 1.5 mL チューブに追加: セルの 1) 100 μ L、ゆで卵、迅速な冷却サケ精子 DNA (10 mg/mL)、3) H2O ボリューム ステップ 3.5、およびプレートの 4) 1 mL の DNA の変換と同等の 2) 40 μ L。
注: これはネガティブ コントロールとなります。 - 軽くピペッティングによる変換をミックス、25 ° C で一晩インキュベートします。
- 熱 25 分 44 ° C の水浴でそれらを配置することによって、細胞に衝撃を与えます。
- ベンチトップ遠心分離機で 2,348 x gで 5 分間スピンし、プレートの混合物の上澄みを削除します。2,348 x gで YPD + Uri と再び 5 分間遠心の 1 mL を追加することで一度洗います。
- YPD + Uri の 0.1 mL のセルを中断し、ローラー ドラムまたは 25 ° C でシェーカーで一晩インキュベートします。
- 200 μ g/mL nourseothricin YPD + Uri のプレート。植民地は、2-5 日に表示されます。
4. 単一コロニーのための縞
-
四分の一に、100 mm × 15 mm のペトリ皿を分割し、各作業領域にラベルを付けます。
- 四半円点の最長の辺の間で生殖不能のつまようじまたはアプリケータ連勝と変換プレートからコロニーの 1 つをタップします。
- 無菌技術を使用して単一コロニーの連勝し、2 日間の 30 ° C で成長するコロニーを許可します。
5. コロニー PCR
-
前方チェック プライマー (FCP) ~ 200 塩基対上流導入された制限サイトのと逆チェック プライマー (RCP) ~ 300 塩基対下流設計します。
- FCP、RCP、耐熱性ポリメラーゼ (ExTaq 1.5 単位) の 0.3 μ L、dNTPs の 3 μ L の 0.3 μ L の 0.3 μ L を追加 (濃度を合計 40 mM)、3 μ ExTaq バッファー、および 23 μ H2O 1.5 mL チューブに。
注:0.5 μ L/反応ジメチルスルホキシド (DMSO) の付加は PCR の効率を向上できます。 - 4.1.2 の手順から単一酵母コロニーの 0.25 μ L を 5.1.1、P10 のピペット チップを使用し、寒天を邪魔しないように世話のステップからの混合物に追加します。
- PCR によって DNA を増幅して増幅が成功すると、管の底で細胞ペレットを邪魔しないように世話を確認するゲルの PCR の 5 μ L を実行します。
- FCP、RCP、耐熱性ポリメラーゼ (ExTaq 1.5 単位) の 0.3 μ L、dNTPs の 3 μ L の 0.3 μ L の 0.3 μ L を追加 (濃度を合計 40 mM)、3 μ ExTaq バッファー、および 23 μ H2O 1.5 mL チューブに。
6. コロニー PCR の制限の消化力
- PCR 製品 (管の底で細胞ペレットを邪魔しないように注意)、バッファーの 3 μ L、制限酵素の 1 μ L、H2O の 16 μ L の 10 μ L を追加し、製造元の指示に従ってを孵化し、対応を識別するために agarose のゲルの解決ct ゲノム編集。
注:ここで使用される制限の酵素、 TPK2特定の修復テンプレートでエンコードされたサイトです。
7. 菌株の保存
- 30 ° C で YPD + Uri の制限の消化力によって確認されている植民地から一晩文化を育てる
- -80 ° C で保存に適した管に 1 mL (25% グリセロールの最終的な集中をもたらす) 50% のグリセロールの文化の 1 mL を追加します。
- -80 ° C で正しいクローンを格納します。
注:正しい系統は、長年にわたって-80 ° C で保存できます。
8. Natrマーカーの削除
- 酵母ペプトン マルトース (YPMaltose) に連勝正しい形質 (2% マルトース)。
- すじ状のプレートからコロニーをピックアップし、液体 YPMaltose 20 g/L マルトースで 30 ° C で 48 h 用酵母を培養します。
- YPMaltose 20 g/L マルトースに 200-400 細胞をプレートし、30 ° C 24 時間で孵化させなさい。
- YPMaltose と YPMaltose 200 μ g/mL 国立にプレートを複製します。
- 30 ° C 24 時間で孵化させなさい。
注:もはや YPMaltose 200 μ g/mL nourseothricin に成長するが、YPMaltose に成長した植民地は、Natrマーカー (CaCAS9)を失っているし、RNA をガイドします。 - Natrマーカー (CaCAS9)とガイド RNA 手順 7.1-7.3 のように失っている系統を保存します。
注:似たようなプラスミッド、pV1393、 MAL2のプロモーターではなくSAP2を使用します。YCB-BSA の成長は、pV1393 遺伝子の編集に使用する場合 flippase と Natrの除去を誘発します。
結果
シーケンスは、ガイド Rna およびC. albicans TPK2、修理テンプレート c AMP キナーゼ触媒サブユニット、上記のガイドラインに従って設計されていた。シーケンスは、(表 1、図 1) のとおりです。ガイド Rna CaCas9 発現ベクターにクローン化し、野生型C. albicansの修理テンプレートと cotransformed。エコRI 制限消化サイト修理テンプレートで終止コドン PAM サイトを混乱させるし、正しい変異体 (図 1) のスクリーニングを容易にします。シングル コロニーの縞し、修理のテンプレート(図 2)の定款のコロニー PCR および制限の消化によって選別されました。制限の消化力は突然変異体シーケンスからすぐに野生型を区別します。
| オリゴヌクレオチド名 | オリゴヌクレオチドのシーケンス |
| 前方ガイド Primer_3 | ATTTGgggtgaactatttgttcgccG |
| 逆引きガイド Primer_3 | AAAACggcgaacaaatagttcacccC |
| テンプレート Forward_3 を修復します。 | tcagcaatatcagcaacaatttcaacaaccgcagcaacaactttatTAAGAATTCggcga |
| テンプレート Reverse_3 を修復します。 | attttgtccagtttgggctgcagcagggtgaactatttgttcgccGAATTCTTAataaag |
| 前方チェック プライマー | ttaaagaaacttcacatcaccaag |
| 逆チェック プライマー | actttgatagcataatatctaccat |
| シーケンス入門 | ggcatagctgaaacttcggccc |
表 1: 本研究の使用の oligo ヌクレオチドの一覧。クローン作成の目的、資本金の追加サイトやガイドのプライマー シーケンスで太字で表示されます。ゲノム DNA を変異修復テンプレートのシーケンスは大文字と太字で表示されます。
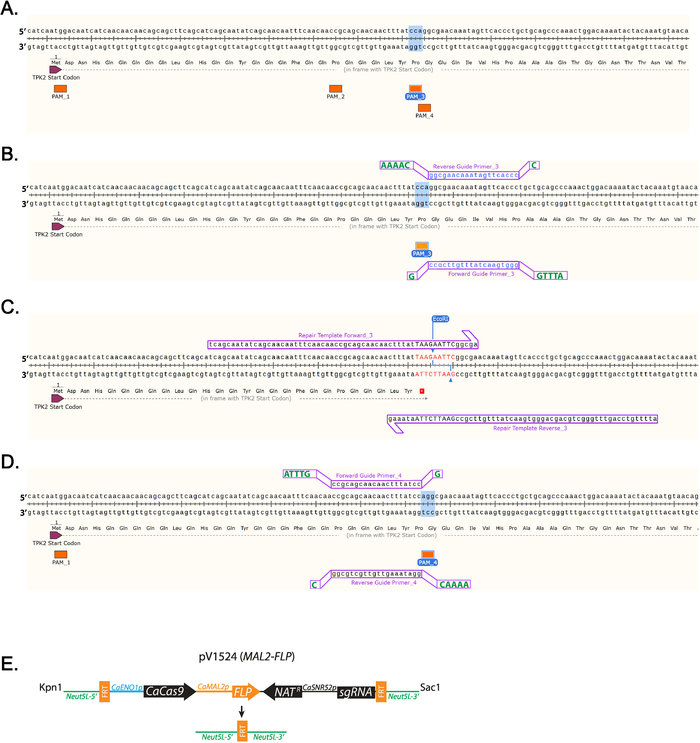
図 1: ガイド RNA および修理のテンプレート デザインの図。(A) TPK2の最初 100 ヌクレオチド内すべての PAM シーケンスのラベリング。本研究で使用されるシーケンスは、PAM シーケンス 3 (PAM_3) がハイライトされます。(B) ガイド RNA は、PAM_3 を使用して設計します。SnapGene を使用して設計した 20 の基本プライマーが小文字と青です。クローン作成に必要な追加の拠点、大文字と緑表示オフセット。(C) 修理 TAA 停止コドンとEcoRI サイト挿入テンプレート プライマーがフレームを読んでTPK2に挿入されます。野生型のシーケンスとは異なる DNA が赤と大文字です。(D) PAM_4 を用いた DNA の肯定的な鎖にガイドを設計する方法の例です。(E) のクローニング ガイド RNA および KpnI と SacI 消化後 pV1524 の模式図。Neut5L 5' と Neut5L 3' C. albicansのゲノムの Neut5L サイトにベクトルを対象します。CaENO1pは、ドライブを酵母に最適化されたCaCas9の発現プロモーターです。Natrは nourseothricin 抵抗カセットです。CaSNR52p は運転ガイド RNA 発現 (sgRNA) プロモーターです。FRT サイトは切断、再結合 flippase (FLP) flippase 式に CRISPR カセットの取り外し。回路図 (E) のようは vyas さんらによって出版されました。11.この図の拡大版を表示するのにはここをクリックしてください。
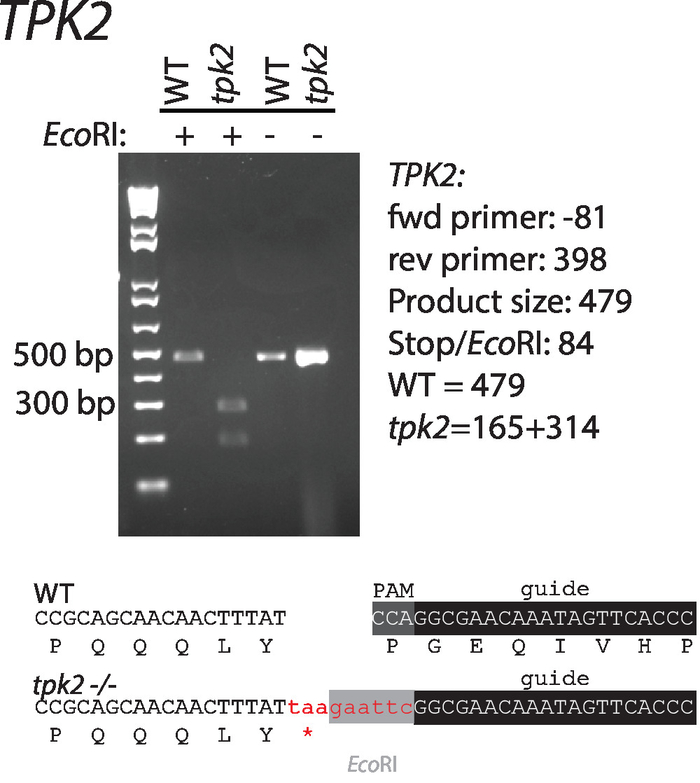
図 2: 導入および停止コドンとTPK2にEcoRI 制限サイトの確認。増幅用プライマーは、表 1のとおりです。ゲルの下野生型と変異型のシーケンスのとおりです。この図は、vyas さんら11から変更されています。この図の拡大版を表示するのにはここをクリックしてください。
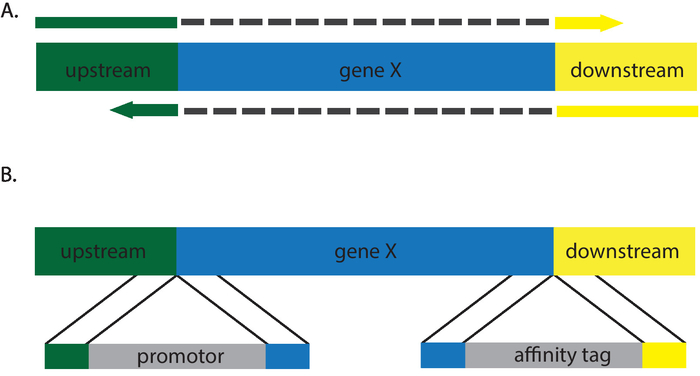
図 3: 削除 (A) と (B) の挿入が生成されます修復テンプレートの説明を漫画します。グレー (a) ダッシュ描写介在シーケンス修理テンプレート プライマーには存在しません。この図の拡大版を表示するのにはここをクリックしてください。
ディスカッション
C. アルビカンスCRISPR は効率的にC. albicansのゲノムを編集します。pV1524 エンコード酵母 Cas9 のコドン最適化し、捜査簡単にCaSNR52プロモーター (図 1)11のガイド RNA シーケンスを下流にクローンを作成するように設計されています。ガイド シーケンスの単一コピーだけがされて複製 CaCas9 発現ベクターに配列、ゲノム編集妨げになる余分なコピーとしてそれが保障されなければなりません。ガイドの複数のコピーが一貫して導入されている場合、は、結紮、焼なましガイドの濃度を下げる必要があります 1 つ。ベクトルと記述されているプロトコルは、あらゆるc.albicans遺伝子のターゲットを許可します。C. アルビカンスは二倍体が、単一の変換のみが遺伝子の両方の対立遺伝子をターゲットに必要です。また、CRISPR CaCas9 ゲノム編集の連続性質は遺伝子家族の複数のメンバーを対象とする研究者をできます。分泌アスパルチル プロテアーゼ (SAP) とタンパク質凝集素のようなシーケンス (ALS) など多くの遺伝子家族はc. アルビカンスの病原性にとって重要です。CRISPR ゲノム編集これらの遺伝子家族の調査を促進します。
上記で説明したプロトコルは、null (図 2) の表現型と同等の開いたリーディング ・ フレームに終止コドンを紹介します。修理テンプレートを変えることによってさまざまな遺伝的交替も可能です。適切な修理テンプレートと再結合を介してナンセンス, ミスセンスとサイレント変異を挿入できます。制限サイトの設立は、それらはなし12,13をシーケンスによって選別する必要があります、形質のスクリーニングを合理化します。さらに、 C. albicans CRISPR により、挿入や削除を生成する研究者それに親和性の札を挿入、プロモーターのスワップを実行し、ノックアウト (図 3) を生成する理想的なシステムを作るします。これらの突然変異のための正しい transformants のスクリーニングは、正しい修理テンプレート定款を確認する編集をシーケンスする必要があるより面倒。さらに、南しみ遺伝子のコピーが追加ゲノム内に挿入されていないことを確認する必要があります。NGG PAM サイトの要件は、対象指定できるゲノムの領域にわずかな制限を配置します。Cpf1 などの代替の核酸を使用する代替の CRISPR システムの開発または Cas9 システムが/が変奏曲14これらの制限の多くを軽減します。この時点で捜査官の知識、これらのシステムはc. アルビカンスにまだ適用されていません。
酵母、 Naumouozyma castellii、ヒトの病原体もカンジダ glabrata11 などの種の様々 な適用することができます上記のプロトコルで説明 CRISPR システムを開発しました。.変換とこれらの酵母の効率的な編集説明プロトコルに若干の変更が必要ですが、これら代替ゲノムの編集のためのフレームワークはc.albicans12で説明したとよく似て。さらに、酵母ゲノムの編集の手順を開発する優れたメカニズムを提供します。酵母、 ADE2変異、アデニン合成経路への前駆物質が蓄積されます、セルを赤きます。この容易に観察可能な表現型では、研究者編集されたセルを識別し、ゲノムのプロトコルを編集を迅速にトラブルシューティングすることができます。菌類の利用可能な広範な分子生物学ツールボックスと組み合わせると、多数の酵母種を編集するためのプロトコルの開発15,16をされています。菌類のゲノム編集技術のような広範なアプリケーション幅広い科学分野に大きく影響する可能性があります。
CRISPR はc. アルビカンス、ゲノム工学の効率を向上、CRISPR までがC. albicansのゲノム広い画面を実行する使用されていません。現在のプロトコルは、 C. albicansの経路を結合する非相同性の終わりは非能率的な12の突然変異を導入する修理テンプレートを必要です。すべての遺伝子の修復テンプレート oligos の生成はゲノム ワイド画面での実行にとって重大な障壁です。DNA 合成と CRISPR 技術の進歩のコストの削減の合流点より現実的削除ライブラリの開発を行います。例えば、CaCas9 ベクトルから修理テンプレートの式はターゲットにすべての遺伝子11持続可能なプラスミド ライブラリの開発のための道を開きます。さらに、 c. アルビカンスゲノムに組み込む CaCas9 発現ベクターを必要としない一過性のカンジダCRISPR プロトコル開発17をされています。また、ガイド式増加ゲノム編集効率18を増加しました。これらと CRISPR 技術その他の進歩c.albicans19,20,21,22のゲノム広い画面の発展に重要です。
C. アルビカンスゲノムは二倍体が A と B の対立遺伝子は、常に同じ5。このようなヘテロ接合では、両方の課題と機会を提供しています。1 つは両方の対立遺伝子を対象とすることを目的と、PAM サイト、ガイド シーケンス、および遺伝子の両方のコピーになる修理テンプレートが使わなければなりません。ただし、一塩基多型遺伝子の存在に応じて、 , C.albicans CRISPR システムは単一の対立遺伝子を対象とする調査をできます。このような精度の対立遺伝子の機能的差異を調べる捜査を許可する可能性があります。または全体の染色体の対立遺伝子のヘテロ接合性 (LOH) の損失が観察されていると、特定の対立遺伝子をターゲットとは、慎重に行う必要があります。シングルc. アルビカンスを編集するとき、対立遺伝子 1 つのクローンが二倍体 SNP プロファイルを維持しているかどうかは、隣接する DNA シーケンスを調べる必要があります。さらに、オフターゲット効果はc.albicans CRISPR のかなり低いのですが、キーの株の全ゲノム配列を考えることができます。
開示事項
著者が明らかに何もありません。
謝辞
著者は読書ありがとう博士 Gennifer メイジャーさんと原稿の役立つコメント。この作品は、ボール州立大学研究所スタートアップ資金と NIH-1R15AI130950-01 D.A.B. にによって支持されました。
資料
| Name | Company | Catalog Number | Comments |
| Chemicals | |||
| Agar | BD Bacto | 214010 | |
| agarose | amresco | 0710-500G | |
| Ampicillan | Sigma-Aldrich | A9518 | |
| Bacto Peptone | BD Bacto | 211677 | |
| Bsmb1 | New England Biolabs | R0580L | |
| Calf intestinal phosphatase (CIP) | New England Biolabs | M0290L | |
| Cut Smart Buffer | New England Biolabs | B7204S | |
| Dimethyl Sulfoxide (DMSO) | Sigma-Aldrich | D8418 | |
| dNTPs | New England Biolabs | N0447L | |
| Ethylenediaminetetraacetic acid (EDTA) | Sigma-Aldrich | 3609 | |
| Glacial Acetic Acid | Sigma-Aldrich | 2810000ACS | |
| Glucose | BDH VWR analytical | BDH9230 | |
| Glycerol | Sigma-Aldrich | 49767 | |
| Kpn1 | New England Biolabs | R3142L | |
| LB-Medium | MP | 3002-032 | |
| Lithium Acetate | Sigma-Aldrich | 517992 | |
| L-Tryptophan | Sigma-Aldrich | T0254 | |
| Maltose | Sigma-Aldrich | M5885 | |
| Molecular Biology Water | Sigma-Aldrich | W4502 | |
| NEB3.1 Buffer | New England Biolabs | B7203S | |
| Nourseothricin | Werner Bioagents | 74667 | |
| Poly(ethylene glycol) PEG 3350 | Sigma-Aldrich | P4338 | |
| Sac1 | New England Biolabs | R3156L | |
| Salmon Sperm DNA | Invitrogen | AM9680 | |
| T4 Polynucleotide kinase | New England Biolabs | M0201S | |
| T4 DNA ligase | New England Biolabs | M0202L | |
| Taq polymerase | New England Biolabs | M0267X | |
| Tris HCl | Sigma-Aldrich | T3253 | |
| uridine | Sigma-Aldrich | U3750 | |
| Yeast Extract | BD Bacto | 212750 | |
| Equipment | |||
| Electrophoresis Appartus | |||
| Incubator | |||
| Microcentifuge | |||
| PCR machine | |||
| Replica Plating Apparatus | |||
| Rollerdrum or shaker | |||
| Spectrophotometer | |||
| Waterbath |
参考文献
- Pfaller, M. A., Diekema, D. J. Epidemiology of invasive candidiasis: a persistent public health problem. Clinincal Microbiology Review. 20 (1), 133-163 (2007).
- Magill, S. S., et al. Multistate point-prevalence survey of health care-associated infections. New England Journal of Medicine. 370 (13), 1198-1208 (2014).
- Wisplinghoff, H., et al. Nosocomial bloodstream infections in US hospitals: analysis of 24,179 cases from a prospective nationwide surveillance study. Clinical Infectious Disease. 39 (3), 309-317 (2004).
- Jones, T., et al. The diploid genome sequence of Candida albicans. Proceedings of the National Academy of Science USA. 101 (19), 7329-7334 (2004).
- Muzzey, D., Schwartz, K., Weissman, J. S., Sherlock, G. Assembly of a phased diploid Candida albicans genome facilitates allele-specific measurements and provides a simple model for repeat and indel structure. Genome Biology. 14 (9), (2013).
- Morschhauser, J., Michel, S., Staib, P. Sequential gene disruption in Candida albicans by FLP-mediated site-specific recombination. Molecular Microbiololgy. 32 (3), 547-556 (1999).
- Lau, V., Davie, J. R. The discovery and development of the CRISPR system in applications in genome manipulation. Biochemistry and Cell Biology. 95 (2), 203-210 (2017).
- Doudna, J. A., Charpentier, E. Genome editing. The new frontier of genome engineering with CRISPR-Cas9. Science. 346 (6213), 1258096 (2014).
- Anders, C., Niewoehner, O., Duerst, A., Jinek, M. Structural basis of PAM-dependent target DNA recognition by the Cas9 endonuclease. Nature. 513 (7519), 569-573 (2014).
- Reardon, S. Welcome to the CRISPR zoo. Nature. 531 (7593), 160-163 (2016).
- Vyas, V. K., et al. New CRISPR Mutagenesis Strategies Reveal Variation in Repair Mechanisms among Fungi. mSphere. 3 (2), (2018).
- Vyas, V. K., Barrasa, M. I., Fink, G. R. A Candida albicans CRISPR system permits genetic engineering of essential genes and gene families. Science Advances. 1 (3), e1500248 (2015).
- Evans, B. A., et al. Restriction digest screening facilitates efficient detection of site-directed mutations introduced by CRISPR in C. albicans UME6. PeerJ. 6, e4920 (2018).
- Kim, D., et al. Genome-wide analysis reveals specificities of Cpf1 endonucleases in human cells. Nature Biotechnology. 34 (8), 863-868 (2016).
- Tarasava, K., Oh, E. J., Eckert, C. A., Gill, R. T. CRISPR-enabled tools for engineering microbial genomes and phenotypes. Biotechnology Journal. , e1700586 (2018).
- Raschmanova, H., Weninger, A., Glieder, A., Kovar, K., Vogl, T. Implementing CRISPR-Cas technologies in conventional and non-conventional yeasts: Current state and future prospects. Biotechnology Advances. 36 (3), 641-665 (2018).
- Min, K., Ichikawa, Y., Woolford, C. A., Mitchell, A. P. Candida albicans Gene Deletion with a Transient CRISPR-Cas9 System. mSphere. 1 (3), (2016).
- Ng, H., Dean, N. Dramatic Improvement of CRISPR/Cas9 Editing in Candida albicans by Increased Single Guide RNA Expression. mSphere. 2 (2), (2017).
- Huang, M. Y., Woolford, C. A., Mitchell, A. P. Rapid Gene Concatenation for Genetic Rescue of Multigene Mutants in Candida albicans. mSphere. 3 (2), (2018).
- Shapiro, R. S., et al. A CRISPR-Cas9-based gene drive platform for genetic interaction analysis in Candida albicans. Nature Microbiology. 3 (1), 73-82 (2018).
- Grahl, N., Demers, E. G., Crocker, A. W., Hogan, D. A. Use of RNA-Protein Complexes for Genome Editing in Non-albicans Candida Species. mSphere. 2 (3), (2017).
- Nguyen, N., Quail, M. M. F., Hernday, A. D. An Efficient, Rapid, and Recyclable System for CRISPR-Mediated Genome Editing in Candida albicans. mSphere. 2 (2), (2017).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved