Method Article
利用 dna 折纸模板将金纳米产物组装成手性质浆细胞分裂体
摘要
我们描述了金纳米棒 dna 起源组装成手性质粒元泡的详细协议, 具有较强的细胞反应。该协议不限于手性配置, 可以很容易地适应各种等离子体结构的制造。
摘要
dna 折纸结构固有的可寻址性使其成为将金属纳米粒子排列成复杂等离子体纳米结构的理想模板。dna 原模板组件的高空间精度允许控制单个粒子的等离子体共振之间的耦合, 并使所构建的纳米结构具有裁剪光学特性。最近, 手性等离子体系统由于等离子体组件的空间结构与其光学响应 (如圆形二色 [cd]) 之间的强相关性而引起了广泛的关注。在该协议中, 我们描述了金纳米棒 (aunr) dna 生成手性组件生成的整个工作流程。该协议包括详细介绍了 dna 折纸模板的制作、aunr 的合成以及原比-aunr 结构的组装的设计原则和实验过程。此外, 还包括利用透射电子显微镜 (tem) 和 cd 光谱对结构进行表征的方法。所述协议不限于手性配置, 可用于各种等离子体结构的构建。
引言
dna 纳米结构, 特别是 dna 折纸, 已被广泛用于排列分子和其他纳米组分 (如蛋白质和纳米粒子 [nps]), 纳米精度几乎任意几何 1,2,3 个,4 个,5. 能够在 dna 折纸模板上排列具有较高的产量和精度的金属 nps, 从而能够制造具有新光学特性的等离子体结构6、7、8、9,10. dna 折纸技术对于手性质粒结构的生成特别有用, 这需要真正的三维结构11,12, 13,14,15,16,17,18,19,20岁。
该协议详细介绍了制备无氮素 dna 原生模板手性组件的整个过程。用于 dna 折纸设计21和结构预测22,23的软件直观且免费提供。创纸制造和 nnr 合成使用常见的生物化学实验室设备 (如热环、凝胶电泳、热板、离心机)。采用标准透射电镜和 cd 光谱对结构进行了表征。
使用自上而下的方法 (例如电子束光刻) 制造类似的等离子体纳米结构将需要相当复杂和昂贵的设备。此外, dna 折纸模板还提供了在等离子体组件 24、25、26、27、28、29中纳入结构可重构性的可能性 , 30,31,32,33,这是极具挑战性的结构制造的光刻技术。与其他基于分子的方法相比,基于 dna的制造提供了高水平的空间精度和可编程性。
研究方案
1. dna 折纸的设计
- 确定所需的 ausnr 相对空间布局和 dna 折纸模板的适当形状 (图 1a)。估计 aunr 和折纸模板的结构参数。找到需要进一步修改的订书针的大致位置 (图 1b)。
- 下载并安装 cadnano18来设计 dna 折纸模板。在 cadnano 中, 根据模板的所需形状路由脚手架和钉线, 并通过单击seq 工具生成钉线序列。单击 "画图工具" 并标记需要进一步修改的装订线 (图 1c)。
- 单击 "导出工具" 将 dna 装订序列 (图 1c) 导出到. csv 文件。
- 设计双链锁, 以固定两个折纸束之间的角度.根据两个束的相对方向, 折纸结构可以适应左手或右手 (lh/rh) 手性空间结构 (图 1b)。
- 将订书针 '. csv 文件导入到电子表格应用程序中。在用于 ausnr 组件 (句柄) 的订书针的末尾添加一个 polya10序列。使用锁序列修改设计的锁站点上的装订线。
注: 代表性结果中的组件包含36个手柄, 突出在短纤维线的 3 ' 端, 18个在每个 dna 折纸束上, 平均分布在两个平行的螺旋每21分。第一个和最后一个手柄位置之间的距离为 168 nt, 大约57纳米 (见所附的 c脱nano 文件)。
2. dna 折纸模板的组装
- 通过混合相同数量的浓度归一化短纤维寡核苷酸 (例如, 100μm), 准备短纤维线 (sm) 的工作库存, 包括带把手和锁的线。
注: 折纸结构通常包含数百根主食。斯台普斯通常是从专门研究多井 (例如96井) 板 dna 寡核苷酸化学合成的供应商那里购买的。 - 对于500μl 的 10 nm 折纸, 混合50μl 的 tris-edta (te, 10x), 100μl 的 mgcl 2 (100 mm), 25μl 的 ncl (100 mm), 175μl 的 h2 o, 100μl sm (0.5μm), 5μl 锁链 (5μm), 以及50μl 支架 (100 nm)。
- 如表 1所示, 将混合物在80°c 至20°c 的热循环器中退火。
3. dna 折纸纯化
注: 本节介绍琼脂糖凝胶纯化的方案。脱氧核糖核酸折纸模板也可以纯化使用替代方法38,39。
- 对于1% 的凝胶, 通过在微波炉中加热混合物, 在100毫升的弗里斯特-硼酸盐-edta (tbe, 0.5倍) 中溶解1克琼脂糖。根据染色规范添加10μl 的 10, 000x dna 染色。为了最大限度地减少在提取步骤 (步骤 3.6) 暴露在紫外线下, 请使用可在蓝色激发下可视化的 dna 染色。
- 将溶液冷却到大约 40°c, 并在晃动时缓慢添加1毫升的 mgcl2 (1.3 m)。在室温下铸造凝胶并孵育30分钟。
- 设置电泳装置, 将冷 (4°c) 运行缓冲液 (0.5倍 tbe, 11 mm mgcl2) 倒入凝胶盒。将凝胶盒放入冰水浴中。
- 在折纸样品中添加加载缓冲液 (6个负载缓冲液在水中含有15% 的多蔗糖400和0.25% 的溴酚蓝)。根据所使用的梳子, 以适当的体积将样品装入井中 (例如,对于厚度为 1.5 mm 的8孔梳子, 为 50μl)。
- 在 80 v 处运行电泳2小时。
注: 要表征折纸并分离开口和闭合结构, 请使用2% 的凝胶而不是 1%, 并将运行时间延长至4小时。 - 用凝胶成像仪对凝胶进行成像 (图 2)。使用蓝光透射灯可视化带, 切割折纸带, 粉碎在准体上的凝胶, 并提取液体。回收率约为40%。
- 将液体移入离心过滤装置, 并以 3, 000 x克旋转 5分钟, 用 uv 可见 (uv-vis) 光谱仪测量260纳米的折纸溶液的吸收。使用 1.3 x 10 8 m-1 ·mcm-1 的消光系数估计折纸的浓度。
注: 琼脂糖凝胶纯化后的折纸溶液的典型浓度为 1-2 nm。 - 将纯化后的折纸模板存放在 4°c, 供以后使用。
4. 金纳米棒的合成
注: nnr 合成协议根据以前的文献40改编, 稍作修改。
- 用水清洗所有玻璃器皿 5分钟, 用水冲洗, 用超纯水将其冲洗干净, 然后在使用前将其擦干。
- 制备 0.2 m 六烷基三甲基溴化物 (ctab)、1 mm hicl 4、4 mmagno 3、64 mm l(+)-抗坏血酸和 6 mm nabh4.使用冷水 (4°c) 溶解 nbh4,并将其保存在4°c 的冰箱中。抗坏血酸溶液必须新鲜制备。
注意: 在皮肤接触 (刺激物)、眼神接触 (刺激物)、摄入和吸入的情况下, ctab 是危险的。穿上合适的防护服。在通风不足的情况下, 穿上合适的呼吸设备。在皮肤接触 (刺激物)、眼神接触 (刺激物)、摄入和吸入的情况下, nbh4 是极其危险的。戴上飞溅护目镜、实验室外套、手套以及蒸汽和灰尘呼吸器。请务必使用经认证的呼吸器或同等标准。 - 准备 au 种子。
- 在玻璃瓶中加入500μl 的 ctab (0.2 m)、250μl 的超纯水和250μl 的 hccl4 (1 mm)。在室温下在450转/分搅拌5分钟。
- 将搅拌速率提高到1200转/分。加入100μl 的冷 nbh4 溶液 (6 mm, 4°c).2分钟后, 停止搅拌, 在30°c 的水浴中孵育溶液30分钟后再使用。
- 准备自动报告。
- 在圆底烧瓶中的温水 (60-65°c) 中溶解 0.55 g 的 ctab 和 0.037 g 的 2, 6-二羟基苯甲酸。将溶液冷却至 30°c, 加入600μl 的 agno 3 (4 mm), 并在 450 rpm下搅拌2分钟。然后, 在30°c 下保持溶液不受干扰15分钟。
- 在溶液中加入15毫升的 haucl 4 (1 mm), 在450转每分钟搅拌 15分钟, 加入 120μl l l l l (+)-抗坏血酸 (64 mm), 然后立即在 1200 rpm 搅拌 30秒, 加入 12μl au 种子, 并在 1200 rpm 下搅拌 30秒.
- 在30°c 的水浴中将溶液培养18小时。不要打扰溶液, 用瓶盖关闭烧瓶。
- 将生成的溶液转移到离心管和离心机, 在 2 0°c 下, 以 9, 500 x克的速度进行12分钟。丢弃上清液, 将颗粒分散在超纯水的20毫升中, 再进行一次离心步骤。
- 将最后的颗粒分散在3毫升的蒸馏水中。利用纵向等离子体共振的消光系数估计 uv-vis 吸收测量的 ausnr 浓度。利用 nnr 形状参数41可以预测消光系数。将 autnr 存放在 4°c, 以便进一步使用。
5. 单链 dna 金纳米棒的功能化
注: 本节介绍了使用单链 dna (ssdna) 进行核能功能化的协议, 遵循了以前文献42所适应的所谓低 ph 值路线。用 dna 覆盖的 ausnr 通过离心纯化;另外, 可以使用琼脂糖凝胶电泳进行纯化。
- 将20微米的硫醇功能化多 t dna 链 (1 mm) 与新鲜制备的 tris(2-羧基乙基) 盐酸磷化氢 (tcep, 14 mm) 培养为 1小时, 以减少二硫键。
注: 硫醇基团与 aunr 形成键, 而与折纸上的 polyT10手柄的多 t 序列杂交, 其中过多或过少的基对可能导致故障或不稳定的组件。
注意: tcep 可导致严重的皮肤灼伤和眼部损伤。穿防护手套/防护服/护眼保护面。 - 混合150μl 的成果 (10nm) 和40μl 的 tcp 处理的硫醇-dna (0.5 mm)。在 nnr 溶液中加入1% 十二烷基硫酸钠 (sds), 最终达到0.05% 的 sds 浓度。使用约1μl 的 hcl (1 m) 将 ph 值调整为2.5-3。
- 在晚上70转的情况下孵化2小时。
注: 根据棒的大小, aunr-dna 比率应在 1:5000-15, 000 左右。对于按照第4节所述协议编写的耐药 (70 纳米 x30 纳米), 建议将硫醇-dna 超过 13, 000。 - 加入氯化钠, 达到 0.5 m 的最终氯化钠协调, 在室温下孵育 4小时, 而在晚上70转的情况下晃动。
注: 此步骤中的颜色变化可能表示 dna 功能化失败。 - 使用 tbe 缓冲液 (10倍) 将 ph 值调整到 ~ 8.5, 并孵育一夜。
- 将样品与1毫升洗涤缓冲液 (0.5倍 tbe 与 0.1% sds) 混合, 以 7, 000 x克离心机清洗, 30分钟内取出上清液, 重新悬浮剩余液体中的 dna-aunr (~ 40μl)。估计 uv-vis 吸收测量中 dna-aunr 的浓度, 如步骤4.4.5 中所述。
注: 由于利用硫醇-dna 从 autnr 表面替换 ctab, 在步骤5.3-5.4 中, 溶液可能会变得略显 "多云"。当加热至 ~ 35°c 5分钟后, 溶液应变得清晰。
6. dna 折纸模板上的金纳米棒组装
- 将 mgcl2添加到纯化的 dna-aunr 溶液中, 最终浓度为 10 mm。将纯化后的 dna-aunr 和折纸混合成10:1 的比例。
注: 较低的比率可能会降低产品产率43。 - 按照表 2中的程序, 将混合物退火在温度控制从40°c 到20°c 的混合器中, 同时在400转/分的情况下晃动。
注: 对于 cd 特性, 样品可以在此步骤之后进行测量, 而无需进一步纯化。 - 使用0.7% 琼脂糖凝胶电泳 (3.5 h 在 80 v), 以净化最终的起源-aunr 结构。
- 使用白光透射光器进行成像。切割产品带 (原生-nar 二聚体) (图 3), 粉碎在副体上的凝胶, 并提取液体。将液体移入离心过滤装置, 以 3, 000 x克的速度旋转 5分钟, 重新将溶液中的原源-ahnr 重新送入溶液。从凝胶中回收产率约为50%。
- 从紫外可见吸收测量中估计原生-aunr 结构的浓度, 如步骤4.4.5 中所述。
7. 透射电子显微镜成像
注: 此尿氨酸甲酸 (ufo) 染色协议改编自以前的文献44。
- 混合 200μl ufo 溶液 (0.75)和1μl 的 naoh (5m) 和涡旋立即 2-3分钟. 离心染色溶液 3-3分钟, 在 14, 000 x克。保护污渍不受光照射 (例如, 将其包裹在铝箔中)。
注意: ufu 如果吸入或吞咽是有毒的, 会引起眼睛刺激。在短暂接触或污染低的情况下, 使用呼吸过滤器装置。在密集或长期接触的情况下, 使用独立的呼吸保护装置。戴上手套。手套材料必须是不透水的, 并且能够抵抗 ufo 及其解决方案。戴上密闭的护目镜。 - 在使用前6个月的发光放电碳包包 tem 网格, 以增加亲水性, 促进结构的粘结。移液器5μl 样品滴在 tem 栅格上, 孵育 5-8, 并通过轻轻触摸滤纸与栅格边缘一起取出。
- 移液器一个大 (约 20μl) 和一个小 (~ 10μl) 滴的染色溶液在一个准叶。将栅格放在小污渍溶液滴上, 然后通过触摸滤芯边缘的滤纸立即干燥。然后, 把它放在大污渍溶液下降30秒。
- 通过触摸带有网格边缘的滤纸, 取出网格上的液体。将网格放在网格支架中。等待网格干燥至少10分钟。
- 用 tem 描述折纸 (图 4)、aunr (图 5) 和折纸-aunr (图 6) 的样品。
8. 圆形二色测量
- 用n2 清洗cd 光谱仪20分钟。
注: 大多数 cd 光谱仪需要在灯具点火前使用n2进行清洗。检查 cd 光谱仪手册。 - 设置带宽、扫描范围和采集步骤。
注: 扫描范围取决于 aunr 的光学特性, 这取决于 ausnr 的大小。 - 使用缓冲区测量空白 cd。
- 测量原位-nnr 样品的 cd 光谱 (图 7)。
注: 使用夸脱或玻璃杯盒进行 cd 测量。塑料试剂盒不适合 cd 光谱。此外, 大多数 cd 光谱仪允许同时采集吸收和 cd 数据。
结果
dna 折纸模板、aunr 和最终折纸-aunr 组件的 tem 图像分别显示在图 4、图 5和图 6a中。由于其对 tem 网格具有约束力的偏好, 起源-aunr 组件通常被视为平行折纸束和棒 (图 6a)。热退火是需要热退火的, 以便在折纸模板上正确对齐才会出现 aunr (图 6a、b)。该协议可将 ausnr 组合成具有强等离子体 cd 响应的手性元壳, 从而实现高产量 (图 7)。
| 温度 (°c) | 时间 |
| 80 | 15分钟 |
| 79-71 | 1°c/1 分钟 |
| 70-66 | 1°c/5 分钟 |
| 65-60 | 1°c/30 分钟 |
| 59-37 | 1°c/60 分钟 |
| 36-30 | 1°c/15 分钟 |
| 29-20 | 1°c/5 分钟 |
| 20 | 按住 |
表 1: dna 折纸模板热退火的温度和速率。
| 温度 (°c) | 时间 (分钟) |
| 40 | 1130 |
| 36 | 180元 |
| 32 | 180元 |
| 22 | 按住 |
表 2: 自动报告和 dna 折纸模板退火的温度和保持时间.各步骤之间的冷却速率设置为0.1°c/min。dna 源-nar 样品在每分钟400转的情况下进行退火。

图 1: dna 原生模板手性元状分子的设计.(a) 确定所需的金纳米棒 (aunr) 的相对空间布局和 dna 折纸模板的适当形状。(b) 估计 aunr (daunr, l失控) 和折纸模板 (w折纸、 l折纸、)的结构参数。找到需要进一步修改的订书针的大致位置。(c) 使用 caDNAno 设计 dna 折纸模板。请点击这里查看此图的较大版本.

图 2: 阿格糖凝胶电泳的阿格山.(a) 用1% 琼脂糖凝胶电泳 2 h 在 80 v (b) 表征与2% 琼脂糖凝胶电泳4小时在 80 v. 请点击这里查看这个数字的更大版本.
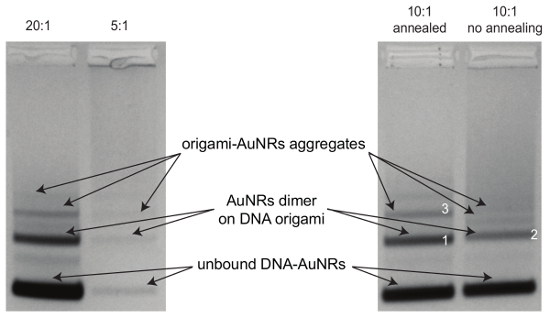
图 3: 琼脂糖凝胶电泳纯化的起源-奥恩.凝胶 (0.7%)在不经过退火程序的情况下, 对于按照不同 dna-aunr-赞同图比 (20x:1, 5:1) 和样品 (10:1 dna-aunrs-to-to-to-作功比) 在组装过程中制备的样品, 在 80 v 时运行了 3.5 h。有关带1、2和3的样本的 tem 图像, 请参见图 6。请点击这里查看此图的较大版本.

图 4: 代表 tem 图像的 dna 折纸模板.折纸结构由两个14螺旋束 (80 纳米 x 16 纳米 x 8 纳米) 连接在一起的脚手架链。请点击这里查看此图的较大版本.

图 5* nr 的代表 tem 形象.合成的成功率为 70 x 30 nm。请点击这里查看此图的较大版本.

图 6: 原集-nnr 组件的透射电镜图像.(a) 退火后对折纸的啊度二等分子 (图3中的波段 1)。(b) 在不退火的情况下对折纸的啊度二等分子 (图3中的波段 2)。(c) 起源-nar 集合体 (图3中的波段 3)。请点击这里查看此图的较大版本.
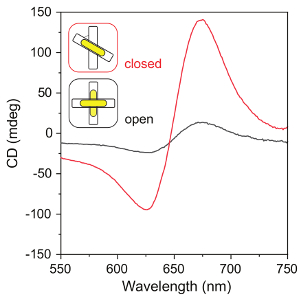
图 7: 原核-nnr 组件的 cd 光谱.闭合的结构的 cd 光谱 (折纸模板由锁链固定入右手配置, 与50°在二个折纸捆绑之间) 和开放结构 (折纸模板没有锁线)。请点击这里查看此图的较大版本.
讨论
该协议介绍了基于 nr 的 dna 特性手性组件的设计、组装、纯化和表征的整个工作流程。协议中使用的 dna 折纸模板特别适用于刺激响应组件的制造。可以将各种类型的响应和功能化合并到定义折纸模板手性状态的锁链中 (图 1b)24、25、26、31。对于静态组件, 更简单的块形状模板通常足够14、45、46、47。
基于 dna 起源的等离子体纳米结构制备方法继承了 dna 折纸技术48的局限性。折纸模板的大小通常受脚手架链大小的限制。在法律条件下, dna 结构的稳定性降低。合成主食的成本仍然相当高。然而, 结构 dna 纳米技术领域的最新发展预计将克服这些限制49、50、51、52、53、54,55岁
与其他基于分子的方法相比, dna 折纸生成了割精 34、35、36、37的手性组件, 它提供了较高的空间精度和可编程性。
为了实现手性组件的可靠和可重复的光学响应, 我们强烈建议调整 nnr 合成40的协议, 因为商业产品的质量和光学性能可能因批次而异。额外的退火 (步骤 6.2) 对于确保将 aunr 正确地连接到 dna 折纸模板通常至关重要 (图 6)。
最后, 这里描述的协议并不限于手性程序集。脱氧核糖核酸折纸为复杂的等离子体纳米结构的制造提供了一个非常灵活的平台 9,10。
披露声明
作者没有什么可透露的。
致谢
作者感谢 s. voutilainen 在 cd 光谱仪方面的帮助。作者承认奥塔纳诺 aalto 大学提供的设施和技术支持----纳米显微镜中心 (aalto-nmc)。这项工作得到了芬兰科学院 (308992 赠款) 和欧洲联盟《地平线2020》研究和创新方案在 marie skvoodowska-curie 第764号赠款协议下的支持。
材料
| Name | Company | Catalog Number | Comments |
| 2,6-Dihydroxybenzoic acid | Sigma-Aldrich | D109606-25 | 98+% |
| AgNO3 | Alfa Aesar | AA1141414 | 99.90% |
| Blue light transilluminator | Nippon Genetics | FG-06 | FastGene LED Transilluminator |
| Bromophenol Blue | Acros Organics | 403160050 | For agarorose gel loading buffer |
| Centrifugal filter units | Merck Millipore | 42600 | DNA extraction from agarose |
| Chirascan CD spectrometer | Applied Photophysics | ||
| Cuvette | Hellma | 105-202-85-40 | Quartz SUPRASIL |
| DNA lobind tubes | Eppendorf | 30108051 | |
| Eppendorf Biospectrometer | Eppendorf | 6135000904 | |
| Eppendorf ThermoMixer C | Eppendorf | 5382000015 | |
| Ficoll 400 | Thermo Fisher Scientific | BP525-10 | Polysucrose 400 (For agarorose gel loading buffer) |
| Gel electrophoresis sets | Thermo Fisher Scientific | ||
| Gel imager | Bio-Rad | Gel Doc XR+ System | |
| HAuCl4•3H2O | Alfa Aesar | AA3640006 | 99.99% |
| HCl | Scharlau | AC07441000 | 1M |
| Hexadecyltrimethylammonium bromide (CTAB) | Sigma-Aldrich | H9151-100 | BioXtra, 98+% |
| L(+)-ascorbic acid | Acros Organics | 401471000 | 99+% |
| M13p7560 scaffold strand | Tilibit nanosystems | ||
| MgCl2•6H2O | Sigma-Aldrich | M2670-500 | BioXtra, 99+% |
| NaBH4 | Acros Organics | 200050250 | 99% |
| NaCl | Sigma-Aldrich | S7653-500 | BioXtra, 99.5+% |
| NaOH | Sigma-Aldrich | S8045-500 | BioXtra, 98+% |
| Parafilm | Sigma-Aldrich | P7668-1EA | PARAFILM M |
| PBS buffer (10X) | Thermo Fisher Scientific | BP3991 | Molecular Biology |
| ProFlex PCR System | Thermo Fisher Scientific | 4484073 | |
| Sodium dodecyl sulfate (SDS) | Sigma-Aldrich | 74255-250 | 99+% |
| Staple strands | Thermo Fisher Scientific | ||
| Sybr Safe | Invitrogen | S33102 | For DNA stain |
| TBE buffer (10X) | Invitrogen | 15581-044 | Molecular Biology |
| TE buffer (10X) | Thermo Fisher Scientific | BP24771 | Molecular Biology |
| TEM | FEI | FEI Tecnai F12 | |
| Thiol-functionalized ssDNA | Biomers.net | ||
| Tris(2-carboxyethyl)phosphine hydrochloride (TCEP-HCl) | Thermo Fisher Scientific | PI20491 | |
| UltraPure Agarose | Invitrogen | 16500-100 | |
| Ultrapure water (Type 1) | Milli-Q Direct 8 system | ||
| Uranyl Formate | Tebu-bio | 24762-1 | |
| White light transilluminator | UVP | TW-26 |
参考文献
- Rothemund, P. W. K. Folding DNA to create nanoscale shapes and patterns. Nature. 440, 297-302 (2006).
- Wang, P., Meyer, T. A., Pan, V., Dutta, P. K., Ke, Y. The Beauty and Utility of DNA Origami. Chem. 2, 359-382 (2017).
- Hong, F., Zhang, F., Liu, Y., Yan, H. DNA Origami: Scaffolds for Creating Higher Order Structures. Chemical Reviews. 117, 12584-12640 (2017).
- Roller, E. -M., Argyropoulos, C., Högele, A., Liedl, T., Pilo-Pais, M. Plasmon-Exciton Coupling Using DNA Templates. Nano Letters. 16, 5962-5966 (2016).
- Acuna, G. P., et al. Fluorescence Enhancement at Docking Sites of DNA-Directed Self-Assembled Nanoantennas. Science. 338, 506-510 (2012).
- Roller, E. -M., et al. DNA-Assembled Nanoparticle Rings Exhibit Electric and Magnetic Resonances at Visible Frequencies. Nano Letters. 15, 1368-1373 (2015).
- Liu, Q., Song, C., Wang, Z. -G., Li, N., Ding, B. Precise organization of metal nanoparticles on DNA origami template. Methods. 67, 205-214 (2014).
- Roller, E. -M., et al. Hotspot-mediated non-dissipative and ultrafast plasmon passage. Nature Physics. 13, 761-765 (2017).
- Liu, N., Liedl, T. DNA-Assembled Advanced Plasmonic Architectures. Chemical Reviews. 118, 3032-3053 (2018).
- Kuzyk, A., Jungmann, R., Acuna, G. P., Liu, N. DNA Origami Route for Nanophotonics. ACS Photonics. 5, 1151-1163 (2018).
- Kuzyk, A., et al. DNA-based self-assembly of chiral plasmonic nanostructures with tailored optical response. Nature. 483, 311-314 (2012).
- Shen, X., et al. Three-Dimensional Plasmonic Chiral Tetramers Assembled by DNA Origami. Nano Letters. 13, 2128-2133 (2013).
- Liu, H., Shen, X., Wang, Z. -G., Kuzyk, A., Ding, B. Helical nanostructures based on DNA self-assembly. Nanoscale. 6, 9331-9338 (2014).
- Shen, X., et al. 3D plasmonic chiral colloids. Nanoscale. 6, 2077-2081 (2014).
- Urban, M. J., et al. Plasmonic Toroidal Metamolecules Assembled by DNA Origami. Journal of the American Chemical Society. 138, 5495-5498 (2016).
- Hentschel, M., Schäferling, M., Duan, X., Giessen, H., Liu, N. Chiral plasmonics. Science Advances. 3, 1602735(2017).
- Cecconello, A., Besteiro, L. V., Govorov, A. O., Willner, I. Chiroplasmonic DNA-based nanostructures. Nature Reviews Materials. 2, 17039(2017).
- Lan, X., Wang, Q. Self-Assembly of Chiral Plasmonic Nanostructures. Advanced Materials. 28, 10499-10507 (2016).
- Shen, C., et al. Spiral Patterning of Au Nanoparticles on Au Nanorod Surface to Form Chiral AuNR@AuNP Helical Superstructures Templated by DNA Origami. Advanced Materials. 29, 1606533(2017).
- Zhou, C., Duan, X., Liu, N. A plasmonic nanorod that walks on DNA origami. Nature Communications. 6, 8102(2015).
- Douglas, S. M., et al. Rapid prototyping of 3D DNA-origami shapes with caDNAno. Nucleic Acids Research. 37, 5001-5006 (2009).
- Kim, D. -N., Kilchherr, F., Dietz, H., Bathe, M. Quantitative prediction of 3D solution shape and flexibility of nucleic acid nanostructures. Nucleic Acids Research. 40, 2862-2868 (2012).
- Maffeo, C., Yoo, J., Aksimentiev, A. De novo reconstruction of DNA origami structures through atomistic molecular dynamics simulation. Nucleic Acids Research. 44, 3013-3019 (2016).
- Kuzyk, A., et al. Reconfigurable 3D plasmonic metamolecules. Nature Materials. 13, 862-866 (2014).
- Kuzyk, A., et al. A light-driven three-dimensional plasmonic nanosystem that translates molecular motion into reversible chiroptical function. Nature Communications. 7, 10591(2016).
- Kuzyk, A., Urban, M. J., Idili, A., Ricci, F., Liu, N. Selective control of reconfigurable chiral plasmonic metamolecules. Science Advances. 3, 1602803(2017).
- Gerling, T., Wagenbauer, K. F., Neuner, A. M., Dietz, H. Dynamic DNA devices and assemblies formed by shape-complementary, non-base pairing 3D components. Science. 347, 1446-1452 (2015).
- Zhou, C., Duan, X., Liu, N. DNA-Nanotechnology-Enabled Chiral Plasmonics: From Static to Dynamic. Accounts of Chemical Research. 50, 2906-2914 (2017).
- Ijäs, H., et al. Dynamic DNA Origami Devices: from Strand-Displacement Reactions to External-Stimuli Responsive Systems. International Journal of Molecular Sciences. 19, 2114(2018).
- Lan, X., et al. DNA-Guided Plasmonic Helix with Switchable Chirality. Journal of the American Chemical Society. 140, 11763-11770 (2018).
- Funck, T., Nicoli, F., Kuzyk, A., Liedl, T. Sensing Picomolar Concentrations of RNA Using Switchable Plasmonic Chirality. Angewandte Chemie International Edition. 57, 13495-13498 (2018).
- Zhou, C., Xin, L., Duan, X., Urban, M. J., Liu, N. Dynamic Plasmonic System That Responds to Thermal and Aptamer-Target Regulations. Nano Letters. 18, 7395-7399 (2018).
- Zhan, P., et al. Reconfigurable Three-Dimensional Gold Nanorod Plasmonic Nanostructures Organized on DNA Origami Tripod. ACS Nano. 11, 1172-1179 (2017).
- Ma, W., et al. Attomolar DNA detection with chiral nanorod assemblies. Nature Communications. 4, 2689(2013).
- Ma, W., et al. Chiral plasmonics of self-assembled nanorod dimers. Scientific Reports. 3, 1934(2013).
- Guerrero-Martínez, A., et al. Intense Optical Activity from Three-Dimensional Chiral Ordering of Plasmonic Nanoantennas. Angewandte Chemie International Edition. 50, 5499-5503 (2011).
- Kumar, J., et al. Detection of amyloid fibrils in Parkinson's disease using plasmonic chirality. Proceedings of the National Academy of Sciences. 115, 3225-3230 (2018).
- Stahl, E., Martin, T. G., Praetorius, F., Dietz, H. Facile and Scalable Preparation of Pure and Dense DNA Origami Solutions. Angewandte Chemie International Edition. 53, 12735-12740 (2014).
- Shaw, A., Benson, E., Högberg, B. Purification of Functionalized DNA Origami Nanostructures. ACS Nano. 9, 4968-4975 (2015).
- Ye, X., et al. Improved Size-Tunable Synthesis of Monodisperse Gold Nanorods through the Use of Aromatic Additives. ACS Nano. 6, 2804-2817 (2012).
- Near, R. D., Hayden, S. C., Hunter, R. E., Thackston, D., El-Sayed, M. A. Rapid and Efficient Prediction of Optical Extinction Coefficients for Gold Nanospheres and Gold Nanorods. The Journal of Physical Chemistry C. 117, 23950-23955 (2013).
- Shi, D., Song, C., Jiang, Q., Wang, Z. -G., Ding, B. A facile and efficient method to modify gold nanorods with thiolated DNA at a low pH value. Chemical Communications. 49, 2533-2535 (2013).
- Gür, F. N., Schwarz, F. W., Ye, J., Diez, S., Schmidt, T. L. Toward Self-Assembled Plasmonic Devices: High-Yield Arrangement of Gold Nanoparticles on DNA Origami Templates. ACS Nano. 10, 5374-5382 (2016).
- Castro, C. E., et al. A primer to scaffolded DNA origami. Nature Methods. 8, 221-229 (2011).
- Lan, X., et al. Bifacial DNA Origami-Directed Discrete, Three-Dimensional, Anisotropic Plasmonic Nanoarchitectures with Tailored Optical Chirality. Journal of the American Chemical Society. 135, 11441-11444 (2013).
- Lan, X., et al. Au Nanorod Helical Superstructures with Designed Chirality. Journal of the American Chemical Society. 137, 457-462 (2015).
- Zhu, C., Wang, M., Dong, J., Zhou, C., Wang, Q. Modular Assembly of Plasmonic Nanoparticles Assisted by DNA Origami. Langmuir. , (2018).
- Pinheiro, A. V., Han, D., Shih, W. M., Yan, H. Challenges and opportunities for structural DNA nanotechnology. Nature Nanotechnology. 6, 763-772 (2011).
- Ducani, C., Kaul, C., Moche, M., Shih, W. M., Högberg, B. Enzymatic production of 'monoclonal stoichiometric' single-stranded DNA oligonucleotides. Nature Methods. 10, 647-652 (2013).
- Praetorius, F., et al. Biotechnological mass production of DNA origami. Nature. 552, 84-87 (2017).
- Wagenbauer, K. F., Sigl, C., Dietz, H. Gigadalton-scale shape-programmable DNA assemblies. Nature. 552, 78-83 (2017).
- Zhang, T., et al. 3D DNA Origami Crystals. Advanced Materials. 30, 1800273(2018).
- Ong, L. L., et al. Programmable self-assembly of three-dimensional nanostructures from 10,000 unique components. Nature. 552, 72-77 (2017).
- Ponnuswamy, N., et al. Oligolysine-based coating protects DNA nanostructures from low-salt denaturation and nuclease degradation. Nature Communications. 8, 15654(2017).
- Agarwal, N. P., Matthies, M., Gür, F. N., Osada, K., Schmidt, T. L. Block Copolymer Micellization as a Protection Strategy for DNA Origami. Angewandte Chemie International Edition. 56, 5460-5464 (2017).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。