Method Article
DNA 종이 접기 서식 파일을 사용 하 여 키 랄 Plasmonic Metamolecules로 금 Nanorods의 조립
요약
우리는 DNA 종이 접기 기반 어셈블리 골드 nanorods의 강한 chiroptical 응답 랄 plasmonic metamolecules에 대 한 상세한 프로토콜을 설명합니다. 프로토콜 랄 구성에 국한 되지 않습니다 하 고 다양 한 plasmonic 아키텍처의 제작을 위해 쉽게 적응 시킬 수 있다.
초록
DNA 종이 접기 구조의 내재 접근성은 그들이 복잡 한 plasmonic nanostructures에 금속 나노 입자의 배열에 대 한 이상적인 서식 파일. DNA 종이 접기 템플릿 어셈블리의 공간 고정밀 제어 개별 입자의 plasmonic 공명 간의 결합 있으며 건설된 nanostructures의 광학 특성에 맞게 수 있습니다. 최근, 키 랄 plasmonic 시스템 plasmonic 어셈블리의 공간 구성 및 그들의 광학 응답 (예를 들어, 원형이 색 성 [CD]) 사이 강한 상관 관계 때문에 관심을 많이 끌었다. 이 프로토콜에서 우리는 금 nanorods (AuNRs)의 종이 접기-기반 랄 어셈블리 DNA의 세대에 대 한 전체 워크플로 설명합니다. 프로토콜 설계 원칙 및 DNA 종이 접기 템플릿 제작, AuNRs, 합성 및 종이 접기-AuNR 구조체의 어셈블리에 대 한 실험 절차에 대 한 자세한 설명이 포함 되어 있습니다. 또한, 전송 전자 현미경 (TEM) 및 CD 분광학을 사용 하 여 구조체의 특성 포함 됩니다. 설명된 프로토콜 랄 구성에 국한 되지 않습니다 하 고 다양 한 plasmonic 아키텍처의 건설에 대 한 적응 시킬 수 있다.
서문
DNA nanostructures, DNA 종이 접기 특히 널리 사용 된 분자와 다른 나노 스케일 구성 요소 (예: 단백질 및 나노 [NPs])을 나노미터 정밀도로 거의 임의의 형상1,2 로 , 3 , 4 , 5. 높은 수율 및 정확도 가진 DNA 종이 접기 템플릿에 금속 NPs를 정렬 하는 기능 수 소설 광 속성6,,78, plasmonic 구조체의 제조 9 , 10. DNA 종이 접기 기술 특히 유용 구조의 키 랄 plasmonic, 진정으로 3 차원 아키텍처11,12,13, 를 요구 하는 세대에 대 한 14 , 15 , 16 , 17 , 18 , 19 , 20.
이 프로토콜 자세하게에서 AuNRs의 DNA 종이 접기 템플릿 랄 어셈블리의 제작의 전체 과정을 설명합니다. 소프트웨어 디자인21 에 대 한 사용 되며 구조 예측22,23 DNA 종이 접기의 직관적이 고 자유롭게 사용할 수 있습니다. 종이 접기 제조 및 AuNR 합성 일반적인 생화학 실험실 장비 (예를 들어, thermocyclers, 젤 전기 이동 법, 핫 플레이트, 원심 분리기)를 사용합니다. 구조는 표준 가장 및 CD 분광학을 사용 하 여 특징 이다.
하향식 방법 (예를 들어, 전자 빔 리소 그래피)와 비슷한 plasmonic nanostructures의 제조는 오히려 복잡 하 고 비싼 장비를 요구할 것입니다. 또한, DNA 종이 접기 템플릿은 plasmonic 어셈블리24,25,26,,2728,29 구조 갖추었을 통합 가능성 ,30,31,,3233, 리소 그래피 기술로 가공 하는 구조에 대 한 매우 도전입니다. 다른 분자 기반 접근34,35,,3637에 비해, DNA 종이 접기-기반 제조 높은 수준의 공간 정밀도 및 프로그래밍 기능을 제공 합니다.
프로토콜
1입니다. 디자인의 DNA 종이 접기
- AuNRs의 원하는 상대 공간 배열 및 DNA 종이 접기 템플릿 (그림 1A)의 적당 한 모양을 식별 합니다. 종이 접기 템플릿과 AuNRs의 구조 매개 변수를 견적 한다. 추가 수정 (그림 1B)를 필요로 하는 먹을거리의 대략적인 위치를 찾습니다.
- 다운로드 및 설치 caDNAno18 DNA 종이 접기 템플릿 디자인. CaDNAno에서 서식 파일의 원하는 모양에 따라 비 계 및 스테이플 가닥 하 고 Seq 도구를 클릭 하 여 주식 가닥 시퀀스를 생성 합니다. 페인트 도구 를 클릭 하 고 수정 (그림 1C)를 추가 해야 하는 주식 가닥을 표시 합니다.
- DNA 주식 순서 (그림 1C).csv 파일로 내보내려면 내보내기 도구 를 클릭 합니다.
- 두 종이 접기 번들 사이 각 Θ 를 해결 하기 위해 더블-좌초 자물쇠 디자인. 두 개의 번들의 상대적 방향에 따라 종이 접기 구성 왼쪽-또는 오른 (LH/RH) 랄 공간 구성 (그림 1B)를 적응할 수 있습니다.
- 스프레드시트 응용 프로그램으로 스테이플.csv 파일을 가져옵니다. PolyA10 시퀀스 스테이플 AuNR 어셈블리 (핸들)에 대 한 사용의 끝에 추가 합니다. 잠금 시퀀스 설계 된 잠금 위치에 스테이플 가닥을 수정 합니다.
참고: 대표 결과에 어셈블리 포함 36 주식 가닥의 3' 끝에 튀어나온 손잡이, 동등 하 게 두 개의 배포 각 DNA 종이 접기 번들에 18 병렬 나선 모든 21 nt. 첫 번째 및 마지막 핸들 위치 사이의 거리는 168 nt, 약 57 nm (caDNAno 첨부 파일 참조).
2입니다. DNA 종이 접기 서식 파일 어셈블리
- 작업 재고 주식 가닥 (SM), 핸들 및 잠금, 물가 포함 하 여 동일한 양의 주식 oligonucleotides 농도 정규화 (예:, 100 µ M)를 혼합 하 여 준비 합니다.
참고: 종이 접기 구조는 보통 주식 물가의 몇몇 수백을 포함합니다. 스테이플은 일반적으로 (예를 들어, 96-잘) multiwell DNA oligonucleotides의 화학 합성에서 전문 공급 업체에서 구입한 접시. - 10 nM 종이 접기의 500 µ L, 트리 스-EDTA (테, 10 배), MgCl2 (100 mM) 100 µ L, NaCl (100 m m), H2O, 100 µ L의 SM의 175 µ L의 25 µ L의 50 µ L 섞는다 (0.5 µ M), 잠금 가닥 (5 µ M)의 5 µ L 그리고 50 µ L 비 계 (100 nM).
- 표 1에 설명 된 대로 20 ° C에 80 ° C에서 thermocycler에 혼합물을 anneal.
3. DNA 종이 접기 정화
참고:이 섹션에서는 agarose 젤 정화에 대 한 프로토콜을 설명합니다. DNA 종이 접기 템플릿은 대체 방법을38,39또한 정화 수 있습니다.
- 1% 젤, 분해 트리 스-borate-EDTA의 100 ml에서 agarose의 1 g (TBE, 0.5 x) 렌지에 혼합물을가 열 하 여. 10000 x DNA 얼룩 얼룩 사양에 따라 10 µ L를 추가 합니다. 추출 단계 (단계 3.6)에 자외선에 노출을 최소화 하기 위해 블루 여기에서 구상 될 수 있다 DNA 얼룩을 사용 합니다.
- 약 40 ° C에 대 한 해결책을 냉각 하 고 천천히 흔들어 동안 MgCl2 (1.3 M)의 1 mL을 추가. 젤을 캐스팅 하 고 실 온에서 30 분 동안 품 어.
- 전기 장치를 설정 하 고 부 어 감기 (4 ° C) 실행 버퍼 (0.5 11 m m MgCl2와 x TBE) 젤 상자에. 얼음 물 목욕에서 젤 상자를 놓습니다.
- (6 x 로딩 버퍼 포함 15% polysucrose 400 및 0.25 %bromophenol 물에 파란색) 종이 접기 샘플을 로딩 버퍼를 추가 합니다. 빗을 사용 (예:, 50 µ L 두께에서 1.5 m m의 8 잘 빗에 대 한)에 따라 적절 한 볼륨으로 우물에는 샘플을 로드 합니다.
- 80 V에 2 시간에 대 한 전기를 실행 합니다.
참고: 종이 접기 특성화 고 열리고 닫힌 구조를 분리, 1 %2% 젤을 사용 하 고 4 h 실행 시간을 연장. - 젤 젤 영상 (그림 2)와 함께 이미지. 블루 빛 transilluminator를 사용 하 여 시각화 하는 밴드, 종이 접기 밴드를 잘라, parafilm에 젤을 버리고 고 액체를 추출. 복구 비중은 약 40%입니다.
- 원심 필터 장치에 액체를 플라스틱 및 3000 x g 5 분에서 스핀 측정 260에서 종이 접기 솔루션의 흡수 UV 표시 (UV-VI) 분석기 nm. 종이 접기 108 M-1·cm-1x 1.3의 소멸 계수를 사용 하 여 농도 견적 한다.
참고: 종이 접기 솔루션 agarose 젤 정화 후의 전형적인 농도 1-2 nM. - 나중에 사용할 4 ° C에서 순화 된 종이 접기 서식 파일을 저장 합니다.
4입니다. 골드 nanorods의 합성
참고: AuNR 합성에 대 한 프로토콜은 사소한 수정 이전 문학40 에서 적응.
- 5 분을 위한 아쿠아 레 지아와 모든 유리 그릇을 씻어, 물으로 씻어, 초순, 함께 sonicate 그리고 사용 하기 전에 건조.
- 준비 0.2 M hexadecyltrimethylammonium 브 로마 이드 (CTAB), 1 mM HAuCl4, 4mm AgNO3, 64 m m L (+)-아 스 코르 빈 산과 6mm NaBH4. 냉 수 (4 ° C)를 사용 하 여 분해 NaBH4 4 ° c.에는 냉장고에 보관을 Ascorbic의 산 성 솔루션 갓 준비 했다.
주의: CTAB (자극성) 피부 접촉, 눈 접촉 (자극), 섭취, 흡입 시 위험입니다. 적절 한 보호복을 착용 하십시오. 부족 한 환기의 경우 적당 한 호흡 장비를 착용 하십시오. NaBH4 가 (자극성) 피부 접촉, 눈 접촉 (자극), 섭취, 흡입 시 매우 위험 합니다. 스플래시 고글, 실험실 외 투, 장갑, 그리고 증기와 먼지 인공 호흡기를 착용 하십시오. 승인/인증 호흡기 또는 해당 사용 해야 합니다. - Au 씨앗을 준비 합니다.
- 유리 제 작은 유리병에 CTAB (0.2 M), 250 µ L의 초순, 고 HAuCl4 (1mm)의 250 µ L의 500 µ L를 추가 합니다. 5 분 동안 실내 온도에 450 rpm에서 저 어.
- 교 반 속도 1200 rpm를 증가. 차가운 NaBH4 솔루션 (6 m m, 4 ° C)의 100 µ L를 추가 합니다. 2 분 후에 교 반 중지 하 고 사용 하기 전에 30 분 동안 30 ° C에서 물 욕조에 솔루션을 품 어.
- AuNRs을 준비 합니다.
- 둥근 바닥 플라스 크에 0.55 g CTAB에의 하 고 따뜻한 물 (60-65 ° C)의 15 ml 2, 6-dihydroxybenzoic 산의 0.037 g를 용 해. 30 ° C에 대 한 해결책을 식히십시오 AgNO3 (4mm)의 600 µ L을 추가 하 고 450 rpm 2 분 저 어. 그런 다음, 솔루션을 30 ° c.에서 15 분 동안 그대로 두고
- 솔루션, HAuCl4 (1mm)의 15 mL를 추가 하 고 15 분 추가 120 µ L L (+)의 대 한 450 rpm 볶음-아 스 코르 빈 산 (64 m m), 즉시, Au 씨앗의 30 s. 추가 12 µ L에 대 한 1200 rpm에서 저 어 그리고 30 1200 rpm에서 계속 저 어 s.
- 18 h 30 ° C에서 물 욕조에 솔루션을 품 어. 안 솔루션을 방해 하 고 모자를 사용 하 여 플라스 크를 닫습니다.
- 원심 분리기 튜브, 및 20 ° c.에 12 분 9500 x g 에서 원심 결과 솔루션을 전송 삭제는 상쾌한, 초순 물 20 mL에 펠 릿을 분산 고 원심 분리 단계를 수행 합니다.
- 마지막 펠 릿 3 mL의 증류수에 분산. 경도 플라스몬 공명에 대 한 소멸 계수를 사용 하 여 대 한 UV 흡수 측정에서 AuNRs의 농도 견적 한다. 소 광 계수 AuNR 모양 매개 변수41을 사용 하 여 예언 될 수 있다. 추가 사용에 대 한 4 ° C에서 AuNRs를 저장 합니다.
5. 단일 가닥 DNA와 금 nanorods의 기능화
참고:이 섹션에서는 이전 문학42에서 적응 하는 소위 낮은 pH 경로 따라 단일 가닥 dna (ssDNA) AuNR 기능화에 대 한 프로토콜을 설명 합니다. DNA로 덮여 AuNRs 원심 분리;에 의해 순화 된다 또는, 정화 agarose 젤 전기 이동 법을 사용 하 여 수행할 수 있습니다.
- 갓의 20 µ L로 thiol 기능성된 polyT DNA 가닥 (1mm)의 20 µ L를 품 어 tris(2-carboxyethyl) 이황화 결합을 줄이기 위해 1 h phosphine 염 (TCEP, 14 m m).
참고: thiol 그룹 형태로 채권 AuNRs, 그리고 polyT 시퀀스에 종이 접기, 너무 많거나 너무 적은 기본적인 쌍 오작동 또는 불안정 한 어셈블리 이어질 수 polyA10 핸들 hybridizes.
주의: TCEP는 심한 피부 화상과 눈 손상을 일으킬 수 있습니다. 보호 장갑/보호 의류/눈 보호/얼굴 보호를 착용 하십시오. - AuNRs의 믹스 150 µ L (10 nM) 및 TCEP 취급 thiol-DNA (0.5 m m)의 40 µ L. 0.05%의 최종 SDS 농도 도달 AuNR 솔루션 1% 나트륨 라우릴 황산 염 (SDS)를 추가 합니다. 2.5-3 (1m) HCl의 ~ 1 µ L로 pH를 조정 합니다.
- 2 h 70 rpm에서 떨고 있는 동안 품 어.
참고: AuNR DNA 비율 1:5, 000-15, 000, 막대의 크기에 따라 순서 대로 해야 합니다. AuNRs에 대 한 (70 nm x 30 nm) 준비 다음 프로토콜 thiol DNA의 4, 13, 000 초과 섹션에 설명 된 것이 좋습니다. - 0.5 M의 최종 NaCl concertation를 도달 하 고 실 온에서 4 h 70 rpm에서 떨고 있는 동안 품 어 NaCl을 추가 합니다.
참고:이 단계에서 색상 변화는 실패 한 DNA 기능화를 나타낼 수 있습니다. - TBE 버퍼 (10 배)와 ~8.5에 pH를 조정 하 고 밤새 품 어.
- DNA-AuNRs 4 워시 샘플 버퍼 세척의 1 mL와 혼합 하 여 x (0.5 0.1 %SDS x TBE), 그리고 원심 분리기 7000 x g 에서 30 분에는 상쾌한을 제거 하 고 남은 액체 (~ 40 µ L)에서 DNA AuNRs resuspend. 4.4.5 단계에 설명 된 대로 대 한 UV 흡수 측정에서 DNA AuNRs의 농도 견적 한다.
참고: 솔루션 thiol DNA로는 AuNRs의 표면에서 CTAB 교체로 인해 5.3 5.4 단계에서 약간 '흐린' 될 수도 있습니다. 솔루션 5 분 동안 최대 ~ 35 ° C 온난 화 시 분명 될 것입니다.
6. DNA 종이 접기 템플릿에 골드 nanorods의 조립
- MgCl2 순화 된 DNA-AuNRs, 10 m m의 최종 농도를 솔루션에 추가 합니다. 순화 된 DNA AuNRs와 종이 접기를 10:1 비율로 섞는다.
참고: 낮은 비율43의 제품 생산량 줄일 수 있습니다. - 표 2의 절차에 따라 400 rpm에서 떨고 있는 동안 20 ° C에 40 ° C에서 온도 제어와 믹서에서 혼합 anneal.
참고: CD 특성에 대 한 샘플 더 정화 없이이 단계 후에 측정할 수 있습니다. - 0.7 %agarose 젤 전기 이동 법 (3.5 h 80 V)를 사용 하 여 최종 종이 접기-AuNR 구조를 정화.
- 흰 빛 transilluminator를 사용 하 여 이미지에 대 한. 제품 밴드 (종이 접기-AuNR 이합체)를 잘라 (그림 3), parafilm에 젤을 버리고, 액체를 추출. 피펫은 원심 필터 단위 및 5 분에 대 한 3000 x g 에서 스핀으로 액체 솔루션에서 종이 접기-AuNRs Resuspend. 젤에서 복구 수율은 약 50%입니다.
- 4.4.5 단계에 설명 된 대로 대 한 UV 흡수 측정에서 종이 접기-AuNR 구조체의 농도 견적 한다.
7. 전송 전자 현미경 이미징
참고:이 uranyl 편대 (UFo) 프로토콜을 얼룩이 지는 이전 문학44에서 적응입니다.
- 믹스 200 µ L의 UFo 솔루션 (0.75%) 그리고 1 µ L NaOH (5 M)와 2-3 분 위한 즉시 소용돌이의 원심 14000 x g에서 3-4 분 동안 얼룩 솔루션. (예를 들어, 알루미늄 호 일에 포장) 하 여 얼룩을 가벼운 노출에서 보호 합니다.
주의: UFo 독성이 흡입 또는 삼 킨 경우 눈 자극을 일으킬 수 있다. 간단한 노출 또는 낮은 오염, 호흡기 필터 장치를 사용 합니다. 집중 또는 더 긴 노출의 경우 자가 호흡기 보호 장치를 사용 합니다. 장갑을 착용 한다. 장갑 재료는 불 침투성 및 UFo와의 솔루션에 저항력이 있다. 단단히 밀봉된 고글을 착용. - 글로우 방전 6 편 격자 탄소/formvar 코팅 화란 증가 및 구조의 튀어나와 홍보를 사용 하기 전에 단지 s. 플라스틱 편 격자에 5 µ L 샘플 상품, 5-8 분 동안 품 어 고 필터 종이 격자의 가장자리를 부드럽게 만져 드롭을 제거 합니다.
- 하나는 parafilm에 얼룩이 솔루션의 드롭 큰 (~ 20 µ L) 및 1 개의 작은 (~ 10 µ L) 플라스틱. 작은 얼룩 솔루션 방울에 그리드를 넣고 그리드의 가장자리 필터 종이 만져 즉시 건조. 그런 다음, 30의 큰 얼룩 솔루션 방울에 넣어 s.
- 격자의 가장자리와 필터 종이 만져에 액체를 제거 합니다. 표에서 보유자에 그리드를 배치 합니다. 적어도 10 분 동안 건조 그리드를 기다립니다.
- 종이 접기 (그림 4), AuNRs (그림 5), 및 종이 접기-AuNRs (그림 6) 가장에 의해 샘플을 특징.
8. 원형이 색 성 측정
- 20 분에 대 한 N2 CD 분석기를 제거.
참고: CD 분석기의 대부분 N2 램프 점화 하기 전에 정화 필요 합니다. CD 분 광 계 설명서를 확인 하십시오. - 대역폭, 검색 범위, 및 수집 단계를 설정 합니다.
참고: 검색 범위는 AuNRs의 크기에 따라 AuNRs의 광학 속성에 따라 달라 집니다. - 버퍼와 측정 빈 Cd입니다.
- 종이 접기-AuNR 샘플 (그림 7)의 CD 스펙트럼을 측정 합니다.
참고: CD 측정에 대 한 쿼트 또는 유리 큐 벳을 사용 합니다. 플라스틱 큐 벳 CD 분광학에 대 한 적합 하지 않습니다. 또한, 대부분 CD 분석기 흡수 및 CD 데이터의 동시 취득을 수 있습니다.
결과
DNA 종이 접기 템플릿, AuNRs, 및 마지막 종이 접기-AuNR 어셈블리의 TEM 이미지는 각각 그림 4, 그림 5, 그림 6A에 표시 됩니다. 때문에 그들의 바인딩 특혜 가장 격자를, 종이 접기-AuNR 어셈블리 일반적으로 병렬 종이 접기 번들 및 막대 (그림 6A)로 보인다. 열 어 닐 링 하는 것은 종이 접기 템플릿 (그림 6A, B)에 AuNRs의 정확한 줄 맞춤에 대 한 필요 합니다. 프로토콜에는 강한 plasmonic CD 반응 (그림 7) 랄 metamolecules로 AuNRs의 어셈블리의 높은 수율 수 있습니다.
| 온도 (° C) | 시간 |
| 80 | 15 분 |
| 79-71 | 1 ° C/1 분 |
| 70-66 | 1 ° C/5 분 |
| 65-60 | 1 ° C/30 분 |
| 59-37 | 1 ° C/60 분 |
| 36-30 | 1 ° C/15 분 |
| 29-20 | 1 ° C/5 분 |
| 20 | 보류 |
표 1: 온도 그리고 DNA 종이 접기 템플릿 열 어 닐 링에 대 한 요금.
| 온도 (° C) | 시간 (분) |
| 40 | 130 |
| 36 | 180 |
| 32 | 180 |
| 22 | 보류 |
표 2: 온도 및 AuNRs 및 DNA 종이 접기 템플릿의 어 닐 링에 대 한 보유 시간. 단계 사이 냉각 속도 0.1 ° C/min에서 설정 됩니다. DNA 종이 접기-AuNR 샘플 400 rpm에서 떨고 있는 동안 단련 됩니다.

그림 1 : DNA 종이 접기 템플릿 랄 metamolecules의 디자인. 골드 nanorods (AuNRs)의 원하는 상대 공간 배열 및 DNA 종이 접기 서식 파일의 적당 한 모양에는 식별을 (A) (B) AuNRs (DAuNR, LAuNR)와 종이 접기 템플릿 (W종이 접기, L종이 접기, Θ)의 구조 매개 변수 견적. 추가 수정이 필요 스테이플의 대략적인 위치를 찾습니다. (C) caDNAno를 사용 하 여 DNA 종이 접기 템플릿의 디자인. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.

그림 2 : 종이 접기의 agarose 젤 전기 이동 법. (A) 1 %agarose 젤 전기 이동 법 (B) 특성화 대 80에 2 h 4 h 80 대에 대 한 2 %agarose 젤 전기 이동 법을 가진 정화 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
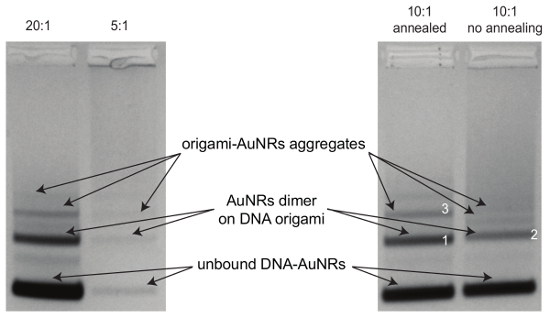
그림 3 : 종이 접기-AuNRs의 agarose 젤 전기 이동 법 정화. 젤 (0.7%) 3.5 h 80 V 다른 DNA-AuNR-에-종이 접기 비율 (20:1, 5:1)와 샘플 (DNA-AuNRs-에-종이 접기 비율이 10:1)와 함께/없이 절차를 어 닐 링 조립 절차에 따라 샘플 준비에서 실행 되었습니다. 밴드 1, 2 및 3에서에서 샘플의 TEM 이미지, 그림 6을 참조 하십시오. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.

그림 4 : DNA 종이 접기의 대표 TEM 이미지. 비 계 가닥에 의해 서로 연결 된 두 개의 14-나선 번들 (80 nm x 16 nm x 8 nm) 종이 접기 구조에 의하여 이루어져 있다. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.

그림 5 :는 AuNRs의 대표 TEM 이미지. 합성된 AuNRs의 평균 크기는 70 x 30 nm. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.

그림 6 : 종이 접기-AuNR 어셈블리의 TEM 이미지. (A) 종이 접기 ( 그림3에서 밴드 1) 어 닐 링 후에 AuNR 이합체. (B) 어 닐 링 (밴드 2 그림3에서) 없이 종이 접기에 AuNR 이합체. (C) 종이 접기-AuNR 집계 (밴드 3 그림3에서). 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
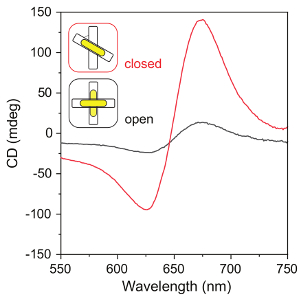
그림 7 : 종이 접기-AuNR 어셈블리의 CD 스펙트럼. 닫힌된 구조 (오른 손잡이 구성, 두 개의 종이 접기 번들 사이 50 °에 잠금 가닥 정한 종이 접기 템플릿)의 CD 스펙트럼 및 개방형 구조 (잠금 가닥 없이 종이 접기 템플릿). 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
토론
프로토콜 설계, 조립, 정화, 및 AuNRs의 종이 접기-기반 랄 어셈블리 DNA의 특성의 전체 워크플로 소개합니다. 프로토콜에 사용 되는 DNA 종이 접기 템플릿에 자극-반응 어셈블리의 제조에 특히 적합 합니다. 다양 한 유형의 응답 functionalizes와 종이 접기 템플릿 (그림 1B)24,25,,2631의 카이 랄 상태를 정의 하는 잠금 가닥으로 통합 될 수 있다. 정적 어셈블리에 대 한 간단한 블록 모양의 서식 파일은 종종 충분 한14,45,,4647.
DNA 종이 접기-기반 접근 plasmonic nanostructure의 제조에 DNA 종이 접기 기법48의 한계를 상속합니다. 종이 접기 서식 파일의 크기는 일반적으로 비 계 가닥의 크기에 의해 제한 됩니다. DNA 구조의 안정성 법-소금 조건 하에서 감소 된다. 합성 스테이플의 비용 보다 높은 남아 가닥. 그러나 구조 DNA 나노기술 분야에서 최근 개발이 제한49,50,51,,5253,54 극복 것으로 예상 된다 , 55.
AuNRs34,35,,3637의 카이 랄 어셈블리를 생성 하기 위한 다른 분자 기반 접근법에 비해 DNA 종이 접기는 공간 정밀도 및 프로그래밍의 높은 수준을 제공 합니다.
카이 랄 어셈블리의 안정적이 고 재현 가능한 광학 응답을 달성 하기 위한 품질 및 상용 제품의 광학 속성 일괄 처리 사이 다를 수 있습니다 이후 AuNR 합성40에 대 한 프로토콜을 적응 것이 좋습니다. 추가 어 닐 링 (단계 6.2)는 종종 DNA 종이 접기 템플릿 (그림 6) AuNRs의 올바른 첨부 파일을 보장 하기 위한 중요 한입니다.
마지막으로, 여기에 설명 된 프로토콜 랄 어셈블리에 국한 되지 않습니다. DNA 종이 접기는 복잡 한 plasmonic nanostructures9,10의 제조에 매우 유연한 플랫폼을 제공합니다.
공개
저자는 공개 없다.
감사의 말
저자는 CD 분석기와 그녀의 지원에 대 한 S. Voutilainen 감사합니다. 저자는 시설 및 OtaNano-Nanomicroscopy 센터 (Aalto-NMC)에서 Aalto 대학 기술 지원의 제공을 인정합니다. 이 작품 (그랜트 308992) 핀란드의 아카데미에 의해 지원 되었다 그리고 유럽 연합의 지평선 2020 연구와 혁신 프로그램 Marie Skłodowska-퀴리 아래 계약 번호 71364 부여.
자료
| Name | Company | Catalog Number | Comments |
| 2,6-Dihydroxybenzoic acid | Sigma-Aldrich | D109606-25 | 98+% |
| AgNO3 | Alfa Aesar | AA1141414 | 99.90% |
| Blue light transilluminator | Nippon Genetics | FG-06 | FastGene LED Transilluminator |
| Bromophenol Blue | Acros Organics | 403160050 | For agarorose gel loading buffer |
| Centrifugal filter units | Merck Millipore | 42600 | DNA extraction from agarose |
| Chirascan CD spectrometer | Applied Photophysics | ||
| Cuvette | Hellma | 105-202-85-40 | Quartz SUPRASIL |
| DNA lobind tubes | Eppendorf | 30108051 | |
| Eppendorf Biospectrometer | Eppendorf | 6135000904 | |
| Eppendorf ThermoMixer C | Eppendorf | 5382000015 | |
| Ficoll 400 | Thermo Fisher Scientific | BP525-10 | Polysucrose 400 (For agarorose gel loading buffer) |
| Gel electrophoresis sets | Thermo Fisher Scientific | ||
| Gel imager | Bio-Rad | Gel Doc XR+ System | |
| HAuCl4•3H2O | Alfa Aesar | AA3640006 | 99.99% |
| HCl | Scharlau | AC07441000 | 1M |
| Hexadecyltrimethylammonium bromide (CTAB) | Sigma-Aldrich | H9151-100 | BioXtra, 98+% |
| L(+)-ascorbic acid | Acros Organics | 401471000 | 99+% |
| M13p7560 scaffold strand | Tilibit nanosystems | ||
| MgCl2•6H2O | Sigma-Aldrich | M2670-500 | BioXtra, 99+% |
| NaBH4 | Acros Organics | 200050250 | 99% |
| NaCl | Sigma-Aldrich | S7653-500 | BioXtra, 99.5+% |
| NaOH | Sigma-Aldrich | S8045-500 | BioXtra, 98+% |
| Parafilm | Sigma-Aldrich | P7668-1EA | PARAFILM M |
| PBS buffer (10X) | Thermo Fisher Scientific | BP3991 | Molecular Biology |
| ProFlex PCR System | Thermo Fisher Scientific | 4484073 | |
| Sodium dodecyl sulfate (SDS) | Sigma-Aldrich | 74255-250 | 99+% |
| Staple strands | Thermo Fisher Scientific | ||
| Sybr Safe | Invitrogen | S33102 | For DNA stain |
| TBE buffer (10X) | Invitrogen | 15581-044 | Molecular Biology |
| TE buffer (10X) | Thermo Fisher Scientific | BP24771 | Molecular Biology |
| TEM | FEI | FEI Tecnai F12 | |
| Thiol-functionalized ssDNA | Biomers.net | ||
| Tris(2-carboxyethyl)phosphine hydrochloride (TCEP-HCl) | Thermo Fisher Scientific | PI20491 | |
| UltraPure Agarose | Invitrogen | 16500-100 | |
| Ultrapure water (Type 1) | Milli-Q Direct 8 system | ||
| Uranyl Formate | Tebu-bio | 24762-1 | |
| White light transilluminator | UVP | TW-26 |
참고문헌
- Rothemund, P. W. K. Folding DNA to create nanoscale shapes and patterns. Nature. 440, 297-302 (2006).
- Wang, P., Meyer, T. A., Pan, V., Dutta, P. K., Ke, Y. The Beauty and Utility of DNA Origami. Chem. 2, 359-382 (2017).
- Hong, F., Zhang, F., Liu, Y., Yan, H. DNA Origami: Scaffolds for Creating Higher Order Structures. Chemical Reviews. 117, 12584-12640 (2017).
- Roller, E. -M., Argyropoulos, C., Högele, A., Liedl, T., Pilo-Pais, M. Plasmon-Exciton Coupling Using DNA Templates. Nano Letters. 16, 5962-5966 (2016).
- Acuna, G. P., et al. Fluorescence Enhancement at Docking Sites of DNA-Directed Self-Assembled Nanoantennas. Science. 338, 506-510 (2012).
- Roller, E. -M., et al. DNA-Assembled Nanoparticle Rings Exhibit Electric and Magnetic Resonances at Visible Frequencies. Nano Letters. 15, 1368-1373 (2015).
- Liu, Q., Song, C., Wang, Z. -G., Li, N., Ding, B. Precise organization of metal nanoparticles on DNA origami template. Methods. 67, 205-214 (2014).
- Roller, E. -M., et al. Hotspot-mediated non-dissipative and ultrafast plasmon passage. Nature Physics. 13, 761-765 (2017).
- Liu, N., Liedl, T. DNA-Assembled Advanced Plasmonic Architectures. Chemical Reviews. 118, 3032-3053 (2018).
- Kuzyk, A., Jungmann, R., Acuna, G. P., Liu, N. DNA Origami Route for Nanophotonics. ACS Photonics. 5, 1151-1163 (2018).
- Kuzyk, A., et al. DNA-based self-assembly of chiral plasmonic nanostructures with tailored optical response. Nature. 483, 311-314 (2012).
- Shen, X., et al. Three-Dimensional Plasmonic Chiral Tetramers Assembled by DNA Origami. Nano Letters. 13, 2128-2133 (2013).
- Liu, H., Shen, X., Wang, Z. -G., Kuzyk, A., Ding, B. Helical nanostructures based on DNA self-assembly. Nanoscale. 6, 9331-9338 (2014).
- Shen, X., et al. 3D plasmonic chiral colloids. Nanoscale. 6, 2077-2081 (2014).
- Urban, M. J., et al. Plasmonic Toroidal Metamolecules Assembled by DNA Origami. Journal of the American Chemical Society. 138, 5495-5498 (2016).
- Hentschel, M., Schäferling, M., Duan, X., Giessen, H., Liu, N. Chiral plasmonics. Science Advances. 3, 1602735(2017).
- Cecconello, A., Besteiro, L. V., Govorov, A. O., Willner, I. Chiroplasmonic DNA-based nanostructures. Nature Reviews Materials. 2, 17039(2017).
- Lan, X., Wang, Q. Self-Assembly of Chiral Plasmonic Nanostructures. Advanced Materials. 28, 10499-10507 (2016).
- Shen, C., et al. Spiral Patterning of Au Nanoparticles on Au Nanorod Surface to Form Chiral AuNR@AuNP Helical Superstructures Templated by DNA Origami. Advanced Materials. 29, 1606533(2017).
- Zhou, C., Duan, X., Liu, N. A plasmonic nanorod that walks on DNA origami. Nature Communications. 6, 8102(2015).
- Douglas, S. M., et al. Rapid prototyping of 3D DNA-origami shapes with caDNAno. Nucleic Acids Research. 37, 5001-5006 (2009).
- Kim, D. -N., Kilchherr, F., Dietz, H., Bathe, M. Quantitative prediction of 3D solution shape and flexibility of nucleic acid nanostructures. Nucleic Acids Research. 40, 2862-2868 (2012).
- Maffeo, C., Yoo, J., Aksimentiev, A. De novo reconstruction of DNA origami structures through atomistic molecular dynamics simulation. Nucleic Acids Research. 44, 3013-3019 (2016).
- Kuzyk, A., et al. Reconfigurable 3D plasmonic metamolecules. Nature Materials. 13, 862-866 (2014).
- Kuzyk, A., et al. A light-driven three-dimensional plasmonic nanosystem that translates molecular motion into reversible chiroptical function. Nature Communications. 7, 10591(2016).
- Kuzyk, A., Urban, M. J., Idili, A., Ricci, F., Liu, N. Selective control of reconfigurable chiral plasmonic metamolecules. Science Advances. 3, 1602803(2017).
- Gerling, T., Wagenbauer, K. F., Neuner, A. M., Dietz, H. Dynamic DNA devices and assemblies formed by shape-complementary, non-base pairing 3D components. Science. 347, 1446-1452 (2015).
- Zhou, C., Duan, X., Liu, N. DNA-Nanotechnology-Enabled Chiral Plasmonics: From Static to Dynamic. Accounts of Chemical Research. 50, 2906-2914 (2017).
- Ijäs, H., et al. Dynamic DNA Origami Devices: from Strand-Displacement Reactions to External-Stimuli Responsive Systems. International Journal of Molecular Sciences. 19, 2114(2018).
- Lan, X., et al. DNA-Guided Plasmonic Helix with Switchable Chirality. Journal of the American Chemical Society. 140, 11763-11770 (2018).
- Funck, T., Nicoli, F., Kuzyk, A., Liedl, T. Sensing Picomolar Concentrations of RNA Using Switchable Plasmonic Chirality. Angewandte Chemie International Edition. 57, 13495-13498 (2018).
- Zhou, C., Xin, L., Duan, X., Urban, M. J., Liu, N. Dynamic Plasmonic System That Responds to Thermal and Aptamer-Target Regulations. Nano Letters. 18, 7395-7399 (2018).
- Zhan, P., et al. Reconfigurable Three-Dimensional Gold Nanorod Plasmonic Nanostructures Organized on DNA Origami Tripod. ACS Nano. 11, 1172-1179 (2017).
- Ma, W., et al. Attomolar DNA detection with chiral nanorod assemblies. Nature Communications. 4, 2689(2013).
- Ma, W., et al. Chiral plasmonics of self-assembled nanorod dimers. Scientific Reports. 3, 1934(2013).
- Guerrero-Martínez, A., et al. Intense Optical Activity from Three-Dimensional Chiral Ordering of Plasmonic Nanoantennas. Angewandte Chemie International Edition. 50, 5499-5503 (2011).
- Kumar, J., et al. Detection of amyloid fibrils in Parkinson's disease using plasmonic chirality. Proceedings of the National Academy of Sciences. 115, 3225-3230 (2018).
- Stahl, E., Martin, T. G., Praetorius, F., Dietz, H. Facile and Scalable Preparation of Pure and Dense DNA Origami Solutions. Angewandte Chemie International Edition. 53, 12735-12740 (2014).
- Shaw, A., Benson, E., Högberg, B. Purification of Functionalized DNA Origami Nanostructures. ACS Nano. 9, 4968-4975 (2015).
- Ye, X., et al. Improved Size-Tunable Synthesis of Monodisperse Gold Nanorods through the Use of Aromatic Additives. ACS Nano. 6, 2804-2817 (2012).
- Near, R. D., Hayden, S. C., Hunter, R. E., Thackston, D., El-Sayed, M. A. Rapid and Efficient Prediction of Optical Extinction Coefficients for Gold Nanospheres and Gold Nanorods. The Journal of Physical Chemistry C. 117, 23950-23955 (2013).
- Shi, D., Song, C., Jiang, Q., Wang, Z. -G., Ding, B. A facile and efficient method to modify gold nanorods with thiolated DNA at a low pH value. Chemical Communications. 49, 2533-2535 (2013).
- Gür, F. N., Schwarz, F. W., Ye, J., Diez, S., Schmidt, T. L. Toward Self-Assembled Plasmonic Devices: High-Yield Arrangement of Gold Nanoparticles on DNA Origami Templates. ACS Nano. 10, 5374-5382 (2016).
- Castro, C. E., et al. A primer to scaffolded DNA origami. Nature Methods. 8, 221-229 (2011).
- Lan, X., et al. Bifacial DNA Origami-Directed Discrete, Three-Dimensional, Anisotropic Plasmonic Nanoarchitectures with Tailored Optical Chirality. Journal of the American Chemical Society. 135, 11441-11444 (2013).
- Lan, X., et al. Au Nanorod Helical Superstructures with Designed Chirality. Journal of the American Chemical Society. 137, 457-462 (2015).
- Zhu, C., Wang, M., Dong, J., Zhou, C., Wang, Q. Modular Assembly of Plasmonic Nanoparticles Assisted by DNA Origami. Langmuir. , (2018).
- Pinheiro, A. V., Han, D., Shih, W. M., Yan, H. Challenges and opportunities for structural DNA nanotechnology. Nature Nanotechnology. 6, 763-772 (2011).
- Ducani, C., Kaul, C., Moche, M., Shih, W. M., Högberg, B. Enzymatic production of 'monoclonal stoichiometric' single-stranded DNA oligonucleotides. Nature Methods. 10, 647-652 (2013).
- Praetorius, F., et al. Biotechnological mass production of DNA origami. Nature. 552, 84-87 (2017).
- Wagenbauer, K. F., Sigl, C., Dietz, H. Gigadalton-scale shape-programmable DNA assemblies. Nature. 552, 78-83 (2017).
- Zhang, T., et al. 3D DNA Origami Crystals. Advanced Materials. 30, 1800273(2018).
- Ong, L. L., et al. Programmable self-assembly of three-dimensional nanostructures from 10,000 unique components. Nature. 552, 72-77 (2017).
- Ponnuswamy, N., et al. Oligolysine-based coating protects DNA nanostructures from low-salt denaturation and nuclease degradation. Nature Communications. 8, 15654(2017).
- Agarwal, N. P., Matthies, M., Gür, F. N., Osada, K., Schmidt, T. L. Block Copolymer Micellization as a Protection Strategy for DNA Origami. Angewandte Chemie International Edition. 56, 5460-5464 (2017).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유