Method Article
通过化学交联质谱法进行四元结构建模:扩展 TX-MS Jupyter 报告
摘要
靶向交联质谱使用使用多达三种不同采集方案获取的质谱数据创建季铵盐蛋白质结构模型。当在Cheetah-MS Web服务器上作为简化的工作流程执行时,结果将在Jupyter Notebook中报告。在这里,我们演示了如何扩展Jupyter Notebook以进行更深入分析的技术方面。
摘要
蛋白质 - 蛋白质相互作用可能具有挑战性,但可以提供对生物系统如何运作的见解。靶向交联质谱(TX-MS)是一种结合四元蛋白质结构建模和化学交联质谱的方法,使用从复杂,不均匀的样品中获得的数据创建高精度结构模型。这消除了蛋白质复合物结构分析的主要障碍之一,因为感兴趣的蛋白质不再需要大量纯化。猎豹-MS网络服务器的开发是为了使社区的协议的简化版本更容易访问。考虑到串联的MS/MS数据,Cheetah-MS生成了一个Jupyter Notebook,这是一份图形报告,总结了最重要的分析结果。扩展Jupyter Notebook可以产生更深入的见解,并更好地了解模型和支持它的质谱数据。这里介绍的技术协议演示了一些最常见的扩展,并解释了可以获得哪些信息。它包含的模块有助于分析串联MS / MS采集数据以及检测到的XL对报告的四元模型的总体影响。此类分析的结果可以应用于使用 NGLView 嵌入在笔记本中的结构模型。
引言
蛋白质-蛋白质相互作用是生物系统结构和功能的基础。获得蛋白质的四级结构可以提供关于两种或多种蛋白质如何相互作用形成高阶结构的见解。不幸的是,获得第四纪结构仍然具有挑战性。这反映在相对较少数量的蛋白质数据库(PDB)条目1中,其中包含一种以上的多肽。蛋白质 - 蛋白质相互作用可以通过X射线晶体学,NMR和冷冻电镜等技术进行研究,但是在可以应用这些方法的条件下获得足够量的纯化蛋白质可能非常耗时。
开发了化学交联质谱法,以获得蛋白质 - 蛋白质相互作用的实验数据,同时减少对样品制备的限制,因为质谱法可用于获取任意复杂样品的数据2,3,4,5,6,7,8,9.然而,数据分析的组合性质和相对较少的交联肽要求在分析之前对样品进行分馏。为了解决这一缺点,我们开发了TX-MS,这是一种将计算建模与化学交联质谱10相结合的方法。TX-MS可用于任意复杂的样品,并且与以前的方法10相比,灵敏度要高得多。它通过将与给定蛋白质 - 蛋白质相互作用相关的所有数据作为一个集合进行评分来实现这一点,而不是独立解释每个MS谱。TX-MS还使用多达三种不同的MS采集协议:高分辨率MS1(hrMS1),数据依赖采集(DDA)和数据独立采集(DIA),进一步提供了通过结合多个观察结果来鉴定交联肽的机会。TX-MS计算工作流程很复杂,原因有几个。首先,它依靠多个MS分析软件程序11、12、13来创建蛋白质结构模型14、15。其次,数据量可能相当可观。第三,建模步骤会消耗大量的计算机处理能力。
因此,TX-MS最好通过Cheetah-MS Web服务器16用作自动化,简化的计算工作流程,该服务器在大型计算基础设施(如计算机云或集群)上运行。为了便于解释结果,我们制作了一个交互式Jupyter Notebook17。在这里,我们演示了如何扩展Jupyter Notebook报告,以对给定结果进行更深入的分析。
研究方案
1. 在 https://txms.org 提交工作流。
- 转到 https://txms.org,然后单击"使用Cheetah-MS"。
- 若要提交工作流,需要提供两个 PDB 文件和一个 MS/MS mzML 或 MGF 文件。您还可以单击"加载示例数据"以查看工作流的演示版本。
注意:请查看 Web 服务器的手册页,了解有关如何提交作业的详细信息。Web服务器支持不同的不可切割交联器代理,多达12个翻译后修改(PTM),与计算建模和MS数据分析相关的选项。提交页面上还设计了小帮助按钮,以显示有关每个选项的更多信息。
2. 运行猎豹-MS。
注意:使用 ProteoWizard MSConvert 软件将供应商特定的格式转换为 mzML 或 MGF19。
- 将 MS 数据上传到 https://txms.org。然后,单击"选择文件"并选择MS数据,其必须采用mzML / MGF数据格式18。
注:示例数据可在 https://txms.org 上找到。这些数据也可以通过 zenodo.org DOI 10.5281/zenodo.3361621 直接访问。 - 将两个 PDB 文件上载到 https://txms.org。单击"选择文件",然后选择要上传的PDB文件。
注意:如果不存在实验结构,请使用例如SWISS-MODEL20(如果同系结构可用)或trRosetta21,22或Robetta23,24 Web服务器进行 从头 结构预测来创建模型。 - 提交新的工作流。单击"提交"以接收作业标识符标签。然后,使用此标记按照表单转到结果部分。
注意:计算结果需要时间,因此请等到工作流完成,并存储作业标识符标记以返回到结果页面。计算在远程计算基础设施上进行。如果要在本地运行 TX-MS,请参阅 Hauri et al.10。 - 使用联机查看器检查 Jupyter Notebook 报告。然后,使用作业标识符标记向下滚动到结果部分中的"报告"。
3. 安装 JupyterHub。
- 按照 https://docs.docker.com/install/ 中的说明安装 docker。
- 下载带有Jupyter openBIS25扩展的JupyterHub docker容器。一般命令是"docker pull malmstroem/jove:latest",但在其他平台上可能会有所不同。
注意:有关如何下载容器的一般讨论,请参阅 https://www.docker.com/get-started。也可以从 zenodo.org DOI 10.5281/zenodo.3361621下载容器。
注意:Jupyter openBIS 扩展源代码可从以下站点获得:https://pypi.org/project/jupyter-openbis-extension/。 - 启动 docker 容器:docker run -p 8178:8000 malmstroem/jove:latest。
注意:JupyterHub 默认使用的端口为 8000。此端口是可配置的,如果更改,则需要相应地调整上述命令。端口 8178 是任意选择,可以更改。下面提供的示例 URL 需要相应地进行调整。 - 转到以下地址:http://127.0.0.1:8178。使用用户名"user"和密码"user"登录。
注意:地址 http://127.0.0.1 表示 docker 容器正在本地计算机上运行。如果 docker 容器在服务器上运行,请使用服务器的 IP 地址或 URL(例如,https://example.com)。Docker容器基于Ubuntu Bionic 18.04,JupyterHub 0.9.6和Jupyter openBIS扩展0.2。可以在其他操作系统中安装它,但尚未对其进行测试。
4. 下载报告。
- 通过单击"新建"来创建新笔记本 |Python 3 使用位于页面右上角附近的菜单。这将打开一个新选项卡,其中包含一个名为 "无标题 "(或类似内容)的笔记本。
- 单击 Jupyter 工具菜单中的"配置 openBIS 连接"。
- 填写名称:txms;网址: https://txms.org;用户:客人;密码:访客通行。
- 点击"连接"。
- 选择新连接,然后单击"选择连接"。
- 搜索报告模板(例如,/CHEETAH/WF70),然后单击 下载。
注意:您需要根据在Cheetah-MS Web服务器上运行作业获得的结果和报告来调整报告模板。 - 通过单击单元格|重新运行报表 全部运行。
5. 扩展报表。
- 在底部添加新单元格: 单元格|在下面插入。
- 键入所需的代码。有关示例,请参阅下面的"代表性结果"部分。
- 通过按"Shift-Enter"来执行单元格。
结果
TX-MS 提供由 MS 派生的实验约束支持的结构输出。它的工作原理是将不同的MS数据采集类型与计算建模相结合。因此,单独解析每个MS数据并提供输出结构的可视化是有帮助的。 补充数据 1 包含一个示例笔记本,可以解析作为 TX-MS 输出生成的 DDA 和 DIA 数据。用户可以选择感兴趣的XL。通过运行笔记本,将显示该XL的MS2光谱,其中不同的颜色有助于区分与第一肽,第二肽和组合片段离子相关的片段。XL还可以使用嵌入在Jupyter Notebook中的NGLView小部件映射到结构。
此笔记本中的另一个单元格可以帮助用户分析和可视化 DIA 数据。但是,可视化 DIA 数据更加困难,因为分析的数据需要以正确的格式进行准备。
图1 显示了M1和白蛋白的示例结构,其顶部XL映射在该结构上。TX-MS在解析hrMS1,DDA和DIA数据后获得了所有XL,RosettaDock协议提供了计算模型。
由于此报表是 Jupyter 笔记本,因此可以将任何有效的 Python 代码添加到新的笔记本单元格中。例如,下面的代码将创建一个基于 MS2 计数的直方图,指示基础数据对每个交叉链接的支持程度。
进口海生生物作为sns
sns.distplot(ms2['count']);
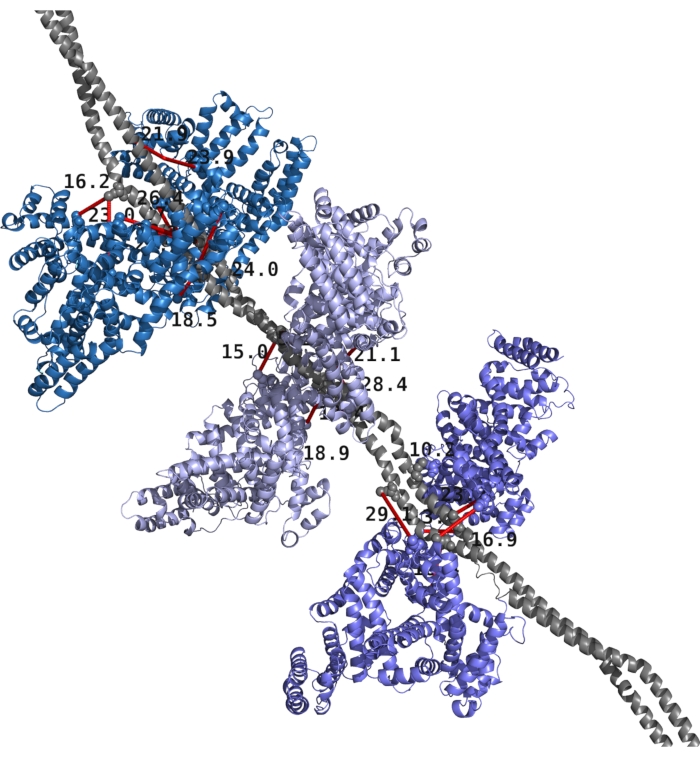
图1: 化脓性链球菌 M1 蛋白和人白蛋白的结构模型,结构上映射了XL。 M1蛋白以灰色显示,构成同源二聚体。六种白蛋白分子以各种深浅不一的蓝色成对呈现。交叉链接和距离以红色和黑色文本给出。 请点击此处查看此图的大图。
补充文件。Jupyter notebook data.请点击此处下载此文件。
讨论
现代计算工作流程通常很复杂,具有来自许多不同供应商的多个工具,复杂的相互依赖性,高数据量和多方面的结果。因此,越来越难以准确记录获得结果所需的所有步骤,因此难以重现给定的结果。在这里,我们将演示一种通用策略,该策略将生成通用报表的自动化工作流的自动化和易用性与以可重现的方式自定义报表的灵活性相结合。
方案需要满足三个要求才能发挥作用:首先,选择用于分析的蛋白质需要以这样的方式相互作用,即化学交联实验能够以足够高的浓度产生交联物质,以便由质谱仪检测;不同的质谱仪具有不同的检测水平,并且还取决于采集方案以及交联试剂的选择。当前版本的TX-MS方案仅允许DSS,一种赖氨酸 - 赖氨酸同源双官能交联试剂。尽管如此,这种限制主要是由于机器学习步骤可能需要针对其他试剂进行调整。这种限制在Cheetah-MS网络服务器中得到了改善,因为可以考虑另外两种交联试剂,但所有三种都是不可切割的试剂。其次,这两种蛋白质需要具有实验确定的结构,或者使用比较建模技术或 从头 技术进行建模。并非所有蛋白质都可以建模,但是改进的软件和PDB中实验结构的恒定沉积的组合扩展了可以建模的蛋白质数量。第三,相互作用的蛋白质应该在它们的结合和未结合状态下保持足够相似,以便TX-MS和Cheetah-MS使用的对接算法可以创建足够质量的四元结构来实现评分。这一要求相对模糊,因为可接受的质量是高度系统依赖性的,其中已知结构的较小蛋白质通常比未知结构的大型蛋白质更容易比较。
如果结果为阴性,首先检查TX-MS是否发现了作为同一多肽链一部分的残基之间的内链,交联。如果没有发现,最可能的解释是样品制备或数据采集出现问题。如果多个距离约束不支持模型,请目视检查模型以确保构象由交联残差支持。没有明显的方法可以在不中断至少一个交联的情况下使其中一个交互者枢轴。如果交联的交联时间长于给定交联试剂的允许距离,请尝试通过合并交联数据来改进相互作用子的建模。
可以使用替代软件应用程序来完成等效的结果,前提是所选软件的灵敏度与TX-MS的灵敏度相当。例如,有RosettaDock,HADDOCK等的在线版本。还可以通过xQuest/xProphet5,6,plink7和SIM-XL26分析化学交联数据。
我们不断将TX-MS和Cheetah-MS应用于新项目27,28,29,从而改进了这些方法生成的报告,以便在不扩大报告的情况下对结果进行更详细的分析。
披露声明
作者没有什么可透露的。
致谢
这项工作得到了Knut和Alice Wallenberg基金会(拨款编号:2016.0023)和瑞士国家科学基金会(拨款编号:2016.0023)的支持。P2ZHP3_191289)。此外,我们感谢苏黎世大学S3IT的计算基础设施和技术支持。
材料
| Name | Company | Catalog Number | Comments |
| Two Protein DataBank files of the proteins of interest. | N/A | N/A | Example files available on txms.org and zenodo.org, DOI 10.5281/zenodo.3361621 |
| An mzML data file acquired on a sample where the proteins of interest were crosslinked. | N/A | N/A | Example files available on txms.org or zenodo.org, DOI 10.5281/zenodo.3361621 |
参考文献
- Berman, H. M., et al. The Protein Data Bank. Acta Crystallographica Section D: Biological Crystallography. 58 (6), 899-907 (2002).
- Herzog, F., et al. Structural Probing of a Protein Phosphatase 2A Network by Chemical Cross-Linking and Mass Spectrometry. Science. 337 (6100), 1348-1352 (2012).
- Hoopmann, M. R., et al. Kojak: efficient analysis of chemically cross-linked protein complexes. Journal of Proteome Research. 14 (5), 2190-2198 (2015).
- Seebacher, J., et al. Protein cross-linking analysis using mass spectrometry, isotope-coded cross-linkers, and integrated computational data processing. Journal of Proteome Research. 5 (9), 2270-2282 (2006).
- Rinner, O., et al. Identification of cross-linked peptides from large sequence databases. Nature Methods. 5 (4), 315-318 (2008).
- Walzthoeni, T., et al. False discovery rate estimation for cross-linked peptides identified by mass spectrometry. Nature Methods. 9 (9), 901-903 (2012).
- Yang, B., et al. Identification of cross-linked peptides from complex samples. Nature Methods. 9 (9), 904-906 (2012).
- Chu, F., Baker, P. R., Burlingame, A. L., Chalkley, R. J. Finding Chimeras: a Bioinformatics Strategy for Identification of Cross-linked Peptides. Molecular & Cellular Proteomics. 9 (1), 25-31 (2010).
- Holding, A. N., Lamers, M. H., Stephens, E., Skehel, J. M. Hekate: Software Suite for the Mass Spectrometric Analysis and Three-Dimensional Visualization of Cross-Linked Protein Samples. Journal of Proteome Research. 12 (12), 5923-5933 (2013).
- Hauri, S., et al. Rapid determination of quaternary protein structures in complex biological samples. Nature Communications. 10 (1), 192 (2019).
- Röst, H. L., et al. OpenSWATH enables automated, targeted analysis of data-independent acquisition MS data. Nature Biotechnology. 32 (3), 219-223 (2014).
- Röst, H. L., et al. OpenMS: a flexible open-source software platform for mass spectrometry data analysis. Nature Methods. 13 (9), 741-748 (2016).
- Quandt, A., et al. Using synthetic peptides to benchmark peptide identification software and search parameters for MS/MS data analysis. EuPA Open Proteomics. 5, 21-31 (2014).
- Bradley, P., et al. Free modeling with Rosetta in CASP6. Proteins: Structure, Function, and Bioinformatics. 61 (S7), 128-134 (2005).
- Gray, J. J. High-resolution protein-protein docking. Current Opinion in Structural Biology. 16 (2), 183-193 (2006).
- Khakzad, H., et al. Cheetah-MS: a web server to model protein complexes using tandem cross-linking mass spectrometry data. Bioinformatics. , (2021).
- Malmström, L. Chapter 15: Computational Proteomics with Jupyter and Python. Methods in Molecular Biology. 15, 237-248 (1977).
- Martens, L., et al. mzML--a community standard for mass spectrometry data. Molecular & Cellular Proteomics. 10 (1), (2011).
- Chambers, M. C., et al. A cross-platform toolkit for mass spectrometry and proteomics. Nature Biotechnology. 30 (10), 918-920 (2012).
- Waterhouse, A., et al. SWISS-MODEL: homology modelling of protein structures and complexes. Nucleic Acids Research. 46 (W1), W296-W303 (2018).
- Yang, J., et al. Improved protein structure prediction using predicted interresidue orientations. Proceedings of the National Academy of Sciences. 117 (3), 1496-1503 (2020).
- Koehler Leman, J., et al. Macromolecular modeling and design in Rosetta: recent methods and frameworks. Nature Methods. 17 (7), 665-680 (2020).
- Chivian, D., et al. Prediction of CASP6 structures using automated Robetta protocols. Proteins: Structure, Function, and Bioinformatics. 61 (S7), 157-166 (2005).
- Chivian, D., et al. Automated prediction of CASP-5 structures using the Robetta server. Proteins: Structure, Function, and Bioinformatics. 53 (S6), 524-533 (2003).
- Bauch, A., et al. openBIS: a flexible framework for managing and analyzing complex data in biology research. BMC Bioinformatics. 12, 468 (2011).
- Lima, D. B., et al. SIM-XL: A powerful and user-friendly tool for peptide cross-linking analysis. Journal of Proteomics. 129, 51-55 (2015).
- Happonen, L., et al. A quantitative Streptococcus pyogenes-human protein-protein interaction map reveals localization of opsonizing antibodies. Nature Communications. 10, 2727 (2019).
- Khakzad, H., et al. Structural determination of Streptococcus pyogenes M1 protein interactions with human immunoglobulin G using integrative structural biology. PLOS Computational Biology. 17 (1), E1008169 (2021).
- Khakzad, H., et al. In vivo cross-linking MS of the complement system MAC assembled on live Gram-positive bacteria. Frontiers in Genetics. 11, (2020).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。