Method Article
Kimyasal Çapraz Bağlama Kütle Spektrometrisi ile Kuaterner Yapı Modellemesi: TX-MS Jupyter Raporlarının Genişletilmesi
Bu Makalede
Özet
Hedeflenen çapraz bağlama kütle spektrometresi, üç farklı edinme protokolü kullanılarak elde edilen kütle spektrometresi verilerini kullanarak kuaterner protein yapı modelleri oluşturur. Cheetah-MS web sunucusunda basitleştirilmiş bir iş akışı olarak yürütüldüğünde, sonuçlar bir Jupyter Not Defterinde raporlanır. Burada, Jupyter Notebook daha derinlemesine bir analiz için nasıl genişletilebileceğinin teknik yönlerini gösteriyoruz.
Özet
Protein-protein etkileşimlerini incelemek zor olabilir, ancak biyolojik sistemlerin nasıl çalıştığına dair içgörüler sağlar. Kuaterner protein yapı modellemesini ve kimyasal çapraz bağlamalı kütle spektrometrisini birleştiren bir yöntem olan hedefli çapraz bağlama kütle spektrometresi (TX-MS), karmaşık, fraksiyone edilmemiş numunelerden elde edilen verileri kullanarak yüksek hassasiyetli yapı modelleri oluşturur. Bu, protein kompleks yapı analizinin önündeki en büyük engellerden birini ortadan kaldırır, çünkü ilgilenilen proteinlerin artık büyük miktarlarda saflaştırılmasına gerek yoktur. Cheetah-MS web sunucusu, protokolün basitleştirilmiş sürümünü topluluk için daha erişilebilir hale getirmek için geliştirilmiştir. Tandem MS / MS verilerini göz önünde bulundurarak, Cheetah-MS, en önemli analiz sonuçlarını özetleyen grafiksel bir rapor olan bir Jupyter Notebook oluşturur. Jupyter Dizüstü Bilgisayarını genişletmek, daha derinlemesine içgörüler sağlayabilir ve modeli ve onu destekleyen kütle spektrometresi verilerini daha iyi anlayabilir. Burada sunulan teknik protokol, en yaygın uzantılardan bazılarını gösterir ve hangi bilgilerin elde edilebileceğini açıklar. Tandem MS / MS toplama verilerini ve tespit edilen XL'lerin bildirilen kuaterner modeller üzerindeki genel etkisini analiz etmeye yardımcı olacak bloklar içerir. Bu tür analizlerin sonucu, NGLView kullanılarak dizüstü bilgisayara gömülü yapısal modellere uygulanabilir.
Giriş
Protein-protein etkileşimleri, biyolojik sistemlerin yapısını ve işlevini destekler. Proteinlerin kuaterner yapılarına erişebilmek, iki veya daha fazla proteinin yüksek dereceli yapılar oluşturmak için nasıl etkileşime girdiğine dair fikir verebilir. Ne yazık ki, kuaterner yapıların elde edilmesi zor olmaya devam etmektedir; bu, birden fazla polipeptit içeren nispeten az sayıda Protein DataBank (PDB) girişi1'e yansır. Protein-protein etkileşimleri, X-ışını kristalografisi, NMR ve kriyo-EM gibi teknolojilerle incelenebilir, ancak yöntemlerin uygulanabileceği koşullar altında yeterli miktarda saflaştırılmış protein elde etmek zaman alıcı olabilir.
Kimyasal çapraz bağlama kütle spektrometrisi, protein-protein etkileşimleri hakkında deneysel veriler elde etmek için geliştirilmiştir ve kütle spektrometresi keyfi olarak karmaşık numuneler hakkında veri elde etmek için kullanılabilir 2,3,4,5,6,7,8,9 . Bununla birlikte, veri analizinin kombinatoryal doğası ve nispeten az sayıda çapraz bağlı peptit, numunelerin analizden önce parçalanmasını gerektirir. Bu eksikliği gidermek için, hesaplamalı modellemeyi kimyasal çapraz bağlama kütle spektrometresi10 ile birleştiren bir yöntem olan TX-MS'yi geliştirdik. TX-MS, keyfi olarak karmaşık numunelerde kullanılabilir ve önceki yöntemlere kıyasla önemli ölçüde daha hassastır10. Bunu, her MS spektrumunu bağımsız olarak yorumlamak yerine, belirli bir protein-protein etkileşimi ile ilişkili tüm verileri bir set olarak puanlayarak gerçekleştirir. TX-MS ayrıca üç farklı MS edinme protokolü kullanır: yüksek çözünürlüklü MS1 (hrMS1), veriye bağımlı edinme (DDA) ve veriden bağımsız edinme (DIA), birden fazla gözlemi birleştirerek çapraz bağlı bir peptidi tanımlamak için fırsatlar sunar. TX-MS hesaplamalı iş akışı çeşitli nedenlerden dolayı karmaşıktır. İlk olarak, protein yapı modelleri14,15 oluşturmak için birden fazla MS analiz yazılımı programı 11,12,13'e dayanır. İkincisi, veri miktarı önemli olabilir. Üçüncüsü, modelleme adımı önemli miktarda bilgisayar işlem gücü tüketebilir.
Sonuç olarak, TX-MS en iyi şekilde, bilgisayar bulutları veya kümeleri gibi büyük hesaplama altyapılarında çalışan Cheetah-MS web sunucusu16 aracılığıyla otomatik, basitleştirilmiş bir hesaplama iş akışı olarak kullanılır. Sonuçların yorumlanmasını kolaylaştırmak için interaktif bir Jupyter Notebook17 hazırladık. Burada, Jupyter Notebook raporunun, belirli bir sonucun daha derinlemesine bir analizini sağlamak için nasıl genişletilebileceğini gösteriyoruz.
Protokol
1. İş akışını https://txms.org'de gönderin.
- https://txms.org gidin ve "Cheetah-MS Kullan" ı tıklayın.
- İş akışı göndermek için iki PDB dosyası ve bir MS/MS mzML veya MGF dosyası sağlamanız gerekir. İş akışının demo sürümünü görmek için "örnek verileri yükle" ye de tıklayabilirsiniz.
NOT: Bir işin nasıl gönderileceği hakkında ayrıntılı bilgi için lütfen web sunucusunun kılavuz sayfasına bakın. Web sunucusu, farklı bölünemeyen çapraz bağlayıcı aracıları, 12 adede kadar çeviri sonrası modifikasyonu (PTM), hesaplamalı modelleme ve MS veri analizi ile ilgili seçenekleri destekler. Her seçenekle ilgili daha fazla bilgi göstermek için gönderme sayfasında küçük yardım düğmeleri de tasarlanmıştır.
2. Çita-MS'i çalıştırın.
NOT: ProteoWizard MSConvert yazılımı19'u kullanarak satıcıya özgü formatları mzML veya MGF'ye dönüştürün.
- MS verilerini https://txms.org'e yükleyin. Ardından, "Dosya seç" e tıklayın ve mzML / MGF veri formatlarında olması gereken MS verilerini seçin18.
NOT: Örnek veriler https://txms.org'da mevcuttur. Bu verilere zenodo.org, DOI 10.5281/zenodo.3361621 adresinden de doğrudan erişilebilir. - https://txms.org iki PDB dosyası yükleyin. "Dosya seç" e tıklayın ve yüklenecek PDB dosyalarını seçin.
NOT: Deneysel yapılar yoksa, örneğin homolog yapılar mevcutsa SWISS-MODEL 20 veya de novo yapı tahminleri için trRosetta21,22 veya Robetta23,24 web sunucularını kullanarak modeller oluşturun. - Yeni bir iş akışı gönderin. İş tanımlayıcı etiketi almak için "Gönder"e tıklayın. Ardından, bu etiketi kullanarak sonuçlar bölümüne giden formu izleyin.
NOT: Sonucun hesaplanması zaman alır, bu nedenle lütfen iş akışı bitene kadar bekleyin ve sonuçlar sayfasına dönmek için iş tanımlayıcı etiketini saklayın. Hesaplama, uzak hesaplama altyapısı üzerinde gerçekleştirilir. TX-MS'i yerel olarak çalıştırmak istiyorsanız, lütfen Hauri et al.10'a bakın. - Çevrimiçi görüntüleyiciyi kullanarak Jupyter Notebook raporunu inceleyin. Ardından, iş tanımlayıcı etiketini kullanarak sonuçlar bölümünde "Raporla"ya ilerleyin.
3. JupyterHub'ı yükleyin.
- Docker'ı https://docs.docker.com/install/'de belirtildiği gibi yükleyin.
- Jupyter openBIS25 uzantısına sahip JupyterHub docker kapsayıcısını indirin. Genel komut "docker pull malmstroem/jove:latest" şeklindedir, ancak diğer platformlarda farklılık gösterebilir.
NOT: Kapsayıcıların nasıl indirileceği hakkında genel bir tartışma için lütfen https://www.docker.com/get-started bakın. Konteyneri zenodo.org, DOI 10.5281/zenodo.3361621 adresinden indirmek de mümkündür.
NOT: Jupyter openBIS uzantısı kaynak kodu burada mevcuttur: https://pypi.org/project/jupyter-openbis-extension/. - docker kapsayıcısını başlatın: docker run -p 8178:8000 malmstroem/jove:sontest.
NOT: JupyterHub'ın varsayılan olarak kullandığı bağlantı noktası 8000'dir. Bu bağlantı noktası yapılandırılabilir ve değiştirilirse yukarıdaki komutların buna göre ayarlanması gerekir. 8178 numaralı bağlantı noktası isteğe bağlı bir seçimdir ve değiştirilebilir. Aşağıda verilen örnek URL'lerin buna göre ayarlanması gerekir. - Şu adrese gidin: http://127.0.0.1:8178. "user" kullanıcı adını ve "user" şifresini kullanarak oturum açın.
NOT: Adres http://127.0.0.1, docker kapsayıcısının yerel bilgisayarda çalıştığını gösterir. docker kapsayıcısı bir sunucuda çalıştırılıyorsa, sunucunun IP adresini veya URL'sini kullanın (ör. https://example.com). Docker kapsayıcısı Ubuntu Bionic 18.04, JupyterHub 0.9.6 ve Jupyter openBIS uzantısı 0.2'yi temel alır. Bunu diğer işletim sistemlerine yüklemek mümkündür, ancak bu test edilmemiştir.
4. Raporu indirin.
- Yeni'yi tıklatarak yeni bir not defteri oluşturun | Python 3, sayfanın sağ üst kısmına yakın bir yerde bulunan menüyü kullanarak. Bu, Adsız (veya benzer bir şey) adlı bir not defterine sahip yeni bir sekme açar.
- Jupyter araç menüsünde "openBIS Bağlantılarını Yapılandır" ı tıklayın.
- Adı girin: txms; URL: https://txms.org; kullanıcı: misafir; şifre: guestpasswd.
- "Bağlan"ı tıklayın.
- Yeni bağlantıyı seçin ve "Bağlantı Seç" i tıklayın.
- Rapor şablonunu arayın (örneğin, /CHEETAH/WF70) ve İndir'i tıklatın.
NOT: Rapor şablonunu, Cheetah-MS web sunucusunda işinizi çalıştırarak elde ettiğiniz sonuçlara ve rapora göre ayarlamanız gerekir. - Hücre |'nı tıklatarak raporu yeniden çalıştırın Tümünü Çalıştır.
5. Raporu genişletin.
- Alt kısımda yeni bir hücre ekleme: Hücre | Aşağıya Ekleyin.
- İstediğiniz kodu yazın. Bir örnek için lütfen aşağıdaki Temsili Sonuçlar bölümüne bakın.
- "Shift-Enter" tuşlarına basarak hücreyi çalıştırın.
Sonuçlar
XT-MS, MS türevi deneysel kısıtlamalarla desteklenen yapısal çıktılar sağlar. Farklı MS veri toplama türlerini hesaplamalı modelleme ile birleştirerek çalışır. Bu nedenle, her MS verisini ayrı ayrı ayrıştırmak ve çıktı yapısının görselleştirilmesini sağlamak yararlıdır. Ek Veri 1, TX-MS çıktısı olarak üretilen DDA ve DIA verilerini ayrıştırabilen örnek bir not defteri içerir. Kullanıcılar ilgilendikleri XL'yi seçebilirler. Not defterini çalıştırarak, bu XL'nin MS2 spektrumu, farklı renklerin birinci peptit, ikinci peptit ve kombinatoryal fragman iyonları ile ilgili parçalar arasında ayrım yapmaya yardımcı olduğu yerlerde gösterilecektir. XL, Jupyter Notebook'a katıştırılmış NGLView widget'ı kullanılarak da yapıya eşlenebilir.
Bu not defterindeki başka bir hücre, kullanıcıların DIA verilerini ayrıştırmasına ve görselleştirmesine yardımcı olabilir. Ancak analiz edilen verilerin doğru formatta hazırlanması gerektiğinden DIA verilerinin görselleştirilmesi daha zordur.
Şekil 1 , M1 ve albüminin örnek bir yapısını, yapı üzerinde üst XL'lerin eşlendiği şekilde göstermektedir. TX-MS, hrMS1, DDA ve DIA verilerini ayrıştırdıktan sonra tüm XL'leri elde etti ve RosettaDock protokolü hesaplama modellerini sağladı.
Bu rapor bir Jupyter Not Defteri olduğundan, herhangi bir geçerli Python kodu yeni not defteri hücrelerine eklenebilir. Örneğin, aşağıdaki kod, MS2 sayımları üzerinden, her çapraz bağlantının temel alınan veriler tarafından ne kadar iyi desteklendiğini gösteren bir histogram oluşturur.
sns olarak denizden ithal
sns.distplot(ms2['sayım']);
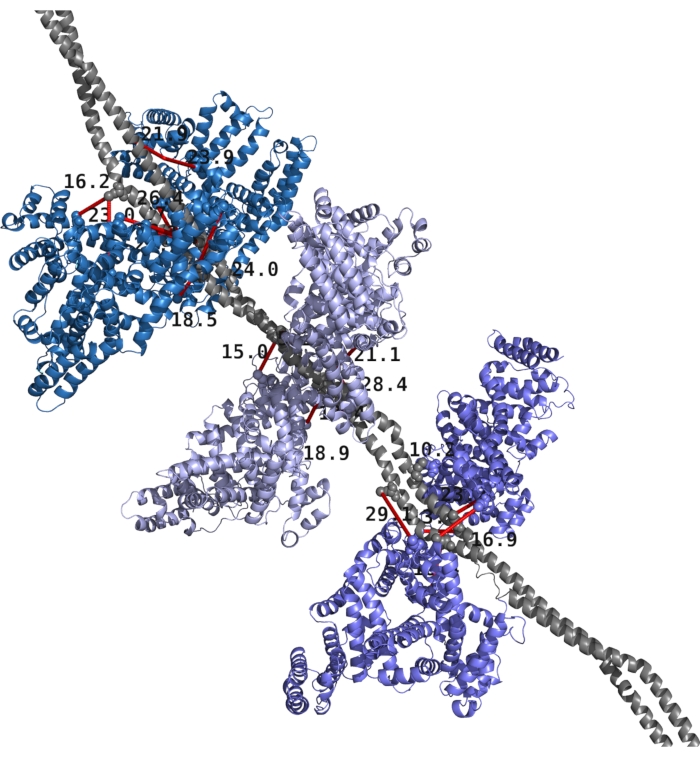
Şekil 1: Streptococcus pyogenes M1 proteininin yapısal modeli ve XL'li insan albümini yapı üzerinde haritalandırılmıştır. M1 proteini gri renkte gösterilir ve bir homodimer oluşturur. Altı albümin molekülü, mavinin çeşitli tonlarında çiftler halinde sunulur. Çapraz bağlantılar ve mesafeler siyah metinle kırmızı renkte verilir. Bu şeklin daha büyük bir versiyonunu görüntülemek için lütfen buraya tıklayın.
Ek Dosya. Jupyter not defteri verileri. Bu Dosyayı indirmek için lütfen tıklayınız.
Tartışmalar
Modern hesaplamalı iş akışları, birçok farklı satıcıdan birden çok araç, karmaşık karşılıklı bağımlılıklar, yüksek veri hacimleri ve çok yönlü sonuçlarla genellikle karmaşıktır. Sonuç olarak, bir sonuç elde etmek için gereken tüm adımları doğru bir şekilde belgelemek giderek zorlaşmakta ve verilen sonucun çoğaltılmasını zorlaştırmaktadır. Burada, genel bir rapor üreten otomatik bir iş akışının otomasyonunu ve kolaylığını, raporu yeniden üretilebilir bir şekilde özelleştirme esnekliğiyle birleştiren genel bir strateji gösteriyoruz.
Protokolün çalışması için üç gereksinimin yerine getirilmesi gerekir: Birincisi, analiz için seçilen proteinlerin, kimyasal çapraz bağlama deneyinin, kütle spektrometresi tarafından tespit edilecek kadar yüksek bir konsantrasyonda çapraz bağlı türler üretebileceği şekilde etkileşime girmesi gerekir; Farklı kütle spektrometreleri farklı algılama seviyelerine sahiptir ve ayrıca edinme protokolüne ve çapraz bağlama reaktifinin seçimine de bağlıdır. TX-MS protokolünün mevcut sürümü yalnızca lizin-lizin homobifonksiyonel çapraz bağlama reaktifi olan DSS'ye izin verir. Yine de, bu sınırlama öncelikle makine öğrenimi adımının diğer reaktifler için ayarlanması gerekme olasılığından kaynaklanmaktadır. Bu sınırlama, Cheetah-MS web sunucusunda iki çapraz bağlama reaktifi daha düşünülebildiği için geliştirilmiştir, ancak üçü de bölünemeyen reaktiflerdir. İkincisi, iki proteinin ya deneysel olarak belirlenmiş bir yapıya sahip olması ya da karşılaştırmalı modelleme teknikleri veya de novo teknikler kullanılarak modellenmesi gerekir. Tüm proteinler modellenemez, ancak geliştirilmiş yazılımın ve PDB'deki deneysel yapıların sürekli birikmesinin bir kombinasyonu, modellenebilecek protein sayısını genişletir. Üçüncüsü, etkileşen proteinler bağlı ve bağlanmamış hallerinde yeterince benzer kalmalıdır, böylece TX-MS ve Cheetah-MS tarafından kullanılan yerleştirme algoritmaları, puanlamayı sağlamak için yeterli kalitede kuaterner yapılar oluşturabilir. Bu gereklilik nispeten belirsizdir, çünkü kabul edilebilir kalite oldukça sisteme bağımlıdır, burada bilinen yapıdaki daha küçük proteinlerin karşılaştırılması genellikle bilinmeyen yapıya sahip daha büyük proteinlerden daha kolaydır.
Negatif bir sonuç durumunda, önce TX-MS'nin aynı polipeptit zincirinin bir parçası olan kalıntılar arasında iç bağlantılar, çapraz bağlantılar bulduğunu kontrol edin. Hiçbiri keşfedilmediyse, en olası açıklama, numune hazırlama veya veri toplama işleminde bir şeylerin ters gittiğindir. Birden fazla mesafe kısıtlaması modelleri desteklemiyorsa, konformasyonun çapraz bağlı kalıntılar tarafından desteklendiğinden emin olmak için modelleri görsel olarak inceleyin. En az bir çapraz bağlantıyı bozmadan interaktörlerden birini döndürmenin açık bir yolu yoktur. Verilen çapraz bağlama reaktifi için izin verilen mesafeden daha uzun çapraz bağlantılar varsa, çapraz bağlama verilerini dahil ederek ara aktörlerin modellemesini geliştirmeye çalışın.
Seçilen yazılımın hassasiyetinin TX-MS'in hassasiyeti ile karşılaştırılabilir olması koşuluyla eşdeğer sonuçlar elde etmek için alternatif yazılım uygulamaları kullanmak mümkündür. Örneğin, RosettaDock, HADDOCK ve diğerlerinin çevrimiçi sürümleri vardır. Kimyasal çapraz bağlama verilerini xQuest/xProphet5,6, plink7 ve SIM-XL26 aracılığıyla analiz etmek de mümkündür.
TX-MS ve Cheetah-MS'iyeni projeler 27,28,29'a sürekli olarak uyguluyoruz, böylece bu yaklaşımlarla üretilen raporları, raporları büyütmeden sonuçların daha ayrıntılı bir analizini sağlayacak şekilde geliştiriyoruz.
Açıklamalar
Yazarların açıklayacak hiçbir şeyleri yoktur.
Teşekkürler
Bu çalışma Knut ve Alice Wallenberg Vakfı (hibe no. 2016.0023) ve İsviçre Ulusal Bilim Vakfı (hibe no. P2ZHP3_191289). Ayrıca, Zürih Üniversitesi S3IT'ye hesaplama altyapısı ve teknik desteği için teşekkür ederiz.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Two Protein DataBank files of the proteins of interest. | N/A | N/A | Example files available on txms.org and zenodo.org, DOI 10.5281/zenodo.3361621 |
| An mzML data file acquired on a sample where the proteins of interest were crosslinked. | N/A | N/A | Example files available on txms.org or zenodo.org, DOI 10.5281/zenodo.3361621 |
Referanslar
- Berman, H. M., et al. The Protein Data Bank. Acta Crystallographica Section D: Biological Crystallography. 58 (6), 899-907 (2002).
- Herzog, F., et al. Structural Probing of a Protein Phosphatase 2A Network by Chemical Cross-Linking and Mass Spectrometry. Science. 337 (6100), 1348-1352 (2012).
- Hoopmann, M. R., et al. Kojak: efficient analysis of chemically cross-linked protein complexes. Journal of Proteome Research. 14 (5), 2190-2198 (2015).
- Seebacher, J., et al. Protein cross-linking analysis using mass spectrometry, isotope-coded cross-linkers, and integrated computational data processing. Journal of Proteome Research. 5 (9), 2270-2282 (2006).
- Rinner, O., et al. Identification of cross-linked peptides from large sequence databases. Nature Methods. 5 (4), 315-318 (2008).
- Walzthoeni, T., et al. False discovery rate estimation for cross-linked peptides identified by mass spectrometry. Nature Methods. 9 (9), 901-903 (2012).
- Yang, B., et al. Identification of cross-linked peptides from complex samples. Nature Methods. 9 (9), 904-906 (2012).
- Chu, F., Baker, P. R., Burlingame, A. L., Chalkley, R. J. Finding Chimeras: a Bioinformatics Strategy for Identification of Cross-linked Peptides. Molecular & Cellular Proteomics. 9 (1), 25-31 (2010).
- Holding, A. N., Lamers, M. H., Stephens, E., Skehel, J. M. Hekate: Software Suite for the Mass Spectrometric Analysis and Three-Dimensional Visualization of Cross-Linked Protein Samples. Journal of Proteome Research. 12 (12), 5923-5933 (2013).
- Hauri, S., et al. Rapid determination of quaternary protein structures in complex biological samples. Nature Communications. 10 (1), 192 (2019).
- Röst, H. L., et al. OpenSWATH enables automated, targeted analysis of data-independent acquisition MS data. Nature Biotechnology. 32 (3), 219-223 (2014).
- Röst, H. L., et al. OpenMS: a flexible open-source software platform for mass spectrometry data analysis. Nature Methods. 13 (9), 741-748 (2016).
- Quandt, A., et al. Using synthetic peptides to benchmark peptide identification software and search parameters for MS/MS data analysis. EuPA Open Proteomics. 5, 21-31 (2014).
- Bradley, P., et al. Free modeling with Rosetta in CASP6. Proteins: Structure, Function, and Bioinformatics. 61 (S7), 128-134 (2005).
- Gray, J. J. High-resolution protein-protein docking. Current Opinion in Structural Biology. 16 (2), 183-193 (2006).
- Khakzad, H., et al. Cheetah-MS: a web server to model protein complexes using tandem cross-linking mass spectrometry data. Bioinformatics. , (2021).
- Malmström, L. Chapter 15: Computational Proteomics with Jupyter and Python. Methods in Molecular Biology. 15, 237-248 (1977).
- Martens, L., et al. mzML--a community standard for mass spectrometry data. Molecular & Cellular Proteomics. 10 (1), (2011).
- Chambers, M. C., et al. A cross-platform toolkit for mass spectrometry and proteomics. Nature Biotechnology. 30 (10), 918-920 (2012).
- Waterhouse, A., et al. SWISS-MODEL: homology modelling of protein structures and complexes. Nucleic Acids Research. 46 (W1), W296-W303 (2018).
- Yang, J., et al. Improved protein structure prediction using predicted interresidue orientations. Proceedings of the National Academy of Sciences. 117 (3), 1496-1503 (2020).
- Koehler Leman, J., et al. Macromolecular modeling and design in Rosetta: recent methods and frameworks. Nature Methods. 17 (7), 665-680 (2020).
- Chivian, D., et al. Prediction of CASP6 structures using automated Robetta protocols. Proteins: Structure, Function, and Bioinformatics. 61 (S7), 157-166 (2005).
- Chivian, D., et al. Automated prediction of CASP-5 structures using the Robetta server. Proteins: Structure, Function, and Bioinformatics. 53 (S6), 524-533 (2003).
- Bauch, A., et al. openBIS: a flexible framework for managing and analyzing complex data in biology research. BMC Bioinformatics. 12, 468 (2011).
- Lima, D. B., et al. SIM-XL: A powerful and user-friendly tool for peptide cross-linking analysis. Journal of Proteomics. 129, 51-55 (2015).
- Happonen, L., et al. A quantitative Streptococcus pyogenes-human protein-protein interaction map reveals localization of opsonizing antibodies. Nature Communications. 10, 2727 (2019).
- Khakzad, H., et al. Structural determination of Streptococcus pyogenes M1 protein interactions with human immunoglobulin G using integrative structural biology. PLOS Computational Biology. 17 (1), E1008169 (2021).
- Khakzad, H., et al. In vivo cross-linking MS of the complement system MAC assembled on live Gram-positive bacteria. Frontiers in Genetics. 11, (2020).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır