Method Article
阵列断层扫描工作流程,用于使用扫描电子显微镜有针对性地采集体积信息
摘要
我们描述了连续切片的色带的制备及其在大型转移支持上的收集,以用作阵列断层扫描样品,以及扫描电子显微镜中的自动成像程序。该协议允许对局部罕见事件进行筛选,检索和有针对性的成像,并采集大量数据。
摘要
电子显微镜在生物学和医学中应用于以纳米分辨率对细胞和结构细节进行成像。从历史上看,透射电子显微镜(TEM)提供了对细胞超微结构的见解,但在最近十年中,现代扫描电子显微镜(SEM)的发展改变了观察细胞内部的方式。尽管当需要蛋白质水平的结构细节时,TEM的分辨率更优越,但SEM分辨率足以解决大多数细胞器水平细胞生物学相关问题。技术的进步使自动体积采集解决方案成为可能,例如串行块面成像(SBF-SEM)和聚焦离子束SEM(FIB-SEM)。然而,直到今天,当识别和导航到感兴趣的区域至关重要时,这些方法仍然效率低下。如果没有在成像前精确定位目标区域的方法,操作员需要获取比他们需要的多得多的数据(在SBF-SEM中),或者更糟糕的是,准备许多网格并对其进行全部成像(在TEM中)。我们提出了在SEM中使用阵列断层扫描的"横向筛选"策略,该策略有助于定位感兴趣区域,然后对总样品体积的相关部分进行自动成像。阵列断层扫描样品在成像过程中是保守的,它们可以被安排成切片库,准备重复成像。展示了几个示例,其中横向筛选使我们能够分析结构细节,这些细节对于使用任何其他方法都非常困难。
引言
尽管EM相关技术的重要性,但掌握它们所需的努力使整个领域仅限于少数专家。一个重大困难是为EM保存的样品中识别和检索感兴趣区域(ROI)。当通过光学显微镜分析和经过EM观察处理后,同一样品的外观有很大不同。化学制备的样品的变化包括脱水步骤后的各向异性样品收缩(每个维度约10%)以及在固定和染色方案中使用锇时荧光的损失(图1A)。对于超薄切片,使用不同的策略将样品嵌入环氧树脂或丙烯酸树脂中(图1B)。为了获得该制备的成功结果,必须将整个样品分馏成不超过1 mm x 1 mm的碎片。为了满足标准透射电子显微镜(TEM)观察条件的要求,样品的这一小部分被进一步切片为50-150nm厚的切片。由此产生的灰度图像比任何其他显微镜技术更详细地显示了整个样品的微小部分的组织组织和细胞器结构(图1C)。典型的TEM数据集提供2D信息,理论上推断以了解细胞和组织中3D空间中自然发生的过程。 图1D 提出了超微结构体积采集的挑战:如果在50nm厚度下切割1,000μm侧的立方体,则需要20,000个切片才能覆盖整个体积;对于500μm的侧立方体,它将是10,000个截面。要覆盖50 μm x 50 μm x 50 μm的体积,可能需要"仅"1,000个部分。手动获取此体积几乎是不可能的,并且对于自动化执行极具挑战性。如果除了样品深度之外,我们还需要覆盖这种假设立方体的整个表面,那么以合理的分辨率覆盖1μm2 表面就成为一个严重的逻辑问题(图1E)。虽然对于非同寻常的大型项目,如连接组学方法,大量的部分至关重要,但对于大多数"普通"的EM项目来说,生成观察所需的更多部分是一个显着的缺点。
有几种方法可以获取3D超微结构信息:连续切片透射电子显微镜(TEM),TEM断层扫描,阵列断层扫描(AT),连续块面成像扫描电子显微镜(SBF-SEM)和聚焦离子束扫描电子显微镜(FIB-SEM)。这些方法之间的主要区别在于切片策略以及图像采集是否耦合到第1代截面。在串行切片TEM中,在插槽网格上收集顺序截面,从这些序列生成TEM图像并对齐2,3,4,5。在TEM断层扫描中,倾斜网格上150-300nm截面的串联,并且当与串联切片时,可提供非常高的分辨率,尽管体积相对较小6,7,8。AT方法使用物理切片,在相对较大的支撑(例如玻璃盖玻片,硅晶圆或特殊胶带)上以各种手动和半自动方式收集切片。对于图像采集,在SEM中分析支持,具有不同的图像采集策略,可用9,10,11,12,13,14,15。 对于SBF-SEM,使用微型切片机实现物理切片,金刚石刀直接设置在SEM腔室内,从树脂块16,17,18,19的表面生成SEM图像。对于FIB-SEM,离子源去除样品的薄层,然后通过SEM20,21对暴露的表面进行自动成像。透射电镜断层扫描和AT产生物理切片,必要时可以重新成像,而FSBF-SEM和FIB-SEM在成像后消除切片。最近由多波束SEM成像的物理部分的组合提供了一种方法的组合,解决了图像采集速度22的"瓶颈"问题。这些技术中的每一种都彻底改变了获取和分析EM数据的方式,并且每种方法都有其与给定研究问题相关的实际影响。
鉴于制备的性质和超微结构尺寸的规模,预测特定目标结构在样品块中的位置并不简单(图1D,E)。ROI定位的一种解决方案是从一开始就以所需的分辨率记录整个块的图像。当远离显微镜时,感兴趣的结构可以在采集的数据量中。与此策略相关的采集时间和数据处理存在问题。需要减少记录的数据量,特别是如果ROI比组织块小得多,即,如果感兴趣的对象是特定类型的细胞(而不是整个器官)。不同的相关光和电子显微镜技术(CLEM)可以成功地在制备之前或之后在同一样品23,24,25,26,27,28,29中保存和定位荧光。然而,即使没有荧光相关性,许多细胞结构也是可识别的,仅基于已知的超微结构。对于这些情况,我们认为横向筛选阵列断层扫描在投资回报率定位和超微结构信息质量方面投入的努力之间提供了平衡的权衡。使用这种策略,在定期间隔内筛选晶圆上的子部分集,这可以根据ROI的大小和性质建立。一旦找到ROI,数据采集就会在锚点部分之前和之后开始的连续一系列部分中设置,从而有针对性地收集相关信息。
我们提出了AT协议,可以简化和加速在众多部分中获取感兴趣的区域或事件,并产生更协调的图像量。横向筛选和多步骤采集可在精确目标区域生成非常高分辨率的数据。我们描述的程序解决了3D EM数据采集的几个挑战,因为它提供了:与各种样品的兼容性,而不会从根本上改变样品制备工作流程;针对切片和扫描电镜采集进行有针对性的本地化;减少设置过程中的时间和精力;对多个部分的区域进行成像,并更好地对齐所得体积;以及平滑的拼接和对齐程序,将不同的图像编译成拼接的马赛克图片。我们选择通过来自已发布和正在进行的项目的几个示例来展示我们方法的强度。我们相信,这种方法可以极大地促进目标EM数据的生成和获取,即使对于EM经验有限的调查人员也是如此。
研究方案
注:样品制备方法在14、30、31的其他地方有描述,本出版物中没有涉及。简而言之,所示实例用戊二醛化学固定,用1%OsO4后固定,然后用1%乙酸铀酰水溶液处理,然后包埋在包埋树脂中。或者,可以使用高压冷冻制备样品,用丙酮中的0.1%乙酸铀酰取代冷冻,并嵌入丙烯酸树脂中。使用平面包埋方法制备样品块,该方法允许清晰地查看样品,便于切片的方向(图1B)。
1. 阵列生成过程
- 嵌入式样品方向和修剪
- 使用双目/显微镜,用剃须刀片轻轻划伤块表面,识别并标记嵌入块表面的投资回报率。这将有助于将样品定向到超薄切片机内,并减少切片表面。
- 将样品夹在超微量组支架上(图2A)。
- 修剪样品周围的树脂,首先使用剃须刀片(粗修剪),然后继续使用金刚石修剪工具(精细修剪; 图 2A)。使用边缘倾斜度为 20° 或 90° 的刀具,以确保块的顶部和底部平行于刀刃。
注:此步骤对于连续切片和获取直带至关重要。 - 以3:1的比例混合二甲苯和胶水。将睫毛连接到牙签上,将这种混合物涂抹在修剪过的块的顶部和底部边缘。让它彻底干燥。
- 准备阵列安装支架A(即硅晶圆)。
- 使用晶圆切割工具(EMS)切割晶圆片,根据项目目标调整其尺寸。2 cm x 4 cm 便于进行光学和电子显微镜观察。
- 在蒸馏水中清洁晶圆以清除碎屑。
- 辉光放电/等离子体使用标准设备清洁表面。精确的参数将取决于所使用的机器。从电网放电的参数开始,并按经验调整时间。此步骤对于在部分干燥时支撑上的部分的良好铺展至关重要,不应省略。
- 准备阵列安装支架B(即玻璃盖玻片)
- 如果计划进行多重免疫标记实验,请将样品转移到盖玻片上,以更好地检测荧光信号。为了提高盖玻片的粘合性,请使用前面9中描述的详细的明胶包衣程序。
- 通过蒸发将载玻片涂上铟锡氧化物(ITO)或金来增加导电性。将准备好的盖玻片保存在干净的环境中。辉光放电样品如1.2.3中所述。
2. 样品切片
- 阿特刀准备
注:要生成阵列,请使用改进的刀(ATS),该刀旨在便于获取长带。为在大型支架上生成截面而设计的Histo-Jumbo刀或类似的刀具也可以用于AT切片。- 使用泡沫胶带将针连接到刀的底部并刺穿它(图2B)。将ATS刀以0°的速度放入超薄片支架中。使用标准程序平行于块表面调整刀的边缘。
- 将修剪过的块放在刀的边缘,使其处于准备切片的位置。
- 将晶圆/盖玻片放入刀盆内,并用水填充到与刀刃相同的水平。让刀的金刚石边缘适当加湿,如有必要,使用连接的注射器抽水(图2C)。
- 阵列切片和传输
- 将切片机设置为所需的切割参数。使用ATS刀,建议使用50-100 nm的范围和0.6-1 mm / s的切割速度。开始切片(图2Di)。
- 获取长度将覆盖目标 z 卷的功能区并停止收集。根据块的大小,组织的均匀性和树脂的类型,色带将相对笔直(图2D-ii)。许多样品不会产生直的丝带,尽管投入了精力并且使用了刀的类型。
- 根据最终目标,使一条或几条长条或几条短丝带并排对齐。2 cm x 4 cm 的支架可以方便地容纳 100 到 1,000 个截面。支撑台阶上缎带的排列是需要灵巧和稳定的手的步骤。然而,这项技能的学习曲线是迅速获得的。
- 使用粘在牙签上的干净,不粘的睫毛尖端将色带从刀刃上拆下(图2Diii)。
- 使用睫毛,轻轻地将丝带移到支撑介质的中心上方。此时,如果需要,请使用氯仿或加热笔拉伸切片。但是,请记住,这种操作可能会导致色带断裂和变形。
- 通过拉动注射器开始排干水。对于更精致的丝带或较慢的水回缩,通过将注射器从软管上拆下,让水滴落。当水位降低到晶圆液位时,控制碳带并在必要时通过轻轻地将碳带推向中心来重新定位。将带状物沉降在晶圆表面上后,继续排水,直到剩余的水完全从盆中收回。
注意:如果不使用底部水缩回ATS刀,请小心减少ATS刀侧面的水,以免引起湍流。 - 将浴缸内支架上的部分部分完全晾干。根据支撑的疏水性和环境湿度水平,水将以不同的速率蒸发(图2Div)。重要的是让样品慢慢干燥以减少或完全避免样品上的所有褶皱。
- 将干样品转移到密闭的盒子中以防止污垢污染,并将其置于60°C的烘箱中至少30分钟。如果需要,使用重金属复色切片以增强整体对比度。
- 在程序结束时,按照制造商的说明谨慎清洁刀具
注:晶圆上多个或串行部分的传输(图2E)与槽栅格上的传输(图2F)相比,完全改变了EM制备体验。在单个支撑上不间断地切片和收集截面,减少了连续分切中常见的剖面和截面收集相关错误的数量。相当于在一个晶圆上收集的100个1000μm x 500μm的切片对应于33个插槽网格(图2G)。
3. 样品观察
注意:本节介绍使用市售软件实现的工作流步骤(请参见 材料表)。可以使用任何配备正确探测器的SEM上的任何图像采集软件,但是,特定的用户操作会有所不同,并且通常需要更多的手动操作。
- 获取SEM图像的概览图,以显示晶圆上的截面位置。内置光学相机图像有助于定义覆盖部分或所有部分的SEM图像镶嵌。通过单击-拖动鼠标在样品的相机图像上创建镶嵌,然后启动 自动采集。
注意:非常粗糙的成像设置,即1-2 μm像素大小和1 μs停留时间就足够了。这一过程在 补充材料 (第2-7页)中进行了说明。 - 使用剖面 查找器自动检测功能查找分区 或手动查找分区。此软件可根据图像匹配自动检索截面位置和轮廓。有关此过程的说明,请参阅 补充材料(第 8-15页)。
- 如果概览图像不能清楚地显示ROI,请获取更高分辨率的切片图像。使用 分区预览 功能自动创建和获取图像。这一过程见 《补充材料》,第16-18页。必须根据用户搜索的 ROI 的性质和大小来选择映像设置。
- 要找到最佳设置,请激活显微镜控制软件中的 实时成像 并导航到一个ROI。更改成像设置,直到图像清楚地显示ROI,但图像采集不会太长。
- 定义映像区域
注:提出了几种不同的策略。- 如果只需要对几个部分进行成像,请使用到目前为止创建的部分的图像导航到相关部分,并使用 可缩放查看器 (显示其原始相对位置中所有采集的图像)来查看所有部分。找到应以高分辨率成像的部分后,使用单击-拖动创建成像区域。选择高分辨率图像处理设置并将设置存储在模板中。将此模板重用于其他部分。
- 要发现特别小或难以检测的罕见事件,请使用横向筛查方法。在每十个部分或每个色带的一个部分上手动创建具有高分辨率成像设置的成像区域,并获取图像。查看软件中包含ROI的图像并标记部分或做笔记。
- 从包含 ROI 的部分开始,在部分集中向前和向后导航,并在相同的相对位置创建高分辨率成像区域,只要结构在截面中仍然可见。这可以手动完成,也可以使用后续步骤中描述的过程来完成。
- 在十多个连续部分中采集图像。缩放图像后 单击"开始位置细化 ",以提高注册截面位置的精度,如手册中所述。这样做会降低图像系列的位置可变性。该过程在 补充材料 第19-21页中进行了说明和解释。
- 单击并拖动任何部分以定义图像区域,同时按住 Alt 键,然后从释放鼠标按钮时打开的上下文菜单中选择" 创建切片集阵列"。 然后,软件会在之前找到或标记的所有部分中的同一相对位置创建映像区域。可以使用"截面范围"滑块将成像限制在特定范围的 截面 。
注意:此过程可以对任意数量的成像区域重复,因此允许记录许多小型高分辨率图像,而不是在每个部分上记录单个较大的图像。 - 创建后,根据需要在每个图像系列中设置像素计数、像素大小、平铺布局、像素停留时间等。创建和设置成像系列后,它们都将列在工作提示中。
- 配置自动功能并开始图像采集
- 使用与上一步中所述相同的方法为自动功能创建单独的图像系列。将图像系列移动到包含高对比度结构的截面上的某个位置。
- 将图像系列设置为 1024 x 884 像素,然后选择与前面步骤中设置的图像系列中使用的最高分辨率相对应的像素大小。在自动功能列表中,选中 自动对焦 和 自动污名器。
- 在采集顺序控件中选择按 部分 ,并确保自动功能图像是列表中的第一个项目。通过单击作业提示旁边的" 运行 "按钮开始图像采集。这些程序见 《补充材料》,第22-23页。
注意:无需手动预先关注每个部分。在记录过程中,每当显微镜前进到新部分时,在本节中记录所有其他图像之前,将执行自动功能。
4. 数据对齐和分析
- 数据导出
- 确保以.tif格式保存数据,因此不需要专用的导出功能。将数据排序到与层树中的层和元素直接对应的文件夹结构中。
- 记录图像镶嵌后,使用 StitchAll 功能自动拼接所有图块。
- 斐济的堆叠对齐和裁剪
注意。许多软件包(免费和商业)可用于处理阵列断层扫描数据。以下步骤与开源程序Fiji32一起显示,因为它广泛可用并且包含所有必需的功能。- 将图像堆栈(或拼接的图像)作为虚拟堆栈导入斐济。
- 如果需要对对比度/亮度进行标准化,请选择 "增强对比度..."从 "进程 "菜单中。将 饱和像素 设置为 0.1 或更低,然后选中 处理所有切片。
- 从插件菜单中,选择 注册|线性堆栈与 SIFT 对齐。
- 从预期转换下拉菜单中选择刚性或仿射。否则,请保留默认设置。通过单击"确定"开始对齐。
注意:将数据加载为虚拟堆栈允许斐济处理任何大小的堆栈。对齐的输出在RAM中创建;但是,这可能会限制可以处理的堆栈的最大大小。在这种情况下,请使用 注册虚拟堆栈切片,这是相同注册算法的文件夹到文件夹实现。注册完成后,将输出数据作为虚拟堆栈加载。 - 通过单击"裁剪"来 裁剪 图像堆栈,使其仅包含 ROI。
- 将堆栈另存为单个.tif图像或一系列.tif图像。
注:阵列断层扫描的关键步骤如图 3所示。
结果
以下示例旨在演示推荐工作流的多功能性。案例研究插图是我们很难用任何其他技术获得满意结果的项目。我们选择了成虫果蝇来说明人们在许多类型的样本中可能遇到的典型挑战。这个管状器官长约6mm,横截面500-1000μm,分为具有独特功能和细胞组成的不同区域(图4A)33。根据切片方向,肠道轮廓的尺寸及其在切片上的外观会有所不同。横向或纵向取向的部分相对较大,并且只能将一对部分放置在单个TEM网格上(图2F)。只有一小部分组织可以在FIB中成像,对于SBF-SEM,难度与任何非均质样品相似。AT为此类样品的分析提供了有效的权衡,而扁平包埋有助于ROI定位。仔细修剪选定区域周围多余的树脂(图4B)对于从相关区域有效收集阵列非常重要(图4C)。可以在单个晶圆上按顺序或随机收集数百个切片(图4D)。根据研究问题,样品筛选和获取将需要不同的策略,我们将其任意分为几种场景。为了更有针对性的方式说明不同的场景,我们从不同的研究项目中选择了几个案例研究。
分析许多随机分布的大结构1-10μm范围(图4E)
通常,需要超微结构数据来验证由几种实验方法产生的假设,比较标准和实验改变的条件。在这些情况下,通常会在网格上随机收集多个部分,并对其进行筛选以定位和成像感兴趣的区域。这种策略通常不太系统化,仅限于少数分析部分。我们建议从给定的色带中记录数十个/数百个中等分辨率部分的概述(图4D)。对于70nm的典型切片,200个切片将跨越约14μm,其中将包含许多细胞,全部或部分,在半小时内完成。作为第一步,记录整个色带的低分辨率概览,并且概述有助于省略显示制备伪影(例如,褶皱,污垢)的部分。之后,可以使用单个或镶嵌成像手动或直接在截面的选定部分或整个截面上进行手动或自动采集,然后进行拼接(图4E)。之后,可以使用高分辨率参数从所选区域获取图像。例如,线粒体,细胞核和微绒毛可以从这种统计改进的方法中受益(图4Ei-iii)。
分析500-1,000 nm范围内的多个小的,稀疏分布的结构(补充电影1)
在这种情况下,无法简单地在低放大倍率概览扫描中识别ROI,并且需要高分辨率图像。在传统的TEM样品中,在找到所需的特征之前,必须对部分进行繁琐的放大和缩小。通常,在统计上,对多个样本中的多个独立位置进行成像比生成单个大体积更相关。在这种情况下,手动获取的复杂性呈指数级增长。虽然一些TEM解决方案可以自动采集或筛选多个网格,但网格的大小和串行切片挑战经常使该方法与许多样品不兼容。对于类似情况,我们生成一个完整的中分辨率图,其中包含多个部分的整体投资回报率,其分辨率足以识别感兴趣的结构。在这个横向筛选步骤中,建议一次跳跃几个部分,目的是在随机接近时至少击中感兴趣的结构的一部分。这在很大程度上取决于结构的整体尺寸:例如,如果结构的整体尺寸为500nm,部分厚度为50nm,则连续至少九个连续部分可能包含感兴趣的结构的一部分。这样,跳过6-7个部分可以有效地在多个区域中找到许多不同类型的结构。自动获取所选部分的已解析镶嵌贴图,可以在获取后仔细筛选这些部分。一旦获得这种高分辨率的地图,就可以裁剪出多个ROI或用于在ROI上定义额外的局部成像区域(补充电影1)。高尔基体,中心粒,连接点,微管,不同类型的囊泡是可能从这种情况中受益的结构的很好的例子(补充电影1)。
分析大样本中稀疏分布的大型ROI(图4F-4H)
这种情况涉及罕见的事件,这些事件通常被描述为"大海捞针",其中问题不在于ROI识别,而在于其定位。对于许多样品,相关方法不是一个有效的选择,但ROI通常具有揭示性的超微结构,并且在局部化时可以以高可靠性进行识别。对于这些样品,必须应用多级采集,从预筛选的样品开始,以中等分辨率进行数十至数百个切片。在此处使用的软件中,有两种不同的策略可用于获取多个部分的图像集:以更高的分辨率录制预览图像或获取具有适当设置的阵列图块集(图4F)。果蝇肠道中的不同特殊细胞类型随机分布(例如茎,肠内分泌细胞),并以随机方向进行薄切片。然而,在筛选使用高分辨率参数从单个部分或作为串行图像集合获得的图像后,可以在视觉上区分它们(图4G)。对齐后,可以使用不同的软件解决方案渲染堆栈(图4H,补充视频2)。
场景1:肠道类器官(图5A)
类器官正迅速成为现代生命科学中最前沿的工具之一。这种近生理3D干细胞衍生的器官模型使得对一系列体内生物过程的准确研究成为可能,包括组织更新,对药物的反应和再生医学。最近推出的微型肠管34开辟了新一代类器官技术,与体内组织生理学,细胞类型组成和稳态非常相似,为疾病建模,宿主 - 微生物相互作用和药物发现提供了广阔的视野。然而,当需要超微结构表征时,使用随机样品在如此大的组织中定位不同的细胞类型可能具有挑战性。此外,在可变的"感染"测定中,确保分析揭示影响组织的不同发育阶段至关重要。对于此类研究,样本的统计显着覆盖率是核心,但使用传统的TEM并网方法很难实现。在这种情况下,AT-scenario 1是有益的:可以在晶圆上生成许多连续的部分(图5Aii),并使用低分辨率参数进行筛选,以定位一般感兴趣的区域(图5Aiii;箭头)。可以使用高级采集参数(图5Aiv 和 图5Av)将这些区域作为进一步分析的目标。当检测到相关结构(通常每100-300个截面中一次为5-10个截面的集群)时,很容易将注意力集中在每个感兴趣的结构上并手动获取单个图像,或者使用自动化功能跨多个截面获取图像量。
场景2:果蝇蛹(图5B)
研究细胞分裂和控制细胞周期进展的机制对于理解多细胞生物中的标准和改变过程至关重要。存在的信息通常来自单细胞系统;然而,该解决方案缺乏组织中细胞之间3D相互作用的关键背景。果蝇幼虫发育中的背部的单细胞单层是一般上皮细胞与细胞分裂之间相互作用的完美模型35。它是分子和细胞相互作用研究的既定模型,使用荧光显微镜和遗传操作提供的数据的组合。脱落是细胞分裂的最后一步,可确保两个分裂细胞之间的最终分离,并且表征脱落期间发生的结构变化对于我们理解有丝分裂至关重要。然而,notum中的有丝分裂不容易在超微结构水平上定位:与脱落区相比,细胞相对较大(图5B)。脱落区的整体大小与要覆盖的部分的表面之间的比率很大(图5Bi)。尽管使用TEM或SBF-SEM方法36可以定位脱落区,但这项任务是费力的。在这种情况下,可以使用20-40个部分的跳跃的自动中分辨率概览图像来定位分割单元格(图5Bii)。当识别出这样的细胞时,这些部分可以作为更仔细检查附近部分的锚点,并且可以找到并选择许多分裂的细胞进行进一步分析。这样,可以定位并整个脱落区成像(图5Biii)。根据问题的不同,可以收集单个高分辨率图像或3-7个图像序列以覆盖结构的深度(图5Biv)。
场景3:小鼠单核细胞神经元(图5C)
小鼠为大脑发育提供了一个完善的模型,并在不同水平上得到了很好的记录,包括EM。尽管不同的自动串行块面方法已被广泛用于研究脑组织,但在某些情况下,AT更适合收集必要的数据。下丘脑是一种成熟的神经科学模型,是大脑中包含多种神经元类型功能的一部分。下丘脑小丘脑细胞代表第三脑室底部的室管膜胶质细胞的特定亚群,具有异常长的过程(高达300μm)和大的端脚(〜5μm)37。这使得它们不方便通过TEM或FIB方法进行分析。当需要定位和分析几个独立的tanycytes时,任务进一步复杂。促进这项任务的方法之一可以是半相关靶向,其中荧光图在固定和嵌入EM之前从荧光标记的样品中获得。通过结合来自荧光样品的位置信息和平埋塑料复制品,对捕获的区域进行切片。之后,可以使用 AT 方案 3:生成高级概览镶嵌图像以显示具有 tanycyte endfeet 簇的区域。随后,软件中的自动化功能用于设置在单个图像或平铺模式下从一个或多个区域采集图像序列。这些图像可以单独分析,作为对齐的堆栈或在此之后渲染。
AT方法的强大功能允许相对轻松地将数据从2D"升级"到3D:地图可从主要采集中获得,并且可以从所选区域及其附近获得体积。生成的堆栈可以对齐并随后呈现。必须事先确定需要哪种分辨率和图像质量才能找到投资回报率。成像应该能够识别ROI,但不能超过此值,因为采集时间与像素停留时间和像素大小的平方反比成比例。
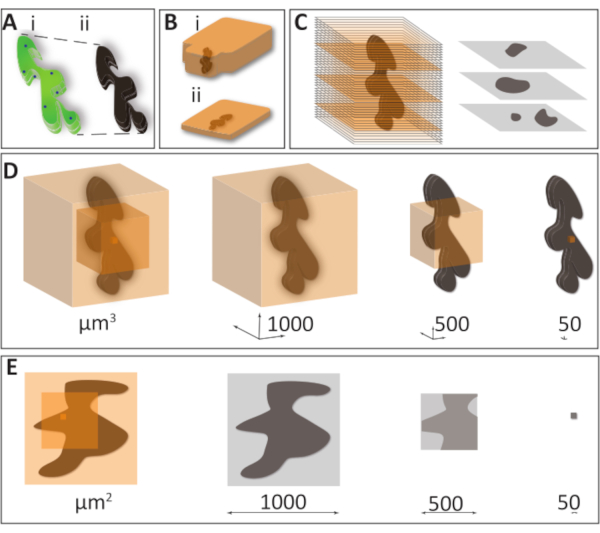
图1:EM样品制备和体积采集的挑战(A)由于样品制备过程中的高重金属浓度和脱水而导致荧光和收缩的损失。(i) 在LM(ii)下观察到的标本的示意图,该标本是为EM准备的同一标本,该标本变得完全不透明,并损失了其体积的10%左右。(B) 样品包埋通常使用环氧树脂或丙烯酸树脂完成。传统模块(i)可以成功地用于不需要特定方向的均质样品。当必须在显微镜下瞄准和定向时,扁平块(ii)是有帮助的,显微镜是一个旨在切片的精确区域,例如,在非均匀样品或相关显微镜程序中。(C)在整个样品体积中,只有有限的部分在单个50nm切片上表示,提供3D样品的2D图像,通常在不熟悉的方向上。(D) 为了说明记录过大体积与精确瞄准的问题,我们选择了三个面为1000,500和50μm的同心立方体,以包括假设的1000 x 500 x 500μm样品(深栗色)。如果用50 nm切片彻底切割这些假设的样品立方体,以覆盖整个体积,则需要总共20,000,10,000和1,000个切片,以及800 tb,100 tb和100 gb(成像分辨率5 nm x 5 nm x 50 nm,8位数据)。这表明了仅为了获取最小必要数量而规划获取EM数据的重要性。(E)以高分辨率覆盖大样品表面积会带来类似于大体积的问题。将多个高分辨率图像拼凑为一个是解决此类问题的有用解决方案。但是,要以10,000x的放大倍率使用2024 x 1048框架覆盖1 mm x 1 mm的表面,将需要大量的瓷砖,这可能变得具有挑战性。此外,如果在切割过程中切片被可变压缩或扭曲,则生成的数据堆栈几乎不可能对齐。请点击此处查看此图的放大版本。
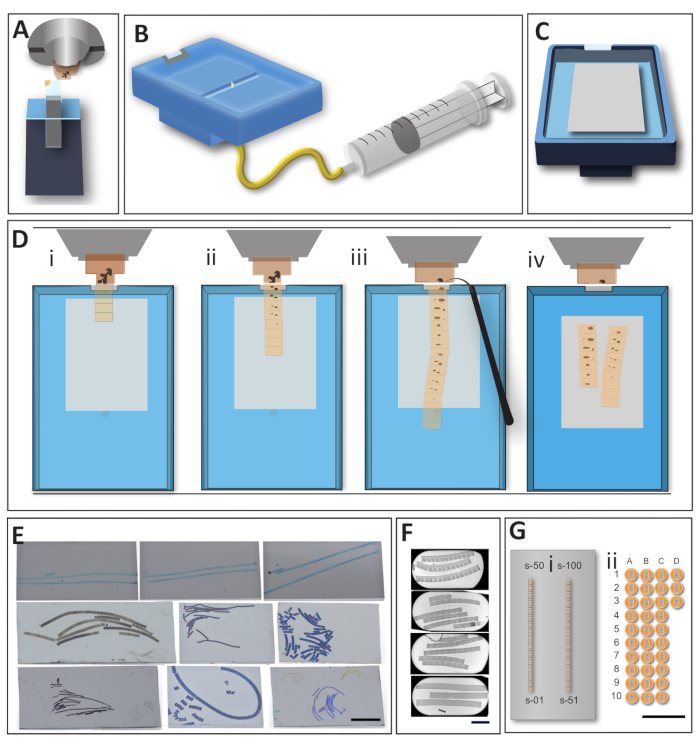
图2:在大支架上直接生成切片阵列的工作流程 (A)为了使用修剪工具进行紧密修剪,块固定在切片机支架内。该步骤有助于确保块的平行侧面,并减少样品周围的空树脂。 (B) 用于AT截面采购的改装刀。一艘大船便于在样品切片和支撑上转移过程中转移截面及其操作。一个大盆地可以操纵截面;排水系统限制了排水步骤中碳带的运动,平坦的底部使支撑的逐渐干燥变得可靠。(C) 将刀,准备切片,将辉光放电的晶圆放置在盆的底部,并将水水平到边缘。刀的结构使针保持嵌入,而不会干扰支撑。(D) 阵列生成,切片机切片区域的顶视图。(i)第一部分通常很容易获得,因为它们彼此粘附并形成规则的丝带。(ii)当向色带中添加更多截面并且变长时,色带将失去稳定性并经常弯曲。保持序列轨迹按顺序组织至关重要,以便为图像采集步骤做准备。(iii)当一条带状部分达到所需长度时,用睫毛小心地将其从刀刃上取下。(四) 水从盆地排出;晶圆留在内部,直到完全风干。这一步是必不可少的,因为它有助于拉直部分并避免形成微褶皱。将晶圆置于60°C的烤箱中至少30分钟,以将部分固定在支撑物上。(E) 具有转移部分的示例晶圆。虽然获得笔直而准确的色带很方便,但在大多数情况下,实际样品可以防止形成这种理想的色带。然而,即使是"草率"的丝带对于大量案例来说也是非常有益的,而"整洁"丝带的重要性将取决于收集这些部分的研究策略。比例尺 1 cm. (F) 带有串行部分的示例插槽网格。即使许多部分被收集在一个网格上,它仍然只是单个晶圆上可以收集的一小部分。掌握网格(特别是狭缝网格)上切片转移所需的技能是掌握电子显微镜样品制备的重要瓶颈。比例尺 500 μm. (G) 无论使用哪种截面采集方法,与并网采集相比,AT 方法的优势在于生成顺序截面的相对容易程度。如果考虑1000 μm x 500 μm样品块,则在2 cm x 4 cm晶圆(i)上安装约100个切片没有问题。插槽网格上相同大小的部分最多只能容纳三个部分/网格(ii)。我们提供了一个缩放图像来显示可能需要多少个网格才能覆盖相同数量的部分,而没有提到在网格上收集串行部分的难度。比例尺 = 1 cm。 请单击此处查看此图的放大版本。
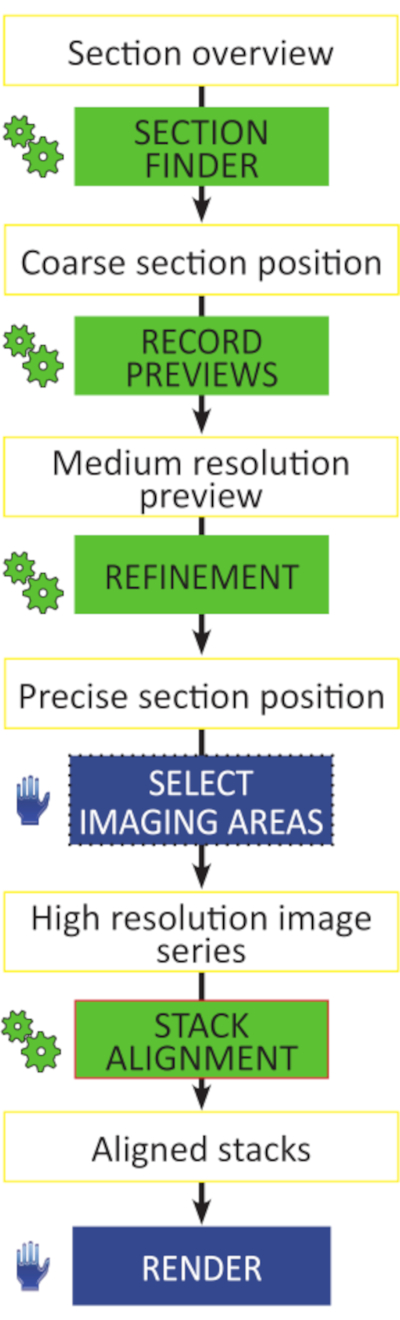
图 3:阵列断层扫描工作流程的关键步骤。 无人值守采集高分辨率图像堆栈的工作流程示意图。所有准备步骤都是自动化的(绿色齿轮符号),不需要逐节手动执行任何操作。图像堆栈可以在任何能够自动刚性或仿射对齐的图像分析软件中对齐。 请点击此处查看此图的放大版本。
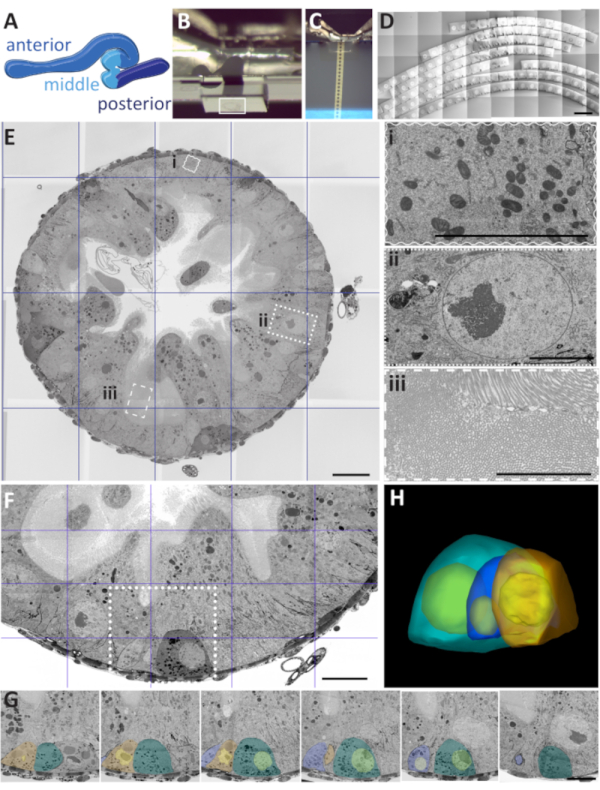
图4:以果蝇成虫肠道为示范模型的三种采集场景。(A)剖析的果蝇中肠的图画,有三个主要区域由不同的颜色指定:前部,中间和后部。(B) 修剪过的扁平块,其中肠胃定向进行横向切片。请注意,在含有感兴趣区域(白色矩形)的组织周围仔细平衡空树脂量。(C)横向连续段漂浮在AT刀的盆地内的水面上。所有图像均使用具有反对比度的镜面探测器在次级电子扫描电镜模式下采集。(D) 晶圆上横向串行部分的拼接马赛克图像。比例尺是1000μm.(E)横截面穿过果蝇肠道。比例尺 20 μm。该图像是7 x 7中档图像的拼接马赛克。插图 - 特定感兴趣区域的更高放大倍率和分辨率图像:(ii)细胞核,(iii)刷子边界和(i)线粒体。比例尺 5 μm 适用于所有。(F) 一个穿过肠道的横向截面的中程图像,其目标是发育中细胞的位置(正方形)。比例尺为 20 μm。(G) 根据在分析面板 F 中存在的部分期间的局部区域收集的串行切片的目标阵列。比例尺为 10 μm。(H) 3D 模型是根据从面板 G 中的目标串行采集中获得的 50 个部分堆栈序列呈现的。请单击此处查看此图的放大版本。

图5:AT应用场景的案例研究(A)肠道类器官中不同细胞结构的定位。(i) 嵌入式硅微芯片。比例尺 = 200 μm. (ii) 通过芯片中央部分的 127 个横截面的拼接马赛克图像。比例尺= 1500μm(iii)通过肠道类器官部分的完整横向截面的四个低分辨率图像。箭头指向潜在的感兴趣地点。(iv)不同的ROI,针对选择用于进一步分析的低分辨率显微照片。比例尺= 10μm.(v)目标感染细胞的高分辨率图像。如果需要,对相邻部分中的同一区域进行分析可以提供有针对性的 3D 信息。比例尺 = 5 μm. (B) 黑腹果蝇蛹蛹的中体定位。(i) 解剖果蝇蛹的示意图。Notum在去除保护性角质层的部分(棕色)后暴露于解剖(米色)。黑线表示剖面的方向(ii)穿过图中显示的区域的横截面。该图像将 3x7 连续拍摄的高分辨率 SEM 图像拼接到一个镶嵌面板上。黑色矩形分隔包含分隔单元格的区域。比例尺为15μm.(iii)从面板ii中分频的单元格上的缩放图像。在这种放大倍率和分辨率下,中体是显而易见的(白色箭头)。分析整个部分以找到分裂的单元格。在横向筛选步骤中,以20-30个截面间隔在不同的切片带之间跳跃,可以定位许多分裂的细胞对。当分割细胞被定位时,比例尺为5μm(iv),从中间体周围的四个部分收集的中体的顺序图像,在面板(iii)中以黄色方块分隔。比例尺为1μm.(C)Tanycytes端脚定位在小鼠下丘脑。(一) 振动切片的荧光图像。Tanycytes表达tdTomato荧光蛋白(红色)。白色矩形分隔感兴趣区域。比例尺500μm.(ii)为EM制备的相同振动切片机部分将根据来自面板(i)的荧光信息的间接相关性在感兴趣区域周围仔细修剪。白色虚线表示超薄切片的区域。两块面板的比例尺均为 50 μm。(iii)在感兴趣区域通过振动切片的横截面。SEM镶嵌图像由75个拼接图像组成。通过横向筛查靶向几个切片,并使用相似的参数进行成像。这些部分被"离线"分析,以找到ROI - tanycyte endfeet。黑色矩形表示包含 tanycyte 端脚的区域。本节将作为进一步分析的锚点。比例尺为15μm.(iv)血管周围单胞细胞端脚的高分辨率,高放大倍率图像。在一个截面上对ROI进行初始定位后,z序列从上游的相邻截面收集到锚点截面(图iii)。比例尺 5 μm.请单击此处查看此图的放大版本。
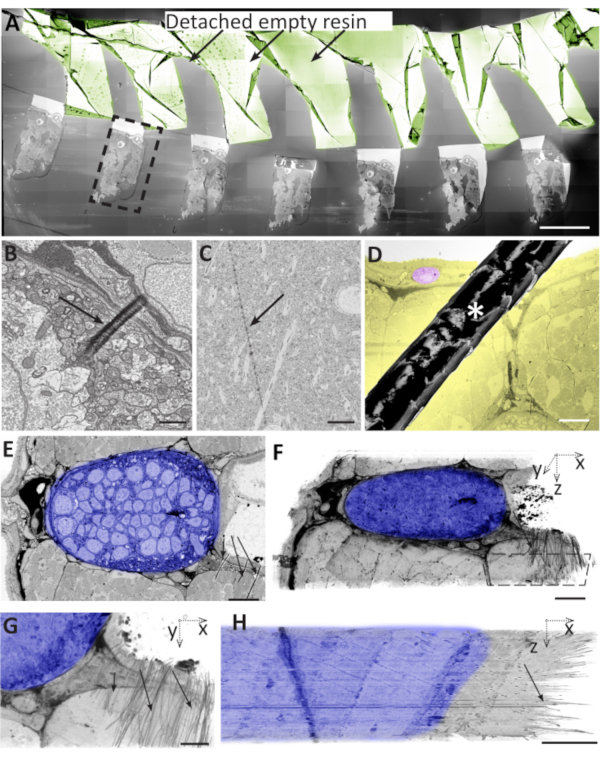
图6:超切片术,切片收集和切片存储过程中的问题可能导致伪影。 大部分空树脂从组织中分离并折叠在自身(棕褐色)上。虚线黑框表示用于包含整个部分的区域。比例尺500μm.(B)在50nm厚的斑马鱼切片表面上的小的局部褶皱。比例尺1μm(C)在70nm小鼠大脑切片的表面上的刀痕。比例尺5μm.(D)晶圆表面上的头发(星号),部分覆盖斑马鱼肌肉部分。在黄色中,组织是分析的目标。粉红色,用于作为受影响区域大小的参考的细胞。比例尺50μm.(E)斑马鱼在右下角有皱纹(黑色箭头),其中致密的神经组织(蓝色阴影)与较软的肌肉组织和空树脂(黑色箭头)接壤。比例尺10μm.(F)对50张图像的堆叠进行体积分割,如E所示,显示该区域在大多数部分都显示出皱纹。虚线多边形勾勒出 G. 比例尺 10 μm. (G) XY 视图中显示的区域,其体积分割与 F 中相同,将褶皱显示为块右半部分的短黑色笔触。请注意,组织其余部分的堆叠对齐不受皱纹的影响。比例尺5μm.(H)XZ投影与G中的相同区域,显示所有50个部分的皱纹。比例尺 5 μm.请单击此处查看此图的放大版本。
补充影片1:通过果蝇前中肠横截面的高分辨率蒙太奇图像。倒置 SE-MD 扫描电镜图像的镶嵌图像。以5 nm分辨率自动采集352个单独的图像图块,并拼接以呈现整个横截面。可以放大以获取更多详细信息,并使用相同的图像获得数据的详尽覆盖。紧密的连接,微管,不同类型的囊泡可以在放大时。比例尺为10μm,请点击这里下载这部电影。
补充电影2: 果蝇 肠道细胞渲染。 五十个对齐的马赛克图像,在分裂肠细胞的区域。细胞边界(蓝色、绿松石色和橙色)和细胞核(白色)的 IMOD 渲染。请按此下载短片。
补充材料。 请点击此处下载此文件。
讨论
电子显微镜可以深入了解细胞和生物体的超微结构,为此,通常需要在其3维环境中对感兴趣的结构进行成像。尽管超微结构分析采用了多种EM策略,但仍然没有"黄金标准"解决方案。主要原因是样品种类繁多,许多生物学问题,通常需要量身定制的方法。建议的AT工作流程旨在最大限度地减少样品处理,数据采集,评估和存储所需的时间。此外,改进的刀为简化阵列采集提供了有用的工具。晶圆上切片的紧凑布局便于观察和随后的样品存储。这种布置通过从色带水平移动到另一个色带并仅扫描每个色带上的一个部分来实现样品的"横向筛选",从而大大减少了定位ROI所需的时间。所提出的数据采集方案有助于瞄准小而随机分布的区域。一旦发现,AT/SEM将高分辨率成像精确地限制在感兴趣的体积,无论是手动执行还是在自动功能的帮助下执行。对于有限体积的AT,可以手动完成,操作员可以浏览样品并逐个定义成像区域。该软件的自动化模块为大截面上的小区域成像提供了灵活的图像采集策略。该软件中的自动化允许在数百个切片上记录大型高分辨率图像,从而实现与SBFI相似的体积。记录所有切片的概述图像可简化ROI定位并减少在显微镜下花费的时间。由于在记录概览和更高分辨率预览期间,这些部分不会受到损害,因此AT/SEM允许重复使用样品以收集其他ROI的进一步数据或以更高的分辨率。
图像采集时间是3D EM最重要(也是最昂贵)的方面之一,因此在实验设计中应予以考虑。虽然大面积成像比小面积成像需要更长的时间并不奇怪,但很容易低估其影响:根据所选的成像参数,每个部分的采集时间可能从几秒钟到几小时不等。关键成像参数包括视场大小、分辨率和停留时间。假设目标分辨率为每像素 10nm,停留时间为 1 μs,则成像 20 μm x 20 μm、100 μm x100 μm 或 500 μm x500 μm 的磁场需要 4 秒、100 秒或 2,500 秒才能记录下来。我们可以将这些每节成像时间乘以截面数,以估计完成成像作业所需的时间。如果切片数量很少或显微镜工具时间无关紧要,则每段成像时间长是可以接受的。
但是,在大多数情况下,有必要将录制时间限制为通宵工作或周末工作。应该考虑的3D EM的一个同样关键的方面是生成的图像数据的数量和结构。在100个部分中记录上述成像场分别生成400 mb,10 gb或250 gb的图像数据;500 μm x 500 μm 图像带来了每个大于 2 gb 的额外问题。许多用于数据评估的软件程序无法打开这种大小的图像。
为了缩短成像时间,选择像素停留时间以满足后续数据评估(例如,重建、跟踪)的信噪比要求非常重要,并将记录限制在定义的ROI内。该软件的AT扩展便于在串行部分的小区域内采集图像。该软件支持手动和自动工作流程以及许多半自动变体:成像区域可以手动定位并聚焦在每个部分,或者用户可以使用自动切片查找器和位置对齐功能。根据样品类型或成像目标选择和支持的自动化水平,在数百个切片中设置图像采集所需的时间可能需要整个工作日(手动完成)或几分钟。原则上,阵列断层扫描使得获得小投资回报率比其他3D EM方法更具挑战性;在连续截面上不精确的区域放置必须通过获取更大的区域来补偿。例如,如果ROI尺寸为20 μm x 20 μm,成像场的截面间位置变异性为10μm,则需要获取40 μm x 40 μm的图像,以确保在每个部分的每个图像中完全捕获ROI。实际图像位置变化范围从 100 μm 到 <10 μm,具体取决于用于位置对齐的软件功能的可用性或质量或用户的耐心。使用此软件,可以在大多数样品中实现10μm,而无需过多的手动干预。
与任何技术一样,AT有几个弱点可以影响成功的数据采集,其中许多类似于其他基于切片的方法。空树脂与组织缺乏均匀分布可能导致弯曲或破碎的阵列。在极端情况下,各部分可以从支撑上分离(图6A)。切割过程中的可变压缩或拉伸会产生褶皱,这些褶皱可能会在后续切片的可变区域破坏样品(图6B)。使用损坏的刀,在收集的部分的表面上可能会出现刀痕(图6C)。切片条件的差异会导致偶尔的切片压缩和厚度差异。灰尘或污垢颗粒可以落在一个部分并部分遮挡感兴趣的区域(图6D)。由于自动对比度、自动对焦和自动污名器功能不完善,图像采集可能会失败。自动创建的成像区域的定位可能是可变的,可能无法捕获所有部分的投资回报率。
在拼接和对准阶段可能会出现几个问题。例如,由于样品内部的大空白空间,马赛克瓷砖采集的自动拼接可能会失败。由于 3D 中形状的巨大变化,图像堆栈可能难以注册。专门开发的程序(例如,IMOD,斐济,TrackEM2,MIB或MAPS-AT)可以促进半自动对齐32,38,39,40。更具挑战性的部分可以使用照片编辑软件手动对齐。遗憾的是,某些数据集可能无法正确对齐。
对于安装在网格上的TEM连续截面来说,大样品具有挑战性;另一方面,许多项目没有理由使用FIB / SEM或SBF-SEM进行长时间的自动采集。AT是繁琐的串行截面TEM的直接替代方案,其中晶圆上串行截面的收集和操作比使用插槽网格更直接。我们开发了几种策略来促进数组的收集,我们分享了扩展现有工具包的方法。在ROI鉴定具有挑战性的情况下,AT-SEM提供了一个基本优势,可以有效地筛选需要50至500个切片的细胞器规模分辨率的样品。对于较大的卷,如果需要更多部分,可以有效地收集自动收集 AT 策略。AT样品可以多次重新成像,便于根据先前获取的概述图像对高分辨率区域进行有针对性的成像。我们认为,这里提出的AT/SEM的针对性分析和减少过采样降低了劳动力和数据存储要求。最终,可以收集和维护章节库,以便以后重用和咨询。对于体积采集,FIB或SBF-SEM方法在块面上易于识别ROI或分析需要大3D体积时,可提供出色的解决方案。但是,当必须有针对性地从定义的 ROI 收集高分辨率堆栈图像时,FIB/SBF-SEM 的效率会降低。综上所述,所提出的AT样品筛选方法和使用中分辨率的概述图像允许将图像采集限制在截面阵列的相关部分。成像区域的精确瞄准可加快数据获取时间并简化数据评估。
总而言之,尽管AT/SEM的概念并不新颖,但其使用仍然不像其优点所暗示的那样广泛。总体而言,它提供了与其他现有EM方法的补充程序。AT/SEM与最广泛的样品制备方案和成像工作流程兼容,可以在任何FIB/SEM或SBF-SEM显微镜上作为附带技术完成。在本文中,我们专注于AT,用于记录来自其他方法不太可能成功解决的样品的超微结构数据。我们希望,所描述的方便收集部分和相当自动化的获取策略的过程将有助于那些从未遇到过该方法的人的首次尝试,并将有助于为那些已经具有一定经验的人完善它。
披露声明
作者Tilman Franke是Thermo Fisher的员工,该公司生产本文中使用的电子显微镜和试点程序。
致谢
我们要感谢洛桑大学EM设施的成员在AT程序不同步骤的开发过程中的支持。我们要感谢Gareth Griffiths,Marta Rodrigues,Urska Repnik,Christel Genoud,Helmut Gnaegi,Einat Zelinger,Paola Moreno-Roman,Lucy O'Brien和Lindsay Lewellyn在手稿准备和批判性阅读期间进行的讨论。我们要感谢贡献用于演示不同场景的样本的团体:Matthias Lutolf,Michail Nikolaev,Devanjali Dutta,Till Matzat和Fanny Langlet。
材料
| Name | Company | Catalog Number | Comments |
| Cutting | |||
| AT sectioning knife | Diatome | DUATS3530 | Diatome Jumbo knife |
| Diamond knife for trimming 90° | Diatome | DTB90 | Diatome trimming 20° Glass knife |
| Pattex contact adhesive | Pattex | PCL3C | |
| Silicon wafer | Ted Pella | 16015 | Resistance: 1-30 Ohms Type P: (Boron) (1 primary flat) Roughness: 2 nm No SiO2 top coating TTV: = <20 µm Wafer is polished on one side |
| Ultramicrotome | Leica UC6 | Alternative: Leica UC7 | |
| Wafer cleaving kit | EMS | 7642 | EMF, Small Sample Cleaver, CatNo. 7652 |
| Image acquisition | |||
| FESEM | Thermo Fischer Helios | 1072419 | Alternatives: Zeiss, Jeol, Hitachi, TESCAN |
| Maps 3 for SEM with Correlative Workflow & Array Tomography | Thermo Fisher Scientific | 1135932 | Maps provides automation of SEM imaging workflows and allows importing of 3rd party data for CLEM and navigation. |
| Image analysis | |||
| Amira x.y | Thermo Fisher Scientific | 1131599 | Amira is a 3D data visualization and analysis software with several practical functions for Array Tomography data reconstruction. |
| Image processing | Open source | Fiji (http://fiji.sc/#download) | IMOD, MIB (See text for refferences) |
参考文献
- Luckner, M., Wanner, G. From light microscopy to analytical scanning electron microscopy (SEM) and focused ion beam (FIB)/SEM in biology: Fixed coordinates, flat embedding, absolute references. Microscopy and Microanalysis. 24 (5), 526-544 (2018).
- White, J. G., Southgate, E., Thomson, J. N., Brenner, S. The structure of the nervous system of the nematode Caenorhabditis elegans. Philosophical Transaction of Royal Society of London B Biological Sciences. 314 (1165), 1 (1986).
- Zheng, Z., et al. A complete electron microscopy volume of the brain of adult Drosophila melanogaster. Cell. 174 (3), 730-743 (2018).
- Mulcahy, B., et al. A pipeline for volume electron microscopy of the caenorhabditis elegans nervous system. Frontiers in Neural Circuits. 12, 94 (2018).
- Schorb, M., Haberbosch, I., Hagen, W. J. H., Schwab, Y., Mastronarde, D. N. Software tools for automated transmission electron microscopy. Nature Methods. 16 (6), 471-477 (2019).
- Baumeister, W., Grimm, R., Walz, J. Electron tomography of molecules and cells. Trends in Cell Biology. 9 (2), 81-85 (1999).
- Hoog, J. L., et al. Organization of interphase microtubules in fission yeast analyzed by electron tomography. Developmental Cell. 12 (3), 349-361 (2007).
- Weber, M. S., Wojtynek, M., Medalia, O. Cellular and structural studies of eukaryotic cells by cryo-electron tomography. Cells. 8 (1), 57 (2019).
- Micheva, K. D., Smith, S. J. Array tomography: a new tool for imaging the molecular architecture and ultrastructure of neural circuits. Neuron. 55 (1), 25-36 (2007).
- Horstmann, H., Korber, C., Satzler, K., Aydin, D., Kuner, T. Serial section scanning electron microscopy (S3EM) on silicon wafers for ultra-structural volume imaging of cells and tissues. PLoS One. 7 (4), 35172 (2012).
- Hayworth, K. J., et al. Imaging ATUM ultrathin section libraries with WaferMapper: a multi-scale approach to EM reconstruction of neural circuits. Frontiers in Neural Circuits. 8, 68 (2014).
- Kubota, Y., et al. A carbon nanotube tape for serial-section electron microscopy of brain ultrastructure. Nature Communication. 9 (1), 437 (2018).
- Wacker, I. U., et al. Multimodal Hierarchical Imaging of Serial Sections for Finding Specific Cellular Targets within Large Volumes. Journal of Visualized Experiments. (133), e57059 (2018).
- Burel, A., et al. A targeted 3D EM and correlative microscopy method using SEM array tomography. Development. 145 (12), (2018).
- Templier, T. MagC, magnetic collection of ultrathin sections for volumetric correlative light and electron microscopy. Elife. 8, 45696 (2019).
- Denk, W., Horstmann, H. Serial block-face scanning electron microscopy to reconstruct three-dimensional tissue nanostructure. PLoS Biology. 2 (11), 329 (2004).
- Knott, G., Marchman, H., Wall, D., Lich, B. Serial section scanning electron microscopy of adult brain tissue using focused ion beam milling. Journal of Neuroscience. 28 (12), 2959-2964 (2008).
- Wanner, A. A., Genoud, C., Masudi, T., Siksou, L., Friedrich, R. W. Dense EM-based reconstruction of the interglomerular projectome in the zebrafish olfactory bulb. Nature Neuroscience. 19 (6), 816-825 (2016).
- Smith, D., Starborg, T. Serial block face scanning electron microscopy in cell biology: Applications and technology. Tissue Cell. 57, 111-122 (2019).
- Kizilyaprak, C., Bittermann, A. G., Daraspe, J., Humbel, B. M. FIB-SEM tomography in biology. Methods in Molecular Biology. 1117, 541-558 (2014).
- Xu, C. S., et al. Enhanced FIB-SEM systems for large-volume 3D imaging. Elife. 6, 25916 (2017).
- Hayworth, K. J., et al. Gas cluster ion beam SEM for imaging of large tissue samples with 10 nm isotropic resolution. Nature Methods. 17 (1), 68-71 (2020).
- Maco, B., et al. Correlative in vivo 2 photon and focused ion beam scanning electron microscopy of cortical neurons. PLoS One. 8 (2), 57405 (2013).
- Russell, M. R., et al. 3D correlative light and electron microscopy of cultured cells using serial blockface scanning electron microscopy. Journal of Cell Science. 130 (1), 278-291 (2017).
- Lucas, M. S., Gunthert, M., Gasser, P., Lucas, F., Wepf, R. Bridging microscopes: 3D correlative light and scanning electron microscopy of complex biological structures. Methods in Cell Biology. 111, 325-356 (2012).
- Koga, D., Kusumi, S., Bochimoto, H., Watanabe, T., Ushiki, T. Correlative light and scanning electron microscopy for observing the three-dimensional ultrastructure of membranous cell organelles in relation to their molecular components. Journal of Histochemistry and Cytochemistry. 63 (12), 968-979 (2015).
- Peddie, C. J., et al. Correlative and integrated light and electron microscopy of in-resin GFP fluorescence, used to localise diacylglycerol in mammalian cells. Ultramicroscopy. 143, 3-14 (2014).
- Markert, S. M., et al. 3D subcellular localization with superresolution array tomography on ultrathin sections of various species. Methods in Cell Biology. 140, 21-47 (2017).
- Kolotuev, I., Schwab, Y., Labouesse, M. A precise and rapid mapping protocol for correlative light and electron microscopy of small invertebrate organisms. Biology of the Cell. 102 (2), 121-132 (2009).
- Kolotuev, I. Positional correlative anatomy of invertebrate model organisms increases efficiency of TEM data production. Microscopy and Microanalysis. 20 (5), 1392-1403 (2014).
- Kato, M., Kolotuev, I., Cunha, A., Gharib, S., Sternberg, P. W. Extrasynaptic acetylcholine signaling through a muscarinic receptor regulates cell migration. Proceedings of the National Academy of Sciences. 118 (1), e1904338118 (2021).
- Schindelin, J., et al. Fiji: An open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Miguel-Aliaga, I., Jasper, H., Lemaitre, B. Anatomy and physiology of the digestive tract of Drosophila melanogaster. Genetics. 210 (2), 357-396 (2018).
- Nikolaev, M., et al. Homeostatic mini-intestines through scaffold-guided organoid morphogenesis. Nature. 585 (7826), 574-578 (2020).
- Knoblich, J. A. Mechanisms of asymmetric stem cell division. Cell. 132 (4), 583-597 (2008).
- Daniel, E., et al. Coordination of septate junctions assembly and completion of cytokinesis in proliferative epithelial tissues. Current Biology. 28 (9), 1380-1391 (2018).
- Pasquettaz, R., et al. Peculiar protrusions along tanycyte processes face diverse neural and non-neural cell types in the hypothalamic parenchyma. Journal of Comparative Neurology. , (2020).
- Kremer, J. R., Mastronarde, D. N., McIntosh, J. R. Computer visualization of three-dimensional image data using IMOD. Journal of Structural Biology. 116 (1), 71-76 (1996).
- Cardona, A., et al. TrakEM2 software for neural circuit reconstruction. PLoS One. 7 (6), 38011 (2012).
- Belevich, I., Joensuu, M., Kumar, D., Vihinen, H., Jokitalo, E. Microscopy image browser: A platform for segmentation and analysis of multidimensional datasets. PLoS Biology. 14 (1), 1002340 (2016).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。