Method Article
大规模多组学全基因组关联研究 (Mo-GWAS):样品制备和归一化指南
摘要
在该协议中,我们提出了一个优化的工作流程,该工作流程结合了许多样品的高效快速样品制备。此外,我们还提供分步指南,以减少代谢GWAS研究高通量评估的分析变异。
摘要
气相色谱-质谱(GC-MS)和液相色谱-质谱(LC-MS)都是广泛用于检测和量化数十万种代谢物特征的代谢组学方法。然而,将这些技术应用于大量样品会受到更复杂的相互作用的影响,特别是对于全基因组关联研究(GWAS)。该协议描述了一种优化的代谢工作流程,它将高效快速的样品制备与豆科植物物种的大量样品分析相结合。这种稍加修饰的提取方法最初是为分析植物和动物组织而开发的,并且基于甲基叔丁基醚的提取:甲醇溶剂,以允许捕获极性和脂质代谢物。此外,我们还为减少分析变异提供了分步指南,这对于GWAS代谢变异的高通量评估至关重要。
引言
大规模的"组学"方法使复杂生物系统1,2,3的分析成为可能,并进一步了解基因型与所得表型4之间的联系。使用超高效液相色谱-质谱(UHPLC-MS)和GC-MS的代谢组学能够检测出过多的代谢物特征,其中只有一些被注释到一定程度,导致高比例的未知代谢物。通过将大规模代谢组学与不同人群的潜在基因型变异相结合,可以探索复杂的相互作用5。然而,处理大型样本集本质上与分析变异有关,扭曲了进一步下游过程的代谢方差评估。具体而言,导致分析变化的主要问题基于机器性能和仪器随时间的变化漂移6。在分析大规模结构化植物种群时,批次间变异的整合具有挑战性,尤其成问题。建议采用多种归一化程序来纠正非生物变异,例如,使用内部、外部和同位素标记的内部标准来纠正分析错误,其中每个标准都与已知问题和陷阱固有关联7,8,9,10。
除了分析变化之外,提取方案的选择通常因分析方法而异。最终,希望通过执行基于相分离的提取方法,降低材料和劳动力成本,以及将同一样品的多个等分试样用于各种分析过程的必要性。这些方法首先使用氯仿引入:甲醇/水溶剂来分馏极性和疏水性化合物11。
该协议描述了一种用于多组学平台的快速高通量管道,用于分析豆科植物物种中的极性代谢物和脂质。此外,它显示了如何在整合基因型信息以通过执行GWAS检测代谢物数量性状位点(QTL)之前,如何针对分析变异适当地校正这些数据集并进行归一化。
研究方案
1. 实验设计与植物栽培
注意:根据实验假设设置实验,例如,使用大规模GWAS群体可以减少多次重复的必要性,因为统计测试将基于所有单个SNP的单倍型而不是加入进行。相比之下,在其他实验方法中,多个重复是必不可少的。在准备实验时必须考虑以下几点。
- 包括足够的生物学重复,这取决于实验假设。
- 块状随机化生物重复,以减少栽培过程中的局部环境偏差,例如温室,田地。
- 确保在生长过程中对植物进行适当的维护。均匀处理植物以减少偏倚。
2. 生物植物材料的制备
- 收获准备
- 标签收集管 (20 mL),包含两个直径为 5 mm 和两个 8 mm 的金属珠,用于均质化。用液氮填充杜瓦瓶。
注意:植物应处于新鲜叶子和根组织收获的营养阶段。
- 标签收集管 (20 mL),包含两个直径为 5 mm 和两个 8 mm 的金属珠,用于均质化。用液氮填充杜瓦瓶。
- 通过在液氮中速冻来收获生物样品。尽快收获以排除昼夜节律振荡对长期收获期间代谢的影响12,13。储存收获的新鲜叶子和根组织,以便在-80°C下进一步处理。
注意:切叶到速冻不应超过几秒钟,因为在叶子切割后,活跃的生物过程会因伤口而改变代谢谱。对于根部,在液氮中速冻之前,用水洗涤来预清洁根部。根部表面的多余水分应用纸巾吸收。干燥的种子可以在室温下储存;无需在液氮中冷冻。 - 使用生活用混合机研磨纸巾。
- 将管架在液氮中预冷几分钟,以在研磨组织时保持低温。
- 从-80°C冰箱中取出生物样品后,用含氮杜瓦瓶运输生物样品。
- 研磨组织以获得均匀的粉末;使用25 Hz 1分钟,如果组织未均匀研磨,则在液氮中冷冻后重复。
- 要研磨干燥的种子,请将种子放入直径为15毫米的金属珠的研磨罐中。使用 2.3.3 中提到的相同频率和时间。
注意:如果没有纸巾搅拌机,可以使用清洁和预冷的研钵和杵。 - 预冷标记的 2 mL 安全锁定微量离心管。使用分析秤称量50毫克,误差为±5毫克新鲜植物材料。将用于在液氮中转移植物材料的工具预冷。确保植物材料在称重过程中保持冷冻状态。
注意:不要将新鲜植物材料暴露在室温下太长时间,因为生物过程会因温度升高而激活,从而改变代谢特征14。 - 通过将每个样品的一定比例混合并称量50mg,将混合的新鲜植物材料混合到预冷的2 mL安全锁定微量离心管中,产生额外的质量控制(QC)样品,误差为±5mg。
注意:建议每60个样品至少提供三个QC样品。QC样品对于下游校正、归一化和分析至关重要。
3. 萃取试剂
- 新鲜组织,例如叶子和根
注意:样品提取基于先前描述的协议15。该协议已根据当前需求进行了修改,例如,多个组织,不同的内部标准和大规模实验。此外,下面提到的所有体积和仪器设置都根据内部分析单元进行调整。协议用户应根据其分析单元和生物样品,根据测试样品调整这些内容。- 萃取混合物 1 (EM1):甲基 叔丁基醚/甲醇 (3:1 v/v)
- 以3:1的比例制备甲基叔丁氧基乙烷/甲基化硅的混合物。对于 100 mL 萃取溶剂,将 75 mL 甲基叔丁氧混合料与 25 mL MeOH 混合在干净的玻璃瓶中。
注意:溶剂应在通风橱中小心处理,并配备适当的安全设备。 - 加入45μL1,2-二绦十六烷酰基-sn-甘油-3-磷胆碱(1mg / mL氯仿)作为基于UHPLC-MS的脂质分析的内部标准,400μL核糖醇(1mg / mL在水中)作为基于GC-MS的分析的内标,以及125μL异维辛(1mg / mL在MeOH /水(1:1 v / v)中)用于基于UHPLC-MS的代谢物分析。
注意:根据分析需要,添加内部标准对于分析后归一化是必要的。由于每个样品需要1 mL EM1,因此根据实验样品大小制备储备溶液,该溶液应用于整个实验。EM1必须储存在-20°C。 检查是否存在使用过的内标,以及与所研究物种中的其他化合物重叠。可以使用多种标准;该协议中内部标准的选择基于先前使用普通豆提取物16的测试。
- 以3:1的比例制备甲基叔丁氧基乙烷/甲基化硅的混合物。对于 100 mL 萃取溶剂,将 75 mL 甲基叔丁氧混合料与 25 mL MeOH 混合在干净的玻璃瓶中。
- 萃取混合物 2 (EM2) 水/ 甲醇 (3:1 v/v)
- 对于 100 mL EM2,在干净的玻璃瓶中加入 75 mL 双蒸馏水和 25 mL MeOH。
- 每个样品加入500μLEM2,并根据实验样品量制备储备溶液,该储备溶液应用于整个实验。将EM2储存在4°C。
- 萃取混合物 1 (EM1):甲基 叔丁基醚/甲醇 (3:1 v/v)
- 干燥的种子
- 萃取混合物 3 (EM3) 甲醇 /水 (7:3 v/v)
- 对于100 mL的EM3,在干净的玻璃瓶中加入70 mL的MeOH和30 mL的双蒸馏水。为每个样品准备1 mL EM3。
- 加入400μL核糖醇(1mg / mL在水中)作为GC-MS分析的内部标准品,并加入125μL异牡荆素(1mg / mL在MeOH /水中(1:1 v / v))用于基于UHPLC-MS的代谢物分析。
注意:根据实验样品量准备储备溶液,并将其用于整个实验。将EM3储存在4°C。
- 萃取混合物 3 (EM3) 甲醇 /水 (7:3 v/v)
4. 样品提取
- 新鲜组织,例如叶子和根
- 为每个样品准备三个1.5 mL安全锁定微量离心管。将 EM1 保存在 -20 °C 的液体冷却系统中。将新鲜样品从-80°C冰箱转移到干冰或液氮中运输。向每 50 mg 等分试样中加入 1 mL 预冷 EM1,并在保持冰上之前短暂涡旋。
- 将样品在轨道振荡器上以800× g 在4°C下孵育10分钟。
- 将样品在冰冷的超声处理浴中超声处理10分钟。
- 使用多通道移液器加入 500 μL EM2,以避免增加体积的变化。
- 在11,200× g 下在4°C下离心5分钟之前,将样品短暂涡旋以混合提取混合物。
- 相分离后,将500μL上含脂相转移到预标记的1.5mL安全锁定微量离心管中。删除上部相位的其余部分。
注意:转移时要小心,因为这个上层相具有很高的蒸气压,并且容易从移液器中泄漏出来。 - 将150μL和300μL含有下极性和半极性代谢物的相分别转移到两个用于GC-MS和UHPLC-MS分析的1.5 mL安全锁定微量离心管中。
- 通过使用真空浓缩器让溶剂蒸发而不加热来浓缩所有提取的馏分,并储存在-80°C。
- 干燥的种子
- 为每个样品准备两个1.5 mL安全锁定微量离心管。将 EM3 保存在冰上。将直径为5 mm的金属珠放入样品等分试样中。
- 在每50mg等分试样中加入1mL EM3,并在25 Hz下将样品均质化2-3分钟,然后将其放在冰上。
- 将样品在冰冷的超声处理浴中超声处理10分钟。
- 在4°C下以11,200× g 离心5分钟之前,将样品短暂涡旋。
- 将 150 μL 和 300 μL 上清液分别转移到两个 1.5 mL 安全锁定微量离心管中,分别用于 GC-MS 和 UHPLC-MS 分析。
- 通过使用真空浓缩器让溶剂蒸发而不加热来浓缩所有提取的馏分,并储存在-80°C。
注意:根据经验,建议用户对干燥种子中的半极性代谢物和衍生化代谢物进行步骤4.2分析。执行提取步骤4.1以进行干燥的种子脂质分析。
5. 使用超高分子色谱-MS分析脂质
- 将干燥的脂质级分重新悬浮在250μL乙腈:2-丙醇(7:3,体积/体积)中。
- 超声处理脂质相5分钟,以11,200× g 离心1分钟。
- 将90μL上清液转移到玻璃小瓶中以进行LC-MS。
- 将2μL提取物注入LC-MS中。
- 在保持在60°C的反相C8柱上进行脂质分馏,流量为400μL / min,淋洗液A和B逐渐变化,如 表1所示。在质量范围为150-1,500 m / z的正电离模式下获取质谱。
- 在所有日常批次中包括多个QC样品和一个空白样品,以确保对分析变化的校正。按顺序逐块随机化样本。
6. 使用 UHPLC-MS 分析极性和半极性代谢物
- 将干燥的极相重新悬浮在180μL UHPLC级甲醇:水(1:1 v / v)中。
- 将极相超声处理2分钟,以11,200× g 离心1分钟。
- 将90μL上清液转移到玻璃小瓶中以进行LC-MS。
- 将3μL提取物注入LC-MS中。
- 在逆相C18柱上进行代谢物分馏,保持在40°C下,以400μL / min的流量运行,淋洗液A和B逐渐变化,如 表1所示。在全MS扫描中,在100-1,500 m / z 的质量范围内获取质谱,并由40 keV的高能碰撞解离(HCD)诱导的所有离子碎片(AIF)。
注:同时使用两种电离模式。然而,由于在运行大量样品时容量有限,因此在两种电离模式下运行测试样品以确定首选的电离模式。 - 在所有日常批次中包括多个QC样品和一个空白样品,以确保对分析变化的校正。按顺序逐块随机化样本。
- 在负电离和正电离模式下,在依赖于数据的 MS2 中运行池化 QC。在后面的步骤(8.5)中使用获得的质谱进行注释。
7. 使用GC-MS17,18分析衍生化代谢物
注意:衍生化代谢物的分析基于先前描述的方案17。处理通风橱中的所有衍生化试剂。确保 N-甲基-N-(三甲基硅基)三氟乙酰胺 (MSTFA) 不与水和湿度接触。
- 衍生化试剂 1 (DR1)
- 将甲氧基胺盐酸盐溶解在吡啶中,以获得浓度为30mg / mL的DR1。每个样品使用40μLDR1。根据样品大小准备储备溶液,并在室温下储存。
- 衍生化试剂 2 (DR2)
- 将 MSTFA 与 20 μL 脂肪酸甲酯 (FAME) 溶解每 1 mL MSTFA。每个样品使用70μLDR2。根据样品量准备储备溶液。将药明纤维物质储存在4°C,将氟甲基目镜储存在-20°C。
注:FAME包括甲基辛酸酯、天竺酸甲酯、癸酸甲酯、月桂酸甲酯、肉豆蔻酸甲酯、棕榈酸甲酯、硬脂酸甲酯、二十二烷酸甲酯、木质素酸甲酯、己二十烷酸甲酯、十八烷酸甲酯和三十一烷酸甲酯,它们分别以0.8 μL/mL或0.4 mg/mL的浓度溶解在CHCl3 中,用于液体或固体标准品。
- 将 MSTFA 与 20 μL 脂肪酸甲酯 (FAME) 溶解每 1 mL MSTFA。每个样品使用70μLDR2。根据样品量准备储备溶液。将药明纤维物质储存在4°C,将氟甲基目镜储存在-20°C。
- 使用真空浓缩器将极相(储存在-80°C)中的沉淀重新干燥30分钟,以避免在储存期间与用于下游衍生化的溶剂产生H2O的任何干扰。
- 加入40微升DR1。
- 使用轨道振荡器在37°C下以950× g 摇动样品2小时,然后对液体进行短时间旋转。
- 加入 70 μL DR2。
- 使用轨道振荡器在37°C下以950× g 再次摇动30分钟。
- 在室温下短暂离心,然后将90μL转移到玻璃瓶中进行GC-MS分析。
- 根据代谢物浓度,将1μL注射至GC-MS无分裂模式,恒定的氦载气流为2 mL / min。使用 30-m 的 MDN-35 毛细管柱将注射温度设置为 230 °C。
注:其他信息,例如温度梯度,可在 表1中找到。质量范围设置为 70-600 m/z,扫描速度为 20 次/分钟。包括拆分模式,以便能够对推定的重载化合物进行定量,从而在这种情况下节省提取物再衍生化的成本和时间。 - 在所有日常批次中包括多个QC样品和一个空白样品,以确保对分析变化的校正。按顺序正确随机化样本。
8. 色谱图处理和化合物注释
- 通过定义强度阈值来过滤化学噪声。在处理色谱图时包括所有QC样品。
注意:对于大规模数据,噪声滤波对于减少计算时间和处理能力至关重要。 - 通过定义保留时间偏移窗口来对齐色谱图。检查每个批次的色谱图,以评估批次内和批次间的变化。
- 根据峰值形状执行峰值检测,例如,在半最大值 (FWHM) 计算下全宽的高度和宽度。
- 对同位素进行聚类以减少冗余信号并滤除单例。
注:有关用于色谱图处理的软件的详细信息,请参阅材料表。提供了有关如何使用各种免费提供的软件工具(例如 MS-DIAL、梅特阿利恩、MzMine 和 Xcalibur 19、20、21)处理色谱图的深入实验方案。 - 使用合并的 QC 样品的 ddMS2 数据进行化合物注释。通过确定单同位素质量并观察常见的中性损失,已知的带电激越和不同类型的裂解(例如,同源或异质解16,22)来评估分子结构。
- 对于报告代谢物数据,请遵循Fernie等人201123中描述的建议。
注意:可以使用不同的计算代谢组学方法来分析代谢组学数据24,25,26。
9. 大规模代谢组学数据集的归一化
- 检查内部标准的分布,并通过校正单个或多个内部标准的响应进行归一化。
- 通过将峰强度除以步骤2.5中等分的均质化样品重量,校正从色谱图获得的峰强度超过精确的样品重量。
- 校正多批次系列的强度漂移。使用 R 执行基于 QC 的校正方法,例如局部估计散点图平滑 (LOESS)27。
注意:有几种工具和软件包可用于解决整个批次28,29采集过程中MS性能的漂移问题。 - 通过数据转换确保性状的正态分布,例如,使用 R 包 MASS 中的箱子考克斯 () 函数执行 GWAS 的箱-考克斯变换30。
- 执行数据缩放,例如帕累托缩放,用于多变量分析,以确保正确称量低丰度化合物31。
注意:如果可能,进行回收测定以避免基质效应,例如,离子抑制14。
10. 全基因组关联研究 (GWAS)32
- 从测序数据33,34中调用单核苷酸多态性(SNP)或结构变体(SV)。
- 过滤小等位基因频率(MAF)的基因型数据<5%,缺失率为>10%,以避免使用Talssel35的低频偏倚。
- 使用 R 包 Ime436 计算实验重复中每个归一化特征的最佳线性无偏预测 (BLUP),以消除源自环境因素(随机效应)的偏差。
- 单独使用每个功能的 BLUP,以使用 R37 中的 rMVP 包执行 GWAS。
注意:每个代谢组学特征在这里被视为一个单独的独立表型。 - 在执行 GWAS 时,使用主成分分析 (PCA) 和按州同一性 (IBS) 或 vanRaden 纠正种群结构,以最大程度地减少混杂效应。此外,请考虑使用混合线性模型 (MLM) 或多轨迹混合模型 (MLMM),因为混合模型包含固定和随机效应。
11. 质量检测
- 在考虑曼哈顿图的情况下,检查显示显著关联的 SNP,以进行连锁不平衡 (LD) 计算以确定潜在的遗传区域。使用 R 封装 LD 热图 或流苏 5 执行 LD 计算。
- 通过检查单倍型之间统计变化的性状水平来检查相关性SNP的效应大小,以找到潜在的因果SNP,例如,SNP导致蛋白质编码序列中的氨基酸变化,这可以解释表型变异。
注意:由于 SNP性状关联不一定产生因果关联,因此确定基因组区域至关重要。通过特征注释进行化合物鉴定可以极大地帮助在特定基因组区域中找到正确的候选基因。我们建议在多效性图谱中结合所有检测到的与某些化合物相关的QTL,以强调遗传区域38,如图 4所示。为了验证候选基因,可以采取几种方法(参见讨论)。
结果
成功的代谢组学GWAS实验应从适当的实验设计开始,然后是样品收集,提取,数据采集和处理,如图 1所示。在该协议中,MTBE方法15用于提取和分析属于几种化合物类别的数百种代谢物。色谱高度依赖于所用色谱柱以及洗脱缓冲液混合物的性质。图2显示了QC样品的色谱图,表明该分析系统中一些主要脂质类别的洗脱模式。 表 1 给出了每个平台应用的梯度。重点放在处理大规模实验中的系统性错误上。进行大规模代谢组学与全身性错误有内在关联。为了进行演示,我们分析了几种常见豆类的脂质组学数据。 补充表 1提供了使用 材料表中指示的软件进行色谱图处理后提取的原始脂质组数据。遵循该协议使我们能够规避处理组学数据的主要问题,特别是在处理大型样本集时。归一化过程可以精确地校正批次分析误差,如图 3所示。虽然增加QC样品的数量会增加归一化的力量,但由于成本和时间的限制,这并不总是可行的。对于具有非靶向代谢特征的高通量代谢组学GWAS,必须适当地说明更多数量的性状- 标志物关联。结合多个GWAS结果的多效性图谱38可用于突出显示与多个性状相关的基因组区域(图4)。
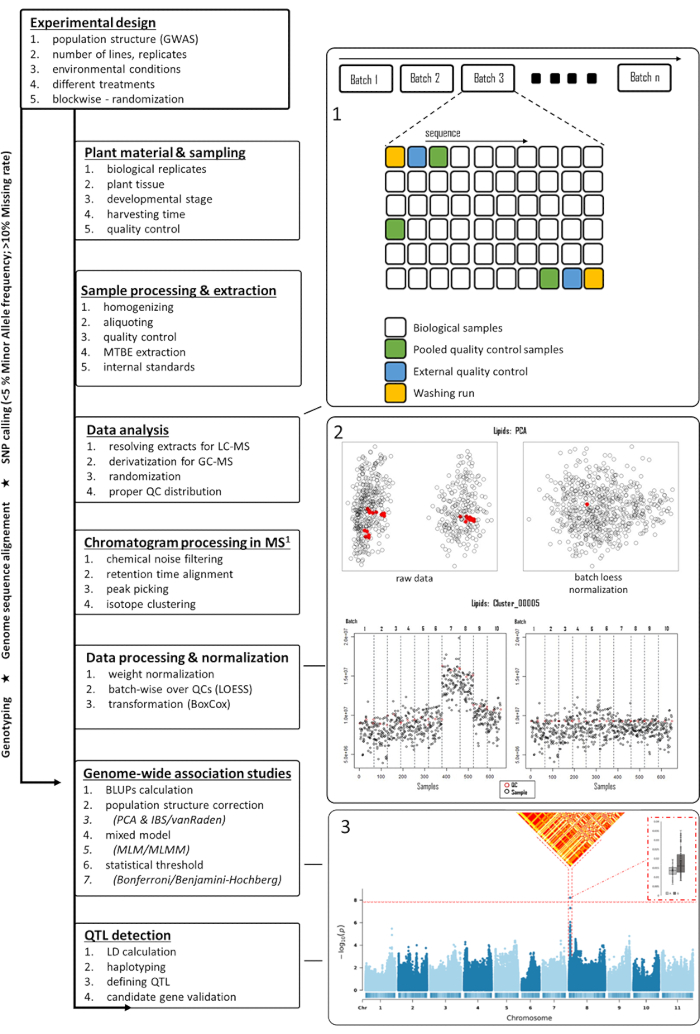
图1:植物中基于代谢组学的GWAS的流程图。 从实验设计到QTL检测的几个步骤显示在左侧面板中。在右侧面板中,显示了多个图形以支持左侧面板中提到的几个步骤。从右上方开始, (1) 显示了LC-MS的建议样本序列, (2) PCA的归一化前和归一化后评分图,包括具有代表性的特征分布预处理和后处理,红色表示QC样本强度,以及 (3) 与LD和单倍型分布具有显着关联的曼哈顿图。缩写:GWAS =全基因组关联研究;QTL = 数量性状位点;PCA = 主成分分析;质量控制= 质量控制;LD = 联动不平衡;MS = 质谱;LC-MS = 液相色谱-质谱;GC-MS = 气相色谱-质谱;黄土 = 局部估计散点图平滑;MLM/MLMM = 混合线性模型/多轨迹混合模型。 请点击此处查看此图的大图。
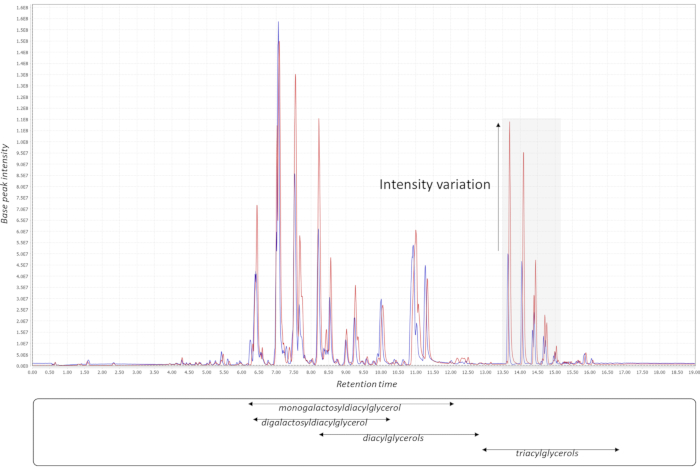
图2:色谱图处理。 来自不同批次的两个QC色谱图(碱基峰,脂质数据)证明了合并QC样品中某些脂质类别的批次变化。四种主要的脂质类别在内部LC-MS系统中用各自的洗脱窗口表示。色谱图是从MzMine21导出的。缩写:QC =质量控制;LC-MS = 液相色谱-质谱。 请点击此处查看此图的大图。
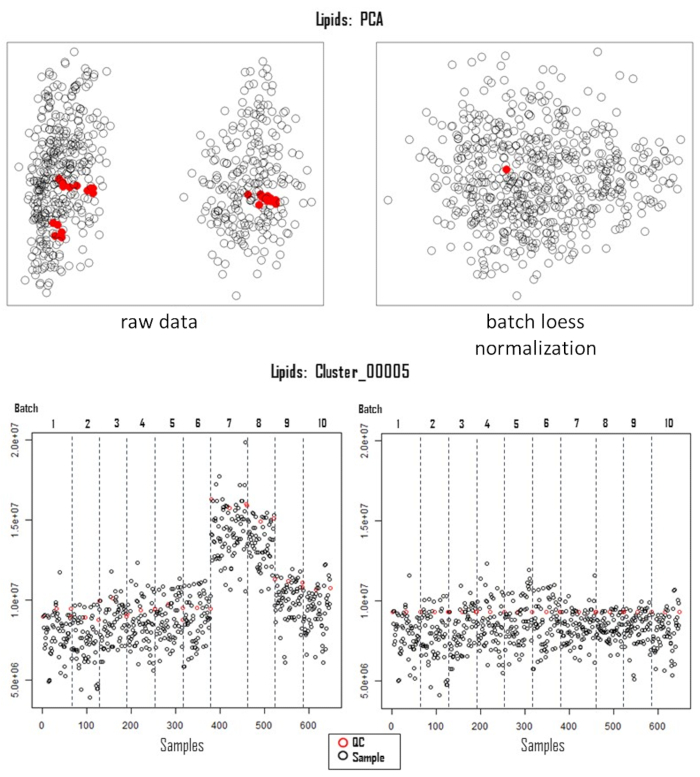
图 3:系统误差的校正。 对采集的脂质组数据进行主成分分析,对系统误差进行前期(左图,原始数据)和校正后(右图,批次黄土)。下图说明了样品 (n=650) 和批次 (n=10) 在分析变异校正前(左)和后(右)校正上的特征(Cluster_00005)分布。缩写:PCA = 主成分分析;质量控制= 质量控制;黄土 = 局部估计的散点图平滑。 请点击此处查看此图的大图。
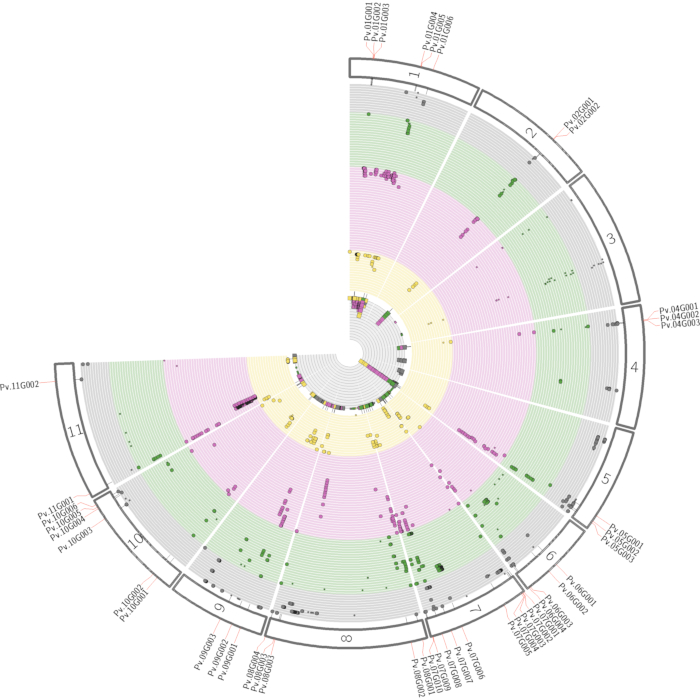
图 4:多效性图,说明 GWAS 综合结果。 多效性图突出显示了整个基因组中与几个性状相关的区域。外环上的数字表示相应的染色体。每个圆圈代表一个具有显著相关性 SNP 的个体特征。颜色代表不同的化合物类别(灰色=化合物类别1;绿色=化合物类别2;紫色=化合物类别3;黄色=化合物类别4)。在具有相同基因组区域的化合物类间关联的情况下,突出显示基因。内侧的灰色圆圈显示了与特定基因组位置相关的所有重要SNP的总和。此图中显示的关联是人为生成的,仅用于说明。缩写:GWAS =全基因组关联研究;SNP = 单核苷酸多态性。 请点击此处查看此图的大图。
| 脂质的超高密度脂蛋白-MS 设置 | ||||
| 时间 [分钟] | 淋洗液 A 至 B [%]* | 信息 | ||
| 0 - 1.00 | 45% 一 | 淋洗液A:1%1M NH4-乙酸盐,0.1%乙酸在水中(UHPLC级) | ||
| 1.00 - 4.00 | lg 45% - 25% A | 淋洗液 B:1% 1M NH4-乙酸盐,0.1% 乙酸乙腈/2-丙醇 7:3(UHPLC 级) | ||
| 4.00 - 12.00 | 25% - 11% A | 流速: 400 μL/分钟 | ||
| 12.00 - 15.00 | lg 11% - 0% A | 注射量: 2 μL | ||
| 15.00 - 19.50 | 连续波 0% A | |||
| 19.50-19.51 | 0% - 45% A | |||
| 19.51-24.00 | 当量 45% | |||
| 极性和半极性代谢物的超高纯化循环-MS/MS 设置 | ||||
| 时间 [分钟] | 淋洗液 A 和 B [%]* | 信息 | ||
| 0 - 1.00 | 99% A | 淋洗液A:水中0.1%甲酸(超高纯碱级) | ||
| 1.00 - 11.00 | 99% -60% A | 淋洗液 B:乙腈中 0.1% 甲酸(UHPLC 级) | ||
| 11.00 - 13.00 | lg 60% - 30% A | 流速: 400 μL/分钟 | ||
| 13.00 - 15.00 | lg 30% - 1% A | 注射量: 3 μL | ||
| 15.00 - 16.00 | 连续 1% A | |||
| 16.00 - 17.00 | LG 1% - 99% A | |||
| 17.00 - 20.00 | 当量 99% A | |||
| 衍生化代谢物的 GC-MS 设置 | ||||
| 时间 [分钟] | 温度 [°C] | 信息 | ||
| 0 - 2.00 | 85 | 载气: 氦 | ||
| 2.00 - 18.66 | lg 80 - 330 | 流速: 2 毫升/分钟 | ||
| 18.66 - 24.66 | 连续波 330 | 温度梯度: 15°C/分钟 | ||
| 24.66 | 快速冷却 | 注射量: 1 μL | ||
表 1:每个分析平台的梯度设置7. 缩写:lg = 线性梯度;cw = 柱洗;等式 = 平衡;超高效液相色谱-质谱= 超高效液相色谱-质谱;超高效液相色谱-MS/MS = 超高效液相色谱-串联质谱;GC-MS = 气相色谱-质谱。* = 百分比值对应于淋洗液 A;剩余百分比值对应于淋洗液B。
补充表1:原始脂质组学数据。 指示每个样本上每个检测到的簇的峰值强度。 请按此下载此表格。
讨论
GC-MS和LC-MS都是广泛用于分析各种代谢物类别的复杂混合物的工具。使用这些工具处理大型数据集固有地与非生物变异(例如,分析变异)相关联,这会干扰和偏倚结果的解释。该协议为全面的代谢分析提供了一个强大且高通量的提取管道,以消除非生物来源的变化并进行大规模的"组学"研究。该方案中使用的体积和浓度针对不同组织中的豆科植物物种进行了调整。然而,这些参数可以稍作修改,并用于来自其他植物物种的大规模代谢样品。
前面描述的15种基于MTBE的提取物可用于分析衍生化代谢物,半极性代谢物和脂质。这可以扩展到蛋白质和植物激素提取39,这超出了本方案的范围。其它萃取方案依赖于二氯甲烷:乙醇混合物40,41。在这些提取方案中,MTBE:甲醇提取方案为现有的基于氯仿的提取方案42提供了有利且危害较小的替代方案,并且不会导致蛋白质沉淀作为极相和脂质相之间的中间相。此外,甲基叔丁基醚方法已在若干研究中用于各种生物样品43,44,45。
该协议讨论了在处理大量样品时可能导致潜在变异的几个关键步骤,例如,在收获过程中12,13,提取14以及随机化46。此外,本方案中尚未讨论的其他问题必须考虑以确保高质量的代谢组学数据,例如基质效应和离子抑制14。
基于QC的归一化方法的强大功能本身取决于每个批次中的QC样品数量。如前所述,尽管增加数量会增加功率,但与这些分析系统中的批次间变化相比,QCs的批次内变化相对较小,如图 3所示。总体而言,还有其他基于 QC 的归一化方法,例如使用随机森林 (SERRF) 的系统误差消除,这些方法已被证明优于大多数其他归一化方法,例如批量比率、使用多个内部标准的最优选择进行归一化 (NOMIS) 和概率商归一化 (PQN)47.然而,SERRF依赖于每批中的多个QC样品,例如,每十分之一的样品,这在处理大量样品时是不可行的。与其他数据驱动或基于内部标准的方法相比,基于QC的归一化的主要优点是它保留了基本的生物学变异,同时适应了不需要的技术变异28。读者可以参考本综述,了解变体28的处理方式。
GWAS的一个主要问题是假阳性率,这主要是由于因果和非因果站点的联系而产生的 48,49。其次,保守的统计校正方法,例如Bonferroni和FDR,对独立测试的数量是正确的,由于近端SNP之间的联系,这不等于GWAS中测定的SNP的数量50,51因此,独立测试的实际数量通常较低。降低保守统计阈值的另一种方法是根据定义的基因组区域上的连锁衰减来减少用于GWAS的测试SNP的数量52。本方案中描述的GWAS集成的高通量代谢组学平台具有广泛的应用。特别是,它将通过改变代谢物/脂质组成来促进作物育种的改进,使其达到工业和营养所需的水平。总体而言,代谢组学提供了对过去几十年中作物驯化过程中发生的大量代谢物和代谢多样化的遗传结构的深入见解,表明代谢组学相关育种的巨大潜力53。用于下游QTL验证的分子生物学方法包括生成CRISPR / Cas9突变品系54,T-DNA插入线55,稳定和/或瞬时过表达线56,VIGS, 离体 代谢组学方法57,仅次于生成交叉F2群体的传统方法以及不同群体中的交叉验证。
通过对上述分析变异进行必要的校正,除了GWAS之外,还可以执行几种综合方法,例如代谢物 - 代谢物,代谢物 - 脂质相关分析,与表型组数据的相关性分析以阐明更复杂的性状,和/或共表达分析以进一步解开生物系统的基础58。
披露声明
作者没有利益冲突需要声明。
致谢
医学博士得到了IMPRS-PMPG"初级代谢和植物生长"的支持。A.R.F.和SA.感谢欧盟地平线2020研究与创新计划、PlantaSYST项目(FPA No. 664620下的SGA-CSA第739582号)和项目"增加"(GA 862862)的财政支持。
材料
| Name | Company | Catalog Number | Comments |
| Reagents and standards | |||
| 1,2-diheptadecanoyl-sn-glycero-3- phosphocholine (17:0 PC) | Avanti Polar Lipids | 850360P | Internal standard for lipids |
| Chloroform | Supleco | 67-66-3 | FAME solvent |
| Isovitexin | Sigma Aldrich | 38953-85-4 | Internal standard for metabolites |
| Lignoceric Acid Methylester | Sigma Aldrich | 2442-49-1 | FAME |
| Methanol (MeOH) | Biosolve Chemicals | 13684102 | ULC-MS grade |
| Methoxyamin -hydrochlorid | Sigma Aldrich | 593-56-6 | Metabolite deriviatization |
| Methyl laurate | Sigma Aldrich | 111-82-0 | FAME |
| Methyl myristate | Sigma Aldrich | 124-10-7 | FAME |
| Methyl palmitate | Sigma Aldrich | 112-39-0 | FAME |
| Methyl stearate | Sigma Aldrich | 112-61-8 | FAME |
| Methyl tert-butyl ether (MTBE) | Biosolve Chemicals | 13890602 | HPLC grade |
| Methyl-caprat | Sigma Aldrich | 110-42-9 | FAME |
| Methylcaprylat | Sigma Aldrich | 111-11-5 | FAME |
| Methyldocosanoat | Sigma Aldrich | 929-77-1 | FAME |
| Methyleicosanoat | Sigma Aldrich | 1120-28-1 | FAME |
| Methyl-hexacosanoat | Sigma Aldrich | 5802-82-4 | FAME |
| Methyl-octacosanoat | Sigma Aldrich | 55682-92-3 | FAME |
| Methyl-pelargonate | Sigma Aldrich | 1731-84-6 | FAME |
| N-Methyl-N-(trimethylsilyl)trifluoracetamid (MSTFA) | Macherey-Nagel | 24589-78-4 | Metabolite deriviatization |
| Pyridine | Supleco | 110-86-1 | Metabolite deriviatization |
| Ribitol | Supleco | 22566-17-2 | Internal standard for derivatized metabolites |
| Triacontanoic Acid Methyl Ester | TCI Chemicals | 629-83-4 | FAME |
| Water | Biosolve Chemicals | 23214102 | ULC-MS grade |
| Equipment | |||
| 1.5 mL Safe-lock microcentrifuge tubes | Eppendorf | 3120086 | |
| 2 mL Safe-lock microcentrifuge tubes | Eppendorf | 3120094 | |
| Balance | Sartorius Corporation | 14 557 572 | |
| DB-35ms, 30 m, 0,25 mm, 0,25 µm | Aglient | 123-3832 | Analysis of derivatized metabolites |
| GC-MS system | Leco Pegasus HT TOF-MS (LECO Corporation) | Analysis of derivatized metabolites | |
| Grinding Balls, Stainless Steel | OPS DIAGNOSTICS | GBSS 196-2500-10 | |
| MS system | Exactive, Orbitrap-type, MS (Exactive, Thermo Fisher Scientific) | Analysis of lipids | |
| MS system | Q Exactive Focus (Q Exactive™ Focus Hybrid Quadrupol-Orbitrap™ Massenspektrometer, Thermo Fisher Scientific) | Analysis of metabolites | |
| Refrigerated microcentrifuge | Eppendorf, model 5427R | 22620701 | |
| Reversed Phase (RP) Bridged Ethyl Hybrid (BEH) C8 column (100 mm × 2.1 mm containing 1.7 μm diameter particles) | Waters | 186002878 | Analysis of lipids |
| RP High Strength Silica (HSS) T3 column (100 mm × 2.1 mm containing 1.8 μm diameter particles) | Waters | 186003539 | Analysis of metabolites |
| Shaker | Eppendorf Thermomixer 5436 | 2050-100-05 | |
| Sonicator | USC 300 TH | 142-0084 | |
| Tissue grinding mixer mill | Retsch, Mixer Mill MM 300 | 20.746.0001 | |
| UPLC system | Waters Acquity UPLC system (Waters) | ||
| Vacuum concentrator | Scan Speed Maxi Vac Alpha Evaporators | 7.008.500.002 | |
| Vortex mixer | Vortex-Genie 2, Model G560 | SI-0236 | |
| Software | |||
| MetAlign | Chromatogram processing | ||
| MzMine | Chromatogram processing | ||
| R package "data.table" | |||
| R package "fujiplot" | pleiotrpoic map | ||
| R package "genetics" | |||
| R package "Ime4" | BLUPs calculation | ||
| R package "LDheatmap" | LD plots | ||
| R package "MASS" | transformation | ||
| R package "rMVP" | GWAS | ||
| R version 4.0.4 | |||
| RefinerMS | Chromatogram processing | ||
| RefinerMS Genedata | Expressionist | Chromatogram processing | |
| Tassel 5 | Genotype filtering | ||
| Xcalibur | Thermo Fisher Scientific | OPTON-30965 | Chromatogram processing |
参考文献
- Doerr, A. Global metabolomics. Nature Methods. 14 (1), 32 (2017).
- Fessenden, M. Metabolomics: Small molecules, single cells. Nature. 540 (7631), 153-155 (2016).
- Oliver, S. G., Winson, M. K., Kell, D. B., Baganz, F. Systematic functional analysis of the yeast genome. Trends in Biotechnology. 16 (9), 373-378 (1998).
- Fiehn, O. Metabolomics-the link between genotypes and phenotypes. Plant Molecular Biology. 48 (1), 155-171 (2002).
- Wu, S., et al. Mapping the Arabidopsis metabolic landscape by untargeted metabolomics at different environmental conditions. Molecular Plant. 11 (1), 118-134 (2018).
- Sysi-Aho, M., Katajamaa, M., Yetukuri, L., Orešič, M. Normalization method for metabolomics data using optimal selection of multiple internal standards. BMC Bioinformatics. 8 (1), 93 (2007).
- Chen, M., Rao, R. S. P., Zhang, Y., Zhong, C. X., Thelen, J. J. A modified data normalization method for GC-MS-based metabolomics to minimize batch variation. SpringerPlus. 3 (1), 439 (2014).
- Dunn, W. B., et al. Metabolic profiling of serum using Ultra Performance Liquid Chromatography and the LTQ-Orbitrap mass spectrometry system. Journal of Chromatography B. 871 (2), 288-298 (2008).
- Fiehn, O., et al. Metabolite profiling for plant functional genomics. Nature Biotechnology. 18 (11), 1157-1161 (2000).
- vander Kloet, F. M., Bobeldijk, I., Verheij, E. R., Jellema, R. H. Analytical error reduction using single point calibration for accurate and precise metabolomic phenotyping. Journal of Proteome Research. 8 (11), 5132-5141 (2009).
- Folch, J., Lees, M., Stanley, G. H. S. A simple method for the isolation and purification of total lipides from animal tissues. Journal of Biological Chemistry. 226 (1), 497-509 (1957).
- Fukushima, A., et al. Impact of clock-associated Arabidopsis pseudo-response regulators in metabolic coordination. Proceedings of the National Academy of Sciences of the United States of America. 106 (17), 7251-7256 (2009).
- Kerwin, R. E., et al. Network quantitative trait loci mapping of circadian clock outputs identifies metabolic pathway-to-clock linkages in Arabidopsis. The Plant Cell. 23 (2), 471-485 (2011).
- Tohge, T., et al. From models to crop species: Caveats and solutions for translational metabolomics. Frontiers in Plant Sciences. 2, 61 (2011).
- Salem, M., Bernach, M., Bajdzienko, K., Giavalisco, P. A simple fractionated extraction method for the comprehensive analysis of metabolites, lipids, and proteins from a single sample. Journal of Visualized Experiments:JoVE. (124), e55802 (2017).
- Tohge, T., Fernie, A. R. Combining genetic diversity, informatics and metabolomics to facilitate annotation of plant gene function. Nature Protocols. 5 (6), 1210-1227 (2010).
- Lisec, J., Schauer, N., Kopka, J., Willmitzer, L., Fernie, A. R. Gas chromatography mass spectrometry-based metabolite profiling in plants. Nature Protocols. 1 (1), 387-396 (2006).
- Osorio, S., Do, P. T., Fernie, A. R., Hardy, N. W., Hall, R. D. . Plant Metabolomics: Methods and Protocols. , 101-109 (2012).
- De Vos, R. C. H., et al. Untargeted large-scale plant metabolomics using liquid chromatography coupled to mass spectrometry. Nature Protocols. 2 (4), 778-791 (2007).
- Perez de Souza, ., Alseekh, L., Naake, S., Fernie, T., A, Mass spectrometry-based untargeted plant metabolomics. Current Protocols in Plant Biology. 4 (4), 20100 (2019).
- Pluskal, T., Castillo, S., Villar-Briones, A., Orešič, M. MZmine 2: Modular framework for processing, visualizing, and analyzing mass spectrometry-based molecular profile data. BMC Bioinformatics. 11 (1), 395 (2010).
- Watson, J. T., Sparkman, D. O. Electron Ionization. Introduction to mass spectrometry: Instrumentation, applications and strategies for data interpretation. , 315 (2007).
- Fernie, A. R., et al. Recommendations for reporting metabolite data. The Plant Cell. 23 (7), 2477 (2011).
- Treutler, H., et al. Discovering regulated metabolite families in untargeted metabolomics studies. Analytical Chemistry. 88 (16), 8082-8090 (2016).
- Wang, M., et al. Sharing and community curation of mass spectrometry data with Global Natural Products Social Molecular Networking. Nature Biotechnology. 34 (8), 828-837 (2016).
- Naake, T., Fernie, A. R. MetNet: Metabolite network prediction from high-resolution mass spectrometry data in R aiding metabolite annotation. Analytical Chemistry. 91 (3), 1768-1772 (2019).
- Chambers, J. M. . Statistical models in S. , (1991).
- Misra, B. B. Data normalization strategies in metabolomics: Current challenges, approaches, and tools. European Journal of Mass Spectrometry. 26 (3), 165-174 (2020).
- Livera, A. M. D., et al. Statistical methods for handling unwanted variation in metabolomics data. Analytical Chemistry. 87 (7), 3606-3615 (2015).
- Sakia, R. M. . The Box-Cox transformation technique: a review. 41 (2), 169-178 (1992).
- vanden Berg, R. A., Hoefsloot, H. C. J., Westerhuis, J. A., Smilde, A. K., vander Werf, M. J. Centering, scaling, and transformations: improving the biological information content of metabolomics data. BMC Genomics. 7, 142 (2006).
- Marees, A. T., et al. A tutorial on conducting genome-wide association studies: Quality control and statistical analysis. International Journal of Methods in Psychiatric Research. 27 (2), 1608 (2018).
- Torkamaneh, D., Laroche, J., Bastien, M., Abed, A., Belzile, F. Fast-GBS: a new pipeline for the efficient and highly accurate calling of SNPs from genotyping-by-sequencing data. BMC Bioinformatics. 18 (1), 5 (2017).
- Zhao, S., Agafonov, O., Azab, A., Stokowy, T., Hovig, E. Accuracy and efficiency of germline variant calling pipelines for human genome data. Scientific Reports. 10 (1), 20222 (2020).
- Bradbury, P. J., et al. TASSEL: software for association mapping of complex traits in diverse samples. Bioinformatics. 23 (19), 2633-2635 (2007).
- Bates, D., Mächler, M., Bolker, B., Walker, S. Fitting linear mixed-effects models using lme4. Journal of Statistical Software. 67 (1), (2015).
- Yin, L., et al. rMVP: A memory-efficient, visualization-enhanced, and parallel-accelerated tool for genome-wide association study. Genomics, Proteomics & Bioinformatics. , (2021).
- Kanai, M., et al. Genetic analysis of quantitative traits in the Japanese population links cell types to complex human diseases. Nature Genetics. 50 (3), 390-400 (2018).
- Salem, M. A., et al. An improved extraction method enables the comprehensive analysis of lipids, proteins, metabolites and phytohormones from a single sample of leaf tissue under water-deficit stress. Plant Journal: for Cell and Molecular Biology. 103 (4), 1614-1632 (2020).
- Balcke, G. U., et al. Multi-omics of tomato glandular trichomes reveals distinct features of central carbon metabolism supporting high productivity of specialized metabolites. The Plant Cell. 29 (5), 960-983 (2017).
- Leonova, T., et al. Does protein glycation impact on the drought-related changes in metabolism and nutritional properties of mature pea (Pisum sativum L.) seeds. International Journal of Molecular Sciences. 21 (2), 567 (2020).
- Alfonsi, K., et al. chemistry tools to influence a medicinal chemistry and research chemistry based organisation. Green Chemistry. 10 (1), 31-36 (2008).
- Bozek, K., et al. Organization and evolution of brain lipidome revealed by large-scale analysis of human, chimpanzee, macaque, and mouse tissues. Neuron. 85 (4), 695-702 (2015).
- Delgado, R., Muñoz, Y., Peña-Cortés, H., Giavalisco, P., Bacigalupo, J. Diacylglycerol activates the light-dependent channel TRP in the photosensitive microvilli of Drosophila melanogaster photoreceptors. The Journal of Neuroscience. 34 (19), 6679 (2014).
- Sharma, D. K., et al. UPLC-MS analysis of Chlamydomonas reinhardtii and Scenedesmus obliquus lipid extracts and their possible metabolic roles. Journal of Applied Phycology. 27 (3), 1149-1159 (2015).
- Dunn, W. B., Wilson, I. D., Nicholls, A. W., Broadhurst, D. The importance of experimental design and QC samples in large-scale and MS-driven untargeted metabolomic studies of humans. Bioanalysis. 4 (18), 2249-2264 (2012).
- Fan, S., et al. Systematic error removal using random forest for normalizing large-scale untargeted lipidomics data. Analytical Chemistry. 91 (5), 3590-3596 (2019).
- Larsson, S. J., Lipka, A. E., Buckler, E. S. Lessons from Dwarf8 on the strengths and weaknesses of structured association mapping. PLOS Genetics. 9 (2), 1003246 (2013).
- Platt, A., Vilhjálmsson, B. J., Nordborg, M. Conditions under which genome-wide association studies will be positively misleading. Genetics. 186 (3), 1045-1052 (2010).
- Nyholt, D. R. A simple correction for multiple testing for single-nucleotide polymorphisms in linkage disequilibrium with each other. American Journal of Human Genetics. 74 (4), 765-769 (2004).
- Teo, Y. Y. Common statistical issues in genome-wide association studies: a review on power, data quality control, genotype calling and population structure. Current Opinion in Lipidology. 19 (2), 133-143 (2008).
- Privé, F., Aschard, H., Ziyatdinov, A., Blum, M. G. B. Efficient analysis of large-scale genome-wide data with two R packages: bigstatsr and bigsnpr. Bioinformatics. 34 (16), 2781-2787 (2018).
- Alseekh, S., et al. Domestication of crop metabolomes: desired and unintended consequences. Trends in Plant Science. 26 (6), 650-661 (2021).
- Yano, K., et al. GWAS with principal component analysis identifies a gene comprehensively controlling rice architecture. Proceedings of the National Academy of Sciences of the United States of America. 116 (42), 21262 (2019).
- Wu, S., et al. Mapping the Arabidopsis metabolic landscape by untargeted metabolomics at different environmental conditions. Molecular Plant. 11 (1), 118-134 (2018).
- Ye, J., et al. An InDel in the promoter of Al-ACTIVATED MALATE TRANSPORTER9 selected during tomato domestication determines fruit malate contents and aluminum tolerance. The Plant Cell. 29 (9), 2249-2268 (2017).
- Zhang, W., et al. Genome assembly of wild tea tree DASZ reveals pedigree and selection history of tea varieties. Nature Communications. 11 (1), 3719 (2020).
- Tohge, T., Fernie, A. R. Annotation of plant gene function via combined genomics, metabolomics and informatics. Journal of Visualized Experiments: JoVE. (64), e3487 (2012).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。