Method Article
Крупномасштабные мультиомные исследования геномных ассоциаций (Mo-GWAS): руководство по подготовке и нормализации образцов
В этой статье
Резюме
В этом протоколе мы представляем оптимизированный рабочий процесс, который сочетает в себе эффективную и быструю пробоподготовку многих образцов. Кроме того, мы предоставляем пошаговое руководство по сокращению аналитических вариаций для высокопроизводительной оценки метаболических исследований GWAS.
Аннотация
Как газовая хроматография-масс-спектрометрия (GC-MS), так и жидкостная хроматография-масс-спектрометрия (LC-MS) являются широко используемыми метаболомическими подходами для обнаружения и количественной оценки сотен тысяч признаков метаболитов. Однако применение этих методов к большому количеству образцов подвержено более сложным взаимодействиям, особенно для общегеномных ассоциативных исследований (GWAS). Этот протокол описывает оптимизированный метаболический рабочий процесс, который сочетает в себе эффективную и быструю пробоподготовку с анализом большого количества образцов на бобовые культуры. Этот слегка модифицированный метод экстракции был первоначально разработан для анализа тканей растений и животных и основан на экстракции в метил-трет-бутиловый эфир: растворитель метанола, позволяющий захватывать полярные и липидные метаболиты. Кроме того, мы предоставляем пошаговое руководство по сокращению аналитических вариаций, которые необходимы для высокопроизводительной оценки метаболической дисперсии в GWAS.
Введение
Крупномасштабные «омические» подходы позволили проанализировать сложные биологические системы 1,2,3 и понять связь между генотипами и результирующими фенотипами4. Метаболомика с использованием сверхвысокоэффективной жидкостной хроматографии-масс-спектрометрии (UHPLC-MS) и GC-MS позволила обнаружить множество признаков метаболитов, из которых только некоторые аннотированы в определенной степени, что приводит к высокой доле неизвестных метаболитов. Сложные взаимодействия могут быть изучены путем объединения крупномасштабной метаболомики с лежащей в основе генотипической вариацией разнообразной популяции5. Однако обработка больших наборов образцов по своей сути связана с аналитическими вариациями, искажающими оценку метаболической дисперсии для дальнейших последующих процессов. В частности, основные проблемы, приводящие к аналитическим изменениям, основаны на производительности машины и инструментальном дрейфе во времени6. Интеграция вариантов от партии к партии является сложной и особенно проблематичной при анализе крупномасштабных структурированных популяций растений. Было предложено несколько процедур нормализации для исправления небиологических вариаций, например, использование внутренних, внешних и изотопных внутренних стандартов для исправления аналитических ошибок, каждая из которых по своей сути связана с известными проблемами и подводными камнями 7,8,9,10.
В дополнение к аналитическим вариациям, выбор протоколов экстракции обычно варьируется в зависимости от аналитического метода. В конечном счете, желательно снизить материальные и трудовые затраты, а также необходимость использования нескольких аликвот одного и того же образца для различных аналитических процессов путем выполнения методов экстракции на основе разделения фаз. Эти методы были впервые введены с использованием хлороформа: метанола/водных растворителей для фракционирования полярных и гидрофобных соединений11.
Этот протокол описывает быстрый высокопроизводительный конвейер для мультиомической платформы для профилирования как полярных метаболитов, так и липидов в бобовых видах. Кроме того, он показывает, как эти наборы данных могут быть соответствующим образом скорректированы для аналитического изменения и нормализованы перед интеграцией генотипической информации для обнаружения локусов количественных признаков метаболитов (QTL) путем выполнения GWAS.
протокол
1. Экспериментальное проектирование и растениеводство
ПРИМЕЧАНИЕ: Постановка эксперимента в зависимости от экспериментальной гипотезы, например, использование крупномасштабной популяции GWAS снижает потребность в множественных репликах, так как статистическое тестирование будет проводиться на основе гаплотипов всех отдельных SNP, а не присоединения. Напротив, множественные реплики незаменимы в других экспериментальных подходах. При подготовке эксперимента необходимо учитывать следующие моменты.
- Включите достаточное количество биологических реплик, в зависимости от экспериментальной гипотезы.
- Рандомизируйте биологические реплики по блокам, чтобы уменьшить локальное смещение окружающей среды во время выращивания, например, теплицы, поля.
- Обеспечьте правильное содержание растения во время роста. Обрабатывайте растения однородно, чтобы уменьшить смещение.
2. Подготовка биологического растительного сырья
- Подготовка урожая
- Этикеточные уборочные трубы (20 мл), содержащие два металлических шарика диаметром 5 мм и два диаметра 8 мм для гомогенизации. Наполните дьюар жидким азотом.
ПРИМЕЧАНИЕ: Растения должны находиться в вегетативной стадии для сбора свежих листьев и корневых тканей.
- Этикеточные уборочные трубы (20 мл), содержащие два металлических шарика диаметром 5 мм и два диаметра 8 мм для гомогенизации. Наполните дьюар жидким азотом.
- Собирайте биологические образцы путем мгновенного замораживания в жидком азоте. Сбор урожая как можно быстрее исключить влияние циркадных колебаний на обмен веществ при длительной продолжительности сбораурожая 12,13. Хранить собранные свежие листья и корневые ткани для дальнейшей обработки при -80 °C.
ПРИМЕЧАНИЕ: Срезание листьев до мгновенного замораживания не должно занимать более нескольких секунд, так как после расщепления листьев активные биологические процессы изменят метаболические профили из-за ранирования. Для корней предварительно очистите корни, промыв водой перед мгновенной заморозкой в жидком азоте. Лишнюю воду на поверхности корня следует запитать бумажной салфеткой. Высушенные семена можно хранить при комнатной температуре; не требуется замораживание в жидком азоте. - Измельчите ткань с помощью мельницы для смешивания тканей.
- Предварительно охладить держатели трубки в жидком азоте в течение нескольких минут для поддержания низкой температуры при измельчении ткани.
- Транспортируйте биологические образцы в азотсодержащем дьюаре после извлечения их из морозильной камеры с температурой -80 °C.
- Измельчить ткани до получения однородного порошка; использовать 25 Гц в течение 1 мин и повторить после замораживания в жидком азоте, если ткань не однородно измельчена.
- Для измельчения высушенных семян поместите семена в шлифовальную банку с металлическим шариком диаметром 15 мм. Используйте те же частоты и время, что и в разделе 2.3.3.
ПРИМЕЧАНИЕ: Чистые и предварительно охлажденные растворы и пестики можно использовать, если мельница для смешивания тканей недоступна. - Предварительно промаркированные микроцентрифужные трубки объемом 2 мл с безопасным замком. Взвешивают 50 мг с погрешностью ±5 мг свежего растительного материала с помощью аналитических весов. Предварительно охладить инструменты, используемые для переноса растительного материала в жидкий азот. Убедитесь, что растительный материал остается замороженным во время процесса взвешивания.
ПРИМЕЧАНИЕ: Не подвергайте свежий растительный материал слишком долго воздействию комнатной температуры, так как биологические процессы активируются повышением температуры, изменяя метаболические профили14. - Создание дополнительных образцов контроля качества (QC) путем объединения части каждого образца и взвешивания 50 мг с погрешностью ±5 мг объединенного свежего растительного материала в предварительно охлажденные микроцентрифужные трубки с безопасным замком 2 мл.
ПРИМЕЧАНИЕ: Рекомендуется по крайней мере три образца QC для каждых 60 образцов. Образцы контроля качества необходимы для последующей коррекции, нормализации и анализа.
3. Экстракционные реагенты
- Свежие ткани, например, листья и корни
ПРИМЕЧАНИЕ: Извлечение образца основано на ранее описанном протоколе15. Этот протокол был модифицирован на основе текущих потребностей, например, нескольких тканей, различных внутренних стандартов и крупномасштабных экспериментов. Кроме того, все объемы и настройки приборов, упомянутые ниже, корректируются для внутренних аналитических блоков. Пользователи протокола должны корректировать их в соответствии со своим аналитическим блоком и биологическими образцами на основе тестовых образцов.- Экстракционная смесь 1 (EM1): метил-трет-бутиловый эфир (МТБЭ)/метанол (MeOH) (3:1 об/об)
- Приготовьте смесь МТБЭ/МеОГ в соотношении 3:1. Для 100 мл экстракционного растворителя смешайте 75 мл МТБЭ с 25 мл MeOH в чистой стеклянной бутылке.
ПРИМЕЧАНИЕ: С растворителями следует обращаться осторожно в вытяжном шкафу с надлежащим предохранительным оборудованием. - Добавьте 45 мкл 1,2-дигептадеканоил-sn-глицеро-3-фосфохолина (1 мг/мл в хлороформе) в качестве внутреннего стандарта для липидного анализа на основе UHPLC-MS, 400 мкл рибитола (1 мг/мл в воде) в качестве внутреннего стандарта для анализа на основе GC-MS и 125 мкл изовитексина (1 мг/мл в MeOH/воде (1:1 в/об)) для анализа метаболитов на основе UHPLC-MS.
ПРИМЕЧАНИЕ: Добавление внутренних стандартов необходимо для нормализации после анализа в соответствии с аналитическими потребностями. Поскольку для каждого образца необходимо 1 мл EM1, подготовьте запасной раствор в соответствии с размером экспериментальной выборки, который следует использовать для всего эксперимента. EM1 необходимо хранить при температуре -20 °C. Проверьте на отсутствие используемого внутреннего стандарта и перекрытия с другими соединениями в исследуемых видах. Можно использовать несколько стандартов; выбор внутренних стандартов в этом протоколе был основан на предыдущих тестах с использованием экстрактов бобов16.
- Приготовьте смесь МТБЭ/МеОГ в соотношении 3:1. Для 100 мл экстракционного растворителя смешайте 75 мл МТБЭ с 25 мл MeOH в чистой стеклянной бутылке.
- Экстракционная смесь 2 (EM2) воды/ метанола (MeOH) (3:1 об/об)
- Для 100 мл EM2 добавьте 75 мл двойной дистиллированной воды и 25 мл MeOH в чистую стеклянную бутылку.
- Добавьте 500 мкл ЭМ2 на образец и подготовьте запасной раствор в соответствии с размером экспериментальной выборки, который следует использовать для всего эксперимента. Хранить EM2 при температуре 4 °C.
- Экстракционная смесь 1 (EM1): метил-трет-бутиловый эфир (МТБЭ)/метанол (MeOH) (3:1 об/об)
- Сушеные семена
- Экстракционная смесь 3 (EM3) метанола (MeOH)/вода (7:3 v/v)
- Для 100 мл EM3 добавьте 70 мл MeOH и 30 мл двойной дистиллированной воды в чистую стеклянную бутылку. Подготовьте 1 мл EM3 для каждого образца.
- Добавьте 400 мкл рибитола (1 мг/мл в воде) в качестве внутренних стандартов для анализа на основе GC-MS и 125 мкл изовитексина (1 мг/мл в MeOH/воде (1:1 в/об)) для анализа метаболитов на основе UHPLC-MS.
ПРИМЕЧАНИЕ: Подготовьте запасной раствор в соответствии с размером экспериментальной выборки и используйте его для всего эксперимента. Хранить EM3 при температуре 4 °C.
- Экстракционная смесь 3 (EM3) метанола (MeOH)/вода (7:3 v/v)
4. Извлечение проб
- Свежие ткани, например, листья и корни
- Подготовьте три микроцентрифужные трубки с безопасным замком объемом 1,5 мл для каждого образца. Держите EM1 в системе жидкостного охлаждения при температуре -20 °C. Перенесите свежие образцы из морозильной камеры с температурой -80 °C в сухой лед или жидкий азот для транспортировки. Добавьте 1 мл предварительно охлажденного EM1 к каждому 50 мг аликвоты и кратковременно вихрь перед тем, как держать на льду.
- Инкубируйте образцы на орбитальном шейкере при 800 × г в течение 10 мин при 4 °C.
- Обжаривайте образцы ультразвуком в охлаждаемой льдом ванне для обработки ультразвуком в течение 10 минут.
- Добавьте 500 мкл EM2 с помощью многоканальной пипетки, чтобы избежать изменения добавленных объемов.
- Вращайте образцы кратковременно, чтобы перемешать экстракционные смеси перед центрифугированием при 11 200 × г в течение 5 мин при 4 °C.
- После разделения фаз переносят 500 мкл верхней липидсодержащей фазы в предварительно маркированную микроцентрифужную трубку с безопасным замком объемом 1,5 мл. Удалите остальную часть верхней фазы.
ПРИМЕЧАНИЕ: Будьте осторожны при переносе, так как эта верхняя фаза имеет высокое давление пара и имеет тенденцию вытекать из пипетки. - Перенос 150 мкл и 300 мкл нижних полярных и полуполярных метаболитсодержащих фаз в две микроцентрифужные трубки с безопасным замком по 1,5 мл, используемые для анализа GC-MS и UHPLC-MS соответственно.
- Сконцентрируйте все экстрагированные фракции, давая растворителям испариться без нагревания с помощью вакуумного концентратора, и храните при -80 °C.
- Сушеные семена
- Подготовьте для каждого образца две микроцентрифужные трубки с безопасным замком объемом 1,5 мл. Держите EM3 на льду. Поместите металлический шарик диаметром 5 мм в образцы аликвоты.
- Добавьте 1 мл EM3 в каждую аликвоту 50 мг и гомогенизируйте образцы при 25 Гц в течение 2-3 мин, прежде чем положить их на лед.
- Обжаривайте образцы ультразвуком в охлаждаемой льдом ванне для обработки ультразвуком в течение 10 минут.
- Вращайте образцы ненадолго перед центрифугированием при 11 200 × г в течение 5 мин при 4 °C.
- Перенос 150 мкл и 300 мкл супернатанта в две микроцентрифужные трубки с безопасным замком по 1,5 мл, используемые для анализа GC-MS и UHPLC-MS соответственно.
- Сконцентрируйте все экстрагированные фракции, позволяя растворителям испаряться без нагревания с помощью вакуумного концентратора, и храните при -80 °C.
ПРИМЕЧАНИЕ: Исходя из опыта, пользователям рекомендуется выполнить этап 4.2 для полуполярных метаболитов и анализ производных метаболитов в сушеных семенах. Выполните этап экстракции 4.1 для анализа липидов высушенных семян.
5. Анализ липидов с помощью UHPLC-MS
- Повторно суспендировать высушенные липидные фракции в 250 мкл ацетонитрила:2-пропанола (7:3, об/об).
- Ультразвуком липидную фазу в течение 5 мин, центрифугу по 11 200 × г в течение 1 мин.
- Переложите 90 мкл супернатанта в стеклянный флакон для LC-MS.
- Вводят 2 мкл экстрактов в LC-MS.
- Выполняют липидное фракционирование на колонне с обратной фазойС8, удерживаемой при 60 °С, работающей с потоком 400 мкл/мин с постепенным изменением элюентов А и В, как показано в таблице 1. Получить масс-спектры в режиме положительной ионизации с диапазоном масс 150-1 500 м/з.
- Включите несколько образцов контроля качества во все ежедневные партии и заготовку, чтобы обеспечить коррекцию аналитических изменений. Рандомизация выборок по блокам в последовательном порядке.
6. Анализ полярных и полуполярных метаболитов с использованием UHPLC-MS
- Повторно суспендировать высушенную полярную фазу в 180 мкл метанола uhPLC: воды (1:1 v/v).
- Обрабатывают ультразвуком полярную фазу в течение 2 мин, центрифугу при 11 200 × г в течение 1 мин.
- Переложите 90 мкл супернатанта в стеклянный флакон для LC-MS.
- Вводят 3 мкл экстрактов в LC-MS.
- Производят фракционирование метаболитов на обратной фазеС18 колонки, удерживаемой при 40 °С, работающей с течением 400 мкл/мин с постепенным изменением элюентов А и В, как показано в таблице 1. Получите масс-спектры в диапазоне масс 100-1 500 м/з при полном ms-сканировании и всех ионных фрагментациях (AIF), индуцированных высокоэнергетической коллизионной диссоциацией (HCD) 40 кэВ.
ПРИМЕЧАНИЕ: Используйте оба режима ионизации. Однако из-за ограниченной емкости при работе с большим количеством образцов запускайте тестовые образцы в обоих режимах ионизации для определения предпочтительного режима ионизации. - Включите несколько образцов контроля качества во все ежедневные партии и заготовку, чтобы обеспечить коррекцию аналитических изменений. Рандомизация выборок по блокам в последовательном порядке.
- Запустите объединенный QC в зависящем от данных MS2 как в отрицательном, так и в положительном режимах ионизации. Используйте полученные масс-спектры на более позднем этапе (8.5) для аннотации.
7. Анализ дериватизированных метаболитов с использованием GC-MS17,18
ПРИМЕЧАНИЕ: Анализ дериватизированных метаболитов основан на ранее описанномпротоколе 17. Обрабатывайте все реагенты дериватизации в вытяжном шкафу. Убедитесь, что N-метил-N-(триметилсилил)трифторацетамид (MSTFA) не контактирует с водой и влажностью.
- Реагент дериватизации 1 (DR1)
- Растворить метоксиамина гидрохлорид в пиридине до получения концентрации 30 мг/мл DR1. Используйте 40 мкл DR1 для каждого образца. Подготовьте запасной раствор в соответствии с размером образца и храните при комнатной температуре.
- Реагент дериватизации 2 (DR2)
- Растворите MSTFA с 20 мкл метиловых эфиров жирных кислот (FAME) на 1 мл MSTFA. Используйте 70 мкл DR2 для каждого образца. Подготовьте запасной раствор в соответствии с размером выборки. Храните MSTFA при температуре 4 °C и FAME при -20 °C.
ПРИМЕЧАНИЕ: ФАЭ включают метилкаприлат, метилпеларгонат, метилкапрат, метиллаурат, метилмиристат, метилпальмитат, метилстеарат, метилэйкозаноат, метилдокозаноат, метиловый эфир лигноцериновой кислоты, метилгексакозаноат, метилоктакосаноат и метилэстер триаконтановой кислоты, которые растворяются в CHCl3 в концентрации 0,8 мкл/мл или 0,4 мг/мл для жидких или твердых стандартов, соответственно.
- Растворите MSTFA с 20 мкл метиловых эфиров жирных кислот (FAME) на 1 мл MSTFA. Используйте 70 мкл DR2 для каждого образца. Подготовьте запасной раствор в соответствии с размером выборки. Храните MSTFA при температуре 4 °C и FAME при -20 °C.
- Повторно высушите гранулу из полярной фазы (хранящуюся при -80 °C) с использованием вакуумного концентратора в течение 30 мин, чтобы избежать каких-либо помехH2O, возникающих во время хранения с растворителями, используемыми для последующей дериватизации.
- Добавьте 40 мкл DR1.
- Встряхните образцы при 950 × г в течение 2 ч при 37 °C с помощью орбитального шейкера с последующим коротким вращением жидкости.
- Добавьте 70 мкл DR2.
- Снова встряхните при 950 × г в течение 30 мин при 37 °C с помощью орбитального шейкера.
- Центрифуг ненадолго при комнатной температуре перед переносом 90 мкл в стеклянные флаконы для анализа GC-MS.
- Впрыскивание 1 мкл в бесплитный режим GC-MS, в зависимости от концентрации метаболита, с постоянным потоком газа-носителя гелия 2 мл/мин. Температура впрыска устанавливается на уровне 230 °C с помощью 30-метровой капиллярной колонны MDN-35.
ПРИМЕЧАНИЕ: Дополнительную информацию, например о градиенте температуры, можно найти в таблице 1. Диапазон масс установлен на уровне 70-600 м/з при 20 сканировании/мин. Включите режимы разделения, чтобы обеспечить количественную оценку предполагаемых перегружающих соединений, экономя затраты и время на редириватизацию экстракта в таких случаях. - Включите несколько образцов контроля качества во все ежедневные партии и заготовку, чтобы обеспечить коррекцию аналитических изменений. Правильно рандомизируйте выборки по блокам в последовательном порядке.
8. Обработка хроматограммы и аннотация соединений
- Фильтрация химического шума путем определения пороговых значений интенсивности. Включите все образцы QC при обработке хроматограмм.
ПРИМЕЧАНИЕ: Для крупномасштабных данных фильтрация шума имеет решающее значение для сокращения вычислительного времени и вычислительной мощности. - Выровняйте хроматограммы, определив окно сдвига времени удержания. Проверьте хроматограммы из каждой партии, чтобы оценить внутри- и межсерийные вариации.
- Выполняйте обнаружение пиков в зависимости от формы пика, например, высоты и ширины для полной ширины при полумаксимальных вычислениях (FWHM).
- Кластерные изотопы для уменьшения избыточных сигналов и фильтрации синглтонов.
ПРИМЕЧАНИЕ: Смотрите Таблицу материалов для получения подробной информации о программном обеспечении, используемом для обработки хроматограммы. Предоставляются подробные протоколы обработки хроматограмм с использованием различных свободно доступных программных средств, например, MS-DIAL, MetAlign, MzMine и Xcalibur 19,20,21. - Используйте данные ddMS2 объединенного образца контроля качества для создания аннотации соединения. Оценить молекулярную структуру путем определения моноизотопной массы и наблюдения общих нейтральных потерь, известных заряженных агликонов и различных типов расщеплений, например, гомолитических или гетеролитических16,22.
- Для представления данных метаболитов следуйте рекомендации, описанной в Fernie et al. 201123.
ПРИМЕЧАНИЕ: Различные вычислительные подходы к метаболомике могут быть использованы для анализа метаболомических данных 24,25,26.
9. Нормализация массива данных крупномасштабной метаболомики
- Проверьте распределение внутреннего стандарта (стандартов) и нормализуйте, исправив для ответа один или несколько внутренних стандартов.
- Скорректируйте пиковые интенсивности, полученные из хроматограммы, по точному весу образца, разделив пиковые интенсивности на аликвотированный гомогенизированный вес образца с шага 2.5.
- Корректируется для дрейфа интенсивности между многосерийными сериями. Выполнение методов коррекции на основе КК, таких как локально оцененное сглаживание диаграмм рассеяния (LOESS)27 с использованием R.
ПРИМЕЧАНИЕ: Доступно несколько инструментов и пакетов для решения проблемы дрейфа производительности MS при приобретении целых партий28,29. - Обеспечить нормальное распределение признаков путем преобразования данных, например, преобразования Box-Cox30 с помощью функции boxcox () из пакета R MASS для выполнения GWAS.
- Выполнение масштабирования данных, например, масштабирование Парето, для многомерного анализа для обеспечения надлежащего взвешивания низкообильных соединений31.
ПРИМЕЧАНИЕ: Если возможно, выполните восстановительный анализ, чтобы избежать матричных эффектов, например, подавления ионов14.
10. Общегеномные ассоциативные исследования (GWAS)32
- Назовем однонуклеотидный полиморфизм (SNP) или структурные варианты (SV) из данных секвенирования33,34.
- Фильтр генотипических данных для частоты малых аллелей (MAF) < 5% и недостающей частоты >10%, чтобы избежать низкочастотного смещения с помощью Tassel35.
- Рассчитайте лучшие линейные непредвзятые прогнозы (BLUP) для каждого нормализованного признака в течение экспериментальных повторений, чтобы устранить смещение, исходящее от факторов окружающей среды (случайных эффектов), используя пакет R Ime436.
- Используйте BLUP каждого компонента по отдельности для выполнения GWAS с использованием пакета rMVP в R37.
ПРИМЕЧАНИЕ: Каждый признак метаболомики рассматривается здесь как индивидуальный автономный фенотип. - При выполнении GWAS корректируйте структуру популяции с использованием анализа главных компонентов (PCA) и идентичности по штатам (IBS) или vanRaden, чтобы свести к минимуму смешанные эффекты. Кроме того, рассмотрите возможность использования смешанной линейной модели (MLM) или смешанной модели с несколькими локусами (MLMM), поскольку смешанные модели содержат фиксированные и случайные эффекты.
11. Обнаружение QTL
- Проверьте SNP, показывающие значительную ассоциацию, принимая во внимание графики Манхэттена, для расчетов неравновесия связей (LD) для определения базовой генетической области. Выполняйте вычисления LD с помощью тепловой карты LD пакета R или Tassel 5.
- Проверьте ассоциированные SNP на величину эффекта над признаком, изучив уровни признаков для статистических изменений между гаплотипами, чтобы найти потенциальные причинные SNP, например, SNP, приводящие к изменению аминокислот в кодирующей белок последовательности, что может объяснить фенотипическую вариацию.
ПРИМЕЧАНИЕ: Поскольку ассоциации SNP-признаков не обязательно дают причинно-следственную связь, крайне важно определить геномную область. Компаундная идентичность по аннотации признаков может очень помочь в поиске правильных генов-кандидатов в определенной геномной области. Мы предлагаем объединить все обнаруженные QTL, связанные с определенными соединениями, в плейотропной карте, чтобы подчеркнуть генетические области38, как показано на рисунке 4. Для валидации генов-кандидатов может быть выполнено несколько подходов (см. обсуждение).
Результаты
Успешные эксперименты по метаболомике GWAS должны начинаться с правильного экспериментального проектирования, за которым следует сбор образцов, извлечение, сбор и обработка данных, как показано на рисунке 1. В этом протоколе метод15 МТБЭ использовался для извлечения и анализа сотен метаболитов, принадлежащих к нескольким классам соединений. Хроматография сильно зависит от свойств используемой колонки, а также элюционных буферных смесей. На рисунке 2 показаны хроматограммы образцов QC, указывающие на картину элюирования некоторых основных классов липидов в этой аналитической системе. Применяемые градиенты для каждой платформы приведены в таблице 1. Большое внимание уделялось обработке системных ошибок в крупномасштабных экспериментах. Выполнение крупномасштабной метаболомики по своей сути связано с системными ошибками. Для демонстрации мы проанализировали липидомические данные по нескольким распространенным видам бобов. В дополнительной таблице 1 приведены извлеченные необработанные липидомные данные, полученные после обработки хроматограммы с использованием программного обеспечения, указанного в Таблице материалов. Следование этому протоколу позволило нам обойти основные проблемы при работе с омическими данными, особенно при обработке больших наборов выборок. Процедура нормализации приводит к точной коррекции пакетных аналитических ошибок, как показано на рисунке 3. Хотя увеличение количества образцов контроля качества увеличит мощность нормализации, это не всегда осуществимо из-за ограничений по стоимости и времени. Для высокопроизводительной метаболомики GWAS с нецелевыми метаболическими особенностями важно соответствующим образом проиллюстрировать более высокое количество ассоциаций признаков-маркеров. Плейотропная карта38, объединяющая несколько результатов GWAS, может быть использована для выделения геномных областей, с которыми связаны несколько признаков (рисунок 4).
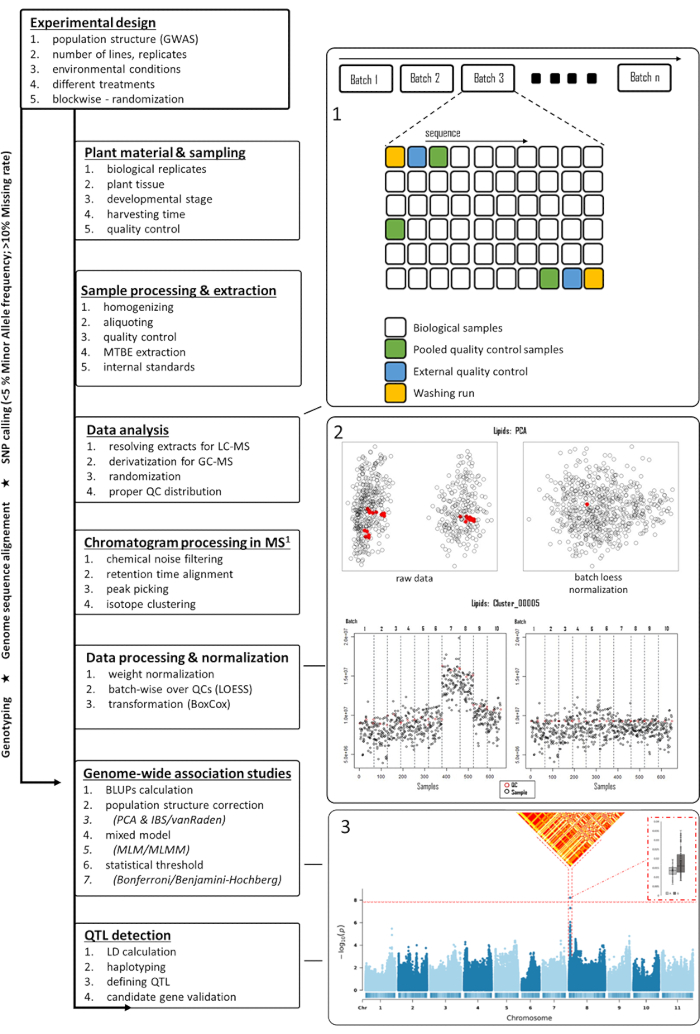
Рисунок 1: Блок-схема ГВАС на основе метаболомики в растениях. Несколько шагов, начиная от экспериментального проектирования до обнаружения QTL, показаны на левой панели. На правой панели показано несколько рисунков, поддерживающих несколько шагов, упомянутых на левой панели. Начиная с правого верха, (1) показана предложенная последовательность образцов для LC-MS, (2) графики PCA до и после нормализации, включая репрезентативное распределение признаков до и после обработки, с красным цветом, указывающим интенсивность выборки QC, и (3) манхэттенский график со значительными ассоциациями, к которым были сгенерированы распределения LD и гаплотипов. Сокращения: GWAS = общегеномные ассоциативные исследования; QTL = количественные локусы признаков; PCA = анализ главных компонентов; QC = контроль качества; LD = неравновесие связи; MS = масс-спектрометрия; LC-MS = жидкостная хроматография-масс-спектрометрия; GC-MS = газовая хроматография-масс-спектрометрия; LOESS = локально оцененное сглаживание диаграммы рассеяния; MLM/MLMM = смешанная линейная модель/мультилокусная смешанная модель. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
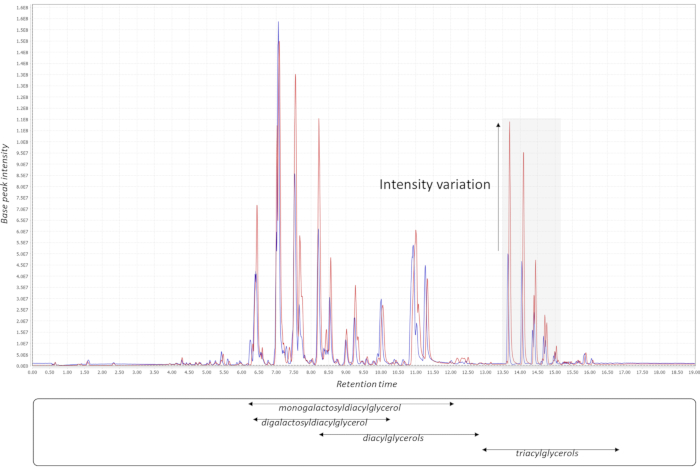
Рисунок 2: Обработка хроматограммы. Две хроматограммы КК (базовый пик; липидные данные) из разных партий демонстрируют вариации по партиям для определенных классов липидов в объединенных образцах КК. Четыре основных класса липидов обозначены соответствующими элюционными окнами в собственной системе LC-MS. Хроматограммы были экспортированы из MzMine21. Сокращения: QC = контроль качества; LC-MS = жидкостная хроматография-масс-спектрометрия. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
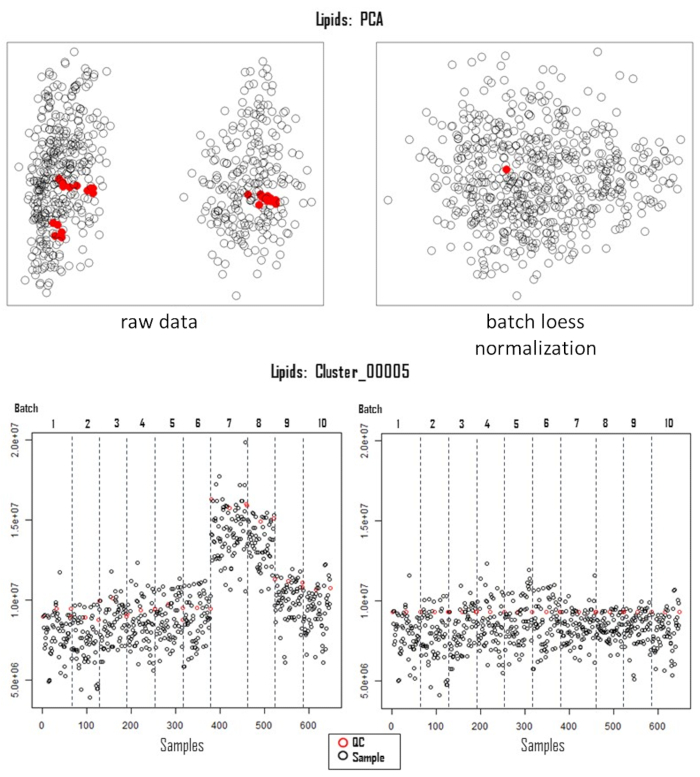
Рисунок 3: Исправление систематической ошибки. Принципиально-компонентный анализ полученных липидомных данных, пре- (левые, необработанные данные) и посткоррекция системных ошибок (правый, пакетный лёсс). Нижние панели иллюстрируют распределение признаков (Cluster_00005) по образцам (n=650) и партиям (n=10) до (слева) и после (справа) для аналитического изменения. Сокращения: PCA = анализ главных компонентов; QC = контроль качества; LOESS = локально оцененное сглаживание диаграммы рассеяния. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
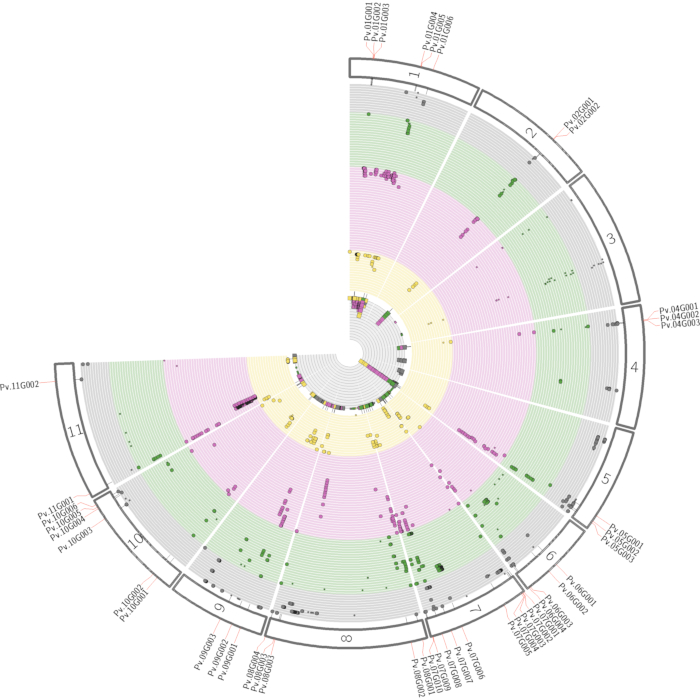
Рисунок 4: Плейотропная карта, иллюстрирующая объединенные результаты GWAS. Плейотропная карта выделяет области во всем геноме, которые связаны с несколькими признаками. Цифры на внешних кольцах указывают на соответствующие хромосомы. Каждый круг представляет собой индивидуальную черту со своими значительно связанными SNP. Цвета представляют различные составные классы (серый = составной класс 1; зеленый = составной класс 2; фиолетовый = составной класс 3; желтый = составной класс 4). В случае межсоединенных классовых ассоциаций с одной и той же геномной областью выделяют гены. Внутренний серый круг показывает сумму всех значимых SNP, связанных с определенным геномным положением. Ассоциации, показанные на этом рисунке, искусственно генерируются только для иллюстрации. Сокращения: GWAS = общегеномные ассоциативные исследования; SNP = однонуклеотидные полиморфизмы. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
| Настройки UHPLC-MS для липидов | ||||
| Время [мин] | Элюент от А до В [%]* | Информация | ||
| 0 - 1.00 | 45% А | Элюент A: 1% 1M NH 4-ацетат, 0,1% уксусной кислоты в воде (марка UHPLC) | ||
| 1.00 - 4.00 | lg 45% - 25% A | Элюент B: 1% 1M NH 4-ацетат, 0,1% уксусной кислоты в ацетонитриле / 2-пропаноле 7: 3 (класс UHPLC) | ||
| 4.00 - 12.00 | lg 25% - 11% A | Расход: 400 мкл/мин | ||
| 12.00 - 15.00 | lg 11% - 0% A | Объем впрыска: 2 мкл | ||
| 15.00 - 19.50 | cw 0% A | |||
| 19.50-19.51 | 0% - 45% А | |||
| 19.51-24.00 | экв. 45% | |||
| Настройки UHPLC-MS/MS для полярных и полуполярных метаболитов | ||||
| Время [мин] | Элюенты A и B [%]* | Информация | ||
| 0 - 1.00 | 99% А | Элюент А: 0,1% муравьиной кислоты в воде (класс UHPLC) | ||
| 1.00 - 11.00 | lg 99% -60% A | Элюент B: 0,1% муравьиной кислоты в ацетонитриле (класс UHPLC) | ||
| 11.00 - 13.00 | lg 60% - 30% A | Расход: 400 мкл/мин | ||
| 13.00 - 15.00 | lg 30% - 1% A | Объем впрыска: 3 мкл | ||
| 15.00 - 16.00 | cw 1% A | |||
| 16.00 - 17.00 | lg 1% - 99% A | |||
| 17.00 - 20.00 | экв. 99% А | |||
| Настройки GC-MS для производных метаболитов | ||||
| Время [мин] | Температура [°C] | Информация | ||
| 0 - 2.00 | 85 | Газ-носитель: Гелий | ||
| 2.00 - 18.66 | лг 80 - 330 | Расход: 2 мл/мин | ||
| 18.66 - 24.66 | cw 330 | Температурный градиент: 15 °C/мин | ||
| 24.66 | быстрое охлаждение | Объем впрыска: 1 мкл | ||
Таблица 1: Градиентные настройки для каждой из аналитических платформ7. Сокращения: lg = линейный градиент; cw = мойка колонны; eq = равновесие; UHPLC-MS = сверхвысокоэффективная жидкостная хроматография-масс-спектрометрия; UHPLC-MS/MS = сверхвысокоэффективная жидкостная хроматография-тандемная масс-спектрометрия; GC-MS = газовая хроматография-масс-спектрометрия. * = процентное значение соответствует элюенту А; оставшееся процентное значение соответствует элюенту B.
Дополнительная таблица 1: Необработанные липидомические данные. Указывает пиковые интенсивности для каждого из обнаруженных кластеров над каждым образцом. Пожалуйста, нажмите здесь, чтобы загрузить эту таблицу.
Обсуждение
Как GC-MS, так и LC-MS являются широко используемыми инструментами для профилирования сложных смесей различных классов метаболитов. Обработка больших наборов данных с помощью этих инструментов по своей сути связана с небиологической вариацией, например, аналитической вариацией, которая мешает и искажает интерпретацию результатов. Этот протокол представляет собой надежный и высокопроизводительный экстракционный конвейер для всестороннего метаболического профилирования для устранения вариаций небиологического происхождения и проведения крупномасштабных исследований «омики». Объемы и концентрации, используемые в этом протоколе, были скорректированы для видов бобовых в различных тканях. Однако эти параметры могут быть слегка изменены и использованы для крупномасштабных метаболических образцов других видов растений.
Ранее15 описанных экстракций на основе МТБЭ могут быть использованы для анализа дериватизированных метаболитов, полуполярных метаболитов и липидов. Это может быть расширено для экстракции белка и растительных гормонов39, которые были вне сферы действия этого протокола. Другие протоколы экстракции основаны на смесях дихлорметана:этанола40,41. Из этих протоколов экстракции протокол МТБЭ:метанол обеспечивает благоприятную и менее опасную альтернативу существующим протоколам экстракции42 на основе хлороформа и не приводит к образованию белковой гранулы в виде интерфазы между полярной и липидной фазами. Кроме того, методы МТБЭ уже использовались в нескольких исследованиях для различных биологических образцов 43,44,45.
В этом протоколе обсуждается несколько важных шагов, которые могут привести к потенциальному изменению при обработке большого количества образцов, например, во время сбора12,13, экстракции14, а также рандомизации46. Кроме того, существуют дополнительные вопросы, которые не обсуждались в настоящем протоколе, которые должны быть рассмотрены для обеспечения высококачественных метаболомных данных, например, матричный эффект и подавление ионов14.
Мощность методов нормализации на основе КК по своей сути зависит от количества образцов КК в каждой партии. Как упоминалось ранее, хотя увеличение числа увеличит мощность, внутрисерийное изменение КК относительно незначительно по сравнению с межсерийным изменением в этих аналитических системах, как показано на рисунке 3. В целом, существуют и другие методы нормализации на основе контроля качества, такие как системное удаление ошибок с использованием случайного леса (SERRF), которые, как было показано, превосходят большинство других методов нормализации, таких как пакетное соотношение, нормализация с использованием оптимального выбора нескольких внутренних стандартов (NOMIS) и вероятностная нормализация коэффициентов (PQN)47. . Тем не менее, SERRF полагается на несколько образцов QC в каждой партии, например, каждый десятый образец, что неосуществимо при обработке большого количества образцов. Основное преимущество нормализации на основе КК по сравнению с другими методами, основанными на данных или внутренними стандартами, заключается в том, что она сохраняет существенные биологические вариации, приспосабливаясь к нежелательным техническим вариациям28. Читатели могут обратиться к этому обзору по обращению с вариантом28.
Одной из основных проблем в GWAS является частота ложных срабатываний, которые возникают в основном из-за связи причинно-следственных и непричинных сайтов48,49. Во-вторых, консервативные подходы статистической коррекции, например, Бонферрони и Рузвельта, корректируют количество независимых тестов, которое не равно количеству анализируемых SNP в GWAS из-за связи между ближайшими SNP 50,51 Поэтому фактическое количество независимых тестов часто ниже. Другим способом снижения консервативного статистического порога могло бы стать уменьшение числа тестируемых SNP, используемых для GWAS, на основе распада связей над определенными геномнымиобластями 52. GwAS-интегрированная высокопроизводительная метаболомическая платформа, описанная в этом протоколе, имеет широкий спектр применений. В частности, это будет способствовать улучшению селекции сельскохозяйственных культур путем изменения состава метаболитов/липидов в соответствии с промышленными и питательными потребностями. В целом, метаболомика дала глубокое понимание генетической архитектуры множества метаболитов и метаболической диверсификации, которые произошли во время одомашнивания сельскохозяйственных культур за последние десятилетия, что указывает на огромный потенциал метаболомики-ассоциированной селекции53. Молекулярно-биологические подходы к последующей валидации QTL включают генерацию мутантных линий54 CRISPR/Cas9, линий вставкиТ-ДНК 55, стабильных и/или преходящих линий сверхэкспрессии56, VIGS, подходов ex vivo метаболомики57 рядом с традиционным подходом к генерации кросс-популяций F2, а также перекрестную валидацию в разных популяциях.
Выполняя необходимую коррекцию аналитических вариаций, как описано выше, в дополнение к GWAS может быть выполнено несколько интегрированных подходов, таких как метаболит-метаболит, метаболит-липидный корреляционный анализ, корреляционный анализ к феномическим данным, чтобы пролить свет на более сложные признаки, и/или анализ коэкспрессии для дальнейшего разгадывания основы биологических систем58.
Раскрытие информации
У авторов нет конфликта интересов, о которых можно было бы заявить.
Благодарности
M.B. поддерживается IMPRS-PMPG «Первичный метаболизм и рост растений». A.R.F. и S.A. признают финансовую поддержку Программы исследований и инноваций ЕС Horizon 2020, проекта PlantaSYST (SGA-CSA No 739582 под FPA No 664620) и проекта INCREASE (GA 862862).
Материалы
| Name | Company | Catalog Number | Comments |
| Reagents and standards | |||
| 1,2-diheptadecanoyl-sn-glycero-3- phosphocholine (17:0 PC) | Avanti Polar Lipids | 850360P | Internal standard for lipids |
| Chloroform | Supleco | 67-66-3 | FAME solvent |
| Isovitexin | Sigma Aldrich | 38953-85-4 | Internal standard for metabolites |
| Lignoceric Acid Methylester | Sigma Aldrich | 2442-49-1 | FAME |
| Methanol (MeOH) | Biosolve Chemicals | 13684102 | ULC-MS grade |
| Methoxyamin -hydrochlorid | Sigma Aldrich | 593-56-6 | Metabolite deriviatization |
| Methyl laurate | Sigma Aldrich | 111-82-0 | FAME |
| Methyl myristate | Sigma Aldrich | 124-10-7 | FAME |
| Methyl palmitate | Sigma Aldrich | 112-39-0 | FAME |
| Methyl stearate | Sigma Aldrich | 112-61-8 | FAME |
| Methyl tert-butyl ether (MTBE) | Biosolve Chemicals | 13890602 | HPLC grade |
| Methyl-caprat | Sigma Aldrich | 110-42-9 | FAME |
| Methylcaprylat | Sigma Aldrich | 111-11-5 | FAME |
| Methyldocosanoat | Sigma Aldrich | 929-77-1 | FAME |
| Methyleicosanoat | Sigma Aldrich | 1120-28-1 | FAME |
| Methyl-hexacosanoat | Sigma Aldrich | 5802-82-4 | FAME |
| Methyl-octacosanoat | Sigma Aldrich | 55682-92-3 | FAME |
| Methyl-pelargonate | Sigma Aldrich | 1731-84-6 | FAME |
| N-Methyl-N-(trimethylsilyl)trifluoracetamid (MSTFA) | Macherey-Nagel | 24589-78-4 | Metabolite deriviatization |
| Pyridine | Supleco | 110-86-1 | Metabolite deriviatization |
| Ribitol | Supleco | 22566-17-2 | Internal standard for derivatized metabolites |
| Triacontanoic Acid Methyl Ester | TCI Chemicals | 629-83-4 | FAME |
| Water | Biosolve Chemicals | 23214102 | ULC-MS grade |
| Equipment | |||
| 1.5 mL Safe-lock microcentrifuge tubes | Eppendorf | 3120086 | |
| 2 mL Safe-lock microcentrifuge tubes | Eppendorf | 3120094 | |
| Balance | Sartorius Corporation | 14 557 572 | |
| DB-35ms, 30 m, 0,25 mm, 0,25 µm | Aglient | 123-3832 | Analysis of derivatized metabolites |
| GC-MS system | Leco Pegasus HT TOF-MS (LECO Corporation) | Analysis of derivatized metabolites | |
| Grinding Balls, Stainless Steel | OPS DIAGNOSTICS | GBSS 196-2500-10 | |
| MS system | Exactive, Orbitrap-type, MS (Exactive, Thermo Fisher Scientific) | Analysis of lipids | |
| MS system | Q Exactive Focus (Q Exactive™ Focus Hybrid Quadrupol-Orbitrap™ Massenspektrometer, Thermo Fisher Scientific) | Analysis of metabolites | |
| Refrigerated microcentrifuge | Eppendorf, model 5427R | 22620701 | |
| Reversed Phase (RP) Bridged Ethyl Hybrid (BEH) C8 column (100 mm × 2.1 mm containing 1.7 μm diameter particles) | Waters | 186002878 | Analysis of lipids |
| RP High Strength Silica (HSS) T3 column (100 mm × 2.1 mm containing 1.8 μm diameter particles) | Waters | 186003539 | Analysis of metabolites |
| Shaker | Eppendorf Thermomixer 5436 | 2050-100-05 | |
| Sonicator | USC 300 TH | 142-0084 | |
| Tissue grinding mixer mill | Retsch, Mixer Mill MM 300 | 20.746.0001 | |
| UPLC system | Waters Acquity UPLC system (Waters) | ||
| Vacuum concentrator | Scan Speed Maxi Vac Alpha Evaporators | 7.008.500.002 | |
| Vortex mixer | Vortex-Genie 2, Model G560 | SI-0236 | |
| Software | |||
| MetAlign | Chromatogram processing | ||
| MzMine | Chromatogram processing | ||
| R package "data.table" | |||
| R package "fujiplot" | pleiotrpoic map | ||
| R package "genetics" | |||
| R package "Ime4" | BLUPs calculation | ||
| R package "LDheatmap" | LD plots | ||
| R package "MASS" | transformation | ||
| R package "rMVP" | GWAS | ||
| R version 4.0.4 | |||
| RefinerMS | Chromatogram processing | ||
| RefinerMS Genedata | Expressionist | Chromatogram processing | |
| Tassel 5 | Genotype filtering | ||
| Xcalibur | Thermo Fisher Scientific | OPTON-30965 | Chromatogram processing |
Ссылки
- Doerr, A. Global metabolomics. Nature Methods. 14 (1), 32 (2017).
- Fessenden, M. Metabolomics: Small molecules, single cells. Nature. 540 (7631), 153-155 (2016).
- Oliver, S. G., Winson, M. K., Kell, D. B., Baganz, F. Systematic functional analysis of the yeast genome. Trends in Biotechnology. 16 (9), 373-378 (1998).
- Fiehn, O. Metabolomics-the link between genotypes and phenotypes. Plant Molecular Biology. 48 (1), 155-171 (2002).
- Wu, S., et al. Mapping the Arabidopsis metabolic landscape by untargeted metabolomics at different environmental conditions. Molecular Plant. 11 (1), 118-134 (2018).
- Sysi-Aho, M., Katajamaa, M., Yetukuri, L., Orešič, M. Normalization method for metabolomics data using optimal selection of multiple internal standards. BMC Bioinformatics. 8 (1), 93 (2007).
- Chen, M., Rao, R. S. P., Zhang, Y., Zhong, C. X., Thelen, J. J. A modified data normalization method for GC-MS-based metabolomics to minimize batch variation. SpringerPlus. 3 (1), 439 (2014).
- Dunn, W. B., et al. Metabolic profiling of serum using Ultra Performance Liquid Chromatography and the LTQ-Orbitrap mass spectrometry system. Journal of Chromatography B. 871 (2), 288-298 (2008).
- Fiehn, O., et al. Metabolite profiling for plant functional genomics. Nature Biotechnology. 18 (11), 1157-1161 (2000).
- vander Kloet, F. M., Bobeldijk, I., Verheij, E. R., Jellema, R. H. Analytical error reduction using single point calibration for accurate and precise metabolomic phenotyping. Journal of Proteome Research. 8 (11), 5132-5141 (2009).
- Folch, J., Lees, M., Stanley, G. H. S. A simple method for the isolation and purification of total lipides from animal tissues. Journal of Biological Chemistry. 226 (1), 497-509 (1957).
- Fukushima, A., et al. Impact of clock-associated Arabidopsis pseudo-response regulators in metabolic coordination. Proceedings of the National Academy of Sciences of the United States of America. 106 (17), 7251-7256 (2009).
- Kerwin, R. E., et al. Network quantitative trait loci mapping of circadian clock outputs identifies metabolic pathway-to-clock linkages in Arabidopsis. The Plant Cell. 23 (2), 471-485 (2011).
- Tohge, T., et al. From models to crop species: Caveats and solutions for translational metabolomics. Frontiers in Plant Sciences. 2, 61 (2011).
- Salem, M., Bernach, M., Bajdzienko, K., Giavalisco, P. A simple fractionated extraction method for the comprehensive analysis of metabolites, lipids, and proteins from a single sample. Journal of Visualized Experiments:JoVE. (124), e55802 (2017).
- Tohge, T., Fernie, A. R. Combining genetic diversity, informatics and metabolomics to facilitate annotation of plant gene function. Nature Protocols. 5 (6), 1210-1227 (2010).
- Lisec, J., Schauer, N., Kopka, J., Willmitzer, L., Fernie, A. R. Gas chromatography mass spectrometry-based metabolite profiling in plants. Nature Protocols. 1 (1), 387-396 (2006).
- Osorio, S., Do, P. T., Fernie, A. R., Hardy, N. W., Hall, R. D. . Plant Metabolomics: Methods and Protocols. , 101-109 (2012).
- De Vos, R. C. H., et al. Untargeted large-scale plant metabolomics using liquid chromatography coupled to mass spectrometry. Nature Protocols. 2 (4), 778-791 (2007).
- Perez de Souza, ., Alseekh, L., Naake, S., Fernie, T., A, Mass spectrometry-based untargeted plant metabolomics. Current Protocols in Plant Biology. 4 (4), 20100 (2019).
- Pluskal, T., Castillo, S., Villar-Briones, A., Orešič, M. MZmine 2: Modular framework for processing, visualizing, and analyzing mass spectrometry-based molecular profile data. BMC Bioinformatics. 11 (1), 395 (2010).
- Watson, J. T., Sparkman, D. O. Electron Ionization. Introduction to mass spectrometry: Instrumentation, applications and strategies for data interpretation. , 315 (2007).
- Fernie, A. R., et al. Recommendations for reporting metabolite data. The Plant Cell. 23 (7), 2477 (2011).
- Treutler, H., et al. Discovering regulated metabolite families in untargeted metabolomics studies. Analytical Chemistry. 88 (16), 8082-8090 (2016).
- Wang, M., et al. Sharing and community curation of mass spectrometry data with Global Natural Products Social Molecular Networking. Nature Biotechnology. 34 (8), 828-837 (2016).
- Naake, T., Fernie, A. R. MetNet: Metabolite network prediction from high-resolution mass spectrometry data in R aiding metabolite annotation. Analytical Chemistry. 91 (3), 1768-1772 (2019).
- Chambers, J. M. . Statistical models in S. , (1991).
- Misra, B. B. Data normalization strategies in metabolomics: Current challenges, approaches, and tools. European Journal of Mass Spectrometry. 26 (3), 165-174 (2020).
- Livera, A. M. D., et al. Statistical methods for handling unwanted variation in metabolomics data. Analytical Chemistry. 87 (7), 3606-3615 (2015).
- Sakia, R. M. . The Box-Cox transformation technique: a review. 41 (2), 169-178 (1992).
- vanden Berg, R. A., Hoefsloot, H. C. J., Westerhuis, J. A., Smilde, A. K., vander Werf, M. J. Centering, scaling, and transformations: improving the biological information content of metabolomics data. BMC Genomics. 7, 142 (2006).
- Marees, A. T., et al. A tutorial on conducting genome-wide association studies: Quality control and statistical analysis. International Journal of Methods in Psychiatric Research. 27 (2), 1608 (2018).
- Torkamaneh, D., Laroche, J., Bastien, M., Abed, A., Belzile, F. Fast-GBS: a new pipeline for the efficient and highly accurate calling of SNPs from genotyping-by-sequencing data. BMC Bioinformatics. 18 (1), 5 (2017).
- Zhao, S., Agafonov, O., Azab, A., Stokowy, T., Hovig, E. Accuracy and efficiency of germline variant calling pipelines for human genome data. Scientific Reports. 10 (1), 20222 (2020).
- Bradbury, P. J., et al. TASSEL: software for association mapping of complex traits in diverse samples. Bioinformatics. 23 (19), 2633-2635 (2007).
- Bates, D., Mächler, M., Bolker, B., Walker, S. Fitting linear mixed-effects models using lme4. Journal of Statistical Software. 67 (1), (2015).
- Yin, L., et al. rMVP: A memory-efficient, visualization-enhanced, and parallel-accelerated tool for genome-wide association study. Genomics, Proteomics & Bioinformatics. , (2021).
- Kanai, M., et al. Genetic analysis of quantitative traits in the Japanese population links cell types to complex human diseases. Nature Genetics. 50 (3), 390-400 (2018).
- Salem, M. A., et al. An improved extraction method enables the comprehensive analysis of lipids, proteins, metabolites and phytohormones from a single sample of leaf tissue under water-deficit stress. Plant Journal: for Cell and Molecular Biology. 103 (4), 1614-1632 (2020).
- Balcke, G. U., et al. Multi-omics of tomato glandular trichomes reveals distinct features of central carbon metabolism supporting high productivity of specialized metabolites. The Plant Cell. 29 (5), 960-983 (2017).
- Leonova, T., et al. Does protein glycation impact on the drought-related changes in metabolism and nutritional properties of mature pea (Pisum sativum L.) seeds. International Journal of Molecular Sciences. 21 (2), 567 (2020).
- Alfonsi, K., et al. chemistry tools to influence a medicinal chemistry and research chemistry based organisation. Green Chemistry. 10 (1), 31-36 (2008).
- Bozek, K., et al. Organization and evolution of brain lipidome revealed by large-scale analysis of human, chimpanzee, macaque, and mouse tissues. Neuron. 85 (4), 695-702 (2015).
- Delgado, R., Muñoz, Y., Peña-Cortés, H., Giavalisco, P., Bacigalupo, J. Diacylglycerol activates the light-dependent channel TRP in the photosensitive microvilli of Drosophila melanogaster photoreceptors. The Journal of Neuroscience. 34 (19), 6679 (2014).
- Sharma, D. K., et al. UPLC-MS analysis of Chlamydomonas reinhardtii and Scenedesmus obliquus lipid extracts and their possible metabolic roles. Journal of Applied Phycology. 27 (3), 1149-1159 (2015).
- Dunn, W. B., Wilson, I. D., Nicholls, A. W., Broadhurst, D. The importance of experimental design and QC samples in large-scale and MS-driven untargeted metabolomic studies of humans. Bioanalysis. 4 (18), 2249-2264 (2012).
- Fan, S., et al. Systematic error removal using random forest for normalizing large-scale untargeted lipidomics data. Analytical Chemistry. 91 (5), 3590-3596 (2019).
- Larsson, S. J., Lipka, A. E., Buckler, E. S. Lessons from Dwarf8 on the strengths and weaknesses of structured association mapping. PLOS Genetics. 9 (2), 1003246 (2013).
- Platt, A., Vilhjálmsson, B. J., Nordborg, M. Conditions under which genome-wide association studies will be positively misleading. Genetics. 186 (3), 1045-1052 (2010).
- Nyholt, D. R. A simple correction for multiple testing for single-nucleotide polymorphisms in linkage disequilibrium with each other. American Journal of Human Genetics. 74 (4), 765-769 (2004).
- Teo, Y. Y. Common statistical issues in genome-wide association studies: a review on power, data quality control, genotype calling and population structure. Current Opinion in Lipidology. 19 (2), 133-143 (2008).
- Privé, F., Aschard, H., Ziyatdinov, A., Blum, M. G. B. Efficient analysis of large-scale genome-wide data with two R packages: bigstatsr and bigsnpr. Bioinformatics. 34 (16), 2781-2787 (2018).
- Alseekh, S., et al. Domestication of crop metabolomes: desired and unintended consequences. Trends in Plant Science. 26 (6), 650-661 (2021).
- Yano, K., et al. GWAS with principal component analysis identifies a gene comprehensively controlling rice architecture. Proceedings of the National Academy of Sciences of the United States of America. 116 (42), 21262 (2019).
- Wu, S., et al. Mapping the Arabidopsis metabolic landscape by untargeted metabolomics at different environmental conditions. Molecular Plant. 11 (1), 118-134 (2018).
- Ye, J., et al. An InDel in the promoter of Al-ACTIVATED MALATE TRANSPORTER9 selected during tomato domestication determines fruit malate contents and aluminum tolerance. The Plant Cell. 29 (9), 2249-2268 (2017).
- Zhang, W., et al. Genome assembly of wild tea tree DASZ reveals pedigree and selection history of tea varieties. Nature Communications. 11 (1), 3719 (2020).
- Tohge, T., Fernie, A. R. Annotation of plant gene function via combined genomics, metabolomics and informatics. Journal of Visualized Experiments: JoVE. (64), e3487 (2012).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены