Method Article
Mit Flachbett-Scanner mit hoher Auflösung zeitversetzt Bilder von der Arabidopsis-Wurzel gravitropen Antwort sammeln
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt ein Verfahren zur raschen Sammlung von Bildern von Arabidopsis Keimlingen Reaktion auf einen Reiz der Schwerkraft unter Verwendung von handelsüblichen Flachbettscanner. Die Methode ermöglicht kostengünstige, High-Volume-Erfassung von hochauflösenden Bildern zugänglich für Downstream-Analyse-Algorithmen.
Zusammenfassung
Die Forschungsanstrengungen in der Biologie erfordern zunehmend den Einsatz von Methoden, die High-Volume-Sammlung von hochauflösenden Daten zu ermöglichen. Eine Herausforderung Labors stehen, ist die Entwicklung und Verwirklichung dieser Methoden. Beobachtung von Phänotypen in einem Prozess von Interesse ist eine typische Ziel der Forschungslabors Studium Gen-Funktion, und dies wird oft durch Bildaufnahme erreicht. Ein besonderer Prozess, der für die Beobachtung mit bildgebenden Verfahren ist die Korrektur Wachstum von einem Sämling Wurzel, die aus der Ausrichtung mit der Schwerkraftvektor verschoben wurde. Imaging-Plattformen verwendet, um die Wurzel gravitropen Reaktion messen kann teuer, im Durchsatz relativ gering ist, und / oder arbeitsintensiv sein. Diese Fragen wurden durch die Entwicklung eines Hochdurchsatz-Bildaufnahmeverfahren mit preiswerten, aber hochauflösende, Flachbett-Scanner angesprochen. Mit dieser Methode können Bilder alle paar Minuten bei 4.800 dpi aufgezeichnet werden. Das aktuelle Setup ermöglicht Sammlung von 216 einzelnen responses pro Tag. Die gesammelten Bilddaten von reichlich Qualität für Bildanalyse.
Einleitung
Sammlung von hochauflösenden phänotypischen Daten sind nützlich in Studien, die das Zusammenspiel von Genetik und Umwelt in der Vermittlung organismal Funktion 1,2 verstehen wollen. Studien dieser Art sind auch von Natur aus in großen Maßstab, so dass es zusätzlich erforderlich, dass für die Messung Phänotypen in diesem Zusammenhang angewandten Methoden in Hochdurchsatz 3,4 sein. In die Methoden zur Phenomics angelegte Forschung, Kompromisse zwischen Durchsatz und Auflösung ins Spiel kommen. Methoden, die in höheren Durchsatz sind, neigen auch zu niedrigeren Auflösung sein, so dass es schwierig ist, geringe Auswirkungen der Genetik oder der Umgebung 5 zu erfassen. Alternativ Methoden, die genauer messen einen gewünschten Phänotyp neigen auch zu niedrigeren Durchsatz, so dass es schwierig ist, genetische und Umwelteinflüsse weitgehend überblicken. Zusätzlich können manuelle Methoden zur Quantifizierung von Phänotypen, einschließlich visueller Inspektion, Veränderungen unterliegen aufgrund der Unterschiede in der menschlichen Pro6 Wahrnehmung.
Imaging-Technologien können eine nützliche Brücke zwischen Durchsatz und Auflösung bei der Beschaffung von phänotypischen Beobachtungen 9.7 bieten. Im allgemeinen ist ein Bild relativ leicht zu erfassen, zu erleichtern Durchsatz, und wenn bei ausreichender Auflösung eingenommen wird, kann feine Phänotypen erkannt werden 1,2,7. Imaging-Technologien neigen modifizierbar, um ein System oder Verfahren von Interesse fit zu sein und sind in der Regel 10-12 skalierbar. Aus diesem Grund sind ideal Imaging-Technologien für die Entwicklung von großen Studien der organismischen Funktion.
Die Reaktion der primären Wurzel zu einem Schwerkraft Stimulus ist ein kompliziertes physiologischer Prozess, der innerhalb eines morphologisch einfaches Organ auftritt. Die Antwort beinhaltet die Aktivierung der Signalwege, die durch die Wurzel Organ ausbreiten und ihr Fortschreiten durch Umwelt-und genetischen Faktoren, wie genetische Faktoren, die durch die Umwelt beeinflusst wird 12-14 . Die Reaktion der primären Wurzel zu einem Schwerpunkt Reiz hat mindestens seit Darwin studiert, aber es gibt viel zu lernen, wie es funktioniert, vor allem in den frühen Signalereignisse und in den Faktoren der Vermittlung Antwort Plastizität 12,14,15. Der Erwerb von einem detaillierten Verständnis der Dynamik dieser Antwort ist bei der Suche nach Möglichkeiten, um die Fähigkeit der Keimlinge, um erfolgreich in einer bestimmten Umgebung 16 etabliert verbessern wichtig. Darüber hinaus ist die Form des Wurzel macht es zugänglich für Bildverarbeitungsanwendungen 8,12,17. Zusammengenommen ist die Wurzel gravitropen Antwort ein ideales System für die Entwicklung von Hochdurchsatz-Imaging-Technologie für die Zwecke der Durchführung der Genomik-Level-Studien der organismischen Funktion.
In diesem Bericht wird ein Hochdurchsatz-und hochauflösende Methode zur Bildaufnahme des Wurzel gravitropen Reaktion unter Verwendung kostengünstiger, handelsüblichen Flachbettscanner vorgestellt. Die Übersicht über dieProtokoll ist in Fig. 1 gezeigt. Sämlinge gepflanzt auf Agar-Platten wurden auf vertikal ausgerichteten Flachbettscanner mit benutzerdefinierten Plexiglasplatte angebracht Halter positioniert. Die Bilder wurden alle paar Minuten bei 4.800 dpi gesammelt und auf einem lokalen Laufwerk oder Daten-Server gespeichert. Metadaten, die mit jeder Bildreihe zugeordnet ist, in einer Datenbank gespeichert, und die gespeicherten Bilder verarbeitet werden. Der Ansatz nutzt die Software VueScan für die Bilderfassung. VueScan kann verwendet werden, um mehr als 2.100 verschiedene Scanner auf Windows-, Mac-oder Linux-Betriebssystemen laufen (siehe Materialien Table) werden. Ein Scanner-Auflösung von 4.800 dpi wurde in dieser Anwendung verwendet, um die in früheren Studien mit festen CCD-Kameras 1,8,12 erreichte Auflösung entsprechen. Die Flexibilität der VueScan-Software zusammen mit der gemeinsamen Schnittstelle verwendet es für jeden Scanner läuft es ermöglicht Benutzern, ohne weiteres auf die in diesem Papier Protokoll verabschieden praktisch jede Scanner-Hardware mit ausreichender Auflösung. Stromdurchsatz ermöglicht die Sammlung von216 Einzelantworten pro Tag. Die Technologie ist anpassungsfähig und skalierbar für den Einsatz in Einrichtungen, die von Hochschulen zu Universitäten erforschen. Darüber hinaus sind die gesammelten Bilder von ausreichender Qualität für die Bildanalyse.
Protokoll
1. Image Acquisition-Protokoll
Überlegungen:
Dieses Protokoll wird am effektivsten mit zwei Personen durchgeführt, obwohl es möglich ist, eine allein arbeiten. Die Anordnung arbeitet am besten in diesem Labor war für eine Person, um Platten für das Scannen vorzubereiten, während andere Werke auf Scanner-Setup, dann beide zusammen arbeiten, um Platten in Scanner platzieren und den Scanvorgang.
Es ist auch wichtig zu beachten, dass die Scanner in diesem Projekt sind vertikal die Scanner-Deckel auf der Rückseite des Scanners ruhen ausgerichtet. Eine benutzerdefinierte Unterstützung wurde gemacht, um Gerichte in dieser vertikalen Position zu halten und wurde auf dem Flachbettfläche mit 3M Befehlsleisten (Abbildung 2) befestigt. Der abnehmbare Dokumentenabdeckung, die mit dem Scanner in diesem Protokoll (ein Epson V700) verwendet wird, stammt wurde auf einer Seite mit schwarzem Filz ausgekleidet. Die Vorlagenabdeckung wurde gegen den Flachbett mit einem Bungee-Seil positionierthalten die Platten an Ort und um den Bildkontrast (Abbildung 3) zu liefern.
Alle Scanner von ausreichender Auflösung könnte für die Bilderfassung verwendet werden. Der Epson Perfection V700 wurde wegen seiner Vierkantprofil (macht es einfach, vertikal positionieren), seine hohe Auflösung, und die zusätzlichen Möglichkeiten, aus der sowohl das Bett und Deckel zu scannen und die Infrarot-Kanal verwenden gewählt. Diese zusätzlichen Optionen wurden nicht in diesem Protokoll verwendet.
Sobald die Platten aus der Wachstumskammer entfernt worden ist, ist es unerlässlich, dass das Protokoll bis zum Ende.
Plattenvorbereitung
Standard-Petrischalen, die 10 ml Medium transparent und 9 Samen in der Mitte jeder Platte gepflanzt wurden verwendet. : Verfahren zur Kennzeichnung Platte, Medien-und Pflanzvorbereitung finden Sie unter http://www.doane.edu/doane-phytomorph
- Rufen Sie die erste Agar-Platte und zu absorbieren gesammelte Kondenswasser am Deckel und Deckelrand von der Agar-Platte mit einem Kimwipe.
- Nehmen Triton X-100 (ein Reinigungsmittel), um den Deckel mit einem Kimwipe - großzügig sein.
(Beachten Sie, dass Triton X-100 verhindert die Ansammlung von Kondenswasser am Deckel, wenn die Platte gescannt. Eine großzügige Anwendung (genug, um einen Film auf der Deckeloberfläche erstellen) wird dazu beitragen, sicherzustellen, dass der Deckel bleibt während der gesamten Lauf Scanner transparent .) - Wickeln Sie die Platte mit Mikroporen-Band, um den Deckel zu sichern und für die Belüftung.
Scanner-Setup-und Bildsammlung
Dieses Protokoll geht davon aus, dass mehr als ein Scanner verwendet wird, und gibt Anweisungen an mehrere Scanner von einem einzigen Computer zu starten.
- Erstellen Sie Ordner zum Speichern von Bildern von jedem Scanner. Jeder Scanner wird zwei Platten zu halten, so bedenken Sie dies bei der Erstellung von Ordnern. Einkönnen wählen, um die Metadaten als Bestandteil der Dateinamen wie eindeutige IDs für jede Platte, Sämling Alter, Größe der Samen, und IDs der Bestände gepflanzt verwenden. Ein Beispiel für einen Ordnernamen in der Datenerhebung verwendet diese Metadaten enthält, ist "1652-2-sm-9-92-17-1653-2-lg-88-79 bis 161".
- Stellen Steckdose Timer für bestimmte Sammelzeit (9 Stunden wurde in diesem Labor verwendet wird). Achten Sie darauf, mehr Zeit (eine Stunde oder so) für die Vorbereitung gesetzt.
(Beachten Sie, dass Scanner sollte in die Steckdose Timer, um die Aufnahmezeit eingestellt gesteckt werden. Während die VueScan Software ermöglicht einem Benutzer, Bilder immer wieder zu sammeln, ist es nicht erlauben dem Benutzer, um anzuzeigen, wie viele Bilder zu sammeln oder wie lange die Bilder für sammeln .) - Schalten Sie die ersten Scanner und warten Sie etwa 10 Sekunden für den Scanner, um durch seine Anfangs warm-ups zu gehen.
- Öffnen Sie das Programm VueScan einmal. VueScan 9.0.20-Version wurde in diesem Protokoll verwendet (siehe Materialien Tabelle), können aber neueren Versionen mit wenig modific verwendet werdenation. Stellen Sie sicher, dass das "Mehr"-Taste ist auf der Unterseite der Benutzerschnittstelle, um die Menüoptionen unten beschrieben angezeigt gedrückt.
- Stellen Sie die automatische Wiederholung: Dropdown-Feld zu keiner unter der Registerkarte Eingabe und unter der Registerkarte Crop gesetzt Vorschaubereich: bis Maximale (Abbildung 4). Drücken Sie 'Vorschau'.
- Erstellen Sie ein Erntefeld, das die Region von Interesse mit der Maus klicken und ziehen Sie in der gesamten Region von Interesse auf der Vorschau-Bild zu erfassen wäre. Die Einstellungen können für die Region von Interesse in der Registerkarte Crop verändert werden. Die typischen Einstellungen für die Crop-Box verwendet wurden, waren: x-Offset 0,675; y-Offset in 1.924, obwohl dies wurde angepasst, um den Keimling Bereich für jeden Scanner zu erfassen. Die Ernte Kastengröße verwendet wurde, war 7,246 in der Breite und 1,1 cm (Abbildung 5).
- Um die Erntefeld zu verschieben, halten Sie die Shift-Taste gedrückt, während Sie mit der Maus. Stellen Sie sicher, dass der Crop-Box enthält alle Sämlinge gescannt werden und beliebige Metadaten, die auf einem Etikett (Abbildung 5) enthalten sein können.
- Unter der Registerkarte Crop, stellen Sie die Vorschau-Bereich: In den Crop-Box und drücken Sie 'Vorschau'.
- Gehen Sie auf die Registerkarte Ausgabe, und wählen Sie die richtige Datei für den Scanner (Abbildung 5).
- Wiederholen Sie die Schritte 1,7-1,12 auf allen Scannern für einen Computer. Wählen Sie die "Ja"-Option, wenn Sie gefragt, ob mehr als eine Instanz von VueScan zu öffnen.
- Gehen Sie durch jede Registerkarte und überprüfen, ob die Einstellungen korrekt sind. (Beachten Sie, dass alle Spezifikationen können geändert werden, um die Bedürfnisse eines einzelnen Labor einschließlich Bild-Farbe, Auflösung, etc. angepasst werden. Allerdings können die Einstellungen in diesem Protokoll direkt an den pa angewendet werdenrticular Scannen Hardware eines bestimmten Labor aufgrund der gemeinsamen Schnittstelle der Software VueScan. Finden Sie in der Liste im Anhang Spezifikationen, die in diesem Projekt verwendeten Parameter mit VueScan 9.0.20 Version) anzuzeigen.
- Unter der Registerkarte Eingabe wählen Dauer im Auto-repeat: Feld, oder wählen Sie ein längeres Zeitintervall zwischen den Bildern, wenn gewünscht. Das Zeitintervall ist die Zeit der Scanner Pausen nach dem Speichern das letzte Bild und beginnt Sammlung des nächsten Bildes. Im Dauerbetrieb kann 3-4 min bei 4.800 dpi Auflösung erhalten werden.
- Wiederholen Sie die Schritte von 1,14 bis 1,15 für den Rest der an einen Computer angeschlossen Scanner.
- Zeigen vorbereitet Platten in den richtigen Scanner mit Sämlinge horizontal ausgerichtet (nicht gravistimulate).
- Vorübergehend legen schwarz, fühlte Hintergrund gegen die Platten, damit sie nicht aus Plexiglas Vorlage fallen. Wiederholen Sie für alle Scan-Partner.
(Anmerkung: In diesem Projekt wurden schwarze Stücke aus Filz, um das Dokument deckt mit der Ausrüstung zur Verfügung gestellt, um die Blendung zu verhindern und Kontrast gegen Wurzelgewebe liefern befestigt Die spezifische Hintergrundfarbe verwendet wird auf die Farbe des Gewebes, das abgebildet abhängen.). - Lassen Sie eine Person drehen die Platten 90 ° (gegen den Uhrzeigersinn Platten wurden in diesem Protokoll eingeschaltet ist) und sofort zu ersetzen, den Filz Hintergrund.
- Die andere Person sollte auf dem Computer zu stehen, so dass sie sofort drücken Sie die "Scan"-Taste.
- Sichern Sie den Hintergrund der Scanner mit einem Bungee-Seil (Abbildung 3). Lassen Sie eine Person halten, die Hintergrund an Ort und Stelle, während ein anderer Positionen der Bungee-Seil.
(Anmerkung: Unmittelbar nach Gravistimulation (Rotation der Platten von 90 °) und die Platzierung des Filzes Hintergrund, "Scan" gedrückt werden sollte). - Wiederholen Sie die Schritte von 1,17 bis 1,21 für den Rest der Scanner auf einem einzelnen Computernäh.
- Wiederholen Sie die Schritte 1,6-1,22 für den nächsten Satz von Scannern falls zutreffend.
- Lassen Sie die Scanner erst mehrere Bilder wurden gesammelt, um sicherzustellen, dass sie richtig sparen.
- Es ist ideal, um die Scanner in einem Bereich, der frei von Störungen für die Scan-Zeit bezeichnet sein wird halten. Es ist auch ratsam, die Umweltbedingungen in der Scan-Bereich betrachten, um ideal phänotypische Reaktionen zu gewährleisten.
- Wenn die Datenerfassung abgeschlossen ist, drücken Sie die grüne Taste abbrechen VueScan auf jedem Fenster, die mit jedem Scanner zusammenfällt.
- Schließen aller Programme auf dem Computer.
- Starten Sie den Computer und schaltete alle Scanner, bevor eine weitere Runde der Bildsammlung.
Ergebnisse
Repräsentative Bilder
Dieser Ansatz ermöglicht eine schnelle Produktion von hochauflösenden Zeitreihen von Arabidopsis Sämling Wachstum. Erste und letzte Bilder von einem Scanner Laufs sind in den 7A und 7B gezeigt. Figuren 7C und 7D optimale Ergebnisse von der Hälfte einer Vollbildscanner. Einige Fragen, die die Bildqualität beeinflussen können, sind in den Figuren 7A und 7B gezeigt. Diese Fragen gehören Variation in Keimung, Variation in Sämling Wachstumskurs zu Beginn der Flucht, und Aufbau von Kondensation beim Scannen. Kondensation kann weitgehend durch eine Erhöhung der Menge an Triton X-100 auf der Innenseite der Deckelplatte aufgebracht gelöst werden. Andere Faktoren, die genaue Bildsammlung hemmen könnten, sind falsche Konfiguration des Erntefeld in Bezug auf die Plattenposition und Positionierungsplatten, so dass sie in Bezug auf das Erntefeld verzerrt.
Bildanalyse-Anwendung: Bildkomprimierung
Sobald eine Zeitsequenz von Scannerbildern erhalten worden ist, muss es sicher in einem Netzwerk zugänglichen Weise gespeichert ist, um die Bildanalyse zu erleichtern. Die Bilddateien mit einer individuellen Lauf Scanner verbunden belegen eine erhebliche Menge an Festplattenspeicher. Eine bei 4.800 dpi gesammelt einzelne TIFF-Datei ist ca. 220 MB und eine typische Scanner Lauf erzeugt 200 Bilddateien. Daher ist etwa 44 GB Festplattenspeicher pro Lauf erforderlich. Den Speicher-und Netzwerkübertragungskosten mit Bildanalyse zu verringern, ist es wünschenswert, die Menge an Platz benötigt wird, um Bilddaten zu speichern, während gleichzeitig die Minimierung der Datenverlust zu reduzieren. Downstream-Analyse Identifizierung jeder Sämling beinhalten in nachfolgenden Bilddateien mit einer experimentellen Lauf verbunden. Daher Segmentierung aus einzelnen Sämlinge aus dem Scanner Bild kann nachgelagerten Analyse erleichtern. Da die Segmentierung des Keimlings weg vom Rest der thBild E kann auch erheblich reduzieren Lagerung von unnötigen Hintergrundpixel führt dieser Ansatz auch deutliche Reduzierung der Datengröße. Außerdem, wenn stromabwärts Analyse auf Wurzelgewebe fokussiert es nicht notwendig sein, um die Farbinformation zu halten, da die Wurzel Pixel sind relativ schmal in ihrer Farbraum. Ein Computer-Bildverarbeitungsprotokoll und Code, um Daten Größe durch die Segmentierung sowohl einzelne Sämlinge und Umwandlung von Bildern in Graustufen zu reduzieren, entwickelt. Der Ansatz führt zu einer Reduktion von 60% in der Speicherplatzbedarf.
Die zur Durchführung dieses Datenkompression zu erreichen Workflow wird in den folgenden Schritten beschrieben:
- Beginnen Sie mit einer Zeitreihe von Scanner-Bild-Dateien in einem einzigen Ordner.
- Für jedes Bild, konvertieren von einem RGB in Graustufen (Abbildung 8, oben).
- Teilen Sie das Bild in linken und rechten Seite.
- Extrahieren Sie jede Keimling aus dem Bild in eine eigene Datei (Abbildung 8).Dies wird durch Anlegen einer Schwelle, um Pixel, die schwarz oder weiß, und dann Berechnen der Gesamtpixelintensität jeder Bildzeile umzuwandeln geführt. Die Zeile mit der höchsten Intensität identifiziert wird und jedes Pixel wird als "Pflanzen" oder "nicht-pflanzlichen 'basierend auf der Intensität der Nachbar klassifiziert. Das Zentrum jeder 'Pflanze' innerhalb dieser Zeile gefunden und von diesem Punkt eine Ernte Feld von einer bestimmten Größe gezeichnet wird (Abbildung 8, unten).
- Erstellen Sie einen separaten Ordner für jede Seite des Bildes (links und rechts) mit separaten Unterordner für die einzelnen Keimling für die Lagerung der einzelnen Zeitbildserie-Dateien.
- Archivieren Sie die resultierende Ordner in eine komprimierte ZIP-Datei.
Ein Code, der diese Schritte vollendet wurde mit der Programmiersprache Python 20 entwickelt. Der Algorithmus ermöglicht eine Reduktion etwa 60% der Datenmenge und ist erfolgreich bei der Identifizierung aller einzelnen Sämlinge in 90% der Scanner imagE-Dateien analysiert, so weit. Die Codes stehen zum Download unter der GNU General Public License Version 3 frei verfügbar (siehe Materialien Tabelle).
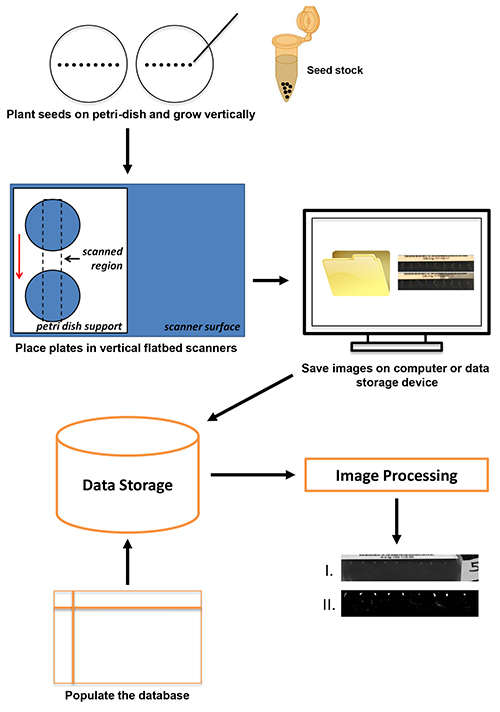
Fig. 1 ist. Der Scanvorgang beginnt mit der Aussaat (bis zu neun Arabidopsis Samen pro Platte) und endet mit Datenspeicherung und Bildverarbeitung. Klicken Sie hier für eine größere Ansicht .
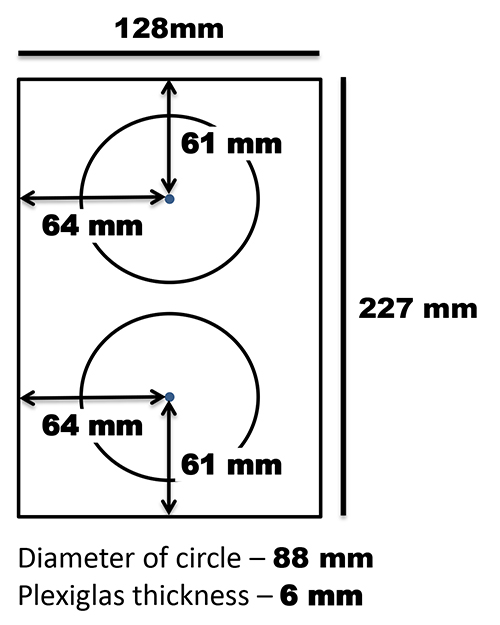
Abbildung 2. Emplate T für den Bau der Petrischale Unterstützung. Ple xiglas wurde geschnitten, so dass die Breite des Flachbett passen (in diesem Fall 227 mm) und die Länge betrug 128 mm. Zwei Kreise mit einem Durchmesser von 88 mm wurden aus dem verbleibenden Teil, so daß sie gleichmäßig über die Breite und Länge des Trägers verteilt geschnitten. Der Träger wurde auf dem Flachbett mit 3M Befehlsleisten befestigt. Klicken Sie hier für eine größere Ansicht .

Abbildung 3. Scanner-Konfiguration nach der Sämlinge wurden gravistimulated und das Dokument Abdeckung positioniert. Dies ist die Konfiguration des Scanners bei Schritt 1.21 von Scanner-Setup-und Bildsammlung."_blank"> Klicken Sie hier für eine größere Ansicht.
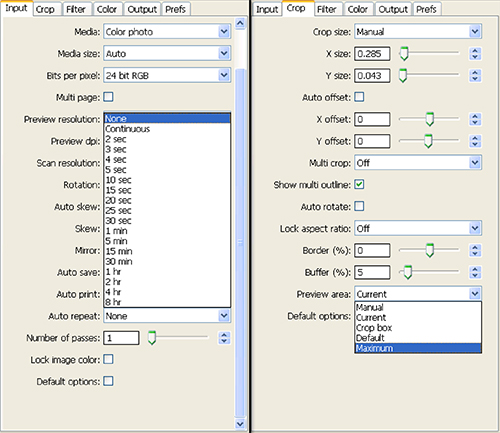
Abbildung 4. Screenshot der Einstellungen für Schritt 1.8 des Scanner-Setup-und Bildsammlung ausgewählt. Klicken Sie hier für eine größere Ansicht .

Abbildung 5. Screenshot der Software VueScan während der Schritte 1.9 und 1.10 der Scanner-Setup-und Bildsammlung. Die rote Box unterstreicht die Pflanzengröße, während der blaue Kasten werden spezifische Einstellungen fo r x-und y-Offset um Sämlinge und Label-Informationen zu erfassen eingesetzt. Der Bereich der Flachbett gescannt werden als gepunktete Linie im Vorschaubereich angezeigt. Klicken Sie hier für eine größere Ansicht .
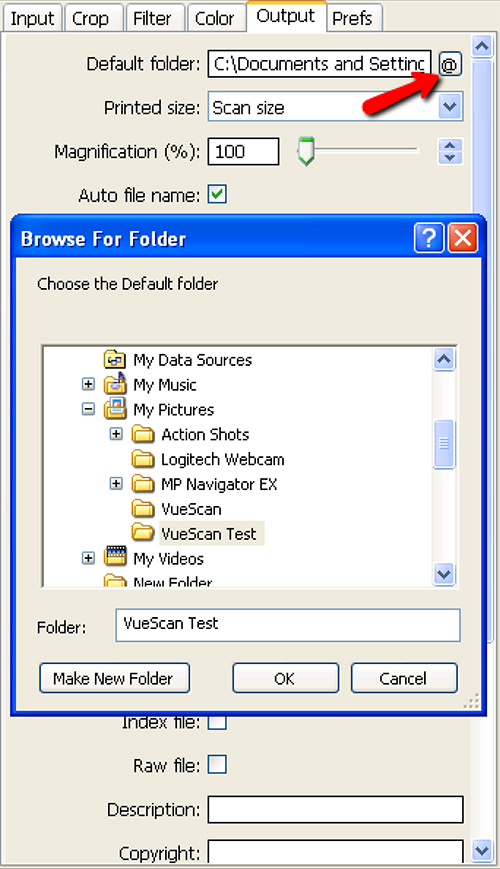
Abbildung 6. Auswahl der Zielordner für Schritt 1.12 des Scanner-Setup-und Bildsammlung. @ Drücken der Taste neben dem Dialogfeld Default Folder (roter Pfeil) kann der Benutzer den entsprechenden Zielordner auswählen. Klicken Sie hier für eine größere Ansicht .
Wieder 7 "fo: content-width =" 5in "fo: src =" / files/ftp_upload/50878/50878fig7highres.jpg "src =" / files/ftp_upload/50878/50878fig7.jpg "width =" 600px "/>
Abbildung 7 (AD). Die obigen Bilder sind Beispiele für die gesammelt unter Verwendung der in diesem Dokument beschriebenen Verfahren. Panels A, B und C, D sind die ersten und letzten Bilder jeweils von einem einzigen Scan Zeitraum. A, B zeigen die Voll gescannten Bereich, während C, D eine beschnittene Bereich des gescannten Bereichs, zeigt eine einzige Platte. Mehrere Inkonsistenzen beobachtet werden. Panel A zeigt die Variation in Keimung und Wachstum Weise. Feld B (die gleichen Sämlinge als Bild A; 9 Stunden später) zeigt, dass die Platten kann sich Kondenswasser ansammeln. Platten C und D gelten als gute Ergebnisse durch robuste Wachstum o seinf Sämlinge und Bildqualität über die gesamte Auflage. Klicken Sie hier für eine größere Ansicht .
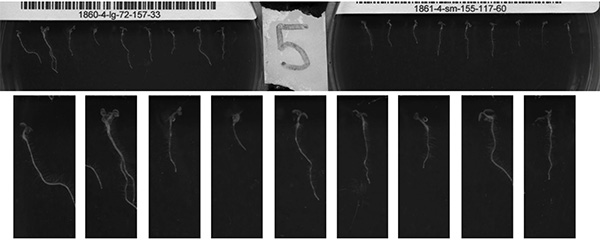
Abbildung 8. Das entwickelte Bild-Kompressions-Algorithmus wandelt ein Bild, um Scanner-Skala (oben) grau. Das Bild wird in rechte und linke Hälften geteilt und Bildgrenzen werden entfernt (nicht gezeigt). Die Positionen der einzelnen Sämlinge auf jeder Hälfte werden von der Suche nach der Zeile mit dem größten Gesamtpixelintensität identifiziert. Diese Positionen werden verwendet, um eine neue Anbaufläche, auf alle Sämlinge auf der Platte (unten) angewendet definieren. Klicken Sie hier für eine größere Ansicht .
Diskussion
Genaue phänotypische Beobachtung ist von entscheidender Bedeutung für das Verständnis der Erscheinungsformen der Gen-Funktion in einem Organismus. Ein Weg, um phänotypische Informationen zu erwerben, ist die Erfassung von hochauflösenden Bilddaten. Der Scanner-basierte Plattform entwickelt hat, Sammlung von vielen Bildern (200 Bilder / Scan-Zeit) mit hoher Auflösung (4800 dpi) über mehrere Stunden aktiviert. Darüber hinaus bietet diese Plattform ist leicht zu einer Vielzahl von Labor und Klassenzimmer Umgebungen aufgrund der Flexibilität der Software VueScan, Tausende von verschiedenen Scannern über eine gemeinsame Schnittstelle laufen 18 angepasst.
Die hier vorgestellte Methode füllt eine Lücke im Hochdurchsatz-Bilderfassung, die von Großanlagen Phänotypisierung und automatisierte Systeme implementierbar in einem einzigen Labor erstreckt. Die Hochdurchsatz-Plattformen verfügbar sind in der Regel spezialisierte Imaging-Hardware, einschließlich Kameras an Roboter Stützen montiert zu verwenden, um hochauflösende Bilder von p zu erfassenrimarily oberirdischen Pflanzenteile (zB Zentrum für Integrative Anlagentechnik und der Scanalyzer HTS durch LemnaTec) 20,21. Spezialisierte Bildsysteme mit Röntgen-und MRT-Technologien haben auch Bild unter der Erde Gewebe mit bemerkenswerter Auflösung entwickelt, wie sie in der Bodenumgebung (zB Zentrum für Integrative Pflanzen Technology) 11,22,23 wachsen. Diese Entwicklung von spezialisierten Technologie ist im Allgemeinen auf Kosten des Durchsatzes, so dass dynamische phänotypische Untersuchungen erschwert. Wichtig ist, dass die Kosten-und Infrastrukturbedarf für diese High-End-Plattformen machen sie meist nicht machbar für den Einsatz in kleineren Labors.
Plattformen sind ebenfalls entwickelt worden, die mehrere Standard-Bildaufnahme-Technologie und sind für die Messung der dynamischen Reaktionen wie die Wurzel Reaktion auf eine Schwerkraftreiz geeignet. Zum Beispiel wurden CCD-Kameras verwendet, um einzelne Sämling Reaktionen auf Licht und Schwerkraft bei hohen erfassenräumliche und zeitliche Auflösung 1,8,12. Andere Systeme wurden entwickelt, ermöglicht daher die Messung Wurzelspitze Ausrichtung von mehreren Wurzeln aus einem einzigen Bild (z. B. durch die RootTipMulti iPlant Collaborative) 17,24. Im ersteren Fall wird der Durchsatz relativ gering da nur ein Sämling von jeder Kamera zu einem Zeitpunkt abgebildet wird, während in dem letzteren Fall Durchsatz ist höher, aber im allgemeinen auf Kosten der Auflösung.
Die in diesem Dokument beschriebene Verfahren stellt eine Plattform für die Erfassung von hochauflösenden Bildern in hoher Durchsatz bei Ausrüstung und Software, die leicht verfügbar und relativ erschwinglich sind. Mit diesem Setup können 1.080 einzelnen Wurzel Antworten jede Woche in einem Labor mit einer Bank von sechs Scannern ausgestattet gesammelt werden. In 15 Monaten sammeln durchschnittlich 864 Einzelantworten pro Woche, wurden insgesamt 41.625 Setzlinge für eine Genomik Studie gescannt. Über 15% der einzelnen Sammlungen durch Setup-Fehler, Netzw gescheitertrk Ausfall oder Fehlfunktionen. Weitere Antworten 22% konnte aufgrund fehlender oder unzureichender Keimwurzelwachstum, eine Wachstumsreaktion hervorzurufen. Der letzte Datensatz besteht aus 27.475 einzelnen Sämling Antworten auf einen Reiz der Schwerkraft von 163 rekombinanten Inzuchtlinien sowie 99 in der Nähe von isogenen Linien. Die Daten wurden in einem Labor gesammelt, so dass dies ein sehr hoher Durchsatz-Ansatz. Auch da der für den Erwerb verwendeten Geräte ist relativ kostengünstig, zuverlässig seit über zwei Jahren auch bei intensiver Nutzung funktioniert hat.
Während dieses Protokoll ist sehr nützlich für die Forschungsziele dieser Gruppe waren, existieren einige Einschränkungen. Aufgrund der Durchsatz von 50 GB unkomprimierter Bilddaten pro Tag, war es offensichtlich, dass eine große Menge an Speicherplatz auf Haus Bilder benötigt, wenn wirksame Kompressionsverfahren entwickelt werden. Das Speicherproblem wurde vorübergehend durch den Kauf von externen Festplatten für jeden Computer gelöst. Darüber hinaus wurden zwei 10 TB Netzwerk verbundenen Speichergeräte angeschafft. Später wurden Kompressionsalgorithmen entwickelt, wie oben beschrieben, die zur Verringerung der Datenmenge um bis zu 60% (Fig. 8). Es ist wichtig zu beachten, dass die Geschwindigkeit, mit der Daten zu einem Netzwerk verbundenen Speichervorrichtung gespeichert werden kann, hängt von der Geschwindigkeit der Netzwerkverbindung. Kompressionsschemata auch auf den Wunsch, um den Verlust von Bilddaten zu verhindern eingeschränkt.
Weitere spezifische zu einem Scanner-basierten Imaging-System Einschränkungen werden auch berücksichtigt. Beispielsweise in einem Scanner-basierte Ansatz Keimlinge Licht hoher Intensität in den weißen und möglicherweise Infrarotbereich während jeder Abtastung belichtet. Dies betrifft wahrscheinlich Sämlingswachstum allerdings Keimlinge noch beobachtet robusten Reaktionen auf eine Schwerkraftreiz unterzogen (Fig. 7) werden. Eine zukünftige Verbesserung könnte beinhalten Programmierung Scanner, so dass nur Infrarot-LEDs aktiv sind. Ein Bereich, in aktiven development ist die Schaffung von Analyse-Algorithmen auch auf die Auflösung und Durchsatz dieser Bilddaten abgeglichen. Die erzeugte mit diesem Scanner-basierte Methode großen Datenmenge ist ideal für die Entwicklung von robusten Werkzeuge für die Hochdurchsatz-Phänotypisierung von Sämling Bilder. Die auf diese in Fig. 7 gezeigten Bilder verwendet Komprimierungsalgorithmus unterstützt die Behauptung, dass sie zugänglich Bildanalyseanwendungen. Zusätzlich können die erzeugten Bilder von der bisher veröffentlichten Algorithmus, RootTrace 17,24 analysiert werden, wenn sie bei geringerer Auflösung (weniger als 1.200 dpi) erhoben werden, und einzelne Setzlinge aus dem Bild unter Verwendung der oben beschriebenen Analyse vor Kompressionsalgorithmus segmentiert. Das Wurzelwachstum Daten könnten von den Bildern zu 1.200 dpi reduziert, während Spitzenwinkel Daten könnten von den Bildern zu 900 dpi (unveröffentlichte Beobachtung) reduziert extrahiert werden extrahiert werden.
Die in diesem Dokument beschriebenen Vorgehensweise passt in seine eigene Nische in der Welt der root-Bildgebung, dass es einen hohen Durchsatz und hohe Auflösung, während immer noch relativ erschwinglich. Ein weiterer Vorteil dieses Ansatzes ist, dass es leicht angepasst werden, um die Imaging-Anforderungen eines bestimmten Forschungsgruppe unterzubringen.
Offenlegungen
Die Autoren erklären, dass sie keine finanziellen Interessen konkurrieren.
Danksagungen
Diese Arbeit wurde durch einen Zuschuss von der National Science Foundation (Verleihungsnummer IOS-1031416) gefördert und wurde in Zusammenarbeit mit Nathan Miller, Logan Johnson und Edgar Spalding von der Universität von Wisconsin und Brian Bockelman, Carl Lundstedt und David Swanson von der durchgeführt worden University of Nebraska Holland Computing Center.
Materialien
| Name | Company | Catalog Number | Comments |
| Epson Perfection V700 Photo Scanners | Epson | B11B178011 | - |
| Plexiglas Scanner Template | - | - | Custom made. See Figure 2. |
| Smart Strap Bungee Cords | SmartStraps | Wal-Mart 1079478 | |
| Brinks Digital Outdoor Timers | Brinks | Wal-Mart 42-1014-2 | |
| VueScan Software | Hamrick Software | http://www.hamrick.com | |
| Segmentation Software | Chris Wentworth, Doane College | https://sites.google.com/a/doane.edu/compphy-doane/projects/root-gravitropism/image-segmentation | |
| 3M Micropore Tape | Fisher Scientific | 19-061-655 | - |
| Holding racks | - | - | Custom made by gluing two cookie racks together. |
Referenzen
- Miller, N. D., Brooks, T. L. D., Assadi, A. H., Spalding, E. P. Detection of a gravitropism phenotype in glutamate receptor-like 3.3 mutants of Arabidopsis thaliana using machine vision and computation. Genetics. 186, 585-593 (2010).
- Clack, N. G. Automated Tracking of Whiskers in Videos of Head Fixed Rodents. PLoS Comp. Biol. 8, (2012).
- Lussier, Y. A., Liu, Y. Computational approaches to phenotyping: high-throughput phenomics. Proc. Am. Thoracic Soc. 4, 18-25 (2007).
- Houle, D. Colloquium Paper: Numbering the hairs on our heads: The shared challenge and promise of phenomics. Proc. Natl. Acad. Sci. U.S.A. 107, 1793-1799 (2009).
- Elwell, A. L., Gronwall, D. S., Miller, N. D., Spalding, E. P., L, T. D. B. Separating parental environment from seed size effects on next generation growth and development in Arabidopsis. Plant Cell Env. 34, 291-301 (2011).
- Silk, W. K. Quantitative Descriptions of Development. Ann. Rev. Plant Physiol. 35, 479-518 (1984).
- Cronin, C. J., Feng, Z., Schafer, W. R. Automated imaging of C. elegans behavior. Methods Mol. Biol. 351, 241-251 (2006).
- Miller, N. D., Parks, B. M., Spalding, E. P. Computer-vision analysis of seedling responses to light and gravity. Plant J. 52, 374-381 (2007).
- Iyer-Pascuzzi, A. S. Imaging and Analysis Platform for Automatic Phenotyping and Trait Ranking of Plant Root Systems. Plant Physiol. 152, 1148-1157 (2010).
- Houle, D., Mezey, J., Galpern, P., Carter, A. Automated measurement of Drosophila wings. BMC Evol. Biol. 3, 25 (2003).
- Jahnke, S. Combined MRI-PET dissects dynamic changes in plant structures and functions. Plant J. 59, 634-644 (2009).
- Durham Brooks, T. L., Miller, N. D., Spalding, E. P. Plasticity of Arabidopsis Root Gravitropism throughout a Multidimensional Condition Space Quantified by Automated Image Analysis. Plant Physiol. 152, 206-216 (2010).
- Perrin, R. M. Gravity signal transduction in primary roots. Ann. Botany. 96, 737-743 (2005).
- Strohm, A. K., Baldwin, K. L., Masson, P. H. Molecular mechanisms of root gravity sensing and signal transduction. Dev. Biol. 1, 276-285 (2012).
- Harrison, B. R., Masson, P. H. ARL2, ARG1 and PIN3 define a gravity signal transduction pathway in root statocytes. Plant J. 53, 380-392 (2007).
- Beeckman, T. Root Development. , Wiley-Blackwell. 157-174 Forthcoming.
- French, A., Ubeda-Tomás, S., Holman, T. J., Bennett, M. J., Pridmore, T. High-throughput quantification of root growth using a novel image-analysis tool. Plant Physiol. 150, 1784-1795 (2009).
- Granier, C. PHENOPSIS, an automated platform for reproducible phenotyping of plant responses to soil water deficit in Arabidopsis thaliana permitted the identification of an accession with low sensitivity to soil water deficit. New Phytol. , 169-623 (2006).
- Walter, A. Dynamics of seedling growth acclimation towards altered light conditions can be quantified via GROWSCREEN: a setup and procedure designed for rapid optical phenotyping of different plant species. New Phytol. 174, 447-455 (2007).
- Gregory, P. J. Non-invasive imaging of roots with high resolution X-ray micro-tomography. Plant Soil. , 255-351 (2003).
- Pierret, A., Kirby, M., Moran, C. Simultaneous X-ray imaging of plant root growth and water uptake in thin-slab systems. Plant Soil. 255, 361-373 (2003).
- Naeem, A., French, A. P., Wells, D. M., Pridmore, T. P. High-throughput feature counting and measurement of roots. Bioinformatics. 27, 1337-1338 (2011).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten