Method Article
RNAscope für
In diesem Artikel
Zusammenfassung
Das Vorhandensein von Hochrisiko-HPV im Kopf-und Halstumorgewebe ist mit den positiven Ergebnissen assoziiert. Die neu entwickelte RNA in situ Hybridisierung Technik namens RNAscope ermöglicht die direkte Visualisierung von HPV E6/E7 mRNA in FFPE-Gewebeschnitten.
Zusammenfassung
Der "Goldstandard" für die onkogenen HPV-Nachweis ist die Demonstration von transkriptionell aktiven Hochrisiko-HPV im Tumorgewebe. , Erkennung von E6/E7 mRNA durch quantitative Reverse Transkription-Polymerase-Kettenreaktion (qRT-PCR) erfordert jedoch die RNA-Extraktion, die das Tumorgewebe Kontext entscheidend für morphologische Korrelation zerstört und war schwierig, in der klinischen Routine verabschiedet werden. Unsere neu entwickelte RNA in situ Hybridisierung Technologie RNAscope, ermöglicht die direkte Visualisierung von RNA in Formalin fixierten und in Paraffin eingebetteten (FFPE) Gewebe mit Einzelmolekülempfindlichkeit und Einzelzellauflösung, die hochsensitive und spezifische in situ-Analyse der RNA Biomarker ermöglicht in der klinischen Routineproben. Die RNAscope HPV-Test wurde entwickelt, um die E6/E7 mRNA von sieben Hochrisiko-HPV-Genotypen (HPV 16, 18, 31, 33, 35, 52 und 58) mit einem Pool von Genotyp-spezifischen Sonden zu detektieren. Es hat eine ausgezeichnete Empfindlichkeit nachgewiesenund Spezifität gegen den Strom "Goldstandard" Verfahren zum Nachweis E6/E7-mRNA durch qRT-PCR. HPV-Status von RNAscope bestimmt ist stark prognos den klinischen Verlauf von Mundrachenkrebs-Patienten.
Einleitung
Hochrisiko-humanen Papillomviren (HR-HPV) macht etwa 5% aller Krebserkrankungen weltweit ein. Die Häufigkeit von HPV-assoziierten oropharyngealen Krebs hat sich in den letzten Jahrzehnten vor allem bei Männern erhöht. HPV-positive Mund-Rachen-Plattenepithelkarzinom (OPSCC) wird wahrscheinlich bilden eine Mehrheit aller Kopf-und Halskrebs in den USA in den nächsten 20 Jahren 2. Oropharyngeale Plattenepithelkarzinome, die durch HPV verursacht werden, mit günstigen Überleben und Tumor HPV-Status ist ein starker und unabhängiger prognostischer Faktor für das Überleben 3.
Beweise für die transkriptionelle Aktivierung der viralen Onkogene E6 und E7 wird als Goldstandard für die Anwesenheit von klinisch relevanten HPV 4 betrachtet. Allerdings ist der Nachweis E6/E7-mRNA aus frischem Tumorgewebe mittels RT-PCR-Technik nicht praktisch in der klinischen Routine und hat zu den Forschungslabors beschränkt. Vor kurzem haben wir have eine neuartige Technologie namens RNA-ISH RNAscope, die Multiplex-Nachweis mit RNA-Einzelmolekülempfindlichkeit in Formalin fixiert Paraffin eingebettete Gewebeproben (FFPE) 5-10 können in einzelnen Zellen. Wir stellten drei Beweislinien für Einzelmoleküldetektion 5. Erstens erlaubt die RNAscope Sondendesign und Signalverstärkungssystem Detektion von einzelnen Kopie HER2 genomischen DNA-Ziele in HeLa-und SK-BR-3-Zelllinien. Zweitens, wenn genomische DNA mit HER2 Signalen verglichen, die Verteilung der Fluoreszenzintensitäten von HER2-mRNA Signal Punkte in HeLa-Zellen war konsistent mit einem Molekül pro Punkt. Drittens, die Anzahl der HER2-mRNA-Signal Punkte pro Zelle eng aufeinander abgestimmt die durch eine lösungsbasierte Quantifizierung Assay geschätzt HER2-mRNA-Kopienzahl, weitere Stützeinzelmoleküldetektion. Außerdem Gegenfärbung der Zellkerne mit DAPI in der Fluoreszenzmikroskopie oder mit Hämatoxylin in Hellfeld-Mikroskopie ermöglicht die Visualisierung der einzelnen Kerne, whICH wiederum ermöglicht den Nachweis und die Quantifizierung von RNA-Zielen auf einer Einzelzellgrundlage 10. Die Fähigkeit, die Genexpression in situ in der klinischen Routineproben analysieren RNAscope macht eine vielversprechende Plattform für diagnostische Pathologie, vor allem diejenigen, FFPE-Gewebeschnitt-basierte Assays 10,11. Wir haben eine RNAscope basierten Assay zum Nachweis HPV E6/E7 mRNA von sieben Hochrisiko-HPV-Genotypen (HPV 16, 18, 31, 33, 35, 52 und 58) mit einem Pool von Genotyp-spezifische Sonden entwickelt. Unsere bisherigen Untersuchungen haben gezeigt, dass OPSCC die RNAscope HPV Assay ist hochempfindlich und spezifisch bei der Bestimmung HPV-Status auf FFPE-Geweben 12-17 und informiert auch Prognose OPSCC 12,16.
Das Prinzip der RNAscope Technik bereits beschrieben worden 5. Hier beschreiben wir die komplette RNAscope Testprotokoll und zeigen ihre Verwendung in der Detektion von HR-HPV in FFPE-Tumorgewebeschnitten.
Protokoll
1. Sample, Ausrüstung und Vorbereitung der Reagenzien
- Cut Gewebeprobe in Blöcke von 3-4 mm Dicke, fix in frischem 10% neutral gepuffertem Formalin für 16 bis 32 Stunden bei Raumtemperatur (25 ° C) und in Paraffin eingebettet werden. Schneiden Sie FFPE in Abschnitte von 5 ± 1 um Dicke aus FFPE-Blöcke, auf Objektträger und der Luft trocknen. Hinweis: Die Folien können bei Raumtemperatur unter Trocknung für 3 Monate gelagert werden. Die montierten Gewebeschnitten in einem trockenen über bei 60 ° C für 1 h vor dem Assay RNAscope gebacken werden.
- Bringen Hybridisierungsofen auf 40 ° C Legen Sie ein Befeuchten von Papier in das Fach Luftfeuchtigkeitskontrolle. 50 ml dH 2 O auf die Befeuchtung Papier, um es vollständig zu benetzen. Legen Sie die abgedeckt Tray in den Ofen, für mindestens 30 Minuten vor der Verwendung vorwärmen.
- Stellen Sie 700 ml 1x vorbehandeln 2 Lösung (10 nl, pH 6 Citrat-Puffer) durch Verdünnen 10x Vorbehandlungslösung in dH 2 O. Erhitzen Sie das vorbehandeln 1x 2 Lösung zu sieden und maintain die Temperatur zwischen 100-104 ° C und verhindert Über-Kochen.
- Machen Sie 3 L 1x Waschpuffer (0,1 x SSC) durch Verdünnen vorgewärmten 50x Waschpuffer in dH 2 O.
- Bereiten Sie 2 x 200 ml Xylol und 2 x 200 ml 100% EtOH für Entparaffinierung unter einer Abzugshaube.
- Bereiten Sie 50% Hämatoxylin Färbelösung und Bläuungsreagenz (0,01% Ammoniakwasser) für Post-Färbung unter einer Abzugshaube.
- Planen 200 ml Xylol, 200 ml 70% und 2 x 200 ml 100% EtOH für die Dehydratisierung unter einem Abzug.
- Vorwärmen Ziel Sonden bei 40 ° C für 10 Minuten und bringen RTU Detection Kit Reagenzien auf Raumtemperatur, einschließlich RTU Amp1, Amp2, Amp3, Amp4, Amp 5-Brown, und Amp6-Brown, außer DAB Chromogen Brown-A-und-B Braun .
2. RNAscope Assay
Entparaffinierung und Dehydration
Nach dem Backen entparaffinieren Gewebeschnitte in Xylol für 2 x 5 min mit häufigem Rühren und entwässernin 100% EtOH für 2 x 3 min mit häufigem Rühren. Luft für 5 Minuten und zeichnen Sie eine hydrophobe Barriere rund um den Gewebeschnitt mit einer hydrophoben Barrier Stift.
Vorbehandlungen
- Inkubieren Gewebeschnitte mit vorbehandeln 1 für 10 min bei RT zum Abschrecken der endogenen Peroxidase-Aktivität, spülen in dH 2 O.
- Inkubieren Gewebeschnitte mit vorbehandeln 2 bei einer Siedetemperatur für 15 min für die RNA-Abruf gehalten gründlich zweimal in dH 2 O.
- Inkubieren Gewebeschnitte mit vorbehandeln 3 für 30 Minuten bei 40 ° C für die Proteinverdauung in HybEZ Ofen gründlich zweimal in dH 2 O.
Zielsondenhybridisierung
Ziel Sonden HPV-HR 7-Pool gehören: HPV16, 18, 31, 33, 35, 52 und 58 auf. In HPV-Sonden, Ubiquitin C (UBC) und bakterielle Gen dapB Sonden separat auf drei nebeneinander Gewebeschnitten. Hybridisierung bei 40 ° C im Ofen für 2hr, dann in 1x Waschpuffer spülen für 2 x 2 min bei RT.
Signalverstärkung
- Inkubieren Gewebeschnitte mit Amp1 (Vorverstärker) 30 min bei 40 ° C, Amp2 (Hintergrund Minderer) für 15 min bei 40 ° C, Amp3 (Verstärker) 30 min bei 40 ° C und Amp4 (Markierungssonde) für 15 min bei 40 ° C im Ofen HybEZ. Nach jedem Hybridisierungsschritt gründlich in 1x Wash Puffer für 2 x 2 min bei RT.
- Inkubieren Gewebeschnitte mit Amp5 für 30 min und Amp6 für 15 min bei RT. Nach jeder Inkubation Schritt gründlich in 1x Wash Puffer für 2 x 2 min bei RT.
Signalerkennung
Inkubieren Gewebeschnitte mit 1:1 DAB Mischung durch Mischen gleicher Volumen von Brown und Brown-A-B für 10 min bei RT gründlich zweimal in dH 2 O.
Gegenfärbung
Stain Gewebeschnitte mit 50% Hämatoxylin-Lösung für 2 min bei RT, spülen mit dH 2 O bis Träger sind klar, während Gewebe lila bleiben. Dip rutscht in 0,01% Ammoniak in dH 2 O für 5x und folgte mit 5 taucht in dH 2 O.Slide Einbau
Entwässern Gewebeschnitte in 70%, 100% und 100% EtOH für je 2 min, 5 min Xylol, montieren mit Xylol-basierten Montage Medien.
Ergebnisse
RNAscope HPV-Test-Workflow
Die RNAscope Assay hat eine hoch optimierten Workflow, die ähnlich IHC ist (Abbildung 1). Es besteht aus vier Hauptschritten: Vorbehandlungen, Hybridisierung Signalverstärkungen und Detektion. Es setzt eine einzigartige Sonde Design-Strategie, die High-Fidelity-Signalverstärkung 5 gewährleistet. Im RNAscope HPV Assay werden die sieben HR-HPV-Sondensätze vereinigt, die alle von der gleichen Signalverstärkungssystem mit Meerrettichperoxidase (HRP) verknüpft erkannt. Spezifische Hybridisierungssignale werden erkannt als braune Niederschläge durch HRP gebildet katalysierte Farbreaktion mit DAB als Substrat, die leicht durch Standard-Hellfeldmikroskopie sichtbar gemacht werden kann.
Repräsentative Färbung für HPV-Nachweis
Abbildung 2 zeigt beispielsweise Bilder von Bunt FFPE Abschnitte des Kopf-Hals-Plattenepithelkarzinom. Bei den HPV-positiven Fall (Abb.Abbildung 2A), erfasst die HR-HPV-Sonden starke punktförmige Signale spezifisch in den Tumorzellen. Die UBC-Sonde nachgewiesen zahlreiche punktförmige zytoplasmatische Signale in beiden Tumorzellen und Stromazellen (2B). Das bakterielle Gen dapB Sonde zeigten einen sauberen Hintergrund (Abbildung 2C). In der HPV-negativen Fall sowohl die HR-HPV-Sonde und der Sonde nachgewiesen dapB keine Signale (Abb. 2D und 2F), während starke Signale wurden unter Verwendung des UBC-Sonde (2E) nachgewiesen. In diesem Test UBC dient als Positivkontrolle, um Gewebe RNA-Qualität und dapB als Negativkontrolle für die Hintergrundsignale zu beurteilen. Die Wertung für HPV-Status Bestimmung erfordert die Prüfung alle drei Rutschen für jeden Fall. Die Färbungspegel auf der negativen Steuerschieber (dapB) Schieber als Grenz verwendet: HPV-Häufigkeit wird durch die Anwesenheit von punktförmigen cytoplasmatischen und / oder Kernfärbung, die über das Signal auf der dabpB Schlitten definiert.

Fig. 1 ist. Flussdiagramm RNAscope-Assay. Illustration von RNAscope Test-Workflow. Die RNAscope Test 4 Hauptschritte, Vorbehandlungen, Zielsondenhybridisierung, Signalverstärkung und Signalerkennung. Das gesamte Testverfahren kann in 8 Stunden abgeschlossen sein. Klicken Sie hier für eine größere Ansicht.
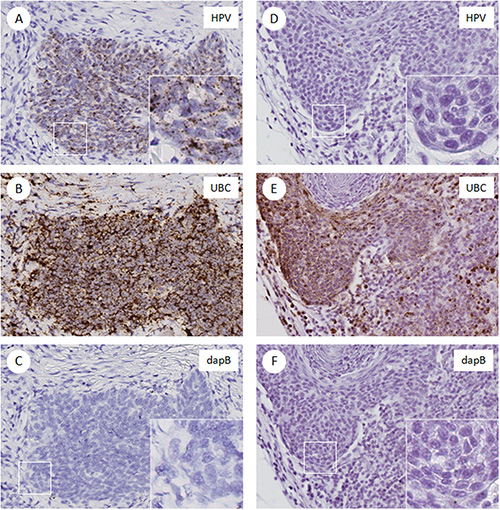
2. Beispiel Bilder von RNAscope-gefärbten Folien. FFPE Abschnitte mit Sonden gebeizt für HR-HPV, UBC (Positivkontrolle) und dapB (Negativkontrolle)von zwei Fällen HNSCC. AC. HPV-positiven Fall. Ein HR-HPV E6/E7 mRNA-Expression in Tumorzellen. Inset, 40-facher Vergrößerung kann punktförmige Signale zeigen. B und C) benachbarten Gewebeschnitten zeigt positive UBC-Färbung (B) und negativ für dapB (C) sind. DF) HPV-negativen Fall. D) keine Färbung für HPV E6/E7 mRNA , ähnlich dem dapB Negativkontrolle (F). E) UBC positive Färbung. Klicken Sie hier für eine größere Ansicht.
Diskussion
Der HPV-Test RNAscope erlaubt die direkte Visualisierung von E6/E7 mRNA in situ in HPV-assoziierte Kopf-Hals-Plattenepithelkarzinom. Die RNAscope Assay ist kompatibel mit routine festen Tumorgewebe und bewahrt Gewebemorphologie für histopathologische Korrelationen (Abbildung 2). Ein wesentlicher Vorteil des Assays über RNAscope CISH herkömmlichen Verfahren ist, dass er spezifisch verstärkt die Hybridisierungssignale (Fig. 2B und 2E) ohne Verstärkung des Hintergrundrauschens (2C und 2F).
In der Praxis kann die RNAscope HPV-Testverfahren innerhalb von 8 Stunden abgeschlossen oder bequem über zwei Tage aufgeteilt werden. Der HPV-Test RNAscope wurde genutzt, um HPV-Status in Kopf-Hals-Plattenepithelkarzinom 12-16 bestimmen, was zeigt, 97% Sensitivität und 93% Spezifität mittels qRT-PCR als Referenzmethode 16. Konventionelle chromogenic ISH für HR-HPV-DNA ist hochspezifisch, sondern hat eine Empfindlichkeit von 12 ~ 80%. Immunhistochemischen (IHC) Färbung für die zelluläre Surrogatmarker p16 zeigt hervorragende Empfindlichkeit, aber können falsche positive Ergebnisse 15,18, vor allem in nonoropharyngeal Kopf-Hals-Tumoren 15 zu generieren. Die aktuelle "Goldstandard"-Methode der qRT-PCR für HPV E6/E7 mRNA-Nachweis erfordert frisch gefrorenem Gewebe für optimale Ergebnisse und ist technisch komplex, was seine Verwendung zu Forschungslabor begrenzt. Darüber hinaus erfordert es die RNA-Extraktion, die es unmöglich macht, HR-HPV E6/E7 mRNA-Expression mit Histopathologie korreliert ist.
Es gibt mehrere kritische Faktoren für den Erfolg der RNAscope Assay. Erstens, um die besten Ergebnisse, Gewebe sind in frischen 10% neutral gepuffertem Formalin bei Raumtemperatur für 16-32 Stunden nach ASCO / CAP 19 Leitlinien festgelegt werden. Zweitens wird der Ofen HybEZ empfohlen, da es zu ermöglichens eine optimale Steuerung der Temperatur und Feuchtigkeit für Sondenhybridisierung und Signalverstärkungsschritten. Drittens ist es wichtig, um überschüssige Rest Puffer vor jedem Schritt zu entfernen, aber immer noch den Gewebeschnitt zu halten vor dem Austrocknen während einer dieser Schritte.
Die hier beschriebene manuelle Verfahren RNAscope wurde vollständig auf einem kommerziellen Auto-Schieber-Färbung System 10 automatisiert. Dies sollte erheblich erleichtern Standardisierung der Testbedingungen und spart wertvolle Handarbeit in der klinischen Pathologie-Laboratorien. Darüber hinaus hat dedizierte Bildanalyse-Software entwickelt, um die Zellen 10 und Färbung Signale auf einer digitalisierten Rutsche, die helfen sollen, die Subjektivität eliminieren und die Reproduzierbarkeit verbessern in Scoring automatisch zu identifizieren.
Zusammenfassend erkennt der HPV-Test RNAscope das Vorhandensein von HR-HPV E6/E7 mRNA-Transkripte in situ in FFPE-Gewebe. Es verfügt über einen Workflow, der vertraut klinischen Pathologie Labor istTories durch Genehmigung der Visualisierung direkt von diesen in Gewebeschnitten. Es bietet eine ideale Plattform für die Prüfung (FFPE) Gewebeproben und kann leicht durch diagnostische Pathologie-Laboratorien angenommen werden.
Offenlegungen
Alle Autoren werden von eigenen Aktien und in Advanced Cell Diagnostics, Inc. beschäftigt
Danksagungen
Unterstützte Teil durch NIH (R43/44CA122444) und DOD BRCP Zuschuss (W81XWH-06-1 bis 0682), um YL.
Materialien
| Name | Company | Catalog Number | Comments |
| SuperFrost Plus slides | Fisher Scientific | 12-550-15 | |
| HybEZ Oven, Tray, and Rack | Advanced Cell Diagnostics | 310011, 310012, 310014 | |
| RNAscope 2.0 FFPE Reagent Kit - Brown | Advanced Cell Diagnostics | 310035 | |
| RNAscope HPV-HR7 Probe for HPV 16, 18, 31, 33, 35, 52, and 58, E6/E7 mRNA | Advanced Cell Diagnostics | 312351 | |
| ImmEdge Hydrophobic Barrier Pen | Vector Laboratory | H-4000 | |
| 100% EtOH | American Master Tech Scientific | ALREAGAL | |
| Xylene | Fisher Scientific | X3P-1GAL | |
| Gill's Hematoxylin I | American Master Tech Scientific | HXGHE1LT | |
| Ammonia hydroxide | Sigma-Aldrich | 320145-500mL | |
| Cover Glass 24 mm x 50 mm | Fisher Scientific | 12-545-F | |
| Hot Plate | Fisher Scientific | 11-300-49SHP | |
| Drying Oven | Capable of holding temperature at 60±1 °C | ||
| Water Bath | Capable of holding temperature at 40±1 °C |
Referenzen
- Parkin, D. M. The global health burden of infection-associated cancers in the year 2002. Int. J. Cancer. 118 (12), 3030-3044 (2006).
- Chaturvedi, A. K., et al. Human papillomavirus and rising oropharyngeal cancer incidence in the United States. J. Clin. Oncol. 29 (32), 4294-4301 (2011).
- Ang, K. K., et al. Human papillomavirus and survival of patients with oropharyngeal cancer. N. Engl. J. Med. 363 (1), 24-35 (2010).
- Smeets, S. J., et al. A novel algorithm for reliable detection of human papillomavirus in paraffin embedded head and neck cancer specimen. Int. J. Cancer. 121 (11), 2465-2472 (2007).
- Wang, F., et al. RNAscope: a novel in situ RNA analysis platform for formalin-fixed, paraffin-embedded tissues. J. Mol. Diagn. 14 (1), 22-29 (2012).
- Liu, X., et al. Specific regulation of NRG1 isoform expression by neuronal activity. J. Neurosci. 31 (23), 8491-8501 (2011).
- Yan, K. S., et al. The intestinal stem cell markers Bmi1 and Lgr5 identify two functionally distinct populations. Proc. Natl. Acad. Sci. U.S.A. 109 (2), 466-471 (2012).
- Payne, R. E., et al. Viable circulating tumour cell detection using multiplex RNA in situ hybridisation predicts progression-free survival in metastatic breast cancer patients. Br. J. Cancer. 106 (11), 1790-1797 (2012).
- Bordeaux, J. M., et al. Quantitative in situ measurement of estrogen receptor mRNA predicts response to tamoxifen. PLoS One. 7 (5), (2012).
- Wang, Z., et al. Automated quantitative RNA in situ hybridization for resolution of equivocal and heterogeneous ERBB2 (HER2) status in invasive breast carcinoma. J. Mol. Diagn. 15 (2), 210-219 (2013).
- Tubbs, R. R., et al. Ultrasensitive RNA in situ Hybridization for Detection of Restricted Clonal Expression of Low Abundance Immunoglobulin Light Chain mRNA in B-Cell Lymphoproliferative Disorders. Am. J. Surg. Pathol. In press, (2013).
- Ukpo, O. C., Flanagan, J. J., Ma, X. J., Luo, Y., Thorstad, W. L., Lewis, J. S. High-risk human papillomavirus E6/E7 mRNA detection by a novel in situ hybridization assay strongly correlates with p16 expression and patient outcomes in oropharyngeal squamous cell carcinoma. Am. J. Surg. Pathol. 35 (9), 1343-1350 (2011).
- Lewis, J. S., et al. Transcriptionally-active high-risk human papillomavirus is rare in oral cavity and laryngeal/hypopharyngeal squamous cell carcinomas--a tissue microarray study utilizing E6/E7 mRNA in situ hybridization. Histopathology. 60 (6), 982-991 (2012).
- Lewis, J. S., et al. Partial p16 staining in oropharyngeal squamous cell carcinoma: extent and pattern correlate with human papillomavirus RNA status. Mod. Pathol. 25 (9), 1212-1220 (2012).
- Bishop, J. A., et al. Detection of transcriptionally active high-risk HPV in patients with head and neck squamous cell carcinoma as visualized by a novel E6/E7 mRNA in situ hybridization method. Am. J. Surg. Pathol. 36 (12), 1874-1882 (2012).
- Schache AG, ., et al. Validation of a novel diagnostic standard in HPV-positive oropharyngeal squamous cell carcinoma. Br. J. Cancer. 108 (6), 1332-1339 (2013).
- Mehrad, M., et al. Papillary Squamous Cell Carcinoma of the Head and Neck: Clinicopathologic and Molecular Features With Special Reference to Human Papillomavirus. Am. J. Surg. Pathol. Jun. , (2013).
- Jordan, R. C., et al. Validation of methods for oropharyngeal cancer HPV status determination in US cooperative group trials. Am. J. Surg. Pathol. 36, 945-954 (2012).
- Hammond, M. E., et al. American Society of Clinical Oncology/College of American Pathologists guideline recommendations for immunohistochemical testing of estrogen and progesterone receptors in breast cancer (unabridged version). Arch. Pathol. Lab. Med. 134 (7), 48-72 (2010).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten