Method Article
Aufbau und Optimierung eines Hochdurchsatz-Setup zum Studium
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Dieses Video Artikel beschreibt das Hochdurchsatz-Pipeline, die erfolgreich etabliert hat, zu infizieren und zu analysieren, eine große Zahl von Zebrafisch-Embryonen bietet eine neue Verbindung leistungsfähiges Werkzeug für Test-und Wirkstoffforschung mit einer ganzen Wirbeltierorganismus.
Zusammenfassung
Zebrafische sind immer ein wertvolles Werkzeug in der präklinischen Phase der Arzneimittelforschung Screenings als Ganzes Tiermodell mit Hochdurchsatzscreening Möglichkeiten. Sie können verwendet werden, um die Lücke zwischen den Assays auf Zellbasis in einem früheren Stadium und in vivo-Validierung in Säugermodellen überbrücken, wodurch auf diese Weise wird die Anzahl der Verbindungen, die durch die Prüfung auf die viel teurer Nagetiermodellen. In diesem Licht ist in der vorliegenden Handschrift ist eine neue Hochdurchsatz-Pipeline mit Zebrafisch in vivo Modellsystem für die Untersuchung von Staphylococcus epidermidis und Mycobacterium marinum Infektion beschrieben. Dieses Setup erlaubt die Erzeugung und Analyse einer großen Anzahl von Synchron Embryonen homogen infiziert. Darüber hinaus ist die Flexibilität der Rohrleitung kann der Benutzer einfach implementieren andere Plattformen, um die Auflösung der Analyse zu verbessern, wenn nötig. Die Kombination aus dem Zebrafisch zusammen mit innovativen Hochdurchsatz-technologies öffnet den Bereich der Droge und Entdeckung neuer Möglichkeiten, nicht nur wegen der Stärke unter Verwendung eines Ganztiermodell, sondern auch wegen der großen Anzahl von transgenen Linien vorhanden, die verwendet werden können, um die Wirkungsweise der neuen Verbindungen zu entschlüsseln.
Einleitung
Um den Zebrafisch stammen (Danio rerio) ist erfolgreich als effizientes Modell, um eine Vielzahl von Infektionskrankheiten ein Studium etabliert. Die Zebrafischembryo bietet einzigartige Möglichkeiten in-vivo-Bildgebung durch ihre Transparenz und der großen Anzahl von vorhandenen transgenen Reporterlinien, die fluoreszierende Proteine. Diese leistungsfähige Kombination macht es möglich, verschiedene Immunzelltypen in der Zeit zu verfolgen, während der Interaktion mit Krankheitserregern wie Mycobacterium marinum, der nächste Verwandte von M. Tuberkulose 2 oder Staphylococcus epidermidis, die Hauptverursacher von Biomaterial-assoziierten Infektionen 5.3. Verschiedene Infektionswege in Zebrafischembryos in Abhängigkeit von den Zwecken der Untersuchung 6 verwendet werden.
Eine dieser Infektionswege ist Dotter Injektion der Bakterien. Der Hauptvorteil dieser Methode im Vergleich zu den anderen ist, dass Dotter infectiauf automatisch über Roboter Injektion durchgeführt werden, eine signifikante Reduzierung der Einspritzzeit und damit hohe Reproduzierbarkeit der Infektion 7, 8.
Frühere Arbeiten unter Verwendung der Zebrafisch ein hoher Durchsatz in vivo Modellsystem für die Untersuchung von S. epidermidis und M. marinum Infektion zeigten erfolgreiche 7, 8 ist. Dieses System ist in der Lage, Krankheitsprogression über Roboter Eigelb Injektion von frühen Embryonen und mittels Fluoreszenzauslese als Maß für die bakterielle Belastung zu screenen. In Übereinstimmung mit dieser Idee, hat dieser Aufbau optimiert und etabliert eine sehr effiziente Hochdurchsatz-Pipeline mit dem Potenzial, eine große Anzahl von homogen infizierte Embryonen zu erzeugen und zu verfolgen den Verlauf der Infektion während der Zeit nach der Behandlung mit einer Anzahl von Verbindungen. Mit der bewährten Setup ist es möglich, erzeugen bis zu 8000 Synchron Embryonen zu Bildschirmzum Fortschreiten der Erkrankung, die Verarbeitung auf diese Weise bis zu 2500 Embryonen pro Stunde. Embryonen werden auf der Grundlage ihrer Bakterienlast sortiert unter Verwendung eines automatisierten Systems, wodurch homogene Gruppen von infizierten Larven. Darüber hinaus, um das Setup zu validieren, die Auswirkungen der Referenz bekannt, Tuberkulose Progression bei Säugetieren zu verhindern, haben an Embryonen mit M. infiziert getestet marinum E11 Belastung oder die virulenter Stamm M 9.
Diese Studie beschreibt im Detail die Hochdurchsatz-Pipeline, die eingerichtet wurde, um eine große Anzahl von infizierten Embryos und die nachfolgende Analyse der Bakterien Progression während der Entwicklung und nach der Behandlung Verbindung zu erzeugen.
Protokoll
1. Bakterienstämme und Wachstumsbedingungen
- Bereiten S. epidermidis Inokulum
- Nehmen Sie mehrere einzelne Kolonien von S. epidermidis-Stamm O-47, enthaltend eine pWVW189 abgeleitet mCherry Expressionsvektor (De Boer L. unveröffentlicht) von einem Luria-Bertani (LB)-Agarplatten mit 10 ug / ml Chloramphenicol und Kultur über Nacht bei 37 ° C in 25 ml ergänzte LB-Medium, ergänzt 10 ug / ml Chloramphenicol Stufe Midlog.
- Zentrifuge 1 ml der Kultur bei 12.000 × g für 1 min und anschließend waschen mit 1 ml steriler phosphatgepufferter Salzlösung (PBS), 3x mit 0,3% (v / v) Tween-80.
- Messung der optischen Dichte bei 600 nm (OD 600) und verdünnt das Bakteriensuspension bis zu einer OD 600 von 0,3 in 2% (w / v) Polyvinylpyrrolidon 40 (PVP 40) in PBS. Hinweis: eine OD 600 von 0,3 entspricht 1,0 × 10 8 koloniebildenden Einheiten / ml (KBE / ml).
- Bereiten M. marinum Inokulum
- Nehmen Sie mehrere einzelne Kolonien von M. marinum Stamm M oder E11, die das pSMT3-mCherry Vektor stabil exprimieren mCherry 10 von einer Middlebrook 7H10-Agar-Platte mit 10% (v / v) Middlebrook-OADC-Anreicherung ergänzt mit 50 ug / ml über Nacht bei 28 ° C Hygromycin und Kultur in 10 ml Middlebrook 7H9-Medium mit 10% (v / v) Middle ADC-Anreicherung mit 50 ug / ml Hygromycin ergänzt.
- Zentrifuge 1 ml der Kultur bei 12.000 × g für 1 min und anschließend Waschen mit 1 ml steriler PBS mit 0,3% (v / v) Tween-80 3x.
- Messung der OD 600 und verdünnt das Bakteriensuspension bis zu einer OD 600 von 0,3 in 2% (w / v) PVP 40 in PBS. Hinweis: eine OD 600 von 1 entspricht 1,0 x 10 8 cfu / ml.
2. Bereiten Zebrafisch-Eier
- Legen Sie ein maximal 70 männliche und 50 weibliche wilde type Zebrafisch getrennt in den großen Zuchtbehälter. Hinweis: Setzen Sie die weiblichen Fische im unteren Teil des großen Zuchtbehälter.
- Entfernen Sie die Trenn am nächsten Tag in der Früh, der Zebrafisch Zucht beginnen zu lassen.
- Sammeln der Eier an der Unterseite des großen Zuchtbehälters durch die Eiersammelanlage in einem 50 ml-Röhrchen mit Ei Wasser (60 &mgr; g / ml Instant Ocean Meersalz) gefüllt.
3. Injektionsnadeln
- Erhalten handelsüblichen Maß Glaskapillaren Nadeln mit einem Innendurchmesser von 10 um.
4. Experimental Outline of Injection
- Kochen Sie 100 ml von 1% (w / v) Agarose in Ei-Wasser, und kühlen, bis ca. 40 º C. Gießen Agarose in den automatisierten Mikroinjektoren Platte und legen Sie die 1024-Well-Stempel in die Agarose. Hinweis: Die Platte ist fertig, wenn abgekühlt zu bedienen.
- Auf der automatisierten Mikroinjektor Betriebssoftware, klicken Sie auf "Kalibrieren Bühne",dann klicken Sie auf '1024 'und Gitter und legen Sie die Agarose-Platte in der Mikroinjektor und kalibrieren die Platte, indem Sie auf dem Bildschirm in der Mittelposition des Brunnens.
- Gehen Sie zu 'Nadel-Menü "und klicken Sie auf" Kalibrieren Nadelhalter.
- Füllen Sie die Injektionsnadel mit einem Microloader Spitze mit entweder 10 ul pvp 40 mit 100 KBE / nl S. epidermidis oder 30 KBE / nl M. marinum oder Verwendung pvp 40 als Scheininjektion.
- Platzieren der Nadel in der automatisierten Mikro Injektor und Kalibrieren der x, y-Position durch Senken oder Bewegen der Nadel und Anklicken auf dem Bildschirm an der Position der Nadel. Dann kalibrieren die z-Position der Nadel, indem Sie auf dem Bildschirm auf die Position der Spitze der Nadel.
- Verteilen Sie die Eier über dem Agarose-Gitter mit einem Kunststoff-Transferpipette, und entfernen Sie überschüssiges Wasser Ei. Legen Sie die Agarose-Netz in der automatisierten Mikroinjektor.
- Gehen Sie auf die 'injection-Menü "und stellen Sie die" Einspritzdruck "-Einstellung auf 200 hPa," Einspritzzeit "0,2 sec und 'Compensation Druck" 15 hPa, die mit 1 nl korreliert, an der FemtoJet Einstellungen-Menü.
- Klicken Sie auf "Inject all 'die gesamte Platte zu injizieren.
- Sammeln der Eier nach der Injektion durch Waschen in einer Petrischale (92 x 16 mm), mit einem Maximum von 70 Embryos pro Petrischale, und Inkubieren bei 28 ° C.
5. Durchflusszytometer Analyse
- Bereiten Sie die große Partikel Durchflusszytometer entsprechend den Anweisungen des Herstellers und füllen Sie die Probenschale und Mantelflüssigkeitsbehälter mit Ei Wasser.
- Bei der Betriebssoftware, gehen Sie auf die "PMT"-Menü und verwenden Sie die folgenden Einstellungen: 650 V für die "Red"-Kanal und 0 V für die "Grün" und "Gelb"-Kanäle. Dann gehen Sie zu "Schwellen"-Menü und verwenden Sie die folgenden Einstellungen: "Optical Dichte 'Schwellensignal: 975 mV (COPAS XL-Wert: 50) und die "Time Of Flight" (TOF) mindestens bis 320 Mikrosekunden (COPAS XL-Wert: 800), um den Einfluss von Schmutz zu reduzieren.
- Für die Analyse ohne Sortierung die Embryonen gehen Sie zu Schritt 5.4, für die Analyse und Sortierung der Embryonen in eine Petrischale mit Schritt 5.5 oder für Analyse-und Sortier die Embryonen in eine 96-Well-Platte gehen Sie zu Schritt 5.6.
- Legen Sie die Embryonen in den Probenbecher und klicken Sie auf "Start", um die Analyse zu starten. Wenn alle Embryonen analysiert werden, stoppen Sie die Analyse durch einen Klick auf "Stop". Speichern Sie die Daten, indem Sie auf "Speichern Sie alle." Hinweis: Alle Daten werden als TXT, LMD-, DAT-, CSV-und BSRT Dateien gespeichert. Folgen Sie dem Protokoll bei Schritt 5.7.
- Legen Sie die Embryonen in den Probenbecher und das Maximum von 70 Embryonen pro Platte durch die Eingabe 70 im Menü 'Sortieren' sortiert werden sollen. Stellen Sie eine leere Petrischale unter dem Sortierer und klicken Sie auf 'Manual Sortieren'. Wenn der Petri di sh gefüllt ist, die Daten zu speichern, indem Sie auf "Speichern Sie alle." Hinweis: Alle Daten werden als TXT, LMD-, DAT-, CSV-und BSRT Dateien gespeichert. Folgen Sie dem Protokoll bei Schritt 5.7.
- Legen Sie die Embryonen in den Probenbecher und die maximal 1 pro Embryo gut durch Eingabe von 1 im Menü 'Sortieren' sortiert werden sollen. Stellen Sie eine leere 96-Well-Platte in der linken Schildhalter und klicken Sie auf 'Füllen Platte. Wenn die 96-Well-Platte gefüllt ist, die Daten zu speichern, indem Sie auf "Speichern Sie alle." Hinweis: Alle Daten werden als TXT, LMD-, DAT-, CSV-und BSRT Dateien gespeichert. Folgen Sie dem Protokoll bei Schritt 5.7.
- Holen Sie sich die TXT-Datei, um die Rohdaten verarbeiten, verwenden Sie die folgenden Daten Filtereinstellungen: 'Status wählen Sie ": 40, und wenn mit der Art Modul' Status-Art ':. 6 Dann nutzen Sie die Zahlen aus der Gesamtfluoreszenzsignal aus dem' Red "Kanal, um den Durchschnitt und die Standardabweichung vom Mittelwert zu berechnen. Zeichnen Sie diese Datensätze in bar oder Streudiagramme.
- Analysieren und Sortieren bei 3 Tage nach der Injektion (dpi), M. marinum infizierten Embryonen in zwei gleich große Gruppen mit dem großen Partikel Durchflusszytometer (Schritt 5.5). Behandeln Sie eine Gruppe mit einer Verbindung der Interesse an seiner Trägerlösungsmittel und andere Träger mit Lösungsmittel allein (Kontrolle). Gelten ähnliche Behandlungen, die injiziert Kontrolle Test für Antibiotika Nebenwirkungen zu verspotten.
- Wiederholen Sie bei 4 und 5 die dpi-Analyse (Schritt 5.5) und aktualisieren Sie die Ei-Wasser-oder Wasser-Ei, das die Verbindung.
7. High Resolution Imaging
- Anesthetize die Embryonen mit 0,02% (w / v) gepufferten 3-Aminobenzoesäureethylester (Tricaine) in Ei-Wasser 10 min vor der Analyse.
- Bereiten Sie die Wirbel Automatisierte Rastertechnologie System und die Große Partikelsammler nach den Anweisungen des Herstellers.
- Wählen Sie die Referenzbilder, die dem Alter der Embryonen aus dem "Imaging - Objekt211; Menü Erkennung Setup '.
- Wählen Sie die Anzahl der Bilder und die Orientierung durch die Wirbel automatisierten Screening-Technologie-System von der "Imaging - Auto-Speichern von Bildern" gemacht werden Menü.
- Legen Sie eine 96-Well-Platte mit Embryonen (aus Schritt 5.6) gefüllt ist, in der linken Schildhalter des Large Partikelsammler, und klicken Sie auf 'Run Platte.
- Wenn ein Embryo erkannt und korrekt positioniert ist; Bild der Kopf und der Schwanz mit dem CLSM separat mit einem 10X Klartrockenobjektiv und nähen die Bilder anschließend mit Hilfe von Bildverarbeitungssoftware.
Ergebnisse
Die vorliegenden Ergebnisse zeigen, dass die Hochdurchsatz-Pipeline nach S. zu studieren epidermidis und M. marinum Infektion erfolgreich hergestellt wurde und das kann zu anderen Infektionsmodellen erweitert werden. Erstens, die Verwendung von großen Zuchtbehälter (1A) auf der Grundlage der veröffentlichten System Adatto et al. (2011) 11, ermöglicht es, eine große Anzahl von Synchron Eier in Einzelereignisse Gewährleistung einer hohen Steuerung der Laichprozess zu erzeugen. Neben der Lage, große Anzahl von Embryonen in einem kurzen Zeitraum zu injizieren, um eine verbesserte Version des zuvor entwickelten automatisierten Mikroinjektionssystem 7 verwendet wurde (Fig. 1A) sein. Um abzuschätzen, welche die beste Entwicklungsstufe für Eigelb Infektion, Injektionen mit S. ist epidermidis und M. marinum wurden bei allen verschiedenen Stufen zwischen 1 und 512 Zellenstadium durchgeführt wird, entsprechend der Beschreibung von Kimmel et al. (1995) 12 .
Injektionen mit 100 KBE S. epidermidis zwischen 16 und 128-Zellen-Stadium, sofern die beste Infektion Muster (Abbildung 2). Die Bakterien in den Dotter vermehrt für 3 Tage und 3 dpi weiter in den Körper ausbreiten. Darstellende Injektionen vor dem 16-Zell-Stadium führte zu der hohen Sterblichkeit bei 4 dpi und nach der Injektion 256-Zellen-Stadium zeigte vor allem das Wachstum von Bakterien in der Dotter mit kaum Bakterien im Körper ausbreitet, des Embryos. Quantifizierung der Bakterienlast wurde durch Fluoreszenzintensitätsanalyse unter Verwendung der großen Partikel Durchflusszytometer durch Veneman et al (2013) 8 (Fig. 3) durchgeführt..
Beobachtungen zeigten, dass die optimale Entwicklungsstadium für die Injektion von 30 KBE M. marinum Injektion ist zwischen 16 bis 128 Zellen-Stadium für die E11-Stamm (4A) und zwischen 16 bis 64-Zell-Stadium mit dem virulenten M Strain (Fig. 4B). Embryonen dieser Stufen injiziert zeigten Bakterienwachstum innerhalb des Eigelbs und Ausbreitung der Bakterien durch den Embryo (Abbildung 7). Die Infektion mit beiden Stämmen in früheren Stadien präsentiert unspezifische generaliBakterienWachstum führt die Embryonen nach 4 dpi sterben. Auf der anderen Seite, in den Embryos in einem späteren Stadium injiziert bakterielle Belastung wurde zu dem Eigelb beschränkt.
Nächstes Vorsortierung mit großen Partikel Durchflusszytometer (Abbildung 1B) erzeugt großen homogenen Gruppen von infizierten Fischen ohne nicht oder stark infizierten Embryonen (5A und 6A). Nach der Vorsortierung, M. marinum infizierten Embryonen wurden mit Rifampicin, eine erste Linie Tuberkulose Medikament behandelt. Frühere Studien zeigten, dass die Behandlung mit Rifampicin in einer Dosis von 200 uM wirksam reduziert M. marinum Infektion in Zebrafisch-7, 13. Unter advantage der Vielzahl von homogen infizierten Embryos mit der hohen Durchsatzaufbau, der Behandlung mit verschiedenen Dosen durchgeführt wurde erzeugt. Embryonen mit M. infiziert marinum M-Stamm und für 48 h bei 12, 24 behandelt, und 200 &mgr; M ergab, Rifampicin, effizient mykobakteriellen Infektion in einer dosisabhängigen Weise (Fig. 5B) zu reduzieren. Im Hinblick auf eine effiziente Reduzierung der Infektion mit Rifampicin in einer Dosis von 200 uM wurde diese Konzentration für die zukünftige Experimente verwendet. Im Einklang mit dem vorherigen Ergebnis, Studium bakterielle Belastung Progression mit M. marinum E11 Belastung eine signifikante Reduktion von 24 h und weiter nach der Behandlung mit Rifampicin 200 uM beobachtet (6B).
Außerdem, wenn hohe Vergrößerung Bildgebung dieser Embryonen erforderlich sind, können sie automatisch in 96-Well-Platten (Fig. 1C), von wo die Proben analysiert werden können unter Verwendung angezeigt werdender Wirbel Automatisierte Rastertechnologie-System mit dem Large Partikelsammler auf eine CLSM montiert.
Der Wirbel Automatisierte Rastertechnologie-System mit dem Large Partikelsammler ist ein System, das entweder auf eine CLSM-oder Stereo-Mikroskop montiert werden kann. Diese Vorrichtung ermöglicht das Laden von lebenden oder fixierte Embryonen aus einem 96-Well-Platte oder Schüttgutbehälter automatisch durch eine Glaskapillare und orientiert sie vor der Kamera in dem gewünschten Winkel (z. B. Rücken-oder Seiten). Bilder des Embryos in allen Orientierungen kann mit der Kamera an Bord oder mit einer externen CLSM (Abbildung 7) vorgenommen werden. Die Embryonen werden in die Kollektion oder Abfallbehälter überführt werden.
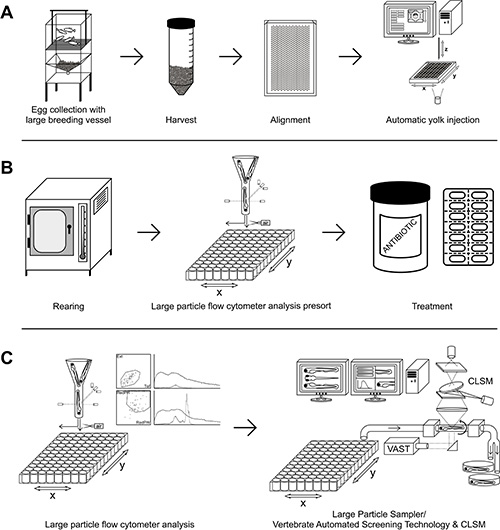
Abbildung 1. Mainstream experimentellen outline. A) Erwachsene Fische sind zusammen zu paaren, Eier werden gesammelt, in einem Agarose-Platte ausgerichtet und eingespritzt. B) Die injizierten Eier werden bei 28 ° C inkubiert und auf mögliche medikamentöse Behandlung vorsortiert werden. C) Die anschließende Analyse von großen Partikel Durchflusszytometer und / oder Groß Partikelsammler / Wirbel Automatisierte Rastertechnologie mit CLSM. Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
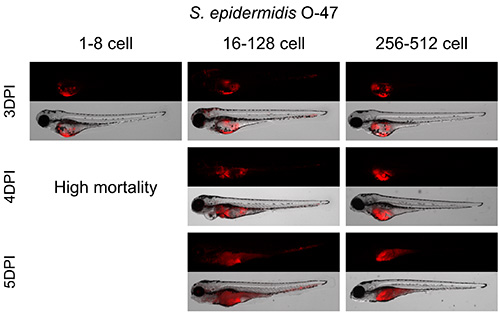
Abbildung 2. Gründung der besten Zellstadium für S. epidermidis Eigelb Injektion. Zebrafisch-Embryonen wurden in den Dotter in verschiedenen Entwicklungsstadien 1-512 Zell-Stadium mit 100 KBE von S. injiziert epidermidis. Embryonen zwischen 1 a eingespritztnd 8-Zell-Stadium zeigte das Bakterienwachstum im Eigelb und hohe Sterblichkeit von 4 dpi. Embryonen zwischen 16 und 128 Zellstadium injiziert zeigte das Bakterienwachstum im Eigelb und im Inneren des Körpers ab 3 dpi. Embryonen zwischen 256 und 512 Zellen-Stadium injiziert zeigten viele Bakterienwachstum in der Dotter. Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
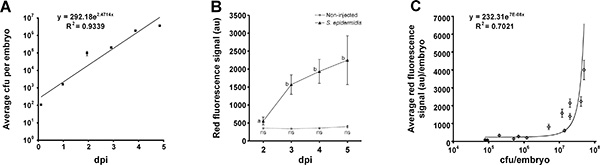
Abbildung 3. Quantifizierung der bakteriellen Belastung mit großen Partikel Durchflusszytometer. 100 KBE von S. epidermidis wurden in den Dotter von Zebrafisch-Embryonen injiziert. A) Bis zu 5 dpi, jeden Tag wurden Gruppen von 10 Embryonen homogenisiert und direkt überzogen, die das durchschnittliche exponentielle Wachstum, das auf zwei biologische Replikate (Fehlerbalken= SEM). B) Große Partikel Durchflusszytometer-Analyse zeigt die durchschnittliche Fluoreszenzsignal aus nicht-eingespritzt und S. epidermidis injizierten Embryonen. 30-160 Embryonen pro Bedingung analysiert (Fehlerbalken = SEM), verschiedene Buchstaben zeigen statistisch signifikante Unterschiede durch eine ANOVA mit Tukey-post-hoc-Test (P <0,001), gefolgt ns:. Keine signifikanten Unterschiede C) Zusammenhang zwischen KBE und durchschnittliche Fluoreszenzsignal von Gruppen von 10 S. epidermidis infizierten Embryonen (Fehlerbalken = SEM). Diese Zahl hat sich von Veneman et al geändert. (2013) 8. Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
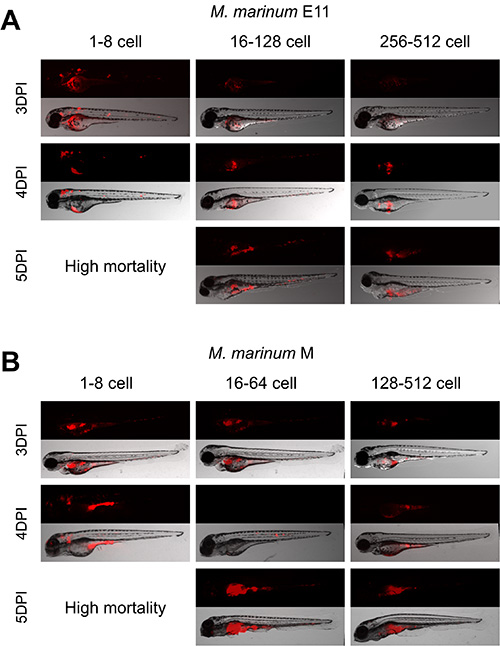
Abbildung 4. Establishment der besten Zellstadium für M. marinum Eigelb Injektion. Zebrafisch-Embryonen wurden auf all die verschiedenen Entwicklungsstufen von 1 bis 512 Zellen-Stadium mit 30 KBE M. injiziert marinum E11 und M-Stamm. A, B) Embryonen injiziert 1-8 Zell-Stadium zeigten ähnliche Verbreitung und Sterblichkeit, die mit beiden Stämmen. A) Embryonen injiziert zwischen 16-128 Zell-Stadium mit E11-Stamm zeigte Bildung von Granulomen und systemischen Infektion, während die eingespritzte 256-512 Zellen-Stadium gehalten bakterielle Belastung in den Dotter. B) Embryonen injiziert zwischen 16-64 Zellstadium mit M-Stamm zeigte die Bildung von Granulom Strukturen und systemischen Infektion, während die 128 bis 512 Zellen-Stadium injiziert gehalten Bakterienlast in den Dotter . Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.

5. Behandlung von M. marinum akute Infektion mit einem First-line-Anti-Tuberkulose-Medikament. Embryonen injiziert, zwischen 16 bis 64-Zell-Stadium mit 30 KBE M. marinum M Stammes wurden durch die großen Teilchen führen Durchflusszytometer bei 3 dpi in beiden Gruppen nach dem Verwerfen der nicht-und / oder stark infizierte Embryonen. A) Die Fluoreszenz der einzelnen Embryonen in beiden Gruppen sortiert werden. b) Embryonen mit Rifampicin (RIF behandelt ) für 48 Stunden in Dosen von 12, 24 und 200 &mgr; M wurden bei 4 dpi analysiert; die bakterielle Belastung erheblich reduziert. C) Repräsentative COPAS Profile von Embryonen mit DMSO und Rifampicin in Dosen von 12, 24 und 200 &mgr; M für 24 Stunden. Bakterielle Belastung und Verteilung wird durch die roten Spitzen angegeben. Blaue Linie stellt das Profil des Elements sortiert (4 dpf Zebrafisch-embryo) von den COPAS. 60-90 Embryonen pro Bedingung wurden analysiert. Jeder Datenpunkt stellt einen einzelnen Embryo. Die Werte sind angegeben als Mittelwert ± SEM. ns: nicht signifikante Unterschiede. Die Analyse der statistischen Signifikanz der Unterschiede wurde von einem ANOVA nach Tukey-Post-hoc-Test durchgeführt, gefolgt. Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
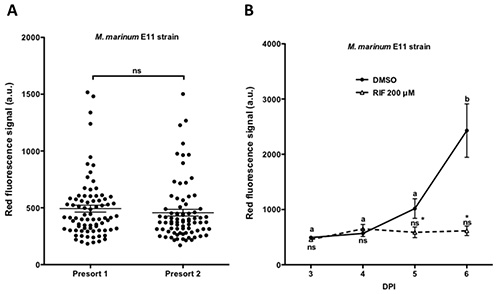
6. Behandlung von M. marinum chronische Infektion mit einer ersten Linie antituberculosis Droge. Embryonen injiziert, zwischen 16 bis 64-Zell-Stadium mit 30 KBE M. marinum E11 Stamm wurden durch die große Partikel Durchflusszytometer bei 3 dpi in beiden Gruppen nach dem Verwerfen der nicht-und / oder stark infizierte Embryonen sortiert werden ausgeführt. A) Die Fluoreszenz der einzelnen Embryonen in beiden Gruppen. B) Embryonen mit Rifampicin (RIF) bei 200 uM 4 Tage lang behandelt wurden, untersucht, die eine deutliche Reduktion der Keimzahl nach dem 1. Tag der Behandlung. 90 Embryonen pro Bedingung wurden analysiert. Die Werte sind angegeben als Mittelwert ± SEM. Unterschiedliche Buchstaben kennzeichnen signifikante Unterschiede zwischen den Zeitpunkten der gleichen Behandlung. * Zeigt signifikante Unterschiede zur Kontrollgruppe. ns: nicht signifikante Unterschiede. Die Analyse der statistischen Signifikanz der Unterschiede wurde von einem ANOVA nach Tukey-Post-hoc-Test durchgeführt, gefolgt. (P <0,05). Abbildung B) von Spaink et al geändert. (2013) 13. Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
hres.jpg "src =" / files/ftp_upload/51649/51649fig7.jpg "/>
Abbildung 7. Ergebnis M. marinum E11 Eigelb Injektion aufgenommen mit Wirbel automatisierten Screening-Technologie und CLSM. Konfokale Z-Stapel (genäht 3 Bilder) aus einem 5-dpi FLI1 egfp 14 Embryos. A) Live-Embryo zeigt Verbreitung von M. marinum E11 Bakterien (rot) im ganzen Körper. B) Fest 5 dpi fli-egfp Embryo zeigt M. marinum E11 Bakterien (rot) im ganzen Körper Co-Lokalisierung mit Leukozyten (hellblau) durch L-15 plastin Immunfärbung nachgewiesen.
Diskussion
Die in diesem Dokument beschriebenen Hochdurchsatz-Methodik ermöglicht eine schnelle und kostengünstige Pipeline zu hohe Anzahl von Fischembryonen und Larven mit verschiedenen Arten von Infektionen zu untersuchen. Mit der großen Zuchtbehälter statt der traditionellen Einzel-oder Familienzuchtbecken erleichtert die Steuerung des Laichprozesses und der Erzeugung von größeren Anzahl von Synchron Eier. Bei einer verbesserten Version des automatisierten Mikroinjektionssystem 7 ist es möglich, bis zu 2500 Eier fast alle innerhalb von 1 h spritzen in der gleichen Zellstadium. Mit diesen Aktualisierungen und verbesserte Software ist es möglich, mehr Eier als dies bisher möglich war, die verwendet werden kann, um große Arzneimittel Bildschirme mit Bakterienproliferation als Ausleseführen zu injizieren. Jedoch ist dieses Verfahren immer noch beschränkt auf Injektion andere Injektionswege beispielsweise von Benard et al Eigelb. (2012) 6, hoffentlich in der automatisierten Mikroinjektionssystem in naher Zukunft aufgenommen werden.
Obwohl diese Methoden für das Screening von Zebrafisch-Benchmarking, wäre es sinnvoll für Anwendungen mit anderen Fischarten als gut. Zum Beispiel hat der Karpfen angedeutet wurde, um Vorteile für Drogen-Bildschirme haben. Wie Zebrafisch, sind Eier und Embryonen im Frühstadium von Karpfen transparent, aber mit dem Vorteil der großen Laich Größe der hunderttausende von Eiern und die Verfügbarkeit von Inzuchtlinien, die eine konstante genetischen Hintergrund 16 anbieten.
Die Analyse von großen Mengen von infizierten Embryos mit dem hohen Durchsatz großer Partikel Durchflußzytometer durchgeführt. Dieses Gerät kann Art in Multi-Well-Platten oder eine Petrischale und ist daher besonders geeignet für die Prüfung einer großen Anzahl von Verbindungen analysiert Embryonen. Wenn eine höhere Bildauflösung erforderlich ist, als die Einrichtung ist so angepaßt, daß die großen Teilchen Durchflusszytometer Technologie kann für Pre-Screening verwendet werden, und anschließend die Proben analysieren zu einem Medium durchsetzen mit einer höheren Auflösung. Dies kann mit dem Wirbel Automatisierte Rastertechnologie 17, 18 durchgeführt werden. Dieses Gerät kann automatisch sammeln Live-oder Fest Embryonen zwischen 2 und 7 Tage nach der Befruchtung aus einer Mehr Well-Platte oder Schüttgut-Container, 360 °-Bild durch eine Kapillare mit CLSM-oder Stereo-Mikroskopie und entsorgen Sie wieder in zwei Großcontainern eine manuelle Sortierung der Embryonen auf Basis auf der mikroskopischen Bilder. Zukünftige Verbesserungen werden die Sortierung der Embryo nach der Bilderzeugung in den Multi-Well-Platte zu ermöglichen, wodurch es möglich ist, automatisch Vielzahl von einzelnen Embryonen im Laufe der Zeit mit CLSM screenen. Unter der Annahme, dass in zukünftigen Anwendungen der Wirbel automatisierten Screening-Technologie-System kann auch auf die große Partikel Durchflusszytometer Technologie ohne die Notwendigkeit von vor Abgabe Larven in Multi-Well-Platten angeschlossen werden, werden zu einer erweiterten Sortier führen.
Dieser Beitrag beschreibt die Einrichtung eind Optimierung eines Hochdurchsatz-Setup, um zu studieren S. epidermidis und M. marinum Infektion als Modell für die Wirkstoffforschung. Sie zeigt, dass das Ergebnis dieser Bakterien in den Dotter injiziert abhängig vom Entwicklungsstadium der Eier bei der Injektion. Injizieren M. marinum E11 bei 16 bis 128 Zellen-Stadium oder die M-Stamm bei 16 bis 64-Zell-Stadium führt zu dem gleichen Muster wie Infektion Schwanzveneninjektion 2, 6. Aber diese Konfiguration ist nicht auf die Proliferation von nur bakterielle Pathogene beschränkt. Es wurde gezeigt, dass, bevor es möglich ist, Roboter zu injizieren Lösungen, die DNA, RNA oder Morpholinos für Transgenese, Überexpression und Gen-Knock-down-Studien, bzw. 13. Weiterhin wurde gezeigt, daß dieser Aufbau ist auch nützlich für das Studium der Krebszellproliferation und Migration. Diese Leitung stellt somit ein vielseitiges Verfahren für Hochdurchsatz-Screening einer Vielzahl von Signalmechanismen im Zusammenhangder angeborenen Immunität, angewendet auf Infektionskrankheiten und die Entwicklung von Krebs. Diese Bildschirme können mit anderen, für die Medizin Entdeckung aber auch mit Analyse der möglichen toxischen Wirkungen von Drogen identifiziert anwendbar kombiniert werden.
Offenlegungen
JS ist Inhaber der Life Science-Methoden BV, die Instrumente in diesem Artikel verwendet produziert.
Danksagungen
Wir sind dankbar, dass Leonie de Boer und Bas Zaat (Academic Medical Centre) für die Bereitstellung der S. epidermidis O-47-Stamm. Wir danken Rico Bongaarts, Francis Smet und Angela Comas (Union Biometrica) um Hilfe und Rat mit der COPAS XL und VAST BioImager Analyse. Wir danken Davy de Witt, Ulrike Nehrdich und Laura van Hulst für Fische Hausmeister und andere Kollegen von der Universität Leiden für hilfreiche Diskussionen. Diese Forschung ist Teil der Projekt P5.03 IBIZA des Forschungsprogramms des Instituts biomedizinischen Materialien, die vom niederländischen Wirtschaftsministerium mitfinanziert, und der Smart-Mix-Programm (NWOA_6QY9BM) des niederländischen Ministeriums für Wirtschaft und des Das niederländische Ministerium für Bildung, Kultur und Wissenschaft. Zusätzliche Unterstützung wurde von dem EU-Projekt ZF-Health (FP7-Health-2009-242048) erhalten, und RMJ wurde von Marie-Curie-Stipendium als erfahrener Forscher in der EU Initial Training Network FishForPharma (PITN-GA-201 unterstützt1-289209). SJR mit Mitteln aus der Innovative Medicines Initiative Joint Undertaking unter Finanzhilfevereinbarung Nr. 115337, Ressourcen, von denen die finanzielle Beteiligung Siebten Rahmenprogramms der Europäischen Union (FP7/2007-2013) und der EFPIA-Unternehmen in Art contribution.The Autoren erkennen weiterhin bestehen finanzielle Unterstützung von der Universität Leiden Fonds (LUF) für Robotik und aus Cyttron, in der Besluit Subventionen Investeringen Kennisinfrastructuur Programm, das wiederum wird finanziell von der niederländischen Organisation für wissenschaftliche Forschung zur Bildgebung Einrichtungen unterstützt. Der Erwerb COPAS System wurde teilweise von der Abteilung für Erd-und Biowissenschaften (ALW) mit finanzieller Hilfe von der niederländischen Organisation für wissenschaftliche Forschung (NWO, 834.10.004) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Middlebrook 7H10 agar | BD Bioscience, Franklin Lakes, New Jersey, USA | 262710 | |
| Middlebrook 7H9 broth | BD Bioscience, Franklin Lakes, New Jersey, USA | 271310 | |
| BBL Middlebrook oleic acid albumin dextrose catalase (OADC) enrichment | BD Bioscience, Franklin Lakes, New Jersey, USA | 211886 | |
| BBL Middlebrook albumin dextrose catalase (ADC) enrichment | BD Bioscience, Franklin Lakes, New Jersey, USA | 211887 | |
| LB agar | Sigma-Aldrich, Saint Louis, Missouri, USA | L5542 | Multiple suppliers |
| LB broth | Sigma-Aldrich, Saint Louis, Missouri, USA | L3022 | Multiple suppliers |
| Chloramphenicol | Bio-connect, Huissen, the Netherlands | 16785.03 | Multiple suppliers |
| Hygromycin | Bio-connect, Huissen, the Netherlands | 25966.01 | Multiple suppliers |
| Tween-80 | Sigma-Aldrich, Saint Louis, Missouri, USA | P1754 | Multiple suppliers |
| Polyvinylpyrrolidone40 | Calbiochem, San Diego, California, USA | 529504 | Multiple suppliers |
| Ethyl 3-aminobenzoate methanesulfonate salt (Tricaine) | Sigma-Aldrich, Saint Louis, Missouri, USA | A5040 | |
| Agarose | Sphaero-Q, Gorinchem, the Netherlands | S103 | Multiple suppliers |
| Instant ocean sea salt | Sera Marin, Heinsberg, Germany | 5460 | |
| iSPAWN | Techniplast, Buguggiate, Italy | iSPAWN | |
| Automated microinjection system | Life Science Methods BV, Leiden, the Netherlands | Automated microinjection system | |
| Complex Object Particle Analyzer and Sorter XL (COPAS XL) | Union BioMetrica Inc., Holliston, Massachusetts, USA | COPAS XL | |
| Vertebrate Automated Screening Technology BioImager (VAST BioImager) | Union BioMetrica Inc., Holliston, Massachusetts, USA | VAST BioImager | |
| LP Sampler | Union BioMetrica Inc., Holliston, Massachusetts, USA | LP Sampler | |
| Confocal laser scanning microscope | Leica Microsystems, Wetzlar, Germany | TCS SL | |
| Injection needle 10 µm inner diameter | Qvotek, Mississauga, Canada |
Referenzen
- Meijer, A. H., Spaink, H. P. Host-pathogen interactions made transparent with the zebrafish model. Curr Drug Targets. 12, 1000-1017 (2011).
- Tobin, D. M., Ramakrishnan, L. Comparative pathogenesis of Mycobacterium marinum and Mycobacterium tuberculosis. Cell Microbiol. 10, 1027-1039 (2008).
- Boelens, J. J., et al. Biomaterial-associated persistence of Staphylococcus epidermidis in pericatheter macrophages. J Infect Dis. 181, 1337-1349 (2000).
- Broekhuizen, C. A., et al. Tissue around catheters is a niche for bacteria associated with medical device infection. Crit Care Med. 36, 2395-2402 (2008).
- Busscher, H. J., et al. Biomaterial-associated infection: locating the finish line in the race for the surface. Sci Transl Med. 4, (2012).
- Benard, E. L., et al. Infection of zebrafish embryos with intracellular bacterial pathogens. J Vis Exp. , (2012).
- Carvalho, R., et al. A high-throughput screen for tuberculosis progression. PLoS One. 6, (2011).
- Veneman, W. J., et al. A zebrafish high throughput screening system used for Staphylococcus epidermidis infection marker discovery. BMC Genomics. 14, 255 (2013).
- Sar, A. M., et al. Mycobacterium marinum strains can be divided into two distinct types based on genetic diversity and virulence. Infect Immun. 72, 6306-6312 (2004).
- Stoop, E. J. M., et al. Zebrafish embryo screen for mycobacterial genes involved in the initiation of granuloma formation reveals a newly identified ESX-1 component. Disease Model., & Mechanisms. 4, 526-536 (2011).
- Adatto, I., et al. A new system for the rapid collection of large numbers of developmentally staged zebrafish embryos. PLoS One. 6, (2011).
- Kimmel, C. B., et al. Stages of embryonic development of the zebrafish. Dev Dyn. 203, 253-310 (1995).
- Spaink, H. P., et al. Robotic injection of zebrafish embryos for high-throughput screening in disease models. Methods. 62, 246-254 (2013).
- Lawson, N. D., Weinstein, B. M. In vivo imaging of embryonic vascular development using transgenic zebrafish. Dev Biol. 248, 307-318 (2002).
- Mathias, J. R., et al. Live imaging of chronic inflammation caused by mutation of zebrafish Hai1. J Cell Sci. 120, 3372-3383 (2007).
- Henkel, C. V., et al. Comparison of the exomes of common carp (Cyprinus carpio) and zebrafish (Danio rerio). Zebrafish. 9, 59-67 (2012).
- Chang, T. -. Y., et al. Fully automated cellular-resolution vertebrate screening platform with parallel animal processing. Lab on a Chip. 12, 711 (2012).
- Pardo-Martin, C., et al. High-throughput in vivo vertebrate screening. Nature Methods. 7, 634-636 (2010).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten