Method Article
Überwachung räumliche Trennung in Oberflächen Colonizing mikrobiellen Populationen
In diesem Artikel
Zusammenfassung
Die Rolle des Sortiments (räumliche Trennung) in evolutionären Szenarien können mit einfachen mikrobiellen Systemen im Labor untersucht werden, die eine Anpassung der räumlichen Verteilung gesteuert ermöglichen. Durch eine Modifikation können die Gründer Zelldichte, verschiedene Sortiment Ebenen fluoreszenzmarkierte Bakterienstämme in der Kolonie Biofilme von Bacillus subtilis verwendet werden sichtbar gemacht .
Zusammenfassung
Microbes provide an intriguing system to study social interaction among individuals within a population. The short generation times and relatively simple genetic modification procedures of microbes facilitate the development of the sociomicrobiology field. To assess the fitness of certain microbial species, selected strains or their genetically modified derivatives within one population, can be fluorescently labelled and tracked using microscopy adapted with appropriate fluorescence filters. Expanding colonies of diverse microbial species on agar media can be used to monitor the spatial distribution of cells producing distinctive fluorescent proteins.
Here, we present a detailed protocol for the use of green- and red-fluorescent protein producing bacterial strains to follow spatial arrangement during surface colonization, including flagellum-driven community movement (swarming), exopolysaccharide- and hydrophobin-dependent growth mediated spreading (sliding), and complex colony biofilm formation. Non-domesticated isolates of the Gram-positive bacterium, Bacillus subtilis can be utilized to scrutinize certain surface spreading traits and their effect on two-dimensional distribution on the agar-solidified medium. By altering the number of cells used to initiate colony biofilms, the assortment levels can be varied on a continuous scale. Time-lapse fluorescent microscopy can be used to witness the interaction between different phenotypes and genotypes at a certain assortment level and to determine the relative success of either.
Einleitung
In den letzten Jahrzehnten wurden Mikroben , die als soziale Gemeinschaften im Zusammenhang mit verschiedenen Ökosysteme auf der Erde 1,2 anerkannt. Im Gegensatz zu Plankton-Kulturen im Allgemeinen Laborpraxis verwendet, Mikroben in der Umwelt zeigen ein breites Spektrum von räumlichen Strukturen Gemeinschaft auf die ökologische Einstellung abhängig. Einfache mikrobiellen Systemen können die Folge von räumlichen Strukturen über die Entwicklung der sozialen Interaktionen 3,4 zu verstehen , genutzt werden. Veröffentlichungen in den letzten 2-3 Jahren sowohl eukaryotische und prokaryotische Modellsystemen unter Verwendung von hervorgehoben , die Auswirkungen der räumlichen Strukturen auf die Stabilität der Zusammenarbeit innerhalb der mikrobiellen Populationen 5-8. Zusätzlich obligat Wechselwirkungen zwischen Mikroben, wie zB Stoffwechselkreuzfütterung, kann auch die räumliche Verteilung der interagierenden Partner 9-11 verändern. Der Einfluss der räumlichen Struktur in diesen Studien wird meist aus Oberfläche angebracht sessile Zellen untersucht, die so bewohnen-called Biofilmen oder in Kolonien auf der Oberfläche eines Agar-Medium wachsen. Genetic drift in hoher räumlicher Zusammenstellung , kann sie in mikrobiellen Kolonien , wo Nährstoffverarmung am Rand einer Zellteilung vermittelten Expansions Ergebnisse in Reihe von genetischen Engpässe festzustellen , dass hohe lokale Fixierung Wahrscheinlichkeit für bestimmte klonalen linages 12 verursacht. Genetische Drift kann daher die Rolle der räumlichen Segregation in mikrobiellen Kolonien zu untersuchen, eingesetzt werden.
In der Umwelt sind Biofilme für mehrere Arten von selbst produzierten Polymermatrix 13 umgeben Gemeinden. Biofilm Struktur, Funktion und Stabilität hängen von einem komplexen Netzwerk von sozialen Interaktionen , wo Bakterien Austauschsignale, Matrixkomponenten und Ressourcen oder für Raum und Nährstoffe mit Toxine und Antibiotika konkurrieren. Bacillus subtilis ist ein im Boden lebenden und Bakterium Wurzel-Kolonisierung , die sehr organisiert entwickelt Biofilmgemeinschaften 14. In Analogie zur sozialenInsekten, B. subtilis - Zellen verwenden eine Arbeitsteilung Strategie, Subpopulationen von extrazellulären Matrix Produzenten und Kannibalen, bewegliche Zellen, ruhende Sporen und anderen Zelltypen entwickeln 15,16. Der Differenzierungsprozess ist dynamisch und kann durch Umgebungsbedingungen 17,18 verändert werden.
Strategien der Oberflächenbesiedlung durch Bakterien kann leicht durch Modifizieren der Agar-Konzentration im Wachstumsmedium unter Laborbedingungen manipuliert werden. Bei niedrigen Agar Ebenen (0,2-0,3%), Bakterien aktiv Geißeln beherbergen können schwimmen, während halbfeste Agar (0,7-1% Agar) Verbreitung flagellum angetrieben Gemeinschaft erleichtert, genannt 19-21 Schwärmen. In Abwesenheit von Flagellen sind bestimmte Bakterienstämme können über halbfesten Medium zu bewegen , über Schiebe-, dh Wachstum abhängige Bevölkerungswachstum 22-24 von Exopolysaccharidmaterial Matrix und anderen abgesondert Hydrophobin Verbindungen erleichtert. Schließlich, die Bakterien sind capable der Biofilmentwicklung bilden architektonisch komplexe Kolonien auf harten Agar - Medium (1,2-2%) 14,17,25. Während diese Merkmale genau im Labor untersucht werden durch Einstellen der Bedingungen, in natürlichen Lebensräume diese Oberfläche ausbreitenden Strategien könnte Transit nach und nach von einem zum anderen in Abhängigkeit von den Umgebungsbedingungen 26. Während einzelne Zelle basierte Motilität ist zu Beginn der Biofilmentwicklung an der Luft-Flüssigkeit - Grenzfläche sowohl Gram-positive und -negative Bakterien 27, komplexe Kolonie Biofilme von B. kritisch subtilis nicht durch Streichung von Flagellen Motilität 28 betroffen. Allerdings räumliche Organisation bei der Entwicklung von B. subtilis Kolonie Biofilmen hängt von der Dichte des bakteriellen Inokulums verwendet , um den Biofilm 8 einzuleiten.
Hier verwenden wir B. subtilis zu zeigen , dass die räumliche Segregation während der Oberflächenbesiedlung hängt von dem Mechanismus der Bevölkerung Ebene motility (dh Schwärmen oder Gleiten) und Kolonie Biofilmentwicklung hängt von der Dichte Gründerzelle. Wir präsentieren ein Fluoreszenz-Mikroskopie-Tool, das kontinuierlich angewendet werden kann, um mikrobiellen Biofilmwachstum, Oberflächenbesiedlung und das Sortiment auf der Makroskala überwachen. Ferner wird ein Quantifizierungsverfahren die relative Dehnungsfluss in der Population zu bestimmen, dargestellt.
Protokoll
1. Herstellung von Kulturmedien, halbfeste Agar und Biofilm-Platten, Pre-Kulturen
- Medium Vorbereitung für Schwärmende und Sliding
- Man löst 2 g Lenox-Bouillon (LB) und 0,7 g Agar-Agar in 100 ml ionenausgetauschtes Wasser und Autoklaven für 20 min bei 120 ° C. Verwenden Sie kleine Volumina (50-200 ml) Reproduzierbarkeit zwischen den Experimenten zu verbessern.
- Unmittelbar nach der Sterilisation, schließen Sie die Kappe des Mediums Flasche für mindestens 2 Stunden Verdunstung und in einem 55 ° C Inkubator zu reduzieren.
- Nachdem die Temperatur des Mediums auf 55 ° C temperiert wurde, gieße 20 ml Agar-LB-Medium in einem 90 mm Durchmesser Petrischale aus Polystyrol unter einem Labor sterile Haube. Für Zeitraffer-Experimente, gießen 5 ml Agar LB-Medium pro 35 mm Durchmesser Petrischale aus Polystyrol.
- Schließen Sie die Petrischale unmittelbar nach dem Gießen, Stapel nicht mehr als 4 Platten auf der jeweils anderen und lassen Sie die Agar-Medium für mindestens 1 Stunde fest werden.
- 2xSG Medium PWiedergutmachung für Colony Biofilme
- Man löst 1,6 g Nutrient Broth, 0,2 g KCl, 0,05 g MgSO 4 7H 2 O und 1,5 g Agar-Agar in 100 ml ionenausgetauschtes Wasser und Autoklaven für 20 min bei 120 ° C. Verwenden Sie kleine Volumina (50-200 ml) Reproduzierbarkeit zwischen den Experimenten zu verbessern.
- Unmittelbar nach der Sterilisation, schließen Sie die Kappe des Mediums Flasche Verdunstung zu reduzieren und die Flasche in einem 55 ° C Inkubator für mindestens 2 Stunden statt.
- Nachdem die Temperatur des Mediums selbstjustierte bis 55 ° C, 0,1 ml Filter hinzufügen sterilisierte 1M Ca (NO 3) 2 -Lösung, 0,1 ml sterilfiltriert 100 mM MnCl & sub2 ; -Lösung, 0,1 ml sterilfiltriert 1 mM FeSO & sub4 ; -Lösung und 0,5 ml sterile 20% ige Glucoselösung.
- In einem Labor sterilen Haube, gießen 20 ml Agar 2x SG Medium pro 90 mm Durchmesser Petrischale aus Polystyrol. Für Zeitraffer-Experimente, gießen 5 ml Agar LB-Medium pro 35 mm Durchmesser Petrischale aus Polystyrol.
- Close die Petrischale unmittelbar nach dem Gießen, stapeln die Platte auf der jeweils anderen, aber nicht mehr als 4 Platten, und lassen Sie die Agar-Medium für mindestens 1 Stunde zu verfestigen.
- Herstellung von Starterkulturen
HINWEIS: Die B. subtilis 168, NCIB 3610 Derivatstämme in den Verfahren unter konstitutiv beschrieben verwendet produzieren grün- oder rot-Fluoreszenz - Proteine und wurden vor 8,27 beschrieben. Stämme werden routinemßig in der -80 ° C Gefrierschrank gelagert.- Impfen Starterkulturen von -80 ° C Bestände in 3 ml LB-Medium und Inkubation über Nacht (16-18 Stunden) bei 37 ° C mit horizontalem Schütteln (225 rpm). Nicht die Kultur brüten länger als 18 Stunden so wild von B. isoliert subtilis sind meist anfällig für einen Biofilm in dem Teströhrchen zu aggregieren und bilden.
2. Co-Inokulation von fluoreszenz Labelled Bakterienstämme für die Oberflächen Verbreitung
- Das Trocknen von halbfesten Agar-Platten für Schwarms undSliding von B. subtilis.
- Trocken Agarplatten für Schwärmen und 20 min vor der Inokulation schieben. Trockenplatten in einer Laminar - Flow - Haube aufgedeckt (siehe Abbildung 1).
HINWEIS: Bacterial swarming und Schiebe hängt von der Trockenheit der halbfestes Agarmedium. Unzureichende Trocknung ermöglicht die Ansammlung von Wasser auf dem Agar-Medium in flagellum vermittelte Schwimmen führt. Eine verlängerte Trocknungszeit führt zu Mangel an Schwärmen.
- Trocken Agarplatten für Schwärmen und 20 min vor der Inokulation schieben. Trockenplatten in einer Laminar - Flow - Haube aufgedeckt (siehe Abbildung 1).
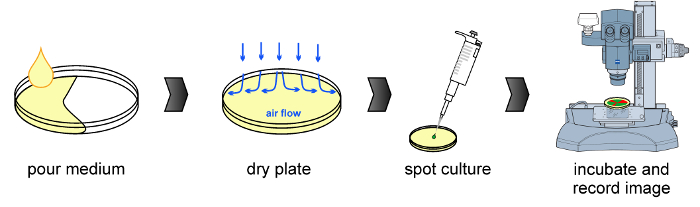
Abbildung 1:.. Experimental Workflow Das einheitliche Verfahren in der Figur dargestellt ist, einschließlich der Vorbereitung des Kulturmedium, um die Platte Trocknen, Inokulation und Fluoreszenzmikroskopie - Detektion (von links nach rechts) Bitte hier klicken , um eine größere Version dieser Figur zu sehen.
- Co-Impfungvon Bakterienkulturen für Schwärmende und Sliding
- Bestimmen die optischen Dichten der über Nacht Starterkulturen bei 600 nm und die Mischungsdichte normalisierten grün- und rot-fluoreszierendes Protein Stämme von B. Herstellung von subtilis NCIB 3610 oder seine Δ hag - Derivat in einem 1,5 ml Reaktionsgefäß. Zum Beispiel mischen 100 ul Stamm 1 mit (100 * [OD 600 der über Nacht Kultur des Stammes 1] / [OD 600 von über Nacht Kultur des Stammes 2]) ul Stamm 2. Mildly Wirbel (3 s bei einer maximalen Drehzahl) zum homogene Verteilung.
HINWEIS: B. subtilis NCIB 3610 Stämme beimpft Schwärmen zu beobachten, während ihre Δ hag - Derivate verwendet werden , zum Gleiten. - Spot 2 ul Mischkultur auf der Mitte eines vorgetrockneten Platte (siehe 1) und trocknen weiter die Platte für 10 min nach der Inokulation.
- Inkubiere Platten bei 37 ° C aufrecht überschüssige Feuchtigkeit zu ermöglichen, sich auf dem Deckel und nicht auf der Agaroberfläche zu kondensieren.
Hinweis: Die Inkubationszeit für B. subtilis Schwärmen ist in der Regel zwischen 16.08 h. Im Allgemeinen erreicht die Kante des Schwarms die Seite der 90 mm-Petrischale in 8 Stunden. Sliding ist ein langsamer Prozess und erfordert mindestens 16 bis 42 Stunden Inkubation. Nach 36 Stunden erreicht die gleitende Vorderseite die Seite des 90 mm Petrischale. - Für Zeitraffer-Experimente, legen Sie die 35 mm Durchmesser Petrischalen in einem vorgewärmten Bühne Inkubationskammer bei 37 ° C eingestellt. Sicherzustellen, dass der Deckel der Petrischale bleibt während der Dauer des Versuchs entfernt. Legen Sie die Abdeckung der Bühne Inkubator bis 40 ° C Feuchtigkeitsbildung auf der Oberseite des Inkubators zu umgehen.
- Bestimmen die optischen Dichten der über Nacht Starterkulturen bei 600 nm und die Mischungsdichte normalisierten grün- und rot-fluoreszierendes Protein Stämme von B. Herstellung von subtilis NCIB 3610 oder seine Δ hag - Derivat in einem 1,5 ml Reaktionsgefäß. Zum Beispiel mischen 100 ul Stamm 1 mit (100 * [OD 600 der über Nacht Kultur des Stammes 1] / [OD 600 von über Nacht Kultur des Stammes 2]) ul Stamm 2. Mildly Wirbel (3 s bei einer maximalen Drehzahl) zum homogene Verteilung.
3. Co-Inokulation von fluoreszenz Labelled Bakterienstämme mit unterschiedlichen Anfangszelldichten
- Das Trocknen von Agar - Platten für Colony Biofilmbildung von B. subtilis.
- Trocknen der Platten zur Koloniebiofilmentwicklung ohne Abdeckung in einer laminaren StrömungHaube für 15 min vor der Impfung.
HINWEIS: Zu wenig Trocknung führt zu einer erhöhten Luftfeuchtigkeit und Schwimmen oder Schwärmen kann möglich 29 sein. Das Trocknen zu lange Ergebnisse in kleinen Biofilm Kolonien.
- Trocknen der Platten zur Koloniebiofilmentwicklung ohne Abdeckung in einer laminaren StrömungHaube für 15 min vor der Impfung.
- Herstellung von 10-fach verdünntem Starterkulturen für Colony Biofilme
- Mischen Sie 100 ul von grün- und rot-fluoreszierenden Proteins über Nacht Starterkulturen von B. subtilis 168 in einem 1,5 ml Reaktionsgefäß und leicht Wirbel für eine homogene Verteilung. Herstellung von 10-fach Verdünnungsreihen in LB-Medium.
- Spot 2 & mgr; l unverdünnter oder 10 1, 10 2, 10 3, 10 4 verdünnt Mischkulturen auf der Platte enthält Biofilm-induzierenden Medium.
HINWEIS: 6 bis 9 Biofilm-Kolonien können auf einem einzigen 90 mm Petrischale eingeleitet werden unter Berücksichtigung, dass die Kolonien in gleichem Abstand voneinander getrennt sind. - Inkubiere Platten bei 30 ° C aufrecht überschüssige Feuchtigkeit zu ermöglichen, sich auf dem Deckel zu kondensieren undnicht auf der Agaroberfläche.
HINWEIS: Die Inkubationszeit für B. subtilis Biofilm zwischen 1 bis 3 Tagen. Im Allgemeinen ist die Kolonie Biofilm von B. subtilis erreicht seine durchschnittliche Größe und komplexe Struktur in 2 Tagen. - Für Zeitraffer-Experimente, legen Sie eine einzelne Inokulum in der Mitte von einem Durchmesser von 35 mm Petrischale und die Schale in einem vorgewärmten Stufe Inkubationskammer bei 30 ° C eingestellt. Stellen Sie sicher, dass die Oberseite der Petrischale bleibt während der Dauer des Versuchs entfernt. Legen Sie die Abdeckung der Bühne Inkubator auf 35 ° C Feuchtigkeitsbildung auf der Oberseite des Inkubators zu umgehen.
4. Fluoreszenz-Mikroskopie Nachweis von Labelled Dehnungen
- Beschreibung Ausrüstung für Imaging.
- Zur Oberflächenbesiedlung und Fluoreszenzsignal, mit einem motorisierten Fluoreszenz Stereo - Zoom - Mikroskop (siehe detaillierte Liste in der Materialtabelle) , ausgestattet mit einem 0,5 - fach PlanApo Ziel erkennen, zwei LEDKaltlichtquellen (eine für Fluoreszenzdetektion und eine für das sichtbare Licht), Filtersätze für GFP (Anregung bei 470/40 nm und einer Emission bei 525/50 nm) und mRFP (Anregung bei 572/25 nm und einer Emission bei 629 / 62 nm) und eine hochauflösende Monochrom-Kamera.
- Führen Sie die Bildaufnahme und Verarbeitung mit entsprechender Software zur Verfügung für den Stereo-Zoom-Mikroskop mit Mehrkanal- und Zeitraffer-Module. Für Verstreichen Experiment Zeit, verwenden Sie eine Standard-Inkubator Heizstufe mit dem Stereo-Zoom-Mikroskop montiert mit einem Adapter.
- Imaging von Schwärmen und Sliding Expansion
- Verwenden Sie die niedrigste Vergrößerung die größtmögliche Fläche der 90-mm-Platte zu erfassen. Stellen Sie den Ursprung der Inokulation (Mitte des 90 mm-Petrischale) an die Ecke des Sichtfeldes zur Überwachung radial Bakterien Expansion und Fluoreszenz.
- Einstellen optimale Belichtungszeit abhängig von der Stärke des Fluoreszenzsignals.
HINWEIS: Für konstitutiv expressed Fluoreszenz - Gene in B. subtilis, grün- und rot-Fluoreszenz mit 1,5 und 3 sec Belichtungszeiten können jeweils verwendet werden. Zusätzlich 10 ms Belichtungszeit ist für sichtbares Licht geeignet.
- Verwenden Sie die Vergrößerung, die die Erfassung des gesamten Biofilm Kolonie und stellen Sie die Kolonie in der Mitte des Sichtfeldes ermöglicht.
HINWEIS: Wie für Schwärmen und Schiebe Ausdehnungen, die optimalen Belichtungszeiten der Fluoreszenzsignale in den Biofilm-Kolonien nachzuweisen, hängt von der Expressionsgrad des fluoreszierenden Proteins kodierenden Gene. Für die repräsentativen Ergebnisse unter, wurde grün- und rot-Fluoreszenz detektiert unter Verwendung von 1 und 3 Sekunden Belichtungsintervalle sind. - Für Zeitraffer-Bildgebung, erhalten Bilder in bestimmten Intervallen konstanter Belichtungszeiten.
- Speichern Sie die Fluoreszenz-Stereo aufgenommenen Bilder in einem Dateiformat, das von ImageJ Software für quantitative Datenanalyse erkannt wird.
5. Dateine Analyse
- Um den Bereich von jedem unterschiedlich markierten fluoreszierenden Stamm besetzt analysieren, öffnen Sie die Datei von Interesse in ImageJ-Software mit einem BioVoxxel Plugin erweitert.
- Wenn ein Fenster "Bio-Formate Import-Optionen" erscheint genannt, wo nur die Optionen "alle Serien Öffnen" und "automatische Skalierung" ausgewählt sind, öffnen Sie die Datei, indem Sie auf "OK" klicken.
HINWEIS: Die Dateien als ein Stapel von drei Bildern angezeigt werden, eine für jeden Kanal verwendet, um ein Bild in dem Mikroskop zu erfassen (grün- roten Fluoreszenz und Hellfeldbilder). - Trennen Sie den Stapel in einzelne Kanal Bilder durch Auswahl von "Bild" - "Stacks" - "Stack auf Bilder" in der ImageJ Bedienfeld.
HINWEIS: Die Bilder erscheinen und werden als 1/3 (grünen Kanal) nummeriert, 2/3 (roter Kanal) und 3/3 (Hellfeld). Hier wird die Hellfeld-Bild wird von der Analyse ausgeschlossen.
- Wenn ein Fenster "Bio-Formate Import-Optionen" erscheint genannt, wo nur die Optionen "alle Serien Öffnen" und "automatische Skalierung" ausgewählt sind, öffnen Sie die Datei, indem Sie auf "OK" klicken.
- Um die Bilder zu analysieren, zu transformieren jeweils in einen 8-Bit-Bild durch Auswahl"Bild" - "Type" - "8-Bit".
- Um die belegte Fläche in Pixel bestimmen 2, setzen den Maßstab der Bilder "Analyze" mit - "Set Scale". Wenn ein Fenster mit verschiedenen Skalierungsoptionen erscheint, stellen Sie die Waage, indem Sie "klicken, Zunder zu entfernen". Aktivieren Sie die Option "Global" den Maßstab für alle geöffneten Bilder zu entfernen.
- Um den Hintergrund zu entfernen, ziehen einen ovalen Bereich (Region of Interest, ROI) außerhalb des fluoreszierenden Bereich der "Oval" Werkzeug in der ImageJ Bedienfeld.
- Um sicherzustellen, dass die Größe des Hintergrund oval das gleiche für alle analysierten Bilder ist, fügen Sie es dem ROI-Manager über das [t] Zeichen der Tastatur. Ein ROI-Manager kommt, wo kann der Hintergrund oval ROI über "Mehr" gespeichert werden - "Speichern" Optionen.
- Wenn der Hintergrund oval ROI auf dem Bild zu sehen ist, messen die Intensität des Bereichs, indem Sie "Analysieren" - "Messen".
HINWEIS: Eine ErgebnisseFenster erscheint unter anderem die mittlere Fluoreszenzintensität, wo in der Spalte mit der Bezeichnung "Mean" angezeigt wird. - Ziehen Sie den Wert der mittleren Hintergrund-Fluoreszenzintensität von dem Bild, das durch den Hintergrund oval ROI Haken entfernen, klicken Sie auf "Prozess" - "Math" - "subtrahieren" und den Messwert eingefügt wird.
- Tragen Sie eine Schwelle auf das Bild, über das "Bild" - "Anpassen" - "Threshold" Option. Wählen Sie die Methode Otsu und Schwarzweiß (B & W). Überprüfen Sie die "Dunkler Hintergrund" Option und verwenden, um die Schwelle, indem Sie auf "Übernehmen".
HINWEIS: Die Bildänderungen in ein binäres Bild, in dem der Bereich oberhalb der Schwelle in weiß dargestellt wird, und dass unterhalb der Schwelle in schwarz dargestellt ist. - Wählen Sie alles über der Schwelle über die "Analyse" - "Analyze Particles" Option. Im Fenster mit den Einstellungen, halten Sie die Standardoptionen und halten Sie die "Ergebnisse anzeigen" und "Summarize "geprüft Optionen. Klicken Sie auf" OK ", um die Zusammenfassung im Ergebnisfenster angezeigt und die Anzeige der belegten Fläche in der Spalte mit der Bezeichnung" Total Area ".
Ergebnisse
Laborsysteme der Bakterienpopulationen bieten einen ansprechenden Ansatz ökologischen oder evolutionären Fragen zu erforschen. Hier sind drei Oberflächenbesiedlung Weisen B. subtilis wurden verwendet , um das Aussehen der Bevölkerung Sortiment, dh die Trennung von genetisch identisch zu untersuchen, aber fluoreszenz verschiedenen markierten Stämme. Das Schwärmen, die eine Geissel abhängige kollektive Oberflächenbewegung von B. ist subtilis, Ergebnisse in einer stark gemischten Population. In diesen Schwärmen Kolonien wurden überlappende die grün- und rot-fluoreszierende Bakterien besiedelt Bereiche (siehe Abbildung 2A). Die schnelle Oberflächenbesiedlung kann in der Zeit (Video 1) verfolgt werden. Während Schwärmen von B subtilis, dehnt sich eine dünne Schicht von Zellen , die aus der Inokulation Zentrum nach einigen Stunden Inkubation (siehe 2B).
4752 / 54752fig2.jpg "/>
Abbildung 2: Schwärmende Expansion von B. . subtilis Die Schwärmen Kolonie enthält grün- und rot-fluoreszierende Stämme , die 1: 1 gemischt wurden vor der Impfung. (A) Nach 15 h, die grün- und rot-Fluoreszenz (GFP und RFP, jeweils) wurden mit geeigneten Fluoreszenzfiltern nachgewiesen. (B) Bilder von dünnen Schicht von Schwärmen B. subtilis werden zu ausgewählten Zeitpunkten extrahiert aus Video Abbildung 1. Maßstabsbalken = 5 mm. Bitte hier klicken , um eine größere Version dieser Figur zu sehen.
Wenn jedoch B. subtilis - Stämme, die funktionelle Flagellen fehlen aber in der Lage , mit Hilfe der erzeugten Exopolysaccharids, Hydrophobin und Surfactin zu verteilen, auf halbfestem Agarmedium getüpfelt wurden, die unterschiedlich markierte Stämme wurden in bestimmten def abgetrenntined Sektoren (siehe 3A). Die Entwicklung der Schiebe Kolonie können in der Zeit (siehe Abbildung 3B oder Video 2) aufgezeichnet werden.

Abbildung 3: Sliding Kolonie von B. . subtilis Die Kolonie enthält grün- und rot-fluoreszierende Stämme , die 1: 1 gemischt wurden vor der Inokulation. (A) Nach 24 Stunden der grün- und rot-Fluoreszenz (GFP und RFP, jeweils) wurden mit geeigneten Fluoreszenzfiltern nachgewiesen. (B) Bilder von der B. subtilis Gleitscheibe werden zu ausgewählten Zeitpunkten extrahiert aus Video Abbildung 2. Maßstabsbalken = 5 mm. Bitte hier klicken , um eine größere Version dieser Figur zu sehen.
Während die Auswahl Ebenen Schwärmen und sliding wachsenden Kolonien konnte nicht geändert werden, werden die räumliche Trennung von unterschiedlich markierten fluoreszierenden Stämme in der Kolonie Biofilm konnte durch die Ausgangszelldichten beeinflusst. Wenn eine Kolonie Biofilm von B. subtilis wurde mit hoher Zelldichte der Mischpopulationen initiiert, die grün- und rot-fluoreszierende Stämme zeigten geringe oder gar keine räumliche Auswahl (siehe Abbildung 4). Im Gegenteil, wenn die Zelldichte der biolfilm niedrig war, klare grün- und rot-Fluoreszenz Sektoren zu initiieren könnte durch Fluoreszenzmikroskopie nachgewiesen werden. Das Sortiment Ebene war eindeutig abhängig von der Verdünnungsgrad des Biofilms Initiieren Bevölkerung. Video 3 und 4 zeigen die Kolonie Erweiterung für die niedrigste und höchste Verdünnung der geimpften Stämme.

Abbildung 4: Sortiment Ebene in Kolonie Biofilme von B. subtilis bei. verschiedene Anfangszelldichten Die Kolonie Biofilme von grün- und rot-Fluoreszenz - Stämme werden nach 2 Tagen gezeigt , die mit unterschiedlichen anfänglichen Zelldichten geimpft wurden (von oben nach unten: nicht verdünnt auf 10 5 - fach verdünnt Kulturen initiiert, beziehungsweise). Maßstabsbalken = 5 mm. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
Das Verhältnis von grün- und rot-fluoreszierende Stämme kann weiter unter Verwendung von Software ImageJ quantifiziert werden, welche die quantitative Charakterisierung der Populationsstruktur und competiveness der Stämme für die Experimente verwendet werden können.
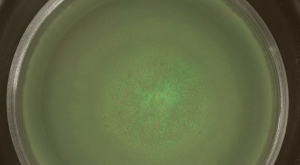
Video 1: Zeitraffer images von Schwärmen B. subtilis initiiert mit . 1: 1 - Mischung aus grün und rot fluoreszierenden Stämme (Rechtsklick zum Download bereit .) Das Video zeigt einen Zeitverlauf von 10 Stunden. Maßstabsbalken = 7 mm.

Video 2: Zeitraffer Bilder von B. Schiebe subtilis initiiert mit . 1: 1 - Mischung aus grün und rot fluoreszierenden Stämme (Rechtsklick zum Download bereit .) Das Video zeigt einen zeitlichen Verlauf von 24 Stunden. Maßstabsbalken = 5 mm.
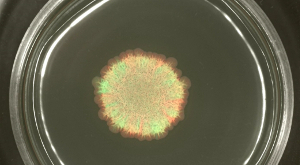
Video 3: Zeitraffer Bilder von B. subtilis Kolonie Biofilmen initiiert mit 1: 1 - Mischung aus grün-. Und rot-fluoreszierende Stämme bei hohen Zelldichten (Rechtsklick zum Download bereit .) Das Video zeigt einen Zeitverlauf von 48 Stunden. Maßstabsbalken = 5 mm.
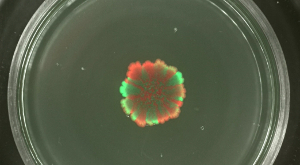
Video Abbildung 4: Zeitraffer Bilder von B. 1 - Mischung aus grün und rot fluoreszierenden Stämme bei niedrigen Zelldichten: Biofilme subtilis Kolonie mit 1 eingeleitet. (Rechtsklick zum Download bereit .) Das Video zeigt einen Zeitverlauf von 48 Stunden. Maßstabsbalken = 5 mm.
Diskussion
Die Verfügbarkeit einer fluoreszierenden Toolbox für Bakterien erleichtert nicht nur die Untersuchung der heterogenen Genexpressions 30,31 und Proteinlokalisation 32, sondern auch die Analyse der räumlichen Verteilung von Stämmen innerhalb einer Population 8. Fluoreszenzmarker mit ausreichend unterschiedlichen Anregungs- und Emissionswellenlängen erlauben, deutlich zwei Stämme zu lokalisieren, die sonst nicht zu unterscheiden sind voneinander, wenn gemischt. Das beschriebene Protokoll kann zur Beobachtung der Populationsdynamik in Mischkulturen eingesetzt werden, beispielsweise Kompetitionsexperimente oder Synergismus zwischen Stämmen oder Spezies. Die Fähigkeit, die relativen Häufigkeiten von fluoreszierend markierten Stämme in einer gemischten Population zu bestimmen, beschränkt sich nicht angebracht Schwärmen an die Oberfläche, Schiebe-, oder Biofilm Kolonien, sondern kann auch für andere mehrzellige Biofilmsystemen verwendet werden, unter Wasser, einschließlich Durchflusszelle oder Luft-Medium-Schnittstelle Biofilme 27,33-35.
_content "> Während die präsentierte Technik ein mächtiges Werkzeug ist die räumliche Verteilung der Belastungen und Designwettbewerb Experimente zu erkennen, sondern ermöglicht auch folgende Genexpression Heterogenität in den Ausbau der Kolonien. Die Kultivierungs hier beschriebenen Bedingungen gelten für B. subtilis und die genauen Parameter für die Expansion auf Agar - Medien könnten 20 Optimierung für andere Tierarten oder Stämme erfordern. die Anordnung der Proben in einer Inkubationskabine während Bildgebung der Experimentator ermöglicht die Populationsdynamik in der Zeit zu folgen, auch wenn die Aufmerksamkeit auf die Feuchtigkeit in der Kammer während der Inkubation gegeben werden sollte.Die hier beschriebenen Techniken erfordern auch die genetische Modifikation der untersuchten Bakterienstämme, so daß die Stämme Fluoreszenzmarker exprimieren, die voneinander unterschieden werden können. Darüber hinaus können neben mit getrennten Anregungs- und Emissionsspektren, wird empfohlen, dass die beiden gewählten Fluoreszenzmarker ähnlich Quant habenum Ausbeuten (dh Verhältnis der absorbierten Photonen, die emittiert werden) und werden in einem vergleichbaren Niveau zum Ausdruck gebracht. Darüber hinaus können relative Intensitätsänderungen in der Zeit zu einem frühen Zeitpunkt, eines Experiments gemessen und normalisiert werden. Die relative Zunahme oder Abnahme kann dann zwischen verschiedenen Fluorophore mit unterschiedlichen Quanteneffizienzen verglichen werden. Für die präsentierten experimentellen System unterschiedliche grün- und rot-fluoreszierende Proteine wurden zuvor 36,37 zu wählen , für den optimalen Fluoreszenzpaare getestet , die in B. nachgewiesen werden können subtilis. Die optimale Belichtungszeit sollte für jeden fluoreszierenden Protein und der Probe bestimmt werden. Bestimmte Zelldichten oder mehrere Schichten von Zellen könnte erforderlich sein, um das Signal effizient innerhalb der Population zu detektieren. Bestimmte fluoreszierende Proteine könnten niedrigen Intensitäten in den Bakterienzellen haben aufgrund ineffizienter Expression und / oder Translation des Proteins und somit eine geringe Quantenausbeute. Solche ineffizienten Fluoreszenzmarkern konnte rdie Empfindlichkeit des Systems educe und die Zeit verlängern möglicherweise die Bakterienstämme in Zytotoxizität durch die Lichtanregung erforderlich zu erfassen führt. Die Fluoreszenzintensitäten, die durch Veränderung des Promotors entsprechend modifiziert werden kann, verwendet, um die fluoreszierenden Reporter codierende Gen zu exprimieren. Ein Expressionspegel, der zu hoch ist in unnötiger Überproduktion des fluoreszierenden Proteins führt zu schädlichen Fitnesskosten für das Bakterium führen könnte. Wenn der Wettbewerb die Durchführung von Experimenten, sollte man die Kosten für bestimmte fluoreszierende Protein in den Zellen in Betracht ziehen. Kontrollexperimente, in denen die Fluoreszenzmarker zwischen konkurrierten Stämmen oder wo zwei isogene Stämme unterscheiden sich nur in ihren Fluoreszenzmarker sind gegeneinander an getauscht werden, sind immer erforderlich, um eine Tendenz zu einem Marker zu bestimmen. Die Lebensdauern der fluoreszierenden Proteine in den Zellen kann auch die gemessene Intensität beeinflussen. Darüber hinaus ist die Autofluoreszenz von bestimmten bakteriellen Speziess könnte die Verwendung von verschiedenen Fluoreszenzmarkern anders als hier beschrieben sind, erfordern.
Genau die räumlichen Verteilungen und Abundanzen der verschiedenen Bakterienstämmen bestimmen, das Hintergrundsignal von dem ersten fluoreszierenden Proteins mit Ursprung während der Fluoreszenzfilter für die zweite Fluoreszenzmarkierung und umgekehrt unter Verwendung sollte individuell auf Monokulturen Proben getestet werden (enthaltend produzierenden Bakterien nur eine Markierung ). Dies ermöglicht die Subtraktion der überlappenden Fluoreszenzsignalintensitäten. Wichtig ist, wie die Stereomikroskopes das Fluoreszenzsignal von oberhalb der wachsenden Kolonie aufzeichnet, ist die dargestellte Protokoll bequem, die räumliche Anordnung in zwei Dimensionen zu bestimmen. Die Architektur der wachsenden Bakterienpopulation könnte in unterschiedlichen Fluoreszenzwerte führen (dh Falten artige Strukturen könnten mehr Zellen enthalten höhere lokale Fluoreszenzintensitäten anzeigt). Daher d die beschriebene Auswertung der Bilderetermines die räumliche Verteilung, aber nicht die Häufigkeit der Stämme innerhalb einer bestimmten Lage. Vorherige Protokolle beschrieben , die Probenvorbereitung für Schwärmen 20 oder Fluoreszenz - Bildgebung der Populationsdynamik in Bakterienkolonien 38, aber unser Protokoll kombiniert diese Techniken. Andere Mikroskopietechniken, die die Beobachtung von dreidimensionalen Auflösung der Bevölkerungsstruktur ermöglichen (zB konfokalen Laser - Scanning - Mikroskopie 39,40 oder strukturierte Beleuchtung Mikroskopie 41) für Proben mit erhöhten strukturellen Komplexität angewendet werden. Diese zusätzlichen Techniken unterstützen auch einzelne Zelle basierte Erkennung der Stämme 31 , die nicht mit Stereomikroskopen ist.
Offenlegungen
Die Autoren erklären, dass sie keine finanziellen Interessen haben.
Danksagungen
Diese Arbeit wurde durch Zuschuss finanziert KO4741 / 3-1 von der Deutschen Forschungsgemeinschaft (DFG). Ferner ist das Labor von Á.TK wurde durch ein Marie-Curie-Sklodowska Karriere Eingliederungszuschuss (PheHetBacBiofilm) und gewähren KO4741 / 2-1 von DFG unterstützt. TH, AD, RG-M., Und EM wurden von der International Max Planck Research School, Alexander von Humboldt-Stiftung, Consejo Nacional de Tecnologia y-Ciencia Deutschen Akademischen Austauschdienst (CONACyT-DAAD) und JSMC Stipendien, jeweils unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Lennox Broth (LB) | Carl Roth GmbH | X964 | |
| Agar-agar, Kobe I | Carl Roth GmbH | 5210 | |
| Petri dish (90 mm diameter) | any | NA | Use Petri dishes without ventillation cams |
| Petri dish (35 mm diameter) | any | NA | Use Petri dishes without ventillation cams |
| Difco Nutrient Broth | BD Europe | 234000 | |
| KCl | any | NA | |
| MgSO,4 7H2O | any | NA | |
| Ca(NO3)2 4H2O | any | NA | |
| MnCl24H2O | any | NA | |
| FeSO4 | any | NA | |
| D-Glucose | any | NA | |
| Fluorescence AxioZoom V16 time-lapse microscope | Carl Zeiss Microscopy GmbH | see bellow detailed description | |
| AxioZoom V16 Microscope body | Carl Zeiss Microscopy GmbH | 435080 9030 000 | |
| Phototube Z 100:0 for Axio Zoom V16 | Carl Zeiss Microscopy GmbH | 435180 9020 000 | without eyepieces |
| Fluar Illuminator Z mot Fluorescence intermediate tube for Axio Zoom.V16 | Carl Zeiss Microscopy GmbH | 435180 9060 000 | |
| Controller EMS 3 | Carl Zeiss Microscopy GmbH | 435610 9010 000 | |
| System Control Panel SYCOP 3 | Carl Zeiss Microscopy GmbH | 435611 9010 000 | |
| Reflector module Z | Carl Zeiss Microscopy GmbH | 435180 9160 000 | For Fluar Illuminator Z mot on Axio Zoom.V16 and SYCOP 3 |
| Filter set 38 HE eGFP shift free (E) | Carl Zeiss Microscopy GmbH | 489038 9901 000 | EX BP 470/40, BS FT 495, EM BP 525/50 |
| Filter set 63 HE mRFP shift free (E) | Carl Zeiss Microscopy GmbH | 489063 0000 000 | EX BP 572/25, BS FT 590, EM BP 629/62 |
| Mount S | Carl Zeiss Microscopy GmbH | 435402 0000 000 | |
| Objektive PlanApo Z 0,5x/0,125 FWD 114 mm | Carl Zeiss Microscopy GmbH | 435280 9050 000 | 164 mm parfocal length; M62x0.75 thread at front |
| Coarse/fine drive with profile column | Carl Zeiss Microscopy GmbH | 435400 0000 000 | 490 mm, 10 kg load capacity, compatible with stand bases 300/450 |
| Stand base 450 | Carl Zeiss Microscopy GmbH | 435430 9902 000 | |
| Cold-light source Zeiss CL 9000 LED CAN | Carl Zeiss Microscopy GmbH | 435700 9000 000 | |
| CAN-bus cable | Carl Zeiss Microscopy GmbH | 457411 9011 000 | 2.5 m length |
| Slit-ring illuminator | Carl Zeiss Microscopy GmbH | 417075 9010 000 | d = 66 mm |
| Flexible light guide 1500 | Carl Zeiss Microscopy GmbH | 417063 9901 000 | 8/1,000 mm |
| Illumination Adapter for light guide | Carl Zeiss Microscopy GmbH | 000000 1370 927 | |
| Lightguide HXP with liquid fill | Carl Zeiss Microscopy GmbH | 000000 0482 760 | ø3 mm x 2,000 mm |
| Camera Adapter 60N-C | Carl Zeiss Microscopy GmbH | 426113 0000 000 | 2/3" 0.63X |
| High Resolution Microscopy Camera AxioCam MRm Rev. 3 FireWire | Carl Zeiss Microscopy GmbH | 426509 9901 000 | |
| AxioCam FireWire Trigger Cable Set | Carl Zeiss Microscopy GmbH | 426506 0002 000 | for direct shutter synchronization |
| ZEN pro 2012 | Carl Zeiss Microscopy GmbH | 410135 1002 120 | Blue edition, requires min. Windows 7 64-bit |
| ZEN Module Time Lapse | Carl Zeiss Microscopy GmbH | 410136 1031 110 | |
| Standard Heating Stage Top Incubator | Tokai Hit | INUL-MS1-F1 | |
| Zeiss Stereo Microscope Base Adapter | Tokai Hit | MS-V12 | |
| Softwares | |||
| ImageJ | National Institute of Health, Bethesda, MD, USA | v 1.49m | |
| BioVoxxel plugin | BioVoxxel | http://www.biovoxxel.de/development/ |
Referenzen
- Nadell, C. D., Xavier, J. B., Foster, K. R. The sociobiology of biofilms. FEMS Microbiol Rev. 33 (1), 206-224 (2009).
- West, S. A., Griffin, A. S., Gardner, A., Diggle, S. P. Social evolution theory for microorganisms. Nat Rev Microbiol. 4 (8), 597-607 (2006).
- Kovács, &. #. 1. 9. 3. ;. T. Impact of spatial distribution on the development of mutualism in microbes. Front Microbiol. 5, 649 (2014).
- Martin, M., Hölscher, T., Dragoš, A., Cooper, V. S., Kovács, &. #. 1. 9. 3. ;. T. Laboratory evolution of microbial interactions in bacterial biofilms. J Bacteriol. , (2016).
- Drescher, K., Nadell, C. D., Stone, H. A., Wingreen, N. S., Bassler, B. L. Solutions to the public goods dilemma in bacterial biofilms. Curr Biol. 24 (1), 50-55 (2014).
- Momeni, B., Waite, A. J., Shou, W. Spatial self-organization favors heterotypic cooperation over cheating. Elife. 2, e00960 (2013).
- van Dyken, J. D., Müller, M. J., Mack, K. M., Desai, M. M. Spatial population expansion promotes the evolution of cooperation in an experimental Prisoner's Dilemma. Curr Biol. 23 (10), 919-923 (2013).
- van Gestel, J., Weissing, F. J., Kuipers, O. P., Kovács, &. #. 1. 9. 3. ;. T. Density of founder cells affects spatial pattern formation and cooperation in Bacillus subtilis biofilms. ISME J. 8 (10), 2069-2079 (2014).
- Momeni, B., Brileya, K. A., Fields, M. W., Shou, W. Strong inter-population cooperation leads to partner intermixing in microbial communities. Elife. 2, e00230 (2013).
- Müller, M. J., Neugeboren, B. I., Nelson, D. R., Murray, A. W. Genetic drift opposes mutualism during spatial population expansion. Proc Natl Acad Sci U S A. 111 (3), 1037-1042 (2014).
- Pande, S., et al. Privatization of cooperative benefits stabilizes mutualistic cross-feeding interactions in spatially structured environments. ISME J. 10, 1413-1423 (2016).
- Hallatschek, O., Hersen, P., Ramanathan, S., Nelson, D. R. Genetic drift at expanding frontiers promotes gene segregation. Proc Natl Acad Sci U S A. 104 (50), 19926-19930 (2007).
- Flemming, H. C., Wingender, J. The biofilm matrix. Nat Rev Microbiol. 8 (9), 623-633 (2010).
- Vlamakis, H., Chai, Y., Beauregard, P., Losick, R., Kolter, R. Sticking together: building a biofilm the Bacillus subtilis way. Nat Rev Microbiol. 11 (3), 157-168 (2013).
- Lopez, D., Kolter, R. Extracellular signals that define distinct and coexisting cell fates in Bacillus subtilis. FEMS Microbiol Rev. 34 (2), 134-149 (2010).
- Lopez, D., Vlamakis, H., Kolter, R. Generation of multiple cell types in Bacillus subtilis. FEMS Microbiol Rev. 33 (1), 152-163 (2009).
- Mhatre, E., Monterrosa, R. G., Kovács, &. #. 1. 9. 3. ;. T. From environmental signals to regulators: Modulation of biofilm development in Gram-positive bacteria. J Basic Microbiol. , (2014).
- Zhang, W., et al. Nutrient depletion in Bacillus subtilis biofilms triggers matrix production. New J Physics. 16, 015028 (2014).
- Kearns, D. B. A field guide to bacterial swarming motility. Nat Rev Microbiol. 8 (9), 634-644 (2010).
- Morales-Soto, N., et al. Preparation, imaging, and quantification of bacterial surface motility assays. J Vis Exp. (98), (2015).
- Angelini, T. E., Roper, M., Kolter, R., Weitz, D. A., Brenner, M. P. Bacillus subtilis spreads by surfing on waves of surfactant. Proc Natl Acad Sci U S A. 106 (43), 18109-18113 (2009).
- Grau, R. R., et al. A Duo of Potassium-Responsive Histidine Kinases Govern the Multicellular Destiny of Bacillus subtilis. MBio. 6 (4), e00581 (2015).
- Park, S. Y., Pontes, M. H., Groisman, E. A. Flagella-independent surface motility in Salmonella enterica serovar Typhimurium. Proc Natl Acad Sci U S A. 112 (6), 1850-1855 (2015).
- van Gestel, J., Vlamakis, H., Kolter, R. From cell differentiation to cell collectives: Bacillus subtilis uses division of labor to migrate. PLoS Biol. 13 (4), e1002141 (2015).
- Abee, T., Kovács, &. #. 1. 9. 3. ;. T., Kuipers, O. P., van der Veen, S. Biofilm formation and dispersal in Gram-positive bacteria. Curr Opin Biotechnol. 22 (2), 172-179 (2011).
- Kovács, &. #. 1. 9. 3. ;. T. Bacterial differentiation via gradual activation of global regulators. Curr Genet. 62 (1), 125-128 (2016).
- Hölscher, T., et al. Motility, chemotaxis and aerotaxis contribute to competitiveness during bacterial pellicle biofilm development. J Mol Biol. 427 (23), 3695-3708 (2015).
- Seminara, A., et al. Osmotic spreading of Bacillus subtilis biofilms driven by an extracellular matrix. Proc Natl Acad Sci U S A. 109 (4), 1116-1121 (2012).
- Patrick, J. E., Kearns, D. B. Laboratory strains of Bacillus subtilis do not exhibit swarming motility. J Bacteriol. 191 (22), 7129-7133 (2009).
- de Jong, I. G., Beilharz, K., Kuipers, O. P., Veening, J. W. Live Cell Imaging of Bacillus subtilis and Streptococcus pneumoniae using Automated Time-lapse Microscopy. J Vis Exp. (53), e3145 (2011).
- Garcia-Betancur, J. C., Yepes, A., Schneider, J., Lopez, D. Single-cell analysis of Bacillus subtilis biofilms using fluorescence microscopy and flow cytometry. J Vis Exp. (60), (2012).
- Turnbull, L., et al. Super-resolution imaging of the cytokinetic Z ring in live bacteria using fast 3D-structured illumination microscopy (f3D-SIM). J Vis Exp. (91), e51469 (2014).
- Barken, K. B., et al. Roles of type IV pili, flagellum-mediated motility and extracellular DNA in the formation of mature multicellular structures in Pseudomonas aeruginosa biofilms. Environ Microbiol. 10 (9), 2331-2343 (2008).
- Oliveira, N. M., et al. Biofilm formation as a response to ecological competition. PLoS Biol. 13 (7), e1002191 (2015).
- Wang, X., et al. Probing phenotypic growth in expanding Bacillus subtilis biofilms. Appl Microbiol Biotechnol. 100, 4607-4615 (2016).
- Detert Oude Weme, R. G., et al. Single cell FRET analysis for the identification of optimal FRET-pairs in Bacillus subtilis using a prototype MEM-FLIM system. PLoS One. 10 (4), e0123239 (2015).
- Overkamp, W., et al. Benchmarking various green fluorescent protein variants in Bacillus subtilis, Streptococcus pneumoniae, and Lactococcus lactis for live cell imaging. Appl Environ Microbiol. 79 (20), 6481-6490 (2013).
- Stannek, L., Egelkamp, R., Gunka, K., Commichau, F. M. Monitoring intraspecies competition in a bacterial cell population by cocultivation of fluorescently labelled strains. J Vis Exp. (83), e51196 (2014).
- Bridier, A., Briandet, R. Contribution of confocal laser scanning microscopy in deciphering biofilm tridimensional structure and reactivity. Methods Mol Biol. 1147, 255-266 (2014).
- Khajotia, S. S., Smart, K. H., Pilula, M., Thompson, D. M. Concurrent quantification of cellular and extracellular components of biofilms. J Vis Exp. (82), e50639 (2013).
- Neu, T. R., Lawrence, J. R. Innovative techniques, sensors, and approaches for imaging biofilms at different scales. Trends Microbiol. 23 (4), 233-242 (2015).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten