Method Article
Zeitliche Reihenfolge der dynamischen Ausdruck von Daten aus Detaillierte Spatial Expression Maps
In diesem Artikel
Zusammenfassung
The segmentation clock drives oscillatory gene expression across the pre-somitic mesoderm (PSM). Dynamic Notch activity is key to this process. We use imaging and computational analyses to extract temporal dynamics from spatial expression data to demonstrate that Delta ligand and Notch receptor expression oscillate in the vertebrate PSM.
Zusammenfassung
During somitogenesis, pairs of epithelial somites form in a progressive manner, budding off from the anterior end of the pre-somitic mesoderm (PSM) with a strict species-specific periodicity. The periodicity of the process is regulated by a molecular oscillator, known as the "segmentation clock," acting in the PSM cells. This clock drives the oscillatory patterns of gene expression across the PSM in a posterior-anterior direction. These so-called clock genes are key components of three signaling pathways: Wnt, Notch, and fibroblast growth factor (FGF). In addition, Notch signaling is essential for synchronizing intracellular oscillations in neighboring cells. We recently gained insight into how this may be mechanistically regulated. Upon ligand activation, the Notch receptor is cleaved, releasing the intracellular domain (NICD), which moves to the nucleus and regulates gene expression. NICD is highly labile, and its phosphorylation-dependent turnover acts to restrict Notch signaling. The profile of NICD production (and degradation) in the PSM is known to be oscillatory and to resemble that of a clock gene. We recently reported that both the Notch receptor and the Delta ligand, which mediate intercellular coupling, themselves exhibit dynamic expression at both the mRNA and protein levels. In this article, we describe the sensitive detection methods and detailed image analysis tools that we used, in combination with the computational modeling that we designed, to extract and overlay expression data from distinct points in the expression cycle. This allowed us to construct a spatio-temporal picture of the dynamic expression profile for the receptor, the ligand, and the Notch target clock genes throughout an oscillation cycle. Here, we describe the protocols used to generate and culture the PSM explants, as well as the procedure to stain for the mRNA or protein. We also explain how the confocal images were subsequently analyzed and temporally ordered computationally to generate ordered sequences of clock expression snapshots, hereafter defined as "kymographs," for the visualization of the spatiotemporal expression of Delta-like1 (Dll1) and Notch1 throughout the PSM.
Einleitung
Somiten sind die ersten in den Verlängerungskörperachse gebildet Segmente in Vertebratenspezies entwickelt und sind die Vorläufer der Wirbelsäule, Rippen und Dermis Gewebe sowie Muskel- und Endothelzellen. Während Somitogenese bilden epithelialen Somiten vom unsegmented präsomitischen Mesoderm (PSM) (beschrieben in Referenz 1). Dieser Prozess wird durch die "Segmentation Uhr" geregelt, die aus einem Netzwerk von oszillatorischen Genen und Proteinen besteht, meist an den Notch-Signalweg gehören. Die Segmentierung Uhr besteht aus verschiedenen negativen Rückkopplungsschleifen, die die pulsatile Produktion von Notch - Aktivität innerhalb einer einzelnen Zelle 2 ( beschrieben in Referenzen 3 - 6) , ermöglichen. Während die intrazelluläre Methode Schwingungs gut charakterisiert ist, ist es immer noch weitgehend unbekannt, wie diese Schwingungen über das PSM Gewebe abgestimmt sind. Es wurde kürzlich gezeigt, durch beide experimentelle und theoretische Untersuchungen, dass diese Schwingungen essentia sindl für den Prozess der Somitogenese und dass der Notch - Signalweg spielt eine wichtige Rolle im Prozess der Segmentierung und sowohl oszillierende Genexpression 7, 8. Jedoch wurde es allgemein , dass Notch - Rezeptor 1 (Notch1) berichteten , und Delta-like - Ligand (Dll) -1 haben statische Gradienten in der PSM 9, 10, 11.
Wir vermuten, dass Notch abhängigen Schwingungen der PSM Segmentierung Uhr hängen von der periodischen Aktivierung des Haupt Notch-Signalweg Rezeptor und Ligand, Notch1 und DLL1 jeweils über die Maus PSM. Die Schlussfolgerungen aus früheren Studien, die eine statische rostral-kaudale Gradienten dieser Proteine berichtet wurden, waren wegen, wir vorhersagen, zu einem Mangel an Sensibilität in Immunfärbungstechniken. Sie waren daher nicht in der Lage mit niedrigem Pegelschwankungen von DLL1 und Notch1 im kaudalen PSM zu erkennen.
Wir have entwickelte ein Verfahren , um mehr genau prüfen , diese Faktoren, experimentelle Daten mit mathematischen Modellen kombiniert , mit dem ein Mechanismus zur Vorhersage der Schwingungen der Proteine von Taktkomponenten über die PSM 12 koordiniert werden.
Das übergeordnete Ziel dieser Methode ist , Low-Level, dynamische Protein - Expression im PSM nachzuweisen und zu quantifizieren und die Expressionsprofile von Proteinen von Interesse zur Karte nach dem Ausdruck des bekannten Uhr - Gen, lunatic fringe (Lfng). Da ein Zyklus des Segmentierungs Uhr im Mausembryo 2 h dauert, werden verschiedene Proben erforderlich , um eine vollständige Raum - Zeit - Profil der DLL1 und Notch1 Protein - Expression während einer Lfng Oszillation in der PSM zu bauen. Damit haben wir dieses Protokoll zu ermöglichen, für die Hochdurchsatz-Nachweis von Low-Level-Proteinexpression in ganz-mount, kontralateralen PSM Explantate entwickelt. Allerdings kann diese Technik auch nützlich sein für Studien tHut Ziel Low-Level-Proteindynamik innerhalb eines embryonalem Gewebe zu charakterisieren, die in kontralateralen Hälften geteilt werden kann.
Protokoll
Alle Experimente wurden unter Projektlizenznummer 6004219 in strikter Einhaltung der Tiere (Scientific Procedures) Act von 1986 und der britischen Home Office Codes of Practice für den Einsatz von Tieren in wissenschaftlichen Verfahren durchgeführt.
1. PSM Explantation Dissection
- Besorgen Sie sich die Schwanzgewebe von Embryonen , die durch die zeitlich Paarung von Wildtyp (CD1) Mäuse 13 hergestellt. Kurz gesagt, bei embryonalen Tag (E) 10.5, euthanize der schwangeren Spendermaus in einer Kohlendioxidkammer. Ernten Sie die Uterushorn und legen Sie sie in 1x sterile phosphatgepufferte Salzlösung (PBS) Lösung in Übereinstimmung mit Home Office License Verfahren oder einem gleichwertigen Regeln. Übertragen Sie das Uterushorn zu einer Gewebekulturschale mit frischen, sterilen PBS. Führen Sie alle nachfolgenden Dissektion Schritte in dieser Lösung.
- Unter einem Stereomikroskop, schneiden Sie die dicke Muskelmembran der Uterushorn gebogenen Schere und jeder Embryo extrahieren feinen Pinzette vorsichtig mit. Pass aufum sicherzustellen, dass die Schwanzgewebe bei diesem Vorgang nicht beschädigt. Mit gebogenen Schere und einer feinen Pinzette, sezieren die Fruchtblase von jedem Embryo entfernt, dabei nicht den Embryo nicht zu beschädigen.
- Verwenden Sie entweder eine chirurgische Nadel oder gebogene Schere den Schwanz Gewebe von jedem Embryo bis zur Ernte durch den Embryo hinter den hinteren Extremitätenknospen zu schneiden.
- Balance der Schwanz Gewebe ventralen Seite nach unten beide Pinzette und eine Nadel. Generieren Paare von PSM Explantate von jedem embryonale Schwanz durch den Schwanz Gewebe in zwei Hälften entlang der Mittellinie seziert; eine sanfte Schaukelbewegung mit einer Nadel durchzuführen. Stellen Sie sicher, dass sich das Neuralrohr, Chorda und PSM Gewebe gleichmäßig zwischen den beiden Explantate unterteilt sind.
- Pipette jeweils kontralateralen PSM Explantat auf die Unterseite eines 35-mm-Kunststoff-Kulturschale Deckel in einem kleinen Volumen von vorgewärmten (37 ° C) Kulturmedium (DMEM-F12 + 0,1% L-Glutamin ersetzt mit 10% fötalem Kälberserum , 10 nM Human-bFGF und 1% Penicillin / Streptomycin).
- Die Schale auf der Oberseite des Deckels und schnell umkehren, so dass das PSM Gewebe aus dem Deckel in einer "hängenden Tropfen" Medium suspendiert ist. Kultur die PSM-Explantaten in einer feuchten Kammer bei 37 ° C für 1 bis 2 h.
- Transfer Paare von PSM Explantate auf die einzelnen Vertiefungen einer 24-Well-Gewebekulturplatte. Inkubieren in 4% Paraformaldehyd in PBS für 1 h bei Raumtemperatur (RT) oder bei 4 ° C über Nacht (O / N). ACHTUNG: Paraformaldehyd ist giftig, und entsprechende Sicherheitsvorkehrungen getroffen werden müssen , wenn sie mit dieser Lösung arbeiten.
ANMERKUNG: Führen Sie alle nachfolgenden Wasch- und Inkubationsschritte in einer 24-Well-Gewebekulturplatte. - Waschen Sie die Proben-Wells in PBS bei RT auf einem Schüttler, eine feine Kunststoff-Pasteur-Pipette mit der PBS-Lösung auf den Proben für frische PBS 3 auszutauschen - 4 mal. Verarbeiten eines PSM Explantat von jedem Paar mittels Immunhistochemie (Schritt 2) und die andere unter Verwendung von Fluoreszenz - in situ - Hybridisierung für eine bekannte Takt Gen (step 3).
2. Immunhistochemie von PSM Explantate
- Waschen Sie ein PSM Explantation von jedem in Schritt erzeugte embryonale Paar 1 in 2% Triton X-100 in PBS für 1 h bei RT auf einem Schüttler, und dann spülen Proben kurz in PBS. Ersetzen Sie den PBS auf die Proben mit Blocking-Lösung (2% Rinderserumalbumin (BSA) und 10% normalem Ziegenserum (NGS) in PBS + 0,1% Tween-20) und Inkubation O / N bei 4 ° C auf einem Schüttler.
Hinweis: Alle nachfolgenden Waschungen und Inkubation Schritte in diesem Abschnitt müssen bei RT auf einem Schüttler durchgeführt werden, sofern nicht anders angegeben. Waschlösungen können einfach über einen spitzen Kunststoff oder Glas Pasteur Pipette verändert. - Verdünne die gewünschten primären Antikörper / Antikörper in Arbeitspuffer (0,1% BSA, 0,3% NGS und 0,2% Triton X-100 in PBS). In diesem Beispiel wird die verdünnte DLL1 und Notch1 Antikörper 01.25 in Puffer arbeiten.
HINWEIS: Die Optimierung wird benötigt, um die entsprechenden Verdünnungsfaktor in thi erforderlich, um zu bestimmens Schritt, wenn alternative Antikörper verwendet werden. - Inkubieren Explantate in der Antikörperlösung für 3 - 5 Tage bei 4 ° C auf einem Schüttler. Achten Sie darauf, einige Proben mit Arbeitspuffer, der keine primären Antikörper enthalten als sekundärer Antikörper Kontrollen zu handeln.
- Wiederherstellen der primären Antikörperlösung in einem 1,5-ml-Speicherrohr mit einer Pipette, und bewahren Sie sie bei 4 ° C.
HINWEIS: Wiederhergestellte primäre Antikörper kann mehrmals verwendet werden, abhängig von der verwendeten Antikörper. - Führen 2 Waschungen der Proben 5 - 10 min jeweils in PBS, gefolgt von 3 Waschungen für 10 min jeweils in 2% Triton X-100 in PBS bei RT auf einer Schüttelplattform.
- Verdünnte fluoreszenzmarkierten sekundären Antikörper / Antikörper (Epitop angepaßten an den primären Antikörper / Antikörper verwendet) in Puffer arbeiten. Gegebenenfalls werden 20 & mgr; g / ml Hoechst 33342 zu dieser Lösung, um die Zellkerne zu Gegenfärbung.
HINWEIS: Die Optimierung kann in diesem Schritt erforderlich, um die entsprechenden Verdünnungsfaktor zu bestimmen, erforderlich. In dieser Example, einem Verdünnungsfaktor von 1: 400 wurde typischerweise verwendet. - Zentrifugieren der sekundären Antikörperlösung für 10 min bei 16 · g, die Bildung von Antikörper-Aggregate zu verhindern. In 250 bis 500 & mgr; l des sekundären Antikörperlösung gut zu jeder Probe, dabei nicht die letzten paar Mikroliter der Lösung zu verwenden, die Antikörper-Aggregate enthalten.
- Decken Sie die Probenplatte mit Alufolie Lichtexposition minimieren und die Proben in der sekundären Antikörperlösung Inkubation für 3 - 5 Tage bei 4 ° C im Dunkeln.
- Vor Montage zu probieren, waschen Sie die Proben zweimal für jeweils 10 Minuten in 0,1% Tween-20 in PBS (PBST) und einmal für 5 min in PBS bei RT auf einem Schüttler (siehe Schritt 4).
3. Fluoreszenz in situ Hybridisierung (FISH) von PSM Explantate
- Wenn in einer alternativen Behälter gespeichert, übertragen Sie die restlichen kontralateralen PSM Explantate zu den einzelnen Wells einer 24-Well-Gewebekulturplatte.
- Waschen Sie die Proben für10 min in 50% Ethanol in PBST und dann 2 Wäschen für jeweils 10 Minuten in 100% Ethanol auf einer Schüttelplattform bei RT durchzuführen um das Gewebe zu entwässern.
Hinweis: Alle nachfolgenden Waschungen und Inkubation Schritte in diesem Abschnitt müssen bei RT auf einem Schüttler durchgeführt werden, sofern nicht anders angegeben. - Rehydrieren das Gewebe durch Waschen für 10 min in 50% Ethanol in PBST, gefolgt von zweimaligem Waschen für jeweils 5 min in PBST.
HINWEIS: Die Schritte 3.2 und 3.3 sind notwendig Fixierungsschritte für dieses Protokoll erforderlich ist und nicht weggelassen werden kann. - Inkubieren der Proben mit 10 & mgr; g / ml Proteinase K in 0,1% Tween-20 in PBS (PBST) für 5 min ohne Rühren. Schnell die Proteinase K zu entfernen und die Proben kurz mit PBST vor post-Fixierung des Gewebes für 30 Minuten in 4% Formaldehyd + 0,1% Glutaraldehyd in PBST spülen. ACHTUNG: Sowohl Formaldehyd und Glutaraldehyd sind giftig, und geeignete Sicherheitsmaßnahmen zu treffen , wenn sie mit diesen Lösungen arbeiten.
HINWEIS: Die folgenden Waschund Inkubationsschritte denen 50% und 100% Hybridisierungsmischungen (Schritte 3,6-3,9) sollte ohne Rühren durchgeführt werden. - die Proben zweimal für jeweils 10 Minuten in PBST, waschen Sie die Proben einmal in 50% Hybridisierungsmischung (geeignet für Intron-Sonden Nach dem Waschen: 50% Formamid, 5 × mit Kochsalzlösung Natriumcitrat (SSC), 5 mM EDTA, 50 ug / ml tRNA, 0,2% Tween-20, 0,1% SDS und 100 ug / ml Heparin) in PBST bei RT hergestellt. Inkubieren der Proben in dieser Lösung für 10 min bei 65 ° C ohne Schütteln.
- Spülen Sie die Proben zweimal mit vorgewärmten (65 ° C) Hybridisierungsmischung bevor die Proben in Hybridisierungsmischung für ≥ 2 h inkubiert (bis zu 48 h) bei 65 ° C (längere Inkubationszeiten verbessern das resultierende Signal-zu-Rausch-Kontrast) . Entfernen Sie die Hybridisierungsmischung aus dem vorherigen Schritt, und ersetzen Sie es mit 0,25 - 0,5 mL vorgewärmtes (65 ° C) Hybridisierungsmischung, die eine Digoxigenin (DIG) -markierten Antisense-RNA-Sonde gegen einen bekannten Segmentierungstaktkomponente.
NICHTE: Zum Beispiel kann ein Intron Lunatic fringe (Lfng (i)) Sonde in einer Konzentration von 20 ul / ml verwendet wurde naszierenden Lfng mRNA zu detektieren. Die Verdünnung in diesem Schritt verwendete Sonde abhängig und Optimierung erfordern. - Verschließen Sie die Platte Klebeband mit Verdunstung zu verhindern und die Proben in der Sondenlösung für zwei Nächte bei 65 ° C inkubiert.
- Mit einem spitzen Kunststoff Pasteurpipette zu erholen, die Sonde für die Wiederverwendung und lagern Sie es bei 20 ° C. Spülen Sie die Proben zweimal mit vorgewärmten (65 ° C) nach der Hybridisierungsmischung (50% Formamid, 0,2% Tween-20 und 1 x SSC), bevor die Proben zweimal für jeweils 20 min bei 65 ° C in Vorwaschen erwärmt nach der Hybridisierung Mix.
- Waschen Sie die Proben für 15 min bei 65 ° C in vorgewärmten 50% Hybridisierungsmischung in 0,1% Tween-20 in Tris-gepufferter Kochsalzlösung (TBST). Spülen Sie die Proben zweimal mit TBST vor auf einer Schüttelplattform in TBST für 30 Minuten bei RT gewaschen.
- Pre-Inkubation der Explantate in blocking Lösung (TBST + 2% Blocking-Puffer-Reagenz (BBR) + 20% wärmebehandelt Ziegenserum) für mindestens 2 h. Ersetzen Sie diese Lösung mit frischen Blocking-Lösung die eine 1: 200-Verdünnung von Meerrettich-Peroxidase (HRP) -konjugierte anti-Digoxigenin-Antikörper. Inkubieren Sie die Proben O / N bei 4 ° C.
- Nach der Antikörper-Inkubation spülen Sie die Proben 3 mal mit TBST bei RT und übertragen sie in die einzelnen Vertiefungen einer neuen 24-Well-Gewebekulturplatte. Waschen der Explantate mit TBST 3 Mal für jeweils 1 h.
- Zu diesem Zeitpunkt übertragen die Proben in 0,5 ml-Vorratsröhrchen oder die einzelnen Vertiefungen einer 48-Well-Gewebekulturplatte das erforderliche Volumen der Tyramid Signalverstärkungs (TSA) Nachweisreagenzien in den folgenden Schritten zu reduzieren.
- Inkubieren Proben in TSA Verstärkung Puffer (die Reagenzien Liste) bei RT für 1 min ohne Rühren so klein ein Volumen wie möglich zu verwenden, um sicherzustellen, dass die Proben in der Lösung vollständig eingetaucht sind.
- In TSA-Reagenz (sieheReagenzien Liste) an die Probe Amplifikationspuffer in einer Verdünnung von 1:50. Schnell die Lösung mischen, bis die TSA Reagenz gleichmäßig verteilt wird, die Platte oder Rohre in Alufolie bedecken, und die Proben für 60 brüten - im Dunkeln 90 min.
- Entfernen Sie die Verstärkungslösung TSA und waschen Sie die Proben in TBST 3 mal für jeweils 5 Minuten. Transfer der Explantate zurück in eine 24-Well-Gewebekulturplatte, die die Waschvolumen und inkubiere die Proben in 1% Wasserstoffperoxid in TBST für 1 h zu erhöhen. Waschen der Proben mit TBST 3 Mal für jeweils 5 Minuten und dann zweimal für jeweils 5 min mit PBST vor Montage abzutasten (siehe Schritt 4).
4. Probenvorbereitung für die Imaging
- Bereiten eines geladenen Haftglasobjektträger für jedes Explantat Paar durch Zugabe von 0,12 mm dicken Abbildungs Spacern, die die Proben verhindert durch die Zugabe von einem Deckglas zerdrückt wird. Entfernen Sie die Klebefolie von einer Oberfläche eines Abstandshalter und legen Sie es Klebstoffseite nach unten auf einen Glasträger, Pressen firmly abzudichten den Abstandshalter an der Folie.
HINWEIS: Für die verbleibenden Schritte, bemüht sich, die Proben bei schwachem Licht oder in der Dunkelheit zu halten Photobleichens zu vermeiden. Pipette Explantat Paare auf eine vorbereitete Objektträger mit einer Glaspasteurpipette in der Mitte des Abstandshalters verwendet, sicherzustellen, dass die sezierten Seite des Explantat den Gleitflächen. Ordnen kontralateralen Paare von Explantaten nebeneinander. - Entfernen Sie so viel Flüssigkeit wie möglich aus der Folie mit einer Glaspasteurpipette und Docht aus Restfeuchtigkeit rund um die Proben ein Stück gefaltet fusselarme Tissue-Papier verwendet wird.
- Lassen Sie die Proben für die 45 an der Folie haften - 60 s, bis das Gewebe klebrig und durchscheinend zu erscheinen beginnt. Während dieser Zeit, entfernen Sie die verbleibenden Kleberbeschichtung aus dem Distanz einer Pinzette. Lassen Sie die Proben zum Trocknen.
- Fügen Sie einen großen Tropfen von Dual-Funktion mountant und Clearing-Lösung (0,5% p-Phenylendiamin und 20 mM Tris, pH 8,8, in 90% Glycerin) zu den Proben in der Mittedes Abstandshalters. HINWEIS: Diese Lösung wird braun / schwarz, wenn erlaubt zu oxidieren.
- Vorsichtig einen kreisförmigen Deckglas (Nr. 1.5) in den Proben zu gewährleisten, dass die mountant gleichmäßig verteilt wird, und dass alle Kanten des Deckglases einen Kontakt mit dem Abstandhalter. Setzen Sie den Deckel-schlüpfte Rutsche Oberseite nach unten auf einige fusselarme Seidenpapier.
- Drücken Sie fest, um sicherzustellen, dass das Deckglas vollständig auf den Abstandshalter haftet, und dass überschüssiges mountant wird entfernt. Wiederholen, bis keine mehr mountant Blots das Papier.
- Sauber und beschriften Sie die Folie (n) entsprechend, und speichern sie in der Dunkelheit bis Bildgebung, kurzzeitig bei -20 ° C oder Langzeit bei -80 ° C. Nachdem die Folien aus dem Speicher zu entfernen, damit sie vor der Abbildung vollständig auftauen.
- Bild die montierten Proben ein konfokales Mikroskop mit Fliesenerfassung und hoher Vergrößerung Ziel verwenden. Bild die Paare Explantation unter Verwendung eines 40X -Ölimmersionsobjektiv bei 4 um z-Intervalle mit 488-nm, 568 nm und 647-nm-Laser lines die grünen, roten und dunkelroten Fluorophore jeweils verwendet für Protein und mRNA - Nachweis in dieser Studie 12 zu erregen.
HINWEIS: Mit Ziegeln gedeckte Bilder wurden nach dem Erwerb eingenäht für die Analyse eines einzelnen Bildes zu bilden.
5. Nach der Akquisition Bildanalyse
- Verwenden Sie die Bildanalyse-Software eine Region von Interesse innerhalb des PSM jeder Versuchsprobe zu definieren.
- In den Expressionsniveaus, subtrahieren Hintergrund und Schwellwertbilder auf dem Niveau einer nicht-primären Kontrollprobe vor der nachfolgenden Quantifizierung quantifizieren. Definieren einen Ursprung, der eine Achse und eine Einheitslänge für jede Probe.
- Berechne die Fluoreszenzintensität als Funktion der Position entlang dem normalisierten rostro-kaudal - Achse für jedes der M Proben 12. Nach der Normalisierung einer Intensität Matrix f (i, j), die die Intensität an der i beschreibt die Intensität Plots, legen Sie die Intensitätsprofile nebeneinander und erhalten th räumliche Position in der j - ten Probe.
6. Zeitliche Reihenfolge der Proben
- Zur zeitlichen Ordnung eines bekannten Taktkomponente abzuleiten, definieren ihre Intensität Matrix. Dann ordnen die Spalten der Intensitätsmatrix, um ein zeitlich periodisches Muster zu erhalten. Dazu definieren Sie die Funktion
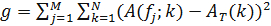
wobei A (f j; k) die Autokorrelationsfunktion der j - ten Spalte von f und A T darstellt , ist eine Funktion Zielautokorrelations, ausgewählt , um die zeitliche Periodizität des Musters zu erzwingen, gegeben durch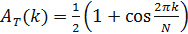
- Verwenden Sie die Metropolis-Hastings (oder eines anderen Minimierungsalgorithmus) 12 die Reihenfolge der Proben zu identifizieren, die die Funktion g minimieren. Somit bestimmen die Reihenfolge derM Proben, die die zeitliche Periodizität eines bekannten Taktkomponente maximiert.
- Unter Verwendung der abgeleiteten zeitlichen Reihenfolge der M Proben, konstruieren eine geordnete kymograph für das Expressionsmuster im Bereich Partnered Kanal 12.
Ergebnisse
Dieses Protokoll ermöglicht die Visualisierung des Raum - Zeit - Profils eines Proteins von Interesse neben Takt Gentranskription in der Maus PSM 12. Beispielsweise DLL1 (1A-C) und Notch1 (1D-F) Proteinexpression gezeigt Lfng aus Synchronität mit dem naszierenden Transkription der Notch-regulierten Segmentierungstakt Gen zu oszillieren. Quantifizierung der DLL1, Notch1 und Lfng (i) Signalintensität in Bezug auf die antero-posterior (AP) -Achse des PSM (1G) zeigt klar oszillatorischen Expressionsdynamik für diese Ziele (Abbildung 1H-J). Die Raum-Zeit-Profil von DLL1 und Notch1 Protein-Expression während des Taktzyklus sind übersichtlich visualisiert und quantifiziert dieses Protokoll durch die Post-Akquisition-Bildanalyse von hochauflösenden fixierten Gewebebilddaten.
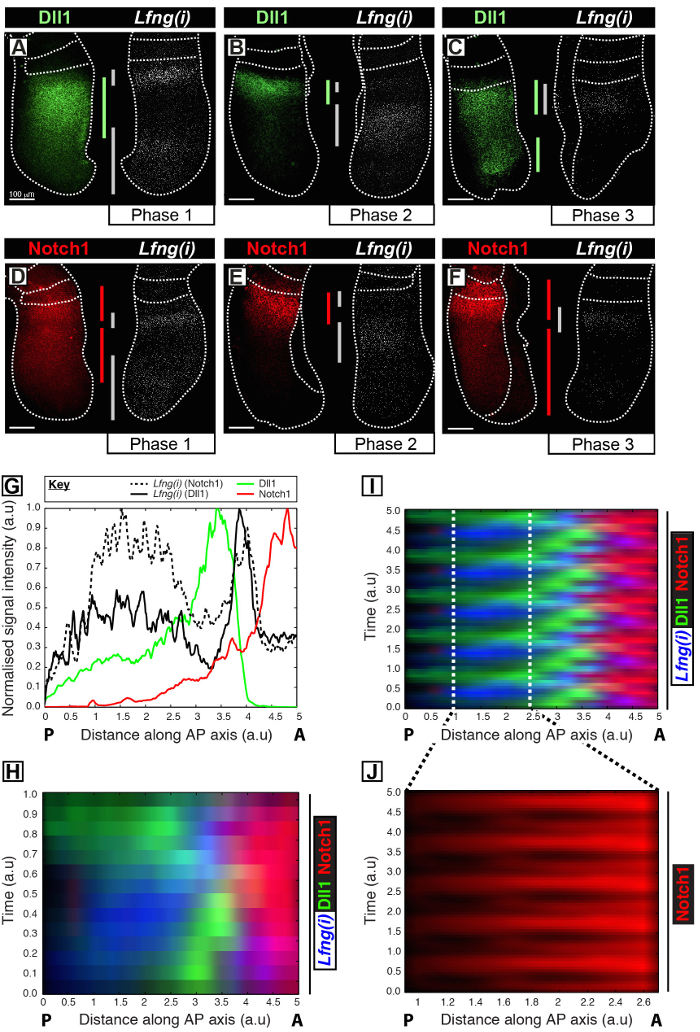
Abbildung 1: Räumlich-zeitliche Visualisierung und Quantifizierung von DLL1 und Protein Expression Dynamics Notch1. (AF) Paare von Explantaten aus sechs E10.5 Embryonen (AF) , um die räumliche Verteilung der DLL1 Protein (AC) oder Notch1 Protein (DF) in eine Hälfte neben der Detektion von Lfng pre-mRNA (Lfng (i)) in das zeigt , entsprechende Gegen Hälfte eines jeden Paares. Platten werden nach Phase 1 (A und D) angeordnet ist , Phase 2 (B und E) und Phase 3 (C und F) des Segmentierungstaktzyklus bestimmt , wie es durch die räumliche Profil Lfng (i) expression. Das Ausmaß der Expressionsdomänen für DLL1 (grün), Notch1 (rot) und Lfng (i) (grau) entlang der anteroposterioren Achse des PSM haben been abgegrenzt durch farbige Balken. Die punktierten Linien kennzeichnen die Positionen der zuletzt gebildeten Somiten (s), die Außenkanten der PSM und dem benachbarten Nervengewebe (C und E). Maßstabsbalken (unten jeder Tafel links, AF) repräsentieren 100 & mgr; m. (G) Ein Beispiel Intensität Grundstück , welches die axiale Variation der Signalintensität über die PSM. Die Daten werden aufgezeichnet von zwei Paaren Explantation Lfng pre-mRNA (schwarz gestrichelte Linie) in einer Explantation im Vergleich zu Notch1 Protein (rot) in der Gegen Explantation (Embryo 1), sowie Lfng pre-mRNA (schwarze Linie) zeigt , die in eine andere Explantation zu DLL1 Protein (grün) in der gegen~~POS=TRUNC Explantation (Embryo 2) verglichen. Gemessene Signalintensität (y-Achse) aufgetragen gegen die axiale Position (x-Achse; anterioren PSM [A] nach rechts und posterior PSM [P] nach links). (H) A kymograph die räumliche Verteilung der DLL1 zeigt, Notch1 und Lfng (i) überzahlreiche PSMs. Jede Zeile der kymograph stellt die Signalintensität eines einzelnen PSM Explantation. Zeilen werden in zeitlicher Reihenfolge angeordnet sind entsprechend der Raum - Zeit - Verteilung der Lfng pre-mRNA (i) die räumlich - zeitliche Verteilung der DLL1, Notch1 und Lfng (i) über mehrere Taktschwingungen durch die periodische Erweiterung der Daten simuliert wird dargestellt in (H) , die Schwingungs Natur der DLL1 und Notch1 Ausdruck Dynamik hervorheben. (J) Pulsatile Notch1 Proteinexpression in der kaudalen PSM wird durch Vergrßerung des Gebiets deutlich in der virtuellen kymograph abgegrenzte in (I) gezeigt. Geändert von Referenz 12. Bitte hier klicken , um eine größere Version dieser Figur zu sehen.

Abbildung 2: Quantifizierung der Räumlich-zeitliche Dynamik der DLL1 und Notch1 Protein Expression. (A) Ein Beispiel Intensität Plot zeigt axiale Variation in der Signalintensität über das PSM. Daten aufgezeichnet von zwei Explantation Paare Lfng prä-mRNA (schwarz gestrichelte Linie) in einer Explantation im Vergleich zu Notch1 Protein (rot) in der Gegen Explantation Hälfte zeigt, sowie Lfng pre-mRNA (schwarze Linie) in einem halben Explantation von einem zweite Schwanz im Vergleich zu DLL1 Protein (grün) in der gegen~~POS=TRUNC Explantation Hälfte des zweiten Schwanz. Gemessenen Intensitäten (y-Achse) aufgetragen gegen axiale Position (x-Achse; rostral [A] nach rechts und caudal [P] nach links). (BH) Kymographen zeigen die räumliche Verteilung von Notch1, DLL1, NiCd- und Lfng (i) in zahlreichen PSMs. (B und C) NICD (B) und DLL1 (C) Ausdruck in PSM Abschnitte; (D und E) Lfng (i) (D) und DLL1 ( E) in kontralateralen Explantation Hälften; (F und G) Lfng (i) (F) und Notch1 (G) in kontralateralen Explantat Hälften. Von Referenz 12. Bitte hier klicken , um eine größere Version dieser Figur zu sehen.
Diskussion
Kritische Schritte im Rahmen des Protokolls
Das vorliegende Protokoll beschreibt ein empfindliches Verfahren die quantitative Analyse von Low-Level-Proteinexpression und Schwingungsdynamik in E10.5 Maus PSM Explantate auszuführen. Ein robustes Protokoll sowohl für die Immunhistochemie und Fluoreszenz - in - situ - Hybridisierung (FISH) wird durch hochauflösende Vollmontage konfokale Abbildung gefolgt, und dann durch Bildanalyse und zeitliche Segmentierung von Kymographen eine Raum - Zeit - Karte von Protein - Expression über die PSM zu erzeugen. Ein hohes Signal-zu-Rausch-Verhältnis in Protein- und mRNA-Nachweis ist von wesentlicher Bedeutung für den Erfolg dieser Technik zu gewährleisten. Es muss sorgfältig getroffen werden, um alle Lösungen effektiv während der Waschschritte auszutauschen und die Temperatur der 65 ° C Waschungen in den entsprechenden Stufen der Schritt zu halten 3. Am günstigsten ist es, die Zeit zu nehmen wirksame Antikörper und RNA-Sonden gegen die Source- Ziele von Interesse und diese Reagenzien zu testengründlich auf ganze-mount Proben vor, dieses Protokoll zu starten.
Technische Änderungen und Fehlerbehebung
Die wichtigsten Probleme, die auftreten können, wenn dieses Protokoll Durchführung ergeben sich aus schlechten Signalerkennung Stärke und Qualität. Dies ist weitgehend abhängig von der Wirksamkeit der verwendeten Antikörper oder RNA-Sonden für die Immunhistochemie oder FISH Schritte in dem Protokoll, respectively. Eine Anzahl von verschiedenen Schritte können Optimierungs erfordern, bevor eine ausreichende Signalerfassung erreicht wird. Eine häufige Ursache für schlechte Signalerkennung ist unsachgemäße Fixierung; es ist zwingend notwendig, dass entweder frisch PFA oder PFA für nicht mehr als eine Woche bei 4 ° C gelagert wird verwendet, um die Proben zu beheben. Die Länge der Fixierung kann auch eine Optimierung erfordern, abhängig von der Antikörper oder RNA-Sonde verwendet. Für Antikörper, ist es ratsam, den Anweisungen des Herstellers, wo möglich, während für die RNA-Sonden zu folgen, wir die Anhörung der veröffentlichten Literatur zu beraten.
In dieser Studie verwendeten wir eine RNA - Sonde, die die prä-mRNA des Takt Gen Lfng spezifisch erkennt. Aufgrund seiner relativen Mangel an Fülle, Erkennung von Lfng pre-mRNA erfordert eine lange Zeit der Inkubation mit der Sonde in Hybridisierungsmischung 5x Saline-Natriumcitrat (SSC) für eine gute Signalerkennung enthält. Dieselben Bedingungen können auf andere Sonden anwenden , die schwach exprimierten mRNAs erkennen, aber in unserer Erfahrung, die Detektion von stabiler mRNA - Ziele kann eine kürzere Sondenhybridisierungsschritt und unteren SSC - Konzentrationen in der Hybridisierungsmischung (beispielsweise 1,3x SSC) erfordern. Für beide Immunhistochemie und FISH, muss das Protokoll zunächst auf ganze Embryonen optimiert werden, und die optimale Konzentration der Antikörper oder Sonde muss empirisch ermittelt werden.
Einschränkungen der Technik
Wie oben erwähnt, ist der Erfolg dieser Technik stark abhängig von der Qualität des Proteins und der mRNA-Detektion. We wurden mehrere Vorschläge dargelegt, wie Protein- und mRNA-Nachweis kann verbessert werden, aber in Abwesenheit von hochwertigen Fluoreszenzsignalerfassung, gibt es keine Möglichkeit der Versuch fortgesetzt werden kann. Die Anzahl der Protein-Targets, die in jeder Gewebeprobe analysiert werden können, wird durch die spektrale Auflösung des konfokalen Mikroskops und durch die Epitope der verwendeten Antikörper beschränkt. In dieser Studie konnten wir auf drei Epitope für Proteindetektion neben einer DNA - Fleck auf jeder Probe 12 verbrauchen. Dieses Protokoll erlaubt nur die Erfassung einer mRNA Ziel, obwohl Strom alternative Verfahren verwendet werden könnten , 14 diese auf bis zu drei Ziele zu erhöhen.
Bedeutung der Technik in Bezug auf bestehende / Alternativmethoden
Das hier beschriebene Verfahren stellt eine empfindliche Technik Low-Level-Protein Schwankungen ganz-mount PSM Explantate zu erkennen. Die Quantifizierung dieser Dynamik istmöglich durch in entsprechenden kontralateralen Explantate FISH für ein bekanntes Uhr-Gen durchgeführt wird. Eine Bibliothek von Kymographen generiert, organisiert über Zyklus eine Segmentierung Uhr werden kann, Hervorhebung der Raum-Zeit-Ausdruck Dynamik ein Ziel von Interesse innerhalb dieses Zeitrahmens. Ein wesentlicher Unterschied in dieser Technik gegenüber anderen ist die Verwendung von Rechen Automatisierung zeitlich große Datenmengen zu bestellen, die die räumlich-zeitliche Dynamik Ausdruck neuer Taktkomponenten ermöglicht in unvoreingenommen analysiert werden. Beispielsweise kann diese Technik bereitgestellt Einblick, wie DLL1 und Notch1 Proteine und ihre Schwingungen über die gesamte PSM co-reguliert. Alternative Methoden in diesem Zusammenhang auch auf die Immunfärbung verlassen, aber sie haben die kleinen Schwankungen in DLL1 und Notch1 Proteinspiegel in der Schwanz PSM nicht erkennen, die offensichtlich mit dieser Methode waren. Stattdessen berichtet sie einen stetigen Steigung des Ausdrucks , die am stärksten in der rostralen Region 9 , 10, 11. Dies könnte auf die Tatsache zurückzuführen sein, dass dieses Protokoll eine längere primäre Antikörper Inkubationszeit (3 - 5 Tage, im Gegensatz zu über Nacht), die erforderlich sein können geringere Mengen an Protein zu detektieren. Da die Ebenen von DLL1 und Notch1 Ausdruck relativ hoch im rostralen PSM sind, haben diese können die Proben, die die Autoren Bild beeinflusst bei einer niedrigeren Belichtungseinstellung als wäre notwendig, um die Schwanzproteinexpression zu erkennen. Ein weiteres Potential Diskrepanz ergibt sich aus der Verwendung von nicht - fixierten Gewebe in der Studie von Chapman et al. In denen die vorübergehende Expression von DLL1 und Notch1 in der Schwanz PSM weniger sein können 9 gut erhalten.
Zukünftige Anwendungen oder Anfahrt nach der Technik Mastering
Sobald dieses Protokoll, Hochdurchsatz-Expressionsanalyse beherrscht wurde für jedes Protein von Interesse in der PSM durchgeführt werden.PSM Explantate aus mehreren Maus Würfen erzeugt wird, kann auf einmal verarbeitet werden, um die hohe Probennummer, die zur Analyse zu erzeugen. Obwohl wir nur Wildtyp-Embryonen in diesen Studien verwendet wurden, ist es möglich, diese Analyse durchzuführen unter Verwendung von gentechnisch veränderten Embryonen, um die Bedeutung eines oder mehrerer Faktoren auf die Proteinexpression Dynamik zu bewerten. Jenseits der PSM kann dieses Protokoll auf andere Systeme angepaßt werden, die aus zwei Hälften zusammengesetzt sind kontralateralen und verwendet werden können, empfindlich auf niedriger Ebene Proteinexpression und Schwingungsdynamik zu erfassen. Ein Beispiel für die dieses Protokoll angepasst werden könnte , ist die Untersuchung der dynamischen Protein - Expression in der Maus Neuralrohr, da kontralateralen Hälften erzeugt werden könnten und kultiviert und Notch - Aktivität wurde sowohl anwesend sein und wichtige gezeigt 15 zum Mustern. Wir ermutigen andere Gruppen dieses Protokoll auf andere Systeme anzupassen und für künftige Verbesserungen, Feedback zu geben.
Offenlegungen
The authors have nothing to disclose.
Danksagungen
Diese Arbeit wurde von einem MRC studentship RAB unterstützt, ein MRC studentship zu CSLB und einem WT Projektförderung zu JKD (WT089357MA). Die Arbeit wurde auch durch ein Wellcome Trust Strategic Auszeichnung (097.945 / Z / 11 / Z) unterstützt. Wir danken Dr. E. Kremmer für die Art Gabe des DLL1 Antikörper und Dr. O. Pourquié für die Lfng RNA - Sonde.
Materialien
| Name | Company | Catalog Number | Comments |
| DMEM-F12 | Gibco (ThermoFisher Scientific) | 11320033 | |
| GlutaMAX™-1 (100x) | Gibco (ThermoFisher Scientific) | 35050 | |
| Fetal Bovine Serum, qualified, E.U.-approved, South America origin | Gibco (ThermoFisher Scientific) | 10270106 | |
| Recombinant Human FGF-basic (154 a.a.) | Peprotech | 100-18B | |
| Penicillin/Streptomycin | Gibco (ThermoFisher Scientific) | 15140122 | |
| anti-mouse monoclonal Notch1 antibody^ | BD Pharmingen | 552466 | |
| anti-rat polyclonal Dll1 antibody^* | N/A | N/A | |
| Lfng intronic anti-sense RNA probe^* | N/A | N/A | |
| 16% paraformaldehyde | Pierce (ThermoFischer Scientific) | PI28908 | |
| Proteinase K, recombinant, PCR grade | Roche (Sigma-Aldrich) | 31158 | |
| Phosphate buffered saline (PBS), pH 7.4 | Made in house | N/A | |
| Triton-X 100 | Sigma-Aldrich | T8787 | |
| Bovine serum albumin (BSA) | Sigma-Aldrich | 5470 | |
| Normal goat serum (NGS) (heat-treated) | Gibco (ThermoFisher Scientific) | 16210072 | |
| Hoechst 33342 | ThermoFischer Scientific | H3570 | |
| Tween-20 | Sigma-Aldrich | P9416 | |
| Ethanol | Sigma-Aldrich | 46139 | |
| Glutaraldehyde | Sigma-Aldrich | 340855 | |
| Formamide | Sigma-Aldrich | F9037 | |
| Saline-sodium citrate (SSC) | Sigma-Aldrich | 93017 | |
| EDTA | Sigma-Aldrich | 798681 | |
| tRNA | Roche (Sigma-Aldrich) | 101095 | |
| Heparin | Sigma-Aldrich | H3149 | |
| Tris-buffered saline (TBS) | Made in house | N/A | |
| Blocking Buffer Reagent | Roche (Sigma-Aldrich) | 11096176001 | |
| anti-DIG horseradish peroxidase (HRP) conjugated antibody | Roche (Sigma-Aldrich) | 11207733910 | |
| Tyramide signal amplification (TSA) kit | Perkin Elmer | NEL744001KT | |
| *The Dll1 antibody and RNA probe used in this study are not commercially available. Please see acknowledgements for sources. | |||
| ^Antibodies/RNA probes should be sourced which are applicable to the research interests of the reader. | |||
Referenzen
- Oates, A. C., Morelli, L. G., Ares, S. Patterning embryos with oscillations: structure, function and dynamics of the vertebrate segmentation clock. Development. 139 (4), 625-639 (2012).
- Krol, A. J., Roellig, D., et al. Evolutionary plasticity of segmentation clock networks. Development. 138 (13), 2783-2792 (2011).
- Dequéant, M. -. L., Ahnert, S., et al. Comparison of Pattern Detection Methods in Microarray Time Series of the Segmentation Clock. PLoS ONE. 3 (8), 2856 (2008).
- Bailey, C., Dale, K. . Somitogenesis in Vertebrate Development. , 1-15 (2015).
- Maroto, M., Bone, R. A., Somitogenesis Dale, J. K. Somitogenesis. Development. 139 (14), 2453-2456 (2012).
- Kageyama, R., Masamizu, Y., Niwa, Y. Oscillator mechanism of notch pathway in the segmentation clock. Developmental Dynamics. 236 (6), 1403-1409 (2007).
- Ferjentsik, Z., Hayashi, S., et al. Notch Is a Critical Component of the Mouse Somitogenesis Oscillator and Is Essential for the Formation of the Somites. PLoS Genetics. 5 (9), 1000662 (2009).
- Wiedermann, G., Bone, R. A., Silva, J. C., Bjorklund, M., Murray, P. J., Dale, J. K. A balance of positive and negative regulators determines the pace of the segmentation clock. eLife. 4, 05842 (2015).
- Chapman, G., Sparrow, D. B., Kremmer, E., Dunwoodie, S. L. Notch inhibition by the ligand DELTA-LIKE 3 defines the mechanism of abnormal vertebral segmentation in spondylocostal dysostosis. Human Molecular Genetics. 20 (5), 905-916 (2011).
- Sparrow, D. B., Chapman, G., et al. A Mechanism for Gene-Environment Interaction in the Etiology of Congenital Scoliosis. Cell. 149 (2), 295-306 (2012).
- Okubo, Y., Sugawara, T., Abe-Koduka, N., Kanno, J., Kimura, A., Saga, Y. Lfng regulates the synchronized oscillation of the mouse segmentation clock via trans-repression of Notch signalling. Nature communications. 3, 1141 (2012).
- Bone, R. A., Bailey, C. S. L., et al. Spatiotemporal oscillations of Notch1, Dll1 and NICD are coordinated across the mouse PSM. Development. 141 (24), 4806-4816 (2014).
- Shea, K., Geijsen, N. Dissection of 6.5 dpc mouse embryos. Journal of Visualized Experiments. (2), 160 (2007).
- Denkers, N., García-Villalba, P., Rodesch, C. K., Nielson, K. R., Mauch, T. J. FISHing for chick genes: Triple-label whole-mount fluorescence in situ hybridization detects simultaneous and overlapping gene expression in avian embryos. Developmental Dynamics. 229 (3), 651-657 (2004).
- Stasiulewicz, M., Gray, S. D., et al. A conserved role for Notch signaling in priming the cellular response to Shh through ciliary localisation of the key Shh transducer Smo. Development. 142 (13), 2291-2303 (2015).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten