Method Article
Gromer Nachweis von Biomarkern mithilfe einer molekularen Prägung basierte kapazitive Biosensor
In diesem Artikel
Zusammenfassung
Hier präsentieren wir ein Protokoll für die Erkennung und Quantifizierung von niedrigen reichlich Moleküle in komplexe Lösungen mit molekularen Prägung in Kombination mit einer Kapazität Biosensor.
Zusammenfassung
Die Fähigkeit zu erkennen und quantitate Biomoleküle in komplexe Lösungen seit jeher begehrte in Naturwissenschaften; für den Nachweis von Biomarkern, Schadstoffe und andere Moleküle des Interesses verwendet wird. Eine häufig verwendete Technik für diesen Zweck ist das Enzym-linked Immunosorbentprobe Assay (ELISA), wo oft ein Antikörper richtet sich an ein bestimmtes Ziel-Molekül, und ein zweite beschriftete Antikörper für den Nachweis des primären Antikörpers verwendet wird, ermöglicht die Absolute Quantifizierung von das Biomolekül unter Studie. Die Nutzung von Antikörpern als Erkennungselemente schränkt jedoch die Robustheit der Methode; genauso wie die Notwendigkeit der Verwendung von beschrifteten Moleküle. Um diese Einschränkungen zu überwinden, wurde molekulare Prägung umgesetzt, Erstellen von künstlichen Anerkennung Websites ergänzen das Vorlage-Molekül und Bilddateien die Notwendigkeit der Verwendung von Antikörpern für die erste Bindung. Darüber hinaus kann für noch höhere Empfindlichkeit, beschrifteten Sekundärantikörper von Biosensoren, die unter Berufung auf die Kapazität für die Quantifizierung des Zielmoleküls ersetzt werden. Dieses Protokoll beschreibt eine Methode, um schnell und markierungsfreie erkennen und quantitate Low-reichlich Biomoleküle (Proteine und Viren) in komplexen Proben mit einer Empfindlichkeit, die deutlich besser ist als die gängigen Systeme wie ELISA. Dies wird alles von molekularen Prägung in Kombination mit einer Kapazität Biosensor vermittelt.
Einleitung
Die Quantifizierung von Biomolekülen ist in vielen verschiedenen Forschungsfeldern innerhalb der Wissenschaft, die Methoden wie die Radioimmunoassay (RIA) oder ELISA1verwendet. Einige dieser Methoden erfordern eine beschriftete Reagenz wie ein Radioisotop oder Enzym beschriftet Antikörper/Antigen, wodurch sie arbeitsintensiv und zeitaufwändig mit komplexen Verfahren2. Darüber hinaus sind die Robustheit, die Selektivität und die Sensitivität dieser Methoden nicht ausreichend für alle Analysen; insbesondere sind sie nicht ausreichend, wenn Attogram Mengen werden, anstatt Piktogramm Mengen3 analysiert müssen. Zu diesem Zweck haben Biosensoren beträchtliches Interesse4,5, insbesondere in Kombination mit molekularen Prägung für eine erhöhte Robustheit gewonnen.
Molekulare Prägung stützt sich auf Hohlräume durch funktionale Monomere um die Vorlage6, Erstellen von künstlichen Anerkennung Websites, die perfekt die Vorlage7ähneln dadurch zu schaffen. Diese Technik ist für verschiedene Anwendungen, einschließlich Drug Delivery Systeme und analytischen Trennung, sondern auch als Biorecognition Elemente in Biosensoren8,9,10verwendet worden. Allerdings gibt es noch einige Schwierigkeiten bei der Gestaltung von molekular geprägte Polymere (MIPs) für makromolekulare Vorlagen wie Proteinen und Zellen11,12. Aus diesem Grund haben viele Forscher konzentrierten sich auf Prägung der Vorlage Protein direkt auf einem Substrat, wodurch eine Fläche, die von der Ziel-Protein-12erkannt wird. Diese Oberflächenbeschichtung-Technik für den Einsatz von Anerkennung Hohlräume für große Moleküle und Baugruppen einschließlich Proteine verwendet wird Microcontact Prägung13,14bezeichnet. Die allgemeine Vorgehensweise der Methode richtet sich nach der Polymerisation zwischen zwei Oberflächen - eine Vorlage-Stempel und eine Polymer-Unterstützung - auf denen die Vorlage adsorbiert an eine Oberfläche15,16und brachte in Kontakt mit der Monomer-behandelte Oberfläche. Auf diese Weise bildet sich eine dünne Polymerfolie auf die Unterstützung durch UV-Polymerisation. Schließlich wird die Vorlage entfernt, hinterlässt Vorlage bestimmte Hohlräume an der Oberfläche der aufgedruckten Elektrode. Diese Methode hat einige Vorteile, unter anderem einen reduzierte Aktivität Verlust des aufgedruckten Moleküls, sowie eine sehr geringe Mengen an Vorlage Moleküle für die Bedruckung16,17verarbeiten. Somit können diese kostengünstige, stabile, empfindlichen und selektiven Oberflächen auf die Sensorflächen, Ausrichtung auf eine Vorlage der Wahl des Benutzers erstellt werden.
Der Biosensor kann für die Erkennung von einzelnen Proteine und viel größere Biomakromoleküle, inklusive Viren verwendet werden. Eine bestimmte Gruppe von Viren, die vor kurzem Interesse zu gewinnen ist die Bakteriophagen, das ist ein Virus, der Bakterien infiziert. Schnelle und empfindliche Detektion von Bakteriophagen ist wichtig bei biotechnologischen und biopharmazeutische Prozesse um die Infektionen von Bakterienkulturen mit Bakteriophagen18zu bestimmen. Die am häufigsten verwendete biologischen Test für Bakteriophage Erkennung ist der Doppelschicht Agar Methode19, mühsam und zeitaufwendig. Mehrere Versuche wurden unternommen, neuartige Diagnose-Tools für Viren (Bakteriophagen) entwickeln wie atomic Force Microscopy (AFM)20, Interferometrie21, Elektrochemie22und Sensor System23, 24. viel Arbeit konzentrierte sich auf Biosensoren aufgrund ihrer Vorteile als einfach zu bedienen, sehr empfindlich, und in der Lage, Echtzeit-Messung15,25. Eine bestimmte Art von Biosensor basiert auf Veränderungen in der Kapazität. Diese kapazitiven Biosensoren sind die elektrochemischen Sensoren zur Messung von Veränderungen in den dielektrischen Eigenschaften wenn ein Analyten mit einem Biorecognition Element auf die Sensoroberfläche interagiert, was zu einer Abnahme der Kapazität2,4 . Kapazitive Biosensoren wurden für den Nachweis der verschiedenen Analyten wie Antigene, Antikörper, Proteine und Schwermetall-Ionen6,26,27,28verwendet. Diese Arten von Biosensoren haben viele Vorteile, wie inhärente Schnelligkeit, hohe Empfindlichkeit, Einfachheit, geringe Kosten, einfache Handhabung und Echtzeit-Messung ohne Kennzeichnung29.
Die hierin beschriebene Methode zielt zu ermöglichen die Erkennung und Quantifizierung von niedrig-reichlich Biomoleküle in hoch komplexen Proben, ohne die Notwendigkeit der Verwendung Kennzeichnung. Insbesondere eignet sich die Technik im Bereich von Atto-Picogram von Biomolekülen, wo nicht andere kommerziell vorhandene Instrumente genau ihr Ziel quantitate.
Protokoll
1. Änderung der Glasabdeckung rutscht (Vorlage Briefmarken)
- Um das Glas Deckel rutscht zu reinigen, tauche sie nacheinander in 10 mL von 1,0 M HCl, deionisiertes Wasser und 1,0 M NaOH, bzw. für 10 min in den einzelnen Schritten in einem Ultraschallreiniger bei Raumtemperatur.
- Trocknen Sie das Glas Deckel rutscht mit Stickstoffgas.
Hinweis: Das Deckel rutscht durch Verdampfung unter dem Strahl des gasförmigen Stickstoff getrocknet.
- Trocknen Sie das Glas Deckel rutscht mit Stickstoffgas.
- Tauchen Sie die gereinigten und getrockneten Cover Liegeplätze auf 10 % (V/V), 10 mL Lösung von 3-amino-Propyl-Triethoxysilane (APTES) in Ethanol für 1 h Aminogruppen auf dem Abdeckung Glas, bei Raumtemperatur einzuführen.
- Spülen Sie das Deckel rutscht mit entionisiertem Wasser.
- Trocknen Sie die Cover-Slips mit Stickstoffgas.
- Tauchen Sie APTES veränderte Cover rutscht in 5 % (V/V), 10 mL Lösung von Glutaraldehyd in 10 mM Phosphatpuffer (pH 7,4) für 2 h um die Aminogruppen auf der Oberfläche, bei Raumtemperatur6,15zu aktivieren.
- Spülen Sie die Deckel Zettel mit 10 mM Phosphatpuffer (pH 7,4) um überschüssige Glutaraldehyd von der Oberfläche zu entfernen.
- Trocknen Sie die Cover-Slips mit Stickstoffgas.
- 1,0 mL der Vorlage (Protein/Bakteriophagen) in 10 mM Phosphatpuffer (pH 7,4) in der Konzentration von 0,1 mg/mL Lösung vorzubereiten.
Hinweis: Auflösung 0,1 mg Protein in 1,0 mL Phosphatpuffer (10 mM, pH 7.4). Wenn nötig, einem Spektrophotometer (280 nm) kann verwendet werden, um die Konzentration des Proteins zu bestimmen.- Drop-200 µL dieses Template-Lösung auf der modifizierten Deckel rutscht und über Nacht bei 4 ° C inkubieren.
Hinweis: Die überschüssige Template-Lösung lässt sich vorbereiten 1-2 weitere Vorlage Briefmarken in der Charakterisierung Studien verwendet werden. - Spülen des Deckel rutscht mit 10 mM Phosphatpuffer (pH 7,4) um die ungebundenen Vorlage von der Oberfläche zu entfernen.
Hinweis: Um die Elektroden zu spülen, waschen Sie sie mit Phosphatpuffer (10 mM, pH 7,4) für 30 s. - Trocknen Sie die Cover-Slips mit Stickstoffgas.
Hinweis: Das Deckel rutscht sollte bei 4 ° C gespeichert werden, bis sie in der Polymerisation Schritt verwendet werden.
- Drop-200 µL dieses Template-Lösung auf der modifizierten Deckel rutscht und über Nacht bei 4 ° C inkubieren.
2. Änderung der kapazitiven Gold-Elektroden
- Reinigen Sie die Elektroden, tauchen die Elektroden in ein kleines Becherglas, sequentiell, mit 5 mL Ethanol (70 %), deionisiertes Wasser, Aceton, deionisiertes Wasser, saure Piranha-Lösung (3:1 H2SO4: H2O2, V/V), und entionisiertem Wasser, jeweils für 10 min in den einzelnen Schritten in einem Ultraschallreiniger bei Raumtemperatur.
- Trocknen Sie die Elektroden mit Stickstoffgas.
- Um die Electropolymerization von Tyramin durchzuführen, bereiten Sie 8 mL 10 mM Tyramin Lösung in 10 mM Phosphat-Puffer (pH 7,4) mit Ethanol (2 mL)15.
Hinweis: Das Gesamtvolumen der Tyramin-Lösung muss 8 mL insgesamt einschließlich 2 mL Ethanol.- Führen Sie zyklische voltammetrischen Scans (15 Zyklen) in diese Lösung mit einem Potentiostaten für ein Potential von 0 - 1,5 V (Ag/AgCl) und eine Scan-Rate von 50 mV/s30.
- Spülen Sie die Elektroden mit entionisiertem Wasser.
- Trocknen Sie die Elektroden mit Stickstoffgas.
- Tauchen Sie die Elektroden in eine Lösung mit 30 mM Acryloyl-Chlorid und 30 mM Trimethylamin in Toluol (VGesamt = 5 mL) bei Raumtemperatur über Nacht6,15,30.
- Spülen Sie die Elektroden mit entionisiertem Wasser.
Hinweis: Um eine bessere Entfernung von nicht umgesetztes Acryloyl-Chlorid und Trimethylamin Rückstände zu gewährleisten, ist es auch möglich, die Oberfläche mit NaOH nach deionisiertes Wasser zu waschen. - Trocknen Sie die Elektroden mit Stickstoffgas.
- Spülen Sie die Elektroden mit entionisiertem Wasser.
3. Vorbereitung der Vorlage bedruckt kapazitive Gold-Elektroden
-
Vorbereitung der Bakteriophagen eingeprägt kapazitive goldene-Elektroden
- Bereiten Sie vor der Polymerisation eine Monomer-Lösung mit Monomer (N-Hydroxymethyl Acrylamid) und Vernetzer (Polyethylenglykol-400-dimethacrylat) in einem Verhältnis von 1:5 (Mol/Mol) in 1 mL 10 mM Phosphatpuffer (pH 7,4).
Hinweis: Die Monomer-Lösung mit Monomer und Vernetzer kann in unterschiedlichen Verhältnissen verwendet werden, oder die Arten von Monomeren und Vernetzer können entsprechend der Vorlage für die Optimierung der spezifischen Interaktion geändert werden. - Fügen Sie 1 mg Foto-Initiator in dieser Lösung.
Hinweis: Erfolgt die Polymerisation unter UV-Licht, muss dann die Foto-Initiator verwendet werden, die Polymerisation zu initiieren. Wenn die Polymerisation durch radikalische Polymerisation erfolgt, muss der Typ des Initiators geändert werden. - Pipette 1,5 µL dieser Lösung auf die Goldoberfläche der modifizierten gold-Elektrode.
Hinweis: Die Goldoberfläche der Elektrode ist schematisch in Abbildung 1dargestellt. - Bringen Sie die Vorlage-Stempel in Kontakt mit dem Monomer-Lösung auf der Oberseite der gold Elektrodenoberfläche.
- Die UV-Polymerisation zu initiieren (365 nm, 400 W) und für 15 min31weiter.
Hinweis: UV-Polymerisation erfolgt in einem kühlen Schrank die bis-25 ° C eingestellt ist, vor dem Beginn der Polymerisation. Dann das UV-Licht aushärten System eingeschaltet ist und die Polymerisation wird für 15 Minuten vor dem Ausschalten das UV-Licht Härtungssystem fortgesetzt. - Mit Pinzette entfernen des Vorlage-Stempels von der Oberfläche.
Hinweis: Beim Entfernen des Vorlage-Stempels von der Oberfläche, die Polymere Film auf der Oberfläche beschädigt werden. Daher sollte der Stempel von der Oberfläche sehr vorsichtig und langsam entfernt werden ohne Gewaltanwendung. - Spülen Sie der Elektrodenoberfläche mit entionisiertem Wasser und trocknen Sie ihn mit Stickstoffgas.
- Tauchen Sie die Elektroden in 1 mL 10 mM 1-Dodecanethiol in Ethanol für 20 min um Nadelstiche auf der Elektrodenoberfläche decken vorbereitet.
- Spülen Sie die Elektroden mit entionisiertem Wasser und trocknen Sie die Elektroden mit Stickstoffgas.
- Bereiten Sie vor der Polymerisation eine Monomer-Lösung mit Monomer (N-Hydroxymethyl Acrylamid) und Vernetzer (Polyethylenglykol-400-dimethacrylat) in einem Verhältnis von 1:5 (Mol/Mol) in 1 mL 10 mM Phosphatpuffer (pH 7,4).
-
Vorbereitung des Proteins eingeprägt kapazitive Gold-Elektroden
- Vor der Polymerisation, bereiten eine Monomer-Lösung mit Monomeren (Acrylamid: 54 mg; N-Hydroxymethylacrylamide: 140 µL; N-isopropylacrylamide: 85,6 mg) und Vernetzer (Methylenebisacrylamide: 9,5 mg) in 820 µL ultrareines Wasser30,32.
- Vorbereiten von 5 % (V/V) N, N, N', N'-Tetramethylethyldiamine (TEMED) in Reinstwasser.
- Fügen Sie 20 µL TEMED Lösung in die Monomer-Lösung und Spülen mit Stickstoffgas für 5 min.
- 10 % (w/V) Ammonium Persulphate (APS) in ultrareines Wasser vorzubereiten.
- 20 µL der APS-Lösung in die Monomer-Lösung hinzufügen.
- Pipette 1,5 µL der Monomer-Lösung auf der modifizierten gold Elektrodenoberfläche.
- Den Vorlage-Stempel mit dem Monomer-behandelten Oberfläche in Kontakt zu bringen.
- Starten Sie Polymerisation bei Raumtemperatur zu und fahren Sie 5 h.
Hinweis: Für die Herstellung von Protein-Aufdruck gold-Elektroden, anstelle von UV-Polymerisation, radikalische Polymerisation bei Raumtemperatur (25 ° C) erfolgt mit APS-TEMED als des Initiator-Katalysators. - Entfernen Sie Vorlage-Stempel von der Oberfläche vorsichtig mit Hilfe einer Pinzette.
- Die Elektrode mit entionisiertem Wasser spülen und trocknen mit Stickstoffgas.
- Tauchen Sie die Elektroden in 1 mL 10 mM 1-Dodecanethiol in Ethanol für 20 min um Nadelstiche auf der Elektrodenoberfläche decken vorbereitet.
- Spülen Sie die Elektroden mit entionisiertem Wasser und trocknen Sie die Elektroden mit Stickstoffgas.
4. Charakterisierung der Elektrodenoberfläche mit Rasterelektronenmikroskopie (SEM)
- Montieren Sie die Exemplare auf Aluminium-Halter mit selbstklebenden Carbon Band.
- Bestreichen Sie die Elektroden mit 10 nm Palladium/Gold.
- Untersuchen Sie die Elektroden mit SEM
5. kapazitive Echtzeitmessungen mit Vorlage bedruckt kapazitive Gold-Elektroden
- Einfügen Sie die aufgedruckten kapazitiven gold-Elektroden in die elektrochemische Messzelle integriert, um eine kapazitive biosensor
- 100 mL Regeneration Puffer (25 mM Glycin-HCl, pH 2.5, einschließlich 50 mM Tween-20) und 1 L laufenden Puffer vorzubereiten (für Bakteriophage eingeprägt System: 10 mM Phosphat, pH 7,4; für Protein-Aufdruck System: 50 mM Tris-HCl, pH 7.4).
- Starten Sie die Analyse mit der Injektion von Regeneration Puffer, das System und laufenden Puffer, um das System für 25 min erneut equilibrate zu regenerieren.
- Bereiten Sie standard-Template-Lösungen im Bereich gewünschte Konzentration im laufenden Puffer vor.
Hinweis: Bereiten Sie zuerst einen Vorrat an der Template-Lösung durch Auflösung 0,1 mg Protein oder ca. 108 Pfu Bakteriophagen, in 1,0 mL Phosphatpuffer (10 mM Phosphat, pH 7.4). Dann bereiten Sie die Standardlösungen für die Eichkurve indem man zehn sequentielle 10-divisibel Verdünnungen aus der Stammlösung. Diese Lösungen werden mit der kapazitiven Biosensor in Schritt 5.5 analysiert werden. Proteinkonzentration kann mit einem Spektralphotometer gemessen werden (280 nm). Um die Bakteriophagen-Konzentration zu messen, eine double-Layer-Agar-Methode eingesetzt werden, die beschrieben wird in Einzelheiten vorher19. - Diese standard-Lösungen gegenüber dem Vorquartal um das System unter optimalen Bedingungen 250 µL zu injizieren (Volumenstrom: 100 µL/min, Temperatur: 25 ° C).
Hinweis: In dieser Anwendung wurden die standard Proteinlösungen vorbereitet im Konzentrationsbereich von 1,0 x 10-4 - 1.0 x 10-14, während die Bakteriophagen-Konzentrationen im Bereich von 1,0 x 101 - 1.0 x 105 Pfu/mL waren. Die Lösungen wurden in das Einspritzventil gelegt und nacheinander in das System, Injektion Proben in Triplicates über die einzelnen Spritzenpumpe und multiport Ventil injiziert. Die Abnahme der Kapazität nach der Injektion der Standardlösungen aus der Bindung der Vorlage an den aufgedruckten Hohlräumen wird automatisch durch die Gerätesoftware überwacht.
Ergebnisse
Anhand des Protokolls gemäß Schaltplan in Abbildung 1wird eine nackte gold Elektrode mit einer Vorlage, eine Biomacromolecule Darstellung bedruckt werden. Diese Elektrode kann in eine kapazitive Biosensor (Abbildung 2), so dass die stabile Anwendung einer Vorlage auf die Elektrode und die Messung von Veränderungen in Kapazität bei der Bindung der Vorlage angewendet werden.
Eine schematische Darstellung der kapazitiven Biosensor ist in Abbildung 2dargestellt. Die MacOS-Pumpe, die für kontinuierliche Injektion von den laufenden Puffer (10 mM Phosphat, pH 7,4) und der Regeneration-Puffer (25 mM Glycin-HCl, pH 2,5) während der Regeneration in der Messzelle verantwortlich ist, ist in der Abbildung deutlich erkennbar. Die Messzelle besteht aus arbeiten, Referenz und Zähler-Elektroden. Das Einspritzventil besteht aus standard Protein/Bakteriophagen-Lösungen, die den Degasser zuerst auf der Durchreise sind und dann nacheinander in das System injiziert. Sobald die Lösungen die Arbeitselektrode eingefügt in der Messzelle erreicht, wird das Ergebnis in Echtzeit überwacht. Die Werte der Kapazität können registriert werden, indem Sie die Sensorgrams auf dem Computer-Bildschirm.
Abb. 3 und Abb. 4 zeigen die Unterschiede zwischen der Oberfläche des nackten und aufgedruckten gold-Elektroden. Diese Charakterisierung Schritt ist wichtig, um sicherzustellen, dass Polymere Hohlräume, gesehen als Rauheit auf der Oberfläche der Elektrode nach Prägung gibt. Abgesehen von SEM, gibt es auch andere Charakterisierungsmethoden AFM, Kontaktwinkel Messungen, einschließlich Ellipsometrie etc., die verwendet werden können, um die Oberfläche nach der Prägung zu charakterisieren. Auf diese Weise kann sichergestellt werden, dass der Prägung Prozess erfolgreich ist und dass Vorlage Vertiefungen auf der Oberfläche gebildet werden. In diesen Hohlräumen kann die Vorlage mit hoher Spezifität und Affinität binden.
Nach Einspritzen der Standardlösungen in der kapazitiven System, wurde im Durchschnitt der letzten fünf Lesungen automatisch von der Software berechnet, und die Kalibrierung Diagramme wurden durch die Veränderung der Kapazität gegen die Konzentration der Auftragen der Analyt. Der Rückgang der registrierten Kapazität entstand aus der Bindung der Vorlage. Je mehr Moleküle, die Bindung an das gold Elektrode Oberfläche, je höher die Verringerung der gesamten Kapazität nach dem allgemeinen Prinzip der kapazitiven Messungen. Abbildung 5 und Abbildung 6 zeigen, dass mit zunehmender Konzentration des Analyten, erhöht sich die ΔC wie erwartet. Der dynamische Bereich (zwischen denen ist der Konzentrationsbereich, wo das System für die Erkennung eines spezifischen Ziels nützlich ist) und der Nachweisgrenze (LOD) können durch die Analyse von diesen Diagrammen ausgewertet werden. Gemäß Abbildung 6, erkennt der Bakteriophagen eingeprägt kapazitive Biosensor Bakteriophagen im Konzentrationsbereich von 101 - 105 Pfu/mL, mit einem LOD-Wert von 10 Pfu/mL in dieser Studie. Abbildung 5 und Abbildung 6 auch Höhepunkt der Notwendigkeit zur Messung der Eichkurve im gleichen Intervall für die Vorlage-Konzentration erforderlich soll, da die Linearität der Regression über die Konzentrationen ( variieren Abbildung 5), oder haben unterschiedliche Neigungen (Abbildung 6). Es sollte auch angemerkt werden, dass aufgrund der niedrigen Konzentrationen verwendet, das System ist sehr empfindlich auf Schwankungen (Trübung in Probe, Luftzug, etc.) und es empfiehlt sich daher, mindestens laufen Triplicates, das Potenzial der einschließlich Ausreißer zu reduzieren . Aus dem gleichen Grund kann die Standardabweichung für stark verdünnte Proben ganz erheblich sein, wie in Abbildung 6zu sehen.
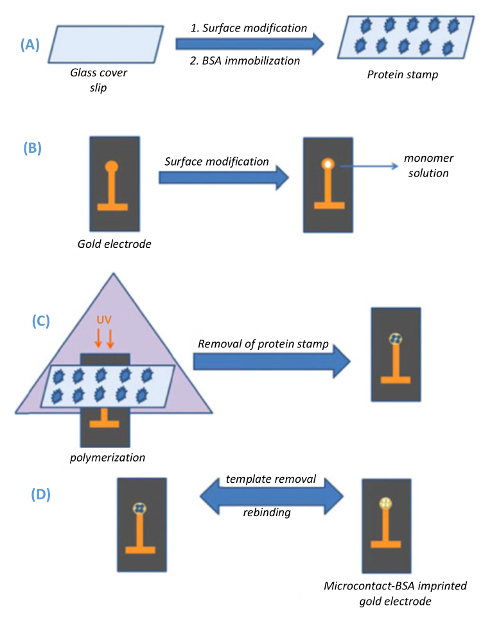
Abbildung 1 . Schematische Darstellung der Methode Prägung Microcontact. (A) Vorbereitung von Glas Deckel rutscht (Vorlage Briefmarken), (B) Vorbereitung der kapazitiven gold Elektroden (C) Microcontact Prägung der Schablone auf das gold Elektrodenoberfläche über UV-Polymerisation und (D) Entfernung von der Vorlage aus der Elektrodenoberfläche (übernommen aus Ertürk Et Al., Biotechnologie Berichte 2014 (3): 65-72 mit Erlaubnis). Bitte klicken Sie hier für eine größere Version dieser Figur.
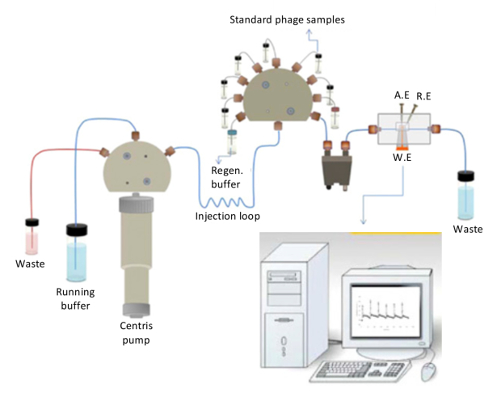
Abbildung 2. Schematische Darstellung der kapazitiven Biosensor. Das allgemeine Layout des kapazitiven Biosensor, die in dieser Studie verwendeten (übernommen aus Ertürk Et Al., Biotechnologie Berichte 2014 (3): 65-72 mit Erlaubnis). Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 3. Scanning Electron Microscopy of Protein eingeprägt Elektroden. REM-Bilder von (A) einer bloßen gold-Elektrode (Maßstabsleiste = 20 µm), und (B) ein Protein eingeprägt kapazitive gold Elektrode (Maßstabsleiste = 10 µm). Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 4. Scanning Electron Microscopy von Bakteriophagen eingeprägt Elektroden. REM-Bilder einer nackten gold Elektrode (Maßstabsleiste = 20 µm) (A), und einem Bakteriophagen eingeprägt kapazitive gold Elektrode in verschiedenen Vergrößerungen (6600 x, Maßstabsleiste = 10 µm) (B), und 11, 500 X, Skala bar = 5 µm) (C); Pfeile bezeichnen eingehalten Bakteriophagen). Bitte klicken Sie hier für eine größere Version dieser Figur.
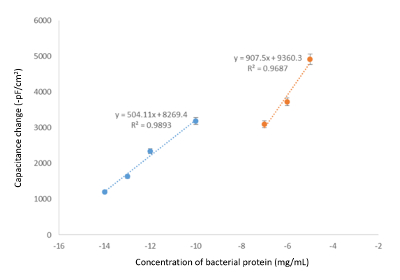
Abbildung 5. Wirkung des Puffers Zusammensetzung für Kalibrierung Graphen. Kalibrierung-Diagramm, das die Änderung in Kapazität vs. einer Proteinkonzentration unter optimalen Bedingungen (Running Puffer: 50 mM Tris-HCl, pH 7.4; Regeneration-Puffer: 25 mM Glycin-HCl, pH 2,5 einschließlich 50 mM Tween-20; Fördermenge: 100 µL/min, Probenvolumen: 250 µL; T: 25 ° C). Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 6. Repräsentative Eichkurve für große Biomakromoleküle. Kalibrierung-Diagramm, das zeigt die Änderung im Kapazität vs. Bakteriophagen Konzentration unter optimalen Bedingungen (Puffer läuft: 10 mM Phosphat, pH 7.4; Regeneration-Puffer: 25 mM Glycin-HCl, pH 2,5 einschließlich 50 mM Tween-20; Fördermenge: 100 µL/min, Probenvolumen: 250 µL; T: 25 ° C) Bitte klicken Sie hier für eine größere Version dieser Figur.
Diskussion
Wenn dieses Verfahren durchgeführt wird, gibt es einige wichtige Schritte, die berücksichtigt werden müssen, während im Anschluss an des Protokolls. Ein wichtiger Schritt ist die Reinigungsschritt mit sauren Piranha-Lösung. Schritt 2.1 darf nicht mehr als 10 Minuten sein. Template-Lösung für Schritt 1.4 darf 0,1 mg/mL nicht überschreiten, da diese Werte bereits optimiert wurden. Zyklische voltammetrische Scans müssen 15 Zyklen nicht überschreiten, um die optimale Dicke zu erhalten. Für Schritt 3.1.3 ist 1,5 µL einer optimierten Wert. Dieser Wert darf nicht höher für diese spezifische Art von Elektrode sein. Wenn die UV-Härtung System eine Leistung von 400 W hat, muss die Polymerisation für maximal 10-15 Minuten durchgeführt werden. Sobald die Lösung nach TEMED APS (Initiator) hinzugefügt wird, muss der nachfolgende Schritt sehr schnell durchgeführt werden, um sofortige Polymerisation (Schritt 3.2.5) zu vermeiden.
Einer der wichtigsten Schritte ist die Entfernung von der Vorlage-Stempel von der Oberfläche nach der UV-Polymerisation. Wenn dieser Schritt nicht ordnungsgemäß ausgeführt wird, gibt es ein Risiko, dass die Polymere Film auf der Oberfläche der Elektrode mit dem Stempel entfernt. Daher empfiehlt es sich, die Elektrode und der Protein-Stempel an der Spitze in einer Lösung von Wasser nach der Polymerisation eintauchen, dann entfernen die Stempel sehr langsam und vorsichtig von der Oberfläche (Schritt 3.1.6).
Basierend auf die Vorlage verwendet wird, können Änderungen in der Art und dem Verhältnis der Monomere verwendet (funktionelle Monomer und Vernetzer) im Hinblick auf die Erzeugung von höheren Empfindlichkeit bewertet werden. Dies muss empirisch ermittelt werden. Darüber hinaus beinhaltet die Affinität Bindung des Moleküls Vorlage in der Regel gleichzeitig mehrere verschiedene Interaktionen. Daher kann dies zu Problemen während der Regeneration Schritte führen. Wenn die gebundene Vorlage nicht richtig von der Oberfläche freigegeben ist, kann dies die Wiederverwendbarkeit von der Elektrode zur weiteren Analyse beeinflussen. Diese multipoint Affinitäten können auch aus der Bindung über schwächere Wechselwirkungen führen. In solchen Systemen kann unspezifische Bindung stattfinden, die die Selektivität des Systems16negativ beeinflussen können. Dies sind gewöhnliche und allgemeine, Grenzen der Methode.
Abgesehen von diesen spezifischen Einschränkungen gibt es viele wesentliche Vorteile der besprochenen Methode gegenüber bestehenden Methoden. RIAs, ELISAs und fluorometrisch Messungen sehr empfindlich sind, benötigen sie die Nutzung des beschrifteten Materials (Vorlage oder Detektor), während der Biosensor ist völlig markierungsfreie. Diese Methoden sind auch teuer und zeitaufwendig. Ein Biosensor-Ansatz ermöglicht schnelle, parallele Synthese von MIPs in verschiedenen Zusammensetzungen in der gleichen Zeit16. Da nur wenige Mikroliter Monomer-Lösung ist für die Vorbereitung erforderlich, eignet sich die Methode Verwendung teuer oder anderweitig beschränkt Monomere. Weitere, einzelne MIP-Elektroden können für ca. 80 Analysen ohne eine signifikante Abnahme der Leistung ist deutlich höher als andere vorhandene Methoden30verwendet werden. Bestehende Methoden leiden auch in unterschiedlichem Ausmaß, geringe Empfindlichkeit und Selektivität, während das beschriebene Verfahren für die Erkennung und Quantifizierung von Molekülen im Bereich von pM mit hoher Selektivität ermöglicht.
Aufgrund der Wirtschaftlichkeit, die einfache Bedienung des Instruments und die sensible Echtzeit-Erkennung in kurzer Zeit im Vergleich zu bestehenden Methoden der Biosensoren sind sehr vielversprechende Punkt der Pflege-Detection-Systeme unter Feldbedingungen; z.B.für die Umweltüberwachung und für Anwendungen in den Entwicklungsländern. In vielen Anwendungen in der Diagnose der Krankheit ist in Echtzeit, sensibel, selektive und schnelle Erkennung eines Biomarkers in einer komplexen Mischung wie Serum benötigt15,25. Biosensoren sind hier besser als bestehende Methoden, insbesondere aufgrund ihrer Robustheit und Empfindlichkeit. Speziell für den Nachweis von Infektionserregern gelten Bakteriophagen kürzlich als alternative Biorecognition Element für Biosensoren aufgrund ihrer Gastgeber Bakterien Spezifität33,34,35. Ersatz von Antikörpern mit Bakteriophagen ist sehr vielversprechend, um die Kosten zu senken und erhöhen die Stabilität noch weitere36. Ein solches System ermöglicht auch die Erkennung und Quantifizierung von spezifischen Phagen in der Umwelt und von klinischen Proben. Aufgrund der Prävalenz von Bakteriophagen und ihre Fähigkeit, Bakterien mit Antibiotika-Resistenz-Gene37,38transduzieren kann diese Methode wertvolle studieren die Verbreitung von resistenten Bakterien sein.
Offenlegungen
Die Autoren haben nichts preisgeben.
Danksagungen
Maria Baumgarten (IQ Biotechnologie Plattform, Infektion Medizin, Universität Lund) ist für die Durchführung und Bereitstellung von scanning Electron Mikrographen anerkannt. Diese Arbeit wurde unterstützt durch Zuschüsse aus The Swedish Research Council Formas (2017-00100) im Rahmen der dritten gemeinsamen Programmierung Initiative antimikrobielle Resistenz (JPIAMR) Bereitschaftsdienst "Übertragung Dynamik." Die Geldgeber hatten keine Rolle im Studiendesign, Interpretation, schreiben, Vorbereitung des Manuskripts, Beschluss einreichen oder Entscheidung, das Werk zu veröffentlichen.
Materialien
| Name | Company | Catalog Number | Comments |
| Glass Cover slips | ThermoFisher | 102222 | protein stamp |
| HCl | Sigma-Aldrich | H1758-500ML | cleaning |
| NaOH | Sigma-Aldrich | 72068-100ML | cleaning |
| Ultrasonic cleaner | Branson Ultrasonic | BRANSONIC M1800- E | cleaning |
| 3-amino-propyl-triethoxysilane (APTES) | Sigma-Aldrich | A3648-100ML | modification |
| EtOH | Sigma-Aldrich | 1009836010 | rinsing/cleaning |
| glutaraldehyde | Sigma-Aldrich | G5882-100ML | cross-linker |
| acetone | Sigma-Aldrich | 34850-1L-M | cleaning |
| H2SO4 | Sigma-Aldrich | 339741-100ML | piranha solution |
| H2O2 | Sigma-Aldrich | H1009-500ML | piranha solution |
| tyramine | Sigma-Aldrich | T90344-5G | modification |
| CompactStat | Ivium Technologies | CompactStat.h: 30mA@10V/3MHz | potentiostat |
| Platinum Counter Electrode Kit | Equilabrium | AFCTR5 | potentiostat |
| Reference Electrode | Equilabrium | RREF0021 | potentiostat |
| acryloyl chloride | EMD Millipore | 8.00826.0100 | modification |
| triethylamine | EMD Millipore | 8.08352.0100 | modification |
| toluene | Sigma-Aldrich | 244511-100ML | modification |
| N-hydroxymethyl acrylamide | Sigma-Aldrich | 245801-100G | functional monomer |
| poly ethylene glycol-400-dimethacrylate | Sigma-Aldrich | 409510-250ML | cross-linker |
| 2-Hydroxy-4′-(2-hydroxyethoxy)-2-methylpropiophenone | Sigma-Aldrich | 410896-50G | functional monomer |
| UV polymerizator | Dymax | Dymax 5000ECE | UV-polymerization |
| forceps | Sigma-Aldrich | Z168777-1EA | consumable |
| 1-dodecanethiol | Sigma-Aldrich | 471364-100ML | blocking agent |
| acrylamide | Sigma-Aldrich | A3553-100G | functional monomer |
| N-hydroxymethylacrylamide | Sigma-Aldrich | 245801-100G | functional monomer |
| N-isopropylacrylamide | Sigma-Aldrich | 415324-50G | functional monomer |
| methylenebisacrylamide | Sigma-Aldrich | 146072-500G | cross-linking monomer |
| N,N,N',N'-tetrametyhlethyldiamine (TEMED) | Sigma-Aldrich | T9281-25ML | catalyst |
| ammonium persulphate | Sigma-Aldrich | A3678-25G | initiator |
| Capacitive biosensor | CapSenze | Equipment | |
| Glycine | Merck | 1042011000 | regeneration buffer |
| Tween-20 | Sigma-Aldrich | P9416-50ML | regeneration buffer |
| Trizma base | Sigma-Aldrich | 93352-1KG | running buffer |
| Na2HPO4 • 2H2O | Calbiochem | 567547-1KG | running buffer |
| NaH2PO4 • 2H2O | Calbiochem | 567549-1KG | running buffer |
| DELPHI correlative light and electron microscope | Phenom-World | equipment | |
| Capacitive gold electrodes | CapSenze Biosystems | consumables | |
| 2,2'-azobis(2-methypropionitrile) | Sigma-Aldrich | 441090-25G | photo-initiator |
| CapSenze Smart Software | CapSenze Biosystems | software program |
Referenzen
- Lin, T. Y., Hu, C. H., Chou, T. C. Determination of albumin concentration by MIP-QCM sensor. Biosensors and Bioelectronics. 20 (1), 75-81 (2004).
- Berggren, C., Bjarnason, B., Johansson, G. Capacitive Biosensors. Electroanalysis. 13 (3), 173-180 (2001).
- Zhang, S., Garcia-D'Angeli, A., Brennan, J. P., Huo, Q. Predicting detection limits of enzyme-linked immunosorbent assay (ELISA) and bioanalytical techniques in general. The Analyst. 139 (2), 439-445 (2014).
- Mattiasson, B., Teeparuksapun, K., Hedström, M. Immunochemical binding assays for detection and quantification of trace impurities in biotechnological production. Trends in Biotechnology. 28 (1), 20-27 (2010).
- Limbut, W., Hedström, M., Thavarungkul, P., Kanatharana, P., Mattiasson, B. Capacitive biosensor for detection of endotoxin. Analytical and Bioanalytical Chemistry. 389 (2), 517-525 (2007).
- Ertürk, G., Berillo, D., Hedström, M., Mattiasson, B. Microcontact-BSA imprinted capacitive biosensor for real-time, sensitive and selective detection of BSA. Biotechnology Reports. 3, 65-72 (2014).
- Arshady, R., Mosbach, K. Synthesis of substrate-selective polymers by host-guest polymerization. Macromolecular Chemistry and Physics. 182 (2), 687-692 (1981).
- Andersson, L. I. Molecular imprinting: developments and applications in the analytical chemistry field. Journal of chromatography. B, Biomedical sciences and applications. 745 (1), 3-13 (2000).
- Li, X., Husson, S. M. Two-dimensional molecular imprinting approach to produce optical biosensor recognition elements. Langmuir: the ACS Journal of Surfaces and Colloids. 22 (23), 9658-9663 (2006).
- Alexander, C., Davidson, L., Hayes, W. Imprinted polymers: artificial molecular recognition materials with applications in synthesis and catalysis. Tetrahedron. 59 (12), 2025-2057 (2003).
- Kryscio, D. R., Peppas, N. A. Surface imprinted thin polymer film systems with selective recognition for bovine serum albumin. Analytica Chimica Acta. 718, 109-115 (2012).
- Inerowicz, H. D., Howell, S., Regnier, F. E., Reifenberger, R. Multiprotein immunoassay arrays fabricated by microcontact printing. Langmuir. 18 (13), 5263-5268 (2002).
- Lin, H. Y., Hsu, C. Y., Thomas, J. L., Wang, S. E., Chen, H. C., Chou, T. C. The microcontact imprinting of proteins: The effect of cross-linking monomers for lysozyme, ribonuclease A and myoglobin. Biosensors and Bioelectronics. 22 (4), 534-543 (2006).
- Liao, P. C., Tyan, Y. C., Wang, C. Y., Hsu, J. F., Chou, T. C., Lin, H. Y. Assessing the binding selectivity of molecularly imprinted polymer artificial antibodies by mass spectrometry-based profiling system. Journal of Biomedical Materials Research Part A. 91 (2), 597-604 (2009).
- Ertürk, G., Hedström, M., Tümer, M. A., Denizli, A., Mattiasson, B. Real-time prostate-specific antigen detection with prostate-specific antigen imprinted capacitive biosensors. Analytica Chimica Acta. 891, 120-129 (2015).
- Ertürk, G., Mattiasson, B. From imprinting to microcontact imprinting-A new tool to increase selectivity in analytical devices. Journal of Chromatography. B, Analytical Technologies in the Biomedical and Life Sciences. 1021, 30-44 (2016).
- Ertürk, G., Mattiasson, B. Molecular Imprinting: The Creation of Biorecognition Imprints on Biosensor Surfaces. Advanced Molecularly Imprinting Materials. , 523-560 (2017).
- Janczuk-Richter, M., et al. Long-period fiber grating sensor for detection of viruses. Sensors and Actuators B: Chemical. 250, 32-38 (2017).
- Kropinski, A. M., Mazzocco, A., Waddell, T. E., Lingohr, E., Johnson, R. P. Enumeration of bacteriophages by double agar overlay plaque assay. Methods in Molecular Biology. 501, 69-76 (2009).
- Meillan, M., et al. Self-assembled monolayer for AFM measurements of Tobacco Mosaic Virus (TMV) at the atomic level. RSC Adv. 4 (23), 11927 (2014).
- Ymeti, A., et al. Fast, ultrasensitive virus detection using a Young interferometer sensor. Nano Letters. 7 (2), 394-397 (2007).
- Wei, Y., Wong, L. P., Toh, C. -. S. Fuel cell virus sensor using virus capture within antibody-coated nanochannels. Analytical Chemistry. 85 (3), 1350-1357 (2013).
- Guliy, O. I., et al. Immunodetection of bacteriophages by a piezoelectric resonator with lateral electric field. Applied biochemistry and microbiology. 52 (4), 457-463 (2016).
- Reta, N., Michelmore, A., Saint, C., Prieto-Simón, B., Voelcker, N. H. Porous silicon membrane-modified electrodes for label-free voltammetric detection of MS2 bacteriophage. Biosensors & Bioelectronics. 80, 47-53 (2016).
- Ertürk, G., Özen, H., Tümer, M. A., Mattiasson, B., Denizli, A. Microcontact imprinting based surface plasmon resonance (SPR) biosensor for real-time and ultrasensitive detection of prostate specific antigen (PSA) from clinical samples. Sensors and Actuators B: Chemical. 224, 823-832 (2016).
- Lebogang, L., Mattiasson, B., Hedström, M. Capacitive sensing of microcystin variants of Microcystis aeruginosa using a gold immunoelectrode modified with antibodies, gold nanoparticles and polytyramine. Microchimica Acta. 181 (9-10), 1009-1017 (2014).
- Loyprasert, S., Hedström, M., Thavarungkul, P., Kanatharana, P., Mattiasson, B. Sub-attomolar detection of cholera toxin using a label-free capacitive immunosensor. Biosensors & Bioelectronics. 25 (8), 1977-1983 (2010).
- Teeparuksapun, K., Hedström, M., Wong, E. Y., Tang, S., Hewlett, I. K., Mattiasson, B. Ultrasensitive detection of HIV-1 p24 antigen using nanofunctionalized surfaces in a capacitive immunosensor. Analytical Chemistry. 82 (20), 8406-8411 (2010).
- Labib, M., Hedström, M., Amin, M., Mattiasson, B. A capacitive biosensor for detection of staphylococcal enterotoxin B. Analytical and Bioanalytical Chemistry. 393 (5), 1539-1544 (2009).
- Ertürk, G., Hedström, M., Mattiasson, B. A sensitive and real-time assay of trypsin by using molecular imprinting-based capacitive biosensor. Biosensors & Bioelectronics. 86, 557-565 (2016).
- Ertürk, G., Uzun, L., Tümer, M. A., Say, R., Denizli, A. Fab fragments imprinted SPR biosensor for real-time human immunoglobulin G detection. Biosensors & Bioelectronics. 28 (1), 97-104 (2011).
- El-Sharif, H. F., Phan, Q. T., Reddy, S. M. Enhanced selectivity of hydrogel-based molecularly imprinted polymers (HydroMIPs) following buffer conditioning. Analytica Chimica Acta. 809, 155-161 (2014).
- Wang, F., et al. Detection of Salmonella Typhimurium on Spinach Using Phage-Based Magnetoelastic Biosensors. Sensors (Basel, Switzerland). 17 (2), (2017).
- Chin, B. A., et al. Rapid detection of small quantities of specific bacteria using phage-based wireless biosensors. 2016 10th International Conference on Sensing Technology (ICST). , 1-5 (2016).
- Park, M. -. K., Chin, B. A. Novel Approach of a Phage-Based Magnetoelastic Biosensor for the Detection of Salmonella enterica serovar Typhimurium in Soil. Journal of Microbiology and Biotechnology. 26 (12), 2051-2059 (2016).
- Mack, J. D., Yehualaeshet, T., Park, M. -. K., Tameru, B., Samuel, T., Chin, B. A. Phage-Based Biosensor and Optimization of Surface Blocking Agents to Detect Salmonella typhimurium on Romaine Lettuce. Journal of food safety. 37 (2), 12299 (2017).
- Colomer-Lluch, M., Jofre, J., Muniesa, M. Antibiotic resistance genes in the bacteriophage DNA fraction of environmental samples. Plos One. 6 (3), 17549 (2011).
- Lood, R., Ertürk, G., Mattiasson, B. Revisiting antibiotic resistance spreading in wastewater treatment plants - bacteriophages as a much neglected potential transmission vehicle. Frontiers in microbiology. 8, (2017).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten