Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Hochgeschwindigkeits-Magnetpinzette für nanomechanische Messungen an kraftempfindlichen Elementen
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Hier beschreiben wir eine magnetische Hochgeschwindigkeitspinzette, die nanomechanische Messungen an kraftsensitiven Biomolekülen mit einer maximalen Rate von 1,2 kHz durchführt. Wir stellen seine Anwendung auf DNA-Haarnadeln und SNARE-Komplexe als Modellsysteme vor, aber es wird auch auf andere Moleküle anwendbar sein, die an mechanobiologischen Ereignissen beteiligt sind.
Zusammenfassung
Einzelmolekül-Magnetpinzetten (MTs) haben sich als leistungsstarke Werkzeuge zur gewaltsamen Abfrage von Biomolekülen wie Nukleinsäuren und Proteinen erwiesen und sind daher bereit, auf dem Gebiet der Mechanobiologie nützlich zu sein. Da die Methode in der Regel auf bildbasiertem Tracking von magnetischen Beads beruht, haben die Geschwindigkeitsbegrenzung bei der Aufnahme und Analyse von Bildern sowie die thermischen Fluktuationen der Beads ihre Anwendung bei der Beobachtung kleiner und schneller struktureller Veränderungen in Zielmolekülen lange Zeit behindert. Dieser Artikel beschreibt detaillierte Methoden für den Aufbau und Betrieb eines hochauflösenden MT-Aufbaus, der die nanoskalige Millisekundendynamik von Biomolekülen und ihren Komplexen auflösen kann. Als Anwendungsbeispiele werden Experimente mit DNA-Haarnadeln und SNARE-Komplexen (Membranfusionsmaschinerie) demonstriert, wobei der Schwerpunkt darauf liegt, wie deren transiente Zustände und Übergänge in Gegenwart von Kräften auf der Piconewton-Skala detektiert werden können. Wir gehen davon aus, dass Hochgeschwindigkeits-MTs weiterhin hochpräzise nanomechanische Messungen an Molekülen ermöglichen werden, die Kräfte in Zellen wahrnehmen, übertragen und erzeugen, und damit unser Verständnis der Mechanobiologie auf molekularer Ebene vertiefen werden.
Einleitung
Zellen nehmen mechanische Reize aktiv wahr und reagieren darauf. Dabei weisen viele Biomoleküle kraftabhängige Eigenschaften auf, die dynamische Strukturveränderungen ermöglichen. Bekannte Beispiele sind mechanosensitive Ionenkanäle und Zytoskelettelemente, die den Zellen wichtige mechanische Informationen aus ihrer Umgebung liefern.
Darüber hinaus können Moleküle, die eine einzigartige krafttragende Natur aufweisen, auch als mechanosensitiv im weiteren Sinne angesehen werden. Zum Beispiel spielen die lokale Bildung und das Schmelzen von Nukleinsäure-Duplexen sowie Strukturen höherer Ordnung wie G-Quadruplexe eine entscheidende Rolle bei der Replikation, Transkription, Rekombination und neuerdings auch bei der Genom-Editierung. Darüber hinaus erfüllen einige neuronale Proteine, die an der synaptischen Kommunikation beteiligt sind, ihre Funktionen, indem sie physikalische Kräfte erzeugen, die über das Niveau typischer intermolekularer Interaktionen hinausgehen. Unabhängig davon, welches Beispiel man untersucht, wird sich die Untersuchung der Nanomechanik der beteiligten Biomoleküle mit hoher raumzeitlicher Präzision als äußerst nützlich erweisen, um molekulare Mechanismen der damit verbundenen mechanobiologischen Prozesse aufzudecken 1,2,3.
Einzelmolekül-Kraftspektroskopie-Methoden haben sich als leistungsstarke Werkzeuge erwiesen, um die mechanischen Eigenschaften der Biomoleküle 2,4,5,6 zu untersuchen. Sie können strukturelle Veränderungen in Nukleinsäuren und Proteinen gleichzeitig mit Krafteinwirkung beobachten und so kraftabhängige Eigenschaften untersuchen. Zwei bekannte Aufbauten sind optische Pinzetten und magnetische Pinzetten (MTs), die mikrometergroße Kügelchen verwenden, um die Moleküle 5,6,7,8 zu manipulieren. In diesen Plattformen werden Polystyrol (für optische Pinzetten) oder magnetische Kügelchen (für MTs) über molekulare "Griffe", die typischerweise aus kurzen Fragmenten doppelsträngiger DNA (dsDNA) bestehen, an Zielmoleküle (z. B. Nukleinsäuren und Proteine) gebunden. Die Kügelchen werden dann bewegt, um Kraft auszuüben, und abgebildet, um ihre Positionen zu verfolgen, die über strukturelle Veränderungen in den Zielmolekülen berichten. Optische und magnetische Pinzetten sind in ihren Anwendungen weitgehend austauschbar, aber es gibt wichtige Unterschiede in ihren Ansätzen zur Kraftkontrolle. Optische Pinzetten sind intrinsische Positionsklemminstrumente, die Perlen in Position halten, wodurch die aufgebrachte Kraft schwankt, wenn sich die Form eines Zielkonstrukts ändert. Eine Verlängerungserhöhung, z. B. durch Entfaltung, lockert das Seil und verringert die Spannung und umgekehrt. Obwohl aktives Feedback implementiert werden kann, um die Kraft in optischen Pinzetten zu steuern, arbeiten MTs im Gegensatz dazu natürlich als Kraftklemmvorrichtung und nutzen die stabilen Fernfeld-Magnetkräfte von Permanentmagneten, die auch Umgebungsstörungen standhalten können.
Trotz ihrer langen Geschichte und ihres einfachen Designs sind MTs bei ihren Anwendungen für hochpräzise Messungen hinter optischen Pinzetten zurückgeblieben, was vor allem auf die technischen Herausforderungen bei der schnellen Perlenverfolgung zurückzuführen ist. In jüngster Zeit haben jedoch mehrere Gruppen gemeinsam eine vielschichtige Verbesserung sowohl der Hard- als auch der Software für MT-Instrumente 2,9,10,11,12,13,14,15,16,17,18,19 durchgeführt . In dieser Arbeit stellen wir ein Beispiel für einen solchen Aufbau vor, der bei 1,2 kHz läuft, und beschreiben, wie man damit nanomechanische Messungen an kraftempfindlichen Biomolekülen durchführen kann. Als Modellsysteme verwenden wir DNA-Haarnadeln und neuronale SNARE-Komplexe und untersuchen deren schnelle, strukturelle Veränderungen im Piconewton-Regime. DNA-Haarnadeln weisen einfache Zwei-Zustands-Übergänge in einem genau definierten Kraftbereich20,21 auf und dienen daher als Spielzeugmodelle, um die Leistung eines Pinzettenaufbaus zu überprüfen. Da sich die SNARE-Proteine zu einem kraftsensitiven Komplex zusammenfügen, der die Membranfusionantreibt 22, wurden sie auch ausgiebig mittels Einzelmolekül-Kraftspektroskopie untersucht 14,23,24,25. Es werden Standardansätze zur Analyse von Daten und zur Extraktion nützlicher Informationen über Thermodynamik und Kinetik vorgestellt. Wir hoffen, dass dieser Artikel die Einführung hochpräziser MTs in mechanobiologischen Studien erleichtern und die Leser motivieren kann, ihre eigenen kraftsensitiven Systeme zu erforschen.
Protokoll
Alle in diesem Protokoll beschriebenen Materialien und Ausrüstungen sind in der Materialtabelle aufgeführt. Die LabVIEW-Software für den Betrieb des unten beschriebenen Hochgeschwindigkeits-MT-Setups sowie die MATLAB-Skripte zur Analyse von Beispieldaten sind auf GitHub (https://github.com/ShonLab/Magnetic-Tweezers) hinterlegt und öffentlich verfügbar.
1. Apparatebau
HINWEIS: Das allgemeine Prinzip der Hochgeschwindigkeits-MT-Konstruktion ähnelt den herkömmlichen Standard-MT-Systemen, mit Ausnahme der Verwendung einer Hochgeschwindigkeits-CMOS-Kamera (Complementary Metal Oxide Semiconductor) und einer kohärenten Hochleistungslichtquelle (Abbildung 1). Weitere Beschreibungen der Standard-MT-Instrumente 5,26,27 finden Sie in anderen Quellen.
- Stellen Sie ein inverses Mikroskop auf einem schwingungsdämpfenden optischen Tisch auf. Installieren Sie eine Hochgeschwindigkeits-CMOS-Kamera und einen Framegrabber.
- Erstellen Sie einen Translationstisch für die Manipulation von Magneten in 3D. Montieren Sie einen motorisierten Lineartisch (>20 mm Verfahrweg) vertikal auf einem manuellen XY-Tisch.
Anmerkungen: Die vertikale Bewegung steuert die Kraft, während der XY-Tisch für die manuelle Ausrichtung der Magnete auf die optische Achse für die anfängliche Konstruktion des Aufbaus vorgesehen ist. - Installieren Sie einen rotierenden Schrittmotor und ein Riemen- und Flaschenzugsystem für rotierende Magnete.
Anmerkungen: Der Riemen überträgt die Drehbewegung zwischen der Motorwelle und den Magneten, die einige Zentimeter voneinander entfernt sind. Die Rotation von Magneten ist Teil der Translationsmanipulation. - Montieren Sie die Magnete. Verwenden Sie einen Acrylhalter (der bei einem Hersteller bestellt wurde; siehe ergänzende Abbildung S1), der zwei identische Magnete parallel mit einem genau definierten Abstand von 1 mm zwischen den Magneten fest aufnehmen kann (Abbildung 1B). Um die maximale Kraft zu nutzen, die mit einem bestimmten Magnetpaar erreicht werden kann, stellen Sie die vertikale Position des Translationstages so ein, dass die Unterseite der Magnete mit der Probenebene ausgerichtet ist, wenn sie in die niedrigste Position bewegt wird.
ANMERKUNG: Siehe Lipfert et al. für weitere Informationen über das Halterdesign und die Konfiguration der Magnete28. Die Höhe und Ausrichtung von Magneten wird von der LabVIEW-Software in Verbindung mit der Datenerfassung gesteuert. - Wenn Sie mit einer Objektivlinse mit geringer Vergrößerung betrachten, richten Sie die Magnete in der Mitte des Sichtfelds aus. Stellen Sie sicher, dass das Drehen der Magnete nicht zu einer großen Verschiebung der Mitte des Magnetpaares führt.
Anmerkungen: Wenn sich der Mittelpunkt zwischen den Magneten um die Drehachse dreht, ist es wahrscheinlich, dass die Magnete aufgrund eines unvollkommenen Halters nicht zentriert sind. Eine geringe Fehlausrichtung im Verhältnis zur Spaltgröße ist tolerierbar, da die Magnetdrehung nur zur Überprüfung der Halteseile und zum Aufbringen von Drehmomenten in bestimmten Anwendungen dient. - Installieren Sie eine Superlumineszenzdiode (SLD) für die Beleuchtung von Perlen. Führen Sie den Strahl durch den 1 mm großen Spalt zwischen den beiden Magneten. Stellen Sie sicher, dass der Strahl richtig kollimiert ist, um in den Spalt zu passen, und die Beleuchtung nicht von den Magneten abgeschattet wird.
- Installieren Sie einen Piezo-Linsenscanner am Objektivrevolver und montieren Sie ein 100-faches Ölimmersionsobjektiv (numerische Apertur [NA]: 1,45) für die Perlenverfolgung. Um mögliche Artefakte in den Tracking-Ergebnissen zu vermeiden, stellen Sie sicher, dass die Beleuchtung gleichmäßig beibehalten wird, wenn die Magnete bewegt werden. Stellen Sie abschließend die Lichtstärke auf die maximale Helligkeit ein, ohne Pixel zu sättigen.
HINWEIS: Für den Vergleich verschiedener Lichtquellen für die Hochgeschwindigkeitsverfolgung von Perlen siehe Dulin et al.29.
2. Kalibrierung der Magnetkraft
- Mittels Polymerase-Kettenreaktion (PCR; siehe Tabelle 1) werden 5 kbp dsDNA-Fragmente (unter Verwendung von Primer B, Primer Z_5k und λ-DNA) hergestellt, die an einem Ende mit Biotin (für die Oberflächenanhaftung) und am anderen Ende mit Azid (für die Bead-Anhaftung) markiert sind.
- Bereiten Sie gemäß Abschnitt 6 eine Durchflusszelle mit den 5 kbp-Molekülen vor.
- Identifizieren Sie gemäß Abschnitt 7 ein gutes Bead-Tether-Konstrukt, indem Sie seine Ausdehnung und Drehung überprüfen. Achten Sie insbesondere darauf, eine Wulst mit einer minimalen Rotationsbahn (d. h. mit einem Radius <200 nm) zu wählen, um den Wulsthöhenversatz aufgrund der dezentrierten Befestigung30,31 zu minimieren. Sobald ein guter Haltegurt identifiziert ist, beginnen Sie mit der Perlenverfolgung gemäß Abschnitt 9.
- Wenn der Aufbau neu ist, charakterisieren Sie sein Rauschen und seine Stabilität für zuverlässige hochauflösende Messungen. Platzieren Sie den Magneten ~3 mm von der Oberfläche der Durchflusszelle entfernt (um >10 pN aufzubringen und die Brownsche Bewegung einer Perle zu unterdrücken), verfolgen Sie die z-Position der Perle bei 1,2 kHz und berechnen Sie die Allan-Abweichung (AD) von der z-Koordinaten-Zeitreihe32,33 (Abbildung 2C). Prüfen Sie, ob AD-Werte von wenigen Nanometern im Hochgeschwindigkeitsbereich (<0,1 s) erreichbar sind und dass die differentielle Nachführung (magnetische Perlenposition relativ zu einer Referenzperle) die AD auf der längeren Zeitskala reduziert.
HINWEIS: Wir erhalten in der Regel einen AD von <3 nm bei maximaler Rate (1,2 kHz oder 0,83 ms Auflösung), und der AD nimmt mindestens bis zu 10 s ab, was eine minimale Drift impliziert. Andere haben ähnliche Werte bei ähnlichen Setups berichtet 9,10,11,12,34. - Wenn sich die Magnete in der Ruheposition (F ~ 0 pN) befinden, zeichnen Sie die x- und y-Koordinaten der angebundenen Perle bei 1,2 kHz auf. Zeichnen Sie die Position für einen ausreichend langen Zeitraum auf (d. h. ausreichend länger als die charakteristische Relaxationszeit der Fluktuation35), damit die Brownsche Bewegung ausreichend abgetastet wird.
HINWEIS: Hier verläuft die x-Richtung entlang der Richtung des Magnetfelds, während die Bewegung in y die transversale Bewegung senkrecht zum Feld darstellt. - Bewegen Sie die Magnete näher an die Durchflusszelle heran und wiederholen Sie die Messungen der Perlenposition, bis die Magnete sanft die Oberseite der Durchflusszelle berühren. Bewegen Sie sich in großen Schritten (z. B. 1-2 mm), wenn die Magnete mehr als 7 mm von der Probenebene entfernt sind (da die aufgebrachte Kraft im Fernfeld der Magnete langsam zunimmt), aber reduzieren Sie die Schrittweite allmählich (z. B. 0,1-0,5 mm), wenn sie sich nähern, um eine feinere Kalibrierung bei höheren Kraftniveaus zu erzielen (Abbildung 2B).
- Berechnen Sie die Kraft an jeder Magnetposition d mit einer der beiden alternativen Methoden (ein MATLAB-Skript "force calibration.m" mit beiden Methoden wird bereitgestellt; siehe Ergänzungsdatei 1).
- Messen Sie die Varianz der y-Koordinaten
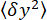 der Perle (Abbildung 2D) und die mittlere z-Position der Perle relativ zur niedrigsten Position
der Perle (Abbildung 2D) und die mittlere z-Position der Perle relativ zur niedrigsten Position 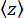 (Abbildung 2B, unten). Verwenden Sie dann Gleichung (1)7,27,36, um die Kraft abzuschätzen (mit einem festen Wulstradius R = 1.400 nm und thermischer Energie kRT = 4,11 pN∙nm):
(Abbildung 2B, unten). Verwenden Sie dann Gleichung (1)7,27,36, um die Kraft abzuschätzen (mit einem festen Wulstradius R = 1.400 nm und thermischer Energie kRT = 4,11 pN∙nm):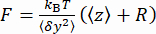 (1)
(1) - Alternativ können Sie die spektrale Leistungsdichte (PSD) der y-Koordinaten Sy berechnen (Abbildung 2E). Bestimmen Sie die aufgebrachte Kraft F, indem Sie ein Doppel-Lorentz-Modell37 mit Gleichung (2) an das gemessene Sy anpassen.
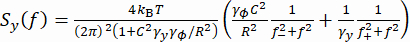 (2)
(2)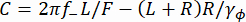 Hier ist , R der Wulstradius, γyund γ φ sind die Translations- bzw. Rotationswiderstandsbeiwerte (geschätzt aus der Stokes-Einstein-Gleichung), kRT ist die thermische Energie, f+ und f- sind zwei charakteristische Frequenzen, die mit Gleichung (3) erhalten werden.
Hier ist , R der Wulstradius, γyund γ φ sind die Translations- bzw. Rotationswiderstandsbeiwerte (geschätzt aus der Stokes-Einstein-Gleichung), kRT ist die thermische Energie, f+ und f- sind zwei charakteristische Frequenzen, die mit Gleichung (3) erhalten werden.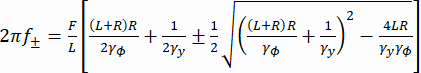 (3)
(3)
HINWEIS: Da die Halteverlängerung L eine Funktion der Kraft ist, die dem etablierten WLC-Modell (Worm-like Chain) folgt, lassen die obigen Ausdrücke F als einzigen Anpassungsparameter übrig (wir legen der Einfachheit halber fest, dass R 1.400 nm beträgt, da er über alle Kraftstufen hinweg geteilt wird und der genaue Wert die Ergebnisse nicht nennenswert beeinflusst). Bei Bedarf müssen Bewegungsunschärfe und Aliasing aus der kamerabasierten Bildaufnahme berücksichtigt werden38,39, aber dieser Effekt ist bei unseren Hochgeschwindigkeitsmessungen über 1 kHz mit 5 kbp Tethers vernachlässigbar.
- Messen Sie die Varianz der y-Koordinaten
- Wiederholen Sie die Schritte 2.4-2.7 für einige weitere Konstrukte. Untersuchen Sie drei bis fünf verschiedene Perlen, um die Kraftvariabilität zwischen den magnetischen Perlen zu ermitteln.
HINWEIS: Die Kraftvariation zwischen den verwendeten magnetischen Perlen sollte berücksichtigt werden, um die richtige Anzahl von Konstrukten für die Mittelwertbildung zu bestimmen. Diese Variabilität ist gering, kann aber selbst bei handelsüblichen Produkten zu einem Fehler von mehr als 1 pN in der gemessenen Kraft führen31. Für die meisten Anwendungen, bei denen die absolute Bestimmung der beteiligten Kräfte nicht entscheidend ist, ist die Mittelung der Kalibrierergebnisse von drei bis fünf Perlen in der Regel ausreichend. Ein alternativer Ansatz, um diese Variation zu berücksichtigen, besteht darin, die Kraft zu Beginn des Experiments mit einzelnen Seilen zu messen, was zeitaufwändig sein kann. Eine weitere Möglichkeit besteht darin, Haarnadelstrukturen einzubetten, die sich bei bekannten Kraftniveaus in jedem Konstrukt31 entpacken. - Zeichnen Sie die gemessene Kraft als Funktion des Magnetabstands auf und passen Sie mit Gleichung (4) eine doppelte Exponentialfunktion an die Daten an (Abbildung 2F).
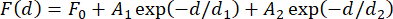 (4)
(4)
Hier sind F0 (Basislinie), A 1 und A 2 (Amplituden) sowie d 1 und d2 (Zerfallskonstanten) Anpassungsparameter. Stellen Sie sicher, dass die Kraftwerte aus den beiden Methoden sowie die resultierenden doppelt-exponentiellen Anpassungen weitgehend übereinstimmen (Abbildung 2F,G).
HINWEIS: Um zu bestätigen, dass die Kraftkalibrierung ordnungsgemäß durchgeführt wird, überprüfen Sie die Kraft-Dehnungs-Beziehung der untersuchten Konstrukte, indem Sie die Ausdehnung im Vergleich zur gemessenen Kraft darstellen. - Um den Wulsthöhenversatz z off zu korrigieren, der sich aus dem kraftabhängigen Verkippen der Magnetperlen30,31 ergibt, schätzen Sie zvom seitlichen Versatz xoff unter Berücksichtigung der Geometrie eines außermittigen Seils mit einem Wulstradius unter Verwendung von Gleichung (5) ab und wenden Sie die Werte auf die gemessenen Dehnungswerte an. Dieser Schritt ist im MATLAB-Skript "force calibration.m" (Zeilen 252-254) implementiert.
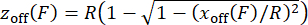 (5)
(5)
ANMERKUNG: Obwohl diese Korrektur kleine Änderungen an der Dehnung vornimmt, insbesondere bei den Perlen mit einem kleinen Rotationsradius (<200 nm), wirkt sich dieser Versatz oft kritisch auf die elastische Reaktion aus, wie in der Änderung von Abbildung 2H zu Abbildung 2I30,31 zu sehen ist. - Überprüfen Sie die Persistenzlänge Lp, indem Sie ein erweiterbares WLC-Modell mit Gleichung (6) an die Daten anpassen.
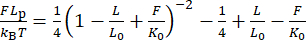 (6)
(6)
Dabei ist L 0 die Konturlänge (1,7 μm für 5 kbp) und K0 der Modul für die enthalpische Streckung.
HINWEIS: Obwohl angenommen wird, dass das L p der dsDNA in einem typischen Puffer wie der phosphatgepufferten Kochsalzlösung (PBS) 40-50 nm beträgt, unterschätzt die WLC-Formel, die auf kurze Moleküle (<5 kbp) angewendet wird, systematisch L p, da L0 um 31,40 abnimmt. Dies liegt daran, dass das klassische WLC-Modell von einem Polymer ausgeht, dessen Kettenlänge ausreichend länger ist als seine Persistenzlänge. Hier erhielten wir L p = 40 ± 3 nm für das 5 kbp-Konstrukt (Abbildung 2H), und die Verlängerungskorrektur ergab weiterhin ein homogenes K0 von 1.100 ± 200 pN (Abbildung 2I). Die Anwendung eines endlichen WLC-Modells 31,40 sowie einer Korrektur für Nicht-Gaußianität in der Erweiterungsverteilung 41 führt zu einer leichten Erhöhung von Lp. - Sobald die Kraftkalibrierung verifiziert ist, wenden Sie die erhaltenen Anpassungsparameter des Doppelexponentialmodells auf die mitgelieferte LabVIEW-Software (Supplemental File 2) an und warten Sie, bis die Software die aktuelle Kraft in Echtzeit aus den Motormesswerten (d. h. der Magnetposition) berechnet. Da kein analytischer Ausdruck für die Umkehrfunktion d(F) verfügbar ist, erstellen Sie eine Nachschlagetabelle von d versus F in 0,1 pN-Schritten durch numerische Schätzung von für die d Zielkraftniveaus. Speichern Sie diese Tabelle auch in der Software, um die Kraftsteuerung zu steuern.
3. Synthese von DNA-Haarnadeln
HINWEIS: DNA-Haarnadelkonstrukte für MT-Experimente werden durch PCR-Amplifikation einer 510 bp-Region in λ-DNA mit zwei kundenspezifischen Primern hergestellt, von denen einer eine Haarnadelstruktur an seinem 5′-Ende enthält (Abbildung 3A). Auf diese Weise wird ein Haarnadelmotiv an einem Ende des PCR-Produkts platziert.
- Bereiten Sie die Grundierungen vor.
- Forward Primer: Primer B_hp, der für die Anhaftung an Glasoberflächen mit 5′-Biotin markiert ist und an λ-DNA bindet. Dieser Primer enthält ein Haarnadelmotiv mit einem 8-bp-Stiel und einer 6-n-Schlaufe, 5′ zur λ-Bindungsregion.
- Reverse-Primer: Primer Z_hp, der für die magnetische Bead-Befestigung mit 5′-Azid markiert ist und 1 kbp vom Vorwärts-Primer entfernt an λ-DNA bindet.
- Richten Sie die PCR mit λ-DNA (Vorlage), nTaq-Polymerase und Standard-PCR-Bedingungen ein und führen Sie sie durch (siehe Tabelle 1). Reinigen Sie das Produkt mit einem handelsüblichen Reinigungsset.
- Messen Sie die DNA-Konzentration durch UV-Absorption bei 260 nm (A260) und führen Sie eine Agarose-Gelelektrophorese (2 % Gel) durch (siehe Tabelle 2), um die Produktgröße zu überprüfen. Eine typische Ausbeute beträgt ~35 μl einer ~600 nM Lösung.
4. Herstellung von SNARE-Proteinen
HINWEIS: Neuronale SNARE-Komplexe werden durch die Kombination von drei gereinigten Rattenproteinen zusammengesetzt, die aus E. coli exprimiert werden: VAMP2/Synaptobrevin-2, Syntaxin-1A und SNAP-25 (Abbildung 3B). Um ihre Assemblierung zu erleichtern, werden Syntaxin und SNAP-25 zusammen mit einem VAMP2-Fragment (ohne die N-terminale Region; als "ΔN-VAMP2" bezeichnet) zu einer Struktur namens "ΔN-Komplex" exprimiert und dann nach der Anheftung des DNA-Griffs mit VAMP2 in voller Länge gemischt, um vollständige Komplexe zu bilden.
- Herstellung von cDNA-haltigen Plasmiden für die Expression von SNARE-Proteinen (DNA-Sequenzen für alle Plasmide sind in der Materialtabelle angegeben).
- Präparation von 6×His-markiertem VAMP2 ohne Transmembrandomäne (2-97; L32C/I97C für Disulfidbindungen), kloniert in einen pET28a-Vektor.
- Präparation von Syntaxin-1A ohne Habc und Transmembrandomäne (191-267, I202C/I266C-Substitutionen für Disulfidbindungen) zusammen mit 6×His-markiertem ΔN-VAMP2 (49-96) in einen pETDuet-1-Vektor.
- Bereiten Sie die SNAP-25-Isoform b (2-206, alle C bis A) in voller Länge vor, die in einen pET28a-Vektor kloniert wurde. Dies wird für die Herstellung von ΔN-Komplexen verwendet.
- Bereiten Sie die 6×His-markierte SNAP-25-Isoform b (1-206, alle C bis A) in voller Länge vor, die in einen pET28a-Vektor kloniert wurde, um sie direkt in den MT-Assay-Puffer zu geben, um die SNARE-Komplexe nach der Entfaltung wieder zusammenzusetzen.
- Bereiten Sie zwei Röhrchen Rosetta (DE3) E. coli-Zellen vor. Transformation einer Gruppe mit VAMP2-Plasmiden (aus Schritt 4.1.1), eine Gruppe mit Syntaxin-1A/ΔN-VAMP2 und unmarkierten SNAP-25-Plasmiden (aus den Schritten 4.1.2 und 4.1.3) zur Expression des ΔN-Komplexes und die andere mit His-markierten SNAP-25-Plasmiden (aus Schritt 4.1.4).
- Die transformierten Zellen werden mit geeigneten Antibiotika (hier Kanamycin und Chloramphenicol für VAMP2 und His-markiertes SNAP-25; Kanamycin, Chloramphenicol und Ampicillin für den ΔN-Komplex) in Luria-Bertani-Brühe (LB) überführt. Bauen Sie sie bei 37 °C in einem Schüttelbrutschrank (220 U/min) an, bis die optische Dichte (OD) der Brühe 0,7-0,9 erreicht.
- Fügen Sie 1 mM Isopropyl β-d-1-thiogalactopyranosid (IPTG) hinzu, um die Proteinexpression zu induzieren, und inkubieren Sie die Zellen für 3-4 h bei 37 °C in einem Schüttelinkubator (220 U/min).
- Pelletieren Sie die Zellen, indem Sie die Kultur bei 4.500 × g für 15 Minuten bei 4 °C zentrifugieren.
- Bereiten Sie Puffer für die Proteinreinigung vor (siehe Tabelle 2).
- SNARE-exprimierende Zellpellets werden in 40 ml eiskaltem Lysepuffer suspendiert und die Zellen durch Beschallung auf Eis lysiert (15 % Amplitude, 5 s an und 5 s aus, insgesamt 30 Minuten).
- Zentrifugieren Sie das Lysat bei 15.000 × g für 30 min bei 4 °C, um unlösliche Stoffe zu entfernen.
- Führen Sie den Überstand durch eine Schwerkraftsäule, die mit 1 ml Ni-NTA-Harz gefüllt ist. Waschen Sie das Harz mit Waschpuffer A, dann mit Waschpuffer B und eluieren Sie die Proteine mit 10 ml Elutionspuffer.
- Entfernen Sie Tris(2-carboxyethyl)phosphin (TCEP) und Imidazol mit Hilfe einer Entsalzungssäule aus dem Eluenten (befolgen Sie die Anweisungen des Herstellers). Elute die Probe mit PBS.
- Konzentrieren Sie die Proteine mit Zentrifugalfiltern (10 kDa Cutoff) auf ~70 μM, während die Proteine in PBS erhalten bleiben (typischerweise 2 ml). Messen Sie die Proteinkonzentration entweder durch ultraviolette (UV) Absorption bei 280 nm (A280) oder mit dem Bradford-Assay.
- Bereiten Sie kleine Aliquots vor, frieren Sie sie in flüssigem Stickstoff ein und lagern Sie sie bis zur Verwendung bei -80 °C.
HINWEIS: Vollständige SNARE-Komplexe werden nach der Konjugation des ΔN-Komplexes an einem DNA-Griff (siehe unten) zusammengesetzt.
5. Befestigung von DNA-Griffen
HINWEIS: Zwei 510 bp dsDNA-Henkel, die an einem Ende primäre Amingruppen enthalten, werden zuerst durch PCR hergestellt, und die Amingruppen werden dann unter Verwendung eines bifunktionellen Vernetzers, SM(PEG)2, in Maleimidgruppen umgewandelt. Die beiden Henkel werden dann über ihre Cysteingruppen kovalent mit SNARE-Komplexen verknüpft, um eine ortsspezifische Konjugation zu ermöglichen (Abbildung 3B).
- Bereiten Sie Grundierungen vor.
- Vorbereiten von Vorwärts-Primern: Primer B (zur Amplifikation von Griff B), der für die Anhaftung an Glasoberflächen mit 5′-Biotin markiert ist und an λ-DNA bindet; Primer Z (zur Verstärkung von Griff Z), der für die magnetische Perlenbefestigung mit 5′-Azid markiert ist und die gleiche Reihenfolge wie Primer B hat.
- Bereiten Sie einen Reverse-Primer vor: Primer N (gemeinsam für Handle B und Handle Z), der für die Proteinkonjugation 5′-Amin-markiert ist und 510 bp vom Forward-Primer entfernt an λ-DNA bindet.
- Richten Sie zwei Sätze von PCR-Reaktionen (18 Röhrchen mit 200 μl-Reaktion für jeden Griff) mit λ-DNA (Vorlage), nTaq-Polymerase und Standard-PCR-Bedingungen ein und führen Sie sie durch (siehe Tabelle 1). Reinigen Sie das Produkt mit einem PCR-Reinigungskit und eluieren Sie jeden Griff mit 45 μl Reinstwasser. Verwenden Sie eine minimale Menge Wasser, um hohe Konzentrationen von Griffen für eine effektive Reaktion in späteren Schritten zu erhalten.
- Messen Sie die DNA-Konzentration mit A260. Die typische Ausbeute beträgt ~650 μl ~2 μM Lösung für jeden Griff. Halten Sie kleine Proben für eine spätere Überprüfung in der Agarose-Gelelektrophorese auseinander.
- Reagieren Sie jeden Griff (1 μM in PBS) mit 5 mM SM(PEG)2. Bei Raumtemperatur mit sanfter Rotation inkubieren. Verwenden Sie nach 1 Stunde ein DNA-Aufreinigungskit, um nicht umgesetztes SM(PEG)2 zu entfernen. Elute jeden Henkel mit 250 μL PBS, um ~2 μM Lösungen zu erhalten.
- Die Lösungen von Handle B und ΔN-Komplex werden im molaren Verhältnis 1:16 (z. B. 1 μM Handle B und 16 μM ΔN-Komplex) in PBS gemischt und 2 h bei Raumtemperatur unter Rühren inkubiert. Bewahren Sie eine kleine Probe für die Agarose-Gelelektrophorese auf.
- Fügen Sie eine Lösung von VAMP2 in einem 2,5-fachen molaren Überschuss über den im vorherigen Schritt verwendeten ΔN-Komplex hinzu. Die Mischung weitere 1 h bei Raumtemperatur unter Rühren inkubieren. In diesem Schritt werden vollständige SNARE-Komplexe zusammengesetzt.
- Freie Proteine durch Pufferaustausch mit frischem PBS und einem Zentrifugalfilter (100 kDa Cutoff) entfernen: 5 min bei 4 °C bei 14.000 × g zentrifugieren, mindestens 6x wiederholen und 15 min für den letzten Schleudergang laufen lassen. Messen Sie den Anstieg des A260/A280-Verhältnisses, um die Entfernung freier Proteine zu überwachen. Bewahren Sie eine kleine Probe für die Agarose-Gelelektrophorese auf.
- Geben Sie Griff Z in einem 15-fachen molaren Überschuss über Griff B in die Lösung. Halten Sie die Konzentration von Griff Z mindestens über 1 μM, um die Reaktion zu erleichtern. Die Mischung über Nacht bei 4 °C unter Rühren inkubieren.
- Verifizieren Sie die Zwischenprodukte (Griff B und seine Proteinkonjugate) und das Endprodukt (SNARE-Komplex mit zwei Griffen) durch Agarose-Gelelektrophorese (Abbildung 3B, Ausschnitt) (siehe Tabelle 2).
HINWEIS: Wenn die Proteine erfolgreich an Griff B angeheftet werden, wird eine Mobilitätsverschiebung festgestellt. Insbesondere kann die Bildung von vollständigen SNARE-Komplexen auf DNA-Griffen durch ihre Resistenz gegen Natriumdodecylsulfat (SDS) bestätigt werden, im Gegensatz zu ΔN-Komplexen, die in SDS zerlegt werden und nur Syntaxin an die DNA gebunden lassen (vgl. b und c in Abbildung 3B). - Bereiten Sie kleine Aliquots vor, frieren Sie sie in flüssigem Stickstoff ein und lagern Sie sie bis zur Verwendung bei -80 °C.
HINWEIS: Obwohl die endgültige Lösung nicht umgesetzte Griffe enthält, wird bei der Probenassemblierung in einer Durchflusszelle nur das gewünschte Konstrukt ausgewählt, das doppelt mit Biotin und Azid markiert ist.
6. Herstellung von Durchflusszellen
HINWEIS: Durchflusszellen für MT-Messungen bestehen aus zwei Glasdeckgläsern, die mit doppelseitigem Klebeband miteinander verbunden sind (Abbildung 3C). Ein Deckglas wird mit einer Mischung aus PEG und biotinyliertem Polyethylenglykol (PEG) beschichtet, um eine unspezifische Bindung zu vermeiden und eine spezifische Anbindung der Zielmoleküle über eine Biotin-NeutrAvidin-Bindung zu ermöglichen (Abbildung 3D). Anschließend werden die Materiallösungen für MT-Experimente mit Hilfe einer Spritzenpumpe sequenziell in eine Durchflusszelle infundiert (Abbildung 3C,D).
- Bereiten Sie zwei Glasdeckgläser vor, je eines für die Oberseite (24 mm × 50 mm, Stärke Nr. 1,5) und die Unterseite (24 mm × 60 mm, Dicke Nr. 1,5). Reinigen Sie die Deckgläser durch Beschallung in 1 M KOH für 30 Minuten. Spülen Sie die Deckgläser nach der Beschallung mit destilliertem Wasser ab und bewahren Sie sie bis zum nächsten Schritt in Wasser auf.
- PEGylieren Sie das untere Deckglas gemäß den veröffentlichten Protokollen42,43. Verwenden Sie N-[3-(Trimethoxysilyl)propyl]ethylendiamin zur Silanisierung und ein 1:100 (ww) Gemisch aus Biotin-PEG-SVA und mPEG-SVA in 100 mM Bicarbonatpuffer. Halten Sie die PEGylated-Deckgläser bei -20 °C trocken und lagern Sie sie einige Wochen.
- Nehmen Sie am Tag der Experimente die PEGylated-Deckgläser heraus und föhnen Sie sie mit einer Stickstoffpistole. Untersuchen Sie sie visuell auf Schmutz, um sicherzustellen, dass sie sauber sind.
- Um die Probenkanäle herzustellen, bereiten Sie ~2 mm breite Streifen doppelseitiges Klebeband vor und legen Sie vier Streifen auf ein unteres Deckglas (PEGylierte Oberfläche nach oben), parallel zueinander und durch ~5 mm voneinander getrennt (Abbildung 3C).
HINWEIS: Auf diese Weise können drei 5 mm breite Probenkanäle in einer einzigen Durchflusszelle erstellt werden. - Platzieren Sie ein oberes Deckglas in der Mitte des unteren Deckglases und lassen Sie an den kurzen Kanten ~5 mm Platz für Kanalein- und -auslässe. Drücken Sie vorsichtig mit einer Pinzette auf die Rückseite des oberen Deckglases, um die Kanäle fest zu verschließen.
- Um ein Einlassreservoir herzustellen, schneiden Sie den Rand einer 200-μl-Pipettenspitze ab. Schneiden Sie ~10 mm aus der breiteren Öffnung aus, um ~200 μl Lösung aufnehmen zu können. Machen Sie drei davon für die drei Strömungskanäle. Um die Auslässe zu konfigurieren, bereiten Sie drei Spritzennadeln vor, die in den Schlauch für die Spritzenpumpe passen.
- Kleben Sie die Behälter und Nadelnaben mit 5 Minuten Epoxidharz auf die Durchflusszelle. Stellen Sie sicher, dass eine vollständige Abdichtung gebildet ist, um Leckagen zu vermeiden, und dass die Kanäle nicht mit überschüssigem Klebstoff blockiert werden. Mindestens 30 Minuten trocknen lassen.
7. Montage von Bead-Tether-Konstruktionen
HINWEIS: Die Materiallösungen für MT-Experimente, einschließlich der Lösungen für Bead-Tether-Konstrukte, werden nacheinander mit Hilfe einer Spritzenpumpe in Durchflusszellen eingebracht (Abbildung 3C,D).
- Bereiten Sie magnetische Perlen vor. Nehmen Sie 5 mg M270-Epoxidkügelchen aus einer Stammlösung (~3,3 × 10 8 Kügelchen in 167,5 μl Dimethylformamid) und ersetzen Sie das Lösungsmittel durch Phosphatpuffer (siehe Tabelle 2) durch magnetische Trennungder Kügelchen.
- Die Kügelchen werden bei ~1,1 × 109 Kügelchen mL−1 in einem Phosphatpuffer mit 1 M Ammoniumsulfat hergestellt und mit 2 mM Dibenzocyclooctin (DBCO)-NH2 umgesetzt. Die Mischung 3 h auf einem rotierenden Mischer bei Raumtemperatur inkubieren. Waschen Sie die Kügelchen nach der Reaktion 3x mit frischem Phosphatpuffer, um nicht umgesetzte Moleküle zu entfernen.
HINWEIS: Die gewaschenen Perlen können vor der Verwendung ohne zusätzliche Rotation bei 4 °C mehrere Wochen gelagert werden. - Verbinden Sie eine Nadel am Ausgang des Durchflusszellenkanals mit der Spritzenpumpe mit einem Polyethylenschlauch. Gleichen Sie die Kanäle mit PBS aus.
- Die folgenden Lösungen werden nacheinander durch Ansaugen mit der Pumpe in den Kanal eingebracht: NeutrAvidin, Target-Konstrukte (DNA-Haarnadeln oder SNARE-Komplexe mit DNA-Griffen), Referenz-Polystyrol-Beads und DBCO-beschichtete Magnet-Beads. Vor der Verwendung die Perlenlösungen gründlich verwirbeln, um potenzielle Perlenaggregate zu dispergieren.
- Waschen Sie ungebundene Perlen mit einer Kraft von 0,1 pN ab.
HINWEIS: Die Aufbringung einer kleinen Aufwärtskraft erleichtert das Entfernen von ungebundenen Perlen und hilft, den Bruch von speziell gebundenen Perlen-Tether-Konstrukten zu vermeiden. - Für Experimente mit SNARE-Komplexen wird 1,5 μM SNAP-25 in den Endpuffer gegeben.
HINWEIS: Die freien SNAP-25-Moleküle können SNARE-Komplexe nach der Entfaltung wieder binden und ermöglichen wiederholte Messungen an einem einzelnen Komplex.
8. Identifizierung von Zielkonstrukten
- Suchen Sie auf der Oberfläche eines Flusszellenkanals nach den magnetischen Kügelchen, die von einzelnen Molekülen des Zielkonstrukts angebunden sind. Stellen Sie sicher, dass sich eine Referenzperle in der Nähe befindet.
- Drehen Sie eine Kandidatenperle und prüfen Sie, ob sie sich frei drehen lässt. Wenn die Perle von mehreren Molekülen angebunden ist, zeigt sie eine eingeschränkte Bewegung.
- Drehen Sie die Wulst für ein paar komplette Umdrehungen und ermitteln Sie den Rotationsradius (diese Funktion ist in der mitgelieferten Software implementiert). Wählen Sie vorzugsweise eine Perle mit kleinem Rotationsradius.
ANMERKUNG: Dieser Radius gibt an, wie stark die Perle von der Halteachse entfernt ist, was während der Perlen-Haltevorrichtung30,31 zufällig bestimmt wird. In allen Experimenten mildert die minimale Dezentrierung einer Perle viele Artefakte, die mit dem von uns verwendeten hohen Verhältnis von Perlenradius zu Halterdehnung verbunden sind. - Erhöhen Sie die Kraft von 0 auf 5 pN, um gute einfach angebundene Perlen zu identifizieren. Achten Sie auf eine große Änderung im Beugungsmuster einer Perle, die sich aus der Dehnung eines 1-kbp-Halteseils (oder der entsprechenden zwei 510-bp-Griffe) ergibt. Wenn sich das Beugungsmuster nicht signifikant ändert, verringern Sie die Kraft auf Null und suchen Sie nach einer anderen Kandidatenperle.
HINWEIS: Die ~300 nm Abhebung einer Perle kann aus den Rohbildern leicht bemerkt werden, ohne dass der Tracking-Prozess tatsächlich gestartet werden muss.
9. Wulstverfolgung für Dehnungsmessungen
HINWEIS: Die Nachverfolgung von Perlen erfolgt durch die Analyse von Perlenbildern in Echtzeit in der LabVIEW-Software, die in diesem Artikel enthalten ist. Die Tracking-Methode und ihre Varianten wurden in den meisten konventionellen MT-Systemen verwendet und werden in der bisherigen Literatur 2,5,7,26 erläutert. Durch die Messung der Position einer magnetischen Perle relativ zu einer festen Referenzperle (d. h. differentielles Tracking) werden die Positionsmessungen extrem robust gegenüber einer externen Störung.
- Sobald sich eine geeignete magnetische Perle zusammen mit einer Referenzperle befindet, klicken Sie auf die Schaltfläche Kalibrieren , um mit den Vorbereitungen für die Perlenverfolgung zu beginnen.
- Klicken Sie auf die Perlen im Bild, um die Positionen der Perlen zu definieren. Die Bilder werden dann auf Regions of Interest (ROIs) zugeschnitten (z. B. 150 x 150 Pixel für eine 3-μm-Perle) um die Perlen herum und dann weiter analysiert, um die genauen Perlenkoordinaten zu extrahieren.
- Warten Sie, bis die Magnetdrehung abgeschlossen ist. Bei diesem Verfahren werden die x- und y-Koordinaten der Wulst aufgezeichnet (durch Berechnung der 2D-Kreuzkorrelation 44 oder durch Verwendung der radialen Symmetrie 45 der Wulstbilder mit vergleichbarer Leistung), während die Magnete gedreht werden, um die außermittige Anbringung der Wulst31 zu dokumentieren.
- Warten Sie für das Tracking in z-Richtung, bis die Software eine Lookup-Tabelle mit Beugungsbildern der Beads in unterschiedlichen Abständen von der Fokusebene generiert hat. Dies geschieht, indem die Objektivlinse mit einem Piezoscanner in äquidistanten Schritten abgestuft wird und an jeder Position schwankungsgemittelte Perlenbilder aufgenommen werden. Anschließend werden die z-Koordinaten der Perlen in tatsächlichen Experimenten bestimmt, indem die Echtzeit-Perlenbilder mit der Lookup-Tabelle mit Interpolation7 verglichen werden.
- Wenn die Generierung der Lookup-Tabelle abgeschlossen ist, aktivieren Sie Tracking und Autofokus (drücken Sie die Tasten Track ? und AF? Schaltflächen) und klicken Sie auf die Schaltfläche Erfassen , um die Aufzeichnung der Perlenpositionen zu starten.
HINWEIS: Der Autofokus ist optional, wird aber empfohlen, um die Stufendrift in z während der Aufnahme zu korrigieren.
10. Anwendungsschemata erzwingen
- Kraft-Rampen-Experimente: Um die Kraft-Dehnungs-Beziehung des Konstrukts zu überprüfen, wenden Sie einen Kraftanstieg nach oben und unten bei konstanter Belastungsrate (± 1,0 pN s−1) an (Abbildung 4A). Wenden Sie z. B. drei Runden eines 0-20-0 pN-Zyklus an, um die Gesamtlänge der Konstruktion und die Kraft-Dehnungs-Kurve der Griffe zu überprüfen.
- Durch die Angabe der Tether-Parameter in der Software wird eine WLC-Kraft-Dehnungs-Kurve über die Messdaten gelegt und festgestellt, ob die Zielperle durch ein echtes Probenkonstrukt mit geeigneten DNA-Griffen angebunden ist. Verwenden Sie die bekannte Konturlänge (z. B. ~340 nm für 1 kbp dsDNA) und die WLC-Persistenzlänge (30-45 nm für kurz dsDNA31) des Konstrukts als Ausgangspunkt. Wenden Sie bei Bedarf die in Schritt 2.11 beschriebene Erweiterungskorrekturmethode an.
- Wenn das Konstrukt verifiziert ist, untersuchen Sie die Kraft-Dehnungs-Reaktion im Detail, um nach zusätzlicher Ausdehnung zu suchen, die sich aus den Zielmolekülen-Haarnadeln oder SNARE-Komplexen ergibt.
- Experimente mit konstanter Kraft: Variieren Sie die aufgebrachte Kraft schrittweise in diskreten Schritten, um die Kraftempfindlichkeit der Zielmoleküle zu untersuchen (Abbildung 4B).
Anmerkungen: MTs ermöglichen einfache und effektive Experimente mit konstanter Kraft, da die aufgebrachte Kraft konstant gehalten wird, wenn die Magnete stillgehalten werden.- Wenden Sie für DNA-Haarnadeln eine Kraft von 4-8 pN in Schritten von 0,2-0,5 pN an und messen Sie die Perlenposition für ~10 s auf jeder Kraftstufe.
- Wenden Sie bei SNARE-Komplexen eine Kraft von 14-16 pN in Schritten von 0,1-0,2 pN an und messen Sie die Wulstposition für ~10 s auf jeder Kraftstufe.
- Kraftsprung-Experimente: Beobachten Sie die Übergangsereignisse von SNARE-Komplexen.
HINWEIS: Kraftsprungexperimente beinhalten wie Experimente mit konstanter Kraft Änderungen der Kraftniveaus. Kraftsprünge nutzen jedoch abruptere Änderungen der ausgeübten Kraft, was die Überwachung von kraftausgelösten Ereignissen in den untersuchten Molekülen ermöglicht, wie z. B. einem plötzlichen Bruch von Proteinkomplexen. Da SNARE-Komplexe beispielsweise eine strukturelle Hysterese im Kraftzyklus23 aufweisen, ist es aufschlussreich, Kraftsprungexperimente durchzuführen und die Latenz bis zum Übergang zu messen (Abbildung 4C).- Entpacken: Ablösen eines VAMP2-Moleküls von einem intakten, ternären SNARE-Komplex, wobei ein binärer Komplex aus Syntaxin-1A und SNAP-25 übrig bleibt.
- Rezipieren: Reißverschluss des entpackten VAMP2-Moleküls zur Regeneration eines intakten SNARE-Komplexes.
- Entfaltung: Vollständige Demontage eines SNARE-Komplexes mit vollständiger Dissoziation von SNAP-25. Nur VAMP2- und Syntaxin-Moleküle verbleiben nach der Entfaltung im Konstrukt.
- Neu faltend: Regeneration eines SNARE-Komplexes nach Bindung eines freien SNAP-25-Moleküls aus dem Puffer.
- Bei 2 pN induzieren Sie die Assemblierung eines intakten SNARE-Komplexes, indem Sie (~30 s) auf die Assoziation eines freien SNAP25-Moleküls warten. Eine plötzliche Abnahme der Ausdehnung wird bei der Bildung eines SNARE-Komplexes beobachtet.
- Um das Entpacken zu beobachten, warten Sie einige Sekunden bei 10-12 pN und wechseln Sie dann abrupt zu 14-15 pN mit der maximal möglichen Motordrehzahl. Abhängig von der Zielkraft zeigt der SNARE-Komplex entweder einen reversiblen Übergang zwischen teilweise entzippten Zwischenprodukten (wie in Experimenten mit konstanter Kraft) oder einen ~25 nm Sprung in einen höheren, entzippten Zustand nach einer zufälligen Wartezeit (oder Latenz).
- Um das Rezipping zu beobachten, verringern Sie die Kraft unmittelbar nach dem Öffnen des Entpackens auf 10-12 pN. Auch hier zeigt der SNARE-Komplex nach einer zufälligen Latenz einen stochastischen Übergang in den unteren, mit Reißverschluss versehenen Zustand. Wenn die Entfaltung nach dem Entpacken stattgefunden hat, kann der Komplex nicht wieder komprimiert werden, da ein SNAP-25-Molekül fehlt.
- Um die sich entfaltenden Ereignisse zu beobachten, warten Sie nach dem Entpacken einen längeren Zeitraum, um eine weitere Zunahme der Ausdehnung (~2 nm) zu erkennen.
11. Datenanalyse
HINWEIS: Die Arten von Analysen, die mit MT-Daten durchgeführt werden können, hängen vom Zielsystem ab. Es gibt jedoch gängige Ansätze, um nützliche Informationen aus den jeweiligen Experimenten zu extrahieren, die in Abbildung 4 beschrieben sind. Alle Analysen werden mit MATLAB (R2021a) unter Verwendung der in diesem Artikel bereitgestellten benutzerdefinierten Codes durchgeführt. Diese Codes generieren Diagramme unter Verwendung der gleichen Daten, die in diesem Artikel vorgestellt wurden. Beachten Sie, dass Rohdaten aus dem 100-Hz-Tracking direkt für die Analyse verwendet wurden, während Daten aus dem 1,2-kHz-Tracking vor der Analyse in der Regel mediangefiltert wurden (mit einem gleitenden Fünf-Punkt-Fenster), um das Rauschen zu reduzieren (mit Ausnahme der Rauschanalyse).
- Kraft-Rampen-Experimente: Analysieren Sie die Kraft-Dehnungs-Beziehung (z. B. Elastizität von Polymeren) und übertragen Sie die Kraft, um Informationen über nanomechanische Eigenschaften zu extrahieren.
- Experimente mit konstanter Kraft: Analysieren Sie die Zustandspopulationen und die Verweildauer (oder Übergangsrate) als Funktion der Kraft, um strukturelle (z. B. am Übergang beteiligte Regionen), thermodynamische (z. B. freie Energiedifferenz) und kinetische (z. B. Energiebarriere) Parameter der Konformationsänderungen zu extrahieren.
- Kraftsprung-Experimente: Analysieren Sie die Bruchkinetik (z. B. Protein-Protein-Interaktionen und Rezeptor-Ligand-Bindung) oder die Lebensdauer von transienten Zwischenprodukten (z. B. Entfaltung von Biomolekülen), um die Stabilität von Zielmolekülen und ihre Zustände zu extrahieren.
- Stellvertretende Anwendungen sind die Analyse der Probendaten für DNA-Haarnadeln und SNARE-Komplexe:
- Zwei-Zustands-Übergänge einer DNA-Haarnadel: Entpackkraft, Öffnungsabstand, Kraftabhängigkeit der Populationsverschiebung sowie Zustandszuweisungs- und Übergangsratenmessungen mit einem versteckten Markov-Modell (HMM) (MATLAB-Codes zur Verfügung gestellt).
- Konformationsänderungen von SNARE-Komplexen: Entpackkraft, Kraftabhängigkeit von Zwischenzuständen und Entpacklatenz, Hysterese beim Rezipping und Entfaltungs-/Rückfaltungsverhalten.
ANMERKUNG: Kraftverlängerungsmodelle für DNA-Griffe, DNA-Haarnadeln und SNARE-Komplex-Konformationen sind in früheren Referenzen14,31 angegeben.
Ergebnisse
Kalibrierung erzwingen
Die Ergebnisse der beiden Kraftmessmethoden (seitliche Verschiebungsvarianz der Perlen und Leistungsspektrumanalyse) unterschieden sich um 0-2 pN (Abbildung 2G). Nach den Ergebnissen in Abbildung 2F können wir mit normalen Neodym-Magneten zuverlässig bis zu 30 pN erreichen.
Zwei-Zustands-Übergänge einer 8 bp DNA-Haarnadel
Wir untersuchten zunächst die Nanomechanik ein...
Diskussion
In dieser Arbeit haben wir einen Einzelmolekül-Kraftspektroskopie-Aufbau vorgestellt, der strukturelle Veränderungen von Biomolekülen mit hoher raumzeitlicher Präzision beobachten kann. Die verwendete Hochgeschwindigkeits-CMOS-Kamera erfasst 1.200 Bilder s−1 bei einer Auflösung von 1.280 x 1.024 und ermöglicht so eine Perlenverfolgung von 1,2 kHz. Die Geschwindigkeit der Messungen ist jedoch derzeit durch die Bead-Tracking-Software begrenzt, so dass der ROI bei Hochgeschwindigkeitsmessungen in der Rege...
Offenlegungen
Die Autoren haben keine Interessenkonflikte zu erklären.
Danksagungen
Diese Arbeit wurde durch einen Zuschuss der National Research Foundation of Korea (NRF) unterstützt, der von der koreanischen Regierung (MSIT) finanziert wird (NRF-2022R1C1C1012176, NRF-2021R1A4A1031754 und NRF-2021R1A6A10042944). S.-H.R. wurde durch den NRF-Zuschuss (2021R1C1C2009717) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Materials for construct synthesis | |||
| Agarose gel electrophoresis system | Advance | Mupid-2plus | |
| DNA ladder | Bioneer | D-1037 | |
| nTaq polymerase | Enzynomics | P050A | |
| PCR purification kit | LaboPass | CMR0112 | |
| PEGylated SMCC crosslinker / SM(PEG)2 | ThermoFisher Scientific | 22102 | For SNARE–DNA coupling |
| Primer B | Bioneer | 5'-Biotin/TCGCCACCATCATTTCCA-3' | For 5-kbp force calibration construct and DNA handles |
| Primer B_hp | IDT | 5'-Biotin/TTTTTTTTTTGTTCTCTATTT TTTTAGAGAAC /AP site/ /AP site/ TCGCCACCATCATTTCCA-3' | For hairpin construct |
| Primer N | Bioneer | 5'-C6Amine/CATGTGGGTGACGCGAAA-3' | For DNA handles |
| Primer Z | Bioneer | 5'-Azide/TCGCCACCATCATTTCCA-3' | For DNA handles |
| Primer Z_5k | Bioneer | 5'-Azide/TTAGAGAGTATGGGTATATGACA TCG-3' | For 5-kbp force calibration construct |
| Primer Z_hp | Bioneer | 5'-Azide/GTGGCAGCATGACACC-3' | For hairpin construct |
| SYBR Safe DNA Gel Stain | ThermoFisher Scientific | S33102 | |
| λ-DNA | Bioneer | D-2510 | Template strand for PCR |
| DNA sequences for SNARE proteins | |||
| 6×His-tagged SNAP-25b (2-206; capitalized) in pET28a | homemade | tggcgaatgggacgcgccctgtagcggcgca ttaagcgcggcgggtgtggtggttacgcgca gcgtgaccgctacacttgccagcgccctagc gcccgctcctttcgctttcttcccttccttt ctcgccacgttcgccggctttccccgtcaag ctctaaatcgggggctccctttagggttccg atttagtgctttacggcacctcgaccccaaa aaacttgattagggtgatggttcacgtagtg ggccatcgccctgatagacggtttttcgccc tttgacgttggagtccacgttctttaatagt ggactcttgttccaaactggaacaacactca accctatctcggtctattcttttgatttata agggattttgccgatttcggcctattggtta aaaaatgagctgatttaacaaaaatttaacg cgaattttaacaaaatattaacgtttacaat ttcaggtggcacttttcggggaaatgtgcgc ggaacccctatttgtttatttttctaaatac attcaaatatgtatccgctcatgaattaatt cttagaaaaactcatcgagcatcaaatgaaa ctgcaatttattcatatcaggattatcaata ccatatttttgaaaaagccgtttctgtaatg aaggagaaaactcaccgaggcagttccatag gatggcaagatcctggtatcggtctgcgatt ccgactcgtccaacatcaatacaacctatta atttcccctcgtcaaaaataaggttatcaag tgagaaatcaccatgagtgacgactgaatcc ggtgagaatggcaaaagtttatgcatttctt tccagacttgttcaacaggccagccattacg ctcgtcatcaaaatcactcgcatcaaccaaa ccgttattcattcgtgattgcgcctgagcga gacgaaatacgcgatcgctgttaaaaggaca attacaaacaggaatcgaatgcaaccggcgc aggaacactgccagcgcatcaacaatatttt cacctgaatcaggatattcttctaatacctg gaatgctgttttcccggggatcgcagtggtg agtaaccatgcatcatcaggagtacggataa aatgcttgatggtcggaagaggcataaattc cgtcagccagtttagtctgaccatctcatct gtaacatcattggcaacgctacctttgccat gtttcagaaacaactctggcgcatcgggctt cccatacaatcgatagattgtcgcacctgat tgcccgacattatcgcgagcccatttatacc catataaatcagcatccatgttggaatttaa tcgcggcctagagcaagacgtttcccgttga atatggctcataacaccccttgtattactgt ttatgtaagcagacagttttattgttcatga ccaaaatcccttaacgtgagttttcgttcca ctgagcgtcagaccccgtagaaaagatcaaa ggatcttcttgagatcctttttttctgcgcg taatctgctgcttgcaaacaaaaaaaccacc gctaccagcggtggtttgtttgccggatcaa gagctaccaactctttttccgaaggtaactg gcttcagcagagcgcagataccaaatactgt ccttctagtgtagccgtagttaggccaccac ttcaagaactctgtagcaccgcctacatacc tcgctctgctaatcctgttaccagtggctgc tgccagtggcgataagtcgtgtcttaccggg ttggactcaagacgatagttaccggataagg cgcagcggtcgggctgaacggggggttcgtg cacacagcccagcttggagcgaacgacctac accgaactgagatacctacagcgtgagctat gagaaagcgccacgcttcccgaagggagaaa ggcggacaggtatccggtaagcggcagggtc ggaacaggagagcgcacgagggagcttcca gggggaaacgcctggtatctttatagtcctgt cgggtttcgccacctctgacttgagcgtcga tttttgtgatgctcgtcaggggggcggagcc tatggaaaaacgccagcaacgcggccttttt acggttcctggccttttgctggccttttgct cacatgttctttcctgcgttatcccctgatt ctgtggataaccgtattaccgcctttgagtg agctgataccgctcgccgcagccgaacgacc gagcgcagcgagtcagtgagcgaggaagcgg aagagcgcctgatgcggtattttctccttac gcatctgtgcggtatttcacaccgcatatat ggtgcactctcagtacaatctgctctgatgc cgcatagttaagccagtatacactccgctat cgctacgtgactgggtcatggctgcgccccg acacccgccaacacccgctgacgcgccctga cgggcttgtctgctcccggcatccgcttaca gacaagctgtgaccgtctccgggagctgcat gtgtcagaggttttcaccgtcatcaccgaaa cgcgcgaggcagctgcggtaaagctcatcag cgtggtcgtgaagcgattcacagatgtctgc ctgttcatccgcgtccagctcgttgagtttc tccagaagcgttaatgtctggcttctgataa agcgggccatgttaagggcggttttttcctg tttggtcactgatgcctccgtgtaaggggga tttctgttcatgggggtaatgataccgatga aacgagagaggatgctcacgatacgggttac tgatgatgaacatgcccggttactggaacgt tgtgagggtaaacaactggcggtatggatgc ggcgggaccagagaaaaatcactcagggtc aatgccagcgcttcgttaatacagatgtaggt gttccacagggtagccagcagcatcctgcga tgcagatccggaacataatggtgcagggcgc tgacttccgcgtttccagactttacgaaaca cggaaaccgaagaccattcatgttgttgctc aggtcgcagacgttttgcagcagcagtcgct tcacgttcgctcgcgtatcggtgattcattc tgctaaccagtaaggcaaccccgccagccta gccgggtcctcaacgacaggagcacgatcat gcgcacccgtggggccgccatgccggcgata atggcctgcttctcgccgaaacgtttggtgg cgggaccagtgacgaaggcttgagcgagggc gtgcaagattccgaataccgcaagcgacagg ccgatcatcgtcgcgctccagcgaaagcggt cctcgccgaaaatgacccagagcgctgccgg cacctgtcctacgagttgcatgataaagaag acagtcataagtgcggcgacgatagtcatgc cccgcgcccaccggaaggagctgactgggtt gaaggctctcaagggcatcggtcgagatccc ggtgcctaatgagtgagctaacttacattaa ttgcgttgcgctcactgcccgctttccagtc gggaaacctgtcgtgccagctgcattaatga atcggccaacgcgcggggagaggcggtttgc gtattgggcgccagggtggtttttcttttca ccagtgagacgggcaacagctgattgccctt caccgcctggccctgagagagttgcagcaag cggtccacgctggtttgccccagcaggcgaa aatcctgtttgatggtggttaacggcgggat ataacatgagctgtcttcggtatcgtcgtat cccactaccgagatatccgcaccaacgcgca gcccggactcggtaatggcgcgcattgcgcc cagcgccatctgatcgttggcaaccagcatc gcagtgggaacgatgccctcattcagcattt gcatggtttgttgaaaaccggacatggcact ccagtcgccttcccgttccgctatcggctga atttgattgcgagtgagatatttatgccagc cagccagacgcagacgcgccgagacagaa cttaatgggcccgctaacagcgcgatttgctgg tgacccaatgcgaccagatgctccacgccca gtcgcgtaccgtcttcatgggagaaaataat actgttgatgggtgtctggtcagagacatca agaaataacgccggaacattagtgcaggcag cttccacagcaatggcatcctggtcatccag cggatagttaatgatcagcccactgacgcgt tgcgcgagaagattgtgcaccgccgctttac aggcttcgacgccgcttcgttctaccatcga caccaccacgctggcacccagttgatcggcg cgagatttaatcgccgcgacaatttgcgacg gcgcgtgcagggccagactggaggtggcaac gccaatcagcaacgactgtttgcccgccagt tgttgtgccacgcggttgggaatgtaattca gctccgccatcgccgcttccactttttcccg cgttttcgcagaaacgtggctggcctggttc accacgcgggaaacggtctgataagagacac cggcatactctgcgacatcgtataacgttac tggtttcacattcaccaccctgaattgactc tcttccgggcgctatcatgccataccgcgaa aggttttgcgccattcgatggtgtccgggat ctcgacgctctcccttatgcgactcctgcat taggaagcagcccagtagtaggttgaggccg ttgagcaccgccgccgcaaggaatggtgcat gcaaggagatggcgcccaacagtcccccggc cacggggcctgccaccatacccacgccgaaa caagcgctcatgagcccgaagtggcgagccc gatcttccccatcggtgatgtcggcgatata ggcgccagcaaccgcacctgtggcgccggtg atgccggccacgatgcgtccggcgtagagga tcgagatctcgatcccgcgaaattaatacga ctcactataggggaattgtgagcggataaca attcccctctagaaataattttgtttaactt taagaaggagatataccATGGGCAGC AGCCATCATCATCATCATCACA GCAGCGGCCTGGTGCCGCGC GGCAGCCATACTAGCGGAGAT ATCGCCGAGGACGCAGACAT GCGCAATGAGCTGGAGGAGA TGCAGAGGAGGGCTGACCAG CTGGCTGATGAGTCCCTGGA AAGCACCCGTCGCATGCTGC AGCTGGTTGAAGAGAGTAAA GATGCTGGCATCAGGACTTT GGTTATGTTGGATGAGCAAG GCGAACAACTGGAACGCATT GAGGAAGGGATGGACCAAAT CAATAAGGACATGAAAGAAG CAGAAAAGAATTTGACGGAC CTAGGAAAATTCGCCGGCCT TGCCGTGGCCCCCGCCAAC AAGCTTAAATCCAGTGATGC TTACAAAAAAGCCTGGGGC AATAATCAGGATGGAGTAGT GGCCAGCCAGCCTGCCCG TGTGGTGGATGAACGGGAG CAGATGGCCATCAGTGGTG GCTTCATCCGCAGGGTAAC AAATGATGCCCGGGAAAAT GAGATGGATGAGAACCTG GAGCAGGTGAGCGGCATC ATCGGAAACCTCCGCCAC ATGGCTCTAGACATGGGCA ATGAGATTGACACCCAGA ATCGCCAGATCGACAGGA TCATGGAGAAGGCTGATT CCAACAAAACCAGAATTG ATGAAGCCAACCAACGTG CAACAAAGATGCTGGGAA GTGGTTAAggatccgaattcgag ctccgtcgacaagcttgcggccgcactc gagcaccaccaccaccaccactgagat ccggctgctaacaaagcccgaaagga agctgagttggctgctgccaccgctgag caataactagcataaccccttggggcct ctaaacgggtcttgaggggttttttgctga aaggaggaactatatccggat | |
| 6×His-tagged VAMP2 (2-97, L32C/I97C; capitalized) in pET28a | homemade | tggcgaatgggacgcgccctgtagcggcgca ttaagcgcggcgggtgtggtggttacgcgca gcgtgaccgctacacttgccagcgccctagc gcccgctcctttcgctttcttcccttccttt ctcgccacgttcgccggctttccccgtcaag ctctaaatcgggggctccctttagggttccg atttagtgctttacggcacctcgaccccaaa aaacttgattagggtgatggttcacgtagtg ggccatcgccctgatagacggtttttcgccc tttgacgttggagtccacgttctttaatagt ggactcttgttccaaactggaacaacactca accctatctcggtctattcttttgatttata agggattttgccgatttcggcctattggtta aaaaatgagctgatttaacaaaaatttaacg cgaattttaacaaaatattaacgtttacaat ttcaggtggcacttttcggggaaatgtgcgc ggaacccctatttgtttatttttctaaatac attcaaatatgtatccgctcatgaattaatt cttagaaaaactcatcgagcatcaaatgaaa ctgcaatttattcatatcaggattatcaata ccatatttttgaaaaagccgtttctgtaatg aaggagaaaactcaccgaggcagttccatag gatggcaagatcctggtatcggtctgcgatt ccgactcgtccaacatcaatacaacctatta atttcccctcgtcaaaaataaggttatcaag tgagaaatcaccatgagtgacgactgaatcc ggtgagaatggcaaaagtttatgcatttctt tccagacttgttcaacaggccagccattacg ctcgtcatcaaaatcactcgcatcaaccaaa ccgttattcattcgtgattgcgcctgagcga gacgaaatacgcgatcgctgttaaaaggaca attacaaacaggaatcgaatgcaaccggcgc aggaacactgccagcgcatcaacaatatttt cacctgaatcaggatattcttctaatacctg gaatgctgttttcccggggatcgcagtggtg agtaaccatgcatcatcaggagtacggataa aatgcttgatggtcggaagaggcataaattc cgtcagccagtttagtctgaccatctcatct gtaacatcattggcaacgctacctttgccat gtttcagaaacaactctggcgcatcgggctt cccatacaatcgatagattgtcgcacctgat tgcccgacattatcgcgagcccatttatacc catataaatcagcatccatgttggaatttaa tcgcggcctagagcaagacgtttcccgttga atatggctcataacaccccttgtattactgt ttatgtaagcagacagttttattgttcatga ccaaaatcccttaacgtgagttttcgttcca ctgagcgtcagaccccgtagaaaagatcaaa ggatcttcttgagatcctttttttctgcgcg taatctgctgcttgcaaacaaaaaaaccacc gctaccagcggtggtttgtttgccggatcaa gagctaccaactctttttccgaaggtaactg gcttcagcagagcgcagataccaaatactgt ccttctagtgtagccgtagttaggccaccac ttcaagaactctgtagcaccgcctacatacc tcgctctgctaatcctgttaccagtggctgc tgccagtggcgataagtcgtgtcttaccggg ttggactcaagacgatagttaccggataagg cgcagcggtcgggctgaacggggggttcgtg cacacagcccagcttggagcgaacgacctac accgaactgagatacctacagcgtgagctatg agaaagcgccacgcttcccgaagggagaaa ggcggacaggtatccggtaagcggcagggtc ggaacaggagagcgcacgagggagcttcca gggggaaacgcctggtatctttatagtcctgt cgggtttcgccacctctgacttgagcgtcga tttttgtgatgctcgtcaggggggcggagcc tatggaaaaacgccagcaacgcggccttttt acggttcctggccttttgctggccttttgct cacatgttctttcctgcgttatcccctgatt ctgtggataaccgtattaccgcctttgagtg agctgataccgctcgccgcagccgaacgacc gagcgcagcgagtcagtgagcgaggaagc ggaagagcgcctgatgcggtattttctccttac gcatctgtgcggtatttcacaccgcatatat ggtgcactctcagtacaatctgctctgatgc cgcatagttaagccagtatacactccgctat cgctacgtgactgggtcatggctgcgccccg acacccgccaacacccgctgacgcgccctga cgggcttgtctgctcccggcatccgcttaca gacaagctgtgaccgtctccgggagctgcat gtgtcagaggttttcaccgtcatcaccgaaa cgcgcgaggcagctgcggtaaagctcatcag cgtggtcgtgaagcgattcacagatgtctgc ctgttcatccgcgtccagctcgttgagtttc tccagaagcgttaatgtctggcttctgataa agcgggccatgttaagggcggttttttcctg tttggtcactgatgcctccgtgtaaggggga tttctgttcatgggggtaatgataccgatga aacgagagaggatgctcacgatacgggttac tgatgatgaacatgcccggttactggaacgt tgtgagggtaaacaactggcggtatggatgc ggcgggaccagagaaaaatcactcagggtc aatgccagcgcttcgttaatacagatgtaggt gttccacagggtagccagcagcatcctgcga tgcagatccggaacataatggtgcagggcgc tgacttccgcgtttccagactttacgaaaca cggaaaccgaagaccattcatgttgttgctc aggtcgcagacgttttgcagcagcagtcgct tcacgttcgctcgcgtatcggtgattcattc tgctaaccagtaaggcaaccccgccagccta gccgggtcctcaacgacaggagcacgatcat gcgcacccgtggggccgccatgccggcgata atggcctgcttctcgccgaaacgtttggtgg cgggaccagtgacgaaggcttgagcgagggc gtgcaagattccgaataccgcaagcgacagg ccgatcatcgtcgcgctccagcgaaagcggt cctcgccgaaaatgacccagagcgctgccgg cacctgtcctacgagttgcatgataaagaag acagtcataagtgcggcgacgatagtcatgc cccgcgcccaccggaaggagctgactgggtt gaaggctctcaagggcatcggtcgagatccc ggtgcctaatgagtgagctaacttacattaa ttgcgttgcgctcactgcccgctttccagtc gggaaacctgtcgtgccagctgcattaatga atcggccaacgcgcggggagaggcggtttgc gtattgggcgccagggtggtttttcttttca ccagtgagacgggcaacagctgattgccctt caccgcctggccctgagagagttgcagcaag cggtccacgctggtttgccccagcaggcgaa aatcctgtttgatggtggttaacggcgggat ataacatgagctgtcttcggtatcgtcgtat cccactaccgagatatccgcaccaacgcgca gcccggactcggtaatggcgcgcattgcgcc cagcgccatctgatcgttggcaaccagcatc gcagtgggaacgatgccctcattcagcattt gcatggtttgttgaaaaccggacatggcact ccagtcgccttcccgttccgctatcggctga atttgattgcgagtgagatatttatgccagc cagccagacgcagacgcgccgagacagaa cttaatgggcccgctaacagcgcgatttgctgg tgacccaatgcgaccagatgctccacgccca gtcgcgtaccgtcttcatgggagaaaataat actgttgatgggtgtctggtcagagacatca agaaataacgccggaacattagtgcaggcag cttccacagcaatggcatcctggtcatccag cggatagttaatgatcagcccactgacgcgt tgcgcgagaagattgtgcaccgccgctttac aggcttcgacgccgcttcgttctaccatcga caccaccacgctggcacccagttgatcggcg cgagatttaatcgccgcgacaatttgcgacg gcgcgtgcagggccagactggaggtggcaac gccaatcagcaacgactgtttgcccgccagt tgttgtgccacgcggttgggaatgtaattca gctccgccatcgccgcttccactttttcccg cgttttcgcagaaacgtggctggcctggttc accacgcgggaaacggtctgataagagacac cggcatactctgcgacatcgtataacgttac tggtttcacattcaccaccctgaattgactc tcttccgggcgctatcatgccataccgcgaa aggttttgcgccattcgatggtgtccgggat ctcgacgctctcccttatgcgactcctgcat taggaagcagcccagtagtaggttgaggccg ttgagcaccgccgccgcaaggaatggtgcat gcaaggagatggcgcccaacagtcccccggc cacggggcctgccaccatacccacgccgaaa caagcgctcatgagcccgaagtggcgagccc gatcttccccatcggtgatgtcggcgatata ggcgccagcaaccgcacctgtggcgccggtg atgccggccacgatgcgtccggcgtagagga tcgagatctcgatcccgcgaaattaatacga ctcactataggggaattgtgagcggataaca attcccctctagaaataattttgtttaactt taagaaggagatataccATGGGCAGC AGCCATCATCATCATCATCAC AGCAGCGGCCTGGTGCCGC GCGGCAGCCATATGGCAGAT CTCTCGGCTACCGCTGCCAC CGTCCCGCCTGCCGCCCCG GCCGGCGAGGGTGGCCCCC CTGCACCTCCTCCAAATCTTA CCAGTAACAGGAGATGCCAG CAGACCCAGGCCCAGGTGG ATGAGGTGGTGGACATCATG AGGGTGAATGTGGACAAGGT CCTGGAGCGAGACCAGAAG CTATCGGAACTGGATGATCG CGCAGATGCCCTCCAGGCA GGGGCCTCCCAGTTTGAAA CAAGTGCAGCCAAGCTCAA GCGCAAATACTGGTGGAAA AACCTCAAGATGATGTGCTA Aggatccgaattcgagctccgtcg acaagcttgcggccgcactcgagcaccacca ccaccaccactgagatccggctgctaacaaa gcccgaaaggaagctgagttggctgctgcca ccgctgagcaataactagcataaccccttgg ggcctctaaacgggtcttgaggggttttttg ctgaaaggaggaactatatccggat | |
| 6×His-tagged ΔN-VAMP2 (49–96; capitalized) and Syntaxin-1A (191–267, I202C/I266C; capitalized) in pETDuet-1 | homemade | ggggaattgtgagcggataacaattcccctc tagaaataattttgtttaactttaagaagga gatataccATGGGCAGCAGCCATCA TCATCATCATCACAGCAGCGG CCTGGAAGTTCTGTTCCAGGG GCCCGGTAATGTGGACAAGGT CCTGGAGCGAGACCAGAAGCT ATCGGAACTGGATGATCGCGC AGATGCCCTCCAGGCAGGGGC CTCCCAGTTTGAAACAAGTGC AGCCAAGCTCAAGCGCAAATAC TGGTGGAAAAACCTCAAGATGAT GTAAgcggccgcataatgcttaagtcgaaca gaaagtaatcgtattgtacacggccgcataa tcgaaattaatacgactcactataggggaat tgtgagcggataacaattccccatcttagta tattagttaagtataagaaggagatatacat ATGGCCCTCAGTGAGATCGAGA CCAGGCACAGTGAGTGCATC AAGTTGGAGAACAGCATCCG GGAGCTACACGATATGTTCAT GGACATGGCCATGCTGGTGG AGAGCCAGGGGGAGATGATT GACAGGATCGAGTACAATGTG GAACACGCTGTGGACTACGTG GAGAGGGCCGTGTCTGACACC AAGAAGGCCGTCAAGTACCAG AGCAAGGCACGCAGGAAGAA GTGCATGATCTAActcgagtc tggtaaagaaaccgctgctgcgaaatttgaa cgccagcacatggactcgtctactagcgcag cttaattaacctaggctgctgccaccgctga gcaataactagcataaccccttggggcctct aaacgggtcttgaggggttttttgctgaaag gaggaactatatccggattggcgaatgggac gcgccctgtagcggcgcattaagcgcggcgg gtgtggtggttacgcgcagcgtgaccgctac acttgccagcgccctagcgcccgctcctttc gctttcttcccttcctttctcgccacgttcg ccggctttccccgtcaagctctaaatcgggg gctccctttagggttccgatttagtgcttta cggcacctcgaccccaaaaaacttgattagg gtgatggttcacgtagtgggccatcgccctg atagacggtttttcgccctttgacgttggag tccacgttctttaatagtggactcttgttcc aaactggaacaacactcaaccctatctcggt ctattcttttgatttataagggattttgccg atttcggcctattggttaaaaaatgagctga tttaacaaaaatttaacgcgaattttaacaa aatattaacgtttacaatttctggcggcacg atggcatgagattatcaaaaaggatcttcac ctagatccttttaaattaaaaatgaagtttt aaatcaatctaaagtatatatgagtaaactt ggtctgacagttaccaatgcttaatcagtga ggcacctatctcagcgatctgtctatttcgt tcatccatagttgcctgactccccgtcgtgt agataactacgatacgggagggcttaccatc tggccccagtgctgcaatgataccgcgagac ccacgctcaccggctccagatttatcagcaa taaaccagccagccggaagggccgagcgca gaagtggtcctgcaactttatccgcctccatc cagtctattaattgttgccgggaagctagag taagtagttcgccagttaatagtttgcgcaa cgttgttgccattgctacaggcatcgtggtg tcacgctcgtcgtttggtatggcttcattca gctccggttcccaacgatcaaggcgagttac atgatcccccatgttgtgcaaaaaagcggtt agctccttcggtcctccgatcgttgtcagaa gtaagttggccgcagtgttatcactcatggt tatggcagcactgcataattctcttactgtc atgccatccgtaagatgcttttctgtgactg gtgagtactcaaccaagtcattctgagaata gtgtatgcggcgaccgagttgctcttgcccg gcgtcaatacgggataataccgcgccacata gcagaactttaaaagtgctcatcattggaaa acgttcttcggggcgaaaactctcaaggatc ttaccgctgttgagatccagttcgatgtaac ccactcgtgcacccaactgatcttcagcatc ttttactttcaccagcgtttctgggtgagcaaa aacaggaaggcaaaatgccgcaaaaaagg gaataagggcgacacggaaatgttgaatact catactcttcctttttcaatcatgattgaag catttatcagggttattgtctcatgagcgga tacatatttgaatgtatttagaaaaataaac aaataggtcatgaccaaaatcccttaacgtg agttttcgttccactgagcgtcagaccccgt agaaaagatcaaaggatcttcttgagatcct ttttttctgcgcgtaatctgctgcttgcaaa caaaaaaaccaccgctaccagcggtggtttg tttgccggatcaagagctaccaactcttttt ccgaaggtaactggcttcagcagagcgcaga taccaaatactgtccttctagtgtagccgta gttaggccaccacttcaagaactctgtagca ccgcctacatacctcgctctgctaatcctgt taccagtggctgctgccagtggcgataagtc gtgtcttaccgggttggactcaagacgatag ttaccggataaggcgcagcggtcgggctgaa cggggggttcgtgcacacagcccagcttgga gcgaacgacctacaccgaactgagataccta cagcgtgagctatgagaaagcgccacgcttccc gaagggagaaaggcggacaggtatccggta agcggcagggtcggaacaggagagcgcac gagggagcttccagggggaaacgcctggtatc tttatagtcctgtcgggtttcgccacctctg acttgagcgtcgatttttgtgatgctcgtca ggggggcggagcctatggaaaaacgccagc aacgcggcctttttacggttcctggccttttg ctggccttttgctcacatgttctttcctgcg ttatcccctgattctgtggataaccgtatta ccgcctttgagtgagctgataccgctcgccgc agccgaacgaccgagcgcagcgagtcagtg agcgaggaagcggaagagcgcctgatgcgg tattttctccttacgcatctgtgcggtatttc acaccgcatatatggtgcactctcagtacaa tctgctctgatgccgcatagttaagccagta tacactccgctatcgctacgtgactgggtca tggctgcgccccgacacccgccaacacccgc tgacgcgccctgacgggcttgtctgctcccg gcatccgcttacagacaagctgtgaccgtct ccgggagctgcatgtgtcagaggttttcacc gtcatcaccgaaacgcgcgaggcagctgcgg taaagctcatcagcgtggtcgtgaagcgatt cacagatgtctgcctgttcatccgcgtccag ctcgttgagtttctccagaagcgttaatgtc tggcttctgataaagcgggccatgttaaggg cggttttttcctgtttggtcactgatgcctc cgtgtaagggggatttctgttcatgggggta atgataccgatgaaacgagagaggatgctca cgatacgggttactgatgatgaacatgcccg gttactggaacgttgtgagggtaaacaactg gcggtatggatgcggcgggaccagagaaaaa tcactcagggtcaatgccagcgcttcgttaa tacagatgtaggtgttccacagggtagccag cagcatcctgcgatgcagatccggaacataa tggtgcagggcgctgacttccgcgtttccag actttacgaaacacggaaaccgaagaccatt catgttgttgctcaggtcgcagacgttttgc agcagcagtcgcttcacgttcgctcgcgtat cggtgattcattctgctaaccagtaaggcaa ccccgccagcctagccgggtcctcaacgaca ggagcacgatcatgctagtcatgccccgcgc ccaccggaaggagctgactgggttgaaggct ctcaagggcatcggtcgagatcccggtgcct aatgagtgagctaacttacattaattgcgtt gcgctcactgcccgctttccagtcgggaaac ctgtcgtgccagctgcattaatgaatcggcc aacgcgcggggagaggcggtttgcgtattgg gcgccagggtggtttttcttttcaccagtga gacgggcaacagctgattgcccttcaccgcc tggccctgagagagttgcagcaagcggtcca cgctggtttgccccagcaggcgaaaatcctg tttgatggtggttaacggcgggatataacat gagctgtcttcggtatcgtcgtatcccacta ccgagatgtccgcaccaacgcgcagcccgga ctcggtaatggcgcgcattgcgcccagcgcc atctgatcgttggcaaccagcatcgcagtgg gaacgatgccctcattcagcatttgcatggt ttgttgaaaaccggacatggcactccagtcg ccttcccgttccgctatcggctgaatttgat tgcgagtgagatatttatgccagccagccag acgcagacgcgccgagacagaacttaatggg cccgctaacagcgcgatttgctggtgaccca atgcgaccagatgctccacgcccagtcgcgt accgtcttcatgggagaaaataatactgttg atgggtgtctggtcagagacatcaagaaata acgccggaacattagtgcaggcagcttccac agcaatggcatcctggtcatccagcggatag ttaatgatcagcccactgacgcgttgcgcga gaagattgtgcaccgccgctttacaggcttc gacgccgcttcgttctaccatcgacaccacc acgctggcacccagttgatcggcgcgagatt taatcgccgcgacaatttgcgacggcgcgtg cagggccagactggaggtggcaacgccaatc agcaacgactgtttgcccgccagttgttgtg ccacgcggttgggaatgtaattcagctccgc catcgccgcttccactttttcccgcgttttc gcagaaacgtggctggcctggttcaccacgc gggaaacggtctgataagagacaccggcata ctctgcgacatcgtataacgttactggtttc acattcaccaccctgaattgactctcttccg ggcgctatcatgccataccgcgaaaggtttt gcgccattcgatggtgtccgggatctcgacg ctctcccttatgcgactcctgcattaggaag cagcccagtagtaggttgaggccgttgagca ccgccgccgcaaggaatggtgcatgcaagga gatggcgcccaacagtcccccggccacgggg cctgccaccatacccacgccgaaacaagcgc tcatgagcccgaagtggcgagcccgatcttc cccatcggtgatgtcggcgatataggcgcca gcaaccgcacctgtggcgccggtgatgccgg ccacgatgcgtccggcgtagaggatcgagat cgatctcgatcccgcgaaattaatacgactc actata | |
| SNAP-25b (1–206, all C to A; capitalized) in pET28a | homemade | tggcgaatgggacgcgccctgtagcggcgca ttaagcgcggcgggtgtggtggttacgcgca gcgtgaccgctacacttgccagcgccctagc gcccgctcctttcgctttcttcccttccttt ctcgccacgttcgccggctttccccgtcaag ctctaaatcgggggctccctttagggttccg atttagtgctttacggcacctcgaccccaaa aaacttgattagggtgatggttcacgtagtg ggccatcgccctgatagacggtttttcgccc tttgacgttggagtccacgttctttaatagt ggactcttgttccaaactggaacaacactca accctatctcggtctattcttttgatttata agggattttgccgatttcggcctattggtta aaaaatgagctgatttaacaaaaatttaacg cgaattttaacaaaatattaacgtttacaat ttcaggtggcacttttcggggaaatgtgcgc ggaacccctatttgtttatttttctaaatac attcaaatatgtatccgctcatgaattaatt cttagaaaaactcatcgagcatcaaatgaaa ctgcaatttattcatatcaggattatcaata ccatatttttgaaaaagccgtttctgtaatg aaggagaaaactcaccgaggcagttccatag gatggcaagatcctggtatcggtctgcgatt ccgactcgtccaacatcaatacaacctatta atttcccctcgtcaaaaataaggttatcaag tgagaaatcaccatgagtgacgactgaatcc ggtgagaatggcaaaagtttatgcatttctt tccagacttgttcaacaggccagccattacg ctcgtcatcaaaatcactcgcatcaaccaaa ccgttattcattcgtgattgcgcctgagcga gacgaaatacgcgatcgctgttaaaaggaca attacaaacaggaatcgaatgcaaccggcgc aggaacactgccagcgcatcaacaatatttt cacctgaatcaggatattcttctaatacctg gaatgctgttttcccggggatcgcagtggtg agtaaccatgcatcatcaggagtacggataa aatgcttgatggtcggaagaggcataaattc cgtcagccagtttagtctgaccatctcatct gtaacatcattggcaacgctacctttgccat gtttcagaaacaactctggcgcatcgggctt cccatacaatcgatagattgtcgcacctgat tgcccgacattatcgcgagcccatttatacc catataaatcagcatccatgttggaatttaa tcgcggcctagagcaagacgtttcccgttga atatggctcataacaccccttgtattactgt ttatgtaagcagacagttttattgttcatga ccaaaatcccttaacgtgagttttcgttcca ctgagcgtcagaccccgtagaaaagatcaaa ggatcttcttgagatcctttttttctgcgcg taatctgctgcttgcaaacaaaaaaaccacc gctaccagcggtggtttgtttgccggatcaa gagctaccaactctttttccgaaggtaactg gcttcagcagagcgcagataccaaatactgt ccttctagtgtagccgtagttaggccaccac ttcaagaactctgtagcaccgcctacatacc tcgctctgctaatcctgttaccagtggctgc tgccagtggcgataagtcgtgtcttaccggg ttggactcaagacgatagttaccggataagg cgcagcggtcgggctgaacggggggttcgtg cacacagcccagcttggagcgaacgacctac accgaactgagatacctacagcgtgagctatg agaaagcgccacgcttcccgaagggagaaa ggcggacaggtatccggtaagcggcagggtc ggaacaggagagcgcacgagggagcttcc agggggaaacgcctggtatctttatagtcctgt cgggtttcgccacctctgacttgagcgtcga tttttgtgatgctcgtcaggggggcggagcc tatggaaaaacgccagcaacgcggccttttt acggttcctggccttttgctggccttttgct cacatgttctttcctgcgttatcccctgatt ctgtggataaccgtattaccgcctttgagtg agctgataccgctcgccgcagccgaacgacc gagcgcagcgagtcagtgagcgaggaagc ggaagagcgcctgatgcggtattttctccttac gcatctgtgcggtatttcacaccgcatatat ggtgcactctcagtacaatctgctctgatgc cgcatagttaagccagtatacactccgctat cgctacgtgactgggtcatggctgcgccccg acacccgccaacacccgctgacgcgccctga cgggcttgtctgctcccggcatccgcttaca gacaagctgtgaccgtctccgggagctgcat gtgtcagaggttttcaccgtcatcaccgaaa cgcgcgaggcagctgcggtaaagctcatcag cgtggtcgtgaagcgattcacagatgtctgc ctgttcatccgcgtccagctcgttgagtttc tccagaagcgttaatgtctggcttctgataa agcgggccatgttaagggcggttttttcctg tttggtcactgatgcctccgtgtaaggggga tttctgttcatgggggtaatgataccgatga aacgagagaggatgctcacgatacgggttac tgatgatgaacatgcccggttactggaacgt tgtgagggtaaacaactggcggtatggatgc ggcgggaccagagaaaaatcactcagggtc aatgccagcgcttcgttaatacagatgtaggt gttccacagggtagccagcagcatcctgcga tgcagatccggaacataatggtgcagggcgc tgacttccgcgtttccagactttacgaaaca cggaaaccgaagaccattcatgttgttgctc aggtcgcagacgttttgcagcagcagtcgct tcacgttcgctcgcgtatcggtgattcattc tgctaaccagtaaggcaaccccgccagccta gccgggtcctcaacgacaggagcacgatcat gcgcacccgtggggccgccatgccggcgata atggcctgcttctcgccgaaacgtttggtgg cgggaccagtgacgaaggcttgagcgagggc gtgcaagattccgaataccgcaagcgacagg ccgatcatcgtcgcgctccagcgaaagcggt cctcgccgaaaatgacccagagcgctgccgg cacctgtcctacgagttgcatgataaagaag acagtcataagtgcggcgacgatagtcatgc cccgcgcccaccggaaggagctgactgggtt gaaggctctcaagggcatcggtcgagatccc ggtgcctaatgagtgagctaacttacattaa ttgcgttgcgctcactgcccgctttccagtc gggaaacctgtcgtgccagctgcattaatga atcggccaacgcgcggggagaggcggtttgc gtattgggcgccagggtggtttttcttttca ccagtgagacgggcaacagctgattgccctt caccgcctggccctgagagagttgcagcaag cggtccacgctggtttgccccagcaggcgaa aatcctgtttgatggtggttaacggcgggat ataacatgagctgtcttcggtatcgtcgtat cccactaccgagatatccgcaccaacgcgca gcccggactcggtaatggcgcgcattgcgcc cagcgccatctgatcgttggcaaccagcatc gcagtgggaacgatgccctcattcagcattt gcatggtttgttgaaaaccggacatggcact ccagtcgccttcccgttccgctatcggctga atttgattgcgagtgagatatttatgccagc cagccagacgcagacgcgccgagacagaa cttaatgggcccgctaacagcgcgatttgctgg tgacccaatgcgaccagatgctccacgccca gtcgcgtaccgtcttcatgggagaaaataat actgttgatgggtgtctggtcagagacatca agaaataacgccggaacattagtgcaggcag cttccacagcaatggcatcctggtcatccag cggatagttaatgatcagcccactgacgcgt tgcgcgagaagattgtgcaccgccgctttac aggcttcgacgccgcttcgttctaccatcga caccaccacgctggcacccagttgatcggcg cgagatttaatcgccgcgacaatttgcgacg gcgcgtgcagggccagactggaggtggcaac gccaatcagcaacgactgtttgcccgccagt tgttgtgccacgcggttgggaatgtaattca gctccgccatcgccgcttccactttttcccg cgttttcgcagaaacgtggctggcctggttc accacgcgggaaacggtctgataagagacac cggcatactctgcgacatcgtataacgttac tggtttcacattcaccaccctgaattgactc tcttccgggcgctatcatgccataccgcgaa aggttttgcgccattcgatggtgtccgggat ctcgacgctctcccttatgcgactcctgcat taggaagcagcccagtagtaggttgaggccg ttgagcaccgccgccgcaaggaatggtgcat gcaaggagatggcgcccaacagtcccccggc cacggggcctgccaccatacccacgccgaaa caagcgctcatgagcccgaagtggcgagccc gatcttccccatcggtgatgtcggcgatata ggcgccagcaaccgcacctgtggcgccggtg atgccggccacgatgcgtccggcgtagagga tcgagatctcgatcccgcgaaattaatacga ctcactataggggaattgtgagcggataaca attcccctctagaaataattttgtttaactt taagaaggagatataccATGGCCGA GGACGCAGACATGCGCAATG AGCTGGAGGAGATGCAGAGG AGGGCTGACCAGCTGGCTGA TGAGTCCCTGGAAAGCACCC GTCGCATGCTGCAGCTGGTT GAAGAGAGTAAAGATGCTGG CATCAGGACTTTGGTTATGTT GGATGAGCAAGGCGAACAAC TGGAACGCATTGAGGAAGGG ATGGACCAAATCAATAAGGAC ATGAAAGAAGCAGAAAAGAAT TTGACGGACCTAGGAAAATTC GCCGGCCTTGCCGTGGCCCC CGCCAACAAGCTTAAATCCAG TGATGCTTACAAAAAAGCCTG GGGCAATAATCAGGATGGAGT AGTGGCCAGCCAGCCTGCCC GTGTGGTGGATGAACGGGAG CAGATGGCCATCAGTGGTGGC TTCATCCGCAGGGTAACAAAT GATGCCCGGGAAAATGAGATG GATGAGAACCTGGAGCAGGT GAGCGGCATCATCGGAAACCT CCGCCACATGGCTCTAGACAT GGGCAATGAGATTGACACCCA GAATCGCCAGATCGACAGGAT CATGGAGAAGGCTGATTCCAA CAAAACCAGAATTGATGAAGC CAACCAACGTGCAACAAAGAT GCTGGGAAGTGGTTAA ctcgagcaccaccaccaccaccactgag atccggctgctaacaaagcccgaaagga agctgagttggctgctgccaccgctgagc aataactagcataaccccttggggcctc taaacgggtcttgaggggttttttgctgaa aggaggaactatatccggat | |
| Materials for protein purificaiton | |||
| 2-Mercaptoethanol | SIGMA | M3148-25ML | |
| Agar | LPS Solution | AGA500 | |
| Ampicillin, Sodium salt | PLS | AC1043-005-00 | |
| Chloramphenicol | PLS | CR1023-050-00 | |
| Competent cells (E. coli) | Novagen | 70956 | Rosetta(DE3)pLysS |
| Glycerol | SIGMA | G5516-500ML | |
| HEPES | SIGMA | H4034-100G | |
| Hydrochloric acid / HCl | SIGMA | 320331-500ML | |
| Imidazole | SIGMA | I2399-100G | |
| Isopropyl β-D-1-thiogalactopyranoside / IPTG | SIGMA | 10724815001 | |
| Kanamycin Sulfate | PLS | KC1001-005-02 | |
| Luria-Bertani (LB) Broth | LPS Solution | LB-05 | |
| Ni-NTA resin | Qiagen | 30210 | |
| PD MiniTrap G-25 (desalting column) | Cytiva | GE28-9180-07 | For instructions, see: https://www.cytivalifesciences.com/en/us/shop/chromatography/prepacked-columns/desalting-and-buffer-exchange/pd-minitrap-desalting-columns-with-sephadex-g-25-resin-p-06174 |
| Phenylmethylsulfonyl fluoride / PMSF | ThermoFisher Scientific | 36978 | |
| Plasmids for SNARE proteins | cloned in house | N/A | Available upon request |
| Protease inhibitor cocktail | genDEPOT | P3100 | |
| Sodium chloride | SIGMA | S5886-500G | |
| Sodium phosphate dibasic / Na2HPO4 | SIGMA | S7907-100G | |
| Sodium phosphate monobasic / NaH2PO4 | SIGMA | S3139-250G | |
| Tris(2-carboxyethyl)phosphine / TCEP | SIGMA | C4706-2G | |
| Trizma base | SIGMA | T1503-250G | |
| Materials for sample assembly | |||
| Biotin-PEG-SVA | LAYSAN BIO | BIO-PEG-SVA-5K-100MG & MPEG-SVA-5K-1g | For PEGylation |
| Dibenzocyclooctyne-amine / DBCO-NH2 | SIGMA | 761540-10MG | For bead coating |
| Double-sided tape | 3M | 136 | For flow cell assembly |
| Epoxy glue | DEVCON | S-208 | For flow cell assembly |
| Glass coverslip for bottom surface | VWR | 48393-251 | Rectangular, 60×24 mm, #1.5 |
| Glass coverslip for top surface | VWR | 48393-241 | Rectangular, 50×24 mm, #1.5 |
| Magnetic bead | ThermoFisher Scientific | 14301 | Dynabeads M-270 Epoxy, 2.8 μm |
| mPEG-SVA | LAYSAN BIO | mPEG-SVA 1g | For PEGylation |
| N,N-Dimethylformamide / DMF | SIGMA | D4551-250ML | For bead coating |
| N-[3-(trimethoxysilyl)propyl]ethylenediamine | SIGMA | 104884-100ML | For PEGylation |
| Neutravidin | ThermoFisher Scientific | 31000 | For sample tethering |
| Phosphate buffered saline / PBS, pH 7.2 | PLS | PR2007-100-00 | |
| Plastic syringe | Norm-ject | A5 | 5 ml, luer tip |
| Polyethylene Tubing | SCI | BB31695-PE/4 | PE-60 |
| Reference bead | SPHEROTECH | SVP-30-5 | Streptavidin-coated Polystyrene Particles; 3.0-3.4 µm |
| Syringe needle | Kovax | 21G-1 1/4'' | 21 G |
| Syringe pump | KD SCIENTIFIC | 788210 | |
| Equipment for magnetic tweezer instrument | |||
| 1-axis motorized microtranslation stage | PI | M-126.PD1 | For vertical positioning of magnets |
| 2-axis manual translation stage | ST1 | LEE400 | For alignment of magnets to the optical axis |
| Acrylic holder for magnets | DaiKwang Precision | custum order | Drawing available upon request |
| Frame grabber | Active Silicon | AS-FBD-4XCXP6-2PE8 | |
| High-speed CMOS camera | Mikrotron | EoSens 3CXP | |
| Inverted microscope | Olympus | IX73P2F-1-2 | |
| Neodymium magnets | LG magnet | ND 10x10x12t | Dimension: 10 mm × 10 mm × 12 mm; two needed |
| Objective lens | Olympus | UPLXAPO100XO | Oil-immersion, NA 1.45 |
| Objective lens nanopositioner | Mad City Labs | Nano-F100S | |
| Rotation stepper motor | AUTONICS | A3K-S545W | For rotating magnets |
| Superluminescent diode | QPHOTONICS | QSDM-680-2 | 680 nm |
| Software | |||
| LabVIEW | National Instruments | v20.0f1 | |
| MATLAB | MathWorks | v2021a |
Referenzen
- Le, S., Liu, R., Lim, C. T., Yan, J. Uncovering mechanosensing mechanisms at the single protein level using magnetic tweezers. Methods. 94, 13-18 (2016).
- Choi, H. -. K., Kim, H. G., Shon, M. J., Yoon, T. -. Y. High-resolution single-molecule magnetic tweezers. Annual Review of Biochemistry. 91 (1), 33-59 (2022).
- Yang, T., Park, C., Rah, S. -. H., Shon, M. J. Nano-precision tweezers for mechanosensitive proteins and beyond. Molecules and Cells. 45 (1), 16-25 (2022).
- Neuman, K. C., Nagy, A. Single-molecule force spectroscopy: optical tweezers, magnetic tweezers and atomic force microscopy. Nature Methods. 5 (6), 491-505 (2008).
- De Vlaminck, I., Dekker, C. Recent advances in magnetic tweezers. Annual Review of Biophysics. 41 (1), 453-472 (2012).
- Bustamante, C. J., Chemla, Y. R., Liu, S., Wang, M. D. Optical tweezers in single-molecule biophysics. Nature Reviews Methods Primers. 1, 25 (2021).
- Gosse, C., Croquette, V. Magnetic tweezers: micromanipulation and force measurement at the molecular level. Biophysical Journal. 82 (6), 3314-3329 (2002).
- Smith, S. B., Finzi, L., Bustamante, C. Direct mechanical measurements of the elasticity of single DNA molecules by using magnetic beads. Science. 258 (5085), 1122-1126 (1992).
- Lansdorp, B. M., Tabrizi, S. J., Dittmore, A., Saleh, O. A. A high-speed magnetic tweezer beyond 10,000 frames per second. Review of Scientific Instruments. 84 (4), 044301 (2013).
- Cnossen, J. P., Dulin, D., Dekker, N. H. An optimized software framework for real-time, high-throughput tracking of spherical beads. Review of Scientific Instruments. 85 (10), 103712 (2014).
- Dulin, D., et al. High spatiotemporal-resolution magnetic tweezers: calibration and applications for DNA dynamics. Biophysical Journal. 109 (10), 2113-2125 (2015).
- Huhle, A., et al. Camera-based three-dimensional real-time particle tracking at kHz rates and Ångström accuracy. Nature Communications. 6 (1), 5885 (2015).
- Popa, I., et al. A HaloTag anchored ruler for week-long studies of protein dynamics. Journal of the American Chemical Society. 138 (33), 10546-10553 (2016).
- Shon, M. J., Kim, H., Yoon, T. -. Y. Focused clamping of a single neuronal SNARE complex by complexin under high mechanical tension. Nature Communications. 9 (1), 3639 (2018).
- Tapia-Rojo, R., Eckels, E. C., Fernández, J. M. Ephemeral states in protein folding under force captured with a magnetic tweezers design. Proceedings of the National Academy of Sciences. 116 (16), 7873-7878 (2019).
- Löf, A., et al. Multiplexed protein force spectroscopy reveals equilibrium protein folding dynamics and the low-force response of von Willebrand factor. Proceedings of the National Academy of Sciences. 116 (38), 18798-18807 (2019).
- Tapia-Rojo, R., Alonso-Caballero, A., Fernandez, J. M. Direct observation of a coil-to-helix contraction triggered by vinculin binding to talin. Science Advances. 6 (21), (2020).
- Rieu, M., et al. Parallel, linear, and subnanometric 3D tracking of microparticles with Stereo Darkfield Interferometry. Science Advances. 7 (6), (2021).
- Rieu, M., Valle-Orero, J., Ducos, B., Allemand, J. -. F., Croquette, V. Single-molecule kinetic locking allows fluorescence-free quantification of protein/nucleic-acid binding. Communications Biology. 4 (1), 1083 (2021).
- Woodside, M. T., et al. Nanomechanical measurements of the sequence-dependent folding landscapes of single nucleic acid hairpins. Proceedings of the National Academy of Sciences. 103 (16), 6190-6195 (2006).
- Camunas-Soler, J., Ribezzi-Crivellari, M., Ritort, F. Elastic properties of nucleic acids by single-molecule force spectroscopy. Annual Review of Biophysics. 45 (1), 65-84 (2016).
- Südhof, T. C., Rothman, J. E. Membrane fusion: grappling with SNARE and SM proteins. Science. 323 (5913), 474-477 (2009).
- Gao, Y., et al. Single reconstituted neuronal SNARE complexes zipper in three distinct stages. Science. 337 (6100), 1340-1343 (2012).
- Zorman, S., et al. Common intermediates and kinetics, but different energetics, in the assembly of SNARE proteins. eLife. 3, e03348 (2014).
- Zhang, Y., Hughson, F. M. Chaperoning SNARE folding and assembly. Annual Review of Biochemistry. 90 (1), 581-603 (2021).
- Vilfan, I. D., Lipfert, J., Koster, D. A., Lemay, S. G., Dekker, N. H. Magnetic tweezers for single-molecule experiments. Handbook of Single-Molecule Biophysics. , 371-395 (2009).
- You, H., Le, S., Chen, H., Qin, L., Yan, J. Single-molecule manipulation of G-quadruplexes by magnetic tweezers. Journal of Visualized Experiments. (127), e56328 (2017).
- Lipfert, J., Hao, X., Dekker, N. H. Quantitative modeling and optimization of magnetic tweezers. Biophysical Journal. 96 (12), 5040-5049 (2009).
- Dulin, D., Barland, S., Hachair, X., Pedaci, F. Efficient illumination for microsecond tracking microscopy. PLoS One. 9 (9), e107335 (2014).
- Klaue, D., Seidel, R. Torsional stiffness of single superparamagnetic microspheres in an external magnetic field. Physical Review Letters. 102 (2), 028302 (2009).
- Shon, M. J., Rah, S. -. H., Yoon, T. -. Y. Submicrometer elasticity of double-stranded DNA revealed by precision force-extension measurements with magnetic tweezers. Science Advances. 5 (6), 1697 (2019).
- Czerwinski, F., Richardson, A. C., Oddershede, L. B. Quantifying noise in optical tweezers by Allan variance. Optics Express. 17 (15), 13255-13269 (2009).
- Lansdorp, B. M., Saleh, O. A. Power spectrum and Allan variance methods for calibrating single-molecule video-tracking instruments. Review of Scientific Instruments. 83 (2), 025115 (2012).
- Ostrofet, E., Papini, F. S., Dulin, D. High spatiotemporal resolution data from a custom magnetic tweezers instrument. Data in Brief. 30, 105397 (2020).
- Yu, Z., et al. A force calibration standard for magnetic tweezers. Review of Scientific Instruments. 85 (12), 123114 (2014).
- Strick, T. R., Allemand, J. -. F., Bensimon, D., Bensimon, A., Croquette, V. The elasticity of a single supercoiled DNA molecule. Science. 271 (5257), 1835-1837 (1996).
- Daldrop, P., Brutzer, H., Huhle, A., Kauert, D. J., Seidel, R. Extending the range for force calibration in magnetic tweezers. Biophysical Journal. 108 (10), 2550-2561 (2015).
- te Velthuis, A. J. W., Kerssemakers, J. W. J., Lipfert, J., Dekker, N. H. Quantitative guidelines for force calibration through spectral analysis of magnetic tweezers data. Biophysical Journal. 99 (4), 1292-1302 (2010).
- Ostrofet, E., Papini, F. S., Dulin, D. Correction-free force calibration for magnetic tweezers experiments. Scientific Reports. 8 (1), 15920 (2018).
- Seol, Y., Li, J., Nelson, P. C., Perkins, T. T., Betterton, M. D. Elasticity of short DNA molecules: theory and experiment for contour lengths of 0.6-7 µm. Biophysical Journal. 93 (12), 4360-4373 (2007).
- Burnham, D. R., Vlaminck, I. D., Henighan, T., Dekker, C. Skewed Brownian fluctuations in single-molecule magnetic tweezers. PLoS One. 9 (9), 108271 (2014).
- Paul, T., Myong, S. Protocol for generation and regeneration of PEG-passivated slides for single-molecule measurements. STAR Protocols. 3 (1), 101152 (2022).
- Lee, H. -. W., et al. Profiling of protein-protein interactions via single-molecule techniques predicts the dependence of cancers on growth-factor receptors. Nature Biomedical Engineering. 2 (4), 239-253 (2018).
- Cheezum, M. K., Walker, W. F., Guilford, W. H. Quantitative comparison of algorithms for tracking single fluorescent particles. Biophysical Journal. 81 (4), 2378-2388 (2001).
- Parthasarathy, R. Rapid, accurate particle tracking by calculation of radial symmetry centers. Nature Methods. 9 (7), 724-726 (2012).
- Woodside, M. T., Block, S. M. Reconstructing folding energy landscapes by single-molecule force spectroscopy. Annual Review of Biophysics. 43 (1), 19-39 (2014).
- Evans, E., Ritchie, K. Dynamic strength of molecular adhesion bonds. Biophysical Journal. 72 (4), 1541-1555 (1997).
- Zhang, Y. Energetics, kinetics, and pathway of SNARE folding and assembly revealed by optical tweezers. Protein Science. 26 (7), 1252-1265 (2017).
- Chen, H., et al. Improved high-force magnetic tweezers for stretching and refolding of proteins and short DNA. Biophysical Journal. 100 (2), 517-523 (2011).
- Cho, S., et al. Tension exerted on cells by magnetic nanoparticles regulates differentiation of human mesenchymal stem cells. Biomaterials Advances. 139, 213028 (2022).
- Shon, M. J., Cohen, A. E. Nano-mechanical measurements of protein-DNA interactions with a silicon nitride pulley. Nucleic Acids Research. 44 (1), 7 (2016).
- Cheng, Y. Single-particle cryo-EM-How did it get here and where will it go. Science. 361 (6405), 876-880 (2018).
- Jumper, J., et al. Highly accurate protein structure prediction with AlphaFold. Nature. 596 (7873), 583-589 (2021).
- Neupane, K., et al. Direct observation of transition paths during the folding of proteins and nucleic acids. Science. 352 (6282), 239-242 (2016).
- Choi, H. -. K., et al. Watching helical membrane proteins fold reveals a common N-to-C-terminal folding pathway. Science. 366 (6469), 1150-1156 (2019).
- Kim, C., et al. Extreme parsimony in ATP consumption by 20S complexes in the global disassembly of single SNARE complexes. Nature Communications. 12 (1), 3206 (2021).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten