Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Generierung und Analyse von High-Parameter-Histologiebildern mit Histoflow-Zytometrie
In diesem Artikel
Zusammenfassung
Hier wird ein Verfahren beschrieben, mit dem fünf oder mehr Fluoreszenzparameter mittels Immunfluoreszenzmikroskopie abgebildet werden können. Es wird eine Analysepipeline für die Extraktion einzelner Zellen aus diesen Bildern und die Durchführung von Einzelzellanalysen durch durchflusszytometrie-ähnliche Gating-Strategien skizziert, mit der Zelluntergruppen in Gewebeschnitten identifiziert werden können.
Zusammenfassung
Die Verwendung der Histologie zur Untersuchung der Immunzelldiversität in Gewebeschnitten, wie sie aus dem Zentralnervensystem (ZNS) stammen, ist durch die Anzahl der Fluoreszenzparameter, die gleichzeitig abgebildet werden können, entscheidend begrenzt. Die meisten Untergruppen von Immunzellen wurden mittels Durchflusszytometrie unter Verwendung komplexer Kombinationen von Proteinmarkern definiert, die oft vier oder mehr Parameter zur endgültigen Identifizierung erfordern, was über die Möglichkeiten der meisten herkömmlichen Mikroskope hinausgeht. Da die Durchflusszytometrie Gewebe dissoziiert und räumliche Informationen verliert, besteht ein Bedarf an Techniken, die räumliche Informationen speichern und gleichzeitig die Rollen komplexer Zelltypen hinterfragen können. Diese Probleme werden hier angegangen, indem eine Methode zur Erweiterung der Anzahl der Fluoreszenzparameter entwickelt wird, die abgebildet werden können, indem die Signale von spektral überlappenden Fluorophoren gesammelt und die Signale jedes einzelnen Fluorophors durch spektrale Entmischung getrennt werden. Diese Bilder werden dann mit einer Analysepipeline verarbeitet, um histologische Bilder mit hohen Parametern aufzunehmen und einzelne Zellen aus diesen Bildern zu extrahieren, so dass die einzigartigen Fluoreszenzeigenschaften jeder Zelle auf Einzelzellebene analysiert werden können. Mit Hilfe von Durchflusszytometrie-ähnlichen Gating-Strategien können Zellen dann in Untergruppen profiliert und auf die Histologieschnitte zurückgeführt werden, um nicht nur ihre Häufigkeit zu quantifizieren, sondern auch festzustellen, wie sie mit der Gewebeumgebung interagieren. Insgesamt wird die Einfachheit und das Potenzial der Verwendung der Histoflow-Zytometrie zur Untersuchung komplexer Immunpopulationen in histologischen Abschnitten demonstriert.
Einleitung
Entzündungen, die von Zellen des Immunsystems und Gliazellen ausgelöst werden, können zu chronischen Erkrankungen des ZNS beitragen, bei denen jede Population die Aktivität der anderen fördern kann 1,2,3. Zu verstehen, wie das Immunsystem mit diesen Elementen des ZNS interagiert, um eine ZNS-Entzündung zu fördern, ist derzeit ein wichtiges Thema von Interesse und wurde durch hochparametrische Techniken wie die Einzelzell-RNA-Sequenzierung erheblich erleichtert. Durch Einzelzell-RNA-Sequenzierung haben wir entdeckt, dass bei mehreren ZNS-Erkrankungen eine umfangreiche Kommunikation zwischen Gliazellen und dem Immunsystem stattfindet 4,5,6. Zu verstehen, wie sich diese Wechselwirkungen auf diese Erkrankungen auswirken, wird entscheidend sein, um die Biologie dieser Krankheiten aufzuklären.
Ein Problem bei Einzelzellsequenzierungsanalysen besteht darin, dass bei diesen Techniken das Gewebe aufgebrochen werden muss, um einzelne Zellen oder Zellkerne zu erhalten, was zu einem vollständigen Verlust der räumlichen Information führt. Zu wissen, wo sich eine Zelle in einem Gewebe befindet, ist entscheidend, um die Rolle der Zelle bei der Entstehung von Entzündungen zu verstehen. Zum Beispiel können sich Immunzellen wie B-Zellen während einer Neuroinflammation im ZNS konzentrieren; sie dringen jedoch selten in das ZNS-Parenchym ein und konzentrieren sich stattdessen in den ZNS-Schranken7. Angesichts ihrer Lokalisation ist es unwahrscheinlich, dass diese Zellen zur ZNS-Entzündung beitragen, indem sie physisch mit Gliazellen im ZNS-Parenchym interagieren, was darauf hindeutet, dass Wechselwirkungen, die sie mit Gliazellen haben könnten, durch sezernierte Faktoren erfolgen würden. Darüber hinaus weist die bei ZNS-Erkrankungen auftretende Pathologie häufig eine Struktur 8,9 auf, so dass die Lokalisation einer Zelle im Gewebe entscheidend bestimmen könnte, ob sie aktiv zu der Störung beiträgt oder ein Zuschauer ist. Daher ist die Verwendung der räumlichen Orientierung zur Bewertung der Rolle einer Zelle in der Pathologie unerlässlich.
Die Untersuchung von Zellen im Gewebe wurde in der Regel mit Hilfe der Immunhistochemie oder der Immunfluoreszenzmikroskopie durchgeführt. Ein Problem bei diesen Techniken besteht darin, dass sie in der Regel nur bis zu vier Parameter gleichzeitig abbilden können. Dies ist eine große Einschränkung dieser Techniken, da wir aus der Durchflusszytometrie und Einzelzell-RNA-Sequenzierungsanalysen wissen, dass viele Zellpopulationen zwei oder mehr Parameter für ihre Identifizierung benötigen; Außerdem erhöht sich die Anzahl der erforderlichen Parameter in der Regel, wenn nach bestimmten Teilmengen einer Zelle vom Typ10 gesucht wird. Daher ist es unpraktisch, Standard-Bildgebungsverfahren zu verwenden, um zu untersuchen, wie Untergruppen von Zellen innerhalb eines Gewebes interagieren können.
Dieses Problem wurde teilweise durch neuere Methoden mit hohen Parametern überwunden, die räumliche Informationen speichern können, wie z. B. die räumliche RNA-Sequenzierung11 und die bildgebende Massenzytometrie12. Diese Techniken sind zwar wertvoll, haben aber mehrere Probleme, wie z. B. dass sie nicht überall verfügbar sind, dreidimensionale Daten in zwei Dimensionen reduzieren und viel Fachwissen für die Ausführung erfordern. Eine andere Technik, die als sequentielle Färbung bekannt ist, bei der Gewebe mit einem Satz Antikörper gefärbt werden, gefolgt von einer Inaktivierung des vorherigen Antikörpersatzes, bevor sie mit einem anderen Satz von Antikörpern gefärbt werden, kann eine Histologie mit hohen Parametern erreichen, ohne dass spezielle Geräte oder Fachwissen erforderlich sind 13,14,15. Die sequentielle Färbung kann jedoch äußerst arbeitsintensiv sein und erfordert eine große Menge an Mikroskopiezeit, was für Labore, die kein persönliches Mikroskop besitzen, unpraktisch sein kann. Daher besteht ein Bedarf an Techniken, die die Anzahl der Fluoreszenzparameter erweitern können, die gleichzeitig auf Mikroskopen abgebildet werden können, die weit verbreitet und zeitnah abgebildet werden können.
Sobald die Daten mit hohen Parametern erfasst wurden, stellt sich ein weiteres Problem: Es ist unwahrscheinlich, dass herkömmliche Bildanalysemethoden die Daten erfolgreich analysieren können. Techniken wie manuelles Zählen oder Schwellenwerte sind nur dann praktikabel, wenn die Analyse aus einem einzigen Parameter besteht oder wenn mehrere Marker die gleiche Lokalisation haben, bei der nur überlappende Signale gezählt werden. Diese Einschränkung macht die herkömmliche Analyse für die Arbeit mit Datasets mit hohen Parametern ungeeignet. Eine erfolgreiche Analyse dieser Datensätze wurde durch die Segmentierung einzelner Zellen aus histologischen Bildern und die anschließende Durchführung durchflusszytometrischer Gating-ähnlicher Gating-Strategien zur Identifizierung von Zelltypen erreicht16,17. Ein weiteres Problem, das diese Analysen betrifft, besteht jedoch darin, dass sie nur für Datensätze funktionieren, bei denen alle interessierenden Zellen physisch voneinander getrennt sind, da diese Techniken keine Methoden verwenden, die Zellen, die in physischem Kontakt stehen, genau trennen können. Daher ist eine neuere Methode erforderlich, die Einzelzellanalysen in histologischen Schnitten auch dann durchführen kann, wenn die Zellen in physischem Kontakt stehen.
In diesem Artikel wird ein einfaches Protokoll namens Histoflow-Zytometrie beschrieben, das zuvor eingeführt wurde18 und das die Anzahl der Fluoreszenzparameter erweitert, die gleichzeitig mit weit verbreiteten Mikroskopen abgebildet werden können. Bei diesem Protokoll werden Gewebe mit spektral überlappenden Farbstoffen gefärbt und dann mit spektraler Kompensation Durchscheinen aus überlappenden Kanälen entfernt, um klare Einzelfärbungen zu erhalten. Um die Analyse von hochparametrischen Histologiebildern zu erleichtern, wird eine detaillierte Analysepipeline beschrieben, die einzelne Zellen aus Gewebeschnitten extrahiert, um Zellen mit Hilfe von Durchflusszytometrie-ähnlichen Gating-Strategien in unterschiedliche Populationen zu sortieren. Dieses Protokoll funktioniert in Geweben, in denen Zellen diffus vorhanden sind, und in Geweben, in denen Zellen eng miteinander verdichtet sind, was diese Technik vielseitig für die Untersuchung von Geweben wie dem ZNS sowohl in der Homöostase als auch in der Neuroinflammation macht. Die Histoflow-Zytometrie ist daher eine nützliche Technik zur Untersuchung von Wechselwirkungen zwischen komplexen Zelltypen, die mehrere Zellmarker benötigen, um Zellen zu definieren und gleichzeitig räumliche Informationen zu erhalten.
Protokoll
Dieses Protokoll gilt nicht für das Schneiden von Geweben für die Histologie; Bitte lesen Sie Jain et al.18 oder19 für Beschreibungen, wie Gewebe für die Histologie geschnitten werden. Dieses Protokoll kann mit allen geschnittenen Geweben auf Objektträgern verwendet werden. In diesem Artikel werden leistenale Lymphknoten verwendet, die von einem immunisierten Tier isoliert wurden, wie zuvor beschrieben18. Das Verfahren und der Zeitplan für dieses Protokoll sind in Abbildung 1 zusammengefasst. Die Einzelheiten zu den Reagenzien und der Ausrüstung, die in dieser Studie verwendet wurden, sind in der Materialtabelle aufgeführt.
1. Färben von Gewebeschnitten
ANMERKUNG: Zusätzlich zum Färben von Geweben mit allen Antikörpermarkierungen, an denen der Forscher interessiert ist, sollte der Forscher auch Einzelfarbkontrollen (eine für jeden Fluoreszenzparameter, der verwendet werden soll) auf benachbarten oder anderweitig identischen Gewebeobjektträgern vorbereiten, wobei jede einzelne Farbkontrolle mit den Reagenzien gefärbt wird, die erforderlich sind, um das Signal eines Fluoreszenzparameters zu erzeugen.
- Perm und Blockierungspuffer sowie Perm und Färbepuffer vorbereiten (Zusammensetzung siehe Tabelle 1 ).
- Tauen Sie Gewebeschnitte auf Objektträgern bei Raumtemperatur auf und wischen Sie das Kondenswasser mit einem Tuch ab. Verwenden Sie einen hydrophoben Marker, um den Rand des Gewebes zu umreißen, das gefärbt werden muss, und lassen Sie die Markierungen trocknen.
- Rehydrieren Sie das Gewebe, indem Sie die Gewebeabschnitte 1 Minute lang in phosphatgepufferter Kochsalzlösung (PBS) einweichen. Schütteln Sie das PBS ab und verwenden Sie ein Tuch, um überschüssiges PBS zu entfernen, ohne das Taschentuch direkt zu berühren.
- Fixieren Sie das Gewebe, indem Sie genügend 4% Paraformaldehyd (PFA) hinzufügen, um das Gewebe vollständig zu bedecken. 20 min bei Raumtemperatur inkubieren.
ACHTUNG: PFA kann giftig sein und sollte in einem Abzug gehandhabt werden.
HINWEIS: Dieser Schritt ist nur für frisches gefrorenes Gewebe erforderlich. Wenn das Gewebe bereits fixiert ist, ignorieren Sie diesen Schritt. Die PFA-Fixierung wird hier beschrieben, aber das Gewebe kann je nach den Anforderungen des Experiments mit anderen Methoden fixiert werden. - Waschen Sie die Taschentücher 1 Minute lang in einem PBS-Bad unter leichtem Schaukeln und nehmen Sie dann den Objektträger aus der Badewanne. Tauschen Sie das PBS aus und wiederholen Sie die Wäsche noch 3 Mal.
- Schütteln Sie überschüssiges PBS ab und verwenden Sie ein Tuch, um überschüssiges PBS zu entfernen, ohne das Taschentuch direkt zu berühren.
- Bestimmen Sie, welche Sekundärantikörper in Schritt 1.12 des Protokolls verwendet werden müssen und von welcher Tierart die Sekundärantikörper gewonnen werden. Reichern Sie den Perm und den Blockierungspuffer an, indem Sie dem Perm Serum von diesen Tieren zusetzen und den Blockierungspuffer auf eine Endkonzentration von 5 % für jede Spezies bringen.
HINWEIS: Die Verwendung von Sekundärantikörpern hilft, die Signalintensität zu verstärken. Daher wird empfohlen, dass Primärantikörper, die schwache Signale erzeugen, mit Sekundärantikörpern gepaart werden sollten, um ihre Signale zu verstärken. Wenn alle Antikörperziele, die gefärbt werden, hell genug sind, um keine Sekundärantikörper zu benötigen, können die Schritte 1.7-1.12 und Schritt 1.15 übersprungen werden. - Blockieren Sie die unspezifische Bindung von Antikörpern, indem Sie genügend angereichertes Perm und Blockierungspuffer hinzufügen, um jedes Gewebe zu bedecken. Inkubieren Sie die Objektträger für 1-8 h bei Raumtemperatur in einer befeuchteten, lichtgeschützten Kammer.
- Geben Sie unkonjugierte Primärantikörper in experimentell bestimmten Verdünnungen zu Perm und Färbepuffer (die Spezies oder Immunglobulin-Isotypen der Primärantikörper sollten alle unterschiedlich und unterscheidbar sein durch die Sekundärantikörper, die der Forscher in Schritt 1.12 verwenden möchte).
- Schütten Sie die Dauerwelle und den Blockierungspuffer ab und verwenden Sie ein Tuch, um überschüssigen Puffer zu entfernen, ohne das Gewebe direkt zu berühren. Geben Sie genügend Dauerwelle und Färbepuffer mit primären Antikörpern in das Gewebe, um es zu bedecken, und inkubieren Sie den Objektträger bei 4 °C für 12-48 h.
HINWEIS: Der Forscher muss den optimalen Zeitpunkt für die Antikörperinkubation bestimmen. - Bereiten Sie den Blockierungspuffer und den Färbepuffer vor (Tabelle 1).
- Dem in Schritt 1.11 hergestellten Färbepuffer werden Sekundärantikörper in experimentell bestimmten Verdünnungen zugesetzt. Die verwendeten Sekundärantikörper sollten auf dieselben Spezies oder Antikörperisotypen abzielen, die in Schritt 1.9 verwendet wurden.
- Bereiten Sie den Abschreckpuffer vor (wie in Tabelle 1).
HINWEIS: Dieser Schritt hilft, die Autofluoreszenz aus dem Gewebe zu reduzieren. Wenn dies nicht erforderlich ist, können die Schritte 1.13 und 1.14 übersprungen werden. - Waschen Sie die Tücher wie in den Schritten 1.5-1.6 beschrieben. Fügen Sie dann genügend Quenchpuffer hinzu, um das Gewebe auf den Objektträgern zu bedecken, und inkubieren Sie die Objektträger 1-2 Minuten lang bei Raumtemperatur.
- Waschen Sie das Taschentuch wie in den Schritten 1.5-1.6 beschrieben. Anschließend werden die in dem in Schritt 1.12 hergestellten Färbepuffer verdünnten Sekundärantikörper so zu den Geweben gegeben, dass das Gewebe vollständig mit dem Färbepuffer bedeckt ist. Objektträger bei 4 °C für 2-8 h inkubieren.
- Bereiten Sie die Blockierungslösung vor, indem Sie 50 μl normales Serum von der in Schritt 1.18 verwendeten Antikörperspezies (5 % jeder Spezies) zu dem in Schritt 1.11 hergestellten Blockierungspuffer hinzufügen.
- Waschen Sie die Tücher wie in den Schritten 1.5-1.6 beschrieben. Geben Sie dann die Blockierungslösung mit normalem Serum, das in Schritt 1.16 hergestellt wurde, in das Gewebe, so dass es vollständig mit Färbepuffer bedeckt ist. Inkubieren Sie die Objektträger bei Raumtemperatur für 1-8 h.
- Die Färbelösung wird durch Zugabe von Fluorophor-konjugierten Primärantikörpern in experimentell bestimmten Verdünnungen zu dem in Schritt 1.11 hergestellten Färbepuffer hergestellt. Geben Sie außerdem einen Kernfleck, wie z. B. Kerngelb, in einer experimentell bestimmten Verdünnung in die Färbelösung.
HINWEIS: Die Schritte 1.18-1.19 sind nur erforderlich, wenn der Forscher zusätzliche Antikörpermarkierungen verwenden muss, die keine Sekundärantikörper für die Signalverstärkung benötigen. Wenn keine primären konjugierten Antikörper verwendet werden, können Kernfärbungen in Schritt 1.12 hinzugefügt werden. - Schütteln Sie den Blockierungspuffer ab und verwenden Sie ein Tuch, um überschüssigen Puffer zu entfernen, ohne das Taschentuch direkt zu berühren. Geben Sie so viel in Schritt 1.18 hergestellte Färbelösung in das Gewebe, dass es bedeckt ist, und inkubieren Sie es bei 4 °C für 12-48 h.
- Waschen Sie die Tücher wie in den Schritten 1.5-1.6 beschrieben. Geben Sie 5 Tropfen Eindeckmittel auf jeden Objektträger, ohne Blasen einzuführen, und kippen Sie vorsichtig und langsam ein Glasdeckglas über das Gewebe, um das Gewebe vollständig mit Eindeckmittel zu bedecken und alle Luftblasen auszuschließen. Lagern Sie die Objektträger bei 4 °C im Dunkeln, bis sie für die Aufnahme bereit sind.
2. Bildgebung der Gewebeschnitte
HINWEIS: In diesem Stadium werden die Gewebeschnitte mit allen Antikörpern gefärbt, die für alle Erkrankungen, die abgebildet werden sollen, von Interesse sind, und einfarbige Kontrollen werden mit einem Fluoreszenzparameter auf jedem Schnitt gefärbt. Die Bildgebung sollte auf einem Mikroskop erfolgen, das über mehrere Detektoren verfügt, die so eingestellt werden können, dass sie bestimmte Lichtbereiche erkennen, und idealerweise sollte es Zugang zu so vielen Laserlinien wie möglich zur Anregung haben.
- Setzen Sie einen der vollständig gefärbten Objektträger in das Mikroskop ein und richten Sie das Mikroskop so ein, dass der Schnitt mit dem Objektiv abgebildet wird, das für die Datenerfassung verwendet werden soll.
- Basierend auf den Fluorophoren, mit denen das Gewebe gefärbt wird, identifizieren Sie die Anregungslaserlinien, die zur Stimulation dieser Fluorophore erforderlich sind, und identifizieren Sie die Emissionsbereiche für jeden Fluorophor, der die größte Signalmenge enthält.
HINWEIS: Online-Tools zur Anzeige von Fluoreszenzspektren (z. B. siehe Materialtabelle) können nützlich sein, um die optimalen Anregungswellenlängen von Fluorophoren und die optimalen Bereiche für die Fluorophordetektion zu identifizieren. Fluorophore, die für die Histoflow-Zytometrie verwendet werden, sollten so gewählt werden, dass sich jedes Fluorophor in der optimalen Laserlinie, durch die das Fluorophor stimuliert wird, oder durch den optimalen Bereich der Emissionswellenlängen unterscheidet. Fluorophore mit engeren Anregungs-/Emissionswellenlängen sind schwieriger zu trennen. Die für diese Studie verwendeten Fluorophore sind im Abschnitt "Ergebnisse" aufgeführt. - Öffnen Sie Software 1 (siehe Materialtabelle) in einem konfokalen Mikroskop und wählen Sie oben den Konfigurationsreiter aus. Wählen Sie dann auf der linken Seite den Hardware-Button aus und ändern Sie die Bittiefe auf 16 Bit.
HINWEIS: Es wird empfohlen, für die Datenerfassung höhere Bittiefen, wie z. B. 16-Bit-Daten, zu verwenden, da dies die Genauigkeit der spektralen Kompensation verbessert. Darüber hinaus verbessern höhere Pixeldichten, höhere Zahlen für Liniendurchschnitte und mehr Schritte im Z-Stapel die Qualität der spektralen Trennung. - Wählen Sie oben auf dem Bildschirm die Registerkarte "Erfassen " aus, und geben Sie die Formateinstellung ein, um die Pixeldichte des Bildes einzubeziehen (in der Regel führen höhere Auflösungen zu besseren Ergebnissen).
- Falls verfügbar, wählen Sie die bidirektionale X-Erfassung , um die Bildgebungszeit zu verkürzen, vorausgesetzt, diese Funktion wurde ausreichend kalibriert.
- Wählen Sie einen Lochblendenwert für das konfokale Mikroskop (typischerweise zwischen 400 und 600 nm), bei dem kleinere Werte in der Regel qualitativ hochwertigere histoflow-zytometrische Ergebnisse liefern, obwohl kleinere Lochblendenwerte die Aufnahme von Bildern erschweren und möglicherweise zusätzliche Bildgebungszeit oder höhere Anregungsintensitäten erfordern, um Bilder zu erzeugen.
- Wählen Sie oben im linken Menü das Bedienfeld "Sequenziellen Scan anzeigen " aus, um die Option für den sequenziellen Scan zu öffnen. Verwenden Sie das '+' -Zeichen, um zusätzliche Sequenzen einzurichten, wobei jede Sequenz eine Anregungswellenlänge enthält, die in Schritt 2.2 festgelegt wurde.
- Schalten Sie alle benötigten Laser ein, indem Sie auf die ON-Tasten klicken, die den gewünschten Lasern in der Mitte des Bildschirms zugeordnet sind, um sie in den ON-Zustand zu versetzen. Gehen Sie in jede spezifische Sequenz und erhöhen Sie die Laserleistung in der Mitte des Bildschirms auf den gewünschten Wert, wobei für jede Sequenz eine Anregungswellenlänge verwendet wird.
- Schalten Sie in jeder Sequenz so viele Detektoren wie nötig im unteren Teil des Bildschirms ein , um alle Fluorophore abzubilden, die auf dieser Laserlinie abgebildet werden. Passen Sie die Bereiche der Detektoren an, indem Sie auf ihre Bereiche doppelklicken und den oberen und unteren Bereich der zu erfassenden Wellenlängen eingeben.
HINWEIS: Es wird empfohlen, nach Möglichkeit hochempfindliche Detektoren zu verwenden, und idealerweise sollten alle Kanäle auf denselben Detektortypen gesammelt werden, obwohl dies nicht erforderlich ist. - Drücken Sie die Live-Taste und fokussieren Sie sich auf das Sample. Verwenden Sie die Schaltfläche "Auto Scale " links neben dem Bild in der Software, um zu bewerten, ob die Kanäle ausreichend mit dem Signal gesättigt sind. Der Benutzer sollte die Verstärkungswerte und Laserintensitäten in jeder Sequenz anpassen, bis die automatische Skalierung den Maximalwert (65535 für 16-Bit-Daten) nicht erreicht, aber für alle Kanäle sehr nahe an der Sättigung liegt.
HINWEIS: Da sich verschiedene Fluorophore in ihrer Helligkeit und verschiedene Antikörperziele in ihrer Häufigkeit unterscheiden können, muss die Empfindlichkeit der Detektoren so angepasst werden, dass sie für helle Signale eine geringe Empfindlichkeit und für schwache Signale eine hohe Empfindlichkeit aufweist, wenn beide Fluorophore durch denselben Laser stimuliert werden (die Laserintensität kann sich in diesem Szenario nicht ändern, da beide Fluorophore betroffen wären). Um optimale Ergebnisse zu erzielen, sollte das Fluoreszenzsignal von jedem Kanal im Experiment so ausgeglichen werden, dass jeder Kanal ungefähr die gleichen Signalmengen hat, was das spektrale Durchscheinen in andere Kanäle reduziert und zu stärkeren Signalen führt. Da sich verschiedene Proben in der Häufigkeit der zu färbenden Marker von Interesse unterscheiden können, wird empfohlen, die Mikroskopeinstellungen unter mehreren Versuchsbedingungen zu testen, um sicherzustellen, dass unter keiner der Versuchsbedingungen ein Bleichen stattfindet. - Sobald die Mikroskopeinstellungen abgeschlossen sind, beladen Sie das Mikroskop mit den einfarbigen Kontrollobjektträgern und finden Sie eine repräsentative Färbung für jeden Fluorophor. Richten Sie einen Z-Stack ein, indem Sie unterhalb des Samples fokussieren und die Starttaste im Z-Stack-Menü drücken, dann oberhalb des Samples fokussieren und die Endtaste drücken.
- Wählen Sie die Anzahl der Schritte und den Abstand zwischen den Schritten (und verwenden Sie die gleiche Anzahl von Schritten und Parametern wie Pixeldichte und Bittiefe, die zum Abbilden der vollständig gefärbten Dias verwendet werden), und drücken Sie dann die Starttaste.
HINWEIS: Im Idealfall sollten diese Bilder die hellsten Signale von echter Färbung enthalten, die auf dem Objektträger zu finden sind. - Nachdem die Einzelfarbregler abgebildet wurden, verwenden Sie dieselben Mikroskopeinstellungen, um alle vollständig gefärbten Proben abzubilden.
3. Fluoreszenz-Kompensation
- Öffnen Sie .lif-Dateien in Software 1 und klicken Sie auf die Registerkarte Prozess.
- Wählen Sie das Modul Farbstofftrennung und dann die Option Automatische Farbstofftrennung . Wählen Sie die manuelle Methode für die Farbtrennung aus, und wählen Sie keine Neuskalierung.
- Öffnen Sie eines der einfarbigen Kontrollbilder. Überprüfen Sie die einfarbigen Steuerelemente manuell auf spektrales Durchscheinen von einem Kanal zum nächsten, indem Sie Graustufenfenster für jeden Kanal beobachten, um Hinweise auf ein Signal in ungeeigneten Kanälen zu finden. Verwenden Sie bei Bedarf die Schaltfläche "Automatisch skalieren ", um schwaches Durchscheinen zu erkennen.
- Beginnen Sie mit dem Entfernen des Durchscheins, indem Sie Zahlen (in der Regel 0-1, wobei 0 für keinen Durchbluten und 1 für 100 % Durchbluten steht) manuell in die Matrix auf dem Bildschirm eingeben und dann auf Anwenden drücken, um zu testen, ob das Durchscheinen ausreichend entfernt wurde. Dies sollte so lange wiederholt werden, bis die Signale in allen anderen Kanälen mit Ausnahme des interessierenden Kanals auf Hintergrundpegel reduziert wurden.
HINWEIS: Eine Überkompensation führt zu ungenauen Ergebnissen, daher muss die Fluoreszenz auf den Hintergrundwert reduziert werden, darf aber nicht darunter liegen. Überkompensierte Daten erscheinen als ein Kanal mit einem Signal und andere Kanäle mit Schwarzen Löchern, wo das Signal von Interesse ist (Abbildung 2). - Sobald eines der einfarbigen Steuerelemente abgeschlossen ist, notieren Sie die Werte in der Matrix, setzen Sie die Matrix zurück und wechseln Sie zum nächsten einfarbigen Steuerelement. Wiederholen Sie diesen Vorgang für jedes einzelne Farbsteuerelement.
HINWEIS: Der erste Farbstoff befindet sich in der Zeile von Farbstoff 1, und die Menge an Fluoreszenz, die von Farbstoff 1 entfernt werden muss, befindet sich in den angrenzenden Spalten. - Assemblieren Sie alle von jedem Steuerelement erfassten Werte in einer Matrix und wenden Sie diese Matrix auf eine vollständig gefärbte Probe an. Wenn die Separation korrekt aussieht, wenden Sie die Einstellungen auf alle Bilder im Test an, andernfalls bewerten Sie die Steuerelemente für einzelne Farben neu.
HINWEIS: Eine effektive Kompensation sollte den größten Teil des Durchscheinens über Kanäle hinweg beseitigen, wie in Abbildung 3 zusammengefasst und dargestellt. - Speichern Sie alle kompensierten Dateien in einer separaten .lif-Datei.
4. Identifizierung von Zellkernen und Zellen mit Ilastik
- Öffnen Sie die kompensierten .lif-Dateien in FIJI20. Aktivieren Sie die Anzeige von Stapeln in einem Hyperstack und wählen Sie ein Bild aus.
- (Fakultativ) Verwenden Sie ein Auswahlwerkzeug wie das Rechteckwerkzeug, um die Teile des Bildes einzuschließen, die analysiert werden sollen, und Bereiche auszuschließen, die nicht analysiert werden. Schneiden Sie das Bild mit der Bild- > Zuschneidefunktion zu.
HINWEIS: Das Reduzieren der Bildgröße ist in den nachfolgenden Schritten nützlich, um den RAM-Verbrauch (Random Access Memory) zu reduzieren und die Verarbeitungsgeschwindigkeit zu erhöhen. - Speichern Sie das Bild als .tif Datei. Speichern Sie das Bild auch als hdf5-Datei, indem Sie das ilastik-Plugin für FIJI verwenden, indem Sie auf Plugins > ilastik > Export hdf5 klicken. Speichern Sie die Datei als "Name der Wahl".h5 mit 0 Komprimierung.
- Öffnen Sie Ilastik21 und erstellen Sie ein Pixelklassifizierungsprojekt. Verwenden Sie die Funktion Neue hinzufügen > Separate Bilder hinzufügen , um mindestens 3 repräsentative Bilder hinzuzufügen, indem Sie neue Eingabedaten hinzufügen.
- Klicken Sie mit der rechten Maustaste auf eines der Bilder, wählen Sie Eigenschaften bearbeiten und ändern Sie den Anzeigemodus in Graustufen.
- Wählen Sie auf der linken Seite des Bildschirms die Registerkarte für die Funktionsauswahl aus. Klicken Sie auf Merkmale auswählen und wählen Sie alle Merkmale bis zum höchsten Sigma-Wert aus, den das Programm Ihnen erlaubt, und drücken Sie dann OK.
- Wählen Sie nun die Registerkarte "Training" aus. Verwenden Sie das Pinselwerkzeug (während die erste Beschriftung ausgewählt ist), um einzelne Zellen von Interesse hervorzuheben (Abbildung 4), sodass das Innere (Zytoplasma und Zellkern) und die Zellmembran in die Hervorhebung einbezogen werden.
- Wiederholen Sie dies für alle Trainingsstichproben, bei denen alle interessierenden Zelltypen ausgewählt sind und aus jeder Probe mehrere Zellen mit unterschiedlichen Intensitäten, Morphologien und Lokalisationen im Gewebe ausgewählt werden sollten.
HINWEIS: Abhängig von den Markern, die für einen einzelnen Satz von Färbungen ausgewählt werden, kann es intrazelluläre Färbungen oder Zellmembranfärbungen für die interessierenden Zellen geben oder nicht. Unabhängig davon, welche Marker verwendet werden, stellen Sie sicher, dass das Highlight die äußerste Färbung auf den interessierenden Zellen und das Zellinnere umfasst, unabhängig davon, ob eine intrazelluläre Färbung verwendet wird. - Verwenden Sie als Nächstes die zweite Beschriftung, um alles hervorzuheben, was nicht die interessierenden Zellen sind (Abbildung 4). Dies kann Leerräume, unmarkierte Zellkerne oder Gewebe umfassen, das keine interessanten Marker aufweist. Stellen Sie sicher, dass der Raum unmittelbar neben den interessierenden Zellen einbezogen wird, um das Erlernen der genauen Zellgrenzen zu fördern.
- Verwenden Sie die Live-Update-Funktion , um die Qualität des Trainings für Bilder von Interesse zu bewerten. Wenn das Training unzureichend ist, setzen Sie das Training in den Schritten 4.7 bis 4.9 fort. Andernfalls klicken Sie bei Zufriedenheit auf die Registerkarte Prediction Export .
- Legen Sie die Quelle auf Wahrscheinlichkeiten fest. Klicken Sie auf die Schaltfläche Einstellungen für den Export von Bildern auswählen und stellen Sie dann den Parameter c auf export 0-1 ein (unter der Annahme, dass Beschriftung 1 die gewünschten Parameter hervorhebt).
- Konvertieren Sie den Datentyp in eine 16-Bit-Ganzzahl und exportieren Sie ihn als hdf5-Datei in das gewünschte Verzeichnis mit dem Namen von Interesse (wir empfehlen die Verwendung von {nickname} mask.h5), und drücken Sie dann OK.
- Klicken Sie auf die Registerkarte Stapelverarbeitung . Klicken Sie auf die Schaltfläche Rohdatendateien auswählen, wählen Sie alle .h5-Dateien von Interesse aus, die verarbeitet werden müssen, und drücken Sie dann Öffnen. Klicken Sie auf Alle Dateien verarbeiten.
- Wiederholen Sie die Schritte 4.4 bis 4.13, nur dass Sie anstelle der Identifizierung von Zellen nun die Zellkerne identifizieren und alles andere ausschließen (Abbildung 4). Exportieren Sie dies als separate Vorhersage zur Identifizierung von Zellkernen.
- Öffnen Sie Ilastik und erstellen Sie ein neues Datenkonvertierungsprojekt. Fügen Sie eine repräsentative hdf5-Datei auf der Registerkarte Eingabedaten hinzu, indem Sie die Funktion Neues hinzufügen > Separates Bild hinzufügen verwenden.
- Klicken Sie auf die Registerkarte Datenexport und wählen Sie die Schaltfläche Exportbildeinstellungen auswählen, wobei die Quelle auf Eingabe eingestellt ist. Ändern Sie das Format in mehrseitiges TIFF, wählen Sie mit der Auswahlfunktion einen Speicherort aus und legen Sie den Namen der Datei auf {Spitzname} fest. Drücken Sie OK.
- Klicken Sie auf die Registerkarte Stapelverarbeitung und wählen Sie alle Ihre hdf5-Dateien mit der Schaltfläche Eingabedatendateien auswählen aus und klicken Sie auf die Schaltfläche Alle Dateien verarbeiten .
- Öffnen Sie Software 2 (siehe Materialtabelle) und fügen Sie alle in den Schritten 4.3 und 4.17 erzeugten tif/tiff-Dateien über die Schaltfläche Dateien hinzufügen hinzu. Wählen Sie einen Speicherort für die Dateiausgabe aus und klicken Sie auf die Schaltfläche Alle starten , um alle Dateien in IMS-Dateien zu konvertieren. Die Dateien sind nun bereit für die Analyse in Software 3 (siehe Materialtabelle).
5. Analyse in Software 3
- Öffnen Sie eines der Bilder, das alle fluoreszierenden Flecken enthält. Wählen Sie Bearbeiten-> Kanäle hinzufügen und fügen Sie die IMS-Dateien hinzu, die den Masken entsprechen, die über Zellkerne und Zellkörper erstellt wurden. Wiederholen Sie diesen Vorgang für alle Proben.
- Wählen Sie die Schaltfläche Neue Zellen hinzufügen aus. Wählen Sie die Option Zellkern und Zelle erkennen und fahren Sie fort.
- Wählen Sie die Maske über den Kernen als Quellkanal für die Erkennung von Kernen aus, und wählen Sie die erweiterte Option zum Aufteilen von Kernen nach Kernpunkten. Wählen Sie einen Wert für den Kerndurchmesser) und fahren Sie fort.
HINWEIS: Der optimale Wert für den Kerndurchmesser muss getestet werden, indem die Simulation ausgeführt und manuell ausgewertet wird, ob das Programm nicht genügend Kerne spaltet (mehrere Kerne werden gruppiert) oder ob es eine Überteilung gibt (ein Kern wird in mehr als einen aufgeteilt). - Wählen Sie einen Wert für die Qualitätsbewertung aus, und fahren Sie fort. In der Regel ist es am besten, alle Zellkerne einzubeziehen, da das Weglassen von Kernen dazu führen kann, dass mehrere Zellen zu einer verschmolzen werden.
- Wählen Sie einen Schwellenwert für die Erstellung von Kernoberflächen und fahren Sie dann fort. Stellen Sie sicher, dass alle Kerne, auch solche, die dunkler sind, noch in den Schwellenwert einbezogen werden. Stellen Sie den Schwellenwert nicht zu niedrig an, da dies die Größe der Kerne überschätzen oder möglicherweise Trümmer als Kernfärbung enthalten kann.
- Wählen Sie einen Wert für die Mindestanzahl von Voxeln pro Kern und fahren Sie fort. In der Regel ist es am besten, alles aufzubewahren, es sei denn, Ablagerungen oder Nicht-Kerne können mit diesem Parameter ausgeschlossen werden.
- Wählen Sie die Option Zellgrenze und Zytoplasma aus, wählen Sie die Maske über den Zellen aus, die als Quellkanal für die Erkennung von Zellkörpern dienen soll, und fahren Sie dann fort.
- Wählen Sie einen Zellenschwellenwert aus, der alle interessierenden Zellen genau erfasst, aber nicht so niedrig ist, dass er den Hintergrund oder eine unspezifische Erkennung von Zellen einschließt. Wählen Sie die Option aus, um berührende Zellen so zu teilen, dass es einen Zellkern pro Zelle gibt, der durch den Abstand zu den Kernen getrennt ist. Wählen Sie auch, Zellen auf Zellkernen zu erweitern und dann fortzufahren.
- Wählen Sie einen Wert für die Mindestanzahl von Voxeln pro Zelle aus, und fahren Sie fort. In der Regel ist es am besten, alles zu behalten, es sei denn, Schmutz oder Nicht-Zellen können mit diesem Parameter ausgeschlossen werden.
- Bewerten Sie, ob die erstellten Oberflächen über den Zellen korrekt sind, indem Sie prüfen, ob mehrere Zellen miteinander verschmolzen wurden oder ob Zellen fehlen. Wenn die Flächen unzureichend sind, gehen Sie zurück zu Schritt 5.2 und probieren Sie neue Parameter aus, bis die optimalen Einstellungen ermittelt sind.
- Sobald die besten Einstellungen festgelegt sind, klicken Sie im linken Menü auf das Objekt der erstellten Zellen , klicken Sie auf die Registerkarte Erstellung und klicken Sie auf die Schaltfläche Parameter für Batch speichern , um die verwendeten Einstellungen zu speichern.
- Öffnen Sie mehrere Fenster von Software 3 und öffnen Sie in jedem Fenster separate Bilder. Drücken Sie in jedem Fenster die neue Schaltfläche Zellen , wählen Sie die gespeicherten Einstellungen im Menü "Favoritenerstellungsparameter " aus und weisen Sie jedes Fenster an, bis zum Abschluss ausgeführt zu werden. Wiederholen Sie dies so oft wie nötig, bis alle Proben analysiert sind.
HINWEIS: Wenn Sie mehrere Instanzen von Software 3 ausführen, wird diese Analyse schneller durchgeführt, aber die Anzahl der Fenster, die parallel ausgeführt werden können, hängt von der Verarbeitungsgeschwindigkeit der Central Processing Unit (CPU) und der Menge an RAM ab, auf der der Computer ausgeführt wird, auf dem die Analyse ausgeführt wird. - Sobald alle Stichproben abgeschlossen sind, klicken Sie auf das erstellte Zellenobjekt im linken Menü in jedem Bild und klicken Sie auf die Registerkarte Statistiken. Klicken Sie auf die Schaltfläche Liste der sichtbaren Statistikwerte konfigurieren , deaktivieren Sie alle Statistiken mit Ausnahme des Werts für den Mittelwert der Zellenintensität und drücken Sie dann OK.
- Klicken Sie für jedes Bild, das analysiert werden muss, auf der Registerkarte "Statistiken" auf die Schaltfläche "Alle Statistiken in Datei exportieren " und speichern Sie die Dateien an einem Speicherort.
6. Histoflowzytometrische Analyse und Kartierung von Populationen auf Gewebeschnitten
- Öffnen Sie Anaconda und JupyterLab innerhalb von Anaconda. Öffnen Sie als Nächstes das Skript, das in der ergänzenden Codierungsdatei 1 bereitgestellt wird. Führen Sie den Code über das Menü Ausführen aus, und wählen Sie Alle Zellen ausführen aus, oder führen Sie die Zellen einzeln in der Reihenfolge aus, indem Sie Ausgewählte Zellen ausführen auswählen.
- Wenn Sie aufgefordert werden, die Quelldatei einzugeben, geben Sie das Dateiverzeichnis für die exportierten Fluoreszenzwerte ein, die in Schritt 5.14 generiert wurden. Das Dateiverzeichnis erhalten Sie, indem Sie sich die Eigenschaften einer der exportierten .csv Dateien ansehen und den Dateipfad in die Eingabeaufforderung kopieren.
- Als nächstes fragt das Programm nach einem Ausgabeort für die verarbeiteten Dateien. Geben Sie das Dateiverzeichnis für einen beliebigen Speicherort ein, der zum Speichern der verarbeiteten Dateien geeignet ist. Das Programm erkennt automatisch die Anzahl der im Ordner enthaltenen Fluoreszenzkanäle.
- Führen Sie den nächsten Abschnitt des Codes aus, um die Daten mit den Namen der einzelnen Kanäle zu versehen. Dieser Code kann nach Bedarf geändert werden, um den Parameter "numberOfChannels" so zu ändern, dass er der Anzahl der in Schritt 6.3 erkannten Kanäle entspricht. Zusätzlich können die Namen der Kanäle für beliebig viele Kanäle geändert werden, indem die Zeile 'ChX':'Marker' so oft wie nötig wiederholt wird und die Namen der Marker geändert werden.
- Beim Ausführen der nächsten Codezeile wird am angegebenen Speicherort eine .csv Datei erstellt, die mit Anmerkungen versehen ist und alle Fluoreszenzparameter im Dataset enthält. Wiederholen Sie die Schritte 6.1 bis 6.5 für jeden Satz von Software 3-Exporten, um .csv Dateien für alle Daten zu erstellen.
- Konvertieren Sie die .csv Dateien in .fcs-Dateien, indem Sie Software 4 öffnen (siehe Materialverzeichnis) und die .csv Dateien in das Programmfenster ziehen. Software 4 beginnt automatisch mit der Konvertierung der Dateien in .fcs-Dateien.
- Doppelklicken Sie auf eines der Beispiele in Software 4, um ein Fenster zu diesem Beispiel zu öffnen. Verwenden Sie die Gating-Tools in Software 4, um eine Gating-Strategie zu erstellen. Es wird empfohlen, mit dem Gating auf einem Marker zu beginnen, der die Abstammungslinie definiert, der alle interessierenden Zellen enthält, dann mit Gates fortzufahren, die zuerst seltenere Teilmengen entfernen, und dann mit den häufigeren Zelltypen zu enden.
- Sobald eine Gating-Strategie eingerichtet ist, testen Sie die Genauigkeit des Gatings, indem Sie mit der rechten Maustaste auf diese Population im Menü klicken und Exportieren auswählen. Exportieren Sie alle Parameter als .csv Datei mit Headern.
- Öffnen Sie JupyterLab in Anaconda. Öffnen Sie als Nächstes das Skript in der ergänzenden Codierungsdatei 2, und führen Sie den Code aus. Wenn Sie aufgefordert werden, den Speicherort der Software 4-Datei einzugeben, geben Sie das Dateiverzeichnis für die exportierte .csv Datei ein, die im vorherigen Schritt generiert wurde. Das Dateiverzeichnis erhalten Sie, indem Sie sich die Eigenschaften der exportierten .csv Datei ansehen und den Dateipfad in die Eingabeaufforderung kopieren.
- Wenn Sie aufgefordert werden, den Ausgabespeicherort hinzuzufügen, wählen Sie ein beliebiges Dateiverzeichnis aus, und stellen Sie sicher, dass das Dateiverzeichnis den Dateinamen am Ende enthält ("Dateiverzeichnis"\filename.txt). Führen Sie den Rest des Codes aus.
- Kopieren Sie den Text in der .txt Datei, und öffnen Sie die entsprechende IMS-Datei in Software 3 für das gleiche Beispiel, auf dem die exportierten Daten basieren. Klicken Sie in der Datei auf das Objekt Zelle , wechseln Sie zur Registerkarte Statistiken, fügen Sie den Text in die Suchleiste ein und starten Sie die Suche. Alle Zellen, die Sie interessieren, werden hervorgehoben.
- Überprüfen Sie manuell, ob alle interessierenden Zellen mit der Gating-Strategie erfasst wurden. Wenn die Anzahl der Zellen unter- oder überschätzt wird (Beispiele in Abbildung 5), gehen Sie zurück zu Schritt 6.7, passen Sie das Gate an und bewerten Sie erneut, ob die Zellen mit dem neuen Gate genau erfasst werden. Wiederholen Sie dies mit allen Gates in der Gating-Strategie und bewerten Sie das Gating an mehreren Samples.
- Wenn alle Gatter validiert sind, exportieren Sie die Daten aus Software 4 mit der Tabelleneditor-Funktion und analysieren Sie die Daten mit der bevorzugten Analysemethode.
HINWEIS: Zellpopulationen können auf die Gewebeschnitte in Software 3 zurückgeführt werden, indem die Gated Population wie in den Schritten 6.8 bis 6.11 identifiziert und dann eine Oberfläche basierend auf den ausgewählten Zellen erstellt wird. Dies kann für jede Gated Population erfolgen, wie in Abbildung 6 und Abbildung 7 dargestellt.
Ergebnisse
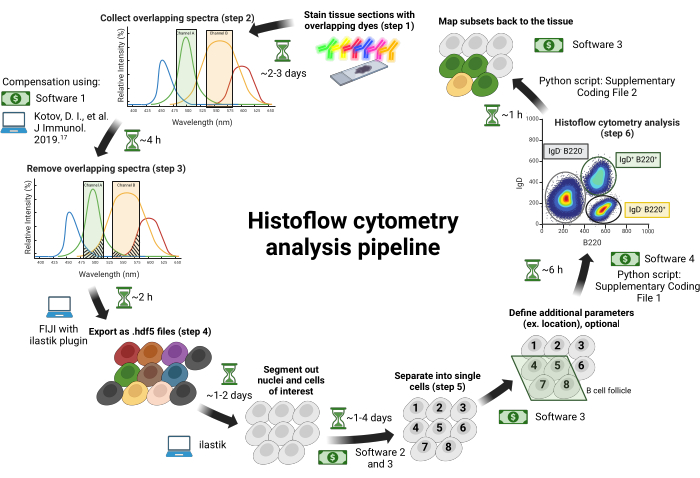
Abbildung 1: Arbeitsablauf der Histoflow-Zytometrie. Die Gewebeschnitte werden mit spektral überlappenden Farbstoffen gefärbt (Schritt 1). Die Bilder werden über einzelne Anregungslaser gesammelt, die mit abstimmbaren Bandpassfiltern gepaart sind, um das spektrale Durchscheinen zwischen den Fluorophoren zu minimieren (Schritt 2). Das spektrale Du...
Diskussion
Hier wird der Einsatz der Histoflow-Zytometrie beschrieben, eine Technik, die bereits zuvor validiert wurde18. Es wird gezeigt, dass bei der Färbung von Gewebeschnitten mit spektral überlappenden Farbstoffen dieses Durchscheinen über Kanäle mittels spektraler Kompensation entfernt werden kann, was dazu führt, dass eine größere Anzahl von Fluoreszenzparametern eindeutig aufgelöst wird, als dies normalerweise mit herkömmlichen Methoden möglich wäre. Da Histologiebilder mit hohen Parameter...
Offenlegungen
Die Autoren haben keine finanziellen Interessenkonflikte.
Danksagungen
Wir danken der Hotchkiss Brain Institute Advanced Microscopy Platform für die Bildgebungsinfrastruktur und -expertise. RWJ wurde durch ein Postdoktorandenstipendium des Eyes High-Programms der University of Calgary und durch ein uneingeschränktes Bildungsstipendium der Multiple Sclerosis Society of Canada und Roche Canada unterstützt. VWY erhielt eine Gehaltsunterstützung aus dem Tier-1-Programm des Canada Research Chair. Diese Arbeit wurde durch Betriebsmittel des Canadian Institutes of Health Research Grant 1049959, des Multiple Sclerosis Society of Canada Grant 3236 und des US-Verteidigungsministeriums des Congressionally Directed Multiple Sclerosis Research Program unterstützt. Abbildung 1 wird mit BioRender.com erstellt. Die in dieser Veröffentlichung angepassten Zahlen wurden ursprünglich im Journal of Immunology veröffentlicht. Rajiv W. Jain, David A. Elliott und V. Wee Yong. 2023. Einzelzellanalyse von hochparametrischen histologischen Bildern mit Histoflow-Zytometrie. J. Immunol. 210: 2038-2049. Urheberrecht © [2023]. Die American Association of Immunologists, Inc.
Materialien
| Name | Company | Catalog Number | Comments |
| 100% Ethanol | Sigma | 676829-1L | |
| 4% PFA | Electron Microscopy Sciences | 157-4 | |
| Anaconda | N/A | N/A | https://www.anaconda.com/download |
| Bovine Serum Albumin | Sigma | A4503-50G | |
| Cold fish stain gelatin | Sigma | G7765 | |
| Collating multichannel data from Imaris.ipynb script | N/A | N/A | https://github.com/elliottcalgary/Histoflow-Cytometry-Analysis- |
| Convert FlowJo output to txt file for Cell selection in Imaris.ipynb script | N/A | N/A | https://github.com/elliottcalgary/Histoflow-Cytometry-Analysis- |
| Donkey anti-rat Alexa Fluor 647 | JacksonImmunoResearch | 712-605-153 | 1:300 concentration |
| Donkey anti-rat DyLight 405 | Jackson ImmunoResearch | 712-475-153 | 1:200 concentration |
| Donkey Serum | JacksonImmunoResearch | 017-000-001 | |
| F(ab')2-Goat anti-Mouse IgG PerCP-eFluor 710 | Thermofisher | 46-4010-82 | 1:25 concentration |
| FIJI | N/A | N/A | https://imagej.net/software/fiji/ |
| FlowJo | FlowJo LLC | Software 4 | |
| Fluorescence spectraviewer | https://www.thermofisher.com/order/fluorescence-spectraviewer/#!/ | ||
| Fluoromount-G | Southern Biotech | 0100-01 | |
| Fresh frozen human tonsil sections | amsbio | HF-707 | |
| Glass coverslip | VWR | 48393 106 | |
| Goat anti-human IgA Alexa Fluor 488 | JacksonImmunoResearch | 109-546-011 | 1:400 concentration |
| Goat anti-human IgG Cy3 | JacksonImmunoResearch | 709-166-098 | 1:400 concentration |
| Goat anti-human IgM Dylight 405 | JacksonImmunoResearch | 109-476-129 | 1:300 concentration |
| Goat anti-rabbit A546 | Thermo Fisher Scientific | A-11035 | 1:250 concentration |
| Goat anti-rabbit IgG PE-Alexa Fluor 610 | Thermofisher | A-20981 | 1:250 concentration |
| Horse Serum | Sigma | H1138 | |
| Ilastik | N/A | N/A | https://www.ilastik.org/ |
| Ilastik FIJI plugin | N/A | N/A | https://www.ilastik.org/documentation/fiji_export/plugin |
| Imaris File Converter | Oxford Instruments | Software 2 | |
| Imaris with cell module | Oxford Instruments | Software 3 | |
| kimwipe | Kimtech | 34155 | |
| LasX Life Science software | Leica | Software 1 | |
| Mouse anti-human CD20 | VWR | CA95024-322 | 1:40 concentration |
| Mouse anti-human CD38 APC-R700 | BD Biosciences | 564980 | 1:20 concentration |
| Normal Goat Serum | JacksonImmunoResearch | 005-000-001 | |
| Normal Mouse Serum | JacksonImmunoResearch | 015-000-001 | |
| Normal Rabbit Serum | JacksonImmunoResearch | 011-000-001 | |
| Normal Rat Serum | JacksonImmunoResearch | 012-000-120 | |
| Nuclear Yellow | Abcam | ab138903 | Dissolve in DMSO at a concentration of 2 mg/ml and store at 4°C in the dark |
| PAP pen | Cedarlane | MU22 | |
| PBS | Gibco | 10010-023 | |
| Rabbit anti-human Ki67 | Abcam | ab15580 | 1:500 concentration |
| Rabbit anti-mouse Iba1 | Wako | 019-19741 | 1:500 concentration |
| Rat anti-human Blimp1 | Thermofisher | 14-5963-82 | 1:40 concentration |
| Rat anti-mouse B220 Alexa Fluor 647 | BioLegend | 103226 | 1:250 concentration |
| Rat anti-mouse CD138 | Biolegend | 142502 | 1:200 concentration |
| Rat anti-mouse CD3 PE-eFluor 610 | Thermo Fisher Scientific | 61-0032-82 | 1:40 concentration |
| Rat anti-mouse CD4 Alexa Fluor 488 | BioLegend | 100529 | 1:200 concentration |
| Rat anti-mouse CD45 allophycocyanin-R700 | BD Biosciences | 565478 | 1:50 concentration |
| Rat anti-mouse IgD PerCP-eFluor 710 | Thermo Fisher Scientific | 46-5993-82 | 1:50 concentration |
| SP8 Confocal microscope | Leica | ||
| Triton X-100 | Sigma | X100-500ml | |
| Trueblack | Biotium | 23007 | |
| Tween-20 | Sigma | P7949-500ml | |
| Ultracomp ebeads | Thermofisher | 01-2222-42 |
Referenzen
- Bar-Or, A., Li, R. Cellular immunology of relapsing multiple sclerosis: interactions, checks, and balances. Lancet Neurol. 20 (6), 470-483 (2021).
- Borst, K., Dumas, A. A., Prinz, M. Microglia: Immune and non-immune functions. Immunity. 54 (10), 2194-2208 (2021).
- Han, R. T., Kim, R. D., Molofsky, A. V., Liddelow, S. A. Astrocyte-immune cell interactions in physiology and pathology. Immunity. 54 (2), 211-224 (2021).
- Absinta, M., et al. A lymphocyte-microglia-astrocyte axis in chronic active multiple sclerosis. Nature. 597 (7878), 709-714 (2021).
- Piwecka, M., Rajewsky, N., Rybak-Wolf, A. Single-cell and spatial transcriptomics: Deciphering brain complexity in health and disease. Nat Rev Neurol. 19 (6), 346-362 (2023).
- Schirmer, L., et al. Neuronal vulnerability and multilineage diversity in multiple sclerosis. Nature. 573 (7772), 75-82 (2019).
- Jain, R. W., Yong, V. W. B cells in central nervous system disease: Diversity, locations and pathophysiology. Nat Rev Immunol. 22 (8), 513-524 (2022).
- Lassmann, H. Multiple sclerosis pathology. Cold Spring Harb Perspect Med. 8 (3), (2018).
- Yong, V. W. Microglia in multiple sclerosis: Protectors turn destroyers. Neuron. 110 (21), 3534-3548 (2022).
- Sharma, S., Boyer, J., Teyton, L. A practitioner's view of spectral flow cytometry. Nat Methods. 21 (5), 740-743 (2024).
- Longo, S. K., Guo, M. G., Ji, A. L., Khavari, P. A. Integrating single-cell and spatial transcriptomics to elucidate intercellular tissue dynamics. Nat Rev Genet. 22 (10), 627-644 (2021).
- Ramaglia, V., et al. Multiplexed imaging of immune cells in staged multiple sclerosis lesions by mass cytometry. Elife. 8, e48051 (2019).
- Bolognesi, M. M., et al. Multiplex staining by sequential immunostaining and antibody removal on routine tissue sections. J Histochem Cytochem. 65 (8), 431-444 (2017).
- Radtke, A. J., et al. IBEX: An iterative immunolabeling and chemical bleaching method for high-content imaging of diverse tissues. Nat Protoc. 17 (2), 378-401 (2022).
- Radtke, A. J., et al. IBEX: A versatile multiplex optical imaging approach for deep phenotyping and spatial analysis of cells in complex tissues. Proc Natl Acad Sci U S A. 117 (52), 33455-33465 (2020).
- Gerner, M. Y., Kastenmuller, W., Ifrim, I., Kabat, J., Germain, R. N. Histo-cytometry: A method for highly multiplex quantitative tissue imaging analysis applied to dendritic cell subset microanatomy in lymph nodes. Immunity. 37 (2), 364-376 (2012).
- Kotov, D. I., Pengo, T., Mitchell, J. S., Gastinger, M. J., Jenkins, M. K. Chrysalis: A new method for high-throughput histo-cytometry analysis of images and movies. J Immunol. 202 (1), 300-308 (2019).
- Jain, R. W., Elliott, D. A., Yong, V. W. Single-cell analysis of high-parameter histology images using histoflow cytometry. J Immunol. 210 (12), 2038-2049 (2023).
- JoVE Science Education Database. General Laboratory Techniques. Histological Sample Preparation for Light Microscopy. JoVE. , (2023).
- Schindelin, J., et al. Fiji: An open-source platform for biological-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Berg, S., et al. ilastik: Interactive machine learning for (bio)image analysis. Nat Methods. 16 (12), 1226-1232 (2019).
- Reichard, A., Asosingh, K. Best practices for preparing a single cell suspension from solid tissues for flow cytometry. Cytometry A. 95 (2), 219-226 (2019).
- Ruhlandt, D., et al. Absolute quantum yield measurements of fluorescent proteins using a plasmonic nanocavity. Commun Biol. 3 (1), 627 (2020).
- Combs, C. A., Shroff, H. Fluorescence microscopy: A concise guide to current imaging methods. Curr Protoc Neurosci. 79, 1-25 (2017).
- Elliott, A. D. Confocal microscopy: Principles and modern practices. Curr Protoc Cytom. 92 (1), e68 (2020).
- Jonkman, J., Brown, C. M., Wright, G. D., Anderson, K. I., North, A. J. Tutorial: Guidance for quantitative confocal microscopy. Nat Protoc. 15 (5), 1585-1611 (2020).
- McNally, J. G., Karpova, T., Cooper, J., Conchello, J. A. Three-dimensional imaging by deconvolution microscopy. Methods. 19 (3), 373-385 (1999).
- Stringer, C., Wang, T., Michaelos, M., Pachitariu, M. Cellpose: A generalist algorithm for cellular segmentation. Nat Methods. 18 (1), 100-106 (2021).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten