JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
Histoflow Cytometry를 이용한 High-parameter Histology 이미지 생성 및 분석
요약
여기에는 면역형광 현미경으로 5개 이상의 형광 파라미터를 이미지화하는 데 사용할 수 있는 방법이 설명되어 있습니다. 이러한 이미지에서 단일 세포를 추출하고 유세포 분석과 같은 게이팅 전략을 통해 단일 세포 분석을 수행하기 위한 분석 파이프라인이 간략하게 설명되어 있으며, 이를 통해 조직 절편의 세포 subset을 식별할 수 있습니다.
초록
중추 신경계(CNS)에서 유래한 것과 같은 조직 절편의 면역 세포 다양성을 조사하기 위해 조직학을 사용하는 것은 한 번에 이미징할 수 있는 형광 매개변수의 수에 의해 매우 제한됩니다. 대부분의 면역 세포 subset은 단백질 마커의 복잡한 조합을 사용하여 유세포 분석을 사용하여 정의되었으며, 종종 결정적으로 식별하기 위해 4개 이상의 매개변수가 필요하며, 이는 대부분의 기존 현미경의 기능을 벗어납니다. 유세포 분석은 조직을 해리하고 공간 정보를 잃기 때문에 복잡한 세포 유형의 역할을 조사하는 동안 공간 정보를 유지할 수 있는 기술이 필요합니다. 여기에서는 스펙트럼적으로 중첩되는 형광단의 신호를 수집하고 스펙트럼 불혼합을 사용하여 각 개별 형광단의 신호를 분리함으로써 이미징할 수 있는 형광 파라미터의 수를 확장하는 방법을 만들어 이러한 문제를 해결합니다. 그런 다음 이러한 이미지는 분석 파이프라인을 사용하여 고파라미터 조직학 이미지를 취하고 이러한 이미지에서 단일 세포를 추출하여 각 세포의 고유한 형광 특성을 단일 세포 수준에서 분석할 수 있습니다. 그런 다음 유세포 분석과 같은 게이팅 전략을 사용하여 세포를 subset으로 프로파일링하고 조직학 섹션에 다시 매핑하여 세포의 존재도를 정량화할 뿐만 아니라 세포 환경과 상호 작용하는 방식을 규명할 수 있습니다. 전반적으로, 조직학 섹션에서 복잡한 면역 집단을 연구하기 위해 histoflow 세포분석을 사용하는 것의 단순성과 잠재력이 입증되었습니다.
서문
면역 체계와 신경교세포의 세포에 의해 유발되는 염증은 각 집단이 다른 1,2,3의 활동을 촉진할 수 있는 CNS의 만성 장애에 기여할 수 있습니다. 면역 체계가 중추신경계의 이러한 요소와 상호 작용하여 중추신경계 염증을 촉진하는 방법을 이해하는 것은 현재 주요 관심 주제이며 단세포 RNA 염기서열 분석과 같은 고분자 기술에 의해 크게 촉진되었습니다. 단일 세포 RNA 염기서열 분석을 통해 우리는 여러 CNS 질환에서 신경교세포와 면역 체계 사이에 광범위한 통신이 발생한다는 것을 발견했습니다 4,5,6. 이러한 상호 작용이 이러한 장애에 어떻게 영향을 미치는지 이해하는 것은 이러한 질병의 생물학을 설명하는 데 중요합니다.
단일 세포 염기서열 분석 분석의 한 가지 문제는 이러한 기술을 사용하여 단일 세포 또는 핵을 얻기 위해 조직을 파괴해야 하므로 공간 정보가 완전히 손실된다는 것입니다. 조직에서 세포가 어디에 존재하는지 아는 것은 염증을 유발하는 세포의 역할을 이해하는 데 중요합니다. 예를 들어, B 세포와 같은 면역 세포는 신경 염증 중에 CNS에 농축 될 수 있습니다. 그러나 그들은 CNS 실질에 거의 들어가지 않고 대신 CNS 장벽7에 집중합니다. 그들의 국소화를 감안할 때, 이러한 세포가 CNS 실질의 신경교세포와 물리적으로 상호 작용하여 CNS 염증에 기여할 가능성은 낮으며, 이는 신경교세포와 가질 수 있는 모든 상호 작용이 분비된 인자를 통해 발생할 것임을 시사합니다. 또한, 중추신경계 장애에서 발생하는 병리학은 종종 구조 8,9를 가지고 있어 조직 내 세포의 국소화가 세포가 장애에 적극적으로 기여하고 있는지 아니면 방관자인지를 비판적으로 결정할 수 있습니다. 따라서 병리학에서 세포의 역할을 평가하기 위해 공간 방향을 사용하는 것이 필수적입니다.
조직 내 세포 연구는 일반적으로 면역조직화학(immunohistochemistry) 또는 면역 형광 현미경(immune fluorescence microscopy)을 사용하여 수행되어 왔습니다. 이러한 기술의 문제점은 일반적으로 최대 4개의 매개변수만 동시에 이미지화할 수 있다는 것입니다. 이는 유세포 분석 및 단일 세포 RNA 염기서열 분석 분석을 통해 많은 세포 집단이 식별을 위해 두 개 이상의 매개변수를 필요로 한다는 것을 알고 있기 때문에 이러한 기술의 주요 제한 사항입니다. 또한, 필요한 파라미터의 수는 세포 유형(10)의 특정 서브셋을 찾을 때 전형적으로 증가한다. 따라서 세포의 부분 집합이 조직 내에서 어떻게 상호 작용할 수 있는지 연구하기 위해 표준 이미징 기술을 사용하는 것은 비실용적입니다.
이 문제는 공간 RNA 염기서열분석(spatial RNA sequencing)11 및 이미징 질량 세포분석(imaging mass cytometry)12과 같은 공간 정보를 유지할 수 있는 새로운 고-파라미터 방법을 통해 부분적으로 극복되었다. 이러한 기술은 유용하지만 널리 사용할 수 없고, 3차원 데이터를 2차원으로 줄이고, 실행하는 데 상당한 전문 지식이 필요한 등 몇 가지 문제가 있습니다. 순차적 염색(sequential staining)으로 알려진 또 다른 기법은 조직을 한 세트의 항체로 염색한 후 다른 항체 세트로 염색하기 전에 이전 항체 세트를 비활성화하는 것으로, 특수 장비나 전문 지식 없이도 고-파라미터 조직학을 달성할 수 있다 13,14,15. 그러나 순차적 염색은 매우 노동 집약적일 수 있으며 많은 현미경 검사 시간이 필요하므로 개인용 현미경이 없는 실험실에는 실용적이지 않을 수 있습니다. 따라서 널리 사용 가능하고 적시에 현미경에서 한 번에 이미지화할 수 있는 형광 파라미터의 수를 확장할 수 있는 기술이 필요합니다.
일단 높은 파라미터 데이터가 수집되면, 또 다른 문제가 발생한다: 기존의 이미지 분석 방법으로는 데이터를 성공적으로 분석할 수 없을 것이다. 수동 카운팅 또는 임계값과 같은 기법은 분석이 단일 파라미터로 구성되거나 여러 마커가 중첩 신호만 카운팅되는 동일한 국소화를 갖는 경우에만 실행 가능합니다. 이러한 제한으로 인해 기존 분석은 매개 변수가 높은 데이터 세트로 작업하기에 적합하지 않습니다. 이러한 데이터 세트의 성공적인 분석은 조직학 이미지에서 단일 세포를 분할한 다음 세포 유형을 식별하기 위해 유세포 분석과 같은 게이팅 전략을 수행함으로써 달성되었습니다16,17. 그러나 이러한 분석에 영향을 미치는 또 다른 문제는 이러한 기술이 물리적으로 접촉하는 세포를 정확하게 분리할 수 있는 방법을 사용하지 않기 때문에 관심 있는 모든 셀이 물리적으로 서로 분리되어 있는 데이터 세트에서만 작동한다는 것입니다. 따라서, 세포가 물리적으로 접촉되어 있더라도 조직학 단면에 대한 단일 세포 분석을 수행할 수 있는 새로운 방법이 필요합니다.
이 기사에서는 이전에 소개된 histoflow cytometry라는 간단한 프로토콜에 대해 설명합니다.18 이 프로토콜은 널리 사용 가능한 현미경을 사용하여 동시에 이미징할 수 있는 형광 파라미터의 수를 확장합니다. 이 프로토콜은 스펙트럼적으로 겹치는 염료로 조직을 염색한 다음 스펙트럼 보정을 사용하여 겹치는 채널에서 블리드스루를 제거하여 선명한 단일 염색을 얻는 방식으로 작동합니다. 고파라미터 조직학 이미지의 분석을 용이하게 하기 위해, 유세포분석과 같은 게이팅 전략을 사용하여 세포를 별개의 집단으로 분류하기 위해 조직 절편에서 단일 세포를 추출하는 상세한 분석 파이프라인이 설명되어 있습니다. 이 프로토콜은 세포가 확산되어 존재하는 조직과 세포가 밀접하게 압축된 조직에서 작동하므로 이 기술은 항상성 및 신경염증 모두에서 CNS와 같은 조직의 연구에 다재다능합니다. 따라서 Histoflow 유세포분석은 공간 정보를 유지하면서 세포를 정의하기 위해 여러 세포 마커가 필요한 복잡한 세포 유형 간의 상호 작용을 연구하는 데 유용한 기술입니다.
프로토콜
이 프로토콜은 조직학을 위한 조직 절편을 다루지 않습니다. 조직학을 위해 조직을 절편하는 방법에 대한 설명은 Jain et al.18 또는19 를 참조하십시오. 이 프로토콜은 유리 슬라이드의 모든 단면 조직과 함께 사용할 수 있습니다. 이 논문은 앞서 설명한 바와 같이 면역이 있는 동물로부터 분리한 서혜부 림프절을 사용한다18. 이 프로토콜의 절차와 일정은 그림 1에 요약되어 있습니다. 이 연구에 사용된 시약 및 장비의 세부 사항은 재료 표에 나열되어 있습니다.
1. 조직 절편 염색
참고: 연구자가 관심 있는 모든 항체 라벨로 조직을 염색하는 것 외에도, 연구자는 인접하거나 동일한 조직 슬라이드에 단일 색상 컨트롤(사용하려는 각 형광 파라미터에 대해 하나씩)을 준비해야 하며, 여기서 각 단일 색상 컨트롤은 하나의 형광 파라미터의 신호를 생성하는 데 필요한 시약으로 염색됩니다.
- 파마 및 차단 완충액 및 파마 및 염색 완충액을 제조한다(조성물에 대한 표 1 참조).
- 유리 슬라이드의 조직 부분을 실온에서 해동하고 물티슈를 사용하여 결로를 닦아냅니다. 소수성 마커를 사용하여 염색해야 하는 조직의 경계를 윤곽을 그리고 마크를 건조시킵니다.
- 조직 절편을 인산염 완충 식염수(PBS)에 1분 동안 담가 조직을 재수화합니다. PBS를 털어내고 티슈를 직접 만지지 않고 물티슈를 사용하여 과도한 PBS를 제거합니다.
- 조직을 완전히 덮을 수 있도록 4%의 파라포름알데히드(PFA)를 충분히 추가하여 조직을 고정합니다. 실온에서 20분 동안 배양합니다.
주의: PFA는 독성이 있을 수 있으므로 흄 후드 내에서 취급해야 합니다.
알림: 이 단계는 신선 냉동 조직에만 필요합니다. 조직이 이미 고정된 경우 이 단계를 무시하십시오. PFA 고정은 여기에 설명되어 있지만 실험의 필요에 따라 다른 방법을 사용하여 조직을 고정할 수 있습니다. - PBS 욕조에서 티슈를 부드럽게 흔들어 1분 동안 씻은 다음 욕조에서 슬라이드를 제거합니다. PBS를 교체하고 세탁을 3회 더 반복합니다.
- 과도한 PBS를 털어내고 티슈를 직접 만지지 않고 물티슈를 사용하여 과도한 PBS를 제거합니다.
- 프로토콜의 1.12단계에서 사용해야 하는 2차 항체와 2차 항체가 파생된 동물 종을 결정합니다. 이 동물의 혈청을 Perm에 추가하고 차단 완충액을 각 종에 대해 최종 농도 5%로 하여 Perm과 차단 완충액을 강화합니다.
참고: 2차 항체를 사용하면 신호 강도를 증폭하는 데 도움이 됩니다. 따라서 약한 신호를 생성하는 1차 항체는 2차 항체와 쌍을 이루어 신호를 증폭하는 것이 좋습니다. 염색되는 모든 항체 표적이 2차 항체가 필요하지 않을 만큼 충분히 밝은 경우 1.7-1.12단계 및 1.15단계를 건너뛸 수 있습니다. - 각 조직을 덮을 수 있도록 충분한 강화 Perm과 차단 완충액을 추가하여 항체의 비특이적 결합을 차단합니다. 빛으로부터 보호되는 가습 챔버에서 실온에서 1-8시간 동안 슬라이드를 배양합니다.
- 실험적으로 결정된 희석액에서 Perm 및 염색 완충액에 비접합 1차 항체를 추가합니다(1차 항체의 종 또는 면역글로불린 동형은 모두 달라야 하며 연구자가 1.12단계에서 사용하려는 2차 항체로 구별할 수 있어야 함).
- 파마와 차단 완충액을 버리고 티슈에 직접 닿지 않고 물티슈를 사용하여 여분의 완충액을 제거합니다. 조직을 덮을 수 있도록 1차 항체가 있는 충분한 Perm과 염색 완충액을 조직에 추가하고 슬라이드를 4°C에서 12-48시간 동안 배양합니다.
참고: 연구자는 항체 배양을 위한 최적의 시간을 결정해야 합니다. - 차단 완충액 및 염색 완충액을 준비합니다(표 1).
- 실험적으로 결정된 희석에서 1.11단계에서 준비한 염색 완충액에 2차 항체를 추가합니다. 사용된 2차 항체는 1.9단계에서 사용된 것과 동일한 종 또는 항체 동형을 표적으로 해야 합니다.
- 담금질 완충액을 준비합니다( 표 1 참조).
참고: 이 단계는 조직의 자가형광을 줄이는 데 도움이 됩니다. 필요하지 않은 경우 1.13 및 1.14 단계를 건너뛸 수 있습니다. - 1.5-1.6단계에 설명된 대로 티슈를 씻습니다. 그런 다음 슬라이드의 조직을 덮을 수 있을 만큼 충분한 담금질 완충액을 추가하고 슬라이드를 실온에서 1-2분 동안 배양합니다.
- 1.5-1.6단계에 설명된 대로 티슈를 씻습니다. 그런 다음 1.12단계에서 준비한 염색 완충액에 희석된 2차 항체를 조직에 첨가하여 조직이 염색 완충액으로 완전히 덮이도록 합니다. 슬라이드를 4 °C에서 2-8 시간 동안 배양합니다.
- 1.18단계에서 사용할 항체 종(각 종의 5%)에서 추출한 50μL의 일반 혈청을 1.11단계에서 준비한 차단 완충액에 첨가하여 차단 용액을 준비합니다.
- 1.5-1.6단계에 설명된 대로 티슈를 씻습니다. 그런 다음 1.16단계에서 준비한 일반 혈청이 포함된 차단 용액을 조직에 추가하여 염색 완충액으로 완전히 덮이도록 합니다. 슬라이드를 실온에서 1-8시간 동안 배양합니다.
- 1.11단계에서 준비한 염색 완충액에 실험적으로 결정된 희석액에서 형광단 복합 1차 항체를 첨가하여 염색 용액을 준비합니다. 또한 핵 황색과 같은 핵 염색을 실험적으로 결정된 희석으로 염색 용액에 첨가합니다.
참고: 1.18-1.19단계는 연구자가 신호 증폭을 위해 2차 항체가 필요하지 않은 추가 항체 라벨을 사용해야 하는 경우에만 필요합니다. primary conjugated antibody를 사용하지 않는 경우 1.12단계에서 핵 염색을 추가할 수 있습니다. - 차단 완충액을 털어내고 티슈를 직접 만지지 않고 물티슈를 사용하여 과도한 완충액을 제거합니다. 1.18단계에서 준비한 염색 용액을 조직에 충분히 추가하여 조직을 덮고 4°C에서 12-48시간 동안 배양합니다.
- 1.5-1.6단계에 설명된 대로 티슈를 씻습니다. 기포가 유입되지 않도록 각 슬라이드에 마운턴트 5방울을 떨어뜨리고 유리 커버슬립을 티슈 위에 부드럽고 천천히 기울여 티슈를 마운턴트로 완전히 덮고 모든 기포를 제거합니다. 이미지를 촬영할 준비가 될 때까지 슬라이드를 4°C의 어두운 곳에 보관하십시오.
2. 조직 단면 이미징
참고: 이 단계에서 조직 절편은 이미징하려는 모든 조건에 대한 모든 관심 항체로 염색되며, 단색 대조군은 각 절편에서 하나의 형광 파라미터로 염색됩니다. 이미징은 특정 범위의 빛을 감지하도록 조정할 수 있는 여러 검출기가 있는 현미경에서 수행해야 하며, 이상적으로는 여기를 위해 가능한 한 많은 레이저 라인에 액세스할 수 있어야 합니다.
- 완전히 염색된 슬라이드 중 하나를 현미경에 삽입하고 데이터 수집에 사용하기 위한 대물렌즈를 사용하여 단면을 이미지화하도록 현미경을 설정합니다.
- 조직이 염색된 fluorophores를 기반으로 이러한 fluorophore를 자극하는 데 필요한 excitation laser line을 식별하고 가장 많은 양의 신호를 포함하는 각 fluorophore의 방출 범위를 식별합니다.
참고: 온라인 형광 스펙트럼 보기 도구(예: 재료 표 참조)는 형광단의 최적 여기 파장을 식별하고 형광단 검출을 위한 최적의 범위를 식별하는 데 유용할 수 있습니다. histoflow cytometry에 사용되는 fluorophore는 각 형광단이 fluorophore에 의해 자극되는 최적의 레이저 라인에서 다르거나 최적의 방출 파장 범위에 따라 다르도록 선택해야 합니다. excitation/emission 파장이 더 가까운 fluorophores는 분리하기가 더 어렵습니다. 이 연구에 사용된 형광단은 결과 섹션에 자세히 설명되어 있습니다. - 컨포칼 현미경에서 소프트웨어 1( 재료 표 참조)을 열고 상단의 구성 탭을 선택합니다. 그런 다음 왼쪽에 있는 하드웨어 단추를 선택하고 비트 깊이 를 16비트로 변경합니다.
참고: 데이터 수집에는 16비트 데이터와 같은 더 높은 비트 심도를 사용하는 것이 권장되며, 이렇게 하면 스펙트럼 보상의 정확도가 향상됩니다. 또한 더 높은 픽셀 밀도, 더 많은 라인 평균, z-stack의 더 많은 스텝은 스펙트럼 분리의 품질을 향상시킵니다. - 화면 상단의 Acquire 탭을 선택하고 이미지의 픽셀 밀도를 포함하도록 형식 설정을 입력합니다(일반적으로 해상도가 높을수록 더 나은 결과를 생성함).
- 사용 가능한 경우, 이 기능이 적절하게 보정된 경우 양방향 X 획득을 선택하여 이미징 시간을 단축하십시오.
- 컨포칼 현미경(일반적으로 400-600nm 사이)의 핀홀 값을 선택하면 일반적으로 값이 작을수록 더 높은 품질의 조직유세포 분석 결과를 얻을 수 있지만, 핀홀 값이 작을수록 이미지를 획득하기가 더 어려워지고 이미지를 생성하기 위해 추가 이미징 시간이나 더 높은 여기 강도가 필요할 수 있습니다.
- 왼쪽 메뉴 상단에 있는 Show Sequential Scan Panel(순차 스캔 패널 표시 )을 선택하여 Sequential scan 옵션을 엽니다. '+' 기호를 사용하여 추가 시퀀스를 설정하며, 각 시퀀스에는 2.2단계에서 결정된 하나의 여기 파장이 포함됩니다.
- 화면 중앙에 있는 관심 레이저와 연결된 ON 버튼을 클릭하여 필요한 모든 레이저를 켜면 ON 상태로 전환됩니다. 각 특정 시퀀스로 이동하여 화면 중앙의 레이저 출력을 원하는 값으로 높이면 각 시퀀스에 하나의 여기 파장이 사용됩니다.
- 각 시퀀스에서 화면 하단 부분에 필요한 만큼의 검출기를 켜 서 해당 레이저 라인에서 이미징할 모든 형광단을 이미징합니다. 검출기의 범위를 두 번 클릭하고 수집해야 하는 파장의 상위 및 하위 범위를 입력하여 검출기의 범위를 조정합니다.
알림: 가능한 경우 고감도 감지기를 사용하는 것이 권장되며, 이상적으로는 필수는 아니지만 모든 채널을 동일한 유형의 감지기에서 수집해야 합니다. - 라이브 버튼을 누른 다음 샘플에 초점을 맞춥니다. 소프트웨어의 이미지 왼쪽에 있는 Auto Scale 버튼을 사용하여 채널이 신호로 적절하게 포화되어 있는지 평가합니다. 사용자는 자동 스케일링이 최대값(16비트 데이터의 경우 65535)에 도달하지 않지만 모든 채널의 포화에 매우 가까워질 때까지 각 시퀀스에서 게인 값과 레이저 강도를 조정해야 합니다.
참고: 서로 다른 형광단의 밝기가 다를 수 있고 다른 항체 표적의 풍부함도 다를 수 있으므로 두 형광단이 동일한 레이저에 의해 자극되는 경우 검출기의 감도를 밝은 신호의 경우 낮은 감도, 희미한 신호의 경우 높은 감도로 조정해야 합니다(이 시나리오에서는 두 형광단이 모두 영향을 받기 때문에 레이저 강도는 변경할 수 없습니다). 최상의 결과를 얻으려면 실험의 각 채널에서 나오는 형광 신호의 균형을 이루어 각 채널이 거의 동일한 양의 신호를 갖도록 해야 하며, 이를 통해 다른 채널로의 스펙트럼 블리드스루를 줄이고 더 강한 신호를 얻을 수 있습니다. 샘플마다 염색되는 관심 마커의 양이 다를 수 있으므로 여러 실험 조건에서 표백이 발생하지 않는지 확인하기 위해 여러 실험 조건에서 현미경 설정을 테스트하는 것이 좋습니다. - 현미경 설정이 완료되면 현미경에 단일 색상 제어 슬라이드를 로드하고 각 형광단에 대한 대표적인 염색을 찾습니다. 샘플 아래에 초점을 맞추고 z 스택 메뉴에서 시작 버튼을 누른 다음 샘플 위에 초점을 맞추고 종료 버튼을 눌러 Z 스택을 설정합니다.
- 단계 수와 단계 사이의 거리를 선택한 다음(완전히 염색된 슬라이드를 이미지화하는 데 사용할 픽셀 밀도 및 비트 심도와 같은 동일한 수의 단계 및 매개변수 사용) 시작 버튼을 누릅니다.
알림: 이상적으로 이러한 이미지에는 슬라이드에서 찾을 수 있는 실제 염색의 가장 밝은 신호가 포함되어야 합니다. - 단일 색상 컨트롤을 이미지화한 후 동일한 현미경 설정을 사용하여 완전히 염색된 모든 샘플을 이미지화합니다.
3. 형광 보상
- 소프트웨어 1에서 .lif 파일을 열고 프로세스 탭을 클릭합니다.
- Dye Separation(염료 분리) 모듈을 선택한 다음 Automatic Dye Separation(자동 염료 분리) 옵션을 선택합니다. 염료 분리(Dye Separation)에 대한 수동 방법을 선택하고 스케일링 없음(no rescaling)을 선택합니다.
- 단색 제어 이미지 중 하나를 엽니다. 부적절한 채널에서 신호의 증거를 찾기 위해 각 채널에 대한 회색조 창을 관찰하여 한 채널에서 다음 채널로의 스펙트럼 블리드스루에 대한 단일 색상 컨트롤을 수동으로 검사합니다. 필요한 경우 Auto Scale 버튼을 사용하여 약한 블리드 스루를 확인합니다.
- 화면의 매트릭스에 숫자(일반적으로 0-1, 0은 블리드스루가 없음을 나타내고 1은 100% 블리드스루를 나타냄)를 수동으로 입력하여 블리드스루를 제거하기 시작합니다. 관심 채널을 제외한 다른 모든 채널의 신호가 배경 수준으로 줄어들 때까지 이 작업을 반복해야 합니다.
참고: 과잉 보정은 부정확한 결과로 이어질 수 있으므로 형광을 배경 수준으로 줄여야 하지만 그 이하로는 안 됩니다. 과보상된 데이터는 신호가 있는 한 채널과 관심 신호가 있는 블랙홀이 있는 것으로 보이는 다른 채널로 나타납니다(그림 2). - 단색 컨트롤 중 하나가 완료되면 매트릭스에 값을 기록한 다음 매트릭스를 재설정 하고 다음 단색 컨트롤로 이동합니다. 각 단일 색상 컨트롤에 대해 이 프로세스를 반복합니다.
알림: 첫 번째 염료는 염료 1 행에 있고 염료 1에서 제거해야 하는 형광의 양은 인접한 열에 있습니다. - 각 대조군에서 획득한 모든 값을 하나의 매트릭스로 조립하고 이 매트릭스를 완전히 염색된 샘플에 적용합니다 . 분리가 정확해 보이면 실험의 모든 이미지에 설정을 적용하고, 그렇지 않으면 단일 색상 컨트롤을 다시 평가합니다.
참고: 효과적인 보정은 그림 3에 요약된 대로 채널 전반에 걸쳐 대부분의 블리드스루를 제거해야 합니다. - 모든 보정된 파일을 별도의 .lif 파일에 저장합니다.
4. ilastik을 이용한 핵 및 세포 식별
- FIJI20에서 보정된 .lif 파일을 엽니다. 하이퍼스택에서 스택 보기를 활성화 하고 이미지를 선택합니다.
- (선택 사항) 사각형 도구와 같은 선택 도구를 사용하여 분석할 이미지 부분을 포함하고 분석하지 않을 영역을 제외합니다. 이미지 > 자르기 기능을 사용하여 이미지를 자릅니다.
참고: 이미지 크기를 줄이는 것은 이후 단계에서 RAM(Random-Access Memory) 소비를 줄이고 처리 속도를 높이는 데 유용합니다. - 이미지를 .tif 파일로 저장합니다. 또한, 플러그인 > ilastik > 내보내기를 클릭하여 FIJI용 ilastik 플러그인을 사용하여 이미지를 hdf5 파일로 저장하십시오. 파일을 0으로 압축하여 "name of choice".h5로 저장합니다.
- Ilastik21 을 열고 픽셀 분류 프로젝트를 만듭니다. 새 입력 데이터 추가로 이동하여 새로 추가 > 별도의 이미지 추가 기능을 사용하여 3개 이상의 대표 이미지를 추가합니다.
- 이미지 중 하나를 마우스 오른쪽 버튼으로 클릭하고 속성 편집을 선택한 다음 디스플레이 모드를 그레이스케일로 변경합니다.
- 화면 왼쪽에서 기능 선택 탭을 선택합니다. Select features(기능 선택 )를 클릭하고 프로그램에서 사용할 수 있는 가장 높은 시그마 값까지 모든 기능을 선택한 다음 OK를 누릅니다.
- 이제 교육 탭을 선택합니다. 페인트 브러시 도구( 레이블 1 을 선택한 상태에서)를 사용하여 내부(세포질 및 핵)와 세포막이 하이라이트에 포함되도록 관심 있는 개별 셀(그림 4)을 강조 표시합니다.
- 관심 있는 모든 세포 유형이 선택되고 조직의 다양한 강도, 형태 및 국소화를 가진 여러 세포를 각 샘플에서 선택해야 하는 모든 훈련 샘플에서 이 작업을 반복합니다.
참고: 개별 염색 세트에 대해 선택한 마커에 따라 관심 세포에 대한 세포 내 염색 또는 세포막 염색이 있을 수도 있고 없을 수도 있습니다. 어떤 마커를 사용하든, 세포 내 염색이 사용되는지 여부에 관계없이 하이라이트에 관심 세포의 가장 바깥쪽 염색이 포함되고 세포 내부가 포함되어야 합니다. - 그런 다음 레이블 2 를 사용하여 관심 있는 셀이 아닌 모든 항목을 강조 표시합니다(그림 4). 여기에는 빈 공간, 라벨링되지 않은 핵 또는 관심 마커가 없는 조직이 포함될 수 있습니다. 정확한 세포 경계에 대한 학습을 촉진하기 위해 관심 세포에 바로 인접한 공간을 포함해야 합니다.
- 라이브 업데이트 기능을 사용하여 관심 이미지에 대한 교육의 품질을 평가하십시오. 훈련이 충분하지 않은 경우 4.7-4.9단계로 훈련을 계속합니다. 그렇지 않고 만족스러우면 Prediction Export 탭을 클릭합니다.
- 소스를 확률로 설정합니다. Choose Export Image Settings 버튼을 클릭한 다음 c 매개 변수를 0-1로 내보냅니다(레이블 1이 관심 있는 매개 변수를 강조한다고 가정).
- 데이터형을 16비트 정수로 변환하고 hdf5 파일로 관심 있는 이름으로 관심 있는 디렉터리에 내보낸 다음({nickname} mask.h5 사용 권장) OK를 누릅니다.
- Batch Processing 탭을 클릭합니다. 원시 데이터 파일 선택 버튼을 누르고 처리가 필요한 모든 .h5 파일을 선택한 다음 열기를 누릅니다. 모든 파일 처리를 클릭합니다.
- 4.4단계에서 4.13단계를 반복하되 세포를 식별하는 대신 핵을 식별하고 다른 모든 단계를 제외합니다(그림 4). 이를 세포핵을 식별하기 위한 별도의 예측으로 내보냅니다.
- Ilastik을 열고 새 데이터 변환 프로젝트를 만듭니다. Add New > Add Separate Image(개별 이미지 추가) 기능을 사용하여 대표적인 hdf5 파일을 입력 데이터(Input data) 탭에 추가합니다.
- Data Export(데이터 내보내기) 탭을 클릭하고 소스가 입력으로 설정된 Choose Export Image Settings(내보내기 이미지 설정 선택) 버튼을 선택합니다. 형식을 여러 페이지로 된 tiff로 변경하고, select 함수로 저장 위치를 선택하고, 파일 이름을 {nickname}으로 설정합니다. OK를 누릅니다.
- 배치 처리 탭을 클릭하고 입력 데이터 파일 선택 버튼을 사용하여 모든 hdf5 파일을 선택하고 모든 파일 처리 버튼을 누릅니다.
- 소프트웨어 2를 열고( 재료 표 참조) 파일 추가 버튼을 사용하여 4.3 및 4.17 단계에서 생성된 모든 tif/tiff 파일을 추가합니다. 파일 출력 위치를 선택하고 모두 시작 버튼을 눌러 모든 파일을 .ims 파일로 변환합니다. 이제 소프트웨어 3에서 파일을 분석할 준비가 되었습니다( 재료 표 참조).
5. 소프트웨어 분석 3
- 모든 형광 염색이 포함된 이미지 중 하나를 엽니다. Edit-> Add Channels를 선택하고 핵 및 세포체 위에 만들어진 마스크에 해당하는 .ims 파일을 추가합니다. 모든 샘플에 대해 이 작업을 반복합니다.
- 새 셀 추가 단추를 선택합니다. detect nucleus and cell(핵 및 세포 감지) 옵션을 선택하고 앞으로 진행합니다.
- 핵을 검출하기 위한 소스 채널로 사용할 핵에 대한 마스크를 선택하고 시드 포인트로 핵을 분할하기 위한 고급 옵션을 선택합니다. 핵 지름 값을 선택)하고 앞으로 진행합니다.
참고: 핵 직경에 대한 최적 값은 시뮬레이션을 실행하고 프로그램이 충분한 핵을 분할하지 않는지(여러 핵이 함께 그룹화됨) 또는 과도한 분할이 있는지(하나의 핵이 둘 이상으로 분할됨) 여부를 수동으로 평가하여 테스트해야 합니다. - 품질 평가를 위한 값을 선택하고 계속 진행합니다. 일반적으로 핵이 누락되면 여러 세포가 하나로 융합될 수 있으므로 모든 핵을 포함하는 것이 가장 좋습니다.
- 핵 표면을 생성하기 위한 임계값을 선택하고 계속 진행합니다. 더 어두운 핵을 포함한 모든 핵이 여전히 임계값에 포함되어 있는지 확인하십시오. 임계값을 너무 낮게 설정하면 핵의 크기가 과대평가되거나 핵 염색으로 파편이 포함될 수 있으므로 임계값을 너무 낮게 만들지 마십시오.
- 핵당 최소 Voxels 수에 대한 값을 선택하고 계속 진행합니다. 일반적으로 이 매개변수를 사용하여 파편이나 비핵을 제외할 수 없는 한 모든 것을 유지하는 것이 가장 좋습니다.
- Cell boundary and cytoplasm(세포 경계 및 세포질) 옵션을 선택하고 cell body 검출을 위한 소스 채널로 사용할 cell over cells를 선택한 다음 계속 진행합니다.
- 관심 있는 모든 세포를 정확하게 캡처하지만 배경 또는 비특이적 세포 검출을 포함할 정도로 낮지 않은 세포 임계값을 선택합니다. 접촉하는 세포를 분할하여 셀당 하나의 핵이 있고 핵으로부터의 거리만큼 분리되도록 하는 옵션을 선택합니다. 또한 핵에서 세포를 확장 하도록 선택한 다음 계속 진행합니다.
- 셀당 최소 복셀 수에 대한 값을 선택하고 계속 진행합니다. 일반적으로 이 매개변수를 사용하여 파편이나 비셀을 제외할 수 없는 한 모든 것을 유지하는 것이 가장 좋습니다.
- 여러 셀이 함께 융합되었는지 또는 셀이 누락되었는지 확인하여 셀 위에 생성된 표면이 정확한지 평가합니다. 표면이 부적절하면 5.2단계로 돌아가서 최적의 설정이 결정될 때까지 새 매개변수를 시도합니다.
- 최상의 설정이 설정되면 왼쪽 메뉴에서 생성된 셀 개체를 클릭하고 생성 탭을 클릭한 다음 배치를 위한 매개변수 저장 버튼을 클릭하여 사용된 설정을 저장합니다.
- 소프트웨어 3의 여러 창을 열고 각 창에서 별도의 이미지를 엽니다. 각 창에서 새 Cells 버튼을 누르고, Favorite Creation Parameters 메뉴에서 저장된 설정을 선택하고, 각 창이 완료될 때까지 실행되도록 지시합니다. 모든 샘플이 분석될 때까지 필요한 만큼 이 작업을 반복합니다.
참고: 소프트웨어 3의 여러 인스턴스를 실행하면 이 분석이 더 빠르게 진행되지만 병렬로 실행할 수 있는 창의 수는 중앙 처리 장치(CPU) 처리 속도와 분석이 실행되는 컴퓨터의 RAM 양에 따라 달라집니다. - 모든 샘플이 완료되면 각 이미지의 왼쪽 메뉴에서 생성된 셀 개체를 클릭하고 통계 탭을 클릭합니다. Configure list of visible Statistics Values 버튼을 클릭하고 'Cell Intensity Mean' 값을 제외한 모든 통계를 선택 취소한 다음 OK를 누릅니다.
- 분석해야 하는 모든 이미지에 대해 통계 탭에서 모든 통계를 파일로 내보내기 버튼을 클릭하고 파일을 특정 위치에 저장합니다.
6. Histoflow 세포분석 분석 및 조직 절편에 집단을 매핑합니다.
- Anaconda 내에서 Anaconda 및 JupyterLab 을 엽니다. 그런 다음 보충 코딩 파일 1에 제공된 스크립트를 엽니다. 실행 메뉴를 사용하여 코드를 실행하고 모든 셀 실행을 선택하거나, 선택한 셀 실행을 선택하여 셀을 순서대로 개별적으로 실행합니다.
- 소스 파일을 입력하라는 메시지가 표시되면 5.14단계에서 생성된 내보낸 형광 값에 대한 파일 디렉토리를 입력합니다. 파일 디렉토리는 내보낸 .csv 파일 중 하나의 속성을 확인하고 파일 경로를 프롬프트에 복사하여 얻을 수 있습니다.
- 다음으로 프로그램은 처리된 파일의 출력 위치를 묻습니다. 처리된 파일을 저장하기에 적합한 위치에 대한 파일 디렉토리를 입력합니다. 프로그램은 폴더에 포함 된 형광 채널의 수를 자동으로 감지합니다.
- 코드의 다음 섹션을 실행하여 각 채널의 이름으로 데이터에 주석을 추가합니다. 필요에 따라 이 코드를 수정하여 6.3단계에서 검색된 채널 수와 일치하도록 "numberOfChannels" 매개 변수를 변경할 수 있습니다. 또한 'ChX':'Marker' 줄을 필요한 만큼 반복하고 마커 이름을 수정하여 필요한 만큼 채널 이름을 수정할 수 있습니다.
- 다음 코드 줄을 실행하면 지정된 위치에 주석이 추가되고 데이터 세트의 모든 형광 매개변수가 포함된 .csv 파일이 생성됩니다. 각 소프트웨어 3 내보내기 세트에 대해 6.1-6.5단계를 반복하여 모든 데이터에 대한 .csv 파일을 만듭니다.
- 소프트웨어 4( 재료 표 참조)를 열고 .csv 파일을 프로그램 창으로 끌어다 놓아 .csv 파일을 .fcs 파일로 변환합니다. 소프트웨어 4는 자동으로 파일을 .fcs 파일로 변환하기 시작합니다.
- 소프트웨어 4의 샘플 중 하나를 두 번 클릭하여 해당 샘플에 대한 창을 엽니다. 소프트웨어 4의 게이팅 도구를 사용하여 게이팅 전략을 만듭니다. 관심 있는 모든 세포를 포함하는 계통 정의 마커에서 게이팅으로 시작한 다음, 먼저 더 희귀한 하위 집합을 제거하는 게이트로 진행한 다음, 더 풍부한 세포 유형으로 마무리하는 것이 좋습니다.
- 게이팅 전략이 설정되면 메뉴에서 해당 모집단 을 마우스 오른쪽 버튼으로 클릭하고 내보내기를 선택하여 게이팅의 정확도를 테스트합니다. 모든 매개 변수를 헤더가 포함된 .csv 파일로 내보냅니다.
- Anaconda에서 JupyterLab을 엽니다. 그런 다음 추가 코딩 파일 2에 제공된 스크립트를 열고 코드를 실행합니다. 소프트웨어 4 파일의 위치를 입력하라는 메시지가 표시되면 이전 단계에서 생성된 내보낸 .csv 파일의 파일 디렉토리를 입력합니다. 파일 디렉토리는 내보낸 .csv 파일의 속성을 확인하고 파일 경로를 프롬프트에 복사하여 얻을 수 있습니다.
- 출력 위치를 추가하라는 메시지가 표시되면 원하는 파일 디렉터리를 선택하고 파일 디렉터리의 끝에 파일 이름('file directory'\filename.txt)이 포함되어 있는지 확인합니다. 코드의 나머지 부분을 실행합니다.
- .txt 파일의 텍스트를 복사하고 소프트웨어 3에서 내보낸 데이터의 기반이 되는 동일한 샘플에 해당하는 .ims 파일을 엽니다. 파일에서 Cell 개체를 클릭하고, 통계 탭으로 전환하고, 텍스트를 검색 창에 붙여넣고, 검색을 시작합니다. 관심 있는 모든 셀이 강조 표시됩니다.
- gating 전략을 사용하여 관심 있는 모든 셀이 캡처되었는지 수동으로 검사합니다. 셀 수가 과소 평가되거나 과대 평가된 경우( 그림 5 참조) 6.7단계로 돌아가서 게이트를 조정하고 새 게이트로 셀이 정확하게 캡처되었는지 다시 평가합니다. 게이팅 전략의 모든 게이트에 대해 이 작업을 반복하고 여러 샘플에서 게이팅을 평가합니다.
- 모든 게이트가 검증되면 테이블 편집기 기능을 사용하여 소프트웨어 4에서 데이터를 내보내고 선호하는 분석 방법을 사용하여 데이터를 분석합니다.
참고: 세포 집단은 6.8-6.11단계에서와 같이 게이트 집단을 식별한 다음 선택한 세포를 기반으로 표면을 생성하여 소프트웨어 3의 조직 섹션에 다시 매핑할 수 있습니다. 이 작업은 그림 6 및 그림 7과 같이 각 게이트 모집단에 대해 수행할 수 있습니다.
결과
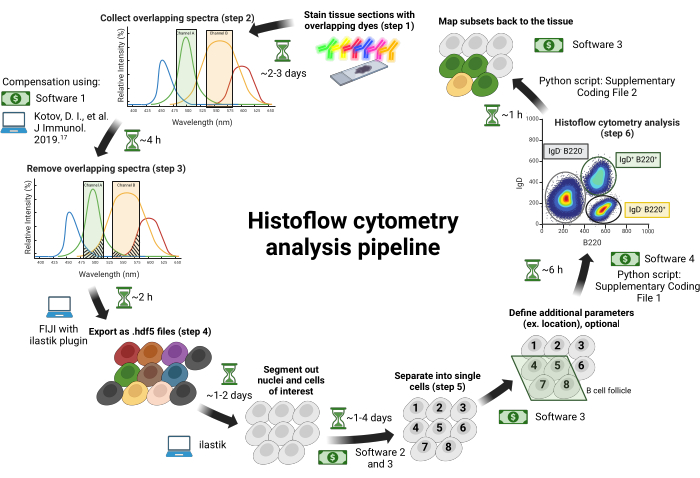
그림 1: Histoflow 유세포분석 워크플로우. 조직 절편은 스펙트럼으로 겹치는 염료로 염색됩니다(1단계). 이미지는 fluorophores 간의 스펙트럼 블리드스루를 최소화하기 위해 tunable bandpass filter와 쌍을 이루는 개별 excitation laser에서 수집됩니다(2단계). 채널 간 스펙트럼 블?...
토론
여기에서는 이전에 검증된 기술인 histoflow cytometry의 사용에 대해 설명합니다(18). 스펙트럼적으로 겹치는 염료로 조직 절편을 염색할 때 스펙트럼 보정을 사용하여 채널을 가로지르는 블리드스루를 제거할 수 있으며, 그 결과 기존 방법을 통해 일반적으로 가능한 것보다 더 많은 수의 형광 매개변수가 명확하게 분해될 수 있음이 입증되었습니다. 고파라미터 조직학 이미지는 기?...
공개
저자는 재정적 이해 상충이 없습니다.
감사의 말
이미징 인프라와 전문 지식을 제공한 Hotchkiss Brain Institute Advanced Microscopy Platform에 감사드립니다. RWJ는 University of Calgary Eyes High 프로그램의 박사후 연구원 기금과 캐나다 다발성 경화증 학회 (Multiple Sclerosis Society of Canada) 및 Roche Canada 무제한 교육 펠로우십의 지원을 받았습니다. VWY는 Canada Research Chair Tier 1 프로그램에서 급여 지원을 받았습니다. 이 연구는 캐나다 보건원(Canadian Institutes of Health) 연구 보조금 1049959, 캐나다 다발성 경화증 학회(Multiple Sclerosis Society of Canada) 보조금 3236 및 미국 국방부(US Department of Defense of the Congressionally Directed Multiple Sclerosis Research Program)의 운영 자금으로 지원되었습니다. 그림 1 은 BioRender.com 로 작성되었습니다. 이 간행물에서 조정된 수치는 원래 The Journal of Immunology에 게재되었습니다. Rajiv W. Jain, David A. Elliott 및 V. Wee Yong. 2023. Histoflow Cytometry를 사용한 High-parameter Histology 이미지의 단일 세포 분석. J. 이뮤놀. 210: 2038-2049. 저작권 © [2023]. 미국 면역학자 협회(American Association of Immunologists, Inc.)
자료
| Name | Company | Catalog Number | Comments |
| 100% Ethanol | Sigma | 676829-1L | |
| 4% PFA | Electron Microscopy Sciences | 157-4 | |
| Anaconda | N/A | N/A | https://www.anaconda.com/download |
| Bovine Serum Albumin | Sigma | A4503-50G | |
| Cold fish stain gelatin | Sigma | G7765 | |
| Collating multichannel data from Imaris.ipynb script | N/A | N/A | https://github.com/elliottcalgary/Histoflow-Cytometry-Analysis- |
| Convert FlowJo output to txt file for Cell selection in Imaris.ipynb script | N/A | N/A | https://github.com/elliottcalgary/Histoflow-Cytometry-Analysis- |
| Donkey anti-rat Alexa Fluor 647 | JacksonImmunoResearch | 712-605-153 | 1:300 concentration |
| Donkey anti-rat DyLight 405 | Jackson ImmunoResearch | 712-475-153 | 1:200 concentration |
| Donkey Serum | JacksonImmunoResearch | 017-000-001 | |
| F(ab')2-Goat anti-Mouse IgG PerCP-eFluor 710 | Thermofisher | 46-4010-82 | 1:25 concentration |
| FIJI | N/A | N/A | https://imagej.net/software/fiji/ |
| FlowJo | FlowJo LLC | Software 4 | |
| Fluorescence spectraviewer | https://www.thermofisher.com/order/fluorescence-spectraviewer/#!/ | ||
| Fluoromount-G | Southern Biotech | 0100-01 | |
| Fresh frozen human tonsil sections | amsbio | HF-707 | |
| Glass coverslip | VWR | 48393 106 | |
| Goat anti-human IgA Alexa Fluor 488 | JacksonImmunoResearch | 109-546-011 | 1:400 concentration |
| Goat anti-human IgG Cy3 | JacksonImmunoResearch | 709-166-098 | 1:400 concentration |
| Goat anti-human IgM Dylight 405 | JacksonImmunoResearch | 109-476-129 | 1:300 concentration |
| Goat anti-rabbit A546 | Thermo Fisher Scientific | A-11035 | 1:250 concentration |
| Goat anti-rabbit IgG PE-Alexa Fluor 610 | Thermofisher | A-20981 | 1:250 concentration |
| Horse Serum | Sigma | H1138 | |
| Ilastik | N/A | N/A | https://www.ilastik.org/ |
| Ilastik FIJI plugin | N/A | N/A | https://www.ilastik.org/documentation/fiji_export/plugin |
| Imaris File Converter | Oxford Instruments | Software 2 | |
| Imaris with cell module | Oxford Instruments | Software 3 | |
| kimwipe | Kimtech | 34155 | |
| LasX Life Science software | Leica | Software 1 | |
| Mouse anti-human CD20 | VWR | CA95024-322 | 1:40 concentration |
| Mouse anti-human CD38 APC-R700 | BD Biosciences | 564980 | 1:20 concentration |
| Normal Goat Serum | JacksonImmunoResearch | 005-000-001 | |
| Normal Mouse Serum | JacksonImmunoResearch | 015-000-001 | |
| Normal Rabbit Serum | JacksonImmunoResearch | 011-000-001 | |
| Normal Rat Serum | JacksonImmunoResearch | 012-000-120 | |
| Nuclear Yellow | Abcam | ab138903 | Dissolve in DMSO at a concentration of 2 mg/ml and store at 4°C in the dark |
| PAP pen | Cedarlane | MU22 | |
| PBS | Gibco | 10010-023 | |
| Rabbit anti-human Ki67 | Abcam | ab15580 | 1:500 concentration |
| Rabbit anti-mouse Iba1 | Wako | 019-19741 | 1:500 concentration |
| Rat anti-human Blimp1 | Thermofisher | 14-5963-82 | 1:40 concentration |
| Rat anti-mouse B220 Alexa Fluor 647 | BioLegend | 103226 | 1:250 concentration |
| Rat anti-mouse CD138 | Biolegend | 142502 | 1:200 concentration |
| Rat anti-mouse CD3 PE-eFluor 610 | Thermo Fisher Scientific | 61-0032-82 | 1:40 concentration |
| Rat anti-mouse CD4 Alexa Fluor 488 | BioLegend | 100529 | 1:200 concentration |
| Rat anti-mouse CD45 allophycocyanin-R700 | BD Biosciences | 565478 | 1:50 concentration |
| Rat anti-mouse IgD PerCP-eFluor 710 | Thermo Fisher Scientific | 46-5993-82 | 1:50 concentration |
| SP8 Confocal microscope | Leica | ||
| Triton X-100 | Sigma | X100-500ml | |
| Trueblack | Biotium | 23007 | |
| Tween-20 | Sigma | P7949-500ml | |
| Ultracomp ebeads | Thermofisher | 01-2222-42 |
참고문헌
- Bar-Or, A., Li, R. Cellular immunology of relapsing multiple sclerosis: interactions, checks, and balances. Lancet Neurol. 20 (6), 470-483 (2021).
- Borst, K., Dumas, A. A., Prinz, M. Microglia: Immune and non-immune functions. Immunity. 54 (10), 2194-2208 (2021).
- Han, R. T., Kim, R. D., Molofsky, A. V., Liddelow, S. A. Astrocyte-immune cell interactions in physiology and pathology. Immunity. 54 (2), 211-224 (2021).
- Absinta, M., et al. A lymphocyte-microglia-astrocyte axis in chronic active multiple sclerosis. Nature. 597 (7878), 709-714 (2021).
- Piwecka, M., Rajewsky, N., Rybak-Wolf, A. Single-cell and spatial transcriptomics: Deciphering brain complexity in health and disease. Nat Rev Neurol. 19 (6), 346-362 (2023).
- Schirmer, L., et al. Neuronal vulnerability and multilineage diversity in multiple sclerosis. Nature. 573 (7772), 75-82 (2019).
- Jain, R. W., Yong, V. W. B cells in central nervous system disease: Diversity, locations and pathophysiology. Nat Rev Immunol. 22 (8), 513-524 (2022).
- Lassmann, H. Multiple sclerosis pathology. Cold Spring Harb Perspect Med. 8 (3), (2018).
- Yong, V. W. Microglia in multiple sclerosis: Protectors turn destroyers. Neuron. 110 (21), 3534-3548 (2022).
- Sharma, S., Boyer, J., Teyton, L. A practitioner's view of spectral flow cytometry. Nat Methods. 21 (5), 740-743 (2024).
- Longo, S. K., Guo, M. G., Ji, A. L., Khavari, P. A. Integrating single-cell and spatial transcriptomics to elucidate intercellular tissue dynamics. Nat Rev Genet. 22 (10), 627-644 (2021).
- Ramaglia, V., et al. Multiplexed imaging of immune cells in staged multiple sclerosis lesions by mass cytometry. Elife. 8, e48051 (2019).
- Bolognesi, M. M., et al. Multiplex staining by sequential immunostaining and antibody removal on routine tissue sections. J Histochem Cytochem. 65 (8), 431-444 (2017).
- Radtke, A. J., et al. IBEX: An iterative immunolabeling and chemical bleaching method for high-content imaging of diverse tissues. Nat Protoc. 17 (2), 378-401 (2022).
- Radtke, A. J., et al. IBEX: A versatile multiplex optical imaging approach for deep phenotyping and spatial analysis of cells in complex tissues. Proc Natl Acad Sci U S A. 117 (52), 33455-33465 (2020).
- Gerner, M. Y., Kastenmuller, W., Ifrim, I., Kabat, J., Germain, R. N. Histo-cytometry: A method for highly multiplex quantitative tissue imaging analysis applied to dendritic cell subset microanatomy in lymph nodes. Immunity. 37 (2), 364-376 (2012).
- Kotov, D. I., Pengo, T., Mitchell, J. S., Gastinger, M. J., Jenkins, M. K. Chrysalis: A new method for high-throughput histo-cytometry analysis of images and movies. J Immunol. 202 (1), 300-308 (2019).
- Jain, R. W., Elliott, D. A., Yong, V. W. Single-cell analysis of high-parameter histology images using histoflow cytometry. J Immunol. 210 (12), 2038-2049 (2023).
- JoVE Science Education Database. General Laboratory Techniques. Histological Sample Preparation for Light Microscopy. JoVE. , (2023).
- Schindelin, J., et al. Fiji: An open-source platform for biological-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Berg, S., et al. ilastik: Interactive machine learning for (bio)image analysis. Nat Methods. 16 (12), 1226-1232 (2019).
- Reichard, A., Asosingh, K. Best practices for preparing a single cell suspension from solid tissues for flow cytometry. Cytometry A. 95 (2), 219-226 (2019).
- Ruhlandt, D., et al. Absolute quantum yield measurements of fluorescent proteins using a plasmonic nanocavity. Commun Biol. 3 (1), 627 (2020).
- Combs, C. A., Shroff, H. Fluorescence microscopy: A concise guide to current imaging methods. Curr Protoc Neurosci. 79, 1-25 (2017).
- Elliott, A. D. Confocal microscopy: Principles and modern practices. Curr Protoc Cytom. 92 (1), e68 (2020).
- Jonkman, J., Brown, C. M., Wright, G. D., Anderson, K. I., North, A. J. Tutorial: Guidance for quantitative confocal microscopy. Nat Protoc. 15 (5), 1585-1611 (2020).
- McNally, J. G., Karpova, T., Cooper, J., Conchello, J. A. Three-dimensional imaging by deconvolution microscopy. Methods. 19 (3), 373-385 (1999).
- Stringer, C., Wang, T., Michaelos, M., Pachitariu, M. Cellpose: A generalist algorithm for cellular segmentation. Nat Methods. 18 (1), 100-106 (2021).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유