Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Eintierische, einröhrige RNA-Extraktion zum Vergleich der relativen Transkriptspiegel mittels qRT-PCR im Bärtierchen Hypsibius exemplaris
In diesem Artikel
Zusammenfassung
In dieser Arbeit wird eine Methode zur schnellen RNA-Extraktion und zum Vergleich auf Transkriptebene zur Analyse der Genexpression im Bärtierchen Hypsibius exemplaris vorgestellt. Diese Hochdurchsatzmethode erfordert mittels physikalischer Lyse ein einzelnes Bärtierchen als Ausgangsmaterial und führt zu einer robusten Produktion von cDNA für die quantitative reverse Transkriptase-Polymerase-Kettenreaktion (qRT-PCR).
Zusammenfassung
Das Bärtierchen Hypsibius exemplaris ist ein aufstrebender Modellorganismus, der für seine Fähigkeit bekannt ist, Umweltextreme zu überleben. Um die molekularen Mechanismen und genetischen Grundlagen einer solchen Extremotoleranz zu erforschen, stützen sich viele Studien auf die RNA-Sequenzierung (RNA-seq), die an Populationen durchgeführt werden kann, die von großen Kohorten bis hin zu einzelnen Tieren reichen. Die Reverse-Transkriptase-Polymerase-Kettenreaktion (RT-PCR) und die RNA-Interferenz (RNAi) werden anschließend verwendet, um die RNA-seq-Befunde zu bestätigen bzw. die genetischen Anforderungen an Kandidatengene zu beurteilen. Solche Studien erfordern eine effiziente, genaue und kostengünstige Methode zur RNA-Extraktion und Messung der relativen Transkriptspiegel mittels quantitativer RT-PCR (qRT-PCR). In dieser Arbeit wird eine effiziente Single-tardigrade-RNA-Extraktionsmethode (STST) vorgestellt, die nicht nur RNA aus einzelnen Bärtierchen zuverlässig isoliert, sondern auch den Zeit- und Kostenaufwand für jede Extraktion reduziert. Diese RNA-Extraktionsmethode liefert Mengen an cDNA, die zur Amplifikation und zum Nachweis mehrerer Transkripte durch quantitative PCR (qRT-PCR) verwendet werden können. Die Methode wurde durch die Analyse dynamischer Veränderungen in der Expression von Genen validiert, die für zwei hitzeschockregulierte Proteine, das Hitzeschockprotein 70 β2 (HSP70 β2) und das Hitzeschockprotein 90α (HSP90α), kodieren, wodurch es möglich ist, ihre relativen Expressionsniveaus bei hitzeexponierten Personen mittels qRT-PCR zu bestimmen. STST ergänzt effektiv die bestehenden Extraktionsmethoden für Bulk- und Einzel-Bärtierchen-RNA und ermöglicht eine schnelle und kostengünstige Untersuchung einzelner Bärtierchen-Transkriptionsspiegel mittels qRT-PCR.
Einleitung
Bärtierchen sind kleine mehrzellige Tiere, die für ihre Fähigkeit bekannt sind, extreme Bedingungen zu überleben, die für die meisten anderen Lebensformen tödlich sind1. Zum Beispiel können diese Tiere fast das 1000-fache der Dosis ionisierender Strahlung überleben, die für den Menschen tödlich ist 2,3,4,5,6,7,8,9,10, fast vollständige Austrocknung 11,12,13,14,15 und Einfrieren ohne Zugabe Kryoprotektiva 16,17,18 und im ausgetrockneten Zustand sogar das Vakuum des Weltraums 19,20. Aufgrund ihrer einzigartigen Fähigkeit, in extremen Umgebungen zu überleben, sind diese Tiere zu grundlegenden Modellen für das Verständnis der Extremotoleranz bei komplexen, mehrzelligen Organismen geworden 1,21,22,23.
Eine stabile genetische Manipulation dieser bemerkenswerten Tiere, einschließlich Transgenese und Genmodifikation in der Keimbahn, blieb bis vor kurzem schwer fassbar24,25. Daher werden die meisten Experimente zur Aufdeckung molekularer Mechanismen der Extremotoleranz durch transkriptionelles Profiling mittels RNA-Sequenzierung durchgeführt. Es existieren viele wertvolle und informative RNA-Sequenzierungsdatensätze für Bärtierchen unter verschiedenen extremen Bedingungen, die von Strahlung 8,9,26,27,28, Hitzestress 29, Froststress12 und Austrocknung 27,30,31,32,33 reichen. Einige dieser Studien haben Methoden zur Extraktion und Reinigung von Massen-RNA verwendet, um unser molekulares Verständnis von Extremotoleranz zu beleuchten. Die Massenextraktion von RNA-Transkripten aus vielen Tieren verhindert jedoch die Analyse der Variation der Genexpression zwischen Individuen, wodurch die potenzielle Fülle verfeinerter Datensätze übersehen wird. Wichtig ist, dass diese Studien oft heterogene Tierpopulationen analysieren, die sowohl Tiere umfassen, die Umweltstressoren überleben, als auch solche, die dies nicht tun. Daher werden diese Studien durch die Mittelung von Expressionsdaten aus mehreren und potenziell dramatisch unterschiedlichen Reaktionszuständen verwirrt. Um dieses Problem zu lösen, entwickelten Arakawa et al., 201634 eine elegante Low-Input-RNA-Seq-Pipeline, die ein RNA-Extraktionskit anwendet, gefolgt von einem linearen PCR-Amplifikationsschritt mit einzelnen 34,35,36 oder mehreren 30,37,38 Tieren als Input. Diese Studien waren grundlegend für unser Verständnis der Bärtierchen-Extremotoleranz22. Interessanterweise wurde dieses Protokoll auch auf die qRT-PCR angewendet, wobei sieben Tiere als Ausgangsmaterialverwendet wurden 24.
In den meisten Modellorganismen wird nach der Identifizierung potenzieller Ziele über RNA-seq eine qRT-PCR durchgeführt, um die durch RNA-seq identifizierten transkriptionellen Veränderungen zu bestätigen und den Expressionszeitverlauf von Kandidatengenen hochauflösend zu beurteilen. Um die Funktion identifizierter Gene zu testen, folgen auf solche Studien häufig der RNAi-vermittelte Knockdown molekularer Ziele39,40 und die Analyse der extremotoleranten Kapazität12,41. Die Wirksamkeit jedes RNAi-Knockdowns wird in der Regel durch qRT-PCR bestätigt, indem die Abnahme der Transkripthäufigkeit direkt überwacht wird. RNAi ist jedoch ein arbeitsintensiver Prozess bei Bärtierchen, da jede dsRNA durch manuelle Mikroinjektion von Individuen verabreicht werden muss 39,40. Aufgrund des geringen Durchsatzes dieser Strategie wäre eine schnelle, kostengünstige RNA-Extraktionsmethode, die für die qRT-PCR von Einzeltieren angepasst ist, für die Bärtierchenforschung sehr wertvoll. Obwohl frühere Methoden zur Extraktion von RNA aus einzelnen Bärtierchen entwickelt wurden, haben diese Protokolle ihre Extraktion nicht mit qRT-PCR kombiniert, sondern sich stattdessen auf auf optische Dichtebasierte Methoden gestützt 12,40,41. Motiviert durch diese Herausforderungen haben wir versucht, ein Protokoll zu entwickeln, das zuverlässig RNA in Quantität und Qualität liefert, die für die qRT-PCR aus einzelnen H. exemplaris verwendet werden kann.
STST basiert auf einem für Caenorhabditis elegans42 entwickelten Einzeltier-RNA-Extraktionsprotokoll und ist für H. exemplaris optimiert. Die Extraktionsmethode besteht aus sechs schnellen Gefrier-Auftau-Schritten, die die Nagelhaut physikalisch aufbrechen und die RNA-Extraktion und die anschließende cDNA-Synthese ermöglichen. Die STST-Methode verkürzt die Extraktionszeit um mehr als das 24-fache im Vergleich zu Bulk-RNA-Extraktionsmethoden, wie von Boothby, 201843 beschrieben, und um 30 % im Vergleich zu einzelnen Bärtierchen-RNA-Extraktionskits, wie von Arakawa et al., 201634 beschrieben. Darüber hinaus wird die Anzahl der Proben-Experimentator-Interaktionen im Vergleich zu RNA-Extraktionskit-Präparaten von 5 auf nur 1 reduziert, wodurch das Risiko einer Kontamination durch exogene Ribonukleasen verringert wird. Bei der Abfrage von hoch exprimierten Genen erzeugt die STST-Methode ausreichend cDNA für 25 quantitative RT-PCR-Reaktionen pro einzelnem Bärtierchen, wobei nur 1 μl des gesamten cDNA-Volumens von 25 μl pro Reaktion benötigt wird. Allerdings müssen die Template-Konzentrationen für Transkripte mit geringerer Abundanz empirisch bestimmt werden.
Die Wirksamkeit der STST-Methode zur Analyse dynamischer Veränderungen in der Genexpression wurde untersucht, indem die differentielle Expression der Gene, die für das Hitzeschockprotein 90α (HSP90α) und das Hitzeschockprotein 70β2 (HSP70β2) kodieren, als Reaktion auf einen kurzfristigen Hitzeschock bei 35 °C für 20 Minuten untersucht wurde. Sowohl HSP70β2 als auch HSP90α werden in den meisten eukaryotischen Organismen nach kurzfristiger Hitzeschockexposition (20 min) schnell hochreguliert42. Die Analyse in H. exemplaris zeigte, dass sowohl die HSP70β2- als auch die HSP90α-kodierende RNAs, die aus einzelnen wärmebehandelten Bärtierchen extrahiert wurden, nach kurzfristiger Hitzeexposition statistisch signifikante Steigerungen der Expression zeigten. Diese Ergebnisse zeigen, dass das STST-Protokoll verwendet werden kann, um dynamische Veränderungen der Genexpression bei einzelnen Tieren im Laufe der Zeit zu analysieren.
Die STST-Extraktionsmethode soll bestehende experimentelle Methoden wie RNA-seq ergänzen, indem sie eine schnelle und kostengünstige RNA-Extraktion und einen anschließenden Vergleich der Transkriptspiegel mittels qRT-PCR ermöglicht. Diese Methode wird auch wertvoll sein, um die Effizienz und Penetranz von RNAi bei manuell injizierten Personen quantitativ zu beurteilen als nur die optische Dichte. Schließlich ist es wahrscheinlich, dass diese Methode aufgrund ihrer ähnlichen kutikulären Strukturen und physikalischen Eigenschaften auch für die Analyse der Genexpression bei anderen Bärtierchenarten wirksam sein wird44.
Access restricted. Please log in or start a trial to view this content.
Protokoll
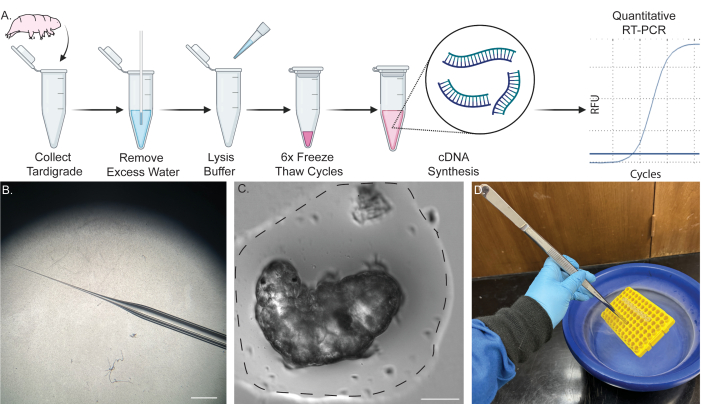
Abbildung 1: Ein-Röhrchen-Pipeline für die RNA-Extraktion aus einem einzelnen Bärtierchen. (A) Schema, das das Protokoll für die RNA-Extraktion aus einem einzelnen Bärtierchen zeigt, einschließlich sechs Frost-Tau-Zyklen und anschließender cDNA-Synthese. Proben können anschließend für die RT-PCR und qRT-PCR verwendet werden. (B) Bild der Mikropipettenverjüngung, die zur Entfernung von Wasser verwendet wird. Maßstabsbalken: 2 mm. (C) Hellfeldbild eines Bärtierchens in einer kleinen Wassermenge (gestrichelte Linie). Die Entfernung des größten Teils des Wassers in der angegebenen Menge ist für eine erfolgreiche Extraktion erforderlich und verhindert eine Verdünnung des Lysepuffers. Maßstab: 50 μm. (D) Das Bild zeigt das Eintauchen von Proben in flüssigen Stickstoff mit einer langen Pinzette, um die Proben schnell und sicher einzufrieren und aufzutauen. Ein Teil der Inhalte wurde in BioRender erstellt. Kirk, M. (2022) BioRender.com/d93s511 Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
HINWEIS: Abbildung 1A zeigt ein Schema des Verfahrens. Detaillierte Informationen zu den Kultivierungsverfahren für Bärtierchen und Algen finden Sie in den zuvor veröffentlichten Berichten 45,46,47.
1. Sterilisation von Quellwasser
- Gießen Sie 2 l Quellwasser aus einem 5-Gallonen-Wasserkrug (siehe Materialtabelle für Einzelheiten) in eine autoklavensichere 2-Liter-Glasflasche.
- Setzen Sie den Verschluss auf die autoklavensichere Flasche und verschließen Sie ihn mit einer kleinen Menge Autoklavenband. Ziehen Sie die Flasche nicht fest; Setzen Sie die Kappe darauf.
- Autoklavieren Sie das Quellwasser 50 Minuten lang in einem Nasszyklus ohne Trocknungsschritt.
- Lassen Sie das Wasser auf Raumtemperatur (RT) kommen und verschließen Sie die Kappe fest, bevor Sie sie bei RT lagern.
2. Ziehen der Mikropipette aus Glas (mit einem Pipettenabzieher)
- Befestigen Sie eine Mikropipette aus Glas (AD: 1 mm, ID: 0,58 mm, Länge: 10 cm) an einem Mikropipettenabzieher. Vermeiden Sie den Kontakt mit dem Heizfaden, da dies die Form der Pipette verändert und den Faden beschädigt.
- Bestimmen Sie das Ziehen der Pipette empirisch für jedes Filament und jeden Pipettenzieher. Als Ausgangspunkt für die Optimierung dienen jedoch 78 °C und ein einzelner Zugschritt mit einem Zuggewicht von 182,2 g.
- Lassen Sie das Filament Hitze und Schwerkraft einwirken, um die Glasmikropipette in zwei Glasmikropipetten mit scharfen Spitzen zu trennen (Abbildung 1B).
- Bewahren Sie diese gezogenen Glasmikropipetten in einer geschlossenen 100-mm-Petrischale mit Wachs oder Ton auf, um sie an Ort und Stelle zu halten und ein Brechen der scharfen Spitzen zu verhindern.
3. Ziehen von Mikropipetten aus Glas (ohne Pipettenabzieher)
- Zünden Sie einen Bunsenbrenner oder eine andere kontrollierte Flammenquelle auf niedriger Stufe an.
- Nehmen Sie eine Mikropipette aus Glas mit einem Ende in jeder Hand.
- Halten Sie die Mitte der Glasmikropipette über die Flamme, bis das Glas zu schmelzen beginnt. Ziehe dann die beiden Enden schnell auseinander. Dadurch entstehen zwei sehr zarte, scharfe Spitzen.
- Brechen Sie die Spitze leicht mit einer sterilen feinen Pinzette.
- Bewahren Sie diese gezogenen Glasmikropipetten in einer geschlossenen 100-mm-Petrischale mit Wachs oder Ton auf, um sie an Ort und Stelle zu halten und ein Brechen der scharfen Spitzen zu verhindern.
4. RNA-Extraktion
- Beziehen Sie 0,5 l flüssigen Stickstoff in einem kryosicheren Behälter.
ACHTUNG: Flüssiger Stickstoff ist kryogenisch und kann Verbrennungen verursachen, wenn er Haut oder Augen ausgesetzt wird. Verwenden Sie bei der Handhabung Schutzkleidung, eine Spritzschutzbrille, Nitrilhandschuhe, Kryohandschuhe, einen Laborkittel und geschlossene Schuhe. Vergewissern Sie sich, dass der Behälter für flüssigen Stickstoff geeignet ist, bevor Sie die Flüssigkeit transportieren. Für diesen Schritt kann auch die Verwendung eines Ethanol-Trockeneisbades möglich sein. - Stellen Sie einen cDNA-Synthese-Mastermix her: eine 10-μl-Lösung, die 1 μl zufälliger Hexamer-Primer, 2 μl DNase, 4 μl 5x RT-Puffer, 1 μl Enzymmix, 1 μl H2O und 1 μl 10 mM dNTPs enthält. Lagern Sie diese Lösung auf Eis.
- Bereiten Sie den Bärtierchen-Lysepuffer (5 mM Tris (pH = 8), 0,5 % (v/v), Detergens 1, 0,5 % (v/v), Detergens 2, 0,25 mM EDTA in sterilem, nukleasefreiem Wasser vor.
HINWEIS: Diese Lösung kann 6 Monate lang auf dem Labortisch gelagert werden. Bewahren Sie jedoch die Sterilität und vermeiden Sie potenzielle RNAse-kontaminierende Quellen. - Aliquot genug Lysepuffer für Extraktionen (2 μL/ Bärtierchen).
- Geben Sie einen RNAse-Inhibitor in die Bärtierchen-Lysepufferlösung bis zu einer Endkonzentration von 4 U/μl.
- Die Lösung wird bei RT auf einer Tischzentrifuge bei einer Geschwindigkeit von 2000 x g 5 s lang vortext und geschleudert, bevor die Lösung auf Eis gelagert wird.
- Mit einer sterilen P1000-Pipette mit Filterspitze werden so viele Bärtierchen, wie für den Versuch benötigt werden, aus der Kultur entnommen und in eine sterile 35-mm-Petrischale gegeben.
HINWEIS: Es können beliebig viele Bärtierchen auf diese Weise verarbeitet werden. In der Regel werden drei Bärtierchen pro Zustand für die Extraktion verarbeitet. - Waschen Sie die Bärtierchen dreimal mit 1 ml autoklaviertem sterilem Quellwasser und einer sterilen P1000-Pipette mit Filterspitze. Das langsame Auf- und Abpipettieren hilft, Algenverunreinigungen zu entfernen.
- Mit einem Präpariermikroskop bei 25- bis 50-facher Vergrößerung wird ein einzelnes Bärtierchen aus dieser gewaschenen Kultur mit einer sterilen P10-Pipette mit Filterspitze in eine neue sterile 35-mm-Petrischale übertragen.
- Verwenden Sie eine sterile P200-Pipette mit Filterspitze, um das einzelne Bärtierchen in 100 μl sterilem nukleasefreiem Wasser zu waschen.
HINWEIS: Dieser Waschschritt wird verwendet, um Verunreinigungen, einschließlich Ribonukleasen, weiter zu entfernen. - Übertragen Sie das gewaschene Bärtierchen mit einer sterilen P10-Pipette mit Filterspitze in 1-2 μl sterilem nukleasefreiem Wasser auf den Boden eines sauberen, sterilen PCR-Röhrchens und achten Sie dabei sorgfältig darauf, dass das Bärtierchen nicht an der Seite der Spitze klebt.
- Visualisieren Sie das Bärtierchen unter einem Präpariermikroskop bei 25-facher Vergrößerung.
- Um die Wasserentnahme zu erleichtern, brechen Sie die Spitze der gezogenen Glasmikropipette leicht außerhalb des Röhrchens. Stellen Sie sicher, dass die Bohrung groß genug ist, um das Wasser hochzuziehen, aber nicht das Bärtierchen.
- Mit der Kapillarwirkung einer gezogenen Glasmikropipette wird das Wasser so lange entfernt, bis das Tier von einer kleinen Wasserblase mit einem Durchmesser von etwa zwei Bärtierchenlängen umgeben ist.
- Überwachen Sie den Wasserentnahmeprozess über das Präpariergerät, um sicherzustellen, dass der Wasserstand angemessen ist und das Bärtierchen hydratisiert bleibt.
HINWEIS: Abbildung 1C zeigt ein Beispiel dafür, wie viel Wasser entfernt werden muss. Dies ist ein entscheidender Schritt. Eine kleine Wasserblase umgibt das Bärtierchen, um ein Austrocknen zu verhindern, aber so viel überschüssiges Wasser wie möglich sollte entfernt werden, um eine Verdünnung des Lysepuffers zu verhindern. Ein Beispiel für die verbleibenden Wasserstände finden Sie in Abbildung 1C. - Unmittelbar nach dem Entfernen des Wassers 2 μl Bärtierchen-Lysepuffer auf den Boden des Röhrchens geben, kurz vortexen und das Röhrchen bei RT 5 s bei 2000 x g auf einer Tischzentrifuge zentrifugieren.
- Legen Sie die Proben, die die Bärtierchen enthalten, sofort in ein PCR-Röhrchengestell und stellen Sie sicher, dass sie vom Gestell fest gehalten werden.
- Fassen Sie das Gestell mit einer langen groben Pinzette an und tauchen Sie das Gestell mit den Proben vorsichtig in den flüssigen Stickstoff, bis es vollständig gefroren ist (Abbildung 1D).
- Nehmen Sie das Gestell vom flüssigen Stickstoff und legen Sie es sofort auf Eis. Lassen Sie die Probe auftauen (dauert ~45 s bis 1 min). Überwachen Sie die Probe alle 15 s, indem Sie sie aus dem Eis nehmen und sichtbar inspizieren. Sobald die Probe sichtbar transparent ist, fahren Sie mit dem nächsten Schritt fort.
- Wiederholen Sie die Schritte 4.18-4.19 noch fünfmal. Für eine maximale Lyse und Extraktion sind insgesamt sechs Gefrier-Auftau-Zyklen erforderlich (Abbildung 2A,B).
- Sobald das Einfrieren und Auftauen abgeschlossen ist, legen Sie die Proben auf Eis und fahren Sie sofort mit dem nächsten Schritt fort. Frieren Sie die Proben zu diesem Zeitpunkt nicht zur Lagerung ein, da dies die verfügbare RNA für die cDNA-Präparation verringert.
5. cDNA-Synthese
- Geben Sie 2 μl cDNA-Synthese-Mastermix in das PCR-Röhrchen mit Bärtierchenlysat. Das Röhrchen kurz schnippen und mit einer Tischzentrifuge 5 s lang bei RT bei 2000 x g drehen, bevor die Proben wieder auf Eis gelegt werden.
- Die Proben werden in einen Thermocycler gegeben und 10 Minuten lang bei 25 °C zu Primern geglüht, 30 Minuten lang bei 55 °C zur reversen Transkription und schließlich Enzyme 5 Minuten lang bei 85 °C hitzeinaktiviert.
- Stellen Sie das Röhrchen nach der Inkubation sofort auf Eis und verdünnen Sie die Probe auf ein Gesamtvolumen von 25 μl, indem Sie 21 μl steriles nukleasefreies Wasser hinzufügen. Bei Transkripten mit niedriger Kopienzahl ist dieser Verdünnungsschritt wie empirisch bestimmt zu ändern.
6. qPCR
- Bestimmen Sie die Annealing-Temperatur des Primer-Sets unter Verwendung von Gesamt-RNA, die aus größeren Mengen von Bärtierchen hergestellt wurde, z. B. mit der in Boothby, 201843 vorgestellten Bulk-Extraktionsmethode.
- Führen Sie einen PCR-Temperaturgradienten durch, um die optimale Annealing-Temperatur zu bestimmen, bevor Sie die qRT-PCR ausführen (für alle in diesem Protokoll verwendeten PCR-Einstellungen siehe Tabelle 1 und Tabelle 2).
- Tauen Sie eine Tube des Indikatorfarbstoffs Super Mix auf Eis auf und isolieren Sie ihn vor Licht. Legen Sie eine 96-Well-qPCR-Platte auf Eis und geben Sie 5 μl Super Mix, 2 μl Wasser, 1 μl jedes Primers (10 μM) und 1 μl cDNA-Produkt in die gewünschte Anzahl von Wells.
- Versiegeln Sie die PCR-Platte mit einer Plattenversiegelung und führen Sie die qRT-PCR mit einer für das Primer-Set geeigneten Glühtemperatur durch (für alle in diesem Dokument verwendeten qRT-PCR-Einstellungen siehe Tabelle 3).
7. Quantifizierung und Interpretation der Ergebnisse
- Vergleichen Sie die Ergebnisse quantitativ mit einem oder mehreren Kontroll-Housekeeping-Genen, von denen erwartet wird, dass ihre Expression unter den auferlegten Bedingungen konstant ist. Für diese Studie wurde das Aktin-Gen verwendet.
- Ermitteln und vergleichen Sie die Ct-Werte oder den Zyklusschwellenwert für jede Vertiefung mit den Ct-Werten der Genreaktionen der Kontrollkontrolle. Berechnen Sie die Faltungsänderung in der Genexpression mit der folgenden Gleichung:
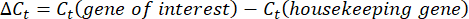
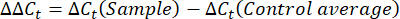
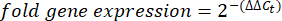
HINWEIS: Die Faltengenexpression wird für jedes Transkript und jedes Bärtierchen als 2-(ΔΔCt)48 aufgetragen. - Um eine grobe Schätzung der Transkriptnummer aus demC-t-Wert zu erhalten, verwenden Sie die folgende Gleichung:
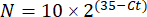
Dabei ist N die Anzahl der Transkripte und 2 die angenommene PCR-Effizienz oder die Faltzunahme der Fluoreszenz pro Zyklus von PCR48.
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
Entwicklung und Optimierung der Extraktion von Single-Tardigrade-RNA
Durch die Anpassung des Protokolls von Ly et al., 201542 für die RNA-Extraktion bei Bärtierchen wird das STST-System optimiert, um die Quantität und Qualität des Präparats zu maximieren (Abbildung 1A). Die RT-PCR wurde für Aktin-Transkripte durchgeführt, wobei die Transkriptausbeute durch Amplifikation einer 527 bp-Region, die die Exons 1 u...
Access restricted. Please log in or start a trial to view this content.
Diskussion
Diese Studie stellt eine effiziente Methode zur Extraktion von RNA für die single-tardigrade qRT-PCR vor. Der direkte Vergleich der STST-Methodik mit einem bestehenden Extraktionskit für bärtierchene RNA zeigte, dass die STST-RNA-Extraktion >200-mal höhere Mengen an Aktin-RNA-Transkripten liefert, die Kosten auf weniger als einen Dollar pro Probe reduziert und die für die Extraktion erforderliche Zeit um 30 % reduziert. Um STST auf eine relevante biologische Fragestellung anzuwenden...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Die Autoren erklären, dass keine Interessenkonflikte offengelegt werden müssen.
Danksagungen
Wir möchten uns bei dem NIH Ruth Kirschstein Fellowship # 5F32AG081056-02 und dem Errett Fisher Post-Doctoral Fellowship bedanken, das Dr. Molly J. Kirk unterstützte, dem Crowe Family Fellowship, das Chaoming Xu unterstützte, und einem Stipendium der University of California, Santa Barbara Academic Senate und NIH Grants #R01GM143771 und #2R01HD081266, die diese Forschungsbemühungen unterstützten. Die Autoren würdigen auch die Nutzung des Biological Nanostructures Laboratory innerhalb des California NanoSystems Institute, das von der University of California, Santa Barbara, und der University of California, Office of the President, unterstützt wird.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| 10 µL Premium Barrier Tips Low Binding, Racked, Sterile | Genesee Scientific | 23-401 | Refered to as Sterile Filter-Tipped P 10 Pipette Tips |
| 1000 µL Premium Pipet Tips, Low Binding, Racked, Sterile | Genesee Scientific | 23-165RS | Refered to as Sterile Filter-Tipped P 1000 Pipette Tips |
| 200 µL Premium Barrier Tips Low Binding, Racked, Sterile | Genesee Scientific | 23-412 | Refered to as Sterile Filter-Tipped P 200 Pipette Tips |
| 4 Star Straight Strong Medium Point Tweezer | Excelta | 00-SA-DC | Refered to as Long forceps |
| 96-Well PCR Rack with Lid Assorted, 5 Racks/Unit | Genesee Scientific | 27-202A | Refered to as PCR Rack |
| Andwin Scientific 3M LEAD FREE AUTOCLAVE TAPE 1" | Thermo Fisher Scientific | NC0802040 | Refered to as Autoclave Tape |
| Autoclave Tape | Thermo Fisher Scientific | AB1170 | Refered to as PCR Plate Seals |
| Benchling v8 | Benchling | N/A | Refered to as Benchling |
| BioRadHard-Shell 96-Well PCR Plate | BioRad | HSS9641 | Refered to as PCR Plate |
| BULWARK FR Lab Coat: | Grainger | 26CF64 | Refered to as Lab Coat |
| C1000 Touch Bio-rad Thermocycler | BioRad | 1851148 | Refered to as Thermocycler |
| C1000 Touch Bio-rad Thermocycler with CFX Optics Module | BioRad | 1845097 | Refered to as qPCR thermocycler |
| Chloroccoccum hypnosporum. | Carolina | 152091 | Refered to as Algae |
| Corning PYREX Reusable Media Storage Bottles | Thermo Fisher Scientific | 06-414-1E | Refered to as 2 L Autoclave-safe Glass Bottle |
| Daigger & Company Vortex-Genie 2 Laboratory Mixer | Thermo Fisher Scientific | 3030A | Refered to as Vortexer |
| Direct-zol Micro Prep | Zymo Research | R2060 | Refered to as RNA extraction kit |
| Dumont 5 Biology Tweezers | Fine Science Tools | 11254-20 | Refered to as Fine Forceps |
| EDTA | Fisher Scientific | S311-500 | Refered to as EDTA |
| FIJI v 2.14.0/1.54f | ImageJ, | N/A | Refered to as FIJI/ImageJ |
| Filament for pippette Puller | Tritech Research | PC-10H | Refered to as Filament |
| Fisherbrand Economy Impact Goggles | Fisher Scientific | 19-181-501 | Refered to as Splash Goggles |
| Glass Micropipette O.D. 1mm ID 0.58, Length 10 cm | TriTech Research | GD-1 | Reffered to as glass micropipette |
| Hypsibius exemplaris Z151 Strain | Carolina | 133960 | Refered to as Tardigrades or H. exemplaris |
| Liquid Nitrogen Dewar 1 L | Agar Scientific | AGB7475 | Refered to as Cryo-safe container |
| Maxima H Minus First Strand cDNA Synthesis Kit | Thermo Fisher Scientific | K1651 | Refered to as cDNA Synthesis Master Mix |
| Narishige Dual-Stage Glass Micropipette Puller | Tritech Research | PC-10 | Refered to as micropipette puller |
| Nitrile Gloves | Fisher Scientific | 17-000-314 | Refered to as Nitrile Gloves |
| PETRI DISH, PS, 35/10 mm, WITH VENTS | Grenier | 627102 | Refered to as 35 mm Petri dish |
| PIPETMAN P10, 1–10 µL, Metal Ejector | Gilson | F144055M | Refered to as P 10 Pipette |
| PIPETMAN P1000, 100–1000 µL, Metal Ejector | Gilson | F144059M | Refered to as P 1000 Pipette |
| PIPETMAN P200, 20–200 µL, Metal Ejector | Gilson | F144058M | Refered to as P 200 Pipette |
| Pound This 4-Color Modeling Clay | American Science Surplus | 96517P001 | Refered to as Clay |
| Prism v10.0 | GraphPad | N/A | Refered to a Prism |
| RNAse-Free, 8 Strip 0.2 mL PCR Tubes with caps | Invitrogen | AM12230 | Refered to as Sterile PCR Tube |
| RNasin Ribonuclease Inhibitor | Promega | N2111 | Refered to as RNAse inhibitor |
| Spring water | Nestle Pure Life | 44221229 | Refered to as Spring Water |
| SsoAdvanced Universal SYBR Green Supermix | BIO RAD | 1725271 | Refered to as Indicator Dye Super mix |
| Stereo-Microscope System w/optics and illumination | TriTech Research | SMT1 | Refered to as Dissecting Microscope |
| Supertek Scientific Tirrill Burners | Thermo Fisher Scientific | S09572B | Refered to as Bunsen Burner |
| Table Top Centrifuge | Qualitron | DW-41-115-NEW | Refered to as Table Top Centrifuge |
| Tempshield Cryo-Gloves | Fisher Scientific | 11-394-305 | Refered to as Cryo Gloves |
| Thermo Scientific Nunc Petri Dishes | Thermo Fisher Scientific | 08-757-099 | Refered to as 100 mm Petri dish |
| Tris base | Fisher Scientific | T395-500 | Refered to as Tris or Tris Base |
| Triton X-100 | Fluka | 93443 | Refered to as Detergent 1 |
| TWEEN 20 | Sigma aldrich | P1379-500 | Refered to as Detergent 2 |
| Water - PCR/RT-PCR certified, nuclease-free | Growcells | PCPW-0500 | Refered to as Sterile Nuclease Free Water |
Referenzen
- Møbjerg, N., Neves, R. C. New insights into survival strategies of tardigrades. Comp Biochem Physiol Part A Mol Integr Physiol. 254, 110890(2021).
- Jönsson, K. I., Harms-Ringdahl, M., Torudd, J. Radiation tolerance in the eutardigrade Richtersius coronifer. Int J Radiat Biol. 81 (9), 649-656 (2005).
- Horikawa, D. D., et al. Radiation tolerance in the tardigrade Milnesium tardigradum. Int J Radiat Biol. 82 (12), 843-848 (2006).
- Bruckbauer, S. T., Cox, M. M. Experimental evolution of extremophile resistance to ionizing radiation. Trends Genet. 37 (9), 830-845 (2021).
- Jönsson, K. I., Hygum, T. L., Andersen, K. N., Clausen, L. K. B., Møbjerg, N. Tolerance to gamma radiation in the marine heterotardigrade, Echiniscoides sigismundi. PLoS One. 11 (12), e0168884(2016).
- Jönsson, K. I. Radiation tolerance in tardigrades: Current knowledge and potential applications in medicine. Cancers (Basel). 11 (9), 1333(2019).
- Yoshida, Y., et al. RNA sequencing data for gamma radiation response in the extremotolerant tardigrade Ramazzottius varieornatus. Data Brief. 36, 107111(2021).
- Clark-Hachtel, C. M., et al. The tardigrade Hypsibius exemplaris dramatically upregulates DNA repair pathway genes in response to ionizing radiation. Curr Biol. 34 (9), 1819-1830.e6 (2024).
- Anoud, M., et al. Comparative transcriptomics reveal a novel tardigrade specific DNA binding protein induced in response to ionizing radiation. Elife. 13, RP92621(2024).
- Jönsson, K. I., Schill, R. O. Induction of Hsp70 by desiccation, ionising radiation and heat-shock in the eutardigrade Richtersius coronifer. Comp Biochem Physiol B Biochem Mol Biol. 146 (4), 456-460 (2007).
- Boothby, T. C. Desiccation of Hypsibius exemplaris. Cold Spring Harb Protoc. 2018 (11), 871-873 (2018).
- Boothby, T. C., et al. Tardigrades use intrinsically disordered proteins to survive desiccation. Mol Cell. 65 (6), 975-984.e5 (2017).
- Horikawa, D. D., Higashi, S. Desiccation tolerance of the tardigrade Milnesium tardigradum collected in Sapporo, Japan, and Bogor, Indonesia. Zoolog Sci. 21 (8), 813-816 (2004).
- Halberg, K. A., Jørgensen, A., Møbjerg, N. Desiccation tolerance in the tardigrade Richtersius coronifer relies on muscle mediated structural reorganization. PLoS One. 8 (12), e3330(2013).
- Sørensen-Hygum, T. L., Stuart, R. M., Jørgensen, A., Møbjerg, N. Modelling extreme desiccation tolerance in a marine tardigrade. Sci Rep. 8 (1), 11495(2018).
- Lyons, A. M., Roberts, K. T., Williams, C. M. Survival of tardigrades (Hypsibius exemplaris) to subzero temperatures depends on exposure intensity, duration, and ice-nucleation - as shown by large-scale mortality dye-based assays. bioRxiv. , (2024).
- Møbjerg, A., et al. Extreme freeze-tolerance in cryophilic tardigrades relies on controlled ice formation but does not involve significant change in transcription. Comp Biochem Physiol Part A Mol Integr Physiol. 271, 111245(2022).
- Tsujimoto, M., Imura, S., Kanda, H. Recovery and reproduction of an Antarctic tardigrade retrieved from a moss sample frozen for over 30 years. Cryobiology. 72 (1), 78-81 (2016).
- Jönsson, K. I. Tardigrades as a potential model organism in space research. Astrobiology. 7 (5), 757-766 (2007).
- Jönsson, K. I., Rabbow, E., Schill, R. O., Harms-Ringdahl, M., Rettberg, P. Tardigrades survive exposure to space in low Earth orbit. Curr Biol. 18 (17), R729-R731 (2008).
- Kasianchuk, N., Rzymski, P., Kaczmarek, Ł The biomedical potential of tardigrade proteins: A review. Biomed Pharmacother. 158, 113983(2023).
- Arakawa, K. Examples of extreme survival: Tardigrade genomics and molecular anhydrobiology. Annu Rev Anim Biosci. 10 (1), 519-542 (2022).
- Hvidepil, L. K. B., Møbjerg, N. New insights into osmobiosis and chemobiosis in tardigrades. Front Physiol. 14, 1274522(2023).
- Tanaka, S., Aoki, K., Arakawa, K. In vivo expression vector derived from anhydrobiotic tardigrade genome enables live imaging in Eutardigrada. Proc Natl Acad Sci U S A. 120 (5), e2216739120(2023).
- Kondo, K., Tanaka, A., Kunieda, T. Single-step generation of homozygous knockout/knock-in individuals in an extremotolerant parthenogenetic tardigrade using DIPA-CRISPR. PloS Genet. 20 (6), e1011298(2024).
- Yoshida, Y., Hirayama, A., Arakawa, K. Transcriptome analysis of the tardigrade Hypsibius exemplaris exposed to the DNA-damaging agent bleomycin. bioRxiv. , (2024).
- Yoshida, Y., et al. Time-series transcriptomic screening of factors contributing to the cross-tolerance to UV radiation and anhydrobiosis in tardigrades. BMC Genomics. 23 (1), 405(2022).
- Yoshida, Y., et al. RNA sequencing data for gamma radiation response in the extremotolerant tardigrade Ramazzottius varieornatus. Data Brief. 36, 107111(2021).
- Neves, R. C., et al. Differential expression profiling of heat stressed tardigrades reveals major shift in the transcriptome. Comp Biochem Physiol Part A Mol Integr Physiol. 267, 111143(2022).
- Yoshida, Y., et al. Comparative genomics of the tardigrades Hypsibius dujardini and Ramazzottius varieornatus. PLoS Biol. 15 (7), e2002266(2017).
- Wang, C., Grohme, M. A., Mali, B., Schill, R. O., Frohme, M. Towards decrypting cryptobiosis - analyzing anhydrobiosis in the tardigrade Milnesium tardigradum using transcriptome sequencing. PLoS One. 9 (3), e92663(2014).
- Mali, B., et al. Transcriptome survey of the anhydrobiotic tardigrade Milnesium tardigradum in comparison with Hypsibius dujardini and Richtersius coronifer. BMC Genomics. 11 (1), 168(2010).
- Förster, F., et al. Transcriptome analysis in tardigrade species reveals specific molecular pathways for stress adaptations. Bioinform Biol Insights. 6, 69-96 (2012).
- Arakawa, K., Yoshida, Y., Tomita, M. Genome sequencing of a single tardigrade Hypsibius dujardini individual. Sci Data. 3 (1), 160063(2016).
- Arakawa, K. Transcriptome assembly of Richtersius coronifer with annotated BLAST result against Ramazzottius varieornatus. Figshare. Dataset. , (2019).
- Yoshida, Y., Konno, S., Nishino, R., Murai, Y., Tomita, M., Arakawa, K. Ultralow input genome sequencing library preparation from a single tardigrade specimen. J Vis Exp. (137), (2018).
- Murai, Y., et al. Multiomics study of a heterotardigrade, Echiniscus testudo, suggests convergent evolution of anhydrobiosis-related proteins in Tardigrada. bioRxiv. , (2020).
- Yoshida, Y., Sugiura, K., Tomita, M., Matsumoto, M., Arakawa, K. Comparison of the transcriptomes of two tardigrades with different hatching coordination. BMC Dev Biol. 19 (1), 24(2019).
- Tenlen, J. R. Microinjection of dsRNA in tardigrades. Cold Spring Harb Protoc. 2018 (11), (2018).
- Tenlen, J. R., McCaskill, S., Goldstein, B. RNA interference can be used to disrupt gene function in tardigrades. Dev Genes Evol. 223 (3), 171-181 (2013).
- Giovannini, I., et al. Production of reactive oxygen species and involvement of bioprotectants during anhydrobiosis in the tardigrade Paramacrobiotus spatialis. Sci Rep. 12 (1), 15888(2022).
- Ly, K., Reid, S. J., Snell, R. G. Rapid RNA analysis of individual Caenorhabditis elegans. MethodsX. 2, 59-63 (2015).
- Boothby, T. C. Total RNA extraction from tardigrades. Cold Spring Harb Protoc. 2018 (11), 905-907 (2018).
- Czerneková, M., Vinopal, S. The tardigrade cuticle. Limnol Rev. 21 (3), 127-146 (2021).
- Goldstein, B. Hypsibius dujardini. collection notes and culture protocol from Bob McNuff. , At http://tardigrades.bio.unc.edu/protocols/CollectionCulture.pdf (2007).
- McNuff, R. Laboratory culture of Hypsibius exemplaris. Cold Spring Harb Protoc. 2018 (11), 867-870 (2018).
- Gabriel, W. N., et al. The tardigrade Hypsibius dujardini, a new model for studying the evolution of development. Dev Biol. 312 (2), 545-559 (2007).
- Ruiz-Villalba, A., Ruijter, J. M., van den Hoff, M. J. B. Use and misuse of cq in qPCR data analysis and reporting. Life (Basel). 11 (6), 508(2021).
- Antonov, J., et al. Reliable gene expression measurements from degraded RNA by quantitative real-time PCR depend on short amplicons and a proper normalization. Lab Invest. 85 (8), 1040-1050 (2005).
- Toussaint, J., et al. Improvement of the clinical applicability of the genomic grade index through a qRT-PCR test performed on frozen and formalin-fixed paraffin-embedded tissues. BMC Genomics. 10, 424(2009).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten