Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Extraction d’ARN sur un seul animal et sur un seul tube pour la comparaison des niveaux relatifs de transcrits par qRT-PCR chez le tardigrade Hypsibius exemplaris
Dans cet article
Résumé
Ce travail présente une méthode d’extraction rapide d’ARN et de comparaison au niveau des transcrits pour analyser l’expression génique chez le tardigrade Hypsibius exemplaris. Utilisant la lyse physique, cette méthode à haut débit nécessite un seul tardigrade comme matériau de départ et permet une production robuste d’ADNc pour la réaction en chaîne par polymérase quantitative à transcription inverse (qRT-PCR).
Résumé
Le tardigrade Hypsibius exemplaris est un organisme modèle émergent réputé pour sa capacité à survivre à des environnements extrêmes. Pour explorer les mécanismes moléculaires et les bases génétiques d’une telle exprémotolérance, de nombreuses études s’appuient sur le séquençage de l’ARN (RNA-seq), qui peut être effectué sur des populations allant de grandes cohortes à des animaux individuels. La réaction en chaîne par polymérase à transcription inverse (RT-PCR) et l’interférence ARN (ARNi) sont ensuite utilisées pour confirmer les résultats du séquençage de l’ARN et évaluer les exigences génétiques pour les gènes candidats, respectivement. De telles études nécessitent une méthode efficace, précise et abordable pour l’extraction de l’ARN et la mesure des niveaux relatifs de transcrits par RT-PCR quantitative (qRT-PCR). Ce travail présente une méthode efficace d’extraction d’ARN à tardigrade unique et à tube unique (STST) qui non seulement isole de manière fiable l’ARN des tardigrades individuels, mais réduit également le temps et le coût requis pour chaque extraction. Cette méthode d’extraction d’ARN produit des quantités d’ADNc qui peuvent être utilisées pour amplifier et détecter plusieurs transcrits par PCR quantitative (qRT-PCR). La méthode est validée en analysant les changements dynamiques dans l’expression des gènes codant pour deux protéines régulées par le choc thermique, la protéine de choc thermique 70 β2 (HSP70 β2) et la protéine de choc thermique 90α (HSP90α), ce qui permet d’évaluer leurs niveaux d’expression relatifs chez les individus exposés à la chaleur par qRT-PCR. STST complète efficacement les méthodes existantes d’extraction d’ARN tardigrade en vrac et unique, permettant un examen rapide et abordable des niveaux transcriptionnels individuels de tardigrade par qRT-PCR.
Introduction
Les tardigrades sont de petits animaux multicellulaires réputés pour leur capacité à survivre à des conditions extrêmes qui sont mortelles pour la plupart des autres formes de vie1. Par exemple, ces animaux peuvent survivre à près de 1000 fois la dose de rayonnement ionisant qui est mortelle pour les humains 2,3,4,5,6,7,8,9,10, une dessiccation presque complète 11,12,13,14,15, la congélation en l’absence d’ajout cryoprotecteurs 16,17,18, et, dans leur état desséché, même le vide de l’espace 19,20. En raison de leur capacité unique de survie dans des environnements extrêmes, ces animaux sont devenus des modèles fondamentaux pour comprendre l’extrémotolérance chez les organismes complexes et multicellulaires 1,21,22,23.
La manipulation génétique stable de ces animaux remarquables, y compris la transgenèse et la modification des gènes germinaux, est restée insaisissable jusqu’à récemment. En tant que tel, la plupart des expériences visant à révéler les mécanismes moléculaires de l’extrémotolérance sont réalisées par profilage transcriptionnel via le séquençage de l’ARN. Il existe de nombreux ensembles de données de séquençage de l’ARN précieux et instructifs pour les tardigrades dans diverses conditions extrêmes, allant du rayonnement 8,9,26,27,28, du stress thermique29, du stress de congélation12 et de la dessiccation 27,30,31,32,33. Certaines de ces études ont utilisé des méthodes d’extraction et de purification de l’ARN en vrac pour éclairer notre compréhension moléculaire de l’extrémotolérance. Cependant, l’extraction en masse des transcrits d’ARN de nombreux animaux empêche l’analyse de la variation de l’expression génique entre les individus, manquant ainsi la richesse potentielle d’ensembles de données plus raffinés. Il est important de noter que ces études analysent souvent des populations hétérogènes d’animaux qui comprennent à la fois des animaux qui survivent aux facteurs de stress environnementaux et ceux qui ne le font pas. En tant que telles, ces études sont faussées par la moyenne des données d’expression provenant d’états de réponse multiples et potentiellement radicalement différents. Pour résoudre ce problème, Arakawa et al., 201634 ont développé un élégant pipeline de séquençage d’ARN à faible entrée qui applique un kit d’extraction d’ARN suivi d’une étape d’amplification PCR linéaire en utilisant un seul34, 35, 36 ou plusieursanimaux 30, 37, 38comme entrée. Ces études ont été fondamentales pour notre compréhension de l’extrémotolérance du tardigrade22. Il est intéressant de noter que ce protocole a également été appliqué à la qRT-PCR en utilisant sept animaux comme matériau de départ24.
Dans la plupart des organismes modèles, après avoir identifié des cibles potentielles via le séquençage de l’ARN, la qRT-PCR est ensuite effectuée pour confirmer les modifications transcriptionnelles identifiées par le séquençage de l’ARN et évaluer l’évolution temporelle de l’expression des gènes candidats de manière à haute résolution. Pour tester la fonction des gènes identifiés, de telles études sont souvent suivies d’une neutralisation de cibles moléculaires par l’ARNi39,40 et d’une analyse de la capacité extremotolérante12,41. L’efficacité de chaque inactivation de l’ARNi est généralement confirmée par qRT-PCR en surveillant directement la diminution de l’abondance des transcrits. Cependant, l’ARNi est un processus à forte intensité de main-d’œuvre chez les tardigrades car chaque ARNdb doit être délivré par micro-injection manuelle d’individus39,40. En raison de la nature à faible débit de cette stratégie, une méthode d’extraction d’ARN rapide et peu coûteuse adaptée à la qRT-PCR à partir d’animaux uniques serait très précieuse pour la recherche sur les tardigrades. Bien que des méthodes antérieures aient été développées pour extraire l’ARN de tardigrades uniques, ces protocoles n’ont pas combiné leur extraction avec la qRT-PCR, s’appuyant plutôt sur des méthodes basées sur la densité optique 12,40,41. Motivés par ces défis, nous avons cherché à développer un protocole qui produit de manière fiable de l’ARN en quantité et en qualité qui peut être utilisé pour la qRT-PCR à partir d’un seul H. exemplaris.
Adapté d’un protocole d’extraction d’ARN sur un seul animal développé pour Caenorhabditis elegans42, STST est optimisé pour H. exemplaris. La méthode d’extraction se compose de six étapes rapides de congélation-décongélation, perturbant physiquement la cuticule, permettant l’extraction de l’ARN et la synthèse ultérieure de l’ADNc. La méthode STST réduit le temps d’extraction de plus de 24 fois par rapport aux méthodes d’extraction d’ARN en vrac, comme décrit par Boothby, 201843, et de 30 % par rapport aux kits d’extraction d’ARN tardigrade unique, comme décrit par Arakawa et al., 201634. De plus, le nombre d’interactions échantillon-expérimentateur est réduit de 5 à seulement 1 par rapport aux préparations de kits d’extraction d’ARN, réduisant ainsi le risque de contamination par des ribonucléases exogènes. Lors de l’interrogation de gènes fortement exprimés, la méthode STST produit suffisamment d’ADNc pour 25 réactions RT-PCR quantitatives par tardigrade, ne nécessitant que 1 μL du volume total de 25 μL d’ADNc par réaction. Cependant, les concentrations de matrice doivent être déterminées empiriquement pour les transcrits de faible abondance.
L’efficacité de la méthode STST pour l’analyse des changements dynamiques dans l’expression génique a été évaluée en étudiant l’expression différentielle des gènes codant pour la protéine de choc thermique 90α (HSP90α) et la protéine de choc thermique 70β2 (HSP70β2) en réponse à un choc thermique à court terme à 35 °C pendant 20 minutes. HSP70β2 et HSP90α chez la plupart des organismes eucaryotes sont rapidement régulés à la hausse après une exposition à court terme à un choc thermique (20 min)42. L’analyse chez H. exemplaris a révélé que les ARN codant pour HSP70β2 et HSP90α extraits de tardigrades uniques traités thermiquement présentaient des augmentations statistiquement significatives de l’expression après une exposition à la chaleur à court terme. Ces résultats démontrent que le protocole STST peut être utilisé pour analyser les changements dynamiques dans l’expression des gènes chez des animaux individuels au fil du temps.
La méthode d’extraction STST devrait compléter les méthodes expérimentales existantes telles que le RNA-seq en facilitant l’extraction rapide et peu coûteuse de l’ARN et la comparaison ultérieure des niveaux de transcrits par qRT-PCR. Cette méthode sera également précieuse pour évaluer l’efficacité et la pénétrance de l’ARNi chez les individus injectés manuellement de manière plus quantitative que la densité optique seule. Enfin, en raison de leurs structures cuticulaires et de leurs caractéristiques physiques similaires, il est probable que cette méthode sera également efficace pour analyser l’expression génique chez d’autres espèces de tardigrades44.
Access restricted. Please log in or start a trial to view this content.
Protocole
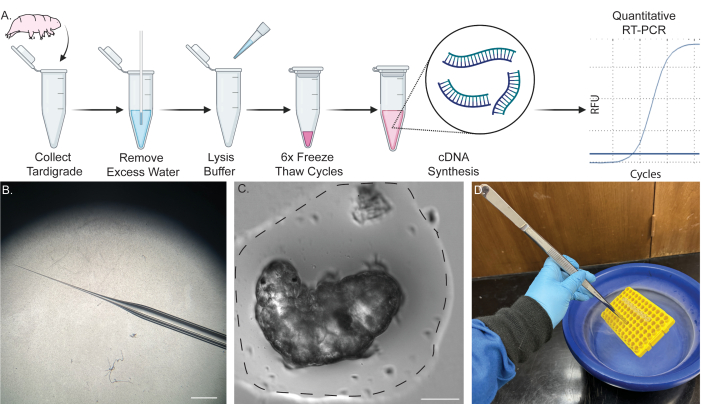
Figure 1 : Pipeline monotube pour l’extraction d’ARN à partir d’un seul tardigrade. (A) Schéma montrant le protocole d’extraction d’ARN à partir d’un seul tardigrade, y compris six cycles de congélation-décongélation et la synthèse ultérieure de l’ADNc. Les échantillons peuvent ensuite être utilisés pour la RT-PCR et la qRT-PCR. (B) Image du cône de la micropipette utilisé pour l’élimination de l’eau. Barre d’échelle : 2 mm. (C) Image en fond clair d’un tardigrade dans un petit volume d’eau (ligne pointillée). L’élimination de la majeure partie de l’eau dans la mesure indiquée est nécessaire pour une extraction réussie et empêche la dilution du tampon de lyse. Barre d’échelle : 50 μm. (D) Image montrant l’immersion d’échantillons dans de l’azote liquide à l’aide de longues pinces pour geler-décongeler rapidement les échantillons en toute sécurité. Une partie du contenu a été créée dans BioRender. Kirk, M. (2022) BioRender.com/d93s511 Veuillez cliquer ici pour voir une version agrandie de cette figure.
REMARQUE : La figure 1A montre un schéma de la procédure. Pour des procédures détaillées d’élevage de tardigrades et d’algues, veuillez consulter les rapports45, 46 et 47 publiés précédemment.
1. Stérilisation de l’eau de source
- Versez 2 L d’eau de source d’une cruche d’eau de 5 gallons (voir le tableau des matériaux pour plus de détails) dans une bouteille en verre de 2 L sans danger pour l’autoclave.
- Placez le bouchon sur la bouteille sans danger pour l’autoclave et fermez-la avec une petite quantité de ruban adhésif pour autoclave. Ne serrez pas la bouteille ; Placez le capuchon sur le dessus.
- Autoclavez l’eau de source pendant 50 min sur un cycle humide sans étape de séchage.
- Laissez l’eau revenir à température ambiante (RT) et fermez fermement le bouchon avant de le ranger à RT.
2. Tirage de micropipettes en verre (avec un extracteur de pipette)
- Fixez une micropipette en verre (D.E. : 1 mm, D.I. : 0,58 mm, Longueur : 10 cm) sur un extracteur de micropipette. Évitez tout contact avec le filament chauffant, car cela modifierait la forme de la pipette et endommagerait le filament.
- Déterminez empiriquement le tirage de la pipette pour chaque filament et extracteur de pipette. Cependant, pour servir de point de départ à l’optimisation, utilisez 78 °C et un seul pas de traction d’un poids de traction de 182,2 g.
- Laissez le filament chauffer et la gravité pour séparer la micropipette en verre en deux micropipettes en verre avec des pointes acérées (Figure 1B).
- Rangez ces micropipettes en verre tiré dans une boîte de Pétri fermée de 100 mm avec de la cire ou de l’argile pour les maintenir en place et éviter que les pointes pointues ne se cassent.
3. Tirage de micropipettes en verre (sans extracteur de pipette)
- Allumez un bec Bunsen ou une autre source de flamme contrôlée à basse température.
- Prenez une micropipette en verre avec une extrémité dans chaque main.
- Maintenez le centre de la micropipette en verre au-dessus de la flamme jusqu’à ce que le verre commence à fondre. Ensuite, séparez rapidement les deux extrémités. Cela créera deux pointes acérées très délicates.
- Cassez légèrement la pointe à l’aide d’une paire de pinces fines stériles.
- Rangez ces micropipettes en verre tiré dans une boîte de Pétri fermée de 100 mm avec de la cire ou de l’argile pour les maintenir en place et éviter que les pointes pointues ne se cassent.
4. Extraction de l’ARN
- Obtenez 0,5 L d’azote liquide dans un récipient cryo-safe.
ATTENTION : L’azote liquide est cryogénique et peut provoquer des brûlures s’il est exposé à la peau ou aux yeux. Lors de la manipulation, utilisez des vêtements de protection, des lunettes anti-éclaboussures, des gants en nitrile, des cryogants, une blouse de laboratoire et des chaussures fermées. Assurez-vous que le récipient est sans danger pour l’azote liquide avant de transporter le liquide. L’utilisation d’un bain d’éthanol-glace carbonique pour cette étape peut également être possible. - Préparez un mélange maître de synthèse d’ADNc : une solution de 10 μL contenant 1 μL d’amorce hexamère aléatoire, 2 μL de DNase, 4 μL de tampon 5x RT, 1 μL de mélange enzymatique, 1 μL de H2O et 1 μL de 10 mM de dNTP Conservez cette solution sur de la glace.
- Préparez un tampon de lyse tardigrade (5 mM de Tris (pH = 8), 0,5 % (v/v) Détergent 1, 0,5 % (v/v) Détergent 2, 0,25 mM d’EDTA dans de l’eau stérile sans nucléases).
REMARQUE : Cette solution peut être conservée sur la paillasse pendant 6 mois. Cependant, maintenez la stérilité et évitez les sources potentielles de contamination de l’ARNase. - Aliquote suffisamment de tampon de lyse pour les extractions (2 μL/ tardigrade).
- Ajouter l’inhibiteur de l’ARNase à la solution tampon de lyse tardive jusqu’à une concentration finale de 4 U/μL.
- Vortex et rotation de la solution à RT sur une centrifugeuse de paillasse à une vitesse de 2000 x g pendant 5 s avant de stocker la solution sur de la glace.
- Retirer de la culture autant de tardigrades que nécessaire pour l’expérience à l’aide d’une pipette P1000 à pointe filtrante stérile et les placer dans une boîte de Pétri stérile de 35 mm.
REMARQUE : N’importe quel nombre de tardigrades peut être traité de cette manière. Habituellement, trois tardigrades par condition sont traités pour l’extraction. - Lavez les tardigrades trois fois, à l’aide de 1 ml d’eau de source stérile autoclavée et d’une pipette P1000 stérile à pointe filtrante. Les pipeter lentement de haut en bas permet d’éliminer les contaminants d’algues.
- À l’aide d’un microscope de dissection à un grossissement de 25x à 50x, transférez un seul tardigrade de cette culture lavée dans une nouvelle boîte de Pétri stérile de 35 mm à l’aide d’une pipette P10 à pointe filtrante stérile.
- À l’aide d’une pipette P200 à pointe filtrante stérile, lavez le tardigrade unique dans 100 μL d’eau stérile sans nucléases.
REMARQUE : Cette étape de lavage est utilisée pour éliminer davantage les contaminants, y compris les ribonucléases. - Transférez le tardigrade lavé au fond d’un tube PCR propre et stérile dans 1 à 2 μL d’eau stérile sans nucléase à l’aide d’une pipette P10 stérile à pointe filtrante, en veillant soigneusement à ce que le tardigrade ne colle pas au côté de la pointe.
- Visualisez le tardigrade sous un microscope de dissection à un grossissement de 25x.
- Pour faciliter l’évacuation de l’eau par l’eau, cassez légèrement l’extrémité de la micropipette en verre tirée à l’extérieur du tube. Assurez-vous que le forage est assez grand pour tirer l’eau mais pas le tardigrade.
- À l’aide de l’action capillaire d’une micropipette en verre tiré, prélever l’eau jusqu’à ce que l’animal soit entouré d’une petite bulle d’eau d’environ deux longueurs de tardigrade de diamètre.
- Surveillez le processus d’élimination de l’eau à l’aide de la lunette de dissection pour vous assurer que le niveau d’eau est approprié et que le tardigrade reste hydraté.
REMARQUE : La figure 1C offre un exemple de la quantité d’eau à évacuer. Il s’agit d’une étape cruciale. Une petite bulle d’eau entourera le tardigrade pour l’empêcher de se dessécher, mais il faut éliminer autant d’eau que possible pour éviter la dilution du tampon de lyse. Pour un exemple des niveaux d’eau restants, veuillez vous référer à la figure 1C. - Immédiatement après avoir retiré l’eau, ajouter 2 μL de tampon de lyse tardigrade au fond du tube, agiter brièvement le vortex et centrifuger le tube à RT pendant 5 s à 2000 x g sur une centrifugeuse de table.
- Placez immédiatement les échantillons contenant les tardigrades dans un support de tube PCR et assurez-vous qu’ils sont bien maintenus par le support.
- Saisissez le support à l’aide d’une paire de longues pinces grossières et trempez doucement le support contenant les échantillons dans l’azote liquide jusqu’à ce qu’il soit complètement congelé (figure 1D).
- Retirez la grille de l’azote liquide et placez-la immédiatement sur de la glace. Laisser décongeler l’échantillon (prendre ~45 s à 1 min). Surveillez l’échantillon toutes les 15 s en le retirant de la glace et en l’inspectant visiblement. Une fois que l’échantillon est visiblement transparent, passez à l’étape suivante.
- Répétez les étapes 4.18-4.19 cinq fois de plus. Au total, six cycles de gel-dégel sont nécessaires pour maximiser la lyse et l’extraction (figures 2A, B).
- Une fois la congélation-décongélation terminée, placez les échantillons sur de la glace et passez immédiatement à l’étape suivante. Ne congelez pas les échantillons à ce stade pour les stocker, car cela diminuerait l’ARN disponible pour la préparation de l’ADNc.
5. Synthèse de l’ADNc
- Ajouter 2 μL de mélange maître de synthèse d’ADNc dans le tube PCR contenant du lysat de tardigrade. Agitez brièvement le tube et faites-le tourner à RT à 2000 x g pendant 5 s avec une centrifugeuse de table avant de replacer les échantillons sur de la glace.
- Placez les échantillons dans un thermocycleur et incubez à 25 °C pendant 10 min pour obtenir des amorces de recuit, à 55 °C pendant 30 min pour effectuer une transcription inverse, et enfin, inactivez thermiquement les enzymes à 85 °C pendant 5 min.
- Après l’incubation, placez immédiatement le tube sur de la glace et diluez l’échantillon jusqu’à un volume total de 25 μL en ajoutant 21 μL d’eau stérile sans nucléases. Pour les transcrits à faible nombre de copies, modifier cette étape de dilution telle qu’elle est déterminée empiriquement.
6. qPCR
- Déterminer la température de recuit de l’ensemble d’amorces à l’aide de l’ARN total préparé à partir de grandes quantités de tardigrades, par exemple, la méthode d’extraction en vrac présentée dans Boothby, 201843.
- Exécutez un gradient de température PCR pour déterminer la température de recuit optimale avant d’exécuter qRT-PCR (pour tous les paramètres PCR utilisés dans ce protocole, reportez-vous au Tableau 1 et au Tableau 2).
- Décongelez un tube de super mélange de colorant indicateur sur de la glace et isolez-le de la lumière. Placez une plaque de qPCR de 96 puits sur de la glace et placez 5 μL de super mélange, 2 μL d’eau, 1 μL de chaque amorce (10 μM) et 1 μL de produit d’ADNc dans le nombre de puits souhaité.
- Scellez la plaque de PCR avec le sceau de plaque et exécutez la qRT-PCR en utilisant une température de recuit appropriée pour le jeu d’amorces (pour tous les paramètres de qRT-PCR utilisés dans cet article, reportez-vous au tableau 3).
7. Quantification et interprétation des résultats
- Comparer quantitativement les résultats à un ou plusieurs gènes de contrôle ménager, dont l’expression est censée être constante dans les conditions imposées. Pour cette étude, le gène de l’actine a été utilisé.
- Obtenez et comparez les valeurst C ou le seuil de cycle pour chaque puits aux valeurs Ct des réactions géniques de contrôle de l’entretien. Calculez le changement de pli dans l’expression des gènes à l’aide de l’équation suivante :
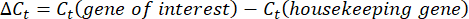
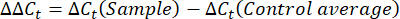
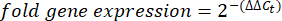
REMARQUE : L’expression du gène de pliage est tracée pour chaque transcrit et tardigrade sous la forme d’un 2-(ΔΔCt)48. - Pour obtenir une estimation approximative du nombre de transcrits à partir de la valeurt C, utilisez l’équation suivante :
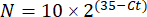
Où N est le nombre de transcrits et 2 est l’efficacité présumée de la PCR ou l’augmentation de la fluorescence par cycle de la PCR48.
Access restricted. Please log in or start a trial to view this content.
Résultats
Développement et optimisation de l’extraction d’ARN tardigrade unique
En adaptant le protocole de Ly et al., 201542 pour l’extraction de l’ARN dans les tardigrades, le système STST est optimisé pour maximiser la quantité et la qualité de la préparation (Figure 1A). La RT-PCR a été réalisée pour les transcrits d’actine, quantifiant le rendement des transcrits en amplifiant une région de 527 pb c...
Access restricted. Please log in or start a trial to view this content.
Discussion
Cette étude présente une méthode efficace pour l’extraction de l’ARN pour la qRT-PCR à tardigrade unique. La comparaison directe de la méthodologie STST à un kit d’extraction d’ARN tardigrade unique existant a révélé que l’extraction de l’ARN STST produit des quantités > 200 fois plus élevées de transcrits d’ARN d’actine, réduit le coût à moins d’un dollar par échantillon et réduit le temps nécessaire à l’extraction de 30 %. Pour appliquer STST à u...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les auteurs ne déclarent aucun conflit d’intérêts à divulguer.
Remerciements
Nous tenons à souligner la bourse Ruth Kirschstein des NIH # 5F32AG081056-02 et la bourse postdoctorale Errett Fisher, qui ont soutenu la Dre Molly J. Kirk, la bourse de la famille Crowe, qui a soutenu Chaoming Xu, ainsi qu’une subvention du Sénat académique de l’Université de Californie à Santa Barbara et des subventions #R01GM143771 et #2R01HD081266 des NIH, qui ont soutenu ces efforts de recherche. Les auteurs reconnaissent également l’utilisation du laboratoire de nanostructures biologiques au sein du California NanoSystems Institute, soutenu par l’Université de Californie à Santa Barbara et l’Université de Californie, Bureau du président.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| 10 µL Premium Barrier Tips Low Binding, Racked, Sterile | Genesee Scientific | 23-401 | Refered to as Sterile Filter-Tipped P 10 Pipette Tips |
| 1000 µL Premium Pipet Tips, Low Binding, Racked, Sterile | Genesee Scientific | 23-165RS | Refered to as Sterile Filter-Tipped P 1000 Pipette Tips |
| 200 µL Premium Barrier Tips Low Binding, Racked, Sterile | Genesee Scientific | 23-412 | Refered to as Sterile Filter-Tipped P 200 Pipette Tips |
| 4 Star Straight Strong Medium Point Tweezer | Excelta | 00-SA-DC | Refered to as Long forceps |
| 96-Well PCR Rack with Lid Assorted, 5 Racks/Unit | Genesee Scientific | 27-202A | Refered to as PCR Rack |
| Andwin Scientific 3M LEAD FREE AUTOCLAVE TAPE 1" | Thermo Fisher Scientific | NC0802040 | Refered to as Autoclave Tape |
| Autoclave Tape | Thermo Fisher Scientific | AB1170 | Refered to as PCR Plate Seals |
| Benchling v8 | Benchling | N/A | Refered to as Benchling |
| BioRadHard-Shell 96-Well PCR Plate | BioRad | HSS9641 | Refered to as PCR Plate |
| BULWARK FR Lab Coat: | Grainger | 26CF64 | Refered to as Lab Coat |
| C1000 Touch Bio-rad Thermocycler | BioRad | 1851148 | Refered to as Thermocycler |
| C1000 Touch Bio-rad Thermocycler with CFX Optics Module | BioRad | 1845097 | Refered to as qPCR thermocycler |
| Chloroccoccum hypnosporum. | Carolina | 152091 | Refered to as Algae |
| Corning PYREX Reusable Media Storage Bottles | Thermo Fisher Scientific | 06-414-1E | Refered to as 2 L Autoclave-safe Glass Bottle |
| Daigger & Company Vortex-Genie 2 Laboratory Mixer | Thermo Fisher Scientific | 3030A | Refered to as Vortexer |
| Direct-zol Micro Prep | Zymo Research | R2060 | Refered to as RNA extraction kit |
| Dumont 5 Biology Tweezers | Fine Science Tools | 11254-20 | Refered to as Fine Forceps |
| EDTA | Fisher Scientific | S311-500 | Refered to as EDTA |
| FIJI v 2.14.0/1.54f | ImageJ, | N/A | Refered to as FIJI/ImageJ |
| Filament for pippette Puller | Tritech Research | PC-10H | Refered to as Filament |
| Fisherbrand Economy Impact Goggles | Fisher Scientific | 19-181-501 | Refered to as Splash Goggles |
| Glass Micropipette O.D. 1mm ID 0.58, Length 10 cm | TriTech Research | GD-1 | Reffered to as glass micropipette |
| Hypsibius exemplaris Z151 Strain | Carolina | 133960 | Refered to as Tardigrades or H. exemplaris |
| Liquid Nitrogen Dewar 1 L | Agar Scientific | AGB7475 | Refered to as Cryo-safe container |
| Maxima H Minus First Strand cDNA Synthesis Kit | Thermo Fisher Scientific | K1651 | Refered to as cDNA Synthesis Master Mix |
| Narishige Dual-Stage Glass Micropipette Puller | Tritech Research | PC-10 | Refered to as micropipette puller |
| Nitrile Gloves | Fisher Scientific | 17-000-314 | Refered to as Nitrile Gloves |
| PETRI DISH, PS, 35/10 mm, WITH VENTS | Grenier | 627102 | Refered to as 35 mm Petri dish |
| PIPETMAN P10, 1–10 µL, Metal Ejector | Gilson | F144055M | Refered to as P 10 Pipette |
| PIPETMAN P1000, 100–1000 µL, Metal Ejector | Gilson | F144059M | Refered to as P 1000 Pipette |
| PIPETMAN P200, 20–200 µL, Metal Ejector | Gilson | F144058M | Refered to as P 200 Pipette |
| Pound This 4-Color Modeling Clay | American Science Surplus | 96517P001 | Refered to as Clay |
| Prism v10.0 | GraphPad | N/A | Refered to a Prism |
| RNAse-Free, 8 Strip 0.2 mL PCR Tubes with caps | Invitrogen | AM12230 | Refered to as Sterile PCR Tube |
| RNasin Ribonuclease Inhibitor | Promega | N2111 | Refered to as RNAse inhibitor |
| Spring water | Nestle Pure Life | 44221229 | Refered to as Spring Water |
| SsoAdvanced Universal SYBR Green Supermix | BIO RAD | 1725271 | Refered to as Indicator Dye Super mix |
| Stereo-Microscope System w/optics and illumination | TriTech Research | SMT1 | Refered to as Dissecting Microscope |
| Supertek Scientific Tirrill Burners | Thermo Fisher Scientific | S09572B | Refered to as Bunsen Burner |
| Table Top Centrifuge | Qualitron | DW-41-115-NEW | Refered to as Table Top Centrifuge |
| Tempshield Cryo-Gloves | Fisher Scientific | 11-394-305 | Refered to as Cryo Gloves |
| Thermo Scientific Nunc Petri Dishes | Thermo Fisher Scientific | 08-757-099 | Refered to as 100 mm Petri dish |
| Tris base | Fisher Scientific | T395-500 | Refered to as Tris or Tris Base |
| Triton X-100 | Fluka | 93443 | Refered to as Detergent 1 |
| TWEEN 20 | Sigma aldrich | P1379-500 | Refered to as Detergent 2 |
| Water - PCR/RT-PCR certified, nuclease-free | Growcells | PCPW-0500 | Refered to as Sterile Nuclease Free Water |
Références
- Møbjerg, N., Neves, R. C. New insights into survival strategies of tardigrades. Comp Biochem Physiol Part A Mol Integr Physiol. 254, 110890(2021).
- Jönsson, K. I., Harms-Ringdahl, M., Torudd, J. Radiation tolerance in the eutardigrade Richtersius coronifer. Int J Radiat Biol. 81 (9), 649-656 (2005).
- Horikawa, D. D., et al. Radiation tolerance in the tardigrade Milnesium tardigradum. Int J Radiat Biol. 82 (12), 843-848 (2006).
- Bruckbauer, S. T., Cox, M. M. Experimental evolution of extremophile resistance to ionizing radiation. Trends Genet. 37 (9), 830-845 (2021).
- Jönsson, K. I., Hygum, T. L., Andersen, K. N., Clausen, L. K. B., Møbjerg, N. Tolerance to gamma radiation in the marine heterotardigrade, Echiniscoides sigismundi. PLoS One. 11 (12), e0168884(2016).
- Jönsson, K. I. Radiation tolerance in tardigrades: Current knowledge and potential applications in medicine. Cancers (Basel). 11 (9), 1333(2019).
- Yoshida, Y., et al. RNA sequencing data for gamma radiation response in the extremotolerant tardigrade Ramazzottius varieornatus. Data Brief. 36, 107111(2021).
- Clark-Hachtel, C. M., et al. The tardigrade Hypsibius exemplaris dramatically upregulates DNA repair pathway genes in response to ionizing radiation. Curr Biol. 34 (9), 1819-1830.e6 (2024).
- Anoud, M., et al. Comparative transcriptomics reveal a novel tardigrade specific DNA binding protein induced in response to ionizing radiation. Elife. 13, RP92621(2024).
- Jönsson, K. I., Schill, R. O. Induction of Hsp70 by desiccation, ionising radiation and heat-shock in the eutardigrade Richtersius coronifer. Comp Biochem Physiol B Biochem Mol Biol. 146 (4), 456-460 (2007).
- Boothby, T. C. Desiccation of Hypsibius exemplaris. Cold Spring Harb Protoc. 2018 (11), 871-873 (2018).
- Boothby, T. C., et al. Tardigrades use intrinsically disordered proteins to survive desiccation. Mol Cell. 65 (6), 975-984.e5 (2017).
- Horikawa, D. D., Higashi, S. Desiccation tolerance of the tardigrade Milnesium tardigradum collected in Sapporo, Japan, and Bogor, Indonesia. Zoolog Sci. 21 (8), 813-816 (2004).
- Halberg, K. A., Jørgensen, A., Møbjerg, N. Desiccation tolerance in the tardigrade Richtersius coronifer relies on muscle mediated structural reorganization. PLoS One. 8 (12), e3330(2013).
- Sørensen-Hygum, T. L., Stuart, R. M., Jørgensen, A., Møbjerg, N. Modelling extreme desiccation tolerance in a marine tardigrade. Sci Rep. 8 (1), 11495(2018).
- Lyons, A. M., Roberts, K. T., Williams, C. M. Survival of tardigrades (Hypsibius exemplaris) to subzero temperatures depends on exposure intensity, duration, and ice-nucleation - as shown by large-scale mortality dye-based assays. bioRxiv. , (2024).
- Møbjerg, A., et al. Extreme freeze-tolerance in cryophilic tardigrades relies on controlled ice formation but does not involve significant change in transcription. Comp Biochem Physiol Part A Mol Integr Physiol. 271, 111245(2022).
- Tsujimoto, M., Imura, S., Kanda, H. Recovery and reproduction of an Antarctic tardigrade retrieved from a moss sample frozen for over 30 years. Cryobiology. 72 (1), 78-81 (2016).
- Jönsson, K. I. Tardigrades as a potential model organism in space research. Astrobiology. 7 (5), 757-766 (2007).
- Jönsson, K. I., Rabbow, E., Schill, R. O., Harms-Ringdahl, M., Rettberg, P. Tardigrades survive exposure to space in low Earth orbit. Curr Biol. 18 (17), R729-R731 (2008).
- Kasianchuk, N., Rzymski, P., Kaczmarek, Ł The biomedical potential of tardigrade proteins: A review. Biomed Pharmacother. 158, 113983(2023).
- Arakawa, K. Examples of extreme survival: Tardigrade genomics and molecular anhydrobiology. Annu Rev Anim Biosci. 10 (1), 519-542 (2022).
- Hvidepil, L. K. B., Møbjerg, N. New insights into osmobiosis and chemobiosis in tardigrades. Front Physiol. 14, 1274522(2023).
- Tanaka, S., Aoki, K., Arakawa, K. In vivo expression vector derived from anhydrobiotic tardigrade genome enables live imaging in Eutardigrada. Proc Natl Acad Sci U S A. 120 (5), e2216739120(2023).
- Kondo, K., Tanaka, A., Kunieda, T. Single-step generation of homozygous knockout/knock-in individuals in an extremotolerant parthenogenetic tardigrade using DIPA-CRISPR. PloS Genet. 20 (6), e1011298(2024).
- Yoshida, Y., Hirayama, A., Arakawa, K. Transcriptome analysis of the tardigrade Hypsibius exemplaris exposed to the DNA-damaging agent bleomycin. bioRxiv. , (2024).
- Yoshida, Y., et al. Time-series transcriptomic screening of factors contributing to the cross-tolerance to UV radiation and anhydrobiosis in tardigrades. BMC Genomics. 23 (1), 405(2022).
- Yoshida, Y., et al. RNA sequencing data for gamma radiation response in the extremotolerant tardigrade Ramazzottius varieornatus. Data Brief. 36, 107111(2021).
- Neves, R. C., et al. Differential expression profiling of heat stressed tardigrades reveals major shift in the transcriptome. Comp Biochem Physiol Part A Mol Integr Physiol. 267, 111143(2022).
- Yoshida, Y., et al. Comparative genomics of the tardigrades Hypsibius dujardini and Ramazzottius varieornatus. PLoS Biol. 15 (7), e2002266(2017).
- Wang, C., Grohme, M. A., Mali, B., Schill, R. O., Frohme, M. Towards decrypting cryptobiosis - analyzing anhydrobiosis in the tardigrade Milnesium tardigradum using transcriptome sequencing. PLoS One. 9 (3), e92663(2014).
- Mali, B., et al. Transcriptome survey of the anhydrobiotic tardigrade Milnesium tardigradum in comparison with Hypsibius dujardini and Richtersius coronifer. BMC Genomics. 11 (1), 168(2010).
- Förster, F., et al. Transcriptome analysis in tardigrade species reveals specific molecular pathways for stress adaptations. Bioinform Biol Insights. 6, 69-96 (2012).
- Arakawa, K., Yoshida, Y., Tomita, M. Genome sequencing of a single tardigrade Hypsibius dujardini individual. Sci Data. 3 (1), 160063(2016).
- Arakawa, K. Transcriptome assembly of Richtersius coronifer with annotated BLAST result against Ramazzottius varieornatus. Figshare. Dataset. , (2019).
- Yoshida, Y., Konno, S., Nishino, R., Murai, Y., Tomita, M., Arakawa, K. Ultralow input genome sequencing library preparation from a single tardigrade specimen. J Vis Exp. (137), (2018).
- Murai, Y., et al. Multiomics study of a heterotardigrade, Echiniscus testudo, suggests convergent evolution of anhydrobiosis-related proteins in Tardigrada. bioRxiv. , (2020).
- Yoshida, Y., Sugiura, K., Tomita, M., Matsumoto, M., Arakawa, K. Comparison of the transcriptomes of two tardigrades with different hatching coordination. BMC Dev Biol. 19 (1), 24(2019).
- Tenlen, J. R. Microinjection of dsRNA in tardigrades. Cold Spring Harb Protoc. 2018 (11), (2018).
- Tenlen, J. R., McCaskill, S., Goldstein, B. RNA interference can be used to disrupt gene function in tardigrades. Dev Genes Evol. 223 (3), 171-181 (2013).
- Giovannini, I., et al. Production of reactive oxygen species and involvement of bioprotectants during anhydrobiosis in the tardigrade Paramacrobiotus spatialis. Sci Rep. 12 (1), 15888(2022).
- Ly, K., Reid, S. J., Snell, R. G. Rapid RNA analysis of individual Caenorhabditis elegans. MethodsX. 2, 59-63 (2015).
- Boothby, T. C. Total RNA extraction from tardigrades. Cold Spring Harb Protoc. 2018 (11), 905-907 (2018).
- Czerneková, M., Vinopal, S. The tardigrade cuticle. Limnol Rev. 21 (3), 127-146 (2021).
- Goldstein, B. Hypsibius dujardini. collection notes and culture protocol from Bob McNuff. , At http://tardigrades.bio.unc.edu/protocols/CollectionCulture.pdf (2007).
- McNuff, R. Laboratory culture of Hypsibius exemplaris. Cold Spring Harb Protoc. 2018 (11), 867-870 (2018).
- Gabriel, W. N., et al. The tardigrade Hypsibius dujardini, a new model for studying the evolution of development. Dev Biol. 312 (2), 545-559 (2007).
- Ruiz-Villalba, A., Ruijter, J. M., van den Hoff, M. J. B. Use and misuse of cq in qPCR data analysis and reporting. Life (Basel). 11 (6), 508(2021).
- Antonov, J., et al. Reliable gene expression measurements from degraded RNA by quantitative real-time PCR depend on short amplicons and a proper normalization. Lab Invest. 85 (8), 1040-1050 (2005).
- Toussaint, J., et al. Improvement of the clinical applicability of the genomic grade index through a qRT-PCR test performed on frozen and formalin-fixed paraffin-embedded tissues. BMC Genomics. 10, 424(2009).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon