Method Article
La combinación de QD-FRET y microfluídica para monitorear Nanocomplex ADN auto-ensamblado en tiempo real
En este artículo
Resumen
Se presenta un nuevo y poderoso de integración de la nanofotónica (QD-FRET) y microfluídica para investigar la formación de polyplexes polielectrolito, que se espera que proporcione un mejor control y síntesis de polyplexes uniforme y personalizable para a base de ácido nucleico futuras terapias.
Resumen
Los avances en genómica continuará impulsando el desarrollo de terapias que pueden dirigirse a la patogénesis a nivel celular y molecular. Por lo general funcional dentro de la célula, basadas en ácidos nucleicos terapéuticos requieren un eficiente sistema de suministro intracelular. Un enfoque ampliamente adoptado es el ADN complejo con un portador del gen para formar nanocomplexes electrostática a través de auto-ensamblaje, facilitando la captación celular de ADN, mientras que lo protege contra la degradación. El desafío radica en el diseño racional de los portadores del gen eficiente, ya que la disociación prematura o excesivamente unión estable sería perjudicial para la captación celular y la eficacia terapéutica. Nanocomplexes sintetizado por el volumen de mezcla mostró una amplia gama de comportamiento intracelular desembalaje y el tráfico, que se atribuyó a la heterogeneidad en el tamaño y la estabilidad de nanocomplexes. Esta heterogeneidad dificulta la evaluación precisa de la cinética de auto-ensamblaje y se suma a la dificultad en la correlación de sus propiedades físicas a las eficiencias de transfección o bioactividad. Se presenta una nueva convergencia de la nanofotónica (es decir, QD-FRET) y microfluídica para caracterizar la cinética en tiempo real de la nanocomplex auto-ensamblaje bajo flujo laminar. QD-FRET proporciona una indicación muy sensible a la aparición de las interacciones moleculares y medida cuantitativa de todo el proceso de síntesis, mientras que microfluidos ofrece un microambiente bien controlados para analizar espacialmente el proceso con una alta resolución temporal (~ milisegundos). Para el sistema de modelo de nanocomplexes poliméricos, dos etapas distintas en el proceso de auto-ensamblaje fueron capturados por esta plataforma analítica. El aspecto cinética del proceso de auto-ensamblaje obtenido en la microescala sería especialmente valioso para microreactor basada en las reacciones que son relevantes para muchas aplicaciones de micro-y nano-escala. Además, nanocomplexes puede ser personalizado mediante un diseño adecuado de los dispositivos de microfludic, y el resultado QD-FRET nanocomplexes polímeros de ADN pueden ser fácilmente aplicada para el establecimiento de relaciones estructura-función.
Protocolo
A. biotinilación de ADN
ADN plásmido se biotinilado covalentemente con las etiquetas de biotina guanina específicas como se describe por el fabricante (Mirus Bio, Madison, WI), pero a escala para que las etiquetas de ~ 1-2 biotina por ADN. ADN plásmido (pEGFP-C1, 4,9 kb, Clontech, Mountain View, CA) fue etiquetado en este protocolo.
- Disolver la cantidad deseada de pDNA en buffer TE de DNasa y RNasa libre (biología molecular, grado de calidad) de agua para hacer una solución de ADN 1μg/μL.
- Llevar a cabo la reacción de marcaje con las mezclas de reacción siguiente. Añadir la etiqueta lo último reactivo.
Para la reacción de ADN 100 microgramos:
| Libre de DNasa y RNasa libre de agua | 75 l |
| Buffer 10X Un etiquetado | 20 l |
| 1μg/μL ADN | 100 L |
| Etiqueta de TI reactivo | 5 l |
| Volumen Total | 200 l |
- Incubar la reacción a 37 ° C durante 1 hora.
- Purificar la muestra marcada por el etanol o isopropanol precipitación siguiendo protocolos estándar.
Nota: Las columnas de filtración en gel base puede conducir a la absorción UV de alta o de fluorescencia de fondo, que puede afectar a la cuantificación del ADN o la caracterización de fluorescencia.
Nota: El nivel de biotinilación puede ser determinado por las pruebas basadas en HABA.
B. Etiquetado de los polímeros catiónicos Cy5-
Quitosano (390 kDa, 83,5% desacetilado, Vanson, Redmond, WA) fue utilizado como un polímero catiónico modelo en este estudio. Las aminas primaria gratuita en la red troncal de polímero quitosano se marcaron con Cy5-NHS (Amersham Biosciences, Piscataway, NJ).
- Para facilitar la conjugación completa de tinte Cy5, calcular la cantidad necesaria de Cy5-NHS de tal manera que la proporción molar de Cy5: aminas primarias es de 1: 200.
- Ajustar el pH de la solución de quitosano (en tampón acetato 25 mM) a ~ 6,5 por adición de NaOH. Tenga en cuenta que la reacción del NHS es más eficiente a pH básico, pero la solubilidad del quitosano aquí limita el rango de pH de trabajo.
- Mientras se agita, se añade lentamente la cantidad calculada de Cy5-NHS (1 mg / ml DMSO) a la solución de quitosano en forma gota a gota.
- Agite la mezcla en la oscuridad a temperatura ambiente durante la noche.
- Para purificar, diálisis con 10k MWCO Slide-A-Lyzer (Pierce) durante 2 horas contra tampón acetato 1% a temperatura ambiente en la oscuridad.
- Vuelva a colocar buffer y otra diálisis de 2 horas a temperatura ambiente en la oscuridad.
- Vuelva a colocar buffer y diálisis durante la noche a 4 ° C en la oscuridad.
- Tienda purificada polímero marcado a -20 ° C.
Nota: En este estudio, una curva estándar se construye mediante la medición de la intensidad de emisión de Cy5-NHS éster a 670 nm. Caracterizar la densidad de etiquetado a través de la medición de la emisiones que se obtengan a 670 nm de Cy5 marcado con quitosano en la curva estándar. Absorbancia también se puede utilizar para determinar el etiquetado de eficiencia, pero no se realizó aquí.
C. Preparación de QD-ADN marcado y Cy5 polímeros
La relación molar de pDNA a QD se mantuvo en exceso (pDNA: QD ≈ 1: 2) para asegurar la conjugación completa de los puntos cuánticos de pDNA. El número de puntos cuánticos etiquetados en cada pDNA se puede estimar a través de TEM imágenes u otros recursos equivalentes. En nuestro estudio, el número de puntos cuánticos por pDNA se estima en ~ 3.1 por TEM y espectroscopia de moléculas individuales. Use un Millipore Milli-Q agua gradiente (> 18,0 MW, 0.2um filtrados) durante la preparación.
- Calcular la cantidad necesaria de quitosano para 10μg pDNA según deseada N / P, la relación teórica de aminas protonadas en la solución de quitosano a los fosfatos negativos en la solución de ADN.
- Añadir estreptavidina-funcionalizados 605QDs (Qdot 605 ITK, Invitrogen, Carlsbad, CA) en la solución de biotina pDNA.
- Incubar la solución a temperatura ambiente en la oscuridad durante 15 min.
- Agregue el QD-ADN marcado en 50 mM solución de sulfato de sodio para hacer el volumen final de 200μL.
- Diluir Cy5-quitosano, según deseada N / P, con agua Milli-Q para hacer que el volumen final de 200μL.
Nota: Mantenga la reacción en la oscuridad para evitar photobleaching posible.
Importante: Tenga cuidado al utilizar el Qdot 605 ITK ™ estreptavidina conjugada (la serie CTI), como puntos cuánticos en este catálogo están diseñados con el propósito de FRET. La serie Qdot regulares se conjugan con una capa de PEG para evitar la unión no específica, especialmente para el etiquetado de celular. Sin embargo, esta capa adicional amplía la distancia donador-aceptor, lo que resulta en rrelucida la eficiencia de transferencia de energía.
D. La fabricación de la SU-8 Masters usando fotolitografía estándar
- Oblea de silicio se pirañas limpia y al horno a 200 ° C durante 5 min.
- Por el espesor de 25μm maestro designado, capa fotosensible girar la negativa (SU-8 2025, Microchem, Newton, MA) en la oblea de Si a 2000 rpm durante 30 segundos.
- Soft hornear la oblea en una placa con una rampa de 65 ° C / hr a 95 ° C.
- Exponga a la luz UV (365 nm) para 250mJ/cm 2 a través de una máscara de la película (CAD / Servicios de arte, Bandon, Oregón) que contiene el diseño de microcanales.
- Después de la exposición hornear la oblea en una placa con una rampa de 65 ° C / hr a 95 ° C.
- Desarrollar la oblea con SU-8 desarrollador fotosensible.
- La oblea patrón es difícil al horno en una placa con una rampa de 65 ° C / hr a 200 ° C. Mantener la oblea a 200 ° C durante al menos 5 horas, luego se enfrían gradualmente la oblea a temperatura ambiente.
Importante: rampa gradual durante el SU-8 proceso de cocción maestro es necesario, de lo contrario el SU-8 puede separarse de la estructura de la oblea de silicio o grietas en la estructura de SU-8 puede ser inducida por el estrés de liberación.
E. Replica Moldeo por inyección de PDMS de los maestros y la vinculación a la tapa de vidrio
- El maestro de SU-8 se coloca en un recipiente pesado.
- Mezcla de silicona y elastómeros agente de curado (poli (dimetilsiloxano), PDMS, Sylgard 184, Dow Corning, Midland, MI) en un 10: 1 relación.
- Vierta la mezcla de PDMS sobre el SU-8 maestros y abandonar el barco con un peso de un secador de vacío para eliminar las burbujas.
- Curar el PDMS a 65 ° C durante 1-2 horas.
- Pelar la franja de PDMS del molde maestro Si.
- Golpe canal entradas y salidas del dispositivo de simulación.
- Limpie la tira de PDMS y cubierta de vidrio con etanol y secar al aire.
- El tratamiento de la limpieza PDMS tira y cubierta de vidrio con plasma de oxígeno (20 W para 1 minuto).
- Inmediatamente de bonos de la franja de PDMS con cubierta de vidrio.
- Deje el chip de microfluidos en condiciones de servidumbre en el horno a 95 ° C durante la noche.
Importante: El tratamiento de plasma y hornear durante la noche son esenciales para la resistencia de unión mejorada.
F. Monitor de la formación de ADN Nanocomplexes En el dispositivo de microfluidos
- Llene el canal de microfluidos con agua (para asegurar que no haya burbujas en el canal de microfluidos), antes de cargar los reactivos para asegurar el flujo suave durante el experimento.
- Cargar el ADN QD-etiquetado y marcado con Cy5 soluciones de quitosano en dos jeringas de vidrio individuales, a través de la tubería se describe en el vídeo.
- Conecte el tubo con las dos entradas de dispositivos de microfluídica. Tenga cuidado de no introducir aire durante el proceso. Establecer la velocidad de flujo en 20nL/min (PHD-2000 bomba de jeringa, Holliston, MA), bajo condiciones de flujo laminar.
- Compruebe los microcanales bajo el microscopio.
- Cuando el flujo es estable (~ 15 a 20 minutos), QD mediada por FRET deben ser observadas en el centro del canal.
- Tome fotografías de fluorescencia (CCD enfriado, Qimaging, BC, Canadá) en diferentes lugares a lo largo del canal.
- Analizar las imágenes de fluorescencia con ImageJ y OriginLab.
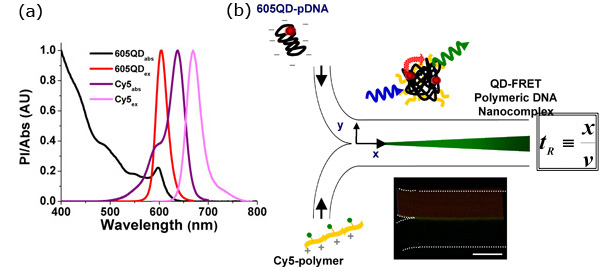
Figura 1. QD-FRET proporciona una indicación sensible de la aparición de Nanocomplexes ADN de auto-ensamblaje
- Puntos cuánticos mediada por la transferencia de energía de resonancia fluorescente (FRET-QD) puede proporcionar una indicación cuantitativa y muy sensible de la estabilidad Polyplex en entornos de extra-o intra-celular, que permite la detección inequívoca de la aparición de las interacciones entre el ADN y el portador del gen. La pareja FRET, 605QD y Cy5, fue elegido basado en la maximización de solapamiento espectral entre el donante y el receptor y minimizar los posibles cross-talk. Para esta pareja, la distancia de Förster es 69.4Å 3.
- Auto-ensamblaje de la nanocomplexes ADN QD-FRET. ADN plásmido aniónicos (pDNA) y el portador del gen catiónico se marcaron con QD (donante de energía) y Cy5 (aceptor de energía), respectivamente. QD-FRET nanocomplexes se formaron a través de coacervación compleja electrostática. A la excitación a 488 nm, QD-FRET mediada Cy5 emisiones indicó la formación de un nanocomplex compacto e intacta. El tiempo de residencia (R t) se puede calcular de acuerdo a la distancia (x), que mide desde donde las dos corrientes se encuentran en la posición de la reacción que se investiga, y la velocidad media del flujo (v). Debido a la naturaleza del flujo laminar, la mezcla sólo se lleva a cabo en la interfaz (el centro de cada imagen), lo que permite calcular de forma precisa el transporte de masa en función de t R. Resolución temporal puede ser ajustado por la variación de la applied caudales. (Recuadro) FRET mediada por la señal se observó inmediatamente en la interfaz cuando las dos corrientes se reunieron, lo que indica que la unión fue rápida, que ocurre dentro de unos pocos milisegundos de acuerdo con los caudales aplicados. Barra de escala: 100μm.
Discusión
- Importancia de nuestro trabajo:
- Este es el primer intento de controlar poliméricos ADN nanocomplex de auto-ensamblaje cinética en tiempo real (resolución de milisegundos) a través de QD-FRET respuestas dentro de un chip de microfluidos sencillos.
- QD mediada por FRET proporciona una indicación muy sensible y cuantitativo de la aparición de las interacciones moleculares y en todo el proceso de auto-ensamblaje, mientras que microfluidos ofrece un microambiente bien controlados para analizar espacialmente el proceso durante la síntesis de ADN nanocomplex.
- La integración de la microfluídica y nanofotónica sugiere un enfoque nuevo e interesante para investigar cualquier tipo de reacciones de complejos.
- El resultado de QD-FRET nanocomplexes ADN poliméricos podría ser fácilmente aplicada para el establecimiento de relaciones estructura-función. 1,2
Divulgaciones
The authors have nothing to disclose.
Agradecimientos
El apoyo financiero proporcionado por el NIH subvención HL89764, subvenciones NSF 0546012, 0730503 y 0725528.
Materiales
| Name | Company | Catalog Number | Comments |
| SU-8 | MicroChem Corp. | SU-8 2025 | |
| PDMS | Dow Corning | Sylgard 184 | |
| Plasmid DNA | Clontech Laboratories | pEGFP-C1 | 4.9 kb, MW = 3.3 x 106 |
| LabelIT Biotin Labeling Kit | Mirus Bio LLC | MIR 3400 | Standard protocol yields labeling efficiency of approximately one label every 20-60 bp of double-stranded DNA, The density of labeling was adjusted in this work. |
| Streptavidin 605QD | Invitrogen | Qdot® 605 ITK® Streptavidin Conjugate | |
| Cy5-NHS Ester | Amersham | PA15101 | |
| Chitosan | Vanson | 390 kDa | 83.5% deacetylated |
| Cover Glass | Fisher Scientific | 12-545C | No. 1; Size: 40 x 22mm |
| Gastight Glass Syringe | Hamilton Co | TLL series | 50μL to 500μL depending on sample volume |
| Tygon Tubing | Small Parts, Inc. | 0.02 ID, 100ft | Tygon Tubes Microbore, 0.02 ID, 100ft |
| Connector | Small Parts, Inc. | HTX-23R | Customized in length of 0.750" |
| Syringe Pump | Harvard Apparatus | PHD-2000 | |
| CCD | QImaging | Intensified Retiga Cooled | |
| Microscope | Olympus Corporation | BX-51 | 100W mercury arc lamp |
| ImageJ | National Institutes of Health | v1.36b | http://rsb.info.nih.gov/ij |
| Origin Pro8 | OriginLab | Student Version | |
| Microscope Filter sets | Omega Optical | 475AF40 | Excitation filter in both channels |
| Microscope Filter sets | Omega Optical | 595AF60 | Emission filter in 605QD channel |
| Microscope Filter sets | Omega Optical | 670DF40 | Emission filter in QD-FRET channel |
| Microscope Filter sets | Omega Optical | 500 DRLP | Long pass dichroic in 605QD channel |
| Microscope Filter sets | Omega Optical | 595DRLP | Long pass dichroic in QD-FRET channel |
Referencias
- Ho, Y. P., Chen, H. H., Leong, K. W., Wang, T. H. Evaluating the intracellular stability and unpacking of DNA nanocomplexes by quantum dots-FRET. J Control Release. 116, 83-89 (2006).
- Chen, H. H. Quantitative comparison of intracellular unpacking kinetics of polyplexes by a model constructed from quantum dot-FRET. Mol. Ther. 16, 324-332 (2008).
- Zhang, C. Y., Yeh, H. C., Kuroki, M., Wang, T. H. Single-Quantum-Dot-Based DNA Nanosensor. Nat Mat. 4, 826-831 (2005).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados