Method Article
Combinant QD-FRET et la microfluidique pour surveiller Nanocomplex ADN auto-assemblage en temps réel
Dans cet article
Résumé
Nous présentons une intégration novatrice et puissante de la nanophotonique (QD-FRET) et de la microfluidique pour étudier la formation des polyplexes polyélectrolyte, qui est censé fournir un meilleur contrôle et une synthèse des polyplexes uniforme et personnalisables pour base d'acide nucléiques thérapeutiques futures.
Résumé
Les progrès de la génomique continuent d'alimenter le développement de thérapies qui peuvent cibler la pathogenèse au niveau cellulaire et moléculaire. Typiquement fonctionnels à l'intérieur de la cellule, base d'acides nucléiques thérapeutiques nécessitent un système de livraison efficace intracellulaire. Une approche largement adoptée est de l'ADN complexe avec un porteur du gène pour former nanocomplexes via l'auto-assemblage électrostatique, ce qui facilite l'absorption cellulaire de l'ADN tout en le protégeant contre la dégradation. Le défi réside dans la conception rationnelle de porteurs du gène efficace, car prématurée dissociation ou trop contraignantes stables serait préjudiciable à l'absorption cellulaire et l'efficacité thérapeutique. Nanocomplexes synthétisés par mélange en vrac a montré une grande diversité de comportements et le trafic intracellulaire de déballage, qui a été attribuée à l'hétérogénéité de la taille et la stabilité de nanocomplexes. Cette hétérogénéité nuit à l'évaluation précise de la cinétique de l'auto-assemblage et ajoute à la difficulté de corréler leurs propriétés physiques à l'efficacité de la transfection ou bioactivités. Nous présentons une convergence roman de nanophotonique (c.-QD-FRET) et de la microfluidique afin de caractériser les cinétiques en temps réel de l'auto-assemblage nanocomplex sous flux laminaire. QD-FRET fournit une indication très sensible de l'apparition des interactions moléculaires et mesure quantitative tout au long du processus de synthèse, alors que la microfluidique offre un microenvironnement bien contrôlée spatialement analyser le processus avec une résolution temporelle élevée (~ millisecondes). Pour le système modèle de nanocomplexes polymères, deux phases distinctes dans le processus d'auto-assemblage ont été capturés par cette plate-forme analytique. L'aspect cinétique du processus d'auto-assemblage obtenu à l'échelle microscopique serait particulièrement précieuse pour microréacteur basée sur des réactions qui sont pertinentes pour de nombreuses applications micro-et nano-échelle. En outre, nanocomplexes peut être personnalisé grâce à une conception appropriée des appareils microfludic, et le résultat QD-FRET nanocomplexes ADN polymères pourrait être facilement appliquée pour l'établissement de relations structure-fonction.
Protocole
A. Biotinylation de l'ADN
L'ADN plasmidique ont été biotinylées covalente avec des étiquettes biotine guanine spécifiques telles que décrites par le fabricant (Mirus Bio, Madison, WI), mais à l'échelle d'avoir ~ 1-2 étiquettes biotine par l'ADN. L'ADN plasmidique (pEGFP-C1, 4,9 kb, Clontech, Mountain View, CA) a été marqué dans ce protocole.
- Dissoudre la quantité désirée de ADNp en tampon TE de DNase et de RNase (biologie moléculaire une qualité) d'eau pour faire une solution d'ADN 1μg/μL.
- Effectuer la réaction de marquage utilisant des mélanges de réaction suivante. Ajouter l'étiquette, il dernier réactif.
Pour la réaction d'ADN 100 ug:
| DNase et RNase-free de l'eau | 75 ul |
| Un tampon de marquage 10X | 20 pl |
| 1μg/μL l'ADN | 100 uL |
| Étiquette, il réactifs | 5 ul |
| Volume total | 200 uL |
- Incuber la réaction à 37 ° C pendant 1 heure.
- Purifier l'échantillon marqué par l'éthanol ou l'isopropanol précipitations suivant des protocoles standard.
Remarque: les colonnes de filtration à base de gel peut conduire à absorbance UV haute ou fond de fluorescence, qui peut affecter la quantification de l'ADN ou la caractérisation de fluorescence.
Remarque: Le niveau de biotinylation peut être déterminée par HABA tests basés.
B. Étiquetage des polymères cationiques Cy5-
Chitosan (390 kDa, 83,5% désacétylé, Vanson, Redmond, WA) a été utilisé comme modèle d'un polymère cationique dans cette étude. Les amines primaires gratuits sur l'épine dorsale polymère de chitosane ont été marquées avec Cy5-NHS (Amersham Biosciences, Piscataway, NJ).
- Afin de faciliter la conjugaison complète de Cy5 colorant, calculer la quantité nécessaire de Cy5-NHS tels que le rapport molaire de Cy5: amines primaires est de 1: 200.
- Ajuster le pH de la solution de chitosan (dans le tampon acétate 25 mM) à ~ 6,5 par addition de NaOH. Notez que la réaction du NHS est plus efficace à pH basique, mais la solubilité du chitosane ici les limites de la gamme de pH de travail.
- Tout en remuant, ajouter lentement le montant calculé de Cy5-NHS (1 mg / ml de DMSO) à la solution de chitosane dans une manière goutte-à-goutte.
- Agiter le mélange dans le noir à température ambiante jusqu'au lendemain.
- Pour purifier, dialyser avec 10k MWCO Slide-A-Lyzer (Pierce) pendant 2 heures contre du tampon acétate de 1% à température ambiante dans l'obscurité.
- Remplacer tampon et dialyser une autre 2 h à température ambiante dans l'obscurité.
- Remplacer tampon et dialyser nuit à 4 ° C dans l'obscurité.
- Boutique purifiée étiquetés polymère à -20 ° C.
Remarque: Dans cette étude, une courbe standard est construite en mesurant l'intensité d'émission de Cy5-NHS ester à 670 nm. Caractériser la densité de marquage en mesurant l'émission à 670 nm obtenue à partir Cy5-étiquetés chitosane dans la courbe standard. Absorbance peut également être utilisé pour déterminer l'efficacité d'étiquetage, mais n'a pas été effectué ici.
C. Préparation des QD-ADN marqué et Cy5-Polymère
Le rapport molaire de ADNp au QD a été maintenu en excès (ADNp: QD ≈ 1: 2) pour assurer la conjugaison complète des QDs à ADNp. Le nombre de boîtes quantiques étiquetés sur chaque ADNp peut être estimée grâce à l'imagerie TEM ou d'autres installations équivalentes. Dans notre étude, le nombre de boîtes quantiques par ADNp est estimé à ~ 1-3 par TEM et spectroscopie molécule unique. Utilisez une Millipore eau Milli-Q gradient (> 18,0 MW, 0.2um filtrée) pendant la préparation.
- Calculer la quantité requise de chitosane pour 10 pg ADNp fonction désirée rapport N / P, le ratio théorique d'amines protonées dans la solution de chitosane à l'phosphates négatifs dans la solution d'ADN.
- Ajouter streptavidine-fonctionnalisés 605QDs (ITK Qdot 605, Invitrogen, Carlsbad, CA) dans la solution biotinylé ADNp.
- Incuber la solution à température ambiante dans l'obscurité pendant 15 min.
- Ajouter le QD-ADN marqué en 50 mM solution de sulfate de sodium pour rendre le volume final 200 pl.
- Diluer Cy5-chitosane, selon désiré rapport N / P, avec eau Milli-Q pour rendre le volume final 200 pl.
Remarque: Conservez la réaction dans l'obscurité pour éviter photoblanchiment possible.
Important: Soyez prudent d'utiliser le Qdot 605 ITK ™ streptavidine conjugué (la série ITK), que les points quantiques dans ce catalogue sont conçus dans le but de FRET. La série Qdot réguliers sont conjugués avec une couche de PEG pour empêcher la liaison non spécifique, en particulier pour l'étiquetage cellulaire. Cependant, ce revêtement supplémentaire élargit la distance donneur-accepteur, résultant en réduite l'efficacité du transfert d'énergie.
D. La fabrication de la SU-8 Maîtrise en utilisant la photolithographie standard
- Plaquette de Si est piranha nettoyé et cuit à 200 ° C pendant 5 min.
- Pour l'épaisseur de 25 um maître désigné, manteau tourner la résine photosensible négative (SU-8 2025, Microchem, Newton, MA) sur la plaquette de Si à 2000 rpm pendant 30 sec.
- Cuire la galette molle sur une plaque chauffante avec une rampe de 65 ° C / hr à 95 ° C.
- Exposer à la lumière UV (365nm) pour 250mJ/cm 2 à travers un film masque (CAO / Art Services, Bandon, OR) contenant de la conception de microcanaux.
- Post-exposition cuire la galette sur une plaque chauffante avec une rampe de 65 ° C / hr à 95 ° C.
- Développer la plaquette en utilisant SU-8 développeur résine photosensible.
- La plaquette est difficile à motifs cuite sur une plaque chauffante avec une rampe de 65 ° C / h à 200 ° C. Maintenir la plaquette à 200 ° C pendant au moins 5 heures, puis refroidir progressivement la plaquette à température ambiante.
Important: montée en puissance progressive lors de la SU-8 cuisson maître est nécessaire, sinon le SU-8 structure peut détacher de la plaquette de silicium ou de fissures sur la structure de SU-8 peut être induite par le stress à la libération.
E. Replica moulage de PDMS par les maîtres et collage sur le verre Couvercle
- Le maître SU-8 est placé dans un bateau pesant.
- Mix de silicone élastomère et de durcisseur (poly (diméthylsiloxane), le PDMS, Sylgard 184, Dow Corning, Midland, MI) en 10: 1 ratio.
- Verser le mélange PDMS sur le SU-8 maître et quitter le bateau pesant dans un dessiccateur sous vide pour éliminer les bulles.
- Guérissez les PDMS à 65 ° C pendant 1-2 heures.
- Peler la bande PDMS de la moule maître de silicium.
- Poinçon entrées et sorties de canal de l'appareil fluidiques.
- Nettoyez la bande de PDMS et couvercle en verre avec de l'éthanol et ensuite sécher à l'air.
- Traiter le PDMS nettoyé bande de verre et couvrir avec un plasma d'oxygène (20W pendant 1min).
- Immédiatement coller la bande PDMS avec couvercle en verre.
- Laisser la puce microfluidique collé dans le four à 95 ° C pour la nuit.
Important: le traitement du plasma et la cuisson pendant la nuit sont essentiels à la force d'adhérence accrue.
F. Surveiller la formation de l'ADN nanocomplexes dans le dispositif microfluidique
- Remplissez le canal microfluidique avec de l'eau (pour s'assurer qu'il n'ya pas de bulles dans le canal microfluidique), avant le chargement des réactifs pour assurer la fluidité lors de l'expérience.
- Chargez l'ADN QD-étiquetés et Cy5-étiquetés solutions chitosane en deux seringues en verre individuel, au travers du tube décrit dans la vidéo.
- Connectez le tube avec les deux entrées de dispositifs microfluidiques. Soyez prudent de ne pas introduire de l'air pendant le processus. Régler le débit à 20nL/min (PHD-2000 pompe seringue, Holliston, MA), dans des conditions d'écoulement laminaire.
- Vérifiez les microcanaux sous le microscope.
- Lorsque le débit est stable (~ 15 à 20 minutes), QD-FRET médiation devrait être observé dans le centre du chenal.
- Prenez des photos de fluorescence (CCD refroidie, QImaging, BC, Canada) à différents endroits le long du canal.
- Analyser les images de fluorescence avec ImageJ et OriginLab.
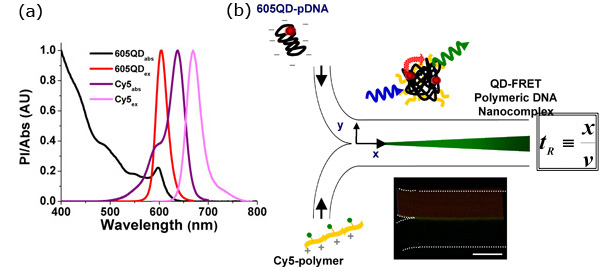
Figure 1. QD-FRET fournit une indication sensible de l'apparition de l'ADN nanocomplexes auto-assemblage
- Quantum dot-médiée Fluorescence Resonance Energy Transfer (QD-FRET) peut fournir une indication quantitative et très sensible de la stabilité dans les deux environnements Polyplex extra-ou intra-cellulaires, permettant de détecter sans ambiguïté de l'apparition d'interactions entre l'ADN et le porteur du gène. La paire FRET, 605QD et Cy5, a été choisie en fonction de la maximisation chevauchement spectral entre le donneur et l'accepteur et de minimiser le potentiel diaphonie. Pour ce couple, la distance Forster est 69.4Å 3.
- Auto-assemblage de l'ADN nanocomplexes QD-FRET. Anioniques ADN plasmidique (ADNp) et le porteur du gène cationiques ont été marqués avec QD (donneur d'énergie) et Cy5 (énergie accepteur), respectivement. QD-FRET nanocomplexes ont été formées par coacervation complexe électrostatique. Lors d'excitation à 488 nm, QD-FRET médiée Cy5 émission indiquées formation d'un nanocomplex compacte et intacte. Le temps de séjour (t R) peut être calculée en fonction de la distance (x) qui mesure où les deux courants se rencontrent pour la position de la réaction de l'enquête, et la vitesse moyenne d'écoulement (v). En raison de la nature de l'écoulement laminaire, le mélange a lieu seulement à l'interface (centre de chaque image), permettant un calcul précis du transport de masse en fonction de t R. La résolution temporelle peut être ajustée en faisant varier la appliedébits d. (En médaillon) FRET médiée signal a été observée immédiatement à l'interface lorsque les deux ruisseaux réunis, indiquant que la liaison a été rapide, survenant en quelques millisecondes en fonction des débits appliqués. Barre d'échelle: 100 microns.
Discussion
- Importance de notre travail:
- C'est la première tentative pour contrôler l'ADN polymérique nanocomplex auto-assemblage cinétiques en temps réel (milliseconde résolution) grâce QD-FRET réponses dans une puce microfluidique simples.
- QD-médiée FRET fournit une indication très sensible et quantitative de l'apparition des interactions moléculaires et tout au long du processus d'auto-assemblage, alors que la microfluidique offre un microenvironnement bien contrôlée spatialement analyser le processus lors de la synthèse nanocomplex ADN.
- L'intégration de la microfluidique et la nanophotonique suggère une approche nouvelle et intéressante pour enquêter sur tout type de réactions de complexation.
- Le résultat QD-FRET nanocomplexes ADN polymères pourrait être facilement appliquée pour l'établissement de relations structure-fonction. 1,2
Déclarations de divulgation
The authors have nothing to disclose.
Remerciements
Le soutien financier fourni par le NIH HL89764, subventions de la NSF 0546012, 0730503 et 0725528.
matériels
| Name | Company | Catalog Number | Comments |
| SU-8 | MicroChem Corp. | SU-8 2025 | |
| PDMS | Dow Corning | Sylgard 184 | |
| Plasmid DNA | Clontech Laboratories | pEGFP-C1 | 4.9 kb, MW = 3.3 x 106 |
| LabelIT Biotin Labeling Kit | Mirus Bio LLC | MIR 3400 | Standard protocol yields labeling efficiency of approximately one label every 20-60 bp of double-stranded DNA, The density of labeling was adjusted in this work. |
| Streptavidin 605QD | Invitrogen | Qdot® 605 ITK® Streptavidin Conjugate | |
| Cy5-NHS Ester | Amersham | PA15101 | |
| Chitosan | Vanson | 390 kDa | 83.5% deacetylated |
| Cover Glass | Fisher Scientific | 12-545C | No. 1; Size: 40 x 22mm |
| Gastight Glass Syringe | Hamilton Co | TLL series | 50μL to 500μL depending on sample volume |
| Tygon Tubing | Small Parts, Inc. | 0.02 ID, 100ft | Tygon Tubes Microbore, 0.02 ID, 100ft |
| Connector | Small Parts, Inc. | HTX-23R | Customized in length of 0.750" |
| Syringe Pump | Harvard Apparatus | PHD-2000 | |
| CCD | QImaging | Intensified Retiga Cooled | |
| Microscope | Olympus Corporation | BX-51 | 100W mercury arc lamp |
| ImageJ | National Institutes of Health | v1.36b | http://rsb.info.nih.gov/ij |
| Origin Pro8 | OriginLab | Student Version | |
| Microscope Filter sets | Omega Optical | 475AF40 | Excitation filter in both channels |
| Microscope Filter sets | Omega Optical | 595AF60 | Emission filter in 605QD channel |
| Microscope Filter sets | Omega Optical | 670DF40 | Emission filter in QD-FRET channel |
| Microscope Filter sets | Omega Optical | 500 DRLP | Long pass dichroic in 605QD channel |
| Microscope Filter sets | Omega Optical | 595DRLP | Long pass dichroic in QD-FRET channel |
Références
- Ho, Y. P., Chen, H. H., Leong, K. W., Wang, T. H. Evaluating the intracellular stability and unpacking of DNA nanocomplexes by quantum dots-FRET. J Control Release. 116, 83-89 (2006).
- Chen, H. H. Quantitative comparison of intracellular unpacking kinetics of polyplexes by a model constructed from quantum dot-FRET. Mol. Ther. 16, 324-332 (2008).
- Zhang, C. Y., Yeh, H. C., Kuroki, M., Wang, T. H. Single-Quantum-Dot-Based DNA Nanosensor. Nat Mat. 4, 826-831 (2005).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon