Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Aplicación de métodos de flujo detenido Cinética para investigar el mecanismo de acción de una proteína de reparación del ADN
En este artículo
Resumen
MSH2, MSH6 es responsable de iniciar la reparación de errores de replicación del ADN. Aquí se presenta un enfoque cinética transitoria hacia la comprensión de cómo funciona esta proteína crítica. El informe pone de manifiesto de flujo detenido experimentos para medir la unión de ADN, acoplado y la cinética de la ATPasa subyacente MSH2, MSH6 mecanismo de acción de reparación del ADN.
Resumen
Análisis de la cinética transitoria es indispensable para entender el funcionamiento de las macromoléculas biológicas, ya que este método proporciona información mecanicista, incluidas las concentraciones del sitio activo y constantes intrínsecas que rigen la función tasa de macromoléculas. En el caso de las enzimas, por ejemplo, las mediciones de estado transitorio o constante pre-identificar y caracterizar los eventos individuales en el camino de reacción, mientras que las mediciones en estado estacionario sólo el rendimiento eficiencia global catalítica y la especificidad. Eventos individuales como las interacciones proteína-proteína o proteína-ligando y la limitación de la velocidad a menudo se producen cambios conformacionales en la escala de tiempo de milisegundos, y se puede medir directamente por flujo detenido y productos químicos apagar los métodos de flujo. Dada una señal óptica, tales como la fluorescencia, se detuvo el flujo sirve como una herramienta poderosa y accesible para el seguimiento de la reacción de la unión del sustrato para liberar el producto y el volumen de negocios 1,2 catalítico.
En este sentido, el informe de aplicación de la cinética de flujo detenido para investigar el mecanismo de acción de MSH2, MSH6, una proteína de reparación del ADN eucariota que reconoce pares de bases y los desajustes de inserción / deleción bucles en el ADN y la reparación de las señales de desequilibrio (MMR) 3-5. De este modo, MSH2, MSH6 aumenta la precisión de la replicación del ADN por tres órdenes de magnitud (frecuencia de errores disminuye a partir de ~ 10 -6 a 10 -9 bases), y por lo tanto ayuda a preservar la integridad genómica. No es de extrañar, defectos humanos MSH2, MSH6 función está asociada con el cáncer de colon hereditario no polipósico y otros cánceres esporádicos 6-8. A fin de comprender el mecanismo de acción de esta proteína ADN metabólica fundamental, que están investigando la dinámica de MSH2, MSH6 interacción con el ADN no coinciden, así como la actividad de la ATPasa de que los combustibles de sus acciones en la MMR. De unión al ADN se mide por la rápida mezcla MSH2, MSH6 con el ADN que contiene un 2-aminopurine (2-Ap) fluoróforo al lado de un G: T desajustes y el control de la consiguiente aumento de la fluorescencia de 2-aminopurine en tiempo real. ADN disociación se mide mediante la mezcla de pre-formado MSH2, MSH6 G: T (2-Ap) complejo de falta de coincidencia con el ADN trampa sin etiquetar y disminuir el control de la fluorescencia en el tiempo 9. Pre-ATPasa constante cinética de estado se mide por la variación en la fluorescencia de 7-dietilamino-3-((((2-maleimidyl) etil) amino) carbonil) cumarina)-etiquetados proteínas de unión a fosfato (MDCC-PBP) de fosfato de unión ( Pi), publicado por MSH2, MSH6 después de la hidrólisis de ATP 9,10.
Los datos revelan una rápida unión de MSH2, MSH6 a un G: no coincide el T y la formación de una larga vida MSH2, MSH6 G: T complejo, que a su vez se traduce en la supresión de la hidrólisis de ATP y la estabilización de la proteína en forma de ATP obligado . La cinética de la reacción proporcionan un claro apoyo a la hipótesis de que la ATP con destino MSH2, MSH6 señales de reparación del ADN en el enlace de un par de bases no coinciden en la doble hélice.
F. Noé Biro y Zhai Jie contribuido a este trabajo por igual.
Protocolo
A. La medición de la cinética MSH2, MSH6 de unión con ADN
1. Preparación de muestras para el experimento MSH2-MSH6 cinética de unión al ADN
Preparación de los reactivos para un experimento de ADN por fluorescencia basada en cinética de unión en un flujo detenido es similar a la de un experimento de equilibrio en un fluorómetro. De hecho el análisis de equilibrio de unión se debe realizar primero para estimar la constante de disociación (KD) para la interacción con el fin de optimizar las condiciones de reacción para el análisis cinético. De flujo detenido experimentos requieren grandes cantidades de materiales biológicos en comparación con los experimentos de equilibrio o de estado estable, por lo tanto, el enfoque es más factible cuando bajas cantidades de miligramo de proteína están disponibles 11,12 y cantidades similares de los ligandos pueden ser preparados o comprados.
- Estamos sobre-expresan S. cerevisiae MSH2, MSH6 en E. coli y purificar cantidades de miligramos de la proteína por cromatografía de intercambio iónico (Fig. 1) 11.
- Reactivos de ADN, con o sin el fluoróforo 2-aminopurine pueden sintetizarse químicamente. Compramos 37 nucleótidos de longitud de cadena simple ADN modificado con 2-aminopurine y recocido dos hebras complementarias para preparar ADN de doble cadena con 2-aminopurina junto a un G: no coincide T (Fig. 2). ADN puede ser purificada por el fabricante o en el laboratorio mediante la desnaturalización electroforesis en gel de poliacrilamida, este último por lo general ofrece un mayor rendimiento.
- Para la cinética de unión al ADN, preparar por separado G: T (2-Ap) de ADN y MSH2, MSH6 muestras de la proteína en tampón de unión al ADN (20 mM Tris-HCl, pH 8,0, MgCl2 5 mM, 100 mM NaCl). MSH2, MSH6 y las concentraciones de ADN son 0,8 M y M, respectivamente, 0,12, lo que produce 0,4 M y 0,06 M concentraciones finales en un solo experimento la mezcla con una proporción de 1:1 de mezcla. De ADN cinética de disociación, preparar una muestra con 0,8 M MSH2-MSH6 y 0,12 M G: T (2-Ap) de ADN, y otra muestra con 8 M sin etiquetar G: ADN-T como una trampa de forma gratuita MSH2-MSH6. Preparar y almacenar las muestras en hielo. En estas reacciones, la concentración de proteínas es muy superior al 10 nM K D para la interacción MSH2, MSH6-ADN y la concentración de ADN es suficiente para una señal de fluorescencia robusto (determinada empíricamente). Un volumen de 400 l por muestra es suficiente para obtener alrededor de 10 huellas cinética (Tabla 1).
- Utilizar productos químicos de alta calidad que estén libres de impurezas fluorescentes de la proteína, el ADN, y los preparativos de amortiguamiento. Filtro de todos los búferes a través de una membrana de 0,2 micras para evitar la obstrucción del flujo detenido con partículas.
- Si el fluoróforo absorbe la luz visible, la preparación y el experimento debe realizarse bajo condiciones de poca luz.
2. Instrumento de preparación para el MSH2, MSH6 la cinética de unión al ADN
Un instrumento de flujo detenido es bastante simple en principio. Utiliza un motor para impulsar rápidamente dos soluciones en jeringuillas unidad en un dispositivo de mezcla, la solución mezclada pasa entonces a una celda de observación para la recogida de datos (Fig. 3). Usamos la KinTek de flujo detenido, lo que requiere un volumen de muestra de baja, permite la detección individual o secuencial de mezcla de reactivos, de una variedad de señales ópticas, y es muy fácil de usar. De flujo detenido instrumentos están disponibles en varios otros fabricantes también.
- Con el fin de preparar el instrumento de flujo detenido para experimentos, asegúrese de que el equipo y el controlador están desactivados. A su vez en el baño de agua circulante ajustado a la temperatura ambiente (25 grados; C) para enfriar la lámpara. Encender la lámpara. Establecer el monocromador a la longitud de onda de excitación deseado (315 nm en el caso de 2-aminopurine). Gire la rueda de corte a la anchura de la apertura deseada (empíricamente excitación balance fluoróforo alta photobleaching baja). Encienda el controlador y el ordenador. A su vez en el baño de agua circulante para llenar la camisa de agua que mantiene los reactivos a la temperatura deseada durante el experimento (30 ° C en el caso de S. cerevisiae, MSH2, MSH6).
- A continuación, ejecutar el programa de flujo detenido. Encienda el detector de elección, que en este caso es un tubo fotomultiplicador (PMT) con un filtro de interferencia apropiado para el fluoróforo (350 nm de corte del filtro en el caso de 2-aminopurine). Aplicar la tensión de la PMT. Medir la corriente oscura para corregir cualquier ruido de fondo eléctrico.
- Las jeringas de unidad de flujo detenido y celda de observación deben ser lavados antes de cargar las muestras. Ajuste de la válvula de carga de la muestra a la posición de carga. Llene una jeringa de 1 ml con agua destilada, filtrada, conéctelo al puerto de carga ubicado debajo de la jeringa de la unidad, y empujar manualmente el agua entre las dos jeringas varias veces. Repite el proceso dos veces por cada jeringa unidad que se utilizará en el experimento. Luego, llene el disco jeringas con agua para lavar la celda de observación. Cambie la válvula de carga de la muestra a la posición de fuego. Use la unidad de jeringa Ajustecomando, que controla el motor y baje la placa de la unidad para impulsar el agua a través de la celda de observación y en la línea de salida. Tenga cuidado de no empujar el émbolo hasta el final de la jeringa de la unidad. Levante la placa de la unidad. Cambie la válvula a la posición de carga. Manual de lavar las jeringas con el tampón de reacción y dejar vacío. El instrumento está ahora listo para su uso.
3. MSH2, MSH6 experimento de unión de ADN y análisis de datos
- Transfiera cada muestra del tubo a una jeringa de nuevo 1. Coloque cada jeringa de la muestra a un puerto de carga y empujar la solución en la jeringa unidad. Tenga cuidado de eliminar todas las burbujas de aire de forma manual presionando la solución entre los dos jeringas de un par de veces con pausas intermitentes. Baje la placa de la unidad hasta que haga contacto con la parte superior de la unidad de jeringas. Deje que los reactivos alcancen la temperatura ambiente durante unos minutos.
- Para la recolección de datos, abra el tiempo establecido / ventana de Canales, seleccione el canal de recogida de datos (PMT), el modo de análisis de datos (Fluorescencia), y el número de trazas (vacunas) para ser recogidos. Introduzca la hora de la recolección de datos durante el cual los puntos de 1000 se recopilarán datos. Una reacción desconocida deben ser controlados durante varios segundos con el fin de detectar cualquier fases lentas. Empíricamente estimar el tiempo requerido para alcanzar el equilibrio o el estado de equilibrio y establecer el tiempo de recolección de datos para la vida media ≥ 6. En el caso de MSH2, MSH6 el marco de tiempo óptimo es de 2 segundos para G: cinética T desajuste vinculante y 60 segundos para G: T cinética de disociación. Ahora colocar la válvula en la posición de fuego y haga clic en el botón Recopilar datos. Esta acción inicia la mezcla de MSH2, MSH6 y el ADN, la entrada de la reacción en la celda de observación, la excitación del fluoróforo 2-aminopurine y recogida de la señal de fluorescencia con el tiempo. Recoge al menos cinco rastros cinética para obtener datos de alta calidad y guardar el archivo.
- Cuando el experimento se ha completado, a su vez de la lámpara. Lave las jeringas y las celdas de observación con el agua como se describió anteriormente. A continuación, apague el resto del equipo, a excepción del baño de agua circulante. El agua se distribuye por otros 15 minutos para enfriar la lámpara.
- Operaciones matemáticas y ajuste de datos se puede realizar con el software KinTek sí mismo, con el fin de tener una idea de la cinética de reacción durante el experimento. El análisis posterior también puede ser realizada por un promedio de varios rastros cinética y guardar y exportar el archivo a un promedio de otros análisis de datos / gráficos de software.
4. Los resultados representativos de la cinética de MSH2, MSH6 de unión al ADN
Los datos cinéticos para MSH2, MSH6 interacciones con G: ADN-T desajuste, puede estar en forma para una función exponencial simple y dar una rápida tasa de k constante de unión en cerca de 3 x 10 7 M-1 segundo-1 (Fig. 4A) y una lenta constante de disociación k OFF de 0,012 segundos -1 (Fig. 4B), que revela que el MSH2, MSH6 se une a G: T desajuste rápidamente y forma un complejo muy estable con una vida media cerca de 60 segundos 13.
B. Medición de MSH2, MSH6 Cinética ATPasa
1. Preparación de muestras para el experimento MSH2-ATPasa MSH6 cinética
- Prepare MSH2, MSH6 y reactivos de ADN como se describió anteriormente, utilizando el ADN no marcado. Además, purificar la proteína de unión a fosfato (PBP) de E. coli y la etiqueta con el fluoróforo MDCC (Fig. 5) 14,15. MDCC-PBP se une fosfatos rápidamente (k ON es igual a 1 x 10 8 M -1 segundo -1) y con alta afinidad (K D = 100 nm), lo que resulta en un aumento dramático en MDCC fluorescencia (Fig. 6). MDCC-PBP es por lo tanto, un periodista de sólida pre-estado estacionario hidrólisis de ATP y la liberación de fosfato de la cinética de 14,15. Tenga en cuenta que es esencial para reducir al mínimo los niveles de base de fosfato para este ensayo, por lo tanto, el uso de vidrio deben ser estrictamente evitados en todas las etapas de preparación de los reactivos.
- Preparar una muestra con 4 M MSH2, MSH6 proteínas con o sin G M 6: T ADN, lo que da 2 M y 3 M concentraciones finales en un solo experimento la mezcla con una proporción de 1:1 de mezcla. Tenga en cuenta que antes de la constante experimentación estatales requieren altas concentraciones de enzimas ya que la señal de interés es de un volumen de negocio único o el primer balón catalizador pocos. Preparar otra muestra que contiene 1 mM recién disuelto ATP y 20 M reportero MDCC-PBP. Añadir 0,10 unidades / ml de purina nucleósido fosforilasa y 200 M de 7 methylguanosine a las muestras, que sirve para limpiar cualquier fosfato contaminantes. Incubar las muestras en hielo durante al menos 20 minutos (Tabla 2).
2. Instrumento de preparación para la cinética de MSH2, MSH6 ATPasa
- Preparación del instrumento de flujo detenido para experimentos como se describió anteriormente. Además, limpiar los contaminantes de fosfato de la unidad de jeringas con 0,5 unidades / ml purina nucleoside fosforilasa y 200 M de 7 methylguanosine durante 20 minutos. La longitud de onda de excitación a 425 nm y 450 nm utilizan un filtro de corte con el PMT para la detección de MDCC-PBP fluorescencia.
3. MSH2, MSH6 ATPasa experimento y análisis de datos
- Cargue el disco jeringas con las muestras como se describe anteriormente. Haga clic en recopilar datos para mezclar MSH2, MSH6 con ATP y MDCC PBP-y la liberación de fosfato en un monitor de MSH2, MSH6 y el aumento acoplados en MDCC-PBP fluorescencia con el tiempo. Recoge al menos cinco rastros cinética para obtener una alta calidad de conjunto de datos y guardar el archivo. Promedio de las huellas cinética múltiples y exportar el archivo de datos para el análisis.
- Preparar una curva de calibración para determinar la relación lineal entre la concentración de fosfato y de fluorescencia MDCC-PBP. Con el fin de hacerlo, mezcla MDCC-PBP con diferentes concentraciones de fosfato en las mismas condiciones experimentales en el flujo detenido, y la medida máxima MDCC-PBP de fluorescencia en cada concentración de fosfato.
- Trazar la máxima MDCC-PBP fluorescencia frente a cada concentración de fosfato para la curva de calibración (Fig. 7) y el uso de la pendiente de obtener la concentración de fosfato en las reacciones de MSH2, MSH6. Parcela concentración de fosfato en función del tiempo y ajustar los datos a una función exponencial y lineal. Esta función se describe una explosión de la pre-estado estacionario hidrólisis de ATP y la liberación de fosfato seguido por el estado de fase lineal constante de la reacción.
4. Los resultados representativos para MSH2, MSH6 cinética ATPasa
Los datos cinéticos indican que MSH2, MSH6 hidroliza ATP y fosfato libera rápidamente a 1,4 segundos -1 en la rotación de catalizador en primer lugar. Esta fase es seguida por un paso lento en la reacción que los límites de pérdidas de balón después de un lanzamiento de 7 veces más lenta gato k constante estado de 0,2 segundos -1 (Fig. 8A). Sin embargo, cuando MSH2, MSH6 se une al ADN no coinciden, el estallido de la hidrólisis de ATP y la liberación de fosfato se suprime, y MSH2, MSH6 permanece en un estado de ATP con destino a un tiempo más largo.

Figura 1. Purificación de S. cerevisiae MSH2, MSH6 de E. coli. MSH2 y MSH6 genes fueron clonados en el vector pET11a y sobre-expresado en E. coli BL21 (DE3) las células. El complejo de la proteína se purificó mediante cromatografía en columna sobre SP-sefarosa, la heparina, y las resinas de Q-sefarosa. SDS-PAGE análisis muestra contiene fracciones de proteína de una columna de Q-sefarosa.
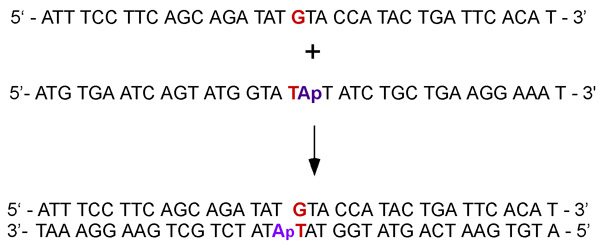
. Figura 2 complementario de cadena sencilla ADN son recocidos para formar un dúplex que contiene G: no coincide con una T de adenosina adyacentes (para los experimentos de la ATPasa) o 2-aminopurine analógico base de fluorescentes de adenosina (para los experimentos de unión al ADN).
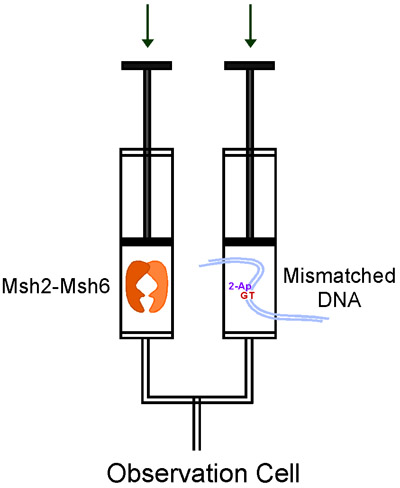
Figura 3. Flujo de reactivos en el KinTek flujo detenido durante un experimento de mezcla única.

Figura 4a Cinética de MSH2, MSH6 interacción con una G:. Desajuste T. La mezcla de ADN de doble cadena (0,12 M) que contiene una G: junto a 2-T con aminopurina MSH2, MSH6 (0,8 M) lleva a un aumento en la fluorescencia con el tiempo, y se obtiene un índice k bimolecular constante ON = 2.4 10 7 M -1 s -1 para la interacción.
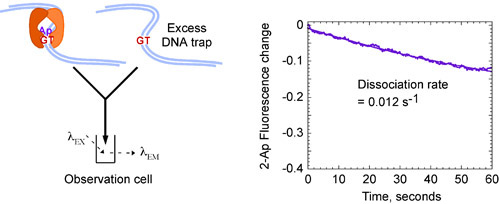
Figura 4b Cinética de MSH2, MSH6 interacción con una G:. Desajuste T. Mezcla preformada MSH2, MSH6 G: T (2-Ap) complejo con el exceso de etiqueta G: T ADN (8 M) que atrapa a cualquiera sin MSH2, MSH6 conduce a la disminución de la fluorescencia con el tiempo, y produce una lenta tasa constante de disociación, k OFF = 0,012 s-1, lo que indica un complejo estable con una vida media larga de 60 segundos. Las concentraciones finales: 0,4 M MSH2-MSH6, 0,06 M de ADN marcado, 4 M sin etiquetar G: trampa T del ADN.
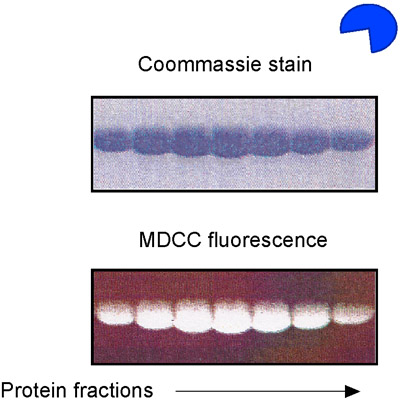
Figura 5. MDCC-PBP preparación. SDS-PAGE análisis de E. coli proteína de unión a fosfato (PBP), purificado y marcado con el fluoróforo MDCC.
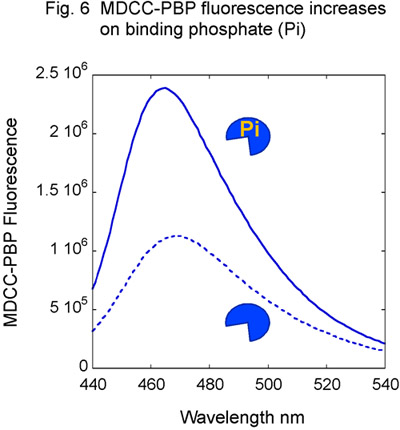
Figura 6. MDCC-PBP respuesta al fosfato. La titulación de MDCC-PBP con fosfato (Pi) resulta en el incremento de fluorescencia MDCC.

Figura 7a. Preparación del fosfato (Pi) curva estándar. MDCC-PBP (20 M) se mezcla con cantidades variables de Pi (0 - 8 M) unad fluorescencia medida a través del tiempo hasta alcanzar el equilibrio. Las concentraciones finales: 10 M MDCC-PBP, 0-4 Pi M.
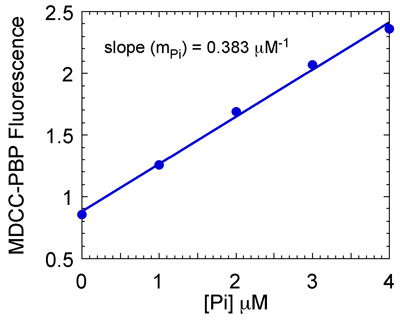
Figura 7b. Preparación del fosfato (Pi) curva estándar. Máximo MDCC-PBP fluorescencia se representa frente [pi] para obtener una curva estándar. La pendiente de la línea (0.383 M -1, en este caso) se utiliza para convertir MDCC-PBP fluorescencia en la concentración de Pi.
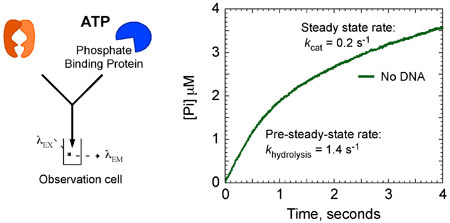
Figura 8a. MSH2, MSH6 ATPasa cinética. MSH2, MSH6 (4 M), en ausencia G: ADN-T, se mezcla rápidamente con el ATP (1 mM) y MDCC-PBP (20 M) muestra una explosión de la hidrólisis de ATP y la liberación Pi. Análisis de los datos se obtiene la tasa (hidrólisis k = 1,4 s -1) y amplitud (2 micras; un sitio por MSH2, MSH6) de la fase de explosión, la cual es seguida por una fase lineal, el estado de equilibrio a un ritmo de 0,4 s M - 1 (k cat = 0,2 s -1).
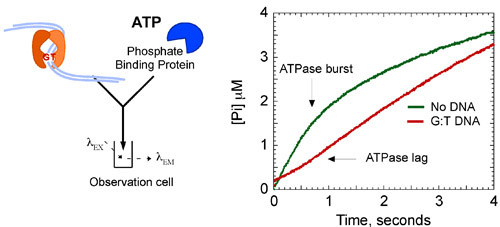
Figura 8b. MSH2, MSH6 cinética ATPasa. Además de la G: ADN-T para la reacción (6 M) suprime el estallido de la hidrólisis de ATP, estabilizar el complejo en un estado de ATP de ruedas. Las concentraciones finales: 2 M MSH2-MSH6, 500 mM ATP, tres de ADN M, 10 M MDCC-PBP. Cinética de ráfaga se ajustan por la siguiente ecuación: [Pi] = A 0 e-t + k Vt, donde [pi] es la concentración de fosfato, A 0 es la amplitud de la explosión, k es la velocidad de tiro observado constante y V es la velocidad de la fase lineal (k cat = V / [MSH2, MSH6]).
| Muestra | Proteína | ADN | ||||
| Reactivo | Valores | De Trabajo | Vol, l | Valores | De Trabajo | Vol, l |
| MSH2, MSH6 | 5 M | 0,8 M | 64 | - | - | - |
| 2ApG: T | - | - | - | 10 M | 0,12 M | 4.8 |
| buffer | 10 veces | 1x | 40 | 10 veces | 1x | 40 |
| ddH2O | - | - | 296 | - | - | 355 |
| Total | 400 | 400 | ||||
Tabla 1 reacción de unión de ADN
| Muestra | Proteína | Proteína-ADN | ATP | ||||||
| Reactivo | Valores | De Trabajo | Vol, l | Valores | De Trabajo | Vol, l | Valores | De Trabajo | Vol, l |
| MSH2, MSH6 | 20 M | 4 M | 80 | 20 M | 4 M | 80 | - | - | - |
| G: T | - | - | - | 100 | 6 | 24 | - | - | - |
| ATP | - | - | - | - | - | - | 50 mM | 1 mM | 8 |
| 7-MEG | 250x | 1x | 1.6 | 250x | 1x | 1.6 | 250x | 1x | 1.6 |
| PNPase | 100 veces | 1x | 4 | 100 veces | 1x | 4 | 100 veces | 1x | 4 |
| PBP-MDCC | 150 M | 20 M | 53.3 | 150 M | 20 M | 53.3 | - | - | - |
| Buffer | 10 veces | 1x | 40 | 10 veces | 1x | 40 | 10 veces | 1x | 40 |
| ddH2O | - | - | 221 | - | - | 197 | - | - | 346 |
| Total | 400 | 400 | 400 | ||||||
Tabla 2 ATPasa reacción
Discusión
El ejemplo de una proteína de unión al ADN desajuste se describe aquí se muestra la eficacia y utilidad de los métodos cinéticos transitoria para el estudio de los mecanismos de las moléculas biológicas. Mediciones de flujo detenido en la escala de tiempo de rotación único previsto una evidencia inequívoca de la unión rápida y específica de la proteína MSH2, MSH6 a un par de bases coincidentes y la formación de un complejo de ADN de larga duración proteína X en la reacción 9. Por otra parte,...
Agradecimientos
Este trabajo fue apoyado por un premio CARRERA NSF (MMH), un Barry M. Goldwater de becas (FNB) y un Premio de Investigación de Pregrado ASBMB (CWD). El clon de la sobre-expresión de PBP fue proporcionado amablemente por el Dr. Martin Webb (MRC, Reino Unido).
Materiales
| ADN nombre | Secuencia |
| 37 G | 5'-TTC TCC AGC ATT TAT AGA G TA CCA TAC TGA ACA TTC T -3 ' |
| 37 T (2-Ap) | 5'-ATG TGA ATC ATG AGT GTA T Ap T TGC ATC TGA AGG AAA T -3 ' |
| 37 T | 5'-ATG TGA ATC ATG AGT GTA T A T TGC ATC TGA AGG AAA -3 T ' |
Referencias
- Johnson, K. A. Advances in transient-state kinetics. Curr Opin Biotechnol. 9 (1), 87-89 (1998).
- Johnson, K. A. E. . Kinetic analysis of macromolecules. , (2003).
- Obmolova, G., Ban, C., Hsieh, P., Yang, W. Crystal structures of mismatch repair protein MutS and its complex with a substrate DNA. Nature. 407 (6805), 703-710 (2000).
- Lamers, M. H. The crystal structure of DNA mismatch repair protein MutS binding to a G x T mismatch. Nature. 407 (6805), 711-717 (2000).
- Warren, J. J. Structure of the human MutSalpha DNA lesion recognition complex. Mol Cell. 26 (4), 579-592 (2007).
- Kunkel, T. A. &. a. m. p. ;. a. m. p., Erie, D. A. . DNA Mismatch Repair. Annu Rev Biochem. 74, 681-710 (2005).
- Jiricny, J. The multifaceted mismatch-repair system. Nat Rev Mol Cell Biol. 7 (5), 335-346 (2006).
- Hsieh, P., Yamane, K. DNA mismatch repair: Molecular mechanism, cancer, and ageing. Mech Ageing Dev. 129 (7-8), 391-407 (2008).
- Jacobs-Palmer, E., Hingorani, M. M. The effects of nucleotides on MutS-DNA binding kinetics clarify the role of MutS ATPase activity in mismatch repair. J Mol Biol. 366 (4), 1087-1098 (2007).
- Antony, E., Khubchandani, S., Chen, S., Hingorani, M. M., M, M. Contribution of Msh2 and Msh6 subunits to the asymmetric ATPase and DNA mismatch binding activities of Saccharomyces cerevisiae Msh2-Msh6 mismatch repair protein. DNA Repair (Amst). 5 (2), 153-162 (2006).
- Antony, E., Hingorani, M. M. Mismatch recognition-coupled stabilization of Msh2-Msh6 in an ATP-bound state at the initiation of DNA repair. Biochemistry. 42 (25), 7682-7693 (2003).
- Finkelstein, J., Antony, E., Hingorani, M. M., O'Donnell, M. Overproduction and analysis of eukaryotic multiprotein complexes in Escherichia coli using a dual-vector strategy. Anal Biochem. 319 (1), 78-87 (2003).
- Zhai, J., Hingorani, M. M. S. cerevisiae Msh2-Msh6 DNA binding kinetics reveal a mechanism of targeting sites for DNA mismatch repair. Proc Natl Acad Sci U S A. 107 (2), 680-685 (2010).
- Brune, M., Hunter, J. L., Corrie, J. E., Webb, M. R. Direct, real-time measurement of rapid inorganic phosphate release using a novel fluorescent probe and its application to actomyosin subfragment 1 ATPase. Biochemistry. 33 (27), 8262-8271 (1994).
- Brune, M. Mechanism of inorganic phosphate interaction with phosphate binding protein from Escherichia coli. Biochemistry. 37 (29), 10370-10380 (1998).
- Antony, E., Hingorani, M. M. Asymmetric ATP binding and hydrolysis activity of the Thermus aquaticus MutS dimer is key to modulation of its interactions with mismatched DNA. Biochemistry. 43 (41), 13115-13128 (2004).
- Gradia, S., Acharya, S., Fishel, R. The human mismatch recognition complex hMSH2-hMSH6 functions as a novel molecular switch. Cell. 91 (7), 995-1005 (1997).
- Mazur, D. J., Mendillo, M. L., Kolodner, R. D., D, R. Inhibition of Msh6 ATPase activity by mispaired DNA induces a Msh2(ATP)-Msh6(ATP) state capable of hydrolysis-independent movement along DNA. Mol Cell. 22 (1), 39-49 (2006).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados