È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Applicazione di metodi Stopped-flow Cinetica per Indagare sulla Meccanismo d'azione di una proteina di riparazione del DNA
In questo articolo
Riepilogo
MSH2-MSH6 è responsabile per l'avvio di riparazione di errori di replicazione del DNA. Qui vi presentiamo un approccio transitorio cinetica verso la comprensione di come lavora questa proteina critica. Il rapporto illustra stopped-flow esperimenti per misurare il legame del DNA accoppiati e cinetica ATPasi sottostante MSH2-MSH6 meccanismo d'azione nella riparazione del DNA.
Abstract
Transitori analisi cinetica è indispensabile per comprendere il funzionamento delle macromolecole biologiche, dal momento che questo approccio produce informazioni meccanicistico, inclusa la concentrazione sito attivo e costanti di velocità intrinseche che regolano la funzione macromolecolari. In caso di enzimi, per esempio, le misurazioni stato transitorio o pre-steady identificare e caratterizzare gli eventi individuali nel percorso di reazione, mentre le misurazioni stato stazionario solo resa complessiva efficienza catalitica e specificità. Singoli eventi, come proteina-proteina o proteina-ligando interazioni e rate-limiting cambiamenti conformazionali spesso si verificano in tempi millisecondo, e può essere misurato direttamente per stopped-flow e chimico-tempra metodi di flusso. Dato un segnale ottico come fluorescenza, stopped-flow è uno strumento potente ed accessibile per il progresso reazione monitoraggio da substrato vincolante al rilascio del prodotto e fatturato 1,2 catalitica.
Qui riportiamo applicazione di stopped-flow cinetica per sondare il meccanismo d'azione di MSH2-MSH6, una proteina eucariotica riparazione del DNA che riconosce base-pair mismatch e inserzione / delezione loop nel DNA e mancata corrispondenza dei segnali di riparazione (MMR) 3-5. In tal modo, MSH2-MSH6 aumenta l'accuratezza della replicazione del DNA da tre ordini di grandezza (frequenza di errore si riduce da ~ 10 -6 a 10 -9 basi), e quindi aiuta a preservare l'integrità genomica. Non sorprende, difettosi umano MSH2-MSH6 funzione è ereditario non associato a poliposi-cancro del colon e altri tumori sporadici 6-8. Al fine di comprendere il meccanismo d'azione di questa proteina fondamentale del metabolismo del DNA, stiamo sondando le dinamiche di MSH2-MSH6 interazione con il DNA non corrispondenti così come l'attività ATPasi che alimenta le sue azioni in MMR. Legame del DNA viene misurata mediante una rapida miscelazione MSH2-MSH6 con il DNA che contiene un 2-aminopurine (2-Ap) fluoroforo adiacente ad un G: T mancata corrispondenza e di monitoraggio il conseguente aumento della 2-aminopurine fluorescenza in tempo reale. DNA dissociazione si misura con la miscelazione pre-formate MSH2-MSH6 G: T (2-Ap) complesso di mancata corrispondenza con il DNA trappola senza etichetta e la diminuzione di monitoraggio della fluorescenza nel tempo 9. Pre-costante cinetica ATPasi statali sono misurati con la variazione di fluorescenza del 7-dietilammino-3-((((2-maleimidyl) etil) ammino) carbonile) cumarina)-etichettati fosfato Binding Protein (MDCC-PBP) su fosfato di legame ( Pi) pubblicato da MSH2-MSH6 seguenti idrolisi dell'ATP 9,10.
I dati rivelano vincolante rapida MSH2-MSH6 ad un G: mancata corrispondenza T e la formazione di una lunga durata MSH2-MSH6 G: T complesso, che a sua volta provoca la soppressione di idrolisi dell'ATP e la stabilizzazione della proteina in un ATP form associato . La cinetica di reazione di fornire un chiaro sostegno per l'ipotesi che l'ATP-bound-MSH2 MSH6 segnali di riparazione del DNA su una coppia di legame non corrispondenti base nella doppia elica.
F. Noè Biro e Jie Zhai contribuito a questo documento allo stesso modo.
Protocollo
A. Misura della MSH2-MSH6 DNA Cinetica Binding
1. Preparazione del campione per il MSH2-MSH6 DNA esperimento di cinetica di binding
Preparazione di reagenti per una fluorescenza basato esperimento DNA cinetica vincolante per uno stopped-flow è simile a quello per un esperimento di equilibrio su un fluorimetro. In effetti l'analisi di equilibrio vincolante dovrebbe essere eseguito prima a stimare la costante di dissociazione (K D) per l'interazione al fine di ottimizzare le condizioni di reazione per l'analisi cinetica. Stopped-flow esperimenti richiedono grandi quantità di materiali biologici rispetto a esperimenti di equilibrio o steady-state, quindi, l'approccio è più fattibile se a bassa quantità dell'ordine di milligrammi di proteine sono disponibili 11,12 e quantità simili di leganti possono essere preparati o acquistati.
- Siamo oltre-express S. cerevisiae MSH2-MSH6 in E. coli e purificare quantità milligrammo della proteina da cromatografia a scambio ionico (Fig. 1) 11.
- Reagenti del DNA, con o senza il 2-Aminopurine fluoroforo può essere sintetizzata chimicamente. Acquistiamo 37 nucleotidi di lunghezza a singolo filamento di DNA modificato con 2-Aminopurine e temprare due elementi complementari per la preparazione del DNA duplex con 2-Aminopurine adiacente ad un G: mancata corrispondenza T (Fig. 2). DNA può essere purificato dal produttore o in laboratorio denaturazione elettroforesi su gel di poliacrilamide, la seconda dà generalmente rendimenti più elevati.
- Per la cinetica di legame al DNA, preparare a parte G: T (2-Ap) DNA e MSH2-MSH6 campioni di proteine in un tampone di legame al DNA (20 mM Tris-HCl, pH 8,0, 5 mM MgCl 2, 100 mM NaCl). MSH2-MSH6 e le concentrazioni di DNA sono 0,8 micron e 0,12 mM, rispettivamente, che produce 0,4 mM e 0,06 mM finale concentrazione in un singolo esperimento di miscelazione con rapporto 1:1 miscelazione. Per DNA cinetica dissociazione, preparare un campione contenente 0,8 micron MSH2-MSH6 e 0,12 mM G: T (2-Ap) del DNA, e un altro campione con 8 mM senza etichetta G: DNA T come una trappola per libero MSH2-MSH6. Preparare e conservare i campioni in ghiaccio. In queste reazioni, concentrazione di proteine è nettamente superiore alla nM 10 K D per MSH2-MSH6-DNA l'interazione e la concentrazione del DNA è sufficiente per un segnale forte fluorescenza (determinato empiricamente). Un volume di 400 microlitri per campione è sufficiente per ottenere circa 10 tracce cinetica (Tabella 1).
- Usare prodotti chimici di alta qualità che siano privi di impurità nella proteina fluorescente, il DNA, e preparati buffer. Filtrare tutti i buffer attraverso una membrana 0,2 micron per evitare di intasare la stopped-flow con il particolato.
- Se il fluoroforo assorbe la luce visibile, la preparazione e la sperimentazione deve essere effettuata in condizioni di scarsa illuminazione.
2. Preparazione dello strumento per la MSH2-MSH6 legano il DNA cinetica
Un stopped-flow strumento è piuttosto semplice in linea di principio. Utilizza un motore per spingere rapidamente due soluzioni in siringhe disco in un dispositivo di miscelazione, la soluzione mista passa successivamente in un cella di osservazione per la raccolta dei dati (Fig. 3). Usiamo il KinTek stopped-flow, che richiede un basso volume del campione, permette la rilevazione singolo o sequenziale miscelazione di reagenti, di una varietà di segnali ottici, ed è molto facile da usare. Stopped-flow strumenti sono disponibili da diversi altri produttori come bene.
- Al fine di preparare la stopped-flow strumento per esperimenti, assicurarsi che il computer e il controller siano spenti. Accendere il bagno d'acqua in circolazione insieme a temperatura ambiente (25 °, C) per raffreddare la lampada. Accendere la lampada. Impostare il monocromatore per la lunghezza d'onda di eccitazione desiderato (315 nm nel caso di 2-Aminopurine). Girare la ruota fessura per la larghezza della fenditura desiderata (empiricamente eccitazione fluoroforo equilibrio alta con photobleaching basso). Accendere il controller e il computer. Accendere il bagno d'acqua in circolazione per riempire la camicia d'acqua che mantiene i reagenti a temperatura desiderato durante l'esperimento (30 ° C in caso di S. cerevisiae MSH6-MSH2).
- Successivamente, eseguire il stopped-flow del programma. Accendere il rivelatore di scelta, che in questo caso è un tubo fotomoltiplicatore (PMT) con un filtro di interferenza appropriato per il fluoroforo (350 nm cut-off del filtro in caso di 2-Aminopurine). Applicare tensione al PMT. Misurare la corrente di buio per correggere qualsiasi rumore di fondo elettrico.
- I stopped-flow siringhe unità e l'osservazione delle cellule devono essere lavate prima di caricare i campioni. Impostare la valvola di carico di esempio per la posizione di carico. Riempire una siringa da 1 ml con acqua deionizzata, acqua filtrata, collegarlo alla porta di carico situato sotto la siringa unità e spingere manualmente acqua tra le due siringhe un paio di volte. Ripetere la procedura due volte per ogni siringa unità da utilizzare nell'esperimento. Successivamente, riempire le siringhe drive con acqua per lavare la cellula di osservazione. Accendere la valvola di carico di esempio per la posizione di FUOCO. Utilizzare l'unità Regolare siringacomando, che controlla il motore, e abbassare il piatto disco per spingere l'acqua attraverso la cella di osservazione e nella linea di uscita. Fare attenzione a non spingere il pistone fino alla fine della siringa unità. Sollevare la piastra di unità. Passa la valvola in posizione LOAD. Manualmente lavare le siringhe con il tampone di reazione e lasciare vuoti. Lo strumento è ora pronto per l'uso.
3. MSH2-MSH6 DNA esperimento vincolante e analisi dei dati
- Trasferire ciascun campione dal tubo di una nuova siringa da 1 ml. Collegare ogni siringa campione a una porta di carico e spingere la soluzione nella siringa unità. Aver cura di rimuovere tutte le bolle d'aria manualmente spingendo la soluzione tra le due siringhe con un paio di volte intermittenti pause. Abbassare la piastra di guidare finché non tocca la parte superiore del disco siringhe. Lasciate che i reagenti a temperatura ambiente per qualche minuto.
- Per la raccolta dati, aprire il Set Time / finestra Canali, selezionare la raccolta dei dati canale (PMT), modalità di analisi dei dati (fluorescenza), e il numero di tracce (Shots) da raccogliere. Inserisci la raccolta dati in tempo durante il quale i punti 1000 dati saranno raccolti. Una reazione sconosciuto deve essere monitorato più di alcuni secondi al fine di rilevare eventuali fasi di lento. Empiricamente stimare il tempo necessario per raggiungere l'equilibrio o stato stazionario e di raccolta dati in tempo a ≥ 6 dimezzamento. Nel caso di MSH2-MSH6 l'intervallo di tempo ottimale è di 2 secondi per G: cinetica mancata corrispondenza T vincolante e 60 secondi per G: T cinetica dissociazione. Ora impostare la valvola in posizione di FUOCO e fare clic sul pulsante Incassa dati. Questa azione avvia miscelazione di MSH2-MSH6 e il DNA, l'ingresso della reazione nella cella di osservazione, l'eccitazione del 2-Aminopurine fluoroforo e la raccolta del segnale di fluorescenza nel tempo. Raccogliere almeno 5 tracce cinetiche di ottenere dati di alta qualità e salvare il file.
- Quando l'esperimento è completa, volta della lampada. Lavare le siringhe e la cella di osservazione con l'acqua come descritto in precedenza. Quindi, spegnere il resto del materiale, ad eccezione del bagno d'acqua in circolo. L'acqua viene fatta circolare per altri 15 minuti per raffreddare la lampada.
- Operazioni matematiche e raccordo dei dati può essere effettuata con il software KinTek stessa, al fine di ottenere un senso della cinetica di reazione durante l'esperimento. Un'ulteriore analisi può essere eseguita anche la media più tracce cinetica e il salvataggio e l'esportazione del file media ad altre analisi dei dati / grafici software.
4. Risultati rappresentativi per MSH2-MSH6 cinetica di legame del DNA
Dati cinetici MSH2-MSH6 interazioni con G: DNA mancata corrispondenza T, può essere adatto a una singola funzione esponenziale e produrre un k veloce tasso di legame costante ON vicino a 3 x 10 7 M-1 secondo-1 (Fig. 4A) e un lento dissociazione costante k OFF di 0,012 secondi -1 (Fig. 4B), che rivela che il MSH2-MSH6 lega una G: mancata corrispondenza T rapidamente e forma un complesso molto stabile, con un'emivita di chiudere a 60 secondi 13.
B. Misure di MSH2-MSH6 Cinetica ATPasi
1. Preparazione del campione per il MSH2-MSH6 esperimento ATPasi cinetica
- Preparare MSH2-MSH6 e reagenti DNA come descritto in precedenza, usando il DNA senza etichetta. Inoltre, purificare Binding Protein fosfato (PBP) da E. coli e l'etichetta con il fluoroforo MDCC (Fig. 5) 14,15. MDCC-PBP lega il fosfato liberi rapidamente (K è uguale a 1 x 10 8 M -1 secondo -1) e con alta affinità (K D = 100 nM), con un conseguente drammatico aumento MDCC fluorescenza (Fig. 6). MDCC-PBP è quindi un reporter robusta di idrolisi dello stato di pre-steady ATP e rilasciare fosfato cinetica 14,15. Si noti che è essenziale per minimizzare i livelli di fosfato di fondo di questo saggio, quindi, l'uso del vetro deve essere rigorosamente evitato in tutte le fasi di preparazione di reagenti.
- Preparare un campione con 4 mM MSH6 proteine MSH2-con o senza mM G 6: T del DNA, che produce 2 mM e 3 mM concentrazioni finali in un singolo esperimento di miscelazione con rapporto 1:1 miscelazione. Si noti che pre-steady esperimenti di stato richiedono elevate concentrazioni di enzimi in quanto il segnale di interesse da un fatturato singolo o il primo fatturati pochi catalitica. Preparare un altro campione contenente 1 mM ATP appena sciolto e 20 mM MDCC PBP-reporter. Aggiungi 0,10 Unità / ml di purine nucleoside fosforilasi e 200 mM 7-methylguanosine ai campioni, che serve ad assorbire qualsiasi fosfato contaminanti. Incubare i campioni in ghiaccio per almeno 20 minuti (Tabella 2).
2. Preparazione dello strumento per la MSH2-MSH6 cinetica ATPasi
- Preparare la stopped-flow strumento per esperimenti come descritto in precedenza. Inoltre, assorbire contaminanti fosfato dalla siringhe disco con 0,5 Unità / ml purina nucleoside fosforilasi e 200 mM 7-methylguanosine per 20 minuti. Impostare la lunghezza d'onda di eccitazione a 425 nm e 450 nm utilizzare un cut-off del filtro con la PMT per il rilevamento di MDCC-PBP fluorescenza.
3. MSH2-MSH6 ATPasi esperimento e analisi dei dati
- Caricare il disco siringhe con i campioni come descritto in precedenza. Fare clic su Raccogliere dati di mescolare MSH2-MSH6 con ATP e MDCC-PBP e rilasciare fosfato monitor MSH2-MSH6 e l'aumento accoppiati in MDCC-PBP fluorescenza nel tempo. Raccogliere almeno 5 tracce cinetica per ottenere un set di alta qualità dei dati e salvare il file. Media tracce multiple cinetica ed esportare i file di dati per l'analisi.
- Preparare una curva di calibrazione per determinare la relazione lineare tra concentrazione di fosfati e MDCC-PBP fluorescenza. Per farlo, mescolare MDCC-PBP con concentrazioni di fosfati differenti nelle stesse condizioni sperimentali in stopped-flow, e la misura massima MDCC-PBP fluorescenza per ogni concentrazione di fosfati.
- Tracciare la massima MDCC-PBP fluorescenza rispetto ai singoli concentrazione di fosfati per la curva di calibrazione (Fig. 7) e utilizzare la pista per determinare la concentrazione di fosfati nel MSH2-MSH6 reazioni. Plot concentrazione di fosfati in funzione del tempo e si adattano ai dati a una funzione esponenziale e lineare. Questa funzione descrive una raffica di idrolisi dello stato di pre-steady ATP e rilasciare fosfato seguita dalla fase lineare allo steady-state della reazione.
4. Risultati rappresentativi per MSH2-MSH6 cinetica ATPasi
I dati cinetici mostrano che MSH2-MSH6 idrolizza ATP e fosfato rilascia rapidamente a 1.4 -1 secondo al giro d'affari prima catalitico. Questa fase è seguita da un passo lento nella reazione che limita fatturati successivamente alla di 7 volte gatto più lento costante k stato di 0,2 secondi -1 (Fig. 8A). Tuttavia, quando MSH2-MSH6 è legato al DNA non corrispondenti, lo scoppio di idrolisi dell'ATP e rilasciare fosfato è soppresso, e MSH2-MSH6 rimane in un ATP-bound stato per un tempo più lungo.

Figura 1. Purificazione di S. cerevisiae MSH2-MSH6 da E. coli. MSH2 e MSH6 geni sono stati clonati in pET11a vettoriali e sovra-espresso in E. coli BL21 (DE3) cellule. Il complesso proteina è stata purificata mediante cromatografia su colonna SP-sefarosio, eparina, e Q-sefarosio resine. SDS-PAGE analisi qui contiene frazioni proteiche da un Q-sefarosio colonna.
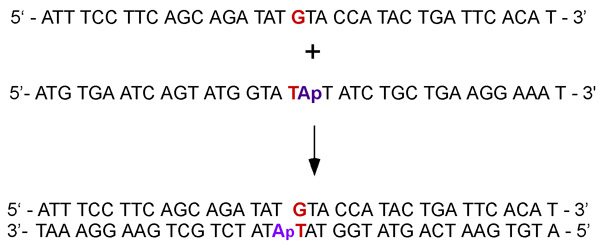
. Figura 2 complementari singolo filamento di DNA sono ricotto a formare un duplex contenente G: mancata corrispondenza T con adenosina adiacente (per esperimenti ATPasi) o 2-Aminopurine analogico di base fluorescente di adenosina (per esperimenti di legame al DNA).
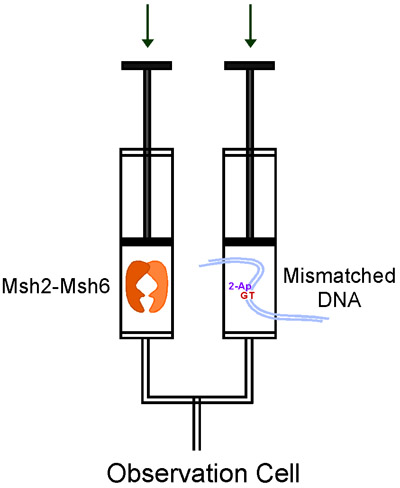
Figura 3. Flusso dei reagenti nel KinTek stopped-flow nel corso di un singolo esperimento di miscelazione.

Figura 4a Cinetica di MSH2-MSH6 interazione con una G:. Mancata corrispondenza T. Miscelazione di DNA duplex (0,12 mM) contenente una G: adiacente al 2-T con Aminopurine MSH2-MSH6 (0,8 mM) porta ad aumento della fluorescenza nel tempo, e produce un tasso costante k bimolecolari ON = 2.4 10 7 M -1 s -1 per l'interazione.
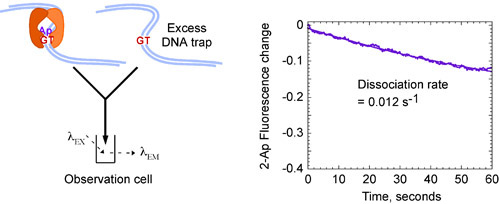
Figura 4b Cinetica di MSH2-MSH6 interazione con una G:. Mancata corrispondenza T. Miscelazione pre-formate MSH2-MSH6 G: T (2-Ap) complesso con eccesso senza etichetta G: T DNA (8 mM) che intrappola qualsiasi libero MSH2-MSH6 porta alla diminuzione della fluorescenza nel tempo, e produce un lento ritmo costante di dissociazione, k OFF = 0,012 s -1, che indica un complesso stabile con una lunga emivita di circa 60 secondi. Concentrazioni finali: 0,4 mM MSH2-MSH6, 0,06 mM DNA marcato, 4 mM senza etichetta G: trappola del DNA T.
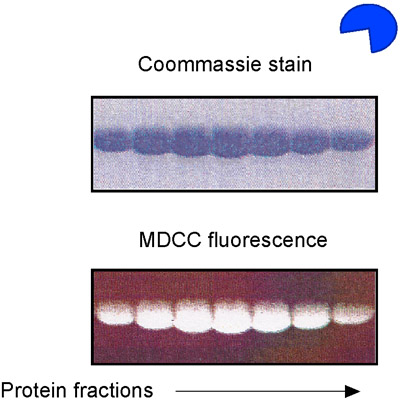
Figura 5. MDCC-PBP preparazione. SDS-PAGE analisi di E. fosfato proteine coli vincolanti (PBP), purificato e marcato con il fluoroforo MDCC.
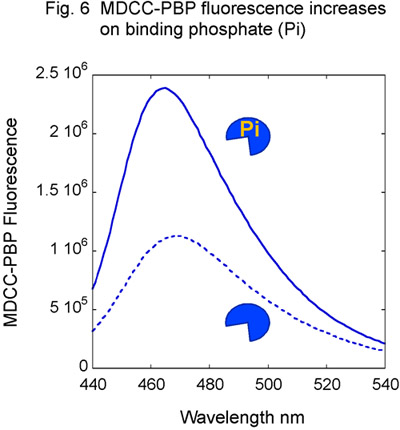
Figura 6. MDCC-PBP risposta al fosfato. Titolazione di MDCC-PBP con fosfato (Pi) si traduce in aumento di fluorescenza MDCC.

Figura 7a. Preparazione del fosfato (Pi) curva standard. MDCC-PBP (20 mM) viene mescolato con quantità variabili di Pi (0 - 8 mM) und fluorescenza misurati nel tempo fino a quando l'equilibrio è raggiunto. Concentrazioni finali: 10 mM MDCC-PBP, 0-4 mM Pi.
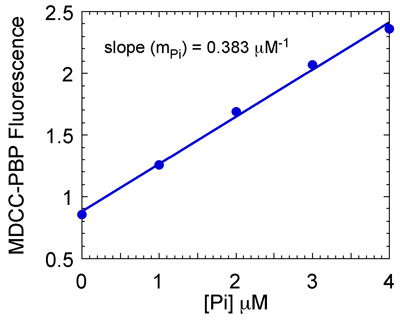
Figura 7b. Preparazione del fosfato (Pi) curva standard. Massima MDCC-PBP fluorescenza riportato in funzione [Pi] per ottenere una curva standard. La pendenza della linea (0,383 mM -1 in questo caso) viene utilizzata per convertire MDCC-PBP fluorescenza in concentrazione Pi.
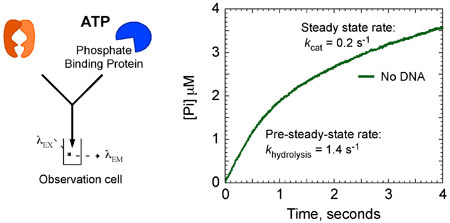
Figura 8 bis. MSH2-MSH6 ATPasi cinetica. MSH2-MSH6 (4 mM), in assenza G: DNA T, mescolato rapidamente con ATP (1 mm) e MDCC-PBP (20 mM) presenta una raffica di idrolisi dell'ATP e rilasciare Pi. L'analisi dei dati raccolti il tasso (idrolisi k = 1,4 s -1) e ampiezza (2 mM, 1 sito per MSH6-MSH2) della fase di scoppio, che è seguita da una lineare, fase di stato stazionario ad un tasso del 0,4 s mM - 1 (k cat = 0,2 s -1).
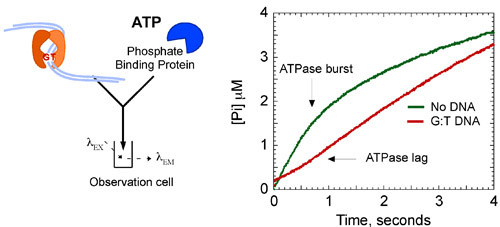
Figura 8b. MSH2-MSH6 cinetica ATPasi. L'aggiunta di G: DNA T per la reazione (6 mM) sopprime la raffica di idrolisi dell'ATP, la stabilizzazione del complesso in un ATP-bound stato. Concentrazioni finali: 2 mM MSH2-MSH6, 500 mM ATP, 3 DNA mM, 10 mM MDCC-PBP. Cinetica scoppio sono idonei dalla seguente equazione: [Pi] = A 0 e-t + k Vt, dove [Pi] è la concentrazione di fosfati, A 0 è l'ampiezza scoppio, k è il tasso di scoppio osservato costante e V è la velocità di la fase lineare (k cat = V / [MSH2-MSH6]).
| Campione | Proteina | DNA | ||||
| Reagente | Stock | Lavoro | Vol, microlitri | Stock | Lavoro | Vol, microlitri |
| MSH2-MSH6 | 5 micron | 0,8 micron | 64 | - | - | - |
| 2ApG: T | - | - | - | 10 micron | 0,12 mM | 4,8 |
| buffer | 10x | 1x | 40 | 10x | 1x | 40 |
| DDH 2 O | - | - | 296 | - | - | 355 |
| Totale | 400 | 400 | ||||
Tabella 1 di reazione al legame del DNA
| Campione | Proteina | Proteina-DNA | ATP | ||||||
| Reagente | Stock | Lavoro | Vol, microlitri | Stock | Lavoro | Vol, microlitri | Stock | Lavoro | Vol, microlitri |
| MSH2-MSH6 | 20 micron | 4 mM | 80 | 20 micron | 4 mM | 80 | - | - | - |
| G: T | - | - | - | 100 | 6 | 24 | - | - | - |
| ATP | - | - | - | - | - | - | 50 mM | 1 mM | 8 |
| 7-MEG | 250x | 1x | 1,6 | 250x | 1x | 1,6 | 250x | 1x | 1,6 |
| PNPase | 100x | 1x | 4 | 100x | 1x | 4 | 100x | 1x | 4 |
| PBP-MDCC | 150 micron | 20 micron | 53,3 | 150 micron | 20 micron | 53,3 | - | - | - |
| Buffer | 10x | 1x | 40 | 10x | 1x | 40 | 10x | 1x | 40 |
| DDH 2 O | - | - | 221 | - | - | 197 | - | - | 346 |
| Totale | 400 | 400 | 400 | ||||||
Tabella 2 ATPasi reazione
Discussione
L'esempio di una proteina di legame al DNA mancata corrispondenza qui descritto illustra la potenza e l'utilità di transitori metodi cinetici per lo studio dei meccanismi di molecole biologiche. Stopped-flow misura sulla scala singolo momento fatturato fornito prove inequivocabili per il legame rapido e specifico di proteine MSH2-MSH6 ad una coppia di basi non corrispondenti e la formazione di una lunga durata complesso proteina X del DNA nella reazione 9. Inoltre, stopped-flow (e dissetare-flo...
Riconoscimenti
Questo lavoro è stato sostenuto da un premio CARRIERA NSF (MMH), una borsa di studio Barry M. Goldwater (FNB) e un premio ASBMB Undergraduate Research (CWD). Il clone di sovra-espressione di PBP è stato gentilmente fornito dal Dr. Martin Webb (MRC, UK).
Materiali
| DNA nome | Sequenza |
| 37 g | 5'-ATT TCC TTC AGC AGA TAT G TA CCA TAC TGA TTC ACA T -3 ' |
| 37 t (2-Ap) | 5'-ATG TGA ATC AGT ATG GTA T Ap T ATC TGC TGA AGG AAA T -3 ' |
| 37 T | 5'-ATG TGA ATC AGT ATG GTA T A T ATC TGC TGA AGG AAA -3 T ' |
Riferimenti
- Johnson, K. A. Advances in transient-state kinetics. Curr Opin Biotechnol. 9 (1), 87-89 (1998).
- Johnson, K. A. E. . Kinetic analysis of macromolecules. , (2003).
- Obmolova, G., Ban, C., Hsieh, P., Yang, W. Crystal structures of mismatch repair protein MutS and its complex with a substrate DNA. Nature. 407 (6805), 703-710 (2000).
- Lamers, M. H. The crystal structure of DNA mismatch repair protein MutS binding to a G x T mismatch. Nature. 407 (6805), 711-717 (2000).
- Warren, J. J. Structure of the human MutSalpha DNA lesion recognition complex. Mol Cell. 26 (4), 579-592 (2007).
- Kunkel, T. A. &. a. m. p. ;. a. m. p., Erie, D. A. . DNA Mismatch Repair. Annu Rev Biochem. 74, 681-710 (2005).
- Jiricny, J. The multifaceted mismatch-repair system. Nat Rev Mol Cell Biol. 7 (5), 335-346 (2006).
- Hsieh, P., Yamane, K. DNA mismatch repair: Molecular mechanism, cancer, and ageing. Mech Ageing Dev. 129 (7-8), 391-407 (2008).
- Jacobs-Palmer, E., Hingorani, M. M. The effects of nucleotides on MutS-DNA binding kinetics clarify the role of MutS ATPase activity in mismatch repair. J Mol Biol. 366 (4), 1087-1098 (2007).
- Antony, E., Khubchandani, S., Chen, S., Hingorani, M. M., M, M. Contribution of Msh2 and Msh6 subunits to the asymmetric ATPase and DNA mismatch binding activities of Saccharomyces cerevisiae Msh2-Msh6 mismatch repair protein. DNA Repair (Amst). 5 (2), 153-162 (2006).
- Antony, E., Hingorani, M. M. Mismatch recognition-coupled stabilization of Msh2-Msh6 in an ATP-bound state at the initiation of DNA repair. Biochemistry. 42 (25), 7682-7693 (2003).
- Finkelstein, J., Antony, E., Hingorani, M. M., O'Donnell, M. Overproduction and analysis of eukaryotic multiprotein complexes in Escherichia coli using a dual-vector strategy. Anal Biochem. 319 (1), 78-87 (2003).
- Zhai, J., Hingorani, M. M. S. cerevisiae Msh2-Msh6 DNA binding kinetics reveal a mechanism of targeting sites for DNA mismatch repair. Proc Natl Acad Sci U S A. 107 (2), 680-685 (2010).
- Brune, M., Hunter, J. L., Corrie, J. E., Webb, M. R. Direct, real-time measurement of rapid inorganic phosphate release using a novel fluorescent probe and its application to actomyosin subfragment 1 ATPase. Biochemistry. 33 (27), 8262-8271 (1994).
- Brune, M. Mechanism of inorganic phosphate interaction with phosphate binding protein from Escherichia coli. Biochemistry. 37 (29), 10370-10380 (1998).
- Antony, E., Hingorani, M. M. Asymmetric ATP binding and hydrolysis activity of the Thermus aquaticus MutS dimer is key to modulation of its interactions with mismatched DNA. Biochemistry. 43 (41), 13115-13128 (2004).
- Gradia, S., Acharya, S., Fishel, R. The human mismatch recognition complex hMSH2-hMSH6 functions as a novel molecular switch. Cell. 91 (7), 995-1005 (1997).
- Mazur, D. J., Mendillo, M. L., Kolodner, R. D., D, R. Inhibition of Msh6 ATPase activity by mispaired DNA induces a Msh2(ATP)-Msh6(ATP) state capable of hydrolysis-independent movement along DNA. Mol Cell. 22 (1), 39-49 (2006).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon