Method Article
Solo lectura y pareadas final Sec ARNm bibliotecas de Illumina de 10 nanogramos ARN total
En este artículo
Resumen
Aquí se describe un método para la preparación de los dos pares de solo lectura y final Illumina Sec-mRNA bibliotecas genómicas para el análisis de expresión genética basado en la amplificación del ARN T7 lineal. Este protocolo requiere solamente 10 nanogramos de ARN total a partir bibliotecas y genera muy consistente que representa transcripciones conjunto.
Resumen
La secuenciación del transcriptoma todo por ARNm-Seq ahora se utiliza ampliamente para llevar a cabo la expresión génica global, la mutación, la expresión específica de alelo y otros análisis en todo el genoma. ARNm-Seq, incluso abre la puerta para el análisis de expresión génica de no secuenciado los genomas. ARNm-Seq ofrece una alta sensibilidad, un rango dinámico amplio y permite la medición del número de copias de transcripción en una muestra. Analizador de Illumina genoma lleva a cabo la secuenciación de un gran número (> 10 7) de la secuencia relativamente corto se lee (<150 pb). El "fin de pares", enfoque, en el que una lectura única y larga es la secuencia en sus dos extremos, permite el seguimiento de los cruces alternativos de empalme , inserciones y eliminaciones, y es útil para de novo de montaje transcriptoma.
Uno de los principales desafíos que enfrentan los investigadores es una cantidad limitada de material de partida. Por ejemplo, en experimentos donde se cosechan las células de micro-disección con láser, disponible a partir del ARN total de may medida en nanogramos. Preparación de las bibliotecas de mRNA-Sec de las muestras se han descrito 1, 2, sino que implica importantes amplificación por PCR que puedan introducir sesgos. Otros ARN-Seq procedimientos de construcción de la biblioteca con un mínimo de amplificación de PCR se han publicado 3, 4, sino que requieren cantidades de microgramos a partir de ARN total.
A continuación se describe un protocolo para la plataforma Illumina Genoma Analyzer II para la secuenciación de ARNm-Seq para la preparación de la biblioteca, que evita importantes amplificación por PCR y sólo requiere de 10 nanogramos de ARN total. Aunque este protocolo ha sido descrito previamente y validado para un solo fin de la secuencia 5, donde se ha demostrado para producir bibliotecas de dirección sin la introducción de un sesgo de amplificación importantes, aquí lo validamos para su utilización como un protocolo de final emparejados. Nos selectivamente amplificar ARN mensajero de partida poliadenilado ARN total utilizando el método basado en Eberwine T7 amplificación lineal, acuñado "T7LA "(T7 amplificación lineal). La amplificación de poli-A ARNm están fragmentados, transcripción reversa y el adaptador de liga para producir la biblioteca de la secuencia final. Para ambas carreras finales individuales de lectura y pares, las secuencias se asignan a los humanos transcriptoma 6 y normalizado para que los datos de varias ejecuciones se pueden comparar. Presentamos la medida de expresión génica en las unidades de las transcripciones por millón (TPM), que es una medida superior a RPKM al comparar muestras 7.
Protocolo
1. Aislar el ARN total
- Añadir 1 ml de Trizol de células / tejidos y homogeneizar con la aguja de gasa 18-22 si es necesario.
- Añadir 200 l de cloroformo, y girar a 14.000 rpm durante 30 minutos a 4 ° C.
- Saque la capa acuosa superior, añadir 0,5 l de acrilamida lineal, a continuación, añadir 500 l de isopropanol. Deje reposar a temperatura ambiente durante 20 minutos.
- Girar a 14.000 rpm a 4 ° C de pellets, lavar con etanol al 70%, y el vacío seco.
- Resuspender en 1 l de agua libre de nucleasa y la transferencia de hasta 200 l tubo de PCR.
2. Preparar cDNA de doble cadena
- A lo anterior agrega un ARN l 1 l de 100 mM de oligo-dT-T7 manual de transcripción inversa (5'-GGCCAGTGAATTGTAATACGACTCACTATAGGGAGGCGGTTTTTTTTTTTTTTTTTTTTTTTT -3 '), el calor a 70 ° C (del bloque de calor) durante 5 min, y presión enfriar en hielo.
- Añadir un buffer l FS 5x, 0.5 TDT l, 0,5 l de mezcla de dNTP y 0,5 RnaseOUT l (a partir de superíndice II kit).
- Calor a 42° C en la máquina de PCR durante 1 minuto, a continuación, añadir 0,5 l Superíndice la transcriptasa inversa II. Incubar durante 1 hora a 42 ° C.
- Calor a 70 ° C durante 10 minutos, enfriar a 4 ° C.
- En el hielo, añadir lo siguiente a la reacción anterior:
- Nucleasa libre de agua - 22,75 l
- Buffer 5X segundo capítulo - 7,5 l
- Mezcla de dNTP - 0,75 l
- E. coli DNA ligase - 0,25 l
- E. coli DNA polimerasa - 1 l
- RNaseH - 0,25 l
Se incuba durante 2 horas a 16 ° C y luego agregar 1 l de polimerasa de ADN T4, y se incuba durante 10 minutos más a 16 ° C.
- Transferencia de hasta 1,5 ml tubo Eppendorf, y añadir 80 l de agua.
- Añadir 0,5 l de acrilamida lineal.
- Añadir 72 l 3 M NH4OAc y 480 l de etanol frío al 100%. Precipitado durante 1 hora a -20 ° C.
- Girar a 14.000 rpm a 4 ° C durante 30 minutos, lavar con 1 ml de etanol al 70%, se centrifuga a 14.000 rpm durante 2minutos, y el vacío en seco durante unos 10 minutos.
3. Amplificar poli-A ARNm por la transcripción in vitro
- Resuspender por encima de cDNA de 3,5 l de agua libre de nucleasa.
- A lo anterior, añadir 1 l cada uno de los dNTPs (un total de 4 l), 1 l de tampón de reacción 10X y 1 l de polimerasa T7, y un 0,5 l de RnaseOUT de kit Megascript.
- Se incuba a 37 ° C en la máquina de PCR durante la noche.
- Listo para el siguiente paso. Pueden ser almacenadas a -80 ° C.
4. Fragmentación de la amplificación de poli-A ARNm
- Añadir 26 l de agua a la reacción anterior y 4 l de reactivo de fragmentación de 10x.
- Calor en la máquina de PCR a 70 ° C durante exactamente 7 minutos.
- Añadir 5 l de tampón fragmentación detener, poner la muestra en hielo.
5. ARN de limpieza
- Para la reacción anterior, se añaden 60 l de agua y 350 l de tampón RLT de Rneasy kit MinElute, y mezclar con la pipeta.
- Añadir 250 l de etanol, mezclar con la pipeta y pipeta en columna de centrifugación.
- Giran a 8000 rcf durante 20 segundos.
- Lavar una vez con el EPR, girar durante 20 segundos, lavar por segunda vez con el 80% de EtOH, girar durante 2 minutos, y columna seca con 5 spin minutos.
- Eluyen ARN en 10 l de agua libre de nucleasa.
6. La síntesis de ADNc
La síntesis de la primera cadena de librería única lectura:
- En un tubo de PCR, añadir lo siguiente:
- La fragmentación de poli-A ARNm - 10 l
- Noti azar Primer nonamer - 1 l
Se incuba a 70 ° C durante 5 minutos en el termociclador, y enfriar rápidamente en hielo. Noti nonamer Primer (5'-TGAATTCGCGGCCGCTCAAGCAGAAGACGGCATACGAGCTCTTCCGATCT NNNNNNNNN -3). La secuencia 5 'proximal es el sitio de restricción NotI mientras que la secuencia siguiente hasta que la región al azar es el complemento inverso de secuencia de Illumina, adaptador B de Chip-Seq kit.
- 5X primer capítulo l buffer -4
- TDT - 2 l
- * La mezcla de dNTP - 1,5 l
Puesto en el termociclador durante 2 minutos a 42 ° C, añadir 1 l de superíndice III transcriptasa inversa, y se incuba a 42 ° C durante 1 hora.
* En lugar de dCTP, 5-metil dCTP fue utilizado en la mezcla de dNTP.
La síntesis de la primera cadena de la biblioteca final emparejados:
- En un tubo de PCR, añadir lo siguiente:
- La fragmentación de poli-A ARNm - 10 l
- Imprimación azar hexamer - 1 l
Se incuba a 65 ° C durante 5 minutos en el termociclador, y enfriar rápidamente en hielo.
- A lo anterior, agregue el siguiente en el hielo (de superíndice firststrand III kit):
- Buffer 5X primer capítulo - 4 l
- TDT - 2 l
- dNTPs (dekit) - 1,5 l
Puesto en el termociclador durante 1 min a 45 ° C, añadir 1 l de superíndice III transcriptasa inversa, y se incuba a 45 ° C durante 1 hora.
Síntesis de segunda línea (para bibliotecas):
- A lo anterior, agregue el siguiente en el hielo (de superíndice II kit):
- Agua (RNasa libre) - 91 l
- Buffer 5X segunda línea - 30 l
- dNTPs (de juego) - 3 l
- E. coli DNA ligase - 1 l
- E. coli DNA polimerasa - 4 l
- E. coli RNasa H - 1 l
Mezclar por inversión del tubo, dar una vuelta corta y se incuba a 16 ° C durante 2 horas.
7. Purificar cDNA
- Purificar la muestra de cDNA con columnas Zymo, eluir en 40 l de agua para lectura individuales y 30 l de agua para la biblioteca de final emparejados.
8. Y reparaciones
Para la biblioteca de una sola lectura:
(Use Illumina Chip-Sec muestra prep kit)
- A lo anterior, 40 l de ADNc, añade:
- T4 ADN ligasa con buffer 10 mM ATP - 5 l
- Mezcla de dNTP - 2 l
- ADN polimerasa T4 - 1 l
- Klenow enzima (diluido 1:5 con agua para 1U / l) - 1 l
- T4 PNK - 1 l
Incubar en el termociclador durante 30 minutos a 20 ° C.
Para la biblioteca de final emparejados:
(Use Illumina muestras pareadas final de preparación del kit)
- A lo anterior, 30 l de ADNc, añade:
- RNasa libre de DNasa agua - 45 l
- T4 ADN ligasa con buffer 10 mM ATP - 10 l
- 10 mM dNTP mix - 4 l
- ADN polimerasa T4 - 5 l
- Klenow enzima - 1 l
- T4 PNK - 5 l
Incubar en el termociclador durante 30 minutos a 20 ° C.
9. limpieza cDNA
- C magra hasta cDNA utilizando columnas Zymo. Eluyen en 34 l de EB para leer individuales y 32 l de EB para la biblioteca de final emparejados.
10. Añadir 'A' bases para el extremo 3 'de los fragmentos de ADN
Para la biblioteca de una sola lectura:
- Prepare la mezcla de reacción siguiente:
- Muestra de ADN - 34 l
- Klenow buffer - 5 l
- dATP - 10 l
- Klenow exo - 1 l
Incubar durante 30 minutos a 37 ° C.
Para la biblioteca de final emparejados:
- Prepare la mezcla de reacción siguiente:
- Muestra de ADN - 32 l
- Klenow buffer - 5 l
- dATP - 10 l
- Klenow exo - 3 l
Incubar durante 30 minutos a 37 ° C.
11. limpieza cDNA
- Limpieza de cDNA utilizando columnas Zymo. Eluyen en 10 l de EB.
12. Adaptador de ligadura
jove_step "> Para leer sola biblioteca:(Use Illumina Chip-Sec muestra prep kit)
- Prepare la mezcla de reacción siguiente:
- Muestra de ADN - 10 l
- Adaptador Oligo Mix - 1 l
- 2X DNA ligase Buffer-l -15
- ADN ligasa - 4 l
Incubar durante 15 minutos a temperatura ambiente. Adaptadores se deben descongelar en hielo y 1:20 diluida.
Para la biblioteca de final emparejados:
(Use Illumina muestras pareadas final de preparación del kit)
- Prepare la mezcla de reacción siguiente:
- Muestra de ADN - 10 l
- PE Adaptador Oligo Mix - 10 l
- 2X DNA ligase Buffer - 25 l
- ADN ligasa - 5 l
Incubar durante 15 minutos a 20 ° C. Adaptadores se deben descongelar en hielo y 1:20 diluida.
13. Reacción de la ligadura de limpieza
- Limpieza de la reacción de ligación con zymo columnas. Eluyen en 44 l de agua para la biblioteca de una sola lectura y 6 l de EB seguido por otro de elución en 5 l de EB para la biblioteca de final emparejados.
Realizar una digestión NotI, SOLO para una sola biblioteca de lectura
- cDNA - 44 l
- ORC buffer 3 a 5 l
- BSA - 0,5 l
- Noti - 1 l
Incubar durante 2 horas a la noche a 37 ° C, y la reacción de purificación con columna Zymo. Eluir con 6 l de tampón EB seguido de una segunda elución con 5 l de EB.
14. Tamaño de la selección / Gel de purificación
Realice el siguiente con un 2% seguro Sybr E-Gel de Invitrogen.
- Ejecutar el Programa 0-PreRun 2 minutos.
- Carga 1kb plus escalera de Invitrogen diluido 1:4, y la carga de 10 microlitros.
- Carga todos los de la muestra de ADN, y llenar los carriles vacíos con 10 l de agua.
- Ejecución del programa 1-Egel 2% de ejecución de 28 minutos.
- Seleccione el tamaño 200-300 pb gel SLICe con una cuchilla de afeitar nueva.
15. Eluir el ADN de rebanada de gel
- Pesar rebanada de gel y agregar 3 volúmenes de tampón QG a un volumen de gel (gel de utilizar el kit de extracción de Qiagen)
- Se incuba a 50 ° C durante 10 minutos o hasta que se corte gel se disuelve.
- Añadir 1 volumen de isopropanol y mezclar por inversión o pipeta.
- Añadir a la columna, rotación durante 1 minuto a máxima velocidad, y descartar el filtrado.
- Añadir 500 l de buffer QG a la columna, rotación durante 1 minuto a máxima velocidad, y desechar el flujo a través de.
- Añadir 750 l de Buffer PE a la columna, rotación durante 1 minuto a máxima velocidad, y desechar el flujo a través de.
- Centrifugar durante 1 minuto a máxima velocidad.
- Eluyen en 36 l de EB para leer individuales y 23 EB l para la biblioteca de final emparejados.
16. PCR
Para la biblioteca de una sola lectura:
(Use Illumina Chip-Sec muestra prep kit)
- Preparar el following mezcla de reacción de PCR:
- ADN - 36 l
- Buffer Phusion 5X - 10 l
- Mezcla de dNTP - 1,5 l
- PCR primer 1,1 a 1 l
- PCR primer 2,1 a 1 l
- Polimerasa Phusion - 0,5 l
Utilice el siguiente protocolo de PCR:
- 30 segundos a 98 ° C
- 10 ciclos de:
- 10 segundos a 98 ° C
- 30 segundos a 65 ° C
- 30 segundos a 72 ° C
- 5 minutos a 72 ° C
- Mantener a 4 ° C
* La primera vez que se usa el kit, diluir PCR primers 1:02 con tampón EB.
Para la biblioteca de final emparejados:
(Use Illumina muestras pareadas final de preparación del kit)
- Prepare la siguiente reacción de PCR:
- ADN - 23 l
- Phusion ADN polimerasa - 25 l
- PCR primer PE 1,1 a 1 l
- PCR primer PE 2.1 -1 l
Amplificar mediante el siguiente protocolo de PCR:
- 30 segundos a 98 ° C
- 10 ciclos de:
- 10 segundos a 98 ° C
- 30 segundos a 65 ° C
- 30 segundos a 72 ° C
- 5 minutos a 72 ° C
- Mantener a 4 ° C
* La primera vez que se usa el kit, diluir PCR primers 1:02 con tampón EB.
17. Biblioteca de la limpieza
- La limpieza de la biblioteca utilizando columnas Zymo. Eluyen en 12 l de EB
18. Cuantificar la biblioteca
- Cuantificar el uso de la biblioteca Qubit. Está listo para la secuenciación. Utilice la secuencia de primers Illumina solo kit leer la generación de cluster V4 o superior para la única biblioteca de lectura y Illumina grupo emparejado final kit de generación V4 o superior para la biblioteca de final emparejados.
19. Análisis de datos
Bowtie 6 se utilizó para mAP dice que el conjunto de genes RefSeq (NCBI Build 36.1). Al final solo lee (30 nucleótidos) y al final se lee par (42 nucleótidos) fueron mapeados que permite hasta 10 partidos con el conjunto de genes, y que permite hasta dos desajustes por lectura. Transcripciones por millón (TPM) se obtuvieron valores para medir la expresión génica utilizando RSEM 7 (ARN-Seq por la expectativa-maximización).
20. Los resultados representativos: Hicimos bibliotecas T7LA tanto para un solo extremo de lectura y pares va de 1 g, g 100, g 10, g 1 y 100 pg a partir de ARN total (Figura 1). Para la evaluación de nuestro protocolo, se hizo un solo de lectura y bibliotecas pares final sin T7 amplificación del ARN a partir de 10 mg de ARN total de estas bibliotecas de control, denominado "MinAmp", han amplificación mínima. La ampliación sólo se someten son los 10 ciclos de PCR hacia el final del protocolo para ligar los adaptadores de Illumina secuencia, un paso común a todas las bibliotecas. Todos utilizan el ARN se aislaron de H14células madre embrionarias humanas 8.
En primer lugar, evaluar el número de genes identificados por las diversas bibliotecas (Tabla 1 y Tabla de apoyo 1). Tanto para las bibliotecas final solo leer y parejas, el 10 ng bibliotecas T7LA identificado casi el mismo número de genes, como las bibliotecas MinAmp 10 mg, con una TPM de 10 o más. En el caso de las bibliotecas de una sola lectura, el 10 ng T7LA biblioteca identificó el 100% de los 8500 genes identificados por la biblioteca de 10 mg no amplificada. Para las bibliotecas de final emparejados, el 10 ng T7LA biblioteca identificó 86% de los genes identificados por la biblioteca de 10 mg sin amplificar (7.961 de 9.267 genes). Bibliotecas a partir de menos de 10 ng no fueron capaces de identificar como muchos genes. Por ejemplo, en el único protocolo de lectura, la biblioteca de 1 ng identifica sólo ~ 50% de los genes identificados por la biblioteca mg 10 MinAmp, lo que llevó a limitar la cantidad más baja del total de ARN para su uso con el protocolo T7LA a 10 ng. Por otra parte, la asignación de un gen de limpieza, GAPDH (Figura 2) muestra que todas las bibliotecas T7LA hecho con por lo menos a partir de ARN 10ng identificados todos los exones, incluyendo el extremo 5 'del exón. La comparación de los 10 ng T7LA único fin bibliotecas de lectura y se combina con las bibliotecas MinAmp muestra un alto grado de similitud (correlación de Spearman, r = 0,90 y 0,95, respectivamente, las figuras 3a y b). También se compararon los dos una sola lectura y bibliotecas pares final a partir de 10 ng de ARN total y tuvieron un coeficiente de correlación muy alta (R = 0,92), demostrando que ambos tipos de bibliotecas realiza mediante el protocolo T7LA producir una firma de expresión de genes muy similares ( Figura 3c.). Por lo tanto, el método T7LA es capaz de producir las bibliotecas de secuenciación que son tan confiables y completas como las bibliotecas MinAmp, pero a partir de 1000 veces menos material de partida.

Figura 1 Esquema de final emparejados y único protocolo de lectura de la biblioteca de preparación.

Figura 2 Una imagen del navegador del genoma de un gen de limpieza, GAPDH, para todas las bibliotecas final solo leer y pareadas. La barra de escala a la izquierda para las bibliotecas solo lectura indica 350 Total de Lecturas. La barra de escala en el centro para las bibliotecas de final emparejados indica total de 5.000 lecturas. El eje horizontal representa la secuencia del genoma de GAPDH.

Figura 3 Correlación de las expresiones de genes entre las diferentes bibliotecas (Spearman). A. Entre única lectura 10 ng T7LA y 10 mg de la biblioteca MinAmp muestra que estas dos bibliotecas tienen un patrón de expresión genética muy similar (R = 0,90) B. Entre el final pares 10 ng T7LA y 10 mg de la biblioteca MinAmp demuestra la similitud de los perfiles de expresión génica (R = 0,95). Correlación C del gen de la exsiones entre 10 ng final pareadas y 10 ng de librerías leer muestran un alto grado de similitud entre estas bibliotecas preparado por el método T7LA (R = 0,92).
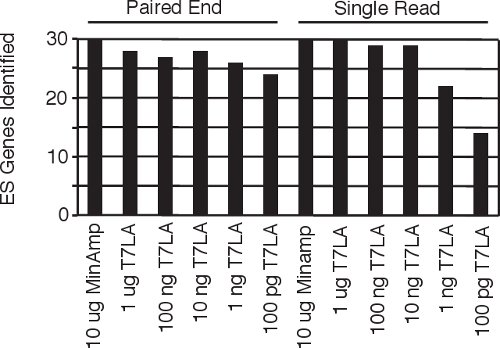
Figura 4 Identificación de los derechos humanos genes5 con células madre embrionarias específicas de todas las bibliotecas de un solo extremo de lectura y pareadas.
| Biblioteca | ° Tipo | Clusters primas | Clusters% al pasar de filtro | Alineación% del genoma | % La tasa de error | Los genes identificados |
| MinAmp 10ug | Lectura única | 225602 + / - 4952 | 65,48 + / - 2,58 | 47,61 + / - 0,53 | 0,62 + / - 0,06 | 8500 |
| 1 ug T7LA | Lectura única | 144818 + / - 6513 | 82,21 + / - 6,45 | 48,09 + / - 0,27 | 0,42 + / - 0,03 | 8757 |
| 100 ng T7LA | Lectura única | 27.385 + / - 1818 | 81,33 + / - 11.75 | 44,46 + / - 4,53 | 0,49 + / - 0,10 | 8709 |
| 10ng T7LA | Lectura única | 11.184 + / - 985 | 60,70 + / - 3,70 | 14,96 + / - 1,15 | 0,99 + / - 0,30 | 8589 |
| 1ng T7LA | Lectura única | 12.695 + / - 1365 | 53,27 + / - 16.76 | 4,08 + / - 0,79 | 2,25 + / - 1,56 | 4720 |
| 100PG T7LA | Lectura única | 10.390 + / - 1398 | 72,99 + / - 2,90 | 1,48 + / - 0,20 | 1,51 + / - 0,39 | 1121 |
| MinAmp 10ug | Fin pares R1 | 95.786 + / - 12.937 | 90,77 + / - 2,79 | 58,50 + / - 0,95 | 0,94 + / - 0,38 | 9267 |
| Fin pares R2 | 95.786 + / - 12.937 | 90,77 + / - 2,79 | 58,13 + / - 1,13 | 0,99 + / - 0,37 | ||
| 1 ug T7LA | Fin pares R1 | 297669 + / - 10.196 | 91,35 + / - 0,36 | 46,89 + / - 0,14 | 0,47 + / - 0,01 | 7334 |
| Fin pares R2 | 297669 + / - 10.196 | 91,35 + / - 0,36 | 45,52 + / - 0,12 | 0,51 + / - 0,01 | ||
| 100 ng T7LA | Fin pares R1 | 205602 + / - 9932 | 90,53 + / - 0,76 | 63,44 + / - 1,00 | 0,48 + / - 0,02 | 8011 |
| Fin pares R2 | 205602 + / - 9932 | 90,53 + / - 0,76 | 61,80 + / - 8,09 | 0.60+ / - 0,36 | ||
| 10ng T7LA | Fin pares R1 | 214622 + / - 11.155 | 89,98 + / - 1,13 | 56,32 + / - 1,94 | 0,80 + / - 0,26 | 7961 |
| Fin pares R2 | 214622 + / - 11.155 | 89,98 + / - 1,13 | 46,41 + / - 18.39 | 2,48 + / - 2,68 | ; | |
| 1ng T7LA | Fin pares R1 | 144 951 + / - 19.841 | 90,54 + / - 1,19 | 3,91 + / - 0,16 | 8,71 + / - 0,86 | 8124 |
| Fin pares R2 | 144 951 + / - 19.841 | 90,54 + / - 1,19 | 3,27 + / - 1,21 | 9,11 + / - 3,52 | ||
| 100PG T7LA | Fin pares R1 | 187600 + / - 11.759 | 89,52 + / - 1,11 | 1,78 + / - 0,05 | 13,42 + / - 0,50 | 6623 |
| Fin pares R2 | 187600 + / - 11.759 | 89,52 + / - 1,11 | 1,99 + / - 0,23 | 15,29 + / - 0,96 | ||
° R1 y R2 son avance y retroceso de las secuencias de una etiqueta
* ≥ 10 TPM
Tabla 1. Información sobre el número de cluster, los genes identificados, la tasa de error, la alineación por ciento de las bibliotecas de un solo extremo de lectura y pareadas.
Que complementa el cuadro 1. Lista de todos los genes y sus valores TPM para todas las muestras, individuales y parejas leer final.
Discusión
Los protocolos actuales para la toma de pares bibliotecas final requieren entre 1 mg 9 a 2,5 mg 10 importe de partida de ARN total. A continuación les presentamos nuestra amplificación lineal T7 basada (T7LA) para preparar los dos una sola lectura y bibliotecas pares Illumina final de secuenciación y demostrar que este método permite la generación de bibliotecas de precios tan bajos como 10 ng de ARN total, la producción de datos que es comparable a la de mínimamente ampliado (MinAmp) bibliotecas de material veces más, a partir 1000 (10 mg de ARN total). Las 10 bibliotecas ng no sólo descubrir las semejanzas en el número total de genes, sino que también producen las firmas de expresión genética que son similares (Figuras 3a yb). Por otra parte, tanto la única lectura y bibliotecas pares final producido por el método T7LA son muy similares entre sí (Figura 3c), que permite a los investigadores comparar los datos generados por las bibliotecas a partir de cualquiera de los protocolos. Desde estas bibliotecas fueron preparados a partir de ARN humano de células madre embrionarias, se realizaron búsquedasde 30 genes de células madre específicas entre las bibliotecas y encontrar que casi todos los de estos genes (93-100%) se identifican por las bibliotecas a partir de al menos 10 ng de RNA total de partida (Figura 4), validando así el protocolo. Creemos que el protocolo sería muy útil para los investigadores, especialmente en circunstancias como las celdas de flujo ordenado o láser micro-disección del tejido en que el material de partida es limitante. En tales circunstancias, el protocolo que permitirá la generación de datos de expresión génica comparable a las bibliotecas a partir de cantidades mucho más grandes de partida ya que el protocolo produce perfiles de expresión comparable a través de al menos 3 órdenes de magnitud a partir de ARN.
Divulgaciones
Los autores revelan que JAT es uno de los fundadores, los accionistas, consultor y miembro del consejo de celulares Dynamics International (CDI). También se desempeña como asesor científico y tiene intereses financieros en las tácticas Empresas II de células madre.
Agradecimientos
Este trabajo fue apoyado por fondos del Instituto Morgridge para la Investigación y la Universidad de Wisconsin Foundation. Damos las gracias a Krista Eastman por su asistencia editorial.
Materiales
| Name | Company | Catalog Number | Comments |
| Empresa | Kit / Reactivo | Catálogo # | Comentarios especiales |
| Ambion | La fragmentación de reactivos | AM8740 | |
| Ambion | Revestimiento de acrilamida | AM9520 | |
| Ambion | MEGAscript T7 Kit | AM1334 | |
| Ambion | DEPC no tratados de agua libre de nucleasa | AM9932 | |
| Fermentas | dNTP conjunto | R0181 | |
| Fermentas | dNTPs metilado | R0431 | Para la preparación de la muestra individual de sólo lectura |
| Illumina | TruSeq SR Grupo Generación kit v5 | GD-203-5001 | Para la preparación de la muestra individual de sólo lectura |
| Illumina | TruSeq Sec Kit v5 36 ciclos | FC-104-5001 | |
| Illumina | Sec. de chips preparación de muestras Kit | IP-102-1001 | Para la preparación de la muestra individual de sólo lectura. Pueden ser reemplazados con los ORC muestra de ADN Mix Master Set de preparación un gato # E6040S |
| Illumina | TruSeq PE Grupo Generación kit v5 | PE-203-5001 | Para la preparación de parejas de la muestra final, sólo |
| Illumina | Muestras pareadas final Prep Kit | PE-102-1001 | Para la preparación de parejas de la muestra final, sólo |
| Invitrogen | 1kb plus escalera | 10787-018 | |
| Invitrogen | E. coli RNasa H | 18021-014 | |
| Invitrogen | RNasa fuera | 10777-019 | |
| Invitrogen | Buffer 5x segunda cadena | 10812-014 | |
| Invitrogen | E. coli DNA polimerasa | 18010-017 | |
| Invitrogen | E-gel SYBR seguro del 2% | G521802 | |
| Invitrogen | Superíndice III (5X buffer FS y TDT 0,1 M) | 18080-085 | |
| Invitrogen | Trizol | 12183555 | |
| Invitrogen | ARN quibit kit de ensayo | Q32852 | |
| Invitrogen | dsDNA SA qubit kit de ensayo | Q32851 | |
| Invitrogen | E. coli ADN ligasa | 18052-019 | |
| Invitrogen | ADN polimerasa T4 | 18005-025 | |
| Invitrogen | Ultra Pure DNasa RNasa agua | 10977-015 | |
| Invitrogen | Superíndice II doble varados cDNA síntesis kit | 11917-020 | |
| Invitrogen | Primer azar (Invitrogen) | 48190-011 | Para la preparación de parejas de la muestra final, sólo |
| IDT | Not1Nonamer B imprimación | N / A | Para un solo sam Leerejemplo de preparación sólo |
| IDT | Oligo dT T7 | N / A | |
| NEB | Not1 digestión kit | R0189S | Para la preparación de la muestra individual de sólo lectura |
| Qiagen | Rneasy MinElute kit | 74204 | |
| Qiagen | RNeasy Mini Kit (50) | 74104 | |
| Qiagen | Gel kit de purificación | 28604 | |
| Qiagen | DNasa conjunto | 79254 | |
| Qiagen | Rneasy MinElute kit | 74204 | |
| Zymo Investigación | ADN limpia y concentrador (250X) | D4014 |
Referencias
- Tang, F., Barbacioru, C., Wang, Y., Nordman, E., Lee, C., Xu, N., Wang, X., Bodeau, J., Tuch, B. B., Siddiqui, A., Lao, K., Surani, M. A. mRNA-Seq whole-transcriptome analysis of a single cell. Nature Methods. 6, 377-382 (2009).
- Armour, C. D., Castle, J. C., Chen, R., Babak, T., Loerch, P., Jackson, S., Shah, J. K., Dey, J., Rohl, C. A., Johnson, J. M., Raymond, C. K. Digital transcriptome profiling using selective hexamer priming for cDNA synthesis. Nature Methods. 6, 647-649 (2009).
- Mortazavi, A., Williams, B. A., McCue, K., Schaeffer, L., Wold, B. Mapping and quantifying mammalian transcriptomes by RNA-Seq. Nature Methods. 5, 621-628 (2008).
- Mamanova, L., Andrews, R. M., James, K. D., Sheridan, E. M., Ellis, P. D., Langford, C. F., Ost, T. W., Collins, J. E., Turner, D. J. FRT-seq amplification-free, strand-specific transcriptome sequencing. Nature Methods. 5, 130-132 (2010).
- Sengupta, S., Ruotti, V., Bolin, J., Elwell, A., Hernandez, A., Thomson, J., Stewart, R. Highly consistent, fully representative mRNA-Seq libraries from ten nanograms of total RNA. Biotechniques. 49, 898-904 (2010).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome. Biology. 10, R25-R25 (2009).
- Li, B., Ruotti, V., Stewart, R. M., Thomson, J. A., Dewey, C. N. RNA-Seq gene expression estimation with read mapping uncertainty. Biotechniques. 26, 493-500 (2010).
- Thomson, J. A., Itskovitz-Eldor, J., Shapiro, S. S., Waknitz, M. A., Swiergiel, J. J., Marshall, V. S., Jones, J. M. Embryonic stem cell lines derived from human blastocysts. Science. 282, 1145-1147 (1998).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados