Method Article
Leia única e emparelhados End mRNA-Seq Bibliotecas Illumina de 10 nanogramas RNA total
Neste Artigo
Resumo
Aqui nós descrevemos um método para a preparação de ambos única leitura e emparelhado final Illumina mRNA Seq-libraries seqüenciamento para análise de expressão gênica com base na amplificação T7 RNA linear. Este protocolo requer apenas 10 nanogramas de começar RNA total e gera bibliotecas altamente consistentes representando transcrições todo.
Resumo
Seqüenciamento do transcriptoma inteiro por mRNA Seq-se amplamente utilizado para realizar a expressão gênica global, mutação, expressão alelo-específico e outras análises genômicas. mRNA-Seq ainda abre o portão para a análise de expressão gênica de genomas seqüenciados não. mRNA Seq oferece alta sensibilidade, uma grande gama dinâmica e permite a medição do número de cópias transcrição em uma amostra. Analisador Illumina genoma realiza sequenciamento de um grande número (> 10 7) da seqüência relativamente curta lê (<150 pb). O "emparelhado fim" abordagem, em que uma leitura única e longa é seqüenciado em ambas as extremidades a sua, permite o rastreamento junções splice alternativo , inserções e deleções, e é útil para a montagem de transcriptoma novo.
Um dos grandes desafios enfrentados pelos pesquisadores é uma quantidade limitada de matéria-prima. Por exemplo, em experimentos onde as células são colhidas por laser micro-dissecção, disponível a partir de RNA total de may medida em nanogramas. Preparação de mRNA Seq-bibliotecas a partir de tais amostras foram descritas 1, 2, mas envolve a amplificação por PCR significativo que pode introduzir viés. Outros RNA-Seq procedimentos de construção da biblioteca com a amplificação PCR mínima foram publicados 3, 4, mas requerem quantidades micrograma de RNA total de partida.
Aqui nós descrevemos um protocolo para a plataforma Illumina Genome Analyzer II para mRNA Seq-seqüenciamento para a preparação de biblioteca que evita a amplificação PCR significativa e requer apenas 10 nanogramas de RNA total. Embora este protocolo foi descrito anteriormente e validado para um único fim de seqüenciamento 5, onde foi mostrado para produzir bibliotecas direcional sem introduzir viés de amplificação significativa, aqui nós validá-lo para utilização como um protocolo final emparelhados. Nós seletivamente amplificam RNA mensageiro polyadenylated de iniciar RNA total usando o T7 com base Eberwine método de amplificação linear, cunhado "T7LA "(T7 amplificação linear). Amplificada O poli-A mRNAs são fragmentados, reverso transcrito e adaptador ligado para produzir a biblioteca seqüenciamento final. Para ambos única corre final ler e emparelhado, as seqüências são mapeados para o transcriptoma humano 6 e normalizados para que dados de várias execuções podem ser comparados. Relatamos a medida de expressão gênica em unidades de transcrições por milhão (TPM), que é uma medida superior a RPKM quando se comparam amostras 7.
Protocolo
1. Isolar RNA total
- Adicionar 1 ml Trizol para as células / tecidos, e homogeneizar com agulha 18-22 gaze se necessário.
- Adicionar 200 mL de clorofórmio, e girar a 14.000 rpm por 30 minutos a 4 ° C.
- Retire camada aquosa superior, adicione 0,5 mL de acrilamida linear, em seguida, adicione 500 mL de isopropanol. Deixe descansar em temperatura ambiente por 20 minutos.
- Giram a 14.000 rpm a 4 ° C, lavar pellet com etanol 70%, e vácuo seco.
- Ressuspender em 1 mL de água nuclease livre e transferir para tubo de 200 mL PCR.
2. Prepare dupla fita de cDNA
- Ao acima de 1 RNA mL adicionar 1 ml de 100 M oligo-dT-T7 cartilha transcrição reversa (5'-GGCCAGTGAATTGTAATACGACTCACTATAGGGAGGCGGTTTTTTTTTTTTTTTTTTTTTTTT -3 '), o calor a 70 ° C (bloco de calor) por 5 min, e encaixe legal no gelo.
- Adicione 1 mL tampão FS 5x, 0,5 mL DTT, 0,5 mL mix dNTP e 0,5 mL RnaseOUT (de SuperScript II kit).
- Calor a 42° C na máquina de PCR por 1 minuto, em seguida, adicionar 0,5 mL Sobrescrito transcriptase reversa II. Incubar por 1 hora a 42 ° C.
- Calor a 70 ° C por 10 minutos, arrefecer a 4 ° C.
- No gelo, adicione o seguinte para a reação acima:
- Água livre de nuclease - 22,75 mL
- 5X tampão vertente Segundo - 7,5 mL
- dNTP mix - 0,75 mL
- E. coli DNA Ligase - 0,25 mL
- E. coli DNA polimerase - 1 ml
- RnaseH - 0,25 mL
Incubar por 2 horas a 16 º C, em seguida, adicionar 1 mL T4 DNA polimerase, e incubar por mais 10 minutos a 16 ° C.
- Transferência para 1,5 tubo eppendorf ml, e adicionar 80 mL de água.
- Adicionar 0,5 mL de acrilamida linear.
- Adicionar 72 mL 3 M e 480 mL NH4OAc de etanol a 100% frio. Precipitar para 1 hr a -20 ° C.
- Giram a 14.000 rpm a 4 ° C por 30 minutos, lave com 1 mL de etanol a 70%, centrifugar a 14.000 rpm por 2minutos, a vácuo e seco por aproximadamente 10 minutos.
3. Amplificar poli-A do mRNA por transcrição in vitro
- Ressuspender acima cDNA em 3,5 mL de água livre de nuclease.
- Ao acima, adicionar 1 ml cada um dos dNTPs (4 mL total), 1 ml de tampão de reação 10X e 1 ml de polimerase T7, e 0,5 mL de RnaseOUT de kit Megascript.
- Incubar a 37 ° C na máquina de PCR durante a noite.
- Pronto para o próximo passo. Podem ser armazenadas a -80 ° C.
4. Fragmentação do amplificada poli-A do mRNA
- Adicionar 26 mL de água a reação acima e 4 mL de reagente fragmentação 10x.
- Calor na máquina de PCR em 70 ° C por exatamente sete minutos.
- Adicionar 5 mL de tampão parar de fragmentação, coloque amostra no gelo.
5. RNA limpar
- Para a reação acima, adicionar 60 mL de água e 350 uL de tampão RLT de kit MinElute Rneasy, e misture por pipetagem.
- Adicionar 250 mL de etanol, a mistura por pipetagem e uma pipeta na coluna spin.
- Giram a 8000 rcf durante 20 segundos.
- Lavar uma vez com RPE, spin por 20 segundos, lave segunda vez com EtOH 80%, spin por 2 minutos, e coluna seca com 5 minutos de rotação.
- Eluir RNA em 10 ml de água livre de nuclease.
6. síntese de cDNA
Síntese primeira vertente para a biblioteca ler single:
- Em um tubo de PCR, adicione o seguinte:
- Fragmentado Poly-A mRNA - 10 ml
- NotI Primer nonamer Random - 1 ml
Incubar a 70 ° C por 5 minutos em termociclador e frio rápido no gelo. NotI Primer Nonamer (-3 5'-TGAATTCGCGGCCGCTCAAGCAGAAGACGGCATACGAGCTCTTCCGATCT NNNNNNNNN '). A 5 'seqüência proximal é o sítio de restrição NotI enquanto a próxima seqüência até que a região aleatória é o complemento reverso da Illumina B do adaptador seqüência de Chip-Seq kit.
- ML tampão 5X primeira vertente -4
- TDT - 2 l
- dNTP mistura * - 1,5 mL
Colocar em termociclador por 2 minutos a 42 ° C, adicione 1 ml de SuperScript transcriptase reversa III, e incubar a 42 ° C por 1h.
* Em vez de dCTP, 5-metil dCTP foi utilizado na mistura dNTP.
Síntese primeira vertente para a biblioteca final emparelhados:
- Em um tubo de PCR, adicione o seguinte:
- Fragmentado Poly-A mRNA - 10 ml
- Cartilha hexâmero aleatória - 1 ml
Incubar a 65 ° C por 5 minutos em termociclador e frio rápido no gelo.
- Ao acima, adicione o seguinte sobre o gelo (de SuperScript firststrand III kit):
- Tampão 5X primeira vertente - 4 mL
- TDT - 2 l
- dNTPs (dekit) - 1,5 mL
Colocar em termociclador por 1 min a 45 ° C, adicione 1 ml de SuperScript transcriptase reversa III, e incubar a 45 ° C por 1 hora.
Síntese segunda vertente (para ambas as bibliotecas):
- Ao acima, adicione o seguinte sobre o gelo (de SuperScript kit II):
- Água (RNase free) - 91 mL
- Tampão 5X Segundo Strand - 30 mL
- dNTPs (de kit) - 3 mL
- E. coli DNA Ligase - 1 ml
- E. coli DNA polimerase - 4 mL
- E. coli RNase H - 1 ml
Misture por inversão do tubo, dá um giro de curto e incubar a 16 ° C por 2 horas.
7. Purify cDNA
- Purify amostra cDNA com colunas Zymo, eluir em 40 mL de água para leitura individuais e 30 l de água para a biblioteca final emparelhados.
8. Reparo final
Para a biblioteca ler single:
(Use Illumina Chip-Seq amostra prep kit)
- Para a 40 mL acima de cDNA, acrescentar:
- Tampão ligase T4 DNA com 10mM ATP - 5 mL
- dNTP mix - 2 l
- DNA polimerase T4 - 1 ml
- Klenow enzima (diluído 1:5 com água para 1U / mL) - 1 ml
- PNK T4 - 1 ml
Incubar no termociclador por 30 minutos a 20 ° C.
Para a biblioteca final emparelhados:
(Use Illumina emparelhado final amostra kit prep)
- À 30 mL acima cDNA, acrescentar:
- RNase água DNase livre - 45 mL
- Tampão ligase T4 DNA com 10mM ATP - 10 ml
- 10mM mix dNTP - 4 mL
- DNA polimerase T4 - 5 mL
- Klenow enzima - 1 ml
- PNK T4 - 5 mL
Incubar no termociclador por 30 minutos a 20 ° C.
9. limpeza cDNA
- C lean up cDNA colunas usando Zymo. Eluir em 34 mL de EB para leitura individuais e 32 mL de EB para a biblioteca final emparelhados.
10. Adicionar 'A' bases para acabar com a 3 'dos fragmentos de DNA
Para a biblioteca ler single:
- Prepare a mistura de reacção seguinte:
- Amostra de DNA - 34 mL
- Klenow buffer - 5 mL
- dATP - 10 ml
- Klenow exo - 1 ml
Incubar por 30 minutos a 37 ° C.
Para a biblioteca final emparelhados:
- Prepare a mistura de reacção seguinte:
- Amostra de DNA - 32 mL
- Klenow buffer - 5 mL
- dATP - 10 ml
- Klenow exo - 3 mL
Incubar por 30 minutos a 37 ° C.
11. limpeza cDNA
- Limpar cDNA colunas zymo usando. Eluir em 10 ml de EB.
12. Ligadura adaptador
jove_step "> Para ler única biblioteca:(Use Illumina Chip-Seq amostra prep kit)
- Prepare a mistura de reacção seguinte:
- Amostra de DNA - 10 ml
- Adaptador Oligo Mix - 1 ml
- 2X DNA Ligase mL-Buffer -15
- DNA ligase - 4 mL
Incubar por 15 minutos em temperatura ambiente. Adaptadores devem ser descongeladas em gelo e 01:20 diluída.
Para a biblioteca final emparelhados:
(Use Illumina emparelhado final amostra kit prep)
- Prepare a mistura de reacção seguinte:
- Amostra de DNA - 10 ml
- PE Adapter Mix Oligo - 10 ml
- 2X tampão de DNA Ligase - 25 mL
- DNA ligase - 5 mL
Incubar por 15 minutos a 20 ° C. Adaptadores devem ser descongeladas em gelo e 01:20 diluída.
13. Reação ligadura limpar
- Limpar a reação utilizando ligadura zycolunas mo. Eluir em 44 mL de água para a biblioteca ler único e 6 mL de EB seguido por outro de eluição em 5 mL de EB para a biblioteca final emparelhados.
Realizar uma digestão NotI, SOMENTE para a biblioteca ler única
- cDNA - 44 mL
- NEB tampão 3-5 mL
- BSA - 0,5 mL
- NotI - 1 ml
Incubar por 2 horas durante a noite a 37 ° C, ea reação purificar utilizando coluna zymo. Eluir com 6 mL de tampão EB seguido por um segundo eluição com 5 mL de EB.
14. Purificação tamanho da seleção / Gel
Execute o seguinte usando a 2% Sybr Seguro E-Gel da Invitrogen.
- Executar Programa 0-PreRun 2 minutos.
- Carga 1kb plus escada da Invitrogen 01:04 diluída, e carregar 10 ml.
- Carregar todos amostra de DNA, e encher pistas vazio com 10 ml de água.
- Executar Programa 1-Egel 2% Run 28 minutos.
- Selecione o tamanho 200-300 bp gel slice com uma lâmina de barbear fresco.
15. Eluir o DNA de fatia gel
- Pesar fatia gel, e adicionar 3 volumes de tampão QG para 1 volume de gel (use kit Extraction Gel de Qiagen)
- Incubar a 50 ° C por 10 minutos ou até que fatia gel se dissolve.
- Adicionar um volume de ispropanol, e misturar por inversão ou pipetagem.
- Adicionar a girar coluna, por 1 minuto em velocidade máxima, e descartar por escoamento.
- Adicionar 500 mL de tampão QG para a coluna, spin por 1 minuto em velocidade máxima, e descartar fluir.
- Adicionar 750 mL de tampão PE à coluna, spin por 1 minuto em velocidade máxima, e descartar fluir.
- Centrifugar por 1 minuto em velocidade máxima.
- Eluir em 36 mL de EB para leitura individuais e 23 mL para a biblioteca EB final emparelhados.
16. PCR
Para a biblioteca ler single:
(Use Illumina Chip-Seq amostra prep kit)
- Prepare o following mistura de reacção da PCR:
- DNA - 36 mL
- Phusion tampão 5X - 10 ml
- dNTP mix - 1,5 mL
- PCR primário 1,1-1 mL
- PCR primário 2,1-1 mL
- Polimerase Phusion - 0,5 mL
Use o seguinte protocolo de PCR:
- 30 segundos a 98 ° C
- 10 ciclos de:
- 10 segundos a 98 ° C
- 30 segundos a 65 ° C
- 30 segundos a 72 ° C
- 5 minutos a 72 ° C
- Segure a 4 ° C
* A primeira vez que o kit é usado, diluir PCR primers 1:2 com tampão EB.
Para a biblioteca final emparelhados:
(Use Illumina emparelhado final amostra kit prep)
- Prepare a reação de PCR seguinte:
- DNA - 23 mL
- Polimerase DNA Phusion - 25 mL
- PCR primário PE 1,1-1 mL
- PCR primário PE 2.1 -1 ml
Amplificar usando o seguinte protocolo de PCR:
- 30 segundos a 98 ° C
- 10 ciclos de:
- 10 segundos a 98 ° C
- 30 segundos a 65 ° C
- 30 segundos a 72 ° C
- 5 minutos a 72 ° C
- Segure a 4 ° C
* A primeira vez que o kit é usado, diluir PCR primers 1:2 com tampão EB.
17. Biblioteca de limpar
- Limpar biblioteca usando colunas zymo. Eluir em 12 mL de EB
18. Quantificar biblioteca
- Biblioteca quantificar usando Qubit. Ele está pronto para o seqüenciamento. Use primers de seqüenciamento de Illumina kit geração leu único cluster V4 ou superior para biblioteca de leitura simples e Illumina emparelhado final kit geração de clusters V4 ou superior para biblioteca final emparelhados.
19. A análise dos dados
Bowtie 6 foi usado para map lê para o conjunto de genes RefSeq (NCBI Construir 36.1). O fim único lê (30 nucleotídeos) e lê o final par (42 nucleotídeos) foram mapeadas, permitindo até 10 partidas para o conjunto de genes, e permitindo até dois desencontros por ler. Transcrições por milhão (TPM) valores foram obtidos para medir a expressão gênica usando RSEM 7 (RNA-Seq por Expectation-Maximization).
20. Resultados representativos: Fizemos T7LA bibliotecas para tanto o fim único de leitura e emparelhado vai de 1 mg, 100 mg, 10 mg, 1 mg e 100 pg começando RNA total (Figura 1). Para avaliação do nosso protocolo, fizemos única leitura e bibliotecas emparelhado fim sem amplificação T7 RNA a partir de 10 mg RNA totais Essas bibliotecas de controle, denominado "MinAmp", têm amplificação mínimo. A amplificação só eles sofrem são os 10 ciclos de PCR perto do final do protocolo para ligadura dos adaptadores Illumina seqüenciamento, um passo comum a todas as bibliotecas. Todos usados RNA foram isoladas de H14células tronco embrionárias humanas 8.
Primeiro, avaliamos o número de genes identificados por várias bibliotecas (Tabela 1 e Tabela Complementar 1). Para ambas as bibliotecas único fim ler e emparelhado, a 10 bibliotecas ng T7LA identificados quase o mesmo número de genes como as bibliotecas MinAmp 10 mg, com uma TPM de 10 ou mais. No caso das bibliotecas única leitura, a 10 ng T7LA biblioteca identificados em 100% dos 8.500 genes identificados pela biblioteca mcg 10 sem amplificação. Para bibliotecas final emparelhados, a 10 ng T7LA biblioteca identificou 86% dos genes identificados pela biblioteca mcg 10 sem amplificação (7.961 de 9267 genes). Bibliotecas feita a partir de menos de 10 ng não foram capazes de identificar como muitos genes. Por exemplo, no protocolo de leitura única, a uma biblioteca ng identificado apenas ~ 50% dos genes identificados pela biblioteca MinAmp 10 mg, levando-nos para limitar a menor quantidade de RNA total para uso com o protocolo T7LA a 10 ng. Além disso, o mapeamento de um gene housekeeping, GAPDH (Figura 2) mostra que todas as bibliotecas T7LA feitas com, pelo menos, 10ng de RNA a partir identificados todos os exons, incluindo os 5 extrema "exon. Comparação dos 10 ng T7LA única bibliotecas final ler e emparelhado com as bibliotecas MinAmp mostra um alto grau de similaridade (correlação de Spearman, R = 0,90 e 0,95, respectivamente, Figuras 3a e b). Também comparamos as duas bibliotecas único fim ler e emparelhado feita a partir de 10 ng de RNA total e eles tiveram um coeficiente de correlação muito alta (R = 0,92), demonstrando que ambos os tipos de bibliotecas feitas usando o protocolo T7LA produzir uma assinatura expressão muito semelhante gene ( Figura 3c.). Assim, o método T7LA é capaz de produzir bibliotecas de sequenciamento que são tão confiáveis e abrangentes como as bibliotecas MinAmp, mas a partir de 1000 vezes menos material de partida.

Figura 1 Esquema de final par e protocolo de preparação única leitura da biblioteca.

Figura 2 Uma imagem browser genoma de um gene housekeeping, GAPDH, para todas as bibliotecas único fim ler e emparelhados. A barra de escala à esquerda para as bibliotecas única leitura indica 350 Total de leituras. A barra de escala no centro para as bibliotecas final emparelhado indica 5000 Total de leituras. O eixo horizontal representa a seqüência do genoma de GAPDH.

Figura 3 Correlação de expressões de genes entre as várias bibliotecas (Spearman):. A. Entre única leia 10 ng T7LA e 10 mg biblioteca MinAmp mostra que ambas as bibliotecas têm um padrão de expressão de genes muito semelhantes (R = 0,90) B. Entre o final emparelhado 10 ng T7LA e 10 mg biblioteca MinAmp demonstra sua semelhança de perfis de expressão gênica (R = 0,95). C. Correlação de gene excompressões entre 10 ng final emparelhados e 10 ng única bibliotecas ler mostram um alto grau de semelhança entre estas bibliotecas preparadas pelo método T7LA (R = 0,92).
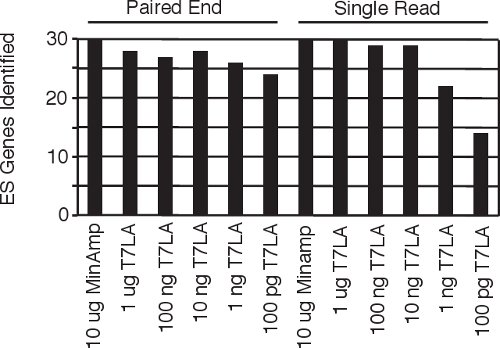
Figura 4 Identificação de genes5 células-tronco embrionárias humanas específicas de todas as bibliotecas único fim ler e emparelhados.
| Biblioteca | Tipo ° | Clusters primas | Clusters% passando filtro | Alinhando% no genoma | Taxa de erro% | Genes identificados |
| MinAmp 10ug | Leitura única | 225.602 + / - 4952 | 65,48 + / - 2,58 | 47,61 + / - 0,53 | 0,62 + / - 0,06 | 8500 |
| 1UG T7LA | Leitura única | 144.818 + / - 6513 | 82,21 + / - 6,45 | 48,09 + / - 0,27 | 0,42 + / - 0,03 | 8757 |
| 100ng T7LA | Leitura única | 27.385 + / - 1818 | 81,33 + / - 11,75 | 44,46 + / - 4,53 | 0,49 + / - 0,10 | 8709 |
| 10ng T7LA | Leitura única | 11.184 + / - 985 | 60,70 + / - 3,70 | 14,96 + / - 1,15 | 0,99 + / - 0,30 | 8589 |
| 1ng T7LA | Leitura única | 12.695 + / - 1365 | 53,27 + / - 16,76 | 4,08 + / - 0,79 | 2,25 + / - 1,56 | 4720 |
| 100PG T7LA | Leitura única | 10.390 + / - 1398 | 72,99 + / - 2,90 | 1,48 + / - 0,20 | 1,51 + / - 0,39 | 1121 |
| MinAmp 10ug | R1 End emparelhado | 95.786 + / - 12937 | 90,77 + / - 2,79 | 58,50 + / - 0,95 | 0,94 + / - 0,38 | 9267 |
| R2 End emparelhado | 95.786 + / - 12937 | 90,77 + / - 2,79 | 58,13 + / - 1,13 | 0,99 + / - 0,37 | ||
| 1UG T7LA | R1 End emparelhado | 297.669 + / - 10196 | 91,35 + / - 0,36 | 46,89 + / - 0,14 | 0,47 + / - 0,01 | 7334 |
| R2 End emparelhado | 297.669 + / - 10196 | 91,35 + / - 0,36 | 45,52 + / - 0,12 | 0,51 + / - 0,01 | ||
| 100ng T7LA | R1 End emparelhado | 205.602 + / - 9932 | 90,53 + / - 0,76 | 63,44 + / - 1,00 | 0,48 + / - 0,02 | 8011 |
| R2 End emparelhado | 205.602 + / - 9932 | 90,53 + / - 0,76 | 61,80 + / - 8,09 | 0,60+ / - 0,36 | ||
| 10ng T7LA | R1 End emparelhado | 214.622 + / - 11155 | 89,98 + / - 1,13 | 56,32 + / - 1,94 | 0,80 + / - 0,26 | 7961 |
| R2 End emparelhado | 214.622 + / - 11155 | 89,98 + / - 1,13 | 46,41 + / - 18,39 | 2,48 + / - 2,68 | ; | |
| 1ng T7LA | R1 End emparelhado | 144.951 + / - 19841 | 90,54 + / - 1,19 | 3,91 + / - 0,16 | 8,71 + / - 0,86 | 8124 |
| R2 End emparelhado | 144.951 + / - 19841 | 90,54 + / - 1,19 | 3,27 + / - 1,21 | 9,11 + / - 3,52 | ||
| 100PG T7LA | R1 End emparelhado | 187.600 + / - 11759 | 89,52 + / - 1,11 | 1,78 + / - 0,05 | 13,42 + / - 0,50 | 6623 |
| R2 End emparelhado | 187.600 + / - 11759 | 89,52 + / - 1,11 | 1,99 + / - 0,23 | 15,29 + / - 0,96 | ||
° R1 e R2 são frente e verso, sequências de uma tag
* ≥ 10 TPM
Tabela Informações 1. Sobre os números de cluster, genes identificados, a taxa de erro, o alinhamento por cento da única bibliotecas final ler e emparelhados.
Tabela Suplementar 1. Lista de todos os genes e seus valores de TPM para todas as amostras, leia única e emparelhado fim.
Discussão
Protocolos atuais para fazer bibliotecas final emparelhados exigem entre 1 mg 9-2,5 mg 10 montante inicial de RNA total. Aqui apresentamos a nossa linear de amplificação T7 método (T7LA) com base para preparar a única bibliotecas final ler e emparelhado Illumina seqüenciamento e mostrar que este método permite a geração de bibliotecas de tão baixo quanto 10 ng de RNA total, produzindo dados que é comparável à de minimamente amplificado (MinAmp) bibliotecas feitas a partir de 1000 o material de partida vezes mais (10 mg RNA total). As 10 bibliotecas ng não só identificar semelhante o número total de genes, mas também produzem assinaturas de expressão gênica que são semelhantes (Figuras 3a e b). Além disso, tanto o single bibliotecas final ler e pares produzidas pelo método T7LA são muito semelhantes entre si (Figura 3c), que permite aos pesquisadores comparar os dados gerados pelas bibliotecas feitas a partir de qualquer protocolo. Uma vez que essas bibliotecas foram preparados a partir de RNA humano de células-tronco embrionárias, buscamosde 30 genes de células-tronco específicas entre as bibliotecas e descobrir que quase todos esses genes (93-100%) são identificadas pelas bibliotecas feita a partir de pelo menos 10 ng de RNA total de partida (Figura 4), validando o nosso protocolo. Acreditamos que o nosso protocolo seria muito útil para pesquisadores, especialmente em circunstâncias como as células de fluxo classificados ou a laser micro-dissecados tecidos onde o material de partida é limitante. Em tais circunstâncias, o nosso protocolo permitiria a geração de dados de expressão gênica comparável às bibliotecas feita a partir de quantidades muito maiores de partida desde o nosso protocolo produz perfis de expressão comparáveis entre pelo menos 3 ordens de magnitude de RNA de partida.
Divulgações
Os autores revelam que JAT é um dos fundadores, stockowner, consultor e membro do conselho da Cellular Dynamics International (CDI). Ele também atua como assessor científico e tem interesses financeiros em Tactics II Ventures células-tronco.
Agradecimentos
Este trabalho foi financiado por fundos do Instituto Morgridge de Pesquisas e da Universidade de Wisconsin Foundation. Agradecemos a Krista Eastman para a assistência editorial.
Materiais
| Name | Company | Catalog Number | Comments |
| Companhia | Kit / Reagente | Catalogo | Comentários especiais |
| Ambion | O reagente de fragmentação | AM8740 | |
| Ambion | A acrilamida Liner | AM9520 | |
| Ambion | MEGAscript T7 Kit | AM1334 | |
| Ambion | DEPC não tratados água livre de nuclease | AM9932 | |
| Fermentas | dNTP set | R0181 | |
| Fermentas | dNTPs metilado | R0431 | Para preparação de amostras Único Somente leitura |
| Illumina | TruSeq SR Cluster Generation kit v5 | GD-203-5001 | Para preparação de amostras Único Somente leitura |
| Illumina | TruSeq Seq Kit v5 36 ciclos | FC-104-5001 | |
| Illumina | Chip Amostra Seq Prep Kit | IP-102-1001 | Para preparação de amostras Único Somente leitura. Pode ser substituído por amostra NEB prep DNA Mix Master Set 1 Cat # E6040S |
| Illumina | TruSeq PE Cluster Generation kit v5 | PE-203-5001 | Para prep emparelhado final amostra apenas |
| Illumina | Emparelhado End Sample Prep Kit | PE-102-1001 | Para prep emparelhado final amostra apenas |
| Invitrogen | 1kb ladder mais | 10787-018 | |
| Invitrogen | E. coli Rnase H | 18021-014 | |
| Invitrogen | Rnase Out | 10777-019 | |
| Invitrogen | Tampão vertente 2 5x | 10812-014 | |
| Invitrogen | E. coli DNA polimerase | 18010-017 | |
| Invitrogen | E-gel SYBR 2% seguro | G521802 | |
| Invitrogen | Sobrescrito III (com 5X FS buffer e DTT 0,1 M) | 18080-085 | |
| Invitrogen | Trizol | 12183555 | |
| Invitrogen | RNA ensaio quibit kit | Q32852 | |
| Invitrogen | dsDNA HS qubit ensaio kit | Q32851 | |
| Invitrogen | E. coli DNA ligase | 18052-019 | |
| Invitrogen | Polimerase DNA T4 | 18005-025 | |
| Invitrogen | Ultra Pure Dnase água Rnase | 10977-015 | |
| Invitrogen | Sobrescrito II dupla kit síntese de cDNA encalhado | 11917-020 | |
| Invitrogen | Primer aleatório (Invitrogen) | 48190-011 | Para prep emparelhado final amostra apenas |
| IDT | Not1Nonamer cartilha B | N / A | Para Single Leia samsoas prep apenas |
| IDT | Oligo dT T7 | N / A | |
| NEB | Not1 kit digestão | R0189S | Para preparação de amostras Único Somente leitura |
| Qiagen | Rneasy Minelute kit | 74204 | |
| Qiagen | RNEasy Mini Kit (50) | 74104 | |
| Qiagen | Kit de purificação de gel | 28604 | |
| Qiagen | DNase set | 79254 | |
| Qiagen | Rneasy MinElute kit | 74204 | |
| Zymo Research | DNA limpo e concentrador (250X) | D4014 |
Referências
- Tang, F., Barbacioru, C., Wang, Y., Nordman, E., Lee, C., Xu, N., Wang, X., Bodeau, J., Tuch, B. B., Siddiqui, A., Lao, K., Surani, M. A. mRNA-Seq whole-transcriptome analysis of a single cell. Nature Methods. 6, 377-382 (2009).
- Armour, C. D., Castle, J. C., Chen, R., Babak, T., Loerch, P., Jackson, S., Shah, J. K., Dey, J., Rohl, C. A., Johnson, J. M., Raymond, C. K. Digital transcriptome profiling using selective hexamer priming for cDNA synthesis. Nature Methods. 6, 647-649 (2009).
- Mortazavi, A., Williams, B. A., McCue, K., Schaeffer, L., Wold, B. Mapping and quantifying mammalian transcriptomes by RNA-Seq. Nature Methods. 5, 621-628 (2008).
- Mamanova, L., Andrews, R. M., James, K. D., Sheridan, E. M., Ellis, P. D., Langford, C. F., Ost, T. W., Collins, J. E., Turner, D. J. FRT-seq amplification-free, strand-specific transcriptome sequencing. Nature Methods. 5, 130-132 (2010).
- Sengupta, S., Ruotti, V., Bolin, J., Elwell, A., Hernandez, A., Thomson, J., Stewart, R. Highly consistent, fully representative mRNA-Seq libraries from ten nanograms of total RNA. Biotechniques. 49, 898-904 (2010).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome. Biology. 10, R25-R25 (2009).
- Li, B., Ruotti, V., Stewart, R. M., Thomson, J. A., Dewey, C. N. RNA-Seq gene expression estimation with read mapping uncertainty. Biotechniques. 26, 493-500 (2010).
- Thomson, J. A., Itskovitz-Eldor, J., Shapiro, S. S., Waknitz, M. A., Swiergiel, J. J., Marshall, V. S., Jones, J. M. Embryonic stem cell lines derived from human blastocysts. Science. 282, 1145-1147 (1998).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados