Method Article
Одноместный Читайте и парные Конец мРНК-Seq Illumina библиотек из 10 нанограмм Всего РНК
В этой статье
Резюме
Здесь мы опишем способ получения как одиночных и парных читать конца Illumina мРНК-Seq секвенирование библиотеки для экспрессии генов анализ, основанный на T7 усиления линейной РНК. Этот протокол предполагает только 10 нанограмм начала тотальной РНК и генерирует очень последовательным библиотек, представляющим весь транскриптов.
Аннотация
Всего секвенирования транскриптома по мРНК-Seq в настоящее время широко используется для выполнения глобальной экспрессии генов, мутации, аллель-специфическая экспрессия и других генома анализа. мРНК-Seq даже открывает ворота для анализа экспрессии генов не-последовательности геномов. мРНК-Seq обеспечивает высокую чувствительность, широкий динамический диапазон измерений и позволяет чисел стенограмму копию в образце. Генома анализатор Illumina это выполняет последовательность большого числа (> 10 7) относительно короткие последовательности операций чтения (<150 б.п.). "Парные конца" подход, при котором один длинный прочитанных последовательно на обоих концах, позволяет для отслеживания альтернативные переходы сращивания , вставки и удаления, и полезно для де сборку заново транскриптома.
Одной из главных задач, стоящих перед исследователями, является ограниченное количество исходного материала. Например, в экспериментах, где клетки собирают лазерной микро-диссекции, доступны с полной РНК май меры в нанограмм. Подготовка мРНК-Seq библиотеки из таких образцов были описаны 1, 2, но требует значительных ПЦР, которая может вносить систематическую ошибку. Другие РНК-Seq библиотеки строительства процедур с минимальным ПЦР были опубликованы 3, 4, но требуют мкг количества начальный суммарный вес РНК.
Здесь мы опишем протокол для Illumina Genome Analyzer II платформу для мРНК-Seq последовательности для подготовки библиотеки, что позволяет избежать значительных ПЦР-амплификации и требует всего 10 нанограмм от общего числа РНК. Хотя этот протокол был описан ранее и проверены одного конца последовательности 5, где было показано, для получения направленного библиотек, не внося значительный уклон усиления, то здесь мы проверить его в дальнейшем для использования в качестве парного конце протокола. Мы избирательно усиливать полиаденилированной РНК посланник начальный суммарный вес РНК с использованием T7 основан Eberwine линейный метод амплификации, придумал "Т7LA "(T7 линейного усиления). Усиливается поли-мРНК фрагментированы, обратной транскрипции и адаптер лигировали для получения окончательного библиотеки последовательности. Для обоих одно чтение и парных работает конца, последовательности отображаются на человека транскриптома 6 и нормированы так, что данные из нескольких работает, можно сравнить. Мы сообщаем измерения экспрессии генов в единицах стенограммы на миллион (TPM), который является превосходной мерой RPKM при сравнении образцов 7.
протокол
1. Изолировать общей РНК
- Добавить 1 мл Trizol в клетки / ткани, и гомогенизации с 18-22 марли иглу, если необходимо.
- Добавить 200 мкл хлороформа, и спина при 14000 оборотов в минуту в течение 30 минут при 4 ° C.
- Выньте верхний водный слой, добавить 0,5 мкл линейной акриламид, затем добавить 500 мкл изопропанола. Дайте настояться при комнатной температуре в течение 20 минут.
- Спиновые при 14000 оборотов в минуту при температуре 4 ° C, мыть гранул с 70% этанола, и вакуумные сухие.
- Ресуспендируют в 1 мкл нуклеазы свободной воды и передачи до 200 мкл ПЦР-пробирку.
2. Подготовка двухцепочечной кДНК
- Для более 1 мкл РНК добавляют 1 мкл 100 мкМ олиго-дТ-T7 обратный праймер транскрипции (5'-GGCCAGTGAATTGTAATACGACTCACTATAGGGAGGCGGTTTTTTTTTTTTTTTTTTTTTTTT -3 '), тепло при 70 ° С (тепловой блок) в течение 5 мин, а оснастку прохладно на льду.
- Добавить 1 мкл 5x буфера FS, 0,5 мкл DTT, 0,5 мкл смеси дНТФ и 0,5 мкл RnaseOUT (от Надстрочный II комплект).
- Тепло до 42° С в машине ПЦР в течение 1 минуты, затем добавьте 0,5 мкл Надстрочный II обратной транскриптазы. Выдержите в течение 1 часа при температуре 42 ° C.
- Тепло при 70 ° С в течение 10 мин, охлаждают до 4 ° C.
- На лед, добавьте следующие строки в указанной реакции:
- Нуклеазы свободной воды - 22,75 мкл
- 5X вторых буфера нить - 7,5 мкл
- дНТФ смеси - 0,75 мкл
- Кишечная палочка ДНК лигазы - 0,25 мкл
- Кишечная палочка ДНК-полимеразы - 1 мкл
- RnaseH - 0,25 мкл
Выдержите в течение 2 часов при 16 ° С, затем добавляют 1 мкл T4 ДНК-полимеразы, и выдержать в течение дополнительных 10 минут при 16 ° С.
- Трансфер до 1,5 мл трубки Эппендорф, и добавить 80 мкл воды.
- Добавить 0,5 мкл линейных акриламида.
- Добавить 72 мкл 3 М NH4OAc и 480 мкл холодного 100% этанола. Осадка в течение 1 часа при температуре -20 ° C.
- Спиновые при 14000 оборотов в минуту при температуре 4 ° С в течение 30 минут, промыть 1 мл 70% этанола, центрифуге при 14000 оборотов в минуту в течение 2минут, и вакуумными сохнуть в течение приблизительно 10 минут.
3. Amplify поли-мРНК в пробирке транскрипции
- Ресуспендируют выше кДНК в 3,5 мкл воды нуклеазы бесплатно.
- Чтобы выше, добавить 1 мкл дНТФ (всего 4 мкл), 1 мкл 10х буфера реакции и 1 мкл полимеразы Т7 и 0,5 мкл RnaseOUT из комплекта Megascript.
- Инкубировать при 37 ° С в машине ПЦР в одночасье.
- Готовы к следующему шагу. Может храниться при температуре -80 ° C.
4. Фрагментация усиливается поли-мРНК
- Добавить 26 мкл воды выше реакции и 4 мкл 10x реагента фрагментации.
- Тепло в машине ПЦР при 70 ° С в течение ровно 7 минут.
- Добавьте 5 мкл буфера фрагментации остановить, поставить образцы на льду.
5. РНК очистки
- Чтобы выше реакции, добавить 60 мкл воды и 350 мкл буфера RLT от Rneasy комплект MinElute и перемешать с помощью пипетки.
- Добавить 250 мкл этанола, смешать с помощью пипетки, а пипетки в спину колонке.
- Спиновые в 8000 RCF в течение 20 секунд.
- Промыть раз с НПП, спина в течение 20 секунд, мыть второй раз с 80% этанола, спина в течение 2 минут, и сухие колонки с 5 минут спина.
- Элюции РНК в 10 мкл воды нуклеазы бесплатно.
6. синтеза кДНК
Первый синтез нить для одной библиотеки читать:
- В ПЦР-пробирку, добавить следующее:
- Фрагментированные Поли-мРНК - 10 мкл
- NotI Случайные Primer nonamer - 1 мкл
Инкубировать при температуре 70 ° С в течение 5 минут в амплификаторе, и быстрое охлаждение на льду. NotI Nonamer Primer (5'-TGAATTCGCGGCCGCTCAAGCAGAAGACGGCATACGAGCTCTTCCGATCT NNNNNNNNN -3 '). 5 'проксимальной последовательности сайта NotI ограничение в то время как до следующей последовательности случайных регион обратном дополнение адаптер последовательность В Illumina из Чип-Seq комплект.
- 5X первой цепи буфера -4 мкл
- DVB-T - 2 мкл
- дНТФ смесь * - 1,5 мкл
Наденьте амплификаторе в течение 2 минут при 42 ° С, добавить 1 мкл Надстрочный транскриптазы III обратном, и инкубировать при температуре 42 ° С в течение 1 час.
* Вместо дЦТФ, 5-метил дЦТФ была использована в смеси дНТФ.
Первый синтез нити для парных конце библиотеки:
- В ПЦР-пробирку, добавить следующее:
- Фрагментированные Поли-мРНК - 10 мкл
- Случайные грунтовки гексамера - 1 мкл
Инкубировать при температуре 65 ° С в течение 5 минут в амплификаторе, и быстрое охлаждение на льду.
- Чтобы выше, добавьте следующие на льду (с Надстрочный firststrand III комплект):
- 5X первый буфер прядь - 4 мкл
- DVB-T - 2 мкл
- дНТФ (откомплект) - 1,5 мкл
Наденьте амплификаторе в течение 1 мин при 45 ° С, добавить 1 мкл Надстрочный транскриптазы III обратном, и инкубировать при 45 ° С в течение 1 часа.
Второй синтез нити (как для библиотек):
- Чтобы выше, добавьте следующие на льду (с Надстрочный II комплект):
- Вода (РНКазы бесплатно) - 91 мкл
- 5X вторых буфера Strand - 30 мкл
- дНТФ (из комплекта) - 3 мкл
- Кишечная палочка ДНК лигазы - 1 мкл
- Кишечная палочка ДНК-полимеразы - 4 мкл
- Кишечная палочка РНКазы H - 1 мкл
Смешайте трубки обращением, дать краткий спина и инкубировать при 16 ° С в течение 2 часов.
7. Purify кДНК
- Purify кДНК образца с колоннами Zymo, элюируются в 40 мкл воды для одно чтение и 30 мкл воды для парных конце библиотеки.
8. Конец ремонта
Для одиночных читать библиотеку:
(Используйте Illumina Чип-Seq образец приготовительный комплект)
- Чтобы выше 40 мкл кДНК, добавьте:
- Т4 ДНК-лигазы буфера 10 мМ ATP - 5 мкл
- дНТФ смеси - 2 мкл
- T4 ДНК-полимеразы - 1 мкл
- Кленова фермента (разбавленный 1:05 водой для 1U / мкл) - 1 мкл
- Т4 PNK - 1 мкл
Инкубируйте в термоциклер в течение 30 минут при 20 ° C.
Для парных конце библиотеки:
(Используйте Illumina парные конце образца приготовительный комплект)
- Чтобы выше 30 мкл кДНК, добавьте:
- РНКазы ДНКазы свободной воды - 45 мкл
- Т4 ДНК-лигазы буфера 10 мМ ATP - 10 мкл
- 10 мМ дНТФ смеси - 4 мкл
- T4 ДНК-полимеразы - 5 мкл
- Кленова фермента - 1 мкл
- Т4 PNK - 5 мкл
Инкубируйте в термоциклер в течение 30 минут при 20 ° C.
9. кДНК очистки
- C опираться на кДНК с использованием столбцов Zymo. Элюции в 34 мкл EB для одно чтение и 32 мкл EB для парных конце библиотеки.
10. Добавить '' баз до конца 3 'фрагментов ДНК
Для одиночных читать библиотеку:
- Подготовка следующей смеси реакции:
- ДНК-пробы - 34 мкл
- Кленова буфера - 5 мкл
- дАТФ - 10 мкл
- Кленова экзо - 1 мкл
Инкубировать 30 минут при 37 ° C.
Для парных конце библиотеки:
- Подготовка следующей смеси реакции:
- ДНК-пробы - 32 мкл
- Кленова буфера - 5 мкл
- дАТФ - 10 мкл
- Кленова экзо - 3 мкл
Инкубировать 30 минут при 37 ° C.
11. кДНК очистки
- Очистка кДНК с использованием столбцов zymo. Элюции в 10 мкл EB.
12. Адаптер лигирования
jove_step "> Для одиноких читать библиотеку:(Используйте Illumina Чип-Seq образец приготовительный комплект)
- Подготовка следующей смеси реакции:
- ДНК-пробы - 10 мкл
- Адаптер Oligo Mix - 1 мкл
- 2X ДНК лигазы буферные -15 мкл
- ДНК-лигазы - 4 мкл
Инкубировать 15 минут при комнатной температуре. Адаптеры должны быть на льду талой и разводили 1:20.
Для парных конце библиотеки:
(Используйте Illumina парные конце образца приготовительный комплект)
- Подготовка следующей смеси реакции:
- ДНК-пробы - 10 мкл
- PE адаптер Oligo Mix - 10 мкл
- 2X ДНК лигазы буфера - 25 мкл
- ДНК-лигазы - 5 мкл
Инкубировать 15 минут при 20 ° C. Адаптеры должны быть на льду талой и разводили 1:20.
13. Лигирование реакции очистки
- Очистка реакции лигирования использовании гудвижение колонны. Элюции в 44 мкл воды для одной библиотеке и читать 6 мкл EB последовала еще одна элюирования в 5 мкл EB для парных конце библиотеки.
Выполните пищеварения NotI, только за одну библиотеку читать
- кДНК - 44 мкл
- NEB буфер 3 - 5 мкл
- BSA - 0,5 мкл
- NotI - 1 мкл
Выдержите в течение 2 часов в течение ночи при 37 ° С, и очистить реакции с помощью zymo колонке. Элюции с 6 мкл буфера EB последовал второй элюирования с 5 мкл EB.
14. Размер выбора / Гель очистки
Выполните следующие использованием 2% SYBR Безопасный E-гель от Invitrogen.
- Запуск программы 0-PreRun 2 минуты.
- Нагрузка 1 КБ плюс лестница из Invitrogen разбавленной 1:4, и нагрузка 10 мкл.
- Нагрузка всех образцов ДНК, а также заполнить пустые полосы по 10 мкл воды.
- Запустите программу 1-Эгель 2% Run 28 минут.
- Размер выберите 200-300 б.п. гель SLICе с свежим лезвием бритвы.
15. Элюции ДНК из геля
- Взвесьте геля и добавить 3 объема буфера QG до 1 объема геля (используйте гель Добыча комплект от Qiagen)
- Инкубировать при температуре 50 ° С в течение 10 минут или до геля растворяется.
- Добавить 1 объем ispropanol, и соединение обращением или пипетки.
- Добавить в колонке, спина течение 1 минуты при максимальной скорости, и отказаться проточных.
- Добавить 500 мкл буфера QG к колонке, спина течение 1 минуты при максимальной скорости, и отказаться проходить через.
- Добавить 750 мкл буфера PE к колонке, спина течение 1 минуты при максимальной скорости, и отказаться проходить через.
- Центрифуга в течение 1 минуты при максимальной скорости.
- Элюции в 36 мкл EB для одно чтение и 23 мкл EB для парных конце библиотеки.
16. ПЦР
Для одиночных читать библиотеку:
(Используйте Illumina Чип-Seq образец приготовительный комплект)
- Подготовка followinг ПЦР реакционную смесь:
- ДНК - 36 мкл
- 5X буфера Phusion - 10 мкл
- дНТФ смеси - 1,5 мкл
- ПЦР праймер 1,1 - 1 мкл
- ПЦР праймер 2,1 - 1 мкл
- Phusion полимераза - 0,5 мкл
Используйте следующие ПЦР протокола:
- 30 секунд при 98 ° C
- 10 циклов:
- 10 секунд при 98 ° C
- 30 секунд при 65 ° C
- 30 секунд при 72 ° C
- 5 минут при 72 ° C
- Держите при температуре 4 ° C
* Впервые комплект используется, развести PCR праймеры 1:2 с буфером EB.
Для парных конце библиотеки:
(Используйте Illumina парные конце образца приготовительный комплект)
- Подготовка следующей реакции ПЦР:
- ДНК - 23 мкл
- Phusion ДНК-полимераза - 25 мкл
- ПЦР праймер PE 1.1 - 1 мкл
- ПЦР праймер PE 2.1 -1 мкл
Amplify, используя следующий ПЦР протокола:
- 30 секунд при 98 ° C
- 10 циклов:
- 10 секунд при 98 ° C
- 30 секунд при 65 ° C
- 30 секунд при 72 ° C
- 5 минут при 72 ° C
- Держите при температуре 4 ° C
* Впервые комплект используется, развести PCR праймеры 1:2 с буфером EB.
17. Библиотека очистки
- Очистка библиотеки с помощью zymo столбцов. Элюции в 12 мкл EB
18. Количественного библиотеки
- Количественного библиотеки с помощью кубита. Он готов для секвенирования. Использование последовательности праймеров от Illumina одного комплекта читать генерации кластеров V4 или выше для одной библиотеки и читать Illumina парные конце генерации кластеров комплект V4 или выше для парных конце библиотеки.
19. Анализ данных
Bowtie 6 был использован для мА. П. читает набор RefSeq генов (NCBI Сборка 36,1). Одного конца читает (30 нуклеотидов) и пара конце читает (42 нуклеотидов) были нанесены на карту позволяет до 10 матчей гена множество, и позволяет до двух несоответствия в чтение. Стенограммы на миллион (TPM) значения были получены для измерения экспрессии генов использованием RSEM 7 (РНК-Seq ожиданием максимизации).
20. Представитель Результаты: Мы сделали T7LA библиотеках как одиночного, так и парные читать конца длится с 1 мкг, 100 мкг, 10 мкг, 1 мкг и 100 мкг, начиная общую РНК (рис. 1). Для оценки нашего протокола, мы сделали одно чтение и парных конце библиотек без T7 РНК усиление, начиная с 10 мкг общей РНК Эти управляющие библиотеки, называется "MinAmp", обладают минимальным усилением. Только усиление они проходят в 10 циклов ПЦР в конце протокола, чтобы перевязывать Illumina секвенирования адаптеры, шаг, общих для всех библиотек. Все РНК использовали были изолированы от H14эмбриональных стволовых клеток человека 8.
Сначала мы оценивали число генов, выявленных различными библиотеками (табл. 1 и дополнительного табл. 1). Для обоих одно чтение и парных библиотеки конца, 10 нг T7LA библиотеки определены почти столько же генов, как 10 библиотек MinAmp мкг, с TPM в 10 и более. В случае одной читать библиотек, 10 нг T7LA библиотеке определили 100% от 8500 генов, определенные 10 мкг неусиленных библиотеки. Для парных библиотеки конца, 10 нг T7LA библиотеке определили 86% генов, выявленных 10 мкг неусиленных библиотека (7961 из 9267 генов). Библиотеки из менее 10 нг не смогли определить, как много генов. Например, в одном протоколе читать, 1 нг библиотеки выявлены только ~ 50% генов, определенные библиотеки MinAmp 10 мкг, что заставило нас ограничить низкое количество общей РНК для использования с протоколом T7LA до 10 нг. Кроме того, картирование генов домашнего хозяйства, GAPDH (рис. 2) показывает, что все T7LA библиотеки сделали, по крайней мере 10ng начала РНК определили все экзонов, в том числе крайних 5 'экзонов. Сравнение 10 нг T7LA одно чтение и парных конце библиотеки с библиотеками MinAmp показывает высокую степень сходства (корреляции Спирмена, R = 0,90 и 0,95 соответственно, 3а, б). Мы также сравнили две односпальные читать и парных конце библиотек из 10 нг общей РНК и у них было очень высоким коэффициентом корреляции (R = 0,92), демонстрируя, что оба типа библиотек с использованием протокола T7LA производят очень похожи подпись экспрессии генов ( 3, в.). Таким образом, метод T7LA способна производить секвенирование библиотек, которые являются надежным и всеобъемлющим, как MinAmp библиотек, но с 1000 раз меньше исходного материала.

Рисунок 1 Схема парных и одиночных конца прочитать протокол библиотеки подготовки.

Рисунок 2 картина генома браузер домашнего хозяйства ген, GAPDH, для всех одно чтение и парных библиотеки конца. Шкалы слева для одной библиотеки читать указывает 350 общего читает. Шкалы в центре для парных библиотек конце 5000 указывается общая говорится в сообщении. Горизонтальная ось представляет геном последовательность GAPDH.

Рисунок 3 Соотношение гена выражения между различными библиотеками (Спирмена). A. С одной читать 10 нг T7LA и 10 мкг MinAmp библиотеки показывает, что обе эти библиотеки имеют очень похожий рисунок экспрессии генов (R = 0,90) B. Между парными конца 10 нг T7LA и 10 мкг MinAmp библиотеки показывает их сходство профилей экспрессии генов (R = 0,95). C. Соотношение гена бывшихвыражений от 10 нг парные конца и 10 нг одной читать библиотек показывают высокую степень сходства между этими библиотеками подготовленных методом T7LA (R = 0,92).
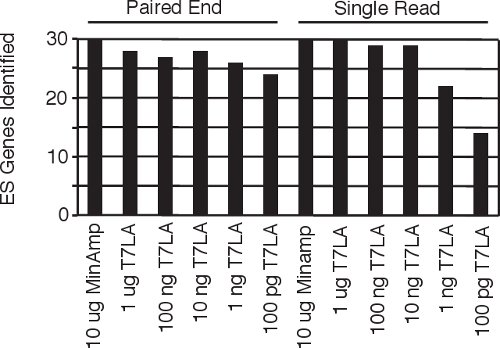
Рисунок 4 Определение эмбриональных стволовых клеток человека конкретных genes5 со всего одно чтение и парных библиотеки конца.
| Библиотека | Типа ¯ | Сырье Кластеры | Кластеры% прохождение фильтра | % Приведение к геному | Оценить% Погрешность | Гены определены |
| MinAmp 10ug | Одноместный читать | 225 602 + / - 4952 | 65,48 + / - 2,58 | 47,61 + / - 0,53 | 0,62 + / - 0,06 | 8500 |
| 1ug T7LA | Одноместный читать | 144 818 + / - 6513 | 82,21 + / - 6,45 | 48,09 + / - 0,27 | 0,42 + / - 0,03 | 8757 |
| 100ng T7LA | Одноместный читать | 27 385 + / - 1818 | 81,33 + / - 11.75 | 44,46 + / - 4,53 | 0,49 + / - 0,10 | 8709 |
| 10ng T7LA | Одноместный читать | 11 184 + / - 985 | 60,70 + / - 3,70 | 14,96 + / - 1,15 | 0.99 + / - 0,30 | 8589 |
| 1ng T7LA | Одноместный читать | 12 695 + / - 1365 | 53,27 + / - 16,76 | 4,08 + / - 0,79 | 2,25 + / - 1,56 | 4720 |
| 100pg T7LA | Одноместный читать | 10 390 + / - 1398 | 72,99 + / - 2,90 | 1,48 + / - 0,20 | 1,51 + / - 0,39 | 1121 |
| MinAmp 10ug | Парные Конец R1 | 95 786 + / - 12937 | 90,77 + / - 2,79 | 58,50 + / - 0,95 | 0,94 + / - 0,38 | 9267 |
| Парные Конец R2 | 95 786 + / - 12937 | 90,77 + / - 2,79 | 58,13 + / - 1,13 | 0.99 + / - 0.37 | ||
| 1ug T7LA | Парные Конец R1 | 297 669 + / - 10196 | 91,35 + / - 0,36 | 46,89 + / - 0,14 | 0,47 + / - 0,01 | 7334 |
| Парные Конец R2 | 297 669 + / - 10196 | 91,35 + / - 0,36 | 45,52 + / - 0,12 | 0,51 + / - 0,01 | ||
| 100ng T7LA | Парные Конец R1 | 205 602 + / - 9932 | 90,53 + / - 0,76 | 63,44 + / - 1,00 | 0,48 + / - 0,02 | 8011 |
| Парные Конец R2 | 205 602 + / - 9932 | 90,53 + / - 0,76 | 61,80 + / - 8,09 | 0,60+ / - 0,36 | ||
| 10ng T7LA | Парные Конец R1 | 214 622 + / - 11155 | 89,98 + / - 1,13 | 56,32 + / - 1,94 | 0,80 + / - 0,26 | 7961 |
| Парные Конец R2 | 214 622 + / - 11155 | 89,98 + / - 1,13 | 46,41 + / - 18,39 | 2,48 + / - 2,68 | ; | |
| 1ng T7LA | Парные Конец R1 | 144 951 + / - 19841 | 90,54 + / - 1,19 | 3,91 + / - 0,16 | 8,71 + / - 0,86 | 8124 |
| Парные Конец R2 | 144 951 + / - 19841 | 90,54 + / - 1,19 | 3,27 + / - 1,21 | 9,11 + / - 3,52 | ||
| 100pg T7LA | Парные Конец R1 | 187 600 + / - 11759 | 89,52 + / - 1,11 | 1,78 + / - 0,05 | 13,42 + / - 0,50 | 6623 |
| Парные Конец R2 | 187 600 + / - 11759 | 89,52 + / - 1,11 | 1,99 + / - 0,23 | 15,29 + / - 0,96 | ||
° R1 и R2 прямой и обратной последовательностей теги
* ≥ 10 TPM
Таблица 1. Информация о номера кластеров, гены определены, уровень ошибок, процент выравнивания одной читать и парных библиотеки конца.
Дополнительная Таблица 1. Список всех генов и их значения TPM для всех образцов, один читал и парных конца.
Обсуждение
Действующие протоколы для изготовления парных библиотеки конца требует от 1 до 9 мкг до 2,5 мкг 10, начиная от общего количество РНК. Здесь мы представляем наш линейного усиления T7 основе (T7LA) для подготовки как одиночного, так и парные читать конца Illumina секвенирование библиотек и показать, что этот метод позволяет получать библиотек на уровне 10 нг общей РНК, производя данных, сравнимую с минимально усиливается (MinAmp) библиотеки из 1000 раз больше исходного материала (10 мкг общей РНК). 10 нг библиотеки не только выявить подобный общее число генов, но также производят экспрессии генов подписи, которые подобны (рис. 3а, б). Более того, и одно чтение и парных конце библиотек, полученных методом T7LA очень похожи друг на друга (рис. 3в), что позволяет исследователям сравнить данные, полученные из библиотеки либо протокол. Поскольку эти библиотеки были изготовлены из человеческих эмбриональных стволовых клеток, РНК, мы искалив течение 30 стволовых клеток, специфических генов среди библиотек и обнаружили, что почти все эти гены (93-100%) идентифицируются библиотек из не менее 10 нг начала тотальной РНК (рис. 4), таким образом, проверка нашего протокола. Мы считаем, что наш протокол будет очень полезной для исследователей, особенно в обстоятельствах, таких как поток отсортированных клеток или лазерной микро-расчлененный ткани, где исходным материалом является предельным. В таких условиях наш протокол позволит поколение данных генной экспрессии сопоставимо с библиотеками из гораздо большего количества, начиная со времени нашего протокола производит профили экспрессии сопоставимы по крайней мере, 3 порядков, начиная РНК.
Раскрытие информации
Авторы раскрывают, что JAT является учредителем, акционерному, консультантом и членом правления Сотовая Динамика International (CDI). Он также выступает в качестве научного советника и имеет финансовые интересы в тактике Ventures II стволовых клеток.
Благодарности
Эта работа была поддержана финансирование из Моргридж Института исследований и Университета Висконсин Foundation. Мы благодарим Криста Eastman за помощь в редактировании.
Материалы
| Name | Company | Catalog Number | Comments |
| Компания | Kit / Реагенты | Каталог # | Специальные Комментарии |
| Амбион | Фрагментация реагента | AM8740 | |
| Амбион | Liner Акриламид | AM9520 | |
| Амбион | MEGAscript T7 Kit | AM1334 | |
| Амбион | Номера DEPC лечение нуклеазы свободной воды | AM9932 | |
| Fermentas | дНТФ набор | R0181 | |
| Fermentas | метилированные дНТФ | R0431 | Для Одноместный Прочитано образец приготовительный только |
| Illumina | TruSeq SR генерации кластеров комплект v5 | GD-203-5001 | Для Одноместный Прочитано образец приготовительный только |
| Illumina | TruSeq Seq Kit v5 36 циклов | FC-104-5001 | |
| Illumina | Чип Пример Seq Prep Kit | IP-102-1001 | Для Одноместный Прочитано образец приготовительный только. Может быть заменен NEB образец ДНК Mix приготовительный Мастер Set 1 Cat # E6040S |
| Illumina | TruSeq PE генерации кластеров комплект v5 | ПЭ-203-5001 | Для парных приготовительный образца конца только |
| Illumina | Парные Конец примера Prep Kit | ПЭ-102-1001 | Для парных приготовительный образца конца только |
| Invitrogen | 1 КБ плюс лестница | 10787-018 | |
| Invitrogen | Кишечная палочка РНКазы Н | 18021-014 | |
| Invitrogen | РНКазы Out | 10777-019 | |
| Invitrogen | Второй буфер прядь 5x | 10812-014 | |
| Invitrogen | Кишечная палочка ДНК-полимеразы | 18010-017 | |
| Invitrogen | E-гель SYBR безопасной 2% | G521802 | |
| Invitrogen | Надстрочный яII (с 5-кратным FS буфера и 0,1 М ДТТ) | 18080-085 | |
| Invitrogen | Trizol | 12183555 | |
| Invitrogen | РНК quibit анализа комплект | Q32852 | |
| Invitrogen | дц HS кубит анализа комплект | Q32851 | |
| Invitrogen | Кишечная палочка ДНК-лигазы | 18052-019 | |
| Invitrogen | T4 ДНК-полимеразы | 18005-025 | |
| Invitrogen | Ultra Pure ДНКазы РНКазы воды | 10977-015 | |
| Invitrogen | Надстрочный II двухцепочечной кДНК комплект синтеза | 11917-020 | |
| Invitrogen | Случайные Primer (Invitrogen) | 48190-011 | Для парных приготовительный образца конца только |
| IDT | Not1Nonamer B грунтовки | N / A | Для Одноместный Прочитано образцовНапример приготовительный только |
| IDT | Oligo дТ T7 | N / A | |
| NEB | Not1 пищеварения комплект | R0189S | Для Одноместный Прочитано образец приготовительный только |
| Qiagen | Rneasy Minelute комплект | 74204 | |
| Qiagen | RNEasy Mini Kit (50) | 74104 | |
| Qiagen | Комплект Гель очистки | 28604 | |
| Qiagen | ДНКазы набор | 79254 | |
| Qiagen | Rneasy MinElute комплект | 74204 | |
| Zymo исследований | ДНК чистой и концентратора (250X) | D4014 |
Ссылки
- Tang, F., Barbacioru, C., Wang, Y., Nordman, E., Lee, C., Xu, N., Wang, X., Bodeau, J., Tuch, B. B., Siddiqui, A., Lao, K., Surani, M. A. mRNA-Seq whole-transcriptome analysis of a single cell. Nature Methods. 6, 377-382 (2009).
- Armour, C. D., Castle, J. C., Chen, R., Babak, T., Loerch, P., Jackson, S., Shah, J. K., Dey, J., Rohl, C. A., Johnson, J. M., Raymond, C. K. Digital transcriptome profiling using selective hexamer priming for cDNA synthesis. Nature Methods. 6, 647-649 (2009).
- Mortazavi, A., Williams, B. A., McCue, K., Schaeffer, L., Wold, B. Mapping and quantifying mammalian transcriptomes by RNA-Seq. Nature Methods. 5, 621-628 (2008).
- Mamanova, L., Andrews, R. M., James, K. D., Sheridan, E. M., Ellis, P. D., Langford, C. F., Ost, T. W., Collins, J. E., Turner, D. J. FRT-seq amplification-free, strand-specific transcriptome sequencing. Nature Methods. 5, 130-132 (2010).
- Sengupta, S., Ruotti, V., Bolin, J., Elwell, A., Hernandez, A., Thomson, J., Stewart, R. Highly consistent, fully representative mRNA-Seq libraries from ten nanograms of total RNA. Biotechniques. 49, 898-904 (2010).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome. Biology. 10, R25-R25 (2009).
- Li, B., Ruotti, V., Stewart, R. M., Thomson, J. A., Dewey, C. N. RNA-Seq gene expression estimation with read mapping uncertainty. Biotechniques. 26, 493-500 (2010).
- Thomson, J. A., Itskovitz-Eldor, J., Shapiro, S. S., Waknitz, M. A., Swiergiel, J. J., Marshall, V. S., Jones, J. M. Embryonic stem cell lines derived from human blastocysts. Science. 282, 1145-1147 (1998).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены