Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Multiplex detección de bacterias en el complejo de muestras clínicas y ambientales utilizando oligonucleótidos de acoplamiento de microesferas fluorescentes
En este artículo
Resumen
Se describe un método multiplex para la detección de microorganismos en una muestra utilizando oligonucleótidos junto perlas fluorescentes. Amplificación de todos los organismos dentro de una muestra se hibridó con un panel de cuentas, junto sonda. Un instrumento Luminex o Bio-Plex se utiliza para consultar cada cuenta para el tipo de cuentas y la señal de hibridación.
Resumen
La vaginosis bacteriana (VB) es un síndrome recurrente polimicrobiana que se caracteriza por un cambio en la "normal" microbiota de Lactobacillus dominado por una microbiota dominado por una serie de especies bacterianas, incluyendo Gardnerella vaginalis, Atopobium vaginas, y 3.1 los demás. Esta condición se asocia con una serie de resultados negativos para la salud, incluyendo la adquisición del VIH 4, y puede ser difícil de manejar clínicamente 5. Además, el diagnóstico de vaginosis bacteriana se ha basado en el uso de la tinción de Gram de los frotis de hisopado vaginal que se califican en diversos criterios numéricos 6,7. Si bien este diagnóstico es sencillo, barato y bien adaptado a entornos con recursos limitados, que pueden sufrir de problemas relacionados con las interpretaciones subjetivas y no le da un perfil detallado de la composición de la microbiota vaginal 8. Los recientes esfuerzos de secuenciación profunda han revelado una microbiota rico, diverso, con la vaginaclaras diferencias entre las muestras tomadas de las personas que son diagnosticadas con BV en comparación con los individuos que se consideran normales 9,10, lo que ha dado como resultado la identificación de una serie de posibles objetivos para el diagnóstico molecular de la BV 11,12. Estos estudios han proporcionado una gran cantidad de información útil, pero la secuencia no es profundo y práctico como un método de diagnóstico en un entorno clínico. Recientemente hemos descrito un método para rápidamente el perfil de la microbiota vaginal en un formato multiplex utilizando oligonucleótidos, junto con la detección de perlas fluorescentes en una plataforma Luminex 13. Este método, como el actual la tinción de Gram basada en métodos, es rápido y sencillo, pero añade la ventaja adicional de la explotación del conocimiento molecular derivados de estudios de secuenciación en el diseño de la sonda. Por lo tanto, este método proporciona una forma de perfil de los microorganismos más importantes que están presentes en una muestra vaginal que se puede utilizar para diagnosticar BV con una alta especificidad y sensibilidad de un borradorared con tinción de Gram, mientras que el suministro de información adicional sobre la presencia y abundancia de especies de una manera semi-cuantitativa y rápida. Este método múltiple se puede ampliar mucho más allá del alcance de los actuales ensayos de PCR cuantitativa a organismos particulares, que actualmente se limita a 5 ó 6 pruebas diferentes en una sola muestra 14. Es importante destacar que el método no se limita a la detección de bacterias en muestras vaginales y pueden ser fácilmente adaptados al perfil rápidamente casi cualquier comunidad microbiana de interés. Por ejemplo, recientemente hemos comenzado a aplicar esta metodología para el desarrollo de herramientas de diagnóstico para su uso en plantas de tratamiento de aguas residuales.
Protocolo
Este método fue utilizado en la investigación publicada en Dumonceaux et al. J. Clin. . Microbiol 47, 4067-4077, doi: 10.1128/jcm.00112-09 (2009).
Un diagrama esquemático que representa el procedimiento general se presenta en la Figura 1.
1. Bolas de acoplamiento
Esto describe los métodos que se utilizan para el acoplamiento de sondas de oligonucleótidos de poliestireno cuentas Luminex (ver Tabla 2). Los volúmenes se adaptó ligeramente para la evaluación de nuevas sondas de captura a modo de prueba, estos volúmenes se indican entre paréntesis.
- Retire 1-etil-3-(3-dimethylamiopropyl) carbodiimida HCl (EDC) en polvo de -20 ° C y caliente desecador a temperatura ambiente.
- Volver a suspender las microesferas por sonciation en un baño de ultrasonidos baño de agua durante 20 segundos, luego agitación unos 20 segundos.
- Transferencia de 400 l (100 l) de microesferas a un tubo Eppendorf (esto corresponde a 5x10 6 o 1,25 x10 6 microesferas). Luminex recomienda el uso de tubos de microcentrífuga de baja proteína vinculante (por ejemplo, tubos Eppendorf LoBind proteína, número de catálogo 0030 108.094) para evitar que las cuentas desacoplado se pegue a los tubos y de interferir con la recuperación de cuentas. No hemos encontrado se trata de un problema con estándar tubos de microcentrífuga de polipropileno, que suelen utilizar (por ejemplo, número de catálogo VWR 87003-298).
- Pellet a 14000 xg durante 1 minuto. Retire y deseche el sobrenadante.
- Resuspender las microesferas en 50 l (12,5 l) de 0,1 a temperatura ambiente M 2 - (N-morfolino) etanosulfónico (MES) de pH 4,5.
- Prepare una solución fresca de la EDC en 10 mg / ml en el agua.
- Preparar a menos de 1 ml de esta solución por un peso de 5-10 mg de EDC en una escala de análisis, a continuación, añadir agua hasta 10 mg / ml.
- Añadir 1 nmol de 5'-amino de oligonucleótidos de captura de C12 modificado (Tabla 2) de microesferas y mezclar mediante agitación. 1 nmol es de 5 l de 200 M oligonucleótidos.
- Añadir 2,5 l de solución fresca EDC a las micro y mezclar mediante agitación durante 5 segundos.
- Incube a temperatura ambiente durante 30 minutos en la oscuridad.
- Deseche la solución EDC (paso 1.6) y preparar una nueva muestra de 10 mg / ml EDC en el agua que el anterior (paso 1.6).
- Añadir otro l 2.5 de nuevo solución de la EDC a las microesferas y agitar durante 5 segundos.
- Incube a temperatura ambiente durante 30 minutos en la oscuridad.
- Lave las cuentas mediante la adición de 1 ml de 0,02% de Tween 20. Vortex (opcional ultrasonidos durante 20 segundos también) para volver a suspender las cuentas.
- Centrífuga 14.000 xg 1 minuto. Retire y deseche el sobrenadante.
- Lave las cuentas de nuevo por la adición de 1 ml de sulfato de sodio 0,1% dodecilsulfato (SDS). Vortex (opcional ultrasonidos durante 20 segundos también) para volver a suspender las cuentas.
- Centrífuga 14.000 xg 1 minuto. Retire y deseche el sobrenadante.
- Volver a suspender las cuentas de 100 l (25 l) de Tris-EDTA (TE) buffer [10 mM Tris-Cl pH 8,0, 1 mM EDTA, pH 8,0].
- Enumerar las cuentas en un hemocitómetro o contador Coulter para determinar la concentración de valores.
- Prepare una mezcla maestra de microesferas mediante la dilución de cada cuenta a una concentración final de 100 bolas / l en buffer TE. Piscina junto cuentas correspondientes a los complejos deseados de la prueba (por ejemplo, para un ensayo de 10 complejos, mezcla de 10 cuentas diferentes, junto a una concentración final de 100/μl de cada cuenta).
- Guarde el Master Mix microesferas a 4 ° C en la oscuridad. La mezcla puede ser almacenado durante meses si se mantiene en estas condiciones.
2. Chaperonina 60 universal, objetivo (cpn60 UT) la producción de amplificación y la generación de cadenas simples.
- Generar cadena de la polimerasa (PCR) de productos para cada muestra. Incluir el fosforotioato y establecer biotina modificado cebador 5 '(Tabla 1). Ver Tabla 3 para el volumen de la mezcla y la concentración.
- Inmediatamente después de la PCR se ha completado, añadir 2 l (20 unidades) de T7 exonuclease a cada tubo de PCR (el buffer de PCR será suficiente para la reacción T7). Incubar la reacción a temperatura ambiente (~ 22-25 ° C) durante 40 minutos.
- Al final de esta incubación, añadir 12,5 l de 0,5 M de etileno diamino tetraacético (EDTA), pH 8,0 y mezclar. Esto da un total de ~ 64.5μl de una sola cadena de productos PCR.
3. La hibridación de una sola cadena de productos PCR de bolas de poliestireno junto oligonucleótidos.
- Precalentar el instrumento a 60 ° C para mantener la temperatura de hibridación durante los análisis. Encienda el calentador de la plataforma utilizando el software del instrumento y asegúrese de usar el calentador de bronce que se ajuste a la placa de PCR de estilo que contiene la mezcla de grano híbrido.
- Volver a suspender las microesferas Mix Master (paso 1,20) con una pipeta, dispense una cantidad adecuada en un tubo Eppendorf, tapar el tubo, y ultrasonidos en un baño de agua sonicador durante 2 minutos. Por otra parte, para garantizar la coherencia absoluta en grano resuspension, la mezcla maestra de microesferas pueden ser resuspendidas por agitación e pipeteo, entonces sonicación de arriba, luego dispensar la cantidad deseada en un tubo de polipropileno Eppendorf.
- Prescindir de 17 l de una sola cadena de productos PCR (paso 2.2) en los pocillos correspondientes de un bajo perfil-96-así Vaina placa PCR (Tabla 2). Añadir 33 l de mezcla resuspendido, cuenta sonciated a cada pocillo. Cubra con una cubierta de silicona (Tabla 2) y golpear suavemente.
- Colocar la placa en el termociclador con un programa de: 95 ° C durante 5 min, 60 ° C durante 10 min, 60 ° C espera, 60 ° C durante 5 minutos, al final. Iniciar el programa.
- Hacer nuevas estreptavidina-ficoeritrina (SA-PE) la solución, usted tendrá 25 l por pocillo (hacer varios pozos adicionales). Haga SA-PE mediante la dilución de la solución de archivo SA-PE (1 mg / ml) de 1:50 a 20 mg / ml con cloruro de tetrametilamonio 1x (TMAC) buffer (TMAC 3 M, 0,1% Sarkosyl, 50 mM Tris-HCl, pH 8,0 , 4 mM EDTA, pH 8,0).
- Cuando el termociclador llega a los 60 ° C mantener el paso,abrir la tapa, quitar la silicona y añadir SA-PE directamente la solución a cada pocillo (no tomar la placa del termociclador). Vuelva a colocar la cubierta de silicona, cierre la tapa del termociclador y reanudar el programa.
- Cuando el programa está completo, tome el plato y rápidamente la transferencia a la máquina BioPlex de leer. La placa debe ser leído en 10 minutos. Leer a los 60 ° C; asegurarse de que el BioPlex ha sido precalentada a esta temperatura. Asegúrese de que la altura de la sonda se ha ajustado para dar cabida a la placa utilizada, tal como se describe en el manual del usuario del instrumento utilizado.
- De precisión de cuentas y detección de la señal, la configuración de la puerta de la BioPlex se debe establecer en función del tipo de microesferas que se utiliza. Perlas de poliestireno BioRad requieren una configuración de puerta de 4,335-10,000 mientras microesferas magnéticas requieren un ajuste de 5,000-25,000. También existe la opción de ejecutar la placa con el ajuste de alta PMT que aumenta el valor de ganancia reportero. Esto lo puedoUMENTAR la intensidad de las señales de menor sin embargo, es importante incluir un control negativo apropiado como señal de fondo también se incrementará.
4. Los resultados representativos:
Uno de nuestros objetivos en el desarrollo y la aplicación de este ensayo fue que sea lo más sencilla y ágil posible. Por lo tanto, optimizar la crítica post-amplificación de los pasos, incluyendo el tiempo de tratamiento T7 exonucleasa y el tiempo de hibridación con el fin de desarrollar un análisis que se puede completar en un plazo razonable. Como se muestra en la Figura 2, el tratamiento T7 de la amplificación es esencial para la generación de señales, como la mayoría de las sondas había poca señal o no con el tratamiento T7 corto o no. La señal de un aumento lineal hasta aproximadamente 40 minutos, momento en que el aumento de la señal de frenado. No hay degradación de la señal se observó incluso a las 2 horas de tiempo de tratamiento T7, lo que indica que la modificación fosforotioato de los cebadores es muy eficaz en la prevención de targedegradación t cadena como se describe 15. Hemos elegido 40 minutos para el tiempo de tratamiento T7 para minimizar el tiempo del protocolo general, sino que se desprende de la Figura 2A que el tratamiento T7 puede continuar durante mucho más tiempo. También se determinó el efecto del tiempo de hibridación en la generación de señales (Figura 2B) y encontró que 10 minutos era suficiente para la máxima señal, ya que ningún nuevo aumento de la señal se observó incluso después de 4 horas de la hibridación. Por lo tanto, una etapa de hibridación 10 minutos fue elegido de nuevo para reducir al mínimo el tiempo de ensayo general. Con estos resultados en mente, y con técnicas rápidas de extracción de ADN, como InstaGene (Bio-Rad), el ensayo general que incluye la extracción de ADN, PCR y análisis de Luminex se puede realizar en 4-5 horas.
En una nota de precaución, el nivel de agregación de las cuentas de poliestireno durante una ejecución de Luminex o BioPlex puede tener un gran impacto en la eficiencia de la prueba. El software BioPlex mostrará el nivel de agregación de cuentas, que ocurre cuandomás de un cordón se detecta en la ruta de láser y los resultados en la exclusión de la señal de hibridación de los agregados. Agregación de cuentas aparente también puede ser causada por cualquier partícula que no es el tamaño adecuado para una sola cuenta, e incluso puede ser causada por las burbujas de aire. En cualquiera de estos eventos, la señal de los agregados de cuentas, burbuja de aire o de partículas se descarta. En la mayoría de los casos, nos encontramos con que la agregación de cuentas es mínima (Figura 3) y el nivel previsto de 100 eventos de conteo para cada cuenta es fácil de alcanzar. Ocasionalmente, sin embargo, las cuentas muestran un incremento moderado (Figura 3B) o grave (Figura 3 C) nivel de agregación. En estos casos, ya que la mayoría de los datos se descartan, el instrumento puede tener problemas para llegar a 100 eventos por tipo de cuentas y los resultados pueden ser cuestionables. El paso de ultrasonidos (paso 3.2) tiene la intención de minimizar la agregación de cuentas. Además, el almacenamiento de las cuentas como una mezcla de microesferas maestro diluida (paso 1,20) puede ayudar. Hemos notado que TMAC excluir de la hybribuffer dization - añadiendo que el diluyente SA-PE en su lugar (paso 3.5) - ayuda a reducir la agregación de cuentas. Por otra parte, aunque no he probado esto, las cuentas más recientes magnética disponible en Luminex o BioRad se cree que mostrar una menor tendencia a agregarse.
Los resultados de la aplicación de un conjunto complejo de 5 Luminex objetivo G. vaginalis, A. Vaginas, L. Iners, L. Crispatus, y L. gasseri junto con los correspondientes Gram frotis vaginal hisopo se muestran en la Figura 4. Estas muestras fueron tomadas de un solo individuo en múltiples momentos. En el momento 0, la persona fue diagnosticada con la VB basado en la tinción de Gram (Figura 4) y los resultados de Luminex de esta misma muestra (Figura 4) muestran que de los organismos representados en la matriz, G. vaginalis fue más frecuente, mientras que A. Vaginas y L. Iners también fueron positivos. Nueve días después, el individuo todavía estaba BV positivo y el signal de G. vaginalis había aumentado considerablemente, mientras que A. Vaginas y L. Iners todavía positivo. Es importante destacar que, después de este tiempo el individuo comenzó a hacer la transición a una microbiota normal como bacilos Gram positivos se detecta en las manchas (Figura 4), mientras que la señal de G. vaginalis disminuyó y la señal de L. Iners aumentado (Figura 4B). Por nuestra definición original de BV con este método (muestras positivas de G. vaginalis y / o A. Vaginas fueron considerados positivos BV) 13, este individuo fue positivo BV en todos los tiempos, aunque la tinción de Gram no pudo detectar G. vaginalis en los puntos de tiempo de estos dos últimos. Con el método de Luminex se describe aquí, las tendencias pueden ser fácilmente comparado con los métodos basados en la secuenciación, y los resultados de la prueba de Luminex general corroborar la tinción de Gram 13, mientras que proporcionar información adicional sobre la identidad de organismo y de la abundancia.

Figura 1. Esquema del protocolo de Luminex para determinar el perfil de la microbiota de una muestra compleja clínicas o ambientales. El protocolo comienza con el ADN de plantilla que se ha extraído de la muestra de interés. (1) Generar productos PCR de ADN de la plantilla con el capítulo específico de biotina, fosforotioato modificados cpn60 UT PCR, junto con modificar cpn60 UT PCR (Tabla 1), (2) Esto genera un conjunto de productos de PCR en representación de la microbiota con la biotina fosforotioato modificación de una cadena, (3) Resumen del producto de doble cadena PCR con exonucleasa T7, que no pueden degradar la cadena fosforotioato modificado y por lo tanto genera una sola cadena de ADN que se ha modificado en el extremo 5 'con biotina, (4) Pareja bolitas de poliestireno para cada especie cpn60 sondas UT - cada cuenta tiene una única dirección espectral (indicado por el color de cuentas) que idiscernible por el Luminex o instrumento Bio-Plex s; (5) híbridos con el producto una sola cadena PCR generados a partir de la muestra de interés para el conjunto de especies específicas, junto oligonucleótidos cuentas; (6) Añadir estreptavidina-ficoeritrina conjugado que se une a la biotina sola cadena de productos PCR, y actúa como un indicador de la hibridación; (7) Determinar la dirección espectral (bolas de identidad) y la intensidad de la señal de hibridación mediante un instrumento Luminex o Bio-Plex. Por lo menos 100 cuentas se cuenta para cada identidad de cuentas y la intensidad media de fluorescencia (MFI) de la señal de ficoeritrina se presenta como la salida. Hasta 100 tipos diferentes de cuentas se puede evaluar de forma simultánea, sino un ensayo que muestra la discriminación de los tres tipos de cuentas es ilustrado. (8) Cuando el MFI de PCR se replica generados a partir de la misma muestra es significativamente mayor que el control negativo para un determinado grano (una cola de la t-test, p <0,05), la muestra se considera positiva para ese organismo.
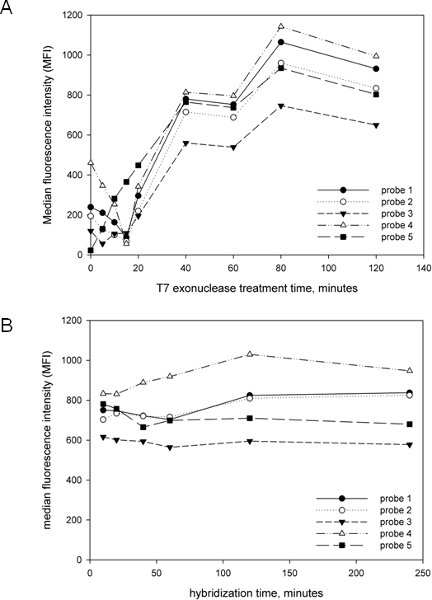
Figura 2. Optimización de los parámetros del ensayo Luminex. Cinco diferentes sondas dirigidas a anaerobius Peptostreptococcus se utilizaron con amplicones generados a partir de una plantilla mixta que comprende plásmidos que contienen el clonado cpn60 UT de 20 bacterias se sabe están asociados con la vagina, incluyendo P. anaerobius. (A). Efecto del tiempo de tratamiento T7 exonucleasa. El protocolo descrito anteriormente fue seguido, pero el tiempo de tratamiento con exonucleasa T7 fue variada antes de la hibridación y la intensidad media de fluorescencia (MFI) se determinó para todas las sondas. (B). Efecto del tiempo de hibridación. El protocolo descrito anteriormente fue seguido con una variedad de tiempos de hibridación. Los mismos conjuntos de sondas se utilizan con una amplificación generados a partir de la misma plantilla que en la A y la intensidad media de fluorescencia (MFI) se determinó para todas las sondas.
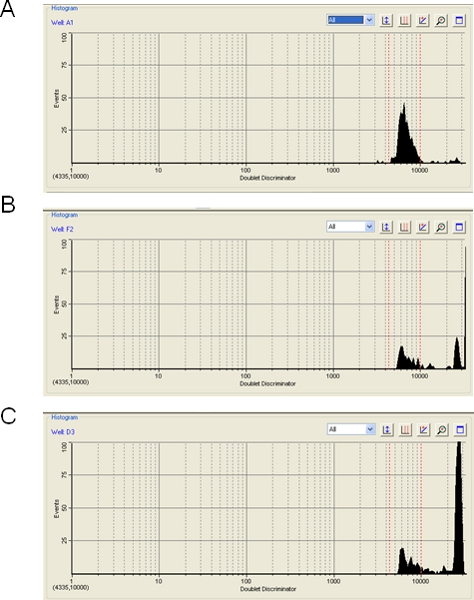
Figura 3. Determinación de la agregación de cuentas el uso de software BioPlex. Tres ejemplos de carreras BioPlex en el que el nivel de agregación de cuentas es (A) aceptable (5%), (B) en el límite (50%); e inaceptable (C) (80%).
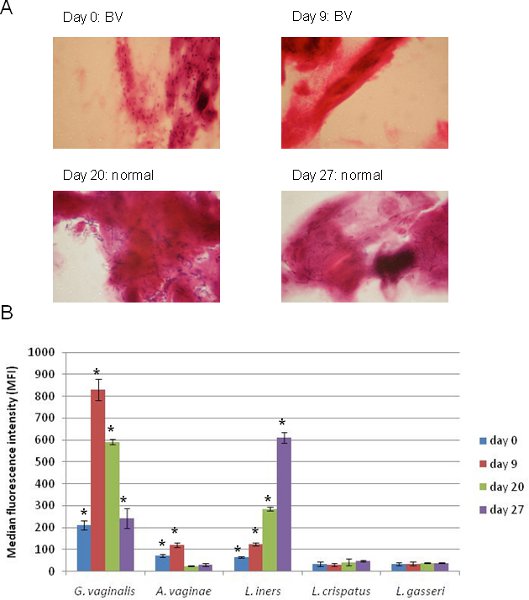
Figura 4. Aplicación de un conjunto complejo de 5 Luminex para el diagnóstico BV en muestras secuenciales tomadas de la misma persona. (A). Tinción de Gram diapositivas que muestran los tradicionales de diagnóstico utilizado para evaluar cada muestra para la vaginosis bacteriana. (B). Aplicación de una serie de 5 complejos Luminex preparado y ejecutado tal como se describe en este protocolo para las mismas cuatro muestras se muestra en (A). Objetivos bacteriana cuya IMF señal fue significativamente positivo según nuestra definición (de una cola de la t-test, p <0,05) se señalan con un asterisco (*).
Discusión
La especificidad de la generación de la señal es de suma importancia, usted debe estar seguro de que la señal observada refleja realmente la detección de amplificación generados a partir de ese organismo. Software como PrimerPlex (Premier Biosoft) pueden ayudar a diseñar las sondas que se hibridan de manera eficiente, pero puede o no hibridizan a las especies no objetivo. Cuando universal de PCR se utilizan como se describe en este protocolo, es importante tener en cuenta que la amplificación generada representa ...
Divulgaciones
No hay conflictos de interés declarado.
Agradecimientos
Agradecemos a Alberto Severini y Goleski Vanessa para ayudar con el desarrollo de ensayos y comentarios críticos sobre este manuscrito. Este trabajo fue financiado por la Agencia de Salud Pública de Canadá y el Programa de Asistencia a la Investigación Industrial (National Research Council de Canadá). El apoyo adicional fue obtenida de la Universidad de Saskatchewan Fondo de Publicación.
Materiales
| Name | Company | Catalog Number | Comments |
| Nombre de oligonucleótidos | Empresa | Secuencia de un | |
| H279BP | Invitrogen, IDT, u otros | Biotina-OEFO GAIIIIGCIGGIGAYGGIACIACIAC | |
| H280 | YKIYKITCICCRAAICCIGGIGCYTT | ||
| H1612BP | Biotina-OEFO GAIIIIGCIGGYGACGGYACSACSAC | ||
| H1613 | CGRCGRTCRCCGAAGCCSGGIGCCTT | ||
| 1O, fosforotioato-C, E, G-fosforotioato; F, fosforotioato-A, I, inosina, Y, C y T, R, A o G, K, T, G, S, C o G. | |||
Tabla 1. Secuencias de los oligonucleótidos modificados para Universal cpn60 PCR.
| Nombre del reactivo | Empresa | Número de catálogo | Comentarios |
| 1-etil-3-(3-dimethylamiopropyl) carbodiimida HCl (EDC) | Atravesar | 22980 | |
| Bolas fluorescentes de poliestireno: Microplex microesferas (Luminex), o Bio-Plex COOH bolas (Bio-Rad) | Luminex o Bio-Rad | Bio-Rad: 171-506xxx donde xxx corresponde a la identificación de cuentas | Partículas magnéticas se están haciendo disponibles para el acoplamiento de oligonucleótidos y pueden ofrecer ciertas ventajas. No han sido juzgados por estos autores. |
| Oligonucleótidos de captura (5 'amino modificado C12) | Invitrogen, IDT, u otros | diferentes | Nivel de pureza desalada es aceptable. Secuencias de las sondas de captura utilizado para caracterizar muestras vaginales se proporcionan en el manuscrito en el que se basa este protocolo 13. |
| T7exonucleasa | New England Biolabs | M0263S | |
| Estreptavidina-R-ficoeritrina (SA-PE) | Invitrogen | S-866 | Tenga cuidado para obtener alta pureza SA-PE, este número de catálogo se recomienda |
| Vaina 96 y placas de PCR | Pescador | CS006509 | encajaban en ambas termociclador de 96 pozos y la máquina BioPlex |
| Vaina de sellado alfombra | Pescador | CS006555 | Puede ser reutilizado, lavar con agua jabonosa, enjuague bien y seque |
| TMAC 5M | Sigma | T3411 | |
| Bio-Plex o instrumento Luminex | Bio-Rad o Luminex | Bio-Rad: 171-000201 | |
| PrimerPlex software de diseño de la sonda | Premier Biosoft | www.premierbiosoft.com | Sugerido para la sonda de Luminexsigno, aunque otras plataformas de software se puede utilizar |
Tabla 2. Reactivos y equipos específicos.
| componente | l / ensayo | ensayos μl/100 | concentración final |
| 10x PCR buffer (Invitrogen) | 5 | 500 | 1x |
| 50 mM MgCl 2 (Invitrogen) | 2.5 | 250 | 2,5 mM |
| 10 mM dNTP | 1 | 100 | 0,2 mM de cada uno |
| H279BP, 25 M | 0.25 | 25 | 375 nM |
| H1612BP, 25 M | 0.75 | 75 | 125 nM |
| H280, 25 M | 0.25 | 25 | 375 nM |
| H1613,25 M | 0.75 | 75 | 125 nM |
| De agua | 34 | 3400 | --- |
| Totales | 44.5 | 4450 |
Tabla 3. Sugerido mezclas de PCR con primers modificados cpn60 UT (Tabla 1). El ensayo está programado para 5 l de ADN y 0,5 l (2.5U) de Taq ADN polimerasa (Invitrogen). Normalmente se preparan grandes volúmenes (por ejemplo, suficiente para 100 ensayos) y almacenados a -20 ° C.
Referencias
- Hale, L. P., Swidsinski, A., Mendling, W. Bacteria associated with bacterial vaginosis. N. Engl. J. Med. 354, 202-203 (2006).
- Hay, P. Life in the littoral zone: lactobacilli losing the plot. Sex. Transm. Infect. 81, 100-102 (2005).
- Morris, M., Nicoll, A., Simms, I., Wilson, J., Catchpole, M. Bacterial vaginosis: a public health review. Brit. J. Obstet. Gynecol. 108, 439-450 (2001).
- Myer, L., Kuhn, L., Stein, Z. A., Wright, T. C., Denny, L. Intravaginal practices, bacterial vaginosis, and women's susceptibility to HIV infection: epidemiological evidence and biological mechanisms. Lancet. Infect. Dis. 5, 786-794 (2005).
- Wilson, J. Managing recurrent bacterial vaginosis. Sex. Transm. Infect. 80, 8-11 (2004).
- Nugent, R. P., Krohn, M. A., Hillier, S. L. Reliability of diagnosing bacterial vaginosis is improved by a standardized method of gram stain interpretation. J. Clin. Microbiol. 29, 297-301 (1991).
- Ison, C. A., Hay, P. E. Validation of a simplified grading of Gram stained vaginal smears for use in genitourinary medicine clinics. Sex. Transm. Infect. 78, 413-415 (2002).
- Money, D. The laboratory diagnosis of bacterial vaginosis. Can. J. Infect. Dis. Med. Microbiol. 16, 77-79 (2005).
- Schellenberg, J. Pyrosequencing of the chaperonin-60 universal target as a tool for determining microbial community composition. Appl. Environ. Microbiol. 75, 2889-2898 (2009).
- Spear, G. T. Comparison of the diversity of the vaginal microbiota in HIV-infected and HIV-uninfected women with or without bacterial vaginosis. J. Infect. Dis. 198, 1131-1140 (2008).
- Brotman, R. M., Ravel, J. Ready or not: the molecular diagnosis of bacterial vaginosis. Clin. Infect. Dis. 47, 44-46 (2008).
- Fredricks, D. N., Fiedler, T. L., Marrazzo, J. M. Molecular identification of bacteria associated with bacterial vaginosis. N. Engl. J. Med. 353, 1899-1911 (2005).
- Dumonceaux, T. J. Multiplex detection of bacteria associated with normal microbiota and with bacterial vaginosis in vaginal swabs by use of oligonucleotide-coupled fluorescent microspheres. J. Clin. Microbiol. 47, 4067-4077 (2009).
- Molenkamp, R., van der Ham, A., Schinkel, J., Beld, M. Simultaneous detection of five different DNA targets by real-time Taqman PCR using the Roche LightCycler480: Application in viral molecular diagnostics. J. Virol. Meth. 141, 205-211 (2007).
- Nikiforov, T. T., Rendle, R. B., Kotewicz, M. L., Rogers, Y. H. The use of phosphorothioate primers and exonuclease hydrolysis for the preparation of single-stranded PCR products and their detection by solid-phase hybridization. PCR. Methods. Appl. 3, 285-291 (1994).
- Hill, J. E., Penny, S. L., Crowell, K. G., Goh, S. H., Hemmingsen, S. M. cpnDB: A Chaperonin Sequence Database. Genome. Res. 14, 1669-1675 (2004).
- Bradshaw, C. S. The association of Atopobium vaginae and Gardnerella vaginalis with bacterial vaginosis and recurrence after oral metronidazole therapy. J. Infect. Dis. 194, 828-836 (2006).
- Dumonceaux, T. J. Enumeration of specific bacterial populations in complex intestinal communities using quantitative PCR based on the chaperonin-60 target. J. Microbiol. Meth. 64, 46-62 (2006).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados