Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Chromosomics: Detección de alteraciones numéricas y estructurales en todos los 24 cromosomas humanos de manera simultánea utilizando un nuevo ensayo FISH OctoChrome
En este artículo
Resumen
Una novela de fluorescencia In situ (FISH), método que al mismo tiempo examina alteraciones cromosómicas numéricas y estructurales, en particular las translocaciones cromosómicas específicas asociadas con la leucemia y el linfoma, de los 24 cromosomas humanos en un solo dispositivo en una hibridación, se describe.
Resumen
Hibridación in situ fluorescente (FISH) es una técnica que permite secuencias específicas de ADN para detectar los cromosomas completos o interfase en el núcleo celular 1. La técnica utiliza sondas de ADN con secuencias únicas que se hibridan con cromosomas enteros o regiones cromosómicas específicas, y sirve como un complemento útil para la citogenética clásica. Por ejemplo, muchos estudios anteriores reportaron la detección de frecuencia de aberraciones cromosómicas aumentaron en los pacientes con leucemia relacionados con la exposición al benceno, los pacientes intoxicación por benceno-, y los trabajadores sanos expuestos al benceno, utilizando el análisis citogenético clásico 2. Mediante FISH, la leucemia, alteraciones cromosómicas específicas se han observado a ser elevados en los trabajadores aparentemente sanos expuestos al benceno 3-6, lo que indica las funciones esenciales de los cambios en el benceno cytogentic inducida leucemogénesis.
Generalmente, una prueba FISH solo examina sólo uno o unos pocos cromosomas enteroso loci específicos por lámina, por lo que múltiples hibridaciones deben llevarse a cabo en varias diapositivas para cubrir todos los cromosomas humanos. Cariotipo espectral (SKY) permite la visualización de todo el genoma al mismo tiempo, pero la necesidad de un software especial y los límites de su aplicación de equipos 7. A continuación, describimos una prueba FISH novela, OctoChrome-FISH, que puede ser aplicado para Chromosomics, que se define aquí como el análisis simultáneo de todos los 24 cromosomas humanos en una diapositiva en estudios en humanos, tales como el estudio de la aneuploidía del cromosoma de ancho (CWA) 8. La base del método, comercializado por Cytocell como el Sistema de Chromoprobe Multiprobe, es un dispositivo OctoChrome que está dividido en 8 casillas, cada una de las cuales lleva tres diferentes sondas enteros pintura cromosoma (Figura 1). Cada una de las tres sondas está directamente etiquetada con un fluoróforo diferente color, verde (FITC), rojo (Texas Red), y azul (cumarina). La disposición de combinaciones cromosómicas en el OctoChromdispositivo electrónico ha sido diseñado para facilitar la identificación de las alteraciones cromosómicas no aleatorias estructurales (translocaciones) que se encuentran en las leucemias y linfomas más comunes, por ejemplo, t (9, 22), t (15; 17), t (8; 21 ), t (14; 18) 9. Por otra parte, los cambios numéricos (aneuploidía) en los cromosomas se pueden detectar al mismo tiempo. La diapositiva plantilla correspondiente también se divide en 8 cuadrados sobre la que se propaga en metafase están enlazados (Figura 2), y está colocado sobre el dispositivo OctoChrome. Las sondas y el ADN diana son desnaturalizado a alta temperatura y se hibridó en una cámara húmeda, y entonces todos los 24 cromosomas humanos pueden ser visualizadas simultáneamente.
PESCADO OctoChrome es una técnica prometedora para el diagnóstico clínico de la leucemia y el linfoma y para la detección de aneuploidías en todos los cromosomas. Hemos aplicado este enfoque nuevo cromosómica en un estudio de CWA de benceno en trabajadores expuestos chinos 8,10.
Protocolo
1. Preparación de la muestra de diapositivas
- PESCADO OctoChrome está diseñado para examinar los cambios cromosómicos en las metafases de las células humanas, incluyendo pero no limitado a cultivos de células de sangre periférica, células madre / progenitoras y líneas celulares. Las muestras se prepararon siguiendo el procedimiento estándar para la recolección de metafases 3,11-13: mediante el empleo de un tratamiento colcemid para detener las células en la metafase, el tratamiento hipotónico de las células se hinchan, y Carnoy para fijar las células en suspensión a aproximadamente 0,5-1 x 10 6 células por ml.
- Limpie la diapositiva 8-cuadrado (siempre en el Kit de OctoChrome, Catálogo #: PMP 803, Cytocell Ltd, Cambridge, Reino Unido, figura 2) por inmersión en etanol puro durante 2 min.
- Antes de hacer la diapositiva muestra, deje caer una pequeña cantidad de células en un portaobjetos para comprobar la densidad celular. Si la densidad de las células es demasiado alta (celdas superpuestas demasiados), diluir la suspensión con fijador. Si la densidad celular es demasiado baja (celda demasiado pocoss en la diapositiva), la desaceleración de las células y vuelva a suspender en un menor volumen de fijador. Pipetear 5-10 l de suspensión celular en cada uno de 8 áreas de la diapositiva de plantilla en una secuencia de cuadrados alternados 1/7 - 2/8 - 3/5 - 4/6. Una vez que los primeros dos cuadrados tiene secada al aire, manchar las plazas restantes en la misma manera. Esto evitará que los spreads de células de interferir unos con otros.
- La diapositiva seco se puede almacenar a -20 ° C en atmósfera de nitrógeno durante mayor que 5 años antes de la hibridación.
2. La localización de metafases utilizando un sistema automatizado
- Aplique 20 l de 4 ',6-diamino-2-fenilindol (0,2 g / ml) en el centro de cada mitad de la diapositiva y luego cubra con un cubreobjetos.
- Realice un escaneo automatizado de la diapositiva muestra a localizar las células en metafase utilizando Metafer de software (MetaSystems, Altlußheim, Alemania). Esto facilita la localización de todas las metafases y el futuro de re-evaluación de todas las imágenes de células.
- Ver los resultados de la exploración manual y eliminar no metafase artefactos.
3. Hibridación
- Preparación de la diapositiva muestra y dispositivo OctoChrome
- Sumergir el portaobjetos de la muestra en tampón 2x SSC a temperatura ambiente durante 30 minutos para lavar DAPI.
- Deshidratar la diapositiva muestra a través de una serie de lavados de etanol (2 minutos cada uno en el 70%, 85% y 100% de etanol), dejar secar y colocar en una placa 37 ° C durante 5 minutos.
- Coloque la cámara de hibridación Chromoprobe Multiprobe (proporcionado en el kit de OctoChrome) en un baño de agua a 37 ° C y deje que se equilibre a 37 ° C (+ / - 1 ° C).
- Mezclar la solución de hibridación (proporcionado en el kit de OctoChrome) mediante pipeteo repetido y precalentar una alícuota de 25 l por dispositivo OctoChrome a 37 ° C.
- Pre-caliente cada dispositivo OctoChrome (proporcionado en el kit de OctoChrome, ver Figura 2) a 37 ° C mediante la colocación de la etiqueta del dispositivo hacia abajo sobre una placa calefactora. NO toque tél superficies en relieve del dispositivo OctoChrome ya que contienen las sondas.
- Posicionamiento de la diapositiva muestra sobre el dispositivo OctoChrome (Figura 2)
- Añadir 2 l de solución de hibridación pre-calentado a cada una de las ocho zonas en el dispositivo OctoChrome pre-calentado mientras permanece a 37 ° C.
- Invierta cuidadosamente los portaobjetos plantilla sobre el dispositivo OctoChrome de tal manera que el número 1, que ahora es al revés, se encuentra sobre el área superior derecha del dispositivo OctoChrome. Para ayudar a localizar una plaza, su posición en el dispositivo se ha marcado en naranja.
- Asegúrese de que el porta está cuidadosamente alineado con las áreas correspondientes en el dispositivo OctoChrome. Baje con cuidado la diapositiva sobre el dispositivo OctoChrome de modo que las gotas de solución de hibridación hacer contacto con la diapositiva. Aplicar suave, incluso presionar para asegurar que la solución de hibridación se extendió a los bordes de cada una de las áreas elevadas en el dispositivo OctoChrome.
- Levantarla diapositiva / OctoChrome, sosteniendo cuidadosamente el extremo esmerilado de la lámina de vidrio, e invertir para que la diapositiva está por debajo del dispositivo OctoChrome. Asegúrese de que el dispositivo no roza con el porta ya que esto podría causar una contaminación cruzada de las sondas.
- Coloque en un 37 ° C (+ / - 1 ° C) placa durante 10 minutos.
- Desnaturalización
- Transferir el dispositivo de muestra de diapositivas / OctoChrome a la zona de cocción con especial cuidado para mantenerla nivelada. Asegúrese de que el deslizamiento de la muestra uniformemente en contacto con la placa calefactora.
- Desnaturalice en la placa caliente a 75 ° C (+ / - 1 ° C) durante 5 minutos.
- Hibridación
- Colocar la muestra de diapositivas / dispositivo OctoChrome en la Cámara Chromoprobe hibridación Multiprobe.
- Vuelva a colocar la tapa e introduzca la cámara en la 37 ° C (+ / - 1 ° C) baño de agua (no - de agitación) durante la noche. Tenga en cuenta: No selle la tapa de la cámara de hibridación, NO selle la tapa en el baño de agua, no se hibridan en unincubadora. Estos pasos son fundamentales para controlar la humedad.
- Hibridación post-lavados
- Preparación de las soluciones de lavado
Solución 1: Preparar un vaso de Coplin / Hellendahl que contenga 0,4 x SSC. Equilibre a 72 ° C (+ / - 1 ° C) en un baño de agua y ajustar el pH a 7,0.
Solución 2: Prepare un vaso de Coplin / Hellendahl contiene 2x SSC y 0,05% de Tween 20. Dejar reposar a temperatura ambiente. - Retire el dispositivo OctoChrome cuidado de la diapositiva y colocarla en la diapositiva en la solución 1 durante 2 minutos.
- Colocar el portaobjetos en la solución 2 durante 30 segundos.
4. Montaje y visualización de los resultados
- Aplique 20 l de DAPI para el centro de cada mitad de la diapositiva y aplicar un cubreobjetos.
- Incubar en la oscuridad durante 10 minutos a temperatura ambiente antes de visualizar por microscopía de fluorescencia.
5. Los resultados representativos
Figude re 3A muestra una célula en metafase normal en la Plaza 2. (1) - (3): Los cromosomas 8, 21 y 12 estaban pintadas de rojo, verde y azul, y se visualizan a través de una Red de Texas, FITC, y el filtro de DEAC, respectivamente, (4): Visualización a través de una DAPI / FITC / Texas Red de triple filtro. En general, los cromosomas pintados con azul no son claras a través de la triple filtro y la necesidad de ser visto bajo el filtro específico DEAC.
La Figura 3B muestra representativas células anormales con leucemia específica translocación cromosómica y aneuploidía. (1): t (8; 21), una translocación cromosómica común en la leucemia mieloide aguda, (2): Trisomía 21, tres copias del cromosoma 21, una aneuploidía común en la leucemia.

Figura 1. Disposición de combinaciones cromosómicas en el dispositivo y los resultados esperados OctoChrome pintura cromosómicas.

Figura 2. Colocación de la muestra de diapositivas sobre el dispositivo OctoChrome.
Figura 3. Los resultados representativosobtenido de pescado OctoChrome (Plaza 2).
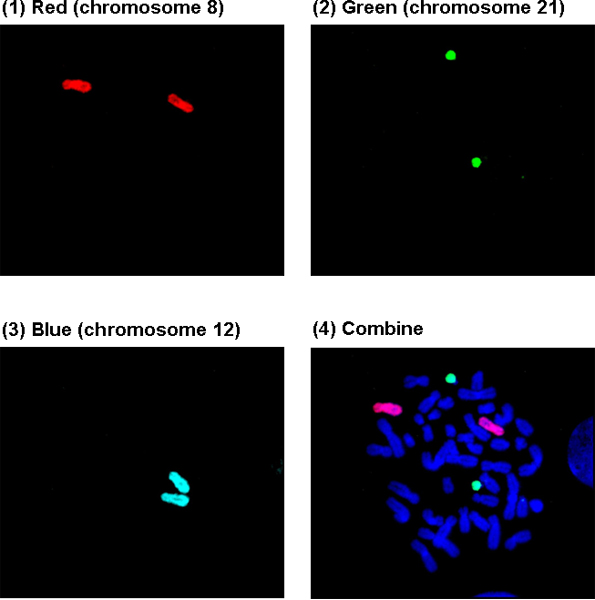
La figura 3A. Una célula normal en los cromosomas 8, 12, 21 pintadas en la plaza de 2.

Figura 3B. El representante de las células anormales con leucemia específica translocación cromosómica y aneuploidía en la plaza de 2.
Discusión
Esta novela OctoChrome-FISH ensayo permite examinar de manera simultánea en los 24 cromosomas humanos, en una hibridación única en una diapositiva. La disposición de combinaciones cromosómicas en los 8 cuadrados ha sido diseñado para facilitar la identificación de los reordenamientos cromosómicos no aleatorios en las leucemias más comunes y los linfomas. Así, el ensayo puede detectar numérica (aneuploidía), así como estructurales (translocaciones) alteraciones cromosómicas concurrentemente.
Divulgaciones
No tenemos nada que revelar.
Agradecimientos
Los autores desean agradecer a la Dra. M. Cliona McHale para la lectura crítica y edición del manuscrito. Este trabajo fue financiado por el Instituto Nacional de Ciencias de Salud Ambiental, Instituto Nacional de Salud de subvención R01ES017452 a L Zhang.
Materiales
| Name | Company | Catalog Number | Comments |
| Nombre del reactivo | Empresa | Número de catálogo | Comentarios (opcional) |
| OctoChrome Kit | Cytocell Ltd, Cambridge, Reino Unido | PMP 803 | |
| fitohemaglutinina (PHA) | Invitrogen Corporation | 10576-015 | |
| Colcemid | Invitrogen Corporation | 15212-012 | |
| Fijador de Carnoy | Metanol: ácido acético glacial = 3: 1 | ||
| Fluorescencia millascopio | Equipado con filtros para ver DAPI, Texas Red, FITC, y el espectro de cumarina de forma individual y una DAPI / FITC / Texas Red de triple filtro para ver los diferentes colores de forma simultánea | ||
| Metafer de software | MetaSystems, Altlußheim, Alemania | Facilitar para localizar todas las metafases y de volver a evaluar las anormalidades |
Referencias
- Levsky, J. M., Singer, R. H. Fluorescence in situ hybridization: past, present and future. J. Cell. Sci. 116, 2833-2838 (2003).
- Zhang, L., Eastmond, D. A., Smith, M. T. The nature of chromosomal aberrations detected in humans exposed to benzene. Crit. Rev. Toxicol. 32, 1-42 (2002).
- Zhang, L. Aberrations in chromosomes associated with lymphoma and therapy-related leukemia in benzene-exposed workers. Environ. Mol. Mutagen. 48, 467-474 (2007).
- Zhang, L. Benzene increases aneuploidy in the lymphocytes of exposed workers: a comparison of data obtained by fluorescence in situ hybridization in interphase and metaphase. Environ. Mol. Mutagen. 34, 260-268 (1999).
- Zhang, L. Increased aneusomy and long arm deletion of chromosomes 5 and 7 in the lymphocytes of Chinese workers exposed to benzene. Carcinogenesis. 19, 1955-1961 (1998).
- Smith, M. T. Increased translocations and aneusomy in chromosomes 8 and 21 among workers exposed to benzene. Cancer. Res. 58, 2176-2181 (1998).
- Bayani, J., Squire, J. A. Advances in the detection of chromosomal aberrations using spectral karyotyping. Clin. Genet. 59, 65-73 (2001).
- Zhang, L. Chromosome-wide aneuploidy study (CWAS) in workers exposed to an established leukemogen, benzene. Carcinogenesis. 32, 605-612 (2011).
- Rowley, J. D. The role of chromosome translocations in leukemogenesis. Semin. Hematol. 36, 59-72 (1999).
- Zhang, L. Use of OctoChrome fluorescence in situ hybridization to detect specific aneuploidy among all 24 chromosomes in benzene-exposed workers. Chem. Biol. Interact. , 153-154 (2005).
- Barch, M. J., Lawce, H. J., Arsham, M. S., Barch, M. J. . In The ACT cytogenetics laboratory. , 17-30 (1991).
- Zhang, L., Yang, W., Hubbard, A. E., Smith, M. T. Nonrandom aneuploidy of chromosomes 1, 5, 6, 7, 8, 9, 11, 12, and 21 induced by the benzene metabolites hydroquinone and benzenetriol. Environ. Mol. Mutagen. 45, 388-396 (2005).
- Smith, M. T. Hydroquinone, a benzene metabolite, increases the level of aneusomy of chromosomes 7 and 8 in human CD34-positive blood progenitor cells. Carcinogenesis. 21, 1485-1490 (2000).
- Smith, M. T. Advances in understanding benzene health effects and susceptibility. Annu. Rev. Public. Health. 31, 133-148 (2010).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados