Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Chromosomics: Erkennung von numerischen und strukturellen Veränderungen in alle 24 menschlichen Chromosomen gleichzeitig unter Verwendung eines neuartigen OctoChrome FISH-Test
In diesem Artikel
Zusammenfassung
Ein neuartiger Fluoreszenz- In-situ- Hybridisierung (FISH)-Methode, die gleichzeitig untersucht sowohl numerische und strukturelle Chromosomen-Veränderungen, insbesondere die spezifische chromosomale Translokationen mit Leukämien und Lymphomen assoziiert, der alle 24 menschlichen Chromosomen auf einem einzigen Gerät in einer Hybridisierung wird beschrieben.
Zusammenfassung
Fluoreszenz in situ Hybridisierung (FISH) ist eine Technik, die spezifische DNA-Sequenzen auf Metaphase oder Interphase-Chromosomen in Zelle Kerne 1 nachgewiesen werden können. Die Technik verwendet DNA-Sonden mit einzigartigen Sequenzen, die auf ganzer Chromosomen oder spezifische chromosomale Regionen hybridisieren, und dient als leistungsfähige Ergänzung zur klassischen Zytogenetik. So berichteten viele frühere Studien der häufige Nachweis von erhöhten Chromosomenaberrationen bei Leukämie-Patienten mit Benzol-Exposition, Benzol-Vergiftung Patienten und gesunde Arbeitnehmer, die Benzol verwandt mit klassischen zytogenetischen Analyse 2. Mit FISH, haben Leukämie-spezifische chromosomale Veränderungen beobachtet worden, um in scheinbar gesunden Arbeitnehmer, die Benzol 3-6 erhöht sein, was auf die kritische Rolle von cytogentic Veränderungen in Benzol-induzierten Leukämogenese.
Generell prüft ein einziger FISH-Test nur eine oder wenige ganze Chromosomenoder spezifische Loci pro Objektträger, so müssen mehrere Hybridisierungen auf mehrere Folien durchgeführt werden, um alle menschlichen Chromosomen bedecken. Spektrale Karyotypisierung (SKY) ermöglicht die Visualisierung des gesamten Genoms gleichzeitig, aber die Forderung nach einer speziellen Software und Ausrüstung Grenzen ihrer Anwendung 7. Hier beschreiben wir eine neuartige FISH-Test, OctoChrome-Fisch, der für Chromosomics angewendet werden können, die wir hier definieren, wie die gleichzeitige Analyse aller 24 menschlichen Chromosomen auf einer Folie in Studien am Menschen, wie Chromosom-weiten Studie Aneuploidie (CWA) 8. Die Grundlage des Verfahrens, von Cytocell als Chromoprobe Multiprobe System vermarktet, ist ein OctoChrome Gerät, das in 8 Quadrate, von denen jede drei verschiedenen ganzen Chromosomen Sonden (Abbildung 1) mit sich führt wird. Jede der drei Sonden wird direkt mit einem anderen farbigen Fluorophor, grün (FITC), rote (Texas Red) und Blau (Cumarin) markiert. Die Anordnung von Chromosom Kombinationen auf dem OctoChromE-Gerät wurde entwickelt, um die Identifizierung der nicht-zufällige strukturelle Chromosomenanomalie, Veränderungen (Translokationen) in den gängigsten Leukämien und Lymphomen gefunden zu erleichtern, z. B. t (9; 22), t (15; 17), t (8; 21 ), t (14; 18) 9. Darüber hinaus können numerische Veränderungen (Aneuploidie) bei gleichzeitig Chromosomen erkannt werden. Die entsprechende Matrizenobjektträger wird auch in 8-Quadraten auf denen Metaphasespreitungen gebunden ist (Abbildung 2) sind, und wird über den OctoChrome Vorrichtung positioniert unterteilt. Die Sonden und Ziel-DNA sind bei hoher Temperatur und hybridisiert in einer feuchten Kammer denaturiert, und dann alle 24 menschlichen Chromosomen können visualisiert.
OctoChrome FISH ist eine vielversprechende Technik für die klinische Diagnostik von Leukämien und Lymphomen und zur Detektion von Aneuploidien in allen Chromosomen. Wir haben dieses neue chromosomale Ansatz in einer Studie CWAs von Benzol-exponierten chinesischen Arbeitern 8,10 angewandt.
Protokoll
1. Musterfolie Vorbereitung
- OctoChrome FISH wurde entwickelt, um chromosomale Veränderungen in Metaphasespreitungen menschlicher Zellen, einschließlich, aber nicht zu untersuchen, kultivierten peripheren Blutzellen, Stammzellen / Vorläuferzellen, und Zelllinien beschränkt. Die Proben sollten nach dem Standard-Verfahren zur Ernte von Metaphasen 3,11-13 vorbereitet werden: durch den Einsatz von Colcemid Behandlung, um Zellen in der Metaphase, hypotonische Behandlung zu verhaften, um Zellen anschwellen und Carnoys Fixativ zu fixieren Zellen in Suspension bei ca. 0,5 bis 1 x 10 6 Zellen pro ml.
- Reinigen Sie den 8-eckigen Schieber (sofern in der OctoChrome Kit, Bestellnummer: PMP-803, Cytocell Ltd, Cambridge, UK, Abbildung 2) durch Eintauchen in reines Ethanol für 2 min.
- Bevor die Probe Rutsche, fallen eine kleine Menge von Zellen auf einem Objektträger, um die Zelldichte zu überprüfen. Wenn die Zelldichte zu hoch ist (zu viele sich überschneidende-Zellen), verdünnen Sie die Suspension mit frischem Fixativ. Wenn die Zelldichte zu niedrig ist (zu wenige Zellens auf Folie), Spin-down und die Zellen in einem kleineren Volumen an frischem Fixativ resuspendieren. Pipette 5-10 &mgr; l der Zellsuspension auf jeweils von 8 Bereiche der Vorlage Folie in einer Folge von alternierenden Plätzen 1/7 - 2/8 - 3/5 - 4/6. Wenn die ersten beiden Plätze hat an der Luft getrocknet, vor Ort die verbliebenen Quadrate in der gleichen Weise. Das wird die Zelle breitet sich von sich gegenseitig zu stören zu verhindern.
- Die getrocknete Folie kann bei -20 ° C in Stickstoffatmosphäre für mehr als 5 Jahre vor der Hybridisierung gespeichert werden.
2. Lokalisierung von Metaphasen mit einem automatisierten System
- Bewerben 20 ul 4 ',6-Diamidino-2-phenylindol (0,2 ug / ml) in die Mitte jeder Hälfte der Rutsche und dann mit einem Deckglas abdecken.
- Führen Sie einen automatisierten Scan der Probe Rutsche zu lokalisieren, die Metaphase-Zellen unter Verwendung Metafer Software (MetaSystems, Altlußheim, Deutschland). Dies erleichtert die Lage aller Metaphasen und zukünftige Neubewertung aller Zell-Bilder.
- Sehen Sie sich die Scan-Ergebnisse manuell zu löschen und nicht-Metaphase Artefakte.
3. Hybridisierung
- Vorbereitung der Probe Rutsche und OctoChrome Gerät
- Tauchen Sie die Probe Folie in 2x SSC-Puffer bei Raumtemperatur für 30 min weg zu waschen DAPI.
- Entwässern der Probe Rutsche durch eine Reihe von Ethanol wäscht (je 2 Minuten in 70%, 85% und 100% Ethanol), trocknen lassen und auf ein 37 ° C Kochplatte für 5 Minuten.
- Setzen Sie den Multiprobe Hybridisierungskammer (sofern in der OctoChrome Kit) in einem 37 ° C warmes Wasserbad und lassen auf 37 ° C (+ / - 1 ° C).
- Mischen Sie die Hybridisierungslösung (sofern in der OctoChrome Kit) durch wiederholtes Pipettieren und vorwärmen einem 25 ul Aliquot pro OctoChrome Gerät bis 37 ° C.
- Pre-warmen jeder OctoChrome Gerät (sofern in der OctoChrome Kit, siehe Abbildung 2) auf 37 ° C, indem das Gerät der beschrifteten Seite nach unten auf einer Heizplatte. Berühren Sie NICHT ter geprägten Oberflächen der OctoChrome Gerät, wie sie die Sonden enthalten.
- Positionierung der Probe einer Rutschpartie über den OctoChrome Gerät (Abbildung 2)
- In 2 ul vorgewärmten Hybridisierungslösung in jede der acht Bereiche auf dem vorgewärmten OctoChrome Gerät, während es bei 37 ° C bleibt
- Vorsichtiges Umdrehen der Vorlage einer Rutschpartie über den OctoChrome derart, dass die Zahl 1, die jetzt auf den Kopf, über der rechten oberen Bereich des OctoChrome Gerät befindet. Zum Auffinden Platz 1, hat seine Position auf dem Gerät in Orange markiert.
- Stellen Sie sicher, dass die Vorlage Dia sorgfältig mit den passenden Bereiche auf der OctoChrome Gerät ausgerichtet sind. Vorsichtig das Bild über den OctoChrome Gerät, so dass die Tropfen der Hybridisierungslösung in Kontakt mit der Folie. Sacht auflegen und gleichmäßig unter Druck zu setzen sicherzustellen, dass die Hybridisierung bis zu den Rändern jedes der erhabenen Flächen auf der OctoChrome Gerät ist zu verbreiten.
- Hebender Schieber / OctoChrome, sorgfältig Halten der bereiften Ende der Objektträger aus Glas, und schwenken, so dass der Schieber unterhalb des OctoChrome Gerät ist. Achten Sie darauf, das Gerät schmiert nicht über den Matrizenobjektträger da dies eine Kreuzkontamination der Sonden verursachen könnte.
- Auf ein 37 ° C (+ / - 1 ° C) Kochplatte für 10 Minuten.
- Denaturierung
- Übertragen Sie die Probe Dia / OctoChrome Gerät auf der Heizplatte und dabei besonders auf das Niveau zu halten. Stellen Sie sicher, dass der Probenobjektträger gleichmäßig in Kontakt mit der Heizplatte.
- Auf der Heizplatte denaturieren bei 75 ° C (+ / - 1 ° C) für 5 Minuten.
- Hybridisierung
- Ort Probenobjektträger / OctoChrome Gerät in der Multiprobe Hybridisierungskammer.
- Setzen Sie den Deckel auflegen und die Kammer in der 37 ° C (+ / - 1 ° C) Wasserbad (nicht - Rühren) über Nacht. Bitte beachten Sie: nicht verschließen den Deckel auf die Hybridisierung Kammer; nicht verschließen Sie den Deckel auf dem Wasserbad; NICHT in ein hybridisierenInkubator. Diese Schritte sind entscheidend für Steuern Luftfeuchtigkeit.
- Post-Hybridisierung Wäschen
- Vorbereitung der Waschlösungen
Lösung 1: Bereiten Sie eine Coplin / Hellendahl Gefäß mit 0,4 x SSC. Äquilibrieren bis 72 ° C (+ / - 1 ° C) in einem Wasserbad und der pH-Wert bis 7,0.
Lösung 2: Einen Coplin / Hellendahl Gefäß mit 2x SSC und 0,05% Tween 20. Zulassen, um bei Raumtemperatur stehen gelassen. - Entfernen Sie die OctoChrome Gerät vorsichtig aus der Folie und den Objektträger in Lösung 1 für 2 Minuten.
- Legen Sie den Objektträger in Lösung 2 für 30 Sekunden.
4. Aufhängungen und Visualisierung der Ergebnisse
- Bewerben 20 ul DAPI auf der Mitte jeder Hälfte des Dia-und Deckglas auflegen.
- Im Dunkeln für 10 Minuten bei Raumtemperatur, bevor Betrachten mit dem Fluoreszenzmikroskop.
5. Repräsentative Ergebnisse
FiguRe 3A zeigt eine normale Zelle in der Metaphase Square 2. (1) - (3): Chromosomen 8, 21 und 12 wurden rot lackiert, Grün und Blau, und sind über einen Texas-Rot, FITC, und DEAC Filter bzw. visualisiert werden, (4): Visualisierung durch eine DAPI / FITC / Texas Red Triple-Filter. Im Allgemeinen sind die Chromosomen mit blau lackierten nicht durch den Dreifach-Filter klar und müssen im Rahmen des spezifischen DEAC Filter betrachtet werden.
3B zeigt repräsentative abnormale Zellen mit Leukämie-spezifische chromosomale Translokation und Aneuploidie. (1): t (8; 21), eine gemeinsame chromosomale Translokation bei akuter myeloischer Leukämie; (2): Trisomie 21, drei Kopien des Chromosoms 21, eine gemeinsame Aneuploidie bei Leukämie.

Abbildung 1. Anordnung der Chromosomen-Kombinationen auf der OctoChrome Gerät und die erwarteten Ergebnisse chromosomale Malerei.

Abbildung 2. Positionierung der Probe einer Rutschpartie über den OctoChrome Gerät.
Abbildung 3. Repräsentative Ergebnisseerhalten aus OctoChrome FISH (Platz 2).
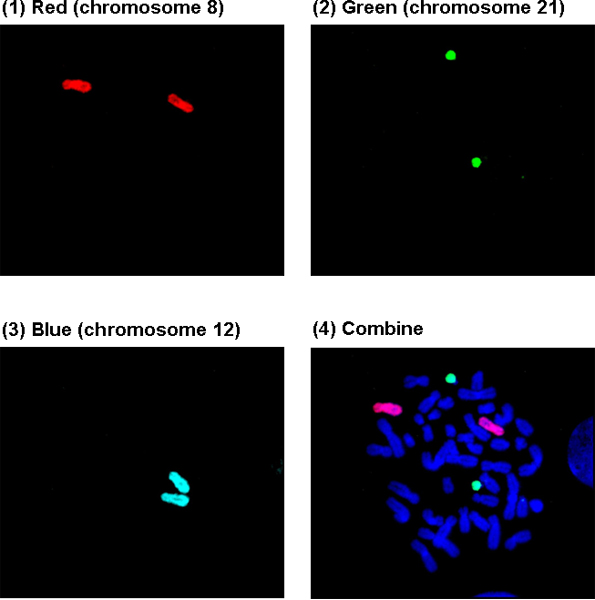
3A. Eine normale Zelle mit Chromosomen 8, 12, 21 in Square 2 gemalt.

3B. Vertreter abnormale Zellen mit Leukämie-spezifische chromosomale Translokation und Aneuploidie bei Square 2.
Diskussion
Dieser Roman OctoChrome-FISH-Test erlaubt es, gleichzeitig zu untersuchen alle 24 menschlichen Chromosomen in einer einzigen Hybridisierung auf einer Folie. Die Anordnung von Chromosom-Kombinationen auf den 8 Feldern wurde entwickelt, um die Identifizierung der nicht-zufällige Chromosomenumlagerungen in den gängigsten Leukämien und Lymphomen zu erleichtern. Somit kann der Assay erkennen numerischen (Aneuploidie) sowie strukturelle (Translokationen) Chromosomenveränderungen gleichzeitig.
...
Offenlegungen
Wir haben nichts zu offenbaren.
Danksagungen
Die Autoren danken Herrn Dr. M. Cliona McHale für die kritische Durchsicht und Bearbeitung des Manuskripts danken. Diese Arbeit wurde vom National Institute of Environmental Health Sciences, National Institute of Health Zuschuss zu R01ES017452 L Zhang finanziert.
Materialien
| Name | Company | Catalog Number | Comments |
| Name des Reagenzes | Firma | Katalog-Nummer | Kommentare (optional) |
| OctoChrome Kit | Cytocell Ltd, Cambridge, UK | PMP 803 | |
| Phytohämagglutinin (PHA) | Invitrogen Corporation | 10576-015 | |
| Colcemid | Invitrogen Corporation | 15212-012 | |
| Carnoys Fixativ | Methanol: Eisessig = 3: 1 | ||
| Fluoreszenz mimikroskop | Ausgerüstet mit Filter DAPI, Texas Red, FITC, und Coumarin-Spektren einzeln und eine DAPI / FITC / Texas Red Triple-Filter sehen, um unterschiedliche Farben gleichzeitig anzeigen | ||
| Metafer Software | MetaSystems, Altlußheim, Deutschland | Erleichtern, um alle Metaphasen finden und neu zu bewerten den Anomalien |
Referenzen
- Levsky, J. M., Singer, R. H. Fluorescence in situ hybridization: past, present and future. J. Cell. Sci. 116, 2833-2838 (2003).
- Zhang, L., Eastmond, D. A., Smith, M. T. The nature of chromosomal aberrations detected in humans exposed to benzene. Crit. Rev. Toxicol. 32, 1-42 (2002).
- Zhang, L. Aberrations in chromosomes associated with lymphoma and therapy-related leukemia in benzene-exposed workers. Environ. Mol. Mutagen. 48, 467-474 (2007).
- Zhang, L. Benzene increases aneuploidy in the lymphocytes of exposed workers: a comparison of data obtained by fluorescence in situ hybridization in interphase and metaphase. Environ. Mol. Mutagen. 34, 260-268 (1999).
- Zhang, L. Increased aneusomy and long arm deletion of chromosomes 5 and 7 in the lymphocytes of Chinese workers exposed to benzene. Carcinogenesis. 19, 1955-1961 (1998).
- Smith, M. T. Increased translocations and aneusomy in chromosomes 8 and 21 among workers exposed to benzene. Cancer. Res. 58, 2176-2181 (1998).
- Bayani, J., Squire, J. A. Advances in the detection of chromosomal aberrations using spectral karyotyping. Clin. Genet. 59, 65-73 (2001).
- Zhang, L. Chromosome-wide aneuploidy study (CWAS) in workers exposed to an established leukemogen, benzene. Carcinogenesis. 32, 605-612 (2011).
- Rowley, J. D. The role of chromosome translocations in leukemogenesis. Semin. Hematol. 36, 59-72 (1999).
- Zhang, L. Use of OctoChrome fluorescence in situ hybridization to detect specific aneuploidy among all 24 chromosomes in benzene-exposed workers. Chem. Biol. Interact. , 153-154 (2005).
- Barch, M. J., Lawce, H. J., Arsham, M. S., Barch, M. J. . In The ACT cytogenetics laboratory. , 17-30 (1991).
- Zhang, L., Yang, W., Hubbard, A. E., Smith, M. T. Nonrandom aneuploidy of chromosomes 1, 5, 6, 7, 8, 9, 11, 12, and 21 induced by the benzene metabolites hydroquinone and benzenetriol. Environ. Mol. Mutagen. 45, 388-396 (2005).
- Smith, M. T. Hydroquinone, a benzene metabolite, increases the level of aneusomy of chromosomes 7 and 8 in human CD34-positive blood progenitor cells. Carcinogenesis. 21, 1485-1490 (2000).
- Smith, M. T. Advances in understanding benzene health effects and susceptibility. Annu. Rev. Public. Health. 31, 133-148 (2010).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten