Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Optogenético La activación de las neuronas somatosensoriales pez cebra utilizando Chef-tdTomato
En este artículo
Resumen
Técnicas optogenético han hecho posible el estudio de la contribución de las neuronas específicas de comportamiento. Se describe un método en larvas de pez cebra para la activación de las neuronas somatosensoriales individuales que expresan una variante canalrodopsina (Chef) con un bombeado por diodos de estado sólido (DPSS) láser y el registro de los comportamientos provocados con una cámara de vídeo de alta velocidad.
Resumen
Larvas de pez cebra se están convirtiendo en un modelo para describir el desarrollo y la función de simples circuitos neuronales. Debido a su fertilización externa, el rápido desarrollo, y la translucidez, en particular el pez cebra se presta a los enfoques optogenético para investigar la función de circuitos neuronales. En este enfoque, sensibles a la luz los canales de iones se expresan en neuronas específicas, permitiendo que el experimentador para activar o inhibir a voluntad y por lo tanto evaluar su contribución a comportamientos específicos. La aplicación de estos métodos en larvas de pez cebra es conceptualmente simple, pero requiere la optimización de los detalles técnicos. Aquí se demuestra un procedimiento para la expresión de una variante en larvas de pez cebra canalrodopsina neuronas somatosensoriales, foto-activación de las células individuales, y el registro de los comportamientos resultantes. Mediante la introducción de algunas modificaciones a los métodos previamente establecidos, este enfoque podría ser utilizado para provocar respuestas de comportamiento de neuronas individuales activados hastaa por lo menos 4 días después de la fertilización-(DPF). En concreto, hemos creado un transgén utilizando una neurona somatosensorial potenciador, CREST3, para conducir la expresión de la variante etiquetados canalrodopsina, Chef-tdTomato. Inyectando este transgén en los embriones de 1-etapa de células resulta en una expresión mosaico en las neuronas somatosensoriales, que se pueden obtener imágenes de microscopía confocal. Illuminating identificado células en estos animales con la luz de un láser DPSS 473 nm, guiado a través de un cable de fibra óptica, provoca comportamientos que se pueden grabar con una cámara de vídeo de alta velocidad y analizados cuantitativamente. Esta técnica podría adaptarse a las conductas de estudio provocados por la activación de cualquier neurona pez cebra. La combinación de este método con perturbaciones genéticas o farmacológico será una manera poderosa para investigar la formación del circuito y la función.
Introducción
El desarrollo de métodos optogenética para promover o inhibir la excitabilidad neuronal con longitudes de onda definidas de luz ha permitido estudiar la función de las distintas poblaciones de neuronas en los circuitos neurales que controlan el comportamiento de 1, 19, 21. Esta técnica se utiliza a menudo para activar grupos de neuronas, pero también puede ser usado para activar las neuronas individuales. Larvas de pez cebra son particularmente susceptibles a estos métodos ya que son translúcidas, su sistema nervioso se desarrolla rápidamente, y la creación de animales transgénicos es rápido y de rutina. Sin embargo, importantes obstáculos técnicos se debe superar para lograr fiablemente activación única neurona.
Para optimizar un procedimiento para la activación de las neuronas optogenético pez cebra individuales, nos hemos centrado en las neuronas somatosensoriales. Larvas de pez cebra detectar una variedad de estímulos somatosensoriales utilizando dos poblaciones de neuronas: las neuronas del trigémino, que inervan la cabeza, y Rohon-(BeardRB), las neuronas que inervan el resto del cuerpo. Cada trigémino y RB neurona proyecta un axón periférico que se ramifica extensamente en la piel para detectar los estímulos y un axón central que se conecta aguas abajo a los circuitos neuronales. Los animales responden a tocar tan pronto como 21 horas después de la fertilización (hpf), lo que indica que los circuitos somatosensoriales coherentes han formado 5, 18. Durante el desarrollo larvario al menos algunas sinapsis las neuronas del trigémino y RB sobre la célula de Mauthner para activar respuestas clásicas de escape, pero la acumulación de evidencia sugiere que hay varias clases de neuronas somatosensoriales con diferentes patrones de conectividad que pueden provocar variaciones en el comportamiento de escape 2, 4, 10, 12, 14, 15, 16, 17. Nuestra motivación para el desarrollo de este método fue caracterizar la función de comportamiento de las diferentes clases de neuronas somatosensoriales, pero este enfoque podría en principio ser usado para estudiar la función de la neurona o casi cualquier población de neuronas en LARval pez cebra.
Douglass et al. Descrito previamente un método para activar canalrodopsina-2-expresando neuronas somatosensoriales con luz azul, provocando comportamiento de escape 3. Su enfoque utiliza un elemento potenciador del gen ISL1 para conducir la expresión de ChR2-EYFP en las neuronas somatosensoriales. Este transgén, sin embargo, se informó de que muestran una fluorescencia relativamente débil, lo que requiere la co-inyección de un segundo reportero, UAS :: GFP, para permitir la visualización de células que expresan ChR2-EYFP. Este enfoque se utilizó para obtener respuestas de comportamiento entre 24-48 HPF, pero nunca pudo obtener una respuesta más allá de 72 HPF. Por lo tanto, mientras que este método funciona para el estudio de los circuitos neuronales en las fases larvarias tempranas (24-48 hpf), es insuficiente para la caracterización de circuitos neurales y las respuestas de comportamiento en las larvas mayores, cuando haya más diversas respuestas de comportamiento son evidentes y circuitos neuronales son más maduros.
Se buscómejorar la sensibilidad de esta técnica con el fin de caracterizar la función de las subpoblaciones de neuronas RB larvales. Para mejorar la expresión se utilizó un potenciador somatosensorial-específico (CREST3) 20 para conducir la expresión de LexA-VP16 y un tramo de secuencias del operador LexA (4xLexAop) 11 para amplificar la expresión de un marcado fluorescentemente y activado por la luz de canal. Esta configuración amplificado expresión del canal, eliminando la necesidad de co-expresión de un segundo reportero y que nos permite determinar directamente la abundancia relativa de la canal en cada neurona. Usando la secuencia de LexA / LexAOp tenía la ventaja adicional de que permite introducir el transgén en líneas indicadoras de pez cebra que utilizan el sistema GAL4/UAS. La expresión transitoria de este transgén resultó en niveles variables de expresión, pero generalmente era lo suficientemente robusta como para visualizar tanto el cuerpo de la célula y las proyecciones axonales de las neuronas individuales en varios días. Para optimizar la sensibilidaddad a la luz se utilizó la luz activado canal Chef, una variante canalrodopsina que consiste en una quimera de channelopsin-1 (Chop1) y channelopsin-2 (Chop2) con un sitio de cruce en hélice bucle EF 13. Este canal se activa a la misma longitud de onda como ChR2, pero requiere una menor intensidad de luz para la activación, por lo que es más sensible que los otros canales de uso común, incluyendo ChR2. La proteína chef fue fusionado a la proteína fluorescente de color rojo, tdTomato, lo que nos permite cribar para la expresión de proteínas sin activar el canal. Como fuente de luz, se utilizó un diodo bombeado de estado sólido (DPSS) láser acoplado a un cable de fibra óptica para proporcionar una precisa, de alta potencia del pulso de luz azul a una región específica de las larvas. Esto nos permitió enfocar la luz láser sobre las neuronas individuales, eliminando la necesidad de encontrar raros animales transgénicos que expresan el canal en una sola neurona. El uso de este enfoque, hemos sido capaces de activar las neuronas individuales RB, registre la respuesta conductuals con una cámara de vídeo de alta velocidad, y la imagen de las neuronas activadas en alta resolución con microscopía confocal.
Protocolo
Preparar el siguiente antes de tiempo.
1. Prepare el cable óptico
- Crear una unidad de almacenamiento para el cable de fibra óptica por fusión del cuello cónico de una pipeta Pasteur de vidrio sobre un mechero de Bunsen para crear un ángulo de ~ 150 °.
- Usando un cortador de alambre o una hoja de afeitar, corte con cuidado el cable de fibra óptica en dos partes. Cada pieza debe tener un extremo con una FC / adaptador de PC y un extremo expuesto. La tienda de una sola pieza como un cable de reserva.
- Pelar el cable de fibra óptica hasta el revestimiento mediante la eliminación de la cubierta de la fibra y fibras de refuerzo (Figura 1A) de 5,0 cm de extremo cortado del cable.
- Inserte el cable óptico en preparados pipeta Pasteur. Asegúrese de cable puede moverse fácilmente dentro y fuera de la punta de la pipeta.
- Con cable de fibra óptica que sobresale de la punta de pipeta Pasteur, corte con cuidado y retire revestimiento de fibra de alrededor de la fibra de vidrio, ~ 2 mm del extremo del corte.
- El uso de un lápiz de diamante / cristal de corte, corte la fibra de vidrio y se rompen off al final para crear un corte limpio / superficie en el extremo de la fibra (Figura 1B).
- Retirar cable óptico en pipeta Pasteur para su almacenamiento. Repita el paso (1,6) si la punta de la fibra óptica se astilla o rompe de manera desigual.
- Cable óptico en posición pipeta Pasteur utilizando un micromanipulador (Figura 1C).
Procedimientos 2-8 describen un método para la inyección de transgenes en embriones generalmente aplicables a muchos experimentos de pez cebra. Variaciones sobre este método, como los descritos en otros vídeos Jové 6, 7, 8, 9, 22 son igualmente eficaces.
2. Tire de agujas de inyección
- El uso de un extractor de aguja, tire de tubos de vidrio borosilicato en dos agujas con punta afilada gradualmente. Extractor de configuración pueden variar. (En un extractor de aguja Sutter Instruments usamos valores: P = 500, Heat = 720, Pull = 50, velocidad = 70 y el Tiempo = 150). Cada tirador de aguja es diferente, así optimizar la configuración del extractor emempíricamente. Para obtener más agujas afiladas, aumente el calor y / o de extracción. Para agujas afiladas menos, aumentar el tiempo y / o Pull disminución.
- Agujas Almacenar en un contenedor seguro (es decir, una placa de Petri con una cinta enrollada, de un lado adhesivo).
3. Vierta Moldes de Inyección
- Derretir 0,5 g de agarosa en embrión de 30 ml / agua azul, hasta que agarosa se disuelva por completo.
- Vierta en la mitad inferior de una placa de Petri.
- Coloque molde rectangular con pozos de montaje (Figura 2) en agarosa, teniendo cuidado de limitar la formación de burbujas alrededor de los pozos.
- Permitir agarosa para solidificar.
- Retire el molde y llenar placa de Petri con azul / agua embrión.
- Guardar en posición vertical, llenado con agua limpia, a la temperatura ambiente para su uso el mismo día o a 4 ° C para su uso futuro.
4. Hacer la mezcla de ADN plasmídico para inyecciones
- Diluir plásmido de ADN a una concentración de 50 ng / ml con rojo fenol 1:10 en ddH 2 O. Paraejemplo:
1,0 ml ADN de plásmido (250 ng / ml) 0,5 ml rojo fenol 3,5 ml ddH 2 O - Mezcla de ADN se pueden mantener a temperatura ambiente si se utiliza inmediatamente o se almacenaron a 4 ° C durante varios días.
5. Configure parejas de apareamiento
Esto se debe hacer la noche antes de que usted planea hacer inyecciones.
- Llene los tanques de cría con agua de la instalación y el lugar peces machos y hembras juntos. Si las inyecciones no puede realizarse tan pronto como las luces se encienden en las instalaciones, pescado separado hombres y mujeres con un divisor.
6. Preparar para inyecciones (puede hacerse mientras se espera para embriones)
- Encienda el inyector de presión de perforación. Asegúrese de que el sistema se ajusta a pulso. Comience con la duración del impulso conjunto a 1.
- Encienda la válvula de aire yAjustar la presión del inyector de aproximadamente 20 psi.
- El uso de un microscopio de disección con el máximo aumento, romper la punta de la aguja con un fórceps o de póquer para crear una abertura ~ 2 micras (Figura 3, Movie 1).
- Rellenar con la mezcla de las agujas del ADN a través de: 1. La colocación de la aguja, lado de la punta hacia arriba, en la mezcla de DNA y dejando que la aguja de llenar por acción capilar o 2. Utilizando una punta de largo alcance para pipetear 1-2 l de la DNA mezclar directamente en la aguja.
- Lugar lleno aguja en un lugar seguro hasta que esté listo para inyectar.
7. Recoger embriones
- Después de las luces de las instalaciones se han encendido, quitar separadores (si corresponde). Recoge los embriones con un colador / tamiz pequeño y transferirlos a una placa de Petri con azul fresca / agua embrión.
8. Se inyecta embriones en la etapa de célula 1-2
- La transferencia de embriones recolectados para moldes de inyección utilizando una pipeta de plástico.
- El uso de un microscopio de disección, empuje suavemente embriones enpozos con fórceps o un póker romo (Figura 4, Movie 2).
- Coloque la aguja cargada en un micromanipulador y posición sobre embriones.
- Calibrar el volumen de inyección mediante el ajuste de la duración del pulso en incrementos de 1-paso hasta conseguir el volumen deseado (1 ~ nl). Esto podría ser calibrado con un micrómetro de portaobjetos, tal como se describe en algunos vídeos anteriores Jove 8, 22, pero para este experimento precisión no es necesaria, ya que una amplia gama de volúmenes de inyección se traducirá en una expresión adecuada.
- Inyectar 1 ~ nl ADN mezclar directamente en la célula y repita para todos los embriones. El ADN también se puede inyectar en la yema de huevo, pero esto tiende a ser menos eficiente (Figura 5, la película 3).
- Retire los embriones inyectados de molde con un chorro suave de azul / agua embrión.
- Tienda inyectado embriones a 28,5 ° C en la oscuridad.
- Retire fecundados, embriones dañados o muertos de forma periódica.
- Tratar a los embriones wiª PTU entre 18-24 HPF para evitar la pigmentación.
9. Pantalla para la expresión transgénica
- Manualmente dechorionate 24-48 HPF embriones utilizando fórceps.
- Anestesie larvas utilizando 0,02% tricaína.
- El uso de un microscopio de fluorescencia de disección, identificar las larvas con RB o expresión de neuronas del trigémino y transferirlos a un nuevo plato con dulce PTU azul / embrión agua. Los embriones con escasa expresión en las células fácilmente identificables son óptimas, pero las neuronas individuales se orientarán para la activación con un cable de fibra óptica para una amplia gama de la densidad de expresión es aceptable.
- Tienda de embriones a 28,5 ° C en la oscuridad hasta la etapa experimental deseado.
10. Monte larvas para experimentos de comportamiento
- Hacer un 1,5% de agarosa de baja fusión en ddH 2 O y guárdelo en un bloque de 42 ° C de calor para evitar que se solidifique.
- Uso de una pipeta Pasteur de vidrio, la transferencia de una de las larvas pre-seleccionados en una tinae del 1,5% de agarosa de baja fusión con tan poco azul / embrión agua como sea posible.
- Transferir las larvas en una gota de agarosa en una pequeña placa Petri.
- Bajo un microscopio de disección, larva posición dorsal hacia arriba.
- Cuando agarosa se ha solidificado, cortar la agarosa con una navaja fina (# 11 bisturí), dejando un trozo de agar alrededor de la larva completa.
- Haga dos cortes diagonales en ambos lados de la yema de huevo, tomar cuidado de no mellar la larva.
- Llene el área que rodea a la agarosa con embrión / agua azul.
- Tire de agarosa de distancia del tronco y la cola de la larva (Figura 6).
11. Preparar cámara de alta velocidad y software de imágenes
- Monte de alta velocidad de la cámara en la disección de alcance.
- Conecte la cámara al ordenador.
- Encienda el ordenador.
- Encienda cámara de alta velocidad.
- Abrir video / imágenes de software (Utilizamos software AOS imagen y se describen los procedimientos para su uso aquí, pero las imágenes de otrosoftware es igualmente aceptable).
- Ajuste la configuración de la cámara en consecuencia (es decir, 1.000 fotogramas por segundo (fps), el 50% de amortiguamiento gatillo u otros ajustes preferidos).
- Inicie la grabación.
12. Activa las neuronas individuales utilizando un láser 473 nm
- Conecte estimulador, láser y cable óptico.
- Encienda el estimulador.
- Establecer estimulador a un máximo de 5 voltios y una duración de pulso de 5 ms.
- Activar el láser de acuerdo con las instrucciones del fabricante.
- El uso de un microscopio de disección para colocar la punta del cable óptico cerca del cuerpo celular de una neurona con Chef-tdTomato expresión (Figura 6).
- Entregar pulso de luz azul para activar la neurona sensorial.
- Comportamiento de grabación con una cámara de alta velocidad en 500 o 1.000 imágenes por segundo.
- Repetir el experimento como se desee, a la espera 1 min entre cada activación para evitar la habituación. (Hemos registrado un mínimo de tres respuestas para cada neurona).
- Alarvas de liberación, separarlas de agarosa con pinzas, cuidando de no lastimar al animal. Este animal se puede permitir seguir desarrollando y el procedimiento se puede repetir en una etapa anterior para caracterizar el desarrollo del comportamiento. El embrión también puede volver a montar de alta resolución de imagen confocal de células activadas por la que se correlacionan con el comportamiento de la estructura celular, como se describe a continuación.
- Traslado larva a la placa de cultivo con azul fresca / agua embrión. Utilizamos una placa de 24 pocillos para realizar un seguimiento de las larvas individual.
13. Imagen Neuron (s) con un microscopio confocal
- Anestesie larvas con 0,02% tricaína.
- Larvas Mount, lado dorsal hacia arriba, en agarosa al 1,2% bajo punto de fusión o en un molde dorsal.
- Imagen Chef-tdTomato neuronas con un láser nm 543 y filtro apropiado y objetivo. Nosotros utilizamos un objetivo de 20x.
- Eliminar las larvas de agarosa y volver al bienestar individual con el azul / agua embrión.
- Almacenar a 28,5 ° C en la oscuridad durante further análisis.
Resultados

Figura 1. Cable óptico configurado. (A) Capas de un cable de fibra óptica. (B) de fibra óptica por cable pelado en una pipeta Pasteur. (C) El cable de fibra óptica en pipeta Pasteur coloca utilizando un micromanipulador.
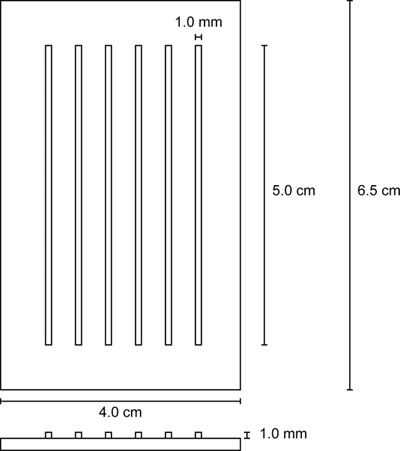
Figura 2. Inyección plantil...
Discusión
Hemos descrito un enfoque para la activación de las neuronas optogenético RB individuales en el pez cebra en vivo. Nuestro método emplea transgénesis transitorio para expresar una variante fluorescente etiquetados canalrodopsina, Chef-tdTomato 13, en determinadas neuronas somatosensoriales. Este enfoque puede ser fácilmente adaptado para el uso en otras poblaciones de larvas de pez cebra celulares.
Usando este enfoque consistente suscitó respuestas conductuales 34 a 48 larva...
Divulgaciones
Los autores declaran que no tienen ningún interés en competencia financiera.
Agradecimientos
Damos las gracias a Fumi Kubo, Thiele y Tod HerwigBaier (UCSF / Max Planck Institute) para el asesoramiento sobre los experimentos de comportamiento y DPSS láser establecidos; Heesoo Kim y Chiara Cerri de la MBL Curso de Neurobiología de ayudar en Chef-tdTomato experimentos; PetronellaKettunen (Universidad de Gotemburgo ) para la colaboración inicial de los experimentos optogenético; BaljitKhakh, Eric Hudson, Baca Mike y John Milligan (UCLA) para el asesoramiento técnico, y Roger Tsien (UCSD) para el chef-tdTomato construir. Este trabajo fue apoyado por un NRSA (5F31NS064817) Premio a AMSP y una subvención de la NSF (RIG: 0819010) a AS.
Materiales
| Name | Company | Catalog Number | Comments |
| Nombre del reactivo / material | Empresa | Número de Catálogo | Comentarios |
| Materiales | |||
| Pipeta Pasteur de vidrio | Pescador | 1367820B | o equivalente (10-15 mm de diámetro) |
| 200 m de fibra óptica | Thorlabs | AFS200/220Y-CUSTOM | Patch Cord, longitud: 3 m, Final A: FC / PC, Final B: FC / PC, Chaqueta: FT030 |
| 50 de fibra óptica micras | Thorlabs | AFS50/125Y-CUSTOM | Patch Cord, longitud: 3 m, Final A: FC / PC, Final B: FC / PC, Chaqueta: FT030 |
| Pelacables ajustable | Thorlabs | AFS900 | o de tres agujeros Stripping Tool (FTS4) |
| Diamond escriba Wedge | Thorlabs | S90W | |
| Flaming / Brown Micropipeta Puller | Sutter Instruments | P-97 | o equivalente |
| Tubos de vidrio borosilicato con filamento | Sutter Instruments | BF-100-78-10 | |
| Molde de inyección | n / a | n / a | Figura 5 |
| 1,5 ml tubos de centrífuga | Cualquier | Cualquier | |
| Placa de Petri (100x15 mm) | Cualquier | Cualquier | |
| Placa de Petri (60x15 mm) | Cualquier | Cualquier | |
| Presión del inyector | ASI | MPPI-3 | o equivalente |
| Micromanipulador y stand de metal | Narashige | M152 | o equivalente |
| Pipetas de plástico desechables | Fisherbrand | 13-711-7 | o equivalente |
| Poker (titular Pin pin Insecto) | Fine Ciencia Tools, Inc. | 26018-17 y 26000-70 | o equivalente |
| Fórceps | Fine Ciencia Tools, Inc. | 11255-20 | o equivalente |
| Microloader puntas de pipeta | Eppendorf | 9300001007 | |
| 28,5 ° C incubadora | cualquier | cualquier | |
| 42 ° C bloque de calor | Cualquier | Cualquier | |
| No estériles hojas de bisturí # 11 | Bellas Herramientas Científicas, Inc. | 10011-00 | o equivalente |
| Disección shacer frente a | Zeiss | Stemi | o equivalente |
| Fluorescente microscopio de disección con filtro estándar | Cualquier | cualquier | o equivalente |
| Microscopio confocal | Zeiss | LSM 510 o 710 | u objetivos equivalentes con láser para las buenas prácticas agrarias y RFP, y 10x, 20x y 40x |
| Cámara de alta velocidad | Tecnologías de la AOS, Inc. | X-PRI (130.025-10) | o equivalente |
| 473 nm láser portátil | Láseres de cristal | CL-473-050 | o poder superior, con la opción TTL |
| S48 estimulador | Astro-Med, Inc. Grass Instrument división | S48K | o equivalente |
| FC / PC con el manguito de acoplamiento FC / PC | Thorlabs | ADAFC1 | May need para la conexión de cable óptico |
| Seguridad de láser Gafas | Thorlabs | LG10 | o equivalente |
| 24 placas de cultivo | Genesee | 25-102 | o equivalente |
| Diapositivas individuales depresión | Pescador | S175201 | O el equivalente |
| Reactivo | |||
| Instantánea océano | Ecosistemas Acuáticos | IS50 | |
| Azul de metileno | Pescador | S71325 | |
| Rojo fenol | Sigma | P4758 | |
| Agarosa | EMD | 2125 | o equivalente |
| Agarosa de bajo Melt | Sigma | A9045 | o equivalente |
| PTU | Sigma | P7629 | |
| Tricaína | Sigma | A5040 | |
| agua azul / embrión | 10 L ddH 2 O 0,6 g Instant Ocean 6 gotas de azul de metileno | ||
| rojo fenol | (5 mg / ml en 0,2 M KCl) | ||
| 100x PTU | 0,150 g PTU 50 ml ddH 2 O disolver a 70 ° C y agitar con frecuencia alicuotar y almacenar a -20 ° C | ||
| 1x PTU | 1 ml 100x PTU 99 ml azul / peces de agua | ||
| Tricaína solución madre | 400 mg tricaína 97,9 ddH 2 O | ||
| ~ 2,1 ml de Tris 1 M, pH 9,0 | ajustar el pH a 7,0 ~ tienda en 4 ° C o -20 ° C para el almacenamiento a largo plazo |
Referencias
- Arrenberg, A. B., Del Bene, F., Baier, H. Optical control of zebrafish behavior with halorhodopsin. Proc. Natl. Acad. Sci. U.S.A. 106 (42), 17968-17973 (2009).
- Burgess, H. A., Johnson, S. L., Granato, M. Unidirectional startle responses and disrupted left-right co-ordination of motor behaviors in robo3 mutant zebrafish. Genes Brain Behav. 8 (5), 500-511 (2009).
- Douglass, A. D., Kraves, S., Deisseroth, K., Schier, A. F., Engert, F. Escape behavior elicited by single, channelrhodopsin-2-evoked spikes in zebrafish somatosensory neurons. Curr. Biol. 18 (15), 1133-1137 (2008).
- Downes, G. B., Granato, M. Supraspinal input is dispensable to generate glycine-mediated locomotive behaviors in the zebrafish embryo. J. Neurobiol. 66 (5), 437-451 (2006).
- Drapeau, P., Saint-Amant, L., Buss, R. R., Chong, M., McDearmid, J. R., Brustein, E. Development of the locomotor network in zebrafish. Prog. Neurobiol. 68 (2), 85-111 (2002).
- Eisenhoffer, G. T., Rosenblatt, J. Live Imaging of Cell Extrusion from the Epidermis of Developing Zebrafish. J. Vis. Exp. (52), e2689 (2011).
- Graeden, E., Sive, H. Live Imaging of the Zebrafish Embryonic Brain by Confocal Microscopy. J. Vis. Exp. (26), e1217 (2009).
- Kague, E., Weber, C., Fisher, S. Mosaic Zebrafish Transgenesis for Evaluating Enhancer Sequences. J. Vis. Exp. (41), e1722 (2010).
- Kemp, H. A., Carmany-Rampey, A., Moens, C. Generating Chimeric Zebrafish Embryos by Transplantation. J. Vis. Exp. (29), e1394 (2009).
- Kohashi, T., Oda, Y. Initiation of Mauthner- or non-Mauthner-mediated fast escape evoked by different modes of sensory input. J. Neurosci. 28 (42), 10641-10653 (2008).
- Lai, S. L., Lee, T. Genetic mosaic with dual binary transcriptional systems in Drosophila. Nat. Neurosci. 9 (5), 703-709 (2006).
- Liao, J. C., Haehnel, M. Physiology of afferent neurons in larval zebrafish provides a functional framework for lateral line somatotopy. J. Neurophysiol. 107 (10), 2615-2623 (2012).
- Lin, J. Y., Lin, M. Z., Steinbach, P., Tsien, R. Y. Characterization of engineered channelrhodopsin variants with improved properties and kinetics. Biophys J. 96 (5), 1803-1814 (2009).
- Liu, K. S., Fetcho, J. R. Laser ablations reveal functional relationships of segmental hindbrain neurons in zebrafish. Neuron. 23 (2), 325-335 (1999).
- Liu, Y. C., Bailey, I., Hale, M. E. Alternative startle motor patterns and behaviors in the larval zebrafish (Danio rerio). J. Comp. Physiol. A. Neuroethol. Sens Neural. Behav. Physiol. 198 (1), 11-24 (2012).
- Palanca, A. M., Lee, S. L., Yee, L. E., Joe-Wong, C., Trinh, L. A., Hiroyasu, E., Husain, M., Fraser, S. E., Pellegrini, M., Sagasti, A. New transgenic reporters identify somatosensory neuron subtypes in larval zebrafish. Dev. Neurobiol. , (2012).
- Pujol-Martí, J., Zecca, A., Baudoin, J. P., Faucherre, A., Asakawa, K., Kawakami, K., López-Schier, H. Neuronal birth order identifies a dimorphic sensorineural map. J. Neurosci. 32 (9), 2976-2987 (2012).
- Saint-Amant, L., Drapeau, P. Time course of the development of motor behaviors in the zebrafish embryo. J. Neurobiol. 37 (4), 622-632 (1998).
- Szobota, S., et al. Remote control of neuronal activity with a light-gated glutamate receptor. Neuron. 54 (4), 535-545 (2007).
- Uemura, O., et al. Comparative functional genomics revealed conservation and diversification of three enhancers of the isl1 gene for motor and sensory neuron-specific expression. Dev. Biol. 278 (2), 587-606 (2005).
- Wyart, C., Del Bene, F., Warp, E., Scott, E. K., Trauner, D., Baier, H., Isacoff, E. Y. Optogenetic dissection of a behavioural module in the vertebrate spinal cord. Nature. 461 (7262), 407-410 (2009).
- Yuan, S., Sun, Z. Microinjection of mRNA and Morpholino Antisense Oligonucleotides in Zebrafish Embryos. J. Vis. Exp. (27), e1113 (2009).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados