È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Attivazione Optogenetic di zebrafish neuroni somatosensoriali con Chef-tdTomato
In questo articolo
Riepilogo
Optogenetic tecniche hanno permesso di studiare il contributo dei neuroni specifici comportamenti. Descriviamo un metodo in zebrafish larvale per l'attivazione di singoli neuroni somatosensoriali che esprimono una variante channelrhodopsin (Chef) con un diodo-pompato allo stato solido (DPSS) laser e registrare i comportamenti suscitate con una telecamera ad alta velocità video.
Abstract
Zebrafish larvali stanno emergendo come modello per descrivere lo sviluppo e la funzione di semplici circuiti neurali. Grazie alla loro fecondazione esterna, rapido sviluppo, e la traslucenza, zebrafish sono particolarmente adatti per gli approcci optogenetic indagare funzione neurale circuito. In questo approccio, fotosensibili canali ionici sono espressi in neuroni specifici, consentendo lo sperimentatore per attivare o inibire a volontà e quindi valutare il loro contributo a specifici comportamenti. L'applicazione di questi metodi in zebrafish larvale è concettualmente semplice, ma richiede l'ottimizzazione dei dettagli tecnici. Qui mostriamo una procedura per esprimere una variante channelrhodopsin in larve di zebrafish neuroni somatosensoriali, singole cellule foto-attivazione, e la registrazione dei comportamenti che ne derivano. Introducendo alcune modifiche ai metodi precedentemente stabiliti, questo approccio può essere utilizzato per ottenere risposte comportamentali da singoli neuroni attivati finoad almeno 4 giorni dopo la fecondazione (DPF). In particolare, abbiamo creato un transgene con un neurone somatosensoriale enhancer, CREST3, per guidare l'espressione del tag channelrhodopsin variante, lo chef-tdTomato. Iniettando questo transgene in 1-cell risultati embrioni allo stadio di espressione mosaico nei neuroni somatosensoriali, che può essere ripreso con microscopia confocale. Illuminante identificato le cellule di questi animali con la luce di un laser 473 nm DPSS, guidati attraverso un cavo in fibra ottica, suscita comportamenti che possono essere registrati con una telecamera ad alta velocità e video analizzati quantitativamente. Questa tecnica potrebbe essere adattato per i comportamenti di studio suscitato attivando ogni neurone zebrafish. La combinazione di questo approccio con perturbazioni genetiche o farmacologici sarà un potente strumento per studiare la formazione di circuiti e la funzione.
Introduzione
Lo sviluppo di metodi optogenetic per promuovere o inibire l'eccitabilità neuronale con lunghezze d'onda definite di luce ha permesso di studiare la funzione di distinte popolazioni di neuroni nei circuiti neurali che controllano il comportamento 1, 19, 21. Questa tecnica viene spesso utilizzato per attivare gruppi di neuroni, ma può anche essere utilizzato per attivare i singoli neuroni. Larve Zebrafish sono particolarmente suscettibili di questi metodi in quanto sono trasparenti, il loro sistema nervoso si sviluppa rapidamente, e la creazione di animali transgenici è veloce e di routine. Tuttavia, notevoli ostacoli tecnici da superare per ottenere l'attivazione affidabile singolo neurone.
Per ottimizzare la procedura per l'attivazione dei neuroni optogenetic zebrafish singoli, ci siamo concentrati sui neuroni somatosensoriali. Larve Zebrafish rilevare una varietà di stimoli somatosensoriali con due popolazioni di neuroni: i neuroni del trigemino, che innervano la testa, e ROHON-Beard (Rb) neuroni che innervano il resto del corpo. Ogni neurone trigeminale e RB proietta un assone periferico che si dirama lungo in pelle di individuare stimoli e un assone centrale che si connette a valle circuiti neurali. Animali risponde al tocco Già il 21 ore post-fecondazione (HPF), indicando che i circuiti coerenti somatosensoriali si sono formati 5, 18. Durante lo sviluppo larvale, almeno un po 'di sinapsi del trigemino e RB neuroni sulla cellula di Mauthner di attivare risposte di fuga classici, ma accumulando evidenza suggerisce che ci sono più classi di neuroni somatosensoriali con diversi modelli di connettività che possono suscitare variazioni sul comportamento di fuga 2, 4, 10, 12, 14, 15, 16, 17. La motivazione per lo sviluppo di questo metodo è di caratterizzare la funzione del comportamento di diverse classi di neuroni somatosensoriali, ma questo approccio potrebbe in linea di principio essere usato per studiare la funzione del neurone o qualsiasi popolazione di neuroni in larval zebrafish.
Douglass et al. Precedentemente descritto un metodo per l'attivazione channelrhodopsin-2-esprimono neuroni somatosensoriali con luce blu, suscitando comportamento di fuga 3. Il loro approccio utilizzato un elemento enhancer del gene isl1 per guidare l'espressione di CHR2-EYFP nei neuroni somatosensoriali. Questo transgene, tuttavia, è stato segnalato per visualizzare fluorescenza relativamente debole, richiedendo co-iniezione di un secondo reporter, UAS :: GFP, per permettere la visualizzazione di cellule esprimenti CHR2-EYFP. Questo approccio è stato utilizzato per ottenere risposte comportamentali tra 24-48 HPF, ma non potrebbe mai ottenere una risposta Dopo 72 HPF. Così, mentre questo metodo funziona per studiare circuiti neurali nelle fasi larvali molto precoci (24-48 HPF), è inadeguato per la caratterizzazione di circuiti neurali e le risposte comportamentali in età superiore larve, quando più risposte diverse comportamentali sono evidenti e circuiti neurali sono più maturi.
Abbiamo cercato dimigliorare la sensibilità di questa tecnica per caratterizzare la funzione delle sottopopolazioni di neuroni RB larvali. Per migliorare l'espressione abbiamo utilizzato un somatosensoriale-specifico enhancer (CREST3) 20 per guidare l'espressione di LexA-VP16 e un tratto di sequenze operatore lexA (4xLexAop) 11 per amplificare l'espressione di una fluorescenza etichettato fotoattivabile canale. Questa configurazione amplificato espressione del canale, eliminando la necessità di co-esprimere un secondo reporter e che ci permette di determinare direttamente l'abbondanza relativa del canale in ogni neurone. Utilizzando il LexA / LexAop sequenza avuto l'ulteriore vantaggio di consentire di introdurre il transgene in linee reporter di zebrafish che utilizzano il sistema Gal4/UAS. Espressione transitoria di questo transgene portato a diversi livelli di espressione, ma di solito era abbastanza robusto per visualizzare sia il corpo cellulare e le proiezioni assonali dei singoli neuroni per diversi giorni. Per ottimizzare la sensibilitàlità alla luce abbiamo usato la luce attivata canale chef, una variante channelrhodopsin costituito da una chimera di channelopsin-1 (Chop1) e channelopsin-2 (Chop2) con un sito di crossover a elica ciclo EF 13. Questo canale è attivato alla stessa lunghezza d'onda CHR2, ma richiede una minore intensità di luce per l'attivazione, il che rende più sensibili di altri canali comunemente utilizzati, compresi CHR2. La proteina chef era fusa alla proteina fluorescente rossa, tdTomato, che ci permette di selezionare per l'espressione della proteina senza attivare il canale. Come sorgente luminosa, abbiamo utilizzato un diodo pompato a stato solido (DPSS) laser accoppiato ad un cavo in fibra ottica per fornire una precisa, potente impulso di luce blu per una regione specifica delle larve. Questo ha permesso di concentrare la luce laser sui neuroni individuali, eliminando la necessità di trovare rare animali transgenici che esprimono il canale in un singolo neurone. Usando questo approccio, siamo stati in grado di attivare i neuroni RB singoli, registrare risposta comportamentales con una telecamera ad alta velocità video e immagine i neuroni attivati ad alta risoluzione con microscopia confocale.
Protocollo
Preparare la seguente prima del tempo.
1. Preparare Optic Cable
- Creare un contenitore per il cavo ottico fondendo il collo rastremata di una pipetta Pasteur di vetro su un becco Bunsen per creare un angolo di ~ 150 °.
- Utilizzando un cutter filo o una lametta, tagliare con cautela il cavo ottico in due pezzi. Ogni pezzo deve avere una estremità con un FC / adattatore PC ed una estremità esposta. Conservare un pezzo come un cavo di riserva.
- Spelare il cavo ottico verso il rivestimento rimuovendo la giacca fibre e le fibre di rinforzo (Figura 1A) da 5,0 centimetri di fine taglio del cavo.
- Inserire il cavo ottico in preparato pipetta Pasteur. Assicurarsi cavo può facilmente muoversi dentro e fuori della punta della pipetta.
- Con cavo ottico che sporge dalla punta della pipetta Pasteur, tagliare con attenzione e rimuovere rivestimenti in fibra di tutto in fibra di vetro, ~ 2 mm dal fine taglio.
- Utilizzando una penna / vetro tagliatore di diamanti, nick la fibra di vetro e rompere off fine di creare un taglio netto / superficie alla fine della fibra (Figura 1B).
- Ritrarre ottica in pipetta Pasteur per la conservazione. Ripetere il punto (1,6), se punta della fibra ottica viene tagliato o si rompe in modo non uniforme.
- Cavo ottico in posizione pipetta Pasteur utilizzando un micromanipolatore (Figura 1C).
Procedure 2-8 descrivono un metodo per l'iniezione di transgeni in embrioni in generale da molti esperimenti zebrafish. Variazioni su questo metodo, come quelli descritti in altri video jove 6, 7, 8, 9, 22 sono ugualmente efficaci.
2. Tirare aghi da iniezione
- Utilizzando un estrattore ago, tirare tubi in vetro borosilicato in due aghi da iniezione con una punta gradualmente ridotta. Impostazioni Puller varia. (Su un estrattore ago Sutter Instruments usiamo impostazioni: P = 500, calore = 720, Pull = 50, = 70 Velocità e Tempo = 150). Ogni estrattore ago è diverso, quindi ottimizzare le impostazioni puller emempiricamente. Per ulteriori aghi conici, aumentare il calore e / o Pull. Per aghi meno conici, aumentare il tempo e / o Pull diminuzione.
- Aghi Conservare in un contenitore sicuro (ad esempio una piastra di Petri con nastro laminato, fuori lato adesivo).
3. Versare stampi ad iniezione
- Sciogliere 0,5 g di agarosio in 30 ml embrione / acqua blu, fino agarosio si è completamente sciolto.
- Versare in metà inferiore di una piastra di Petri.
- Posizionare stampo rettangolare con montaggio pozzetti (Figura 2) in agarosio, facendo attenzione a limitare la formazione di bolle intorno ai pozzi.
- Lasciare agarosio a solidificare.
- Rimuovere stampo e riempire piastra di Petri con blu / acqua embrione.
- Conservare in posizione verticale, riempita con acqua pulita, a temperatura ambiente per il giorno stesso o a 4 ° C per un uso futuro.
4. Fare Mix plasmide DNA per preparazioni iniettabili
- Diluire DNA plasmidico ad una concentrazione di 50 ng / ml con 1:10 rosso fenolo in DDH 2 O. PerEsempio:
1,0 ml DNA plasmidico (250 ng / ml) 0,5 ml rosso fenolo 3,5 ml DDH 2 O - Mix DNA può essere conservato a temperatura ambiente se utilizzare immediatamente o conservato a 4 ° C per diversi giorni.
5. Impostare Coppie di accoppiamento
Questo dovrebbe essere fatto la sera prima avete intenzione di fare iniezioni.
- Riempire vasche di allevamento con l'acqua del sistema e luogo pesci di sesso maschile e femminile insieme. Se le iniezioni non può essere eseguita appena luci accende nella struttura, separata pesce maschio e femmina con un divisore.
6. Preparati per iniezioni (può essere fatto in attesa di embrioni)
- Accendere la pressione dell'iniettore rig. Assicurarsi che il sistema è impostato su DC. Inizia con il set di durata dell'impulso a 1.
- Aprire la valvola AIR eRegolare la pressione iniettore a ~ 20 psi.
- Utilizzando un ambito dissezione con il massimo ingrandimento, rompere punta dell'ago con una pinza o un poker per creare un 2 micron di apertura ~ (Figura 3, Movie 1).
- Riempire aghi con mix di DNA: 1. Posizionando il, lato punta dell'ago in alto, nel mix DNA e lasciando l'ago riempire per azione capillare o 2. Utilizzando un lungo raggiungere punta di pipettare 1-2 microlitri del DNA miscelare direttamente nell'ago.
- Luogo pieno ago in un luogo sicuro fino al momento di iniettare.
7. Raccogliere embrioni
- Dopo impianto luci si sono rivolti, rimuovere divisori (se applicabile). Raccogliere gli embrioni con un colino / setaccio piccolo e trasferirli in una capsula di Petri con blu dolce / acqua embrione.
8. Iniettare embrioni allo Stage cella 1-2
- Trasferimento embrioni raccolti per stampi ad iniezione con una pipetta di plastica.
- Utilizzando un microscopio da dissezione, spingere delicatamente embrioni inpozzetti con forcipe o un poker smussato (Figura 4, Movie 2).
- Inserire l'ago caricato in un micromanipolatore e la posizione più embrioni.
- Calibrare volume di iniezione regolando la durata dell'impulso di 1 step con incrementi fino a raggiungere il volume desiderato (~ 1 nl). Questo potrebbe essere tarato con un vetrino micrometrico, come descritto in un video precedenti Giove 8, 22, ma per questo esperimento precisione non è necessaria, poiché una vasta gamma di volumi di iniezione comporterà espressione adeguata.
- Iniettare ~ 1 nl DNA miscelare direttamente nella cella e ripetere per tutti gli embrioni. DNA può anche essere iniettato nel tuorlo, ma tende ad essere meno efficiente (Figura 5, Movie 3).
- Rimuovere gli embrioni iniettati da stampo con una leggera corrente di blu / acqua embrione.
- Conservare iniettato embrioni a 28,5 ° C al buio.
- Rimuovere non fecondate, gli embrioni morti o periodicamente.
- Trattare embrioni with PTU tra 18-24 HPF per prevenire la pigmentazione.
9. Schermo per espressione del transgene
- Manualmente dechorionate 24-48 embrioni HPF con pinze.
- Anestetizzare larve con 0,02% tricaine.
- Utilizzando un microscopio a fluorescenza dissezione, identificare larve con RB o un'espressione neurone trigeminale e trasferirli in un nuovo piatto fresco con PTU blu / embrione acqua. Embrioni con scarsa espressione in cellule facilmente identificabili sono ottimali, ma singoli neuroni saranno destinati per l'attivazione di un cavo a fibre ottiche così un'ampia gamma di densità espressione è accettabile.
- Embrioni Conservare a 28.5 ° C al buio fino a quando desiderato fase sperimentale.
10. Montare larve per esperimenti di comportamento
- Fai 1,5% bassa fusione agarosio in DDH 2 O e conservare in un blocco termico 42 ° C per evitare che la solidificazione.
- Utilizzando una pipetta Pasteur di vetro, trasferimento uno dei pre-screening larve in una vascae del 1,5% di agarosio a bassa fusione con un minimo di blu / embrione acqua possibile.
- Trasferire larve in una goccia di agarosio su una piccola scatola di Petri.
- Sotto un microscopio da dissezione, posizione larva fino dorsale.
- Quando agarosio si è solidificato, tagliare via la agarosio con una lama di rasoio sottile (bisturi # 11), lasciando un cuneo di agar intorno alla larva intera.
- Fare due tagli diagonali ai lati del tuorlo, prendere attenzione a non intaccare la larva.
- Riempire l'area circostante l'agarosio con embrione / acqua blu.
- Tirare agarosio lontano dal tronco e coda della larva (Figura 6).
11. Preparare ad alta velocità della fotocamera e software di imaging
- Montare ad alta velocità, fotocamera sulla dissezione portata.
- Collegare la fotocamera al computer.
- Accendere il computer.
- Attiva telecamera ad alta velocità.
- Apri video / software di imaging (Usiamo software di imaging AOS e descrivere le procedure per l'utilizzo di qui, ma imagingIl software è ugualmente accettabile).
- Regolare le impostazioni della fotocamera di conseguenza (vale a dire 1.000 fotogrammi al secondo (fps), tampone di trigger 50% o altre impostazioni preferite).
- Avviare la registrazione.
12. Attivare singoli neuroni con un laser 473 nm
- Allega stimolatore, laser e ottica.
- Accendere stimolatore.
- Impostare stimolatore ad un massimo di 5 volt e una durata di impulso di 5 msec.
- Accendere il laser secondo le istruzioni del produttore.
- Utilizzare un microscopio da dissezione per posizionare la punta del cavo ottico vicino al corpo cellulare di un neurone con lo chef-tdTomato espressione (Figura 6).
- Consegnare impulso di luce blu per attivare neurone sensoriale.
- Record comportamento utilizzando un telecamera ad alta velocità fissato a 500 o 1.000 fotogrammi al secondo.
- Ripetere esperimento come desiderato, in attesa 1 minuto tra ogni attivazione per evitare assuefazione. (Si registra un minimo di tre risposte per ogni neurone).
- Alarve di rilascio, indagare separatamente agarosio con una pinza, facendo attenzione a non ferire l'animale. Questo animale può essere consentito di sviluppare ulteriormente e la procedura può essere ripetuta in una fase precedente per caratterizzare sviluppo del comportamento. L'embrione può anche essere rimontato per alta risoluzione confocale della cella attivata per correlare il comportamento con struttura cellulare, come descritto di seguito.
- Trasferimento larva di piastra di coltura con il blu dolce / acqua embrione. Usiamo una piastra a 24 pozzetti per tenere traccia delle larve individuale.
13. Immagine Neuron (s) con un microscopio confocale
- Anestetizzare larve con 0,02% tricaine.
- Monte larve, dorso alto, in basso fuso 1,2% agarosio o in uno stampo dorsale.
- Immagine chef-tdTomato neuroni con un laser a 543 nm e filtro equo ed obiettivo. Usiamo un obiettivo 20x.
- Rimuovere larve di agarosio e tornare al benessere individuale con il blu / acqua embrione.
- Conservare a 28.5 ° C al buio per il further analisi.
Risultati

Figura 1. Cavo ottico impostato. (A) Livelli di un cavo in fibra ottica. (B) cavo in fibra ottica in Stripped una pipetta Pasteur. (C) del cavo in fibra ottica in pipetta Pasteur posizionata utilizzando un micromanipolatore.
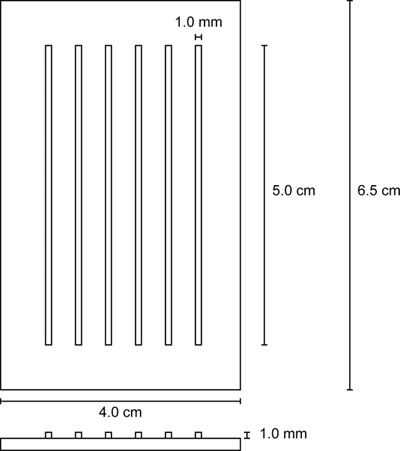
Figura 2. Iniezione stampo m...
Discussione
Abbiamo descritto un approccio per l'attivazione dei neuroni optogenetic RB singole in zebrafish vivo. Il nostro metodo utilizza transgenesi transitoria per esprimere una variante fluorescente tag channelrhodopsin, chef-tdTomato 13, in specifici neuroni somatosensoriali. Questo approccio potrebbe essere facilmente adattato per l'uso nelle popolazioni larvali zebrafish altre cellule.
Utilizzando questo approccio abbiamo costantemente suscitato risposte comportamentali 34-48...
Divulgazioni
Gli autori dichiarano di non avere alcun interesse finanziario concorrente.
Riconoscimenti
Ringraziamo Fumi Kubo, Tod Thiele e HerwigBaier (UCSF / Max-Planck-Institut) per un parere sulla sperimentazione dei comportamenti e di laser DPSS impostati; Heesoo Kim e Chiara Cerri dal Corso di MBL Neurobiologia per l'assistenza nella Chef-tdTomato esperimenti; PetronellaKettunen (Università di Göteborg ) per la collaborazione iniziale di esperimenti optogenetic, BaljitKhakh, Eric Hudson, Mike Baca e John Milligan (UCLA) per un parere tecnico, e Roger Tsien (UCSD) per il cuoco-tdTomato costruire. Questo lavoro è stato supportato da una (5F31NS064817) premio NRSA a AMSP e una borsa di studio dalla NSF (RIG: 0.819.010) ad AS.
Materiali
| Name | Company | Catalog Number | Comments |
| Nome del reagente / materiale | Azienda | Numero di catalogo | Commenti |
| Materiale | |||
| Vetro pipetta Pasteur | Pescatore | 1367820B | o equivalente (10-15 mm di diametro) |
| 200 micron fibra ottica | Thorlabs | AFS200/220Y-CUSTOM | Patch cord, Lunghezza: 3 m, Fine A: FC / PC, End B: FC / PC, Jacket: FT030 |
| Fibra da 50 micron ottica | Thorlabs | AFS50/125Y-CUSTOM | Patch cord, Lunghezza: 3 m, Fine A: FC / PC, End B: FC / PC, Jacket: FT030 |
| Regolabile Stripping Tool | Thorlabs | AFS900 | o tre fori Stripping Tool (FTS4) |
| Diamante Wedge scriba | Thorlabs | S90W | |
| Flaming / Brown Micropipetta Puller | Sutter Instruments | P-97 | o equivalente |
| Tubi di vetro borosilicato con filamento | Sutter Instruments | BF-100-78-10 | |
| Iniezione stampo | n / a | n / a | Figura 5 |
| Centrifuga da 1,5 ml provette | Qualsiasi | Qualsiasi | |
| Petri (100x15 mm) | Qualsiasi | Qualsiasi | |
| Petri (60x15 mm) | Qualsiasi | Qualsiasi | |
| Pressione iniettore | ASI | MPPI-3 | o equivalente |
| Micromanipolatore e metallo cavalletto | Narashige | M152 | o equivalente |
| Pipette di plastica usa e getta | FISHERBRAND | 13-711-7 | o equivalente |
| Poker (supporto del perno e perno Insect) | Bene Scienza Strumenti, Inc. | 26018-17 e 26000-70 | o equivalente |
| Forcipe | Bene Scienza Strumenti, Inc. | 11255-20 | o equivalente |
| Microloader puntali per pipette | Eppendorf | 9300001007 | |
| 28.5 ° C incubatore | qualsiasi | qualsiasi | |
| 42 ° C di calore di blocco | Qualsiasi | Qualsiasi | |
| Lame di bisturi non sterili # 11 | Belle Strumenti Scientifici, Inc. | 10011-00 | o equivalente |
| Dissezione sfar fronte | Zeiss | Stemi | o equivalente |
| Fluorescente portata dissezione con filtro standard | Qualsiasi | qualsiasi | o equivalente |
| Microscopio confocale | Zeiss | LSM 510 o 710 | o obiettivi equivalenti con i laser per la GFP e RFP, e 10x, 20x e 40x |
| Ad alta velocità della fotocamera | AOS Technologies, Inc. | X-PRI (130025-10) | o equivalente |
| 473 nm laser portatile | Cristallo laser | CL-473-050 | o potenza superiore, con opzione TTL |
| S48 Stimolatore | Astro-Med, Inc. Grass divisione strumento | S48K | o equivalente |
| FC / PC FC / PC manicotto di accoppiamento | Thorlabs | ADAFC1 | Maggio need per il collegamento del cavo ottico |
| Sicurezza laser Occhiali | Thorlabs | LG10 | o equivalente |
| 24 piastre di coltura | Genesee | 25-102 | o equivalente |
| Diapositive depressione singoli | Pescatore | S175201 | O equivalente |
| Reagente | |||
| Istantanea oceano | Ecosistemi acquatici | IS50 | |
| Blu di metilene | Pescatore | S71325 | |
| Rosso fenolo | Sigma | P4758 | |
| Agarosio | EMD | 2125 | o equivalente |
| Low Melt agarosio | Sigma | A9045 | o equivalente |
| PTU | Sigma | P7629 | |
| Tricaine | Sigma | A5040 | |
| blu acqua / embrione | 10 L DDH 2 O 0,6 g istantanea Ocean 6 gocce di blu di metilene | ||
| rosso fenolo | (5 mg / ml in 0,2 M KCl) | ||
| 100x PTU | 0,150 g PTU 50 ml DDH 2 O sciogliere a 70 ° C, agitare spesso aliquota e conservare a -20 ° C | ||
| 1x PTU | 1 ml 100x PTU 99 ml blu / pesci d'acqua | ||
| Tricaine soluzione stock | 400 mg tricaine 97,9 DDH 2 O | ||
| ~ 2,1 ml 1M Tris, pH9.0 | aggiustare il pH a ~ 7.0 negozio a 4 ° C o -20 ° C per una conservazione a lungo termine |
Riferimenti
- Arrenberg, A. B., Del Bene, F., Baier, H. Optical control of zebrafish behavior with halorhodopsin. Proc. Natl. Acad. Sci. U.S.A. 106 (42), 17968-17973 (2009).
- Burgess, H. A., Johnson, S. L., Granato, M. Unidirectional startle responses and disrupted left-right co-ordination of motor behaviors in robo3 mutant zebrafish. Genes Brain Behav. 8 (5), 500-511 (2009).
- Douglass, A. D., Kraves, S., Deisseroth, K., Schier, A. F., Engert, F. Escape behavior elicited by single, channelrhodopsin-2-evoked spikes in zebrafish somatosensory neurons. Curr. Biol. 18 (15), 1133-1137 (2008).
- Downes, G. B., Granato, M. Supraspinal input is dispensable to generate glycine-mediated locomotive behaviors in the zebrafish embryo. J. Neurobiol. 66 (5), 437-451 (2006).
- Drapeau, P., Saint-Amant, L., Buss, R. R., Chong, M., McDearmid, J. R., Brustein, E. Development of the locomotor network in zebrafish. Prog. Neurobiol. 68 (2), 85-111 (2002).
- Eisenhoffer, G. T., Rosenblatt, J. Live Imaging of Cell Extrusion from the Epidermis of Developing Zebrafish. J. Vis. Exp. (52), e2689 (2011).
- Graeden, E., Sive, H. Live Imaging of the Zebrafish Embryonic Brain by Confocal Microscopy. J. Vis. Exp. (26), e1217 (2009).
- Kague, E., Weber, C., Fisher, S. Mosaic Zebrafish Transgenesis for Evaluating Enhancer Sequences. J. Vis. Exp. (41), e1722 (2010).
- Kemp, H. A., Carmany-Rampey, A., Moens, C. Generating Chimeric Zebrafish Embryos by Transplantation. J. Vis. Exp. (29), e1394 (2009).
- Kohashi, T., Oda, Y. Initiation of Mauthner- or non-Mauthner-mediated fast escape evoked by different modes of sensory input. J. Neurosci. 28 (42), 10641-10653 (2008).
- Lai, S. L., Lee, T. Genetic mosaic with dual binary transcriptional systems in Drosophila. Nat. Neurosci. 9 (5), 703-709 (2006).
- Liao, J. C., Haehnel, M. Physiology of afferent neurons in larval zebrafish provides a functional framework for lateral line somatotopy. J. Neurophysiol. 107 (10), 2615-2623 (2012).
- Lin, J. Y., Lin, M. Z., Steinbach, P., Tsien, R. Y. Characterization of engineered channelrhodopsin variants with improved properties and kinetics. Biophys J. 96 (5), 1803-1814 (2009).
- Liu, K. S., Fetcho, J. R. Laser ablations reveal functional relationships of segmental hindbrain neurons in zebrafish. Neuron. 23 (2), 325-335 (1999).
- Liu, Y. C., Bailey, I., Hale, M. E. Alternative startle motor patterns and behaviors in the larval zebrafish (Danio rerio). J. Comp. Physiol. A. Neuroethol. Sens Neural. Behav. Physiol. 198 (1), 11-24 (2012).
- Palanca, A. M., Lee, S. L., Yee, L. E., Joe-Wong, C., Trinh, L. A., Hiroyasu, E., Husain, M., Fraser, S. E., Pellegrini, M., Sagasti, A. New transgenic reporters identify somatosensory neuron subtypes in larval zebrafish. Dev. Neurobiol. , (2012).
- Pujol-Martí, J., Zecca, A., Baudoin, J. P., Faucherre, A., Asakawa, K., Kawakami, K., López-Schier, H. Neuronal birth order identifies a dimorphic sensorineural map. J. Neurosci. 32 (9), 2976-2987 (2012).
- Saint-Amant, L., Drapeau, P. Time course of the development of motor behaviors in the zebrafish embryo. J. Neurobiol. 37 (4), 622-632 (1998).
- Szobota, S., et al. Remote control of neuronal activity with a light-gated glutamate receptor. Neuron. 54 (4), 535-545 (2007).
- Uemura, O., et al. Comparative functional genomics revealed conservation and diversification of three enhancers of the isl1 gene for motor and sensory neuron-specific expression. Dev. Biol. 278 (2), 587-606 (2005).
- Wyart, C., Del Bene, F., Warp, E., Scott, E. K., Trauner, D., Baier, H., Isacoff, E. Y. Optogenetic dissection of a behavioural module in the vertebrate spinal cord. Nature. 461 (7262), 407-410 (2009).
- Yuan, S., Sun, Z. Microinjection of mRNA and Morpholino Antisense Oligonucleotides in Zebrafish Embryos. J. Vis. Exp. (27), e1113 (2009).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon