Method Article
La identificación basada en microarrays de expresión individual HERV Loci: Aplicación al descubrimiento de biomarcadores en el cáncer de próstata
En este artículo
Resumen
Retrovirus endógenos humanos (HERV), que ocupan 8% del genoma humano, conservan la capacidad de codificación de los escasos pero cien mil repeticiones terminales largas (LTRs). A Affymetrix microarray costumbre fue diseñado para identificar la expresión locus HERV individual y se utilizó en los tejidos de cáncer de próstata como una prueba de concepto para futuros estudios clínicos.
Resumen
El antígeno específico de la próstata (PSA) es el principal biomarcador de diagnóstico para el cáncer de próstata en el uso clínico, pero carece de especificidad y sensibilidad, particularmente en los valores de dosificación bajos 1. "Cómo usar PSA 'continúa siendo un tema de actualidad, ya sea para el diagnóstico como una zona gris que corresponde a una concentración en suero de 2,5 a 10 ng / ml, que no permite una clara diferenciación que debe hacerse entre el cáncer y no cáncer 2 o para el seguimiento de pacientes -como el análisis de PSA postoperatorio parámetros cinéticos pueden plantear considerables retos para su aplicación en la práctica 3,4. Alternativamente, noncoding RNAs (ncRNAs) están emergiendo como moléculas clave en el cáncer humano, con el potencial de servir como nuevos marcadores de la enfermedad, por ejemplo, de PCA3 en el cáncer de próstata y 5,6 para revelar aspectos de la biología del tumor no caracterizados. Por otra parte, los datos del proyecto ENCODE publicados en 2012 mostraron que los diferentes tipos de ARN cubren cerca del 62% de la generaciónome. También parece que la cantidad de motivos reguladores de la transcripción es al menos 4,5 veces más alto que el correspondiente a los exones codificantes de proteínas. Por lo tanto, repeticiones terminales largas (LTR) de retrovirus endógenos humanos (HERV) constituyen una amplia gama de putativo / candidatos secuencias reguladoras de la transcripción, ya que es su función primaria en los retrovirus infecciosos. HERVs, repartidas a lo largo del genoma humano, se originan en infecciones ancestrales e independientes dentro de la línea germinal, seguidos de procesos de propagación de copiar y pegar, y que conducen a MultiCopy familias que ocupan el 8% del genoma humano (tenga en cuenta que los exones abarcan el 2% de nuestro genoma ). Algunos HERV loci todavía expresan proteínas que se han asociado con diversas patologías incluyendo el cáncer de 7-10. Hemos diseñado un microarray de alta densidad, en formato de Affymetrix, con el objetivo de caracterizar de manera óptima expresión loci HERV individual, con el fin de entender mejor si pueden ser activos, si conducen transcripción ncRNA o modulate de codificación de la expresión génica. Esta herramienta se ha aplicado en el campo del cáncer de próstata (Figura 1).
Introducción
Retrovirus endógenos humanos (también llamados HERVs) se extienden por todo nuestro genoma. Se originan en infecciones ancestrales e independientes dentro de la línea germinal, seguidos de procesos de propagación de copiar y pegar, y que llevan a las familias de múltiples copias. Hoy en día, que no son más infecciosa pero que ocupan 8% del genoma humano; como un punto de comparación, los exones abarcan 2% del genoma humano. Los datos del proyecto ENCODE publicados en 2012 mostraron que los diferentes tipos de ARN cubren alrededor del 62% del genoma, incluyendo un tercio de las regiones intergénicas. Por otra parte, parece que la cantidad de motivos reguladores de la transcripción es al menos 4,5 veces más alto que el correspondiente a los exones codificantes de proteínas. HERV repeticiones terminales largas (LTR) representan una amplia gama de potenciales elementos reguladores de la transcripción, ya que es su función habitual en los retrovirus infecciosos. Históricamente, aparte de unos pocos loci expresado en la placenta o los testículos, se cree comúnmente que HERV son silenciosa debido a EPregulación igenetic. Por lo tanto, hemos diseñado un microarray de alta densidad, en formato de Affymetrix, con el objetivo de caracterizar de manera óptima expresión loci HERV individual, con el fin de entender mejor si están activas, si conducen transcripción lncRNA o modulan la expresión de genes de codificación. Esta herramienta apodada HERV-V2 GeneChip integra 23583 probesets HERV y puede discriminar 5.573 elementos HERV distintas compuestas de LTR en solitario, así como provirus completos y parciales (Figura 2).
Diagnóstico, Evaluación y Plan:
El diagnóstico de cáncer de próstata se basa en la dosis del antígeno específico de la próstata (PSA) biomarcador en el laboratorio clínico, un examen rectal digital para evaluar la alteración morfológica de la próstata y, finalmente, las biopsias de próstata observadas por el patólogo. La falta de especificidad y la sensibilidad suficiente entre biomarcadores de cáncer convencionales, tales como PSA para el cáncer de próstata, ha sido ampliamente reconocido AFter varias décadas de implicaciones clínicas 1. Inicialmente, el PSA se propuso para el diagnóstico y el tratamiento de adenocarcinoma de la próstata 11. Era esta última propuesto para la detección del cáncer y seguimiento de la evolución de la enfermedad 12. Sin embargo, sigue habiendo una pregunta que se hace con regularidad: 'cómo utilizar PSA'. (I) Una zona gris correspondiente a una concentración en suero de 2,5 a 10 ng / ml no permite una clara diferencia que se hace entre el cáncer y no cáncer 2, (ii) dos estudios de cohortes grandes inscribir a cientos de miles de personas en Europa y EE.UU. no pudo llegar a una conclusión clara sobre la utilidad del cribado en cuanto a la mortalidad específica de la enfermedad 13,14, (iii) el análisis de los parámetros cinéticos del PSA después de la operación como el despacho de PSA, velocidad de PSA y el tiempo de duplicación, aunque simple en teoría, puede plantear considerables retos en 3,4 la aplicación práctica. Podemos esperar que en los próximos años, la aplicación de biomarcadorescaciones apoyarán una opción clínica entre la espera vigilante y más o tratamientos menos agresivos dependiendo de fenotipo tumoral. En relación con el diagnóstico emitido por el patólogo, un primer factor de limitación proviene de un diagnóstico de falsos negativos del 20% dentro de las biopsias de próstata (muchos tipos de cáncer se pierden por muestreo). Una segunda preocupación tiene que ver con la necesidad de un procedimiento de biopsia adicional después de una negativa, que pueden presentar efectos adversos.
La prostatectomía radical es en la actualidad uno de los tratamientos estándar para el cáncer de próstata. Se propone en pacientes sanos, envejecimiento de 45 a 65 años, especialmente en el caso de patrones agresivos (Gleason 7 a 10), tumor multifocal o tumor palpable. En la actualidad se realiza en nuestro departamento mediante cirugía asistida por robot. Debido a la creciente evidencia de que los marcadores moleculares tendrán suma importancia en los próximos años, se decidió proponer a todos nuestros pacientes la posibilidad de participar en un programa para la próstatabancos de tejidos. Más precisamente, los programas de investigación en expansión moleculares en el cáncer de próstata se han traducido en una creciente necesidad de acceso a los tejidos de alta calidad tumorales frescas de las piezas de prostatectomía. Esta investigación, en particular los enfoques genómicos, requiere grandes muestras de alta calidad de ADN / ARN. Tumoral y tejidos adyacentes 'no' tumorales del mismo paciente se necesitan. Recomendaciones para el manejo y procesamiento de prostatectomías radicales están diseñados para preservar las características patológicas que determinan el escenario y estado de los márgenes y por lo tanto el potencial más tratamiento y el pronóstico. Cualquier método de muestreo de tejido fresco, por lo tanto, no debe comprometer evaluaciones patológicas posteriores con el fin de ser aceptable para el diagnóstico. Disección macroscópica de la próstata es difícil y requiere gran atención debe prestarse a los tejidos de margen y la invasión capsular: ninguna disección para la banca de próstata debe ser siempre realizada por un uropathologist entrenados de acuerdo a un acuerdod protocolo. El comité de ética de la Facultad de Medicina y de la junta médica del estado de acuerdo a estas investigaciones y el consentimiento informado se obtuvo de todos los pacientes incluidos en la banca tejidos de la próstata.
Protocolo
1. Cirugía
Una vez eliminado por el cirujano, mantener la próstata en hielo hasta que se toma en cargo de un patólogo.
2. Manejo de los tejidos de la próstata
- Respetar el retraso de la isquemia perioperatoria, transferir piezas de prostatectomía radical en el hielo al laboratorio por personal dedicado a los 30 minutos después de la ablación quirúrgica. El retraso de bloqueo no debe ser menos de 20-30 minutos (Figura 3A).
- Pesar y manchar la próstata según el protocolo habitual (por ejemplo, verde en el lado derecho, negro en el lado izquierdo, véanse las Figuras 3B y 3C).
- Realice una sección transversal grande de la glándula en el lado posterior (Figura 3D). Oriente la próstata y lo puso en la cara anterior. Realice una sección transversal grande de la glándula en el lado posterior con un cuchillo quirúrgico estéril.
- Diseccionar piezas de tejido en la zona de transición,en las zonas periféricas izquierda y derecha, dejando los márgenes intacta (Figura 3E).
- Ponga los núcleos de tejido en un tubo Eppendorf, snap congelación y la conservación en nitrógeno líquido (Figura 3F). Si usted no está haciendo biobanco proceder directamente al paso 2.7.
- Realizar la banca de próstata sólo si la longitud total de cáncer en biopsias es superior a 10 mm. Utilice un hilo de sutura para cerrar la próstata y para evitar la distorsión de la glándula y una interrupción mínima del margen quirúrgico (Figura 3G). A continuación, fije la pieza de prostatectomía con formol y de inserción en parafina de acuerdo con el procedimiento habitual para el análisis histológico.
- Montar núcleos de tejido congelado verticalmente sobre un pequeño montículo de octubre y hacer secciones en un criostato. Tome un primer single sección congelada 5 micras y mancharlo con azul de toluidina.
- Realizar un examen histológico rápida de analizar la naturaleza del tejido (es decir. Benigno o maligno). Para tumoraltejido, estimar la cantidad de células tumorales y seleccionar sólo los núcleos con más de 80% de las células tumorales.
- A raíz de esto, realice un nuevo single sección congelada 5 micras y mancharlo con hematoxilina, eosina y Safran. Luego, cortar 15 secciones x 30 micras y colocarlo en un tubo Eppendorf de RNAsa libre.
- Tome una última sección congelada 5 micras para hematoxilina, eosina y Safran y manchar para controlar la cantidad de células tumorales al final del procedimiento.
- Coloque el tubo de Eppendorf en hielo seco y enviar la muestra al laboratorio de biología molecular.
3. Extracción de ARN, purificación y control de calidad
- La homogeneización. Realizar homogeneización en presencia de 1 ml de mg de tejido Trizol/100 hasta que el tejido esté completamente disuelto en solución. Añadir la solución de Trizol gradualmente y proceder con cuidado en hielo usando un molinillo de mano. Una vez homogeneizada, alícuotas de la solución a tubos Eppendorf y dejar en Trizol a temperatura ambiente duranteCinco minutos.
- La separación de fases. Añadir 300 l de cloroformo (o 150 l BCP/1.5 ml Trizol). Vortex 15 segundos y luego dejar a temperatura ambiente durante 2-3 min. Se centrifuga a 12.000 xg durante 15 minutos a 2-8 ° C.
- La precipitación de ARN. Transferir cuidadosamente la fase acuosa superior a un nuevo tubo. Añadir 750 l de isopropanol. Incubar a temperatura ambiente durante 10 min (agitar por inversión). Incubar 2 horas a -19 ° C/-31 ° C. Centrifugar las muestras a 12.000 xg durante 30 minutos a 2-8 ° C.
- Lavado de ARN y la suspensión. Después de la centrifugación, eliminar el sobrenadante. Lave ARN pellet con 1 ml de EtOH al 80% (retroceso suavemente los tubos). Centrifugar las muestras a 7500 xg durante 10 minutos a 2-8 ° C. Aspirar el sobrenadante con P1000 y P10. Permitir restante EtOH para secar al aire durante 2-3 min. Añadir 100 RNasa libre de agua l, tubos de transferencia de calor a 70 ° C de bloque y deje reposar durante 2-3 minutos para disolver el precipitado. A continuación, poner en hielo. Almacenar a -19 ° C/-31 almacenamiento ° C para el corto plazo y -80 ° C para el almacenamiento a largo plazo.
- La purificación del ARN. Se purifica ARN utilizando el kit RNeasy Mini (Qiagen). En pocas palabras, empezar añadiendo 350 l RLT buffer para la muestra de ARN 100 l y mezclar bien, a continuación, siga el procedimiento de Qiagen. Por último, tome un l 3 (de un total de 50 l) alícuota del producto purificado para los controles de calidad (paso 3.6). RNA Conservar a -19 ° C/-31 ° C durante un periodo de corto plazo oa -80 ° C para un almacenamiento a largo plazo.
- ARN QC (Figura 4 A). Comprobación de la calidad del ARN y la integridad del ARN usando un Bioanalyzer (Agilent) y un Nanodrop (Thermo), de acuerdo con las instrucciones del fabricante. El Número de la integridad del ARN (RIN) se utiliza para evaluar la calidad del ARN. En particular, el éxito en la detección de 18S y 28S picos se recomienda encarecidamente el uso de las muestras en otros pasos.
4. WT-ovación de amplificación del ARN
Recomendaciones para realizar las etapas de amplificación utilizando el WT-Ovkit de amplificación ación en óptimas condiciones:
- Correr no menos de ocho muestras de amplificación a la vez para asegurar la precisión de pipeteo. A continuación, dar cuenta de 1 volumen de residuos en la preparación de mezclas maestras que requieren una escisión del kit en 3 lotes de 8 reacciones.
- Mantenga siempre los reactivos descongelados y tubos de reacción en hielo salvo instrucción en contrario.
- Utilice sólo una solución de etanol al 80% fresco para la purificación.
- No detenerse en cualquier etapa del protocolo.
- Diluir ARN total para obtener una concentración de 25 ng / l. Proceso 2 l de la muestra diluida.
- Preparar la poli-Una solución de ARN espiga-en el control por una dilución en serie de la poli-A ARN Stock con poli-A de Control de tampón de dilución (Affymetrix), para lograr una dilución de 1:25.000.
Paso 1: En primer lugar Strand cDNA Synthesis 4,3-4,8. Los reactivos mencionados son referidos por el proveedor de la siguiente manera: A1 (First Strand Primer Mix), A2 (First Strand Buffer Mix), A3 (First Strand Enzyme Mix).
- Descongele A1 y A2 a temperatura ambiente. Mezclar utilizando un vórtice-mezclador durante 2 segundos y centrifugado durante 2 segundos. Luego, coloque rápidamente en hielo. Coloque A3 en hielo.
- Ponga 2 l de ARN total (50 ng) en un tubo de PCR de 0,2 ml y añadir 2 l de A1 (volumen final: 4 mu l). Cap y hacer girar el tubo durante 2 seg.
- Se incuba a 65 ° C durante 5 minutos y luego colocar el tubo en hielo.
- Preparar la primera hebra de ADNc de mezcla maestra de la siguiente manera (dado para una sola reacción). Mezclar con la pipeta y centrifugar brevemente la mezcla maestra. Inmediatamente, colocar en hielo.
Reactivo Volumen First Strand Buffer Mix (A2) 5 l Poli-A Control de ARN (1:25.000) 0.5 l La primera cadena de mezcla de enzimas (A3) 0.5 l - Añadir 6 & #181, l de la mezcla maestra a la / tubo conteniendo Primer-ARN. Mezclar con un movimiento rápido del tubo, giro durante 2 segundos y colocar rápidamente en hielo (volumen final: 10 l).
- Incubar a 4 º C durante 1 min, luego 25 º C durante 10 min, luego 42 º C durante 10 min y luego 70 º C durante 15 min. Mantener en lugar fresco a 4 ° C. Retire el tubo de reacción del termociclador, girar brevemente y mantener en hielo. Continúe inmediatamente con el paso de la segunda cadena de cDNA Synthesis.
Paso 2: Second Strand cDNA Synthesis 4,8-4,12. Los reactivos mencionados son referidos por el proveedor de la siguiente manera: B1 (Segunda Strand Buffer Mix) y B2 (la segunda cadena Enzyme Mix).
- Decantar B2 y B3 durante 2 segundos y rápidamente colocar en hielo. Descongelar B1 a temperatura ambiente. Mezclar utilizando un vórtice-mezclador durante 2 segundos, girar durante 2 segundos y rápidamente colocar en hielo.
- Preparar una segunda hebra de mezcla maestra de la siguiente manera (dado para una sola reacción). Mezclar con la pipeta y girar hacia abajo el brie Master Mixvolar. Inmediatamente, colocar en hielo.
Reactivo Volumen En segundo lugar Strand Buffer Mix (B1) 9,75 l En segundo lugar Strand mezcla de enzimas (B2) 0,25 l - Añadir 10 l de la mezcla maestra a cada tubo de reacción de la primera cadena. Mezclar con la pipeta de 3 aumentos, centrifugado durante 2 segundos y el lugar en el hielo (volumen final: 20 l).
- Incubar a 4 º C durante 1 min, luego 25 º C durante 10 min, luego 50 º C durante 30 min, y luego 70 º C durante 5 min. Mantener en lugar fresco a 4 ° C. Retire el tubo de reacción del termociclador, girar brevemente y mantener en hielo. Continúe inmediatamente con el paso posterior a la segunda cadena de mejora.
Paso 3: Post-Segundo Strand Enhancement 4,13-4,15. Los reactivos mencionados son referidos por el proveedor de la siguiente manera: B1 (Segunda Strand Buffer Mix), B3 (Reacción Enhancement Enzyme Mix).
- Preparar una mezcla maestra mediante la combinación de B1 y B3 de la mezcla de la siguiente manera (dado para una sola reacción). Mezclar con la pipeta y centrifugar brevemente la mezcla maestra. Inmediatamente, colocar en hielo.
Reactivo Volumen En segundo lugar Strand Buffer Mix (B1) 1.9 l Reacción de mejora de mezcla de enzimas (B3) 0.1 l - Añadir 2 l de la mezcla maestra a cada tubo de reacción de la segunda cadena. Mezclar con la pipeta de 3 aumentos, centrifugado durante 2 segundos y el lugar en el hielo (volumen final: 22 l).
- Incubar a 4 º C durante 1 min, luego 37 º C durante 15 min, y luego 80 º C durante 20 min. Mantener en lugar fresco a 4 ° C. Retire el tubo de reacción del termociclador, girar brevemente y colocar en hielo. Continúe inmediatamente con el paso SPIA amplificación.
Paso 4: cDNA sola hebra (sscDNA) síntesis por procedimiento SPIA de 4,16 -4.19. Los reactivos mencionados son referidos por el proveedor de la siguiente forma: C1 (SPIA Primer Mix), C2 (SPIA Buffer Mix), C3 (SPIA Enzyme Mix).
- Descongelar la C1 y C2 a temperatura ambiente. Mezclar utilizando un vórtice-mezclador, giro durante 2 segundos y colocar rápidamente en hielo. Descongele C3 en hielo. Mezcle el contenido invirtiendo suavemente 5x. Asegúrese de no introducir burbujas de aire. Luego, girar durante 2 segundos y el lugar en el hielo.
- Prepare una mezcla SPIA-Master, lo que representa un volumen de 0,5 residuos, de la siguiente manera. Mezclar con la pipeta y centrifugar brevemente la mezcla maestra. Inmediatamente, colocar en hielo.
Reactivo Volumen SPIA-Buffer Mix (C2) 5 l SPIA-Primer Mix (C1) 5 l SPIA-Enzyme Mix (C3) 10 l - Añadir 20 l de la mezcla maestra SPIA para la Mejora de la segunda cadena de reacción tube. Mezclar con la pipeta 6-8x, girar y colocar rápidamente en hielo (volumen final: 42 l).
- Incubar a 4 º C durante 1 min, luego 47 º C durante 60 min y luego 95 º C durante 5 min. Mantener en lugar fresco a 4 ° C. Sacar el tubo del termociclador, girar brevemente y colocar en hielo.
5. sscDNA purificación y control de calidad
- sscDNA purificación. Purificar sscDNA usando el kit de purificación de QIAquik de PCR (Qiagen). En pocas palabras, empezar añadiendo 200 l de tampón PB a los 42 l de cDNA producto amplificado, mezcla y carga sobre la columna. A continuación, siga el procedimiento de Qiagen. Por último, tome un l 3 (de un total de 30 l) alícuota del producto purificado sscDNA para los controles de calidad (paso 5.2).
- sscDNA producir y verificación distribución de tamaño (Figura 4B). Compruebe sscDNA rendimiento y la distribución del tamaño usando un Bioanalyzer y un Nanodrop, de acuerdo con las instrucciones del fabricante. El tamaño de la distribución de ADNc amplificados debe be típicamente comprendida entre 100 y 1500 bases de longitud, con un pico alrededor de 600 bases.
6. sscDNA Fragmentación
- Prepare 2 g de cDNA en 30 l, ajustando el volumen con agua libre de nucleasa.
- Preparar 1x Una-Phor-All Buffer PLUS (OPA), a partir de un tampón 10x OPA PLUS solución.
- Preparar 0,2 U / l de DNasa I (dilución de 5 veces de 1 U / l de DNasa I).
- Prepare una mezcla maestra de fragmentación de la siguiente manera (dada por una sola reacción):
Reactivo Volumen 10X One-Phor-All Buffer PLUS 3.6 l DNasa I (0,2 U / l) 3 l - Añadir 6,6 l de fragmentación Mix a los 30 l de sscDNA.
- Centrifugado y se incuba a 37 ° C durante 10 min, a continuación inactivar la DNasa I a 95 ° C durante 10 min y mantener en hielo. Alícuota 1 &# 181; l del cDNA fragmentada de tamaño basada Agilent verificación distribución.
- sscDNA verificación distribución de tamaño (Figura 4C). Compruebe la distribución del tamaño sscDNA mediante un Bioanalyzer (Agilent). La distribución del tamaño de cDNA fragmentada debe típicamente comprendida entre 35 y 200 bases.
7. Etiquetado de Fragmentado sscDNA
- Diluir la DLR-1a 7,5 mM a 5 mM en agua DEPC.
- Prepare una mezcla maestra de etiquetado de la siguiente manera (dada por una sola reacción):
Reactivo Volumen Tampón de reacción 5x TdT 14 l CoCl2 (25 mM) 14 l DLR-1a (5 mM) 1 l Transferasa terminal (400 U / l) 4.4 l - Añadir 33,4 l de la mezcla de etiquetadoa cada muestra de cDNA fragmentado.
- Mezclar con un movimiento rápido del tubo, girar brevemente y se incuba a 37 ° C durante 60 minutos, a continuación, mantener en hielo.
8. La hibridación con la micromatriz HERV chip
- Prehumedecido el GeneChip HERV con 200 l de prehibridación Mix (Affymetrix) y se incuba a 50 ° C, 60 rpm, durante 10 min.
- Preparar la mezcla de hibridación de la siguiente manera (dado para una sola reacción):
Reactivo Volumen Control de Oligo B2 (3 nM) 3.3 l 20x Eukaryotic hibridación de control 10 l 2x Hibridación Mix 100 l 99,9% de DMSO 17,7 l - Añadir los 131 l de mezcla de hibridación a los 69 l de fragmentada y ADNc marcado a temperatura ambiente para hacer que un VO definitivaLume de 200 l.
- Mezclar y desnaturalizar durante 2 min a 95 ° C, a continuación se incuba a 50 ° C durante 5 min y se centrifuga a velocidad máxima durante 5 min.
- Vacíe el GeneChip HERV prehumedecida y cargar el objetivo de preparación 200 l. Aplique TOUGH-manchas en los dos tabiques.
- Hibridar a 50 º C, 60 rpm, durante 18 h.
- Después de 18 horas, vaciar el GeneChip HERV y almacenar la solución de hibridación recogido a 4 ° C. Llene el conjunto de sondas con 250 l de tampón de lavado A. Si las fichas no se ejecutan inmediatamente en los fluidos, almacenadas a 4 ° C.
9. Lavado y tinción
- Ejecute las Fluidics la barra de menús del SMOC. En el cuadro de diálogo de fluidos, seleccionar la emisora de interés (1 - 4), a continuación, seleccione Shutdown_450 para todos los módulos, a continuación, ejecute. Sumerja las 3 líneas de aspiración Técnica de fluidos en agua Milli-Q. Siga las instrucciones de la pantalla LCD.
- Aplicar el programa Prime_450 a todos los módulos. Coloque el tubo de tampón de lavado A enuna botella que contiene 400 ml de tampón de lavado A, y el otro para el tampón de lavado B en un frasco con 200 ml de tampón de lavado B. Por otra parte, siga las instrucciones de la pantalla LCD.
- Levante las agujas y colocar cóctel 600 l mancha 1 (SAPE Solución Mix) y 600 l de cóctel mancha 2 (solución de anticuerpos Mix) que contienen tubos de microcentrífuga en las posiciones # 1 y # 2, y 800 l arreglo que contiene solución tampón en la posición # 3 .
- Asigne el chip derecho de cada módulo, seleccione el protocolo FS450-004 y ejecutar cada módulo, siguiendo las instrucciones que aparecen en pantalla.
10. Exploración
- Caliente el escáner GS3000. Está listo para escanear cuando la luz cambie a verde.
- Aplicar difíciles-spots en los tabiques para evitar fugas a continuación, cargar el chip en el cargador automático o bien directamente en el escáner. Inicie el escaneo.
- Después de escanear el chip,. Se generan archivos cel. Compruebe la imagen y alinear la cuadrícula para el punto de identificar las celdas sonda ( Figuras 4D-F).
11. Análisis de Datos
- El control de calidad. Se refiere al criterio Affymetrix controles para verificar que las fichas HERV-V2 cumplen con los criterios de control de calidad. Para ello, las siguientes representaciones se pueden utilizar: la distribución de valores de intensidad log (densidad de parcelas y diagramas de caja), la desviación media absoluta (MAD) versus la mediana intensidad (MAD-Med) parcelas, las parcelas de fondo, el error estándar sin escala normalizada (nuse) parcelas y la expresión log relativa (RLE) parcelas.
- La normalización. Además, el conjunto de datos debe ser explorado para poner de relieve los efectos inesperados de lote y para corregirlos antes del análisis estadístico. Así pues, el preprocesamiento de datos incluye una corrección de fondo (por ejemplo. Basado en la señal de línea de base de la sonda triptófano), seguido por la normalización de RMA y resumen 15.
- La minería de datos y la búsqueda de genes diferenciales expresado. Normalizar las virutas y aplicar una clusteri jerárquicaenfoque ng para explorar el conjunto de datos (Figura 6A). A continuación, realice una búsqueda de genes expresados diferencialmente (DEG) por el uso de un análisis clásico significativa de microarrays (SAM) procedimiento 16 seguido de una tasa de falso descubrimiento (FDR) de corrección 17. Tenga en cuenta que estos pasos se integran plenamente en algunas suites de software de análisis como Partek GS pero, alternativamente, se pueden realizar utilizando el software estadístico R 18 con los conjuntos del proyecto Bioconductor 19. Tras el análisis estadístico, se filtra el conjunto de datos para excluir a los probesets para los que los valores de expresión son menos de 2 6.
- La visualización y la interpretación. Interpretar los resultados de los microarrays HERV-V2 en una interfaz dedicada utilizando bases de datos de anotación.
Resultados
El valor de los estudios de transcriptómica reside principalmente en la calidad del material biológico de partida. Si la extracción de ARN se lleva a cabo en condiciones óptimas, el Número de la integridad del ARN (RIN) es típicamente de 7 o mayor (Figura 4A). La necesidad de hibridar 2 g de ADNc en el chip Affymetrix HERV-V2 implica el uso de un proceso de amplificación. Una etapa de amplificación exitosa conduce a una distribución en forma de campana (figura 4B). Entonces, la fragmentación Dnase1 se lleva a cabo con el fin de homogeneizar la distribución de tamaño de alrededor de 100 nucleótidos de ADNc antes de la hibridación (Figura 4C). Después de la hibridación y de barrido (Figura 4D), una inspección visual de la imagen permite a uno para comprobar si la red está bien alineado con los puntos (Figura 4E) y si los controles de hibridación son consistentes (Figura 4F). Este paso también es útil con el fin de excluir a los microarrays en el que las burbujas de aire o errores ocurrido durante el experimento.
Una vez que las fichas han pasado QC (Figura 5) y después de la normalización, el análisis estadístico de los 5 partidos par tumoral y normal de las muestras de ARN de la próstata en el Hospital de Lyon-Sud llevado a la identificación de 207 probesets HERV con los valores de expresión diferencial (p.val <0,05) (Figura 6A). Para apoyar estos registros y para obtener información específico de la próstata, se añadieron 35 muestras partido pares adicionales (colon, ovario, testículo, mama, pulmón y próstata) para el análisis y el procedimiento SAM-FDR (Franklin Delano Roosevelt = 20%) finalmente identificado 44 prostáticos probesets HERV específicos. Entre ellos, las 10 estructuras HERV más relevantes se describen (Figura 6B). Se necesitan más estudios clínicos para evaluar los valores de sensibilidad y especificidad de estos biomarcadores candidatos.
pload/50713/50713fig1.jpg "width =" 500px "/>
. Figura 1 Esquema del procedimiento general de la clínica (1: prostatectomía por el médico y la preparación del tejido por el patólogo) al banco (2-6: preparación de muestras, la preparación de destino, el procesamiento de microarrays) que conduce a la identificación de biomarcadores candidatos (7: análisis de la bioinformática de los microarrays HERV). Los ácidos nucleicos derivados de tejido normal se representan en color naranja; ácidos nucleicos derivados de área tumoral se componen de una mezcla de la normalidad (naranja) y específicos del tumor (negro) los ácidos nucleicos. Haga clic aquí para ver la imagen más grande .
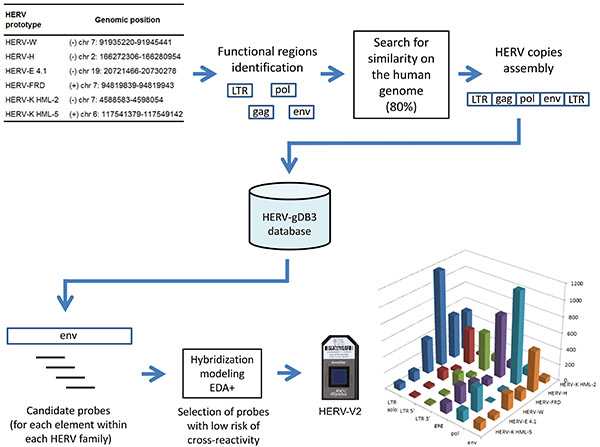
. Figura 2 Concepción y el contenido del chip de HERV-V2: secuencias HERVobtenidos del genoma humano se almacenan en una base de datos llamada HERV-gDB3, entonces las sondas candidatos 25-mer pasan a través de un procedimiento de modelado hibridación dedicado (EDA +) antes de ser finalmente sintetizado en la matriz (las sub-regiones seleccionadas resultantes se representan para cada familia). Haga clic aquí para ver la imagen más grande .
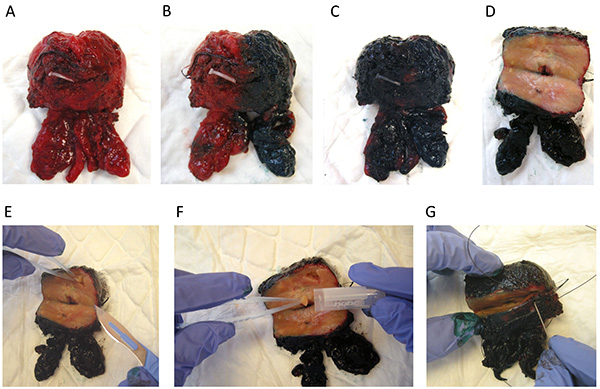
Figura 3. Manejo de próstata por el patólogo. (A) muestra la prostatectomía radical fresca se transfiere al laboratorio. (BC) La próstata está manchada (verde en el lado derecho, negro en la parte izquierda). (D) la sección transversal grande de la glándula en el lado posterior. (E) Dejando al margen intacto, piecES de tejidos se disecan a partir de diferentes áreas de la glándula de la próstata. (F) Los núcleos de tejido se colocan en un tubo Eppendorf. (G) hilo de sutura se utiliza para cerrar la próstata y para evitar la distorsión de la glándula y una interrupción mínima del margen quirúrgico. Entonces, el espécimen de prostatectomía radical está listo para la fijación en formol de acuerdo con el procedimiento habitual para el análisis histológico. Haga clic aquí para ver la imagen más grande .
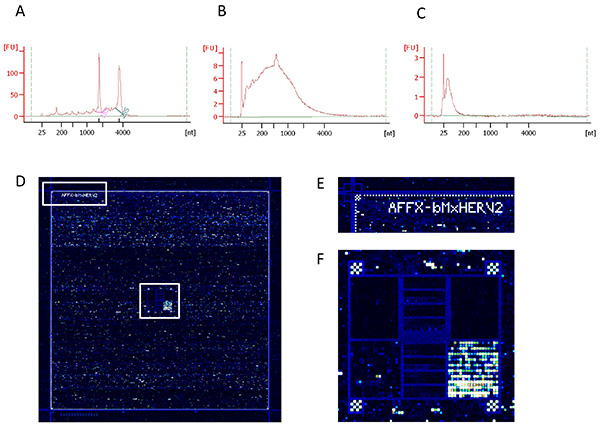
Controles de la Figura 4. Calidad de la preparación de ácido nucleico y la eficiencia de hibridación. (A) la integridad del ARN, (B) de ADNc de objetivos y (c) objetivos fragmentados utilizadas en la etapa de hibridación amplificada. ThESE tres controles de calidad se obtuvieron con el Bioanalyzer utilizando chips de nano ARN y el ensayo del eucariota Nano Serie II. (D) la imagen general de la zona HERV-V2 microarrays de hibridación después de la digitalización, (E) la ampliación de las esquinas superior izquierda que muestra los controles de alineación de cuadrícula y (F) ampliación de la zona central que muestra la detección de controles de hibridación. Haga clic aquí para ver la imagen más grande .
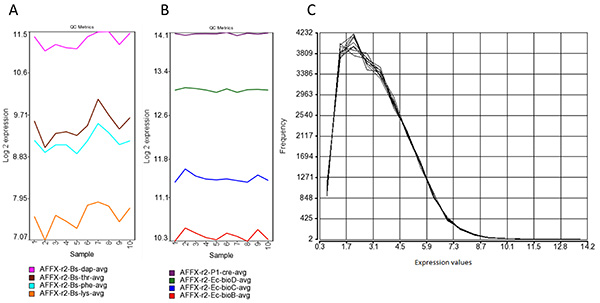
Figura 5. Procesamiento de señales. (A) de Affymetrix polyA espiga-en los controles de amplificación. El poli controla Dap, Thr, Phe y Lys transcripciones de B. genes subtilis se dispararon en la muestra de RNA y sirven para evaluar el éxito general de los pasos de preparación de destino. La intensidad debe ser detectado con valores decrecientes de entre estos controles pico-a para asegurarse de que no había ningún sesgo durante la amplificación WT-Ovation entre altamente y genes de baja-expresado. (B) de Affymetrix espiga-en los controles de hibridación. Estos objetivos aislados a partir de E. coli y bacteriófago P1 se dispararon antes de que el procedimiento de etiquetado. El aumento de los valores de BioB, BioC, bioD y Cre indican el éxito general de la hibridación. (C) la distribución de intensidad de las señales de chips después de la normalización de RMA. La mayoría de los probesets señales de exhibición con valores inferiores a 2 6 (al fondo), lo que indica una expresión general restringido principalmente a algunos loci HERV específico. Haga clic aquí para ver la imagen más grande .
ftp_upload/50713/50713fig6.jpg "width =" 500px "/>
Figura 6. Análisis de los datos. (A) Análisis de agrupación jerárquica de las muestras normales y tumorales. Agrupación de particionamiento se aplicó a los valores de expresión normalizados utilizando un algoritmo de función de distancia euclidiana, agrupando probesets en un máximo (rojo) - y hacia abajo (azul)-regulación entre muestras normales y tumorales. (B) La selección de los 10 mejores estructuras HERV identificados como biomarcador candidato del cáncer de próstata. Para cada elemento HERV, la familia HERV relacionado, se dan las coordenadas genómicas (NCBI 36/hg18) y una breve descripción de la estructura HERV. Haz clic aquí para ver la imagen más grande .
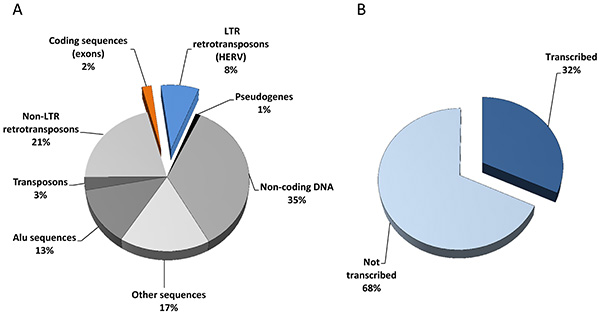
Figura 7. El repertorio HERV. (A) La secuenciación del genoma humano reveló 25.000 genes codificadores de proteínas (exones, 2%) y una gran cantidad de elementos de transposición, incluyendo 200.000 de larga repetición terminal (LTR) retrotransposones (HERV, 8%). (B) La extrapolación de HERV-V2 contenido de chip y los datos de expresión asociados (79 muestras procedentes de 8 normales frente a los tipos de tejidos tumorales) sugieren que una tercera parte del repertorio HERV es transcripcionalmente activo. Haga clic aquí para ver la imagen más grande .
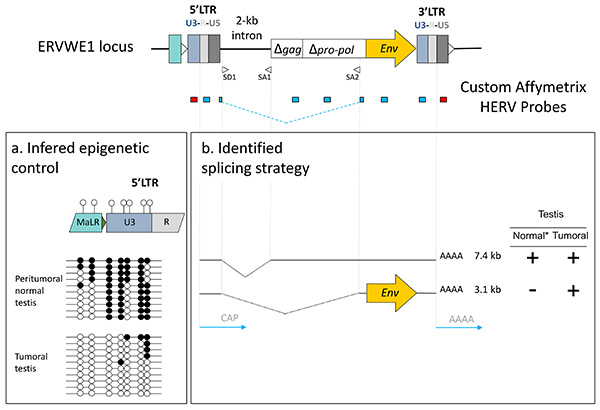
Figura 8. Interpretación funcional de señales desde el chip. (A) la identificación y el control epigenético Promotor: señal negativa U3 (sonda roja, 5'LTR) Versus señal positiva R-U5 (azul sonda, 5'LTR) sugieren la transcripción U3-impulsado, con el apoyo de los diferentes metilación CpG (círculos negros sólidos) contenido de U3 en peritumoral tejidos normales frente tumorales. (B) La estrategia de fusión: la supuesta dotación de 3,1 kb que codifica ARNm expresa exclusivamente en el tumor se identifica usando SD1/SA2 empalme sonda superpuestas. * Deducida por la comparación con otros tejidos no placentarios. Haz clic aquí para ver la imagen más grande .
Discusión
Durante los últimos 10 años, la mayor parte de los intentos para la medición de la expresión de HERV se han utilizado técnicas de RT-PCR, ya sea para centrarse en un locus específico 20-24 o basado en la conservación relativa de los genes pol para evaluar las tendencias generales dentro de HERV géneros 25,26 . Además, las amplificaciones de PCR usando cebadores altamente degenerados junto con microarrays de baja densidad destinados a detectar y cuantificar la expresión de las familias HERV 27,28. Con el fin de rastrear la expresión de locus individual dentro de una familia, los enfoques basados en la amplificación por PCR de regiones conservadas en combinación con la posterior clonación y secuenciación habilitar distintos elementos transcripcionalmente activos de los HML-2 29,30 o HERV-E4.1 31 familias a ser identificado. También termina por etapas de clonación y secuenciación, la técnica de monitorización expresión repetida del genoma con el objetivo de identificar a los promotores entre repeticiones identificados activos HML-2 LTR solitarios humanos específicos 32,33. Hemos desarrollado sucesivamente dos generaciones de micromatrices de alta densidad dedicados al análisis del transcriptoma de HERV, la introducción de metodologías adecuadas para el diseño de la sonda elemento que se repite con el fin de minimizar las reacciones cruzadas entre los elementos parálogos dentro de una familia 34,35. El chip HERV-V2 que se enfoca en 2690 provirus distintas y 2883 LTR en solitario de los HERV-W, HERV-H, HERV-E 4.1, HERV-FRD, HERV-K HML-2 y HERV-K HML-5 familias, dio a conocer el expresión de 1718 HERV loci (Figuras 7A y B) en una amplia gama de tejidos 35, ilustrados en este documento por la identificación de biomarcadores putativos de cáncer de próstata. Además, el uso de múltiples probesets en un locus dado es informativo acerca de su regulación transcripcional. En primer lugar, una señal negativa U3 en conjunción con uno positivo U5 clasifica la LTR como un promotor, y señales negativas a la inversa U3 y U5 positivos puede reflejar un papel de poliadenilación. Así, identified 326 promotor LTR en una amplia gama de tejidos 35 y, basándose en esta información dicotómica U3-U5 proporcionada por la matriz, propusimos y experimentalmente confirmada para algunos casos seleccionados que dicha transcripción autónoma fue controlada por un proceso epigenético de metilación dependientes 34 (Figura 8). En segundo lugar, la detección de señales de, por ejemplo LTR, gag y env probesets independientes o emitidos desde las sondas dirigidas a empalme específica es informativo acerca de la estrategia de empalme proviral, como lo ilustra el perfil de expresión ERVWE1/Syncytin1 en la placenta o en el testículo tumoral 34. Esto indica que el proceso de selección de la sonda específica de HERV es lo suficientemente robusta como para soportar la identificación de la estrategia de corte y empalme asociado de tejido, la manera más eficiente para los genes convencionales 36 (Figura 8).
Este método es el primer intento de identificar la expresión locus HERV de forma individualutilizando un microarray personalizada de alta densidad basada en la tecnología de Affymetrix. Las ventajas claramente identificados de la formato de micromatriz para descifrar transcriptoma HERV consiste en (i) la exploración coordinada de varias familias HERV y (ii) el análisis simultáneo e independiente de las diferentes regiones para cada locus, por ejemplo. U3 y U5 dominios para solista y LTR proviral, regiones gag o env y posibles uniones empalmadas asociados con estructuras proviral, sin ningún a priori sobre la funcionalidad del elemento HERV. Las perspectivas se basan en una mejora de las anotaciones de las herramientas bioinformáticas microarrays asociada. Esto debería permitir a uno para convertir las señales de chips en hipótesis biológicas tales como si HERVs activos evidenciados en coche lncRNA transcripción o modulan más o menos proximal codificación de la expresión génica. De hecho, tal hipótesis está apoyada por estudios recientes que identificaron próstata transcripciones ncRNA casos de cáncer asociados con componentes de vORFs víricas de la familia HERV-K endógena retrovirus o porciones de una región promotor LTR viral 37, así como dos eventos de fusión de genes a saber HERV-K22q11-Etv1 y HERV-K17-ETV 38,39. En su conjunto, este enfoque transcriptoma todo combinado con la función de LTR y las identificaciones de estrategia de empalme puede ayudar a descifrar el marcador frente a los componentes de activación de la expresión HERV en crónica y las enfermedades infecciosas 40,41 42,43.
Divulgaciones
Este trabajo fue apoyado por bioMérieux SA, Hospices Civils de Lyon y la agencia pública francesa OSEO (Diagnóstico avanzado de enfoques en Nueva terapéuticas, un programa financiado por el gobierno francés dedicado a la medicina personalizada). PP, VC, GO, NM, y FM son empleados de bioMérieux SA. PP, NM y FM han presentado solicitudes de patente de las conclusiones de este trabajo.
Agradecimientos
Agradecemos Cecile Montgiraud, Juliette Gimenez, Magali Jaillard y Bertrand Bonnaud por su contribución al desarrollo inicial y la optimización del protocolo HERV-V2. También queremos dar las gracias a Hader Haidous por su orientación sobre consideraciones éticas.
Materiales
| Name | Company | Catalog Number | Comments |
| Trizol | Invitrogen | 15596-026 | |
| RNA poly-A control stock | Affymetrix | 900433 | |
| DNAse 1 | Promega | M6101 | 1,000 U (1 U/µl) |
| Terminal transferase | Roche | 3333574001 | 400 U. Including enzyme and coenzyme (CoCl2). |
| DLR-1a | Affymetrix | 900542 | |
| Hybridization internal controls B2 and 20x Eukaryotic Hybridization Control | Affymetrix | 900454 | |
| GeneChip Hybridization, Wash and staining | Affymetrix | 900720 | Including PreHybridization Mix and 2x Hybridization Mix for 30 reactions |
| 10x One-Phor-All Buffer PLUS | Composition in DEPC-treated water: 100 mM Tris-acetate pH 7.5; 100 mM magnesium acetate; 500 mM potassium acetate. | ||
| RNeasy Mini kit | Qiagen | 74104 | RNA cleanup protocol |
| WT-Ovation RNA amplification system | Nugen | 2210-24 | |
| QIAquik PCR purification kit | Qiagen | 28104 | |
| EQUIPMENT | |||
| Material Name | Company | Catalogue Number | Comments |
| Nanodrop 1000 | Thermo Scientific | ||
| GeneChip Scanner 3000 7G | Affymetrix | GS30007G | Optional: autoloader |
| GeneChip Fluidics Station 450 | Affymetrix | FS450 | |
| GeneChip Hybridization 640 Oven | Affymetrix | 640 | Includes 4 GeneChip Probe array carriers |
| Workstation loaded with GeneChip Operating Software (GCOS) including the GeneChip Scanner 3000 High-Resolution Scanning Patch | |||
| HERV-V2 chip | Affymetrix | Custom array. For microarray availability (for research use only), please contact: François Mallet Laboratoire Commun de Recherche Hospices Civils de Lyon-bioMérieux Medical Diagnostic Discovery Department Centre Hospitalier Lyon Sud, Bâtiment 3F 69495, Pierre Bénite cedex France Phone: 33 (0)4 72 67 87 85 Email: francois.mallet@biomerieux.com | |
| HERV-V2 conception Dedicated database and annotations The construction of a dedicated database, grouping genomic HERV sequences belonging to 6 HERV families, has been achieved by the following procedure: (i) the most complete and representative sequence of each HERV family was selected from the literature and defined as a prototype sequence (Figure 2). (ii) The 6 prototypes were functionally annotated with reference to their LTR (U3/R/U5) and internal parts (gag/pol/env). (iii) RepeatMasker 44 was then applied using these functional sequences as input libraries. A genome-wide search of all related sequences was performed over the human genome on the basis of a minimum 80% homology (NCBI 36/hg18). (iv) Finally, the functional sequences retrieved by this process were assembled into distinct loci on the basis of their genomic location and eventually implemented in a dedicated HERV database. This database, called HERV-gDB3, contains 10,035 individual HERV loci35. Locus-specific probes design Starting from HERV-gDB3, overlapping tracks of 25-mer candidate probes were firstly generated. Each candidate probe was then aligned against the human genome using KASH 45 in order to assess the cross-hybridization potentialities. This latter estimation was performed by a model developed specifically for this purpose and referred to as EDA+. Briefly, the principle of EDA+ is to take into account the instability brought by mismatches and gaps in a 25-mer target/probe hybridization complex. Candidate probes exhibiting low cross-hybridization risks (i.e. a low number of non-specific genomic targets) are selected and lastly assembled into probesets. Custom HERV GeneChip microarray The custom HERV GeneChip integrates 23,583 HERV probesets and can discriminate 5,573 distinct HERV elements, composed of solo LTRs, complete and partial proviruses (Figure 2). The standard Affymetrix control probes for unbiased amplification and hybridization were also included in the microarray. | |||
Referencias
- Artibani, W. Landmarks in prostate cancer diagnosis: the biomarkers. BJU Int. 110, Suppl 1. 8-13 (2012).
- Girometti, R., et al. Negative predictive value for cancer in patients with "gray-zone" PSA level and prior negative biopsy: preliminary results with multiparametric 3.0 Tesla MR. J. Magn. Reson. Imaging. 36, 943-950 (2012).
- You, B., et al. Prognostic value of modeled PSA clearance on biochemical relapse free survival after radical prostatectomy. Prostate. 69, 1325-1333 (2009).
- Vickers, A. J., Brewster, S. F. PSA Velocity and Doubling Time in Diagnosis and Prognosis of Prostate Cancer. Br. J. Med. Surg. Urol. 5, 162-168 (2012).
- Bussemakers, M. J., et al. DD3: a new prostate-specific gene, highly overexpressed in prostate cancer. Cancer Res. 59, 5975-5979 (1999).
- Vlaeminck-Guillem, V., Ruffion, A., Andre, J., Devonec, M., Paparel, P. Urinary prostate cancer 3 test: toward the age of reason. Urology. 75, 447-453 (2010).
- Buscher, K., et al. Expression of the human endogenous retrovirus-K transmembrane envelope, Rec and Np9 proteins in melanomas and melanoma cell lines. Melanoma Res. 16, 223-234 (2006).
- Ishida, T., et al. Identification of the HERV-K gag antigen in prostate cancer by SEREX using autologous patient serum and its immunogenicity. Cancer Immun. 8, 15(2008).
- Sauter, M., et al. Human endogenous retrovirus K10: expression of Gag protein and detection of antibodies in patients with seminomas. J. Virol. 69, 414-421 (1995).
- Pérot, P., Bolze, P. A., Mallet, F. Viruses: Essential Agents of Life. Witzany, G. , Springer-Verlag. 325-361 (2012).
- Stamey, T. A., Kabalin, J. N. Prostate specific antigen in the diagnosis and treatment of adenocarcinoma of the prostate. I. Untreated patients. Untreated patients. J. Urol. 141, 1070-1075 (1989).
- Catalona, W. J., et al. Measurement of prostate-specific antigen in serum as a screening test for prostate cancer. New Engl. J. Med. 324, 1156-1161 (1991).
- Schroder, F. H. Prostate cancer around the world. An overview. Urol. Oncol. 28, 663-667 (2010).
- Klotz, L. Active surveillance for favorable-risk prostate cancer: background, patient selection, triggers for intervention, and outcomes. Curr. Urol. Rep. 13, 153-159 (2012).
- Irizarry, R. A., et al. Exploration, normalization, and summaries of high density oligonucleotide array probe level data. Biostatistics. 4, 249-264 (2003).
- Tusher, V. G., Tibshirani, R., Chu, G. Significance analysis of microarrays applied to the ionizing radiation response. Proc. Natl. Acad. Sci. U.S.A. 98, 5116-5121 (2001).
- Storey, J. D., Tibshirani, R. Statistical significance for genomewide studies. Proc. Natl. Acad. Sci. U.S.A. 100, 9440-9445 (2003).
- Team, R. D. C. R: A language and environment for statistical computing. , (2008).
- Gentleman, R. C., et al. Bioconductor: open software development for computational biology and bioinformatics. Genome Biol. 5, R80(2004).
- de Parseval, N., Lazar, V., Casella, J. F., Benit, L., Heidmann, T. Survey of human genes of retroviral origin: identification and transcriptome of the genes with coding capacity for complete envelope proteins. J. Virol. 77, 10414-10422 (2003).
- Wang-Johanning, F., et al. Detecting the expression of human endogenous retrovirus E envelope transcripts in human prostate adenocarcinoma. Cancer. 98, 187-197 (2003).
- Smallwood, A., et al. Temporal regulation of the expression of syncytin (HERV-W), maternally imprinted PEG10, and SGCE in human placenta. Biol. Reprod. 69, 286-293 (2003).
- Okahara, G., et al. Expression analyses of human endogenous retroviruses (HERVs): tissue-specific and developmental stage-dependent expression of HERVs. Genomics. 84, 982-990 (2004).
- Buscher, K., et al. Expression of human endogenous retrovirus K in melanomas and melanoma cell lines. Cancer Res. 65, 4172-4180 (2005).
- Forsman, A., et al. Development of broadly targeted human endogenous gammaretroviral pol-based real time PCRs Quantitation of RNA expression in human tissues. J. Virol. Methods. 129, 16-30 (2005).
- Muradrasoli, S., Forsman, A., Hu, L., Blikstad, V., Blomberg, J. Development of real-time PCRs for detection and quantitation of human MMTV-like (HML) sequences HML expression in human tissues. J. Virol. Methods. 136, 83-92 (2006).
- Seifarth, W., et al. Comprehensive analysis of human endogenous retrovirus transcriptional activity in human tissues with a retrovirus-specific microarray. J. Virol. 79, 341-352 (2005).
- Pichon, J. P., Bonnaud, B., Mallet, F. Quantitative multiplex degenerate PCR for human endogenous retrovirus expression profiling. Nat. Protoc. 1, 2831-2838 (2006).
- Flockerzi, A., et al. Expression pattern analysis of transcribed HERV sequences is complicated by ex vivo recombination. Retrovirology. 4, 39(2007).
- Flockerzi, A., et al. Expression patterns of transcribed human endogenous retrovirus HERV-K(HML-2) loci in human tissues and the need for a HERV Transcriptome Project. BMC Genomics. 9, 354(2008).
- Gosenca, D., et al. HERV-E-Mediated Modulation of PLA2G4A Transcription in Urothelial Carcinoma. PLoS ONE. 7, e49341(2012).
- Buzdin, A., Kovalskaya-Alexandrova, E., Gogvadze, E., Sverdlov, E. GREM, a technique for genome-wide isolation and quantitative analysis of promoter active repeats. Nucleic Acids Res. 34, e67(2006).
- Buzdin, A., Kovalskaya-Alexandrova, E., Gogvadze, E., Sverdlov, E. At least 50% of human-specific HERV-K (HML-2) long terminal repeats serve in vivo as active promoters for host nonrepetitive DNA transcription. J. Virol. 80, 10752-10762 (2006).
- Gimenez, J., et al. Custom human endogenous retroviruses dedicated microarray identifies self-induced HERV-W family elements reactivated in testicular cancer upon methylation control. Nucleic Acids Res. 38, 2229-2246 (2010).
- Pérot, P., et al. Microarray-based sketches of the HERV transcriptome landscape. PLoS ONE. 7, e40194(2012).
- Fehlbaum, P., Guihal, C., Bracco, L., Cochet, O. A microarray configuration to quantify expression levels and relative abundance of splice variants. Nucleic Acids Res. 33, e47(2005).
- Prensner, J. R., et al. Transcriptome sequencing across a prostate cancer cohort identifies PCAT-1, an unannotated lincRNA implicated in disease progression. Nat. Biotechnol. 29, 742-749 (2011).
- Tomlins, S. A., et al. Distinct classes of chromosomal rearrangements create oncogenic ETS gene fusions in prostate cancer. Nature. 448, 595-599 (2007).
- Hermans, K. G., et al. Truncated ETV1, fused to novel tissue-specific genes, and full-length ETV1 in prostate cancer. Cancer Res. 68, 7541-7549 (2008).
- Brodziak, A., et al. The role of human endogenous retroviruses in the pathogenesis of autoimmune diseases. Med. Sci. Monit. 18, RA80-RA88 (2012).
- Mullins, C. S., Linnebacher, M. Human endogenous retroviruses and cancer: Causality and therapeutic possibilities. World J. Gastroenterol. 18, 6027-6035 (2012).
- Young, G. R., et al. Resurrection of endogenous retroviruses in antibody-deficient mice. Nature. 491, 774-778 (2012).
- Vander Kuyl, A. C. HIV infection and HERV expression: a review. Retrovirology. 9, 6(2012).
- Smit, A. F. A., Hubley, R. RepeatMasker Open-3.0. , (1996).
- Navarro, G., Raffinot, M. Flexible Pattern Matching in Strings: Practical On-Line Search Algorithms for Texts and Biological Sequences. , Cambridge University Press. (2002).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados