Method Article
Un método de monitoreo asequible VIH-1 Resistencia a los medicamentos para los entornos con recursos limitados
En este artículo
Resumen
Pruebas de resistencia a los medicamentos para el VIH-1 en individuos infectados no respondían al tratamiento antirretroviral (ART) puede guiar futuras terapias y mejorar los resultados del tratamiento. Optimización de los resultados de salud individual y poblacional en la alta prevalencia del VIH, sino entornos con recursos limitados, en última instancia requerirá genotipado resistencia a los medicamentos asequibles y accesibles y los métodos de interpretación.
Resumen
VIH-1 resistencia a los medicamentos tiene el potencial de comprometer seriamente la eficacia y el impacto de la terapia antirretroviral (ART). Como los programas de TAR en África subsahariana siguen expandiéndose, las personas en tratamiento antirretroviral deben ser estrechamente monitorizados para la aparición de resistencia a los medicamentos. La vigilancia de la farmacorresistencia transmitida para rastrear la transmisión de cepas virales resistentes a los antirretrovirales ya es también crítico. Desafortunadamente, las pruebas de resistencia a los medicamentos aún no es de fácil acceso en los entornos con recursos limitados, ya que la genotipificación es caro y requiere de laboratorio sofisticado y la infraestructura de gestión de datos. Un acceso abierto método de seguimiento de la resistencia genotípica al fármaco para gestionar personas y evaluar la resistencia a fármacos de transmisión se describe. El método utiliza el software libre de código abierto para la interpretación de los patrones de resistencia a los medicamentos y la generación de informes individuales de los pacientes. El protocolo de determinación del genotipo tiene una tasa de amplificación de más de un 95% para las muestras de plasma con AVcarga iral> 1000 VIH-1 RNA copias / ml. La sensibilidad disminuye significativamente para cargas virales <1.000 copias de ARN del VIH-1 / ml. El método descrito aquí se valida con un método para el VIH-1 resistencia a los fármacos de ensayo aprobados por la Food and Drug Administration de los Estados Unidos (FDA), el método de genotipificación ViroSeq. Limitaciones del método descrito aquí incluyen el hecho de que no está automatizado y que también falló para amplificar la forma circulante CRF02_AG recombinante a partir de un panel de validación de muestras, aunque se amplificó subtipos A y B de la misma panel.
Introducción
La epidemia de VIH en el sur de África ha ido evolucionando rápidamente 1 con un aumento exponencial concomitante en los individuos en terapia antirretroviral (ART), especialmente en el sur de África 2, 3. Como evidencia del impacto epidemiológico de los programas de tratamiento a gran escala en la reducción de la incidencia de 4 y el aumento de la esperanza de vida en los entornos con recursos limitados (RLS) 5 continúa acumulándose, se intensificarán los esfuerzos para aumentar la cobertura de ART. La evolución de las directrices para el uso del tratamiento como herramienta de prevención 6, 7, bajo la prueba y los programas de tratamiento significa que el número absoluto de personas en tratamiento se incrementará aún más. Un gran número de personas estarán en ART durante períodos más largos de tiempo ya que la esperanza media de vida de las personas en tratamiento antirretroviral se acerca a la de la población no infectada por VIH 8. El desarrollo y la transmisión de la farmacorresistencia del VIH tiene always sido considerado una amenaza para los logros de la ART 12.9. Por lo tanto, hay una necesidad de más rigurosa vigilancia y el control de la resistencia a fármacos como se inician más individuos en ART.
Efectuar pruebas de resistencia genotípica de drogas (TRB) se ha utilizado con éxito en los países desarrollados, tanto para la vigilancia y el monitoreo del VIH-1 resistencia a los medicamentos en las personas que reciben tratamiento antirretroviral. En esta configuración, GRT se ha integrado en la continuidad de la atención para el VIH-1 en individuos infectados. La mayoría de las guías internacionales recomiendan TRB para adultos y pacientes pediátricos en su defecto ART (de primera línea y de segunda línea) 13-15, los pacientes pediátricos expuestos a la prevención de la transmisión materno-infantil (PTMI) regímenes pero posteriormente infectan 16, y en entornos con altos niveles de resistencia a los medicamentos de transmisión entre las personas con infección aguda 13-15. Sin embargo, los requisitos de coste, la tecnología y la infraestructura han limitado la implementaciónción de enfoques similares para la supervisión resistencia a los medicamentos en el SPI.
El tratamiento de Sudáfrica el VIH y las directrices de seguimiento no se recomienda actualmente el uso de toneladas de registro bruto en la orientación de la elección de la terapia antirretroviral para las personas que fallan regímenes de primera línea 17. Las personas se conectan basado principalmente en virológica parámetros (VIH-1 RNA carga viral). Sin embargo en 2012, el sur de África Los clínicos Sociedad VIH publicó las primeras directrices de las pruebas de resistencia de drogas ARV África del Sur 18. Estas directrices recomiendan las pruebas TRB para todos los adultos en su defecto de primera línea y de segunda línea y para los bebés y niños infectados expuestos a pMTCT 18. Sin embargo, GRT, no se recomienda para las personas de 18 infectados en forma aguda debido a que no hay evidencia actual de altos niveles de resistencia a los medicamentos de transmisión en el sur de África 19-29. Se espera que algunas de estas recomendaciones se integrarán con el tiempo en el tre nacionaldirectrices tamiento y seguimiento de los distintos países de la región. Ya en los 2013 las directrices de tratamiento de Sudáfrica ahora hay recomendación del GRT en el momento del fallo de segunda línea para adultos y al momento de la falla régimen basado en IP de primera o de segunda línea para los niños 30.
Se ha demostrado que la incorporación de TRB en las guías de tratamiento en Sudáfrica sería potencialmente costo-neutral. Teniendo en cuenta el costo de los medicamentos de segunda línea régimen que son relativamente más caros que los fármacos de primera línea, utilizando TRB para identificar a los pacientes que realmente necesitan ser cambiado a la terapia de segunda línea no supondrá ningún coste adicional para el programa. Además, GRT también puede identificar otras razones para el fracaso, la conservación de las opciones de tratamiento y generar información sobre los patrones de resistencia emergentes 31. Por lo tanto, es necesario para reducir el coste de los métodos de monitoreo de resistencia a fármacos aún más con el fin de mejorar el acceso, la calidad de un cuidadolos resultados d.
A continuación, presentamos un método TRB destinados a utilizar (de código abierto) cebadores genéricos para la transcripción reversa, reacción en cadena de la polimerasa (PCR) y la secuenciación (Tabla 1), así como software de código abierto en su mayoría para la interpretación resistencia a los medicamentos. Para la gestión clínica, el protocolo se complementa con un método integral y notificando a la interpretación especialista del informe resistencia a los medicamentos de laboratorio con una estrecha adhesión a las directrices nacionales de tratamiento. El protocolo se divide en cuatro componentes diferentes: 1) Ácido VIH ribonucleico (ARN) Extracción, 2) transcripción inversa y reacción en cadena de la polimerasa (PCR) de amplificación de dianas virales, 3) Secuenciación y 4) los métodos de bioinformática para el análisis de los cromatogramas, alineación, curación y la interpretación de los datos de secuencia.
Protocolo
1. Ácido etilendiaminotetraacético (EDTA) Procesamiento de sangre entera
Nota: La sangre puede ser procesado inmediatamente después de la toma de se puede almacenar a 4 ° C durante no más de 24 horas.
- Trabajar en un gabinete de bioseguridad, permita que el EDTA muestra de sangre total alcance la temperatura ambiente.
- Para cada muestra, etiquetar suficientes crioviales con la identificación de la muestra (ID), material de almacenamiento (plasma), y la fecha.
- Centrifugar las muestras durante 10 min a 1000 x g. No utilice los frenos para detener la centrifugadora. Esto dará lugar a tres capas (de arriba a abajo): plasma, leucocitos (capa leucocitaria) - una capa muy fina - y eritrocitos, incluyendo plaquetas.
- Aspirar cuidadosamente el sobrenadante (plasma) y alícuota de 500 ml en cada criovial. Tenga cuidado de no perturbar la capa de células (capa leucocitaria) o transferir cualquier célula.
- Almacenar a -80 ° C hasta que se necesite para la extracción de ARN o proceder a la extracción de RNA de inmediato.
- Preparar una hoja de extracción con los ID de las muestras para ser extraídos, incluidos los controles de plasma positivos y negativos.
- Para cada muestra a extraer, etiquetar un tubo de microcentrífuga estéril de 1,5 ml con el ID de la muestra, fecha de extracción y "ARN". También etiquetar un tubo de la columna y la recolección de ensamblado, así como un tubo de microcentrífuga de 2 ml que contenía solución de lisis de trabajo con los números correspondientes de la hoja de trabajo de extracción.
- El trabajo en el Gabinete de Bioseguridad, añadir 200 l de muestra en el correspondiente tubo de microcentrífuga de 2 ml de solución de lisis de trabajo.
- Así vórtice y se incuba durante 10 min a temperatura ambiente.
- Después de 10 minutos, centrifugar el tubo brevemente.
- Añadir 800 ml de etanol absoluto para cada uno de los tubos.
- Mezclar por agitación pulso y centrifugar brevemente.
- Transferir 600 l de esta solución para el conjunto de tubo de la columna / colección correspondiente. Se centrifuga a 6.000 xg durante 1 min.
- Transferir la columna a un nuevo tubo de recogida y desechar el tubo de recogida de edad que contiene el filtrado. Repita el paso anterior 2.8 (arriba) dos veces más.
- Añadir 500 l AW1 tampón de lavado a cada columna y centrifugar a 6000 xg durante 1 min.
- Deseche el tubo de filtrado y la recolección y transferencia de la columna a un nuevo tubo de recogida.
- Añadir 500 l era AW2 tampón y se centrifuga a 20.000 xg durante 3 min. Repita el paso 2.11.
- Se centrifuga en un nuevo tubo de recogida a 20.000 xg durante 2 min.
- Deseche la columna filtrado y colocar en 1,5 ml tubo de microcentrífuga.
- Añadir 60 l tampón AVE (RNasa libre de agua) para el medio de la columna de asegurar que usted no dispensar el líquido en el lado de la columna.
- Incubar a temperatura ambiente durante 1 min.
- Se centrifuga a 6.000 xg durante 2 min.
- Deseche la columna y tape los tubos de microcentrífuga de 1,5 ml.
- Las muestras están ahora listas para reverla transcripción se.
- Si el análisis se va a realizar de inmediato, se almacenan a 4 ° C durante un máximo de 6 horas. Sin embargo, si la prueba es que se retrase luego coloque a -80 ° C inmediatamente. NB: no congelar / descongelar las muestras de más de 3x.
3. Preparación de los reactivos para la transcripción inversa
- Antes de comenzar, calcular los volúmenes de cada uno de los reactivos necesarios para el número de muestras que se están procesando incluyendo, los controles de plasma positivos y negativos. También agregue un control reactivo.
- Uso de los volúmenes calculados desde el paso 3.1 (arriba), preparar el trifosfato de desoxirribonucleótido mezcla (dNTP)-cebador en una estéril 200 l tubo limpio, la PCR seguido de agitación con vórtex brevemente pulso. Cada muestra debe tener 0,5 l de cebador inverso RT21 y 0,5 l de dNTP, ver Tabla 2.
- Alícuota de 1,0 l de la mezcla dNTP imprimación para 200 tubos de PCR l.
- Preparar la transcriptasa inversa (RT) de mezcla de enzimas mediante la adición de 1 μ; L del tampón de transcripción inversa 10x, 1 l de DTT 0,1 M y 2 l de 25 mM de MgCl 2 a un tubo estéril seguido de agitación con vórtex y centrifugar brevemente, ver Tabla 3.
- Añadir 0,5 l cada una de las enzimas RNaseOUT y Superíndice III de la transcriptasa reversa al tubo de mezcla de enzimas a continuación, toque el tubo suavemente para mezclar.
- Mantener los tubos con las mezclas-dNTP imprimación y mezcla de enzimas en un bloque frío y pasar a la estación de ARN.
4. Transcripción inversa
- Añadir 6 l de la muestra de ARN para el tubo de mezcla de dNTP-cebador seguido por vórtex brevemente para mezclar.
- Después de la adición de la ARN, mover a la sala de PCR con tanto de dNTP / cebador / mezcla de ARN y RT mezcla de enzimas tubos en un bloque frío o hielo.
- Brevemente centrifugar los tubos de dNTP / mezcla de cebadores / ARN (desde el paso 4.2) y colocarlos en un termociclador.
- Se calienta a 65 ° C durante 5 min para desnaturalizar el ARN.
- Rápidamente fría a 4 ° C, mantener durante 2min.
- Pausa el termociclador cuando aún estaba en 4 º C; sacar los tubos.
- Rápidamente añadir 5 l de la mezcla de enzimas, manteniendo los tubos en un bloque frío.
- Mezclar suavemente golpeando ligeramente el tubo y centrifugar brevemente los tubos y volver al termociclador.
- Mantenga los tubos a 50 ° C durante 60 minutos para transcribir inversamente el ARN, seguido de la desnaturalización de la enzima a 85 ° C durante 5 min para detener la transcripción inversa.
- Enfriar a 37 ° C. Tan pronto la temperatura llega a 37 ° C, una pausa y tomar el metro fuera del termociclador.
- Añadir rápidamente 0,5 l de ARNasa H a los tubos y volver a la termociclador.
- Se mantiene a 37 ° C durante 20 minutos y luego se enfría a 4 ° C.
- El ADN complementario (ADNc) se puede utilizar inmediatamente o se puede almacenar a -20 ° C o menos hasta que se necesite. Sin embargo, el almacenamiento a largo plazo de ADNc debería ser a -80 ° C.
5. Preparación de los reactivos para la PCR
- Before de partida, calcular los volúmenes de cada uno de los reactivos necesarios para el número de muestras que se están procesando y los controles. Además de los tres controles (positivo, negativo y de reactivo), también puede agregar un control de PCR (ADN HIV). Las primera y segunda mezclas de PCR redondas se pueden preparar de forma simultánea y la segunda mezcla maestra se almacenaron a -20 ° C hasta que se necesite. Las mezclas se pueden almacenar durante aproximadamente 8 horas.
- Añadir 18,4 l de agua, 2,5 l de 10x tampón, 1,0 l de MgCl 2, 0,5 mu l de dNTP, y 0,25 l de cada uno de los cebadores como se muestra en la Tabla 4 y vórtice.
- Añadir 0,1 l de Platinum Taq polimerasa (5U/μl) y mezclar suavemente el tubo golpeándolo.
- Alícuota de 23 l de la mezcla maestra para 200 tubos de PCR l.
- Con los tubos de mezcla maestra sobre un bloque de hielo o fría traslado a la sala de PCR.
6. Nested PCR
- Añadir 2 l del cDNA de 23 l de la primera ronda de PCR master mezclar.
- Cerrar los tubos, poner las muestras en el termociclador y utilizar las siguientes condiciones de ciclado de PCR: 94 ° C durante 2 min, 30 ciclos de 95 ° C durante 30 seg, 58 ° C durante 20 seg, y 72 ° C durante 2 min, seguido de una extensión final a 72 ° C durante 10 min, como se muestra en la Figura 1.
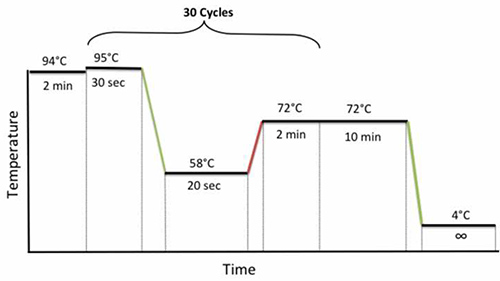
Figura 1. Condiciones de ciclos de PCR anidada. Haz click aquí para ver la imagen más grande.
- Continuar a la segunda etapa de PCR redondo o almacenar los productos de primera ronda de PCR a -20 ° C o menos hasta que sea necesario en una etapa posterior.
- Por segunda ronda de PCR, añadir 2 l de la primera ronda de PCR producto a 23 l de la segunda ronda de una mezcla maestra de PCRd utilizar el mismo programa de PCR en la Figura 1.
7. Electroforesis en gel
- Preparación de un gel
- Añadir un 0,5 g de la tableta de agarosa a un matraz de vidrio de 250 ml y añadir 50 ml de tampón 1x TBE al matraz.
- El calor en el microondas hasta que hierva; agitar con frecuencia (aproximadamente cada 30 segundos) hasta que esté completamente disuelta. Utilice un agarre de silicona o silicona horno guante para agarrar el frasco caliente. La solución de agarosa puede hervir del matraz con mucha facilidad por lo que vigilar de cerca este proceso.
- Enfriar a temperatura ambiente durante aproximadamente 10 min.
- Pour de agarosa en una bandeja de colada de gel que contiene peine tamaño apropiado; gel es listo para usar en aproximadamente 20-30 min.
- Coloque gel en la cámara de electroforesis y ejecutar según lo recomendado por el fabricante.
- La electroforesis en gel y visualización.
- Vortex Novel jugo durante 10 s antes de su uso.
- Diluir 1 l de jugo de Novela con 5 l de DMuestra de NA y la mezcla.
- Diluir 3 l de jugo de Novela con 3 l de marcador de peso molecular y mezclar.
- Cargar las mezclas de las secciones 7.2.2 y 7.2.3 (arriba) y ejecutar el gel a 100 V y 400 mA durante 40 min para evaluar la amplificación por PCR.
- Amplificación positiva puede ser visualizado a la luz ultravioleta como 1315 bp fragmento, la Figura 2.
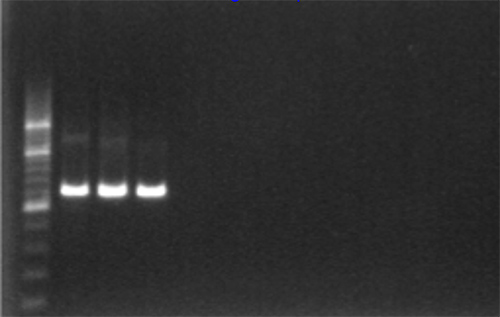
Figura 2. Confirmación Gel de amplificación por PCR utilizando 1% electroforesis en gel de agarosa y una escalera de 200 pb. Haga clic aquí para ver la imagen más grande.- No debería haber ninguna amplificación en los controles negativos y de reactivos, lo que indica ausencia de contaminación.
8. PCR Cleanup Producto
- En la preparación para la reacción de secuenciación, los segundos productos de PCR redondas positivos se limpian utilizando el kit de purificación de PCR PureLink.
- Añadir 80 l de tampón de unión de trabajo de alta Cutoff (B3) a 20 l de producto de PCR y la combinación de la pipeta.
- Añadir la muestra mezclada con el tampón de unión a una columna de centrifugación en un tubo de recogida.
- Centrifugar la columna a 10.000 xg durante 1 min. Transferir la columna en un nuevo tubo de recogida.
- Lavar la columna con 650 l de tampón de lavado con etanol.
- Centrifugar la columna a 10.000 xg durante 1 min. Transferir la columna en un nuevo tubo de recogida.
- Centrifugar la columna a máxima velocidad durante 2-3 minutos para eliminar cualquier solución de lavado residual.
- Colocar la columna de centrifugación en un tubo de 1,7 ml de elución limpio suministrado con el kit.
- Añadir 40 l de tampón de elución al centro de la columna y se incuba la columna a temperatur habitaciónE para 1 min.
- Centrifugar la columna a la velocidad máxima durante 2 min (> 10.000 xg).
- El tubo de elución contiene su producto de PCR purificado listo para la secuenciación. Deseche la columna.
- Determinar la concentración y la calidad del ADN usando una Nanodrop.
- Si no hay instalaciones de secuenciación de la casa están disponibles, los productos de PCR purificados se pueden enviar a un laboratorio de secuenciación comercial en esta etapa.
9. Reacciones de secuenciación
- Los productos de PCR se secuenciaron utilizando el gran versión del kit de tinte terminador de 3,1 y 4 cebadores para cada muestra (dos adelante y dos inversas). Las secuencias de los cebadores se muestran en la Tabla 2. Por lo tanto, después de la carrera de secuenciación, cada muestra tendrá cuatro secuencias para su montaje en un contig.
- Configurar las reacciones de secuenciación como se indica en la Tabla 5 para cada uno de los cuatro cebadores.
- Mezclar el tampón de secuenciación y los cebadores por agitación antes de su uso.
- Mezclar laagua, tampón y el cebador antes de la adición de la gran secuenciación de colorante. Mezclar por agitación.
- Mezcle suavemente la mezcla maestra después de añadir la gran mezcla de secuenciación tinte invirtiendo el tubo o tocando suavemente.
- Alícuota de 9 l de la mezcla maestra en una placa óptica de 96 pocillos.
- Con el fin de realizar 24 muestras / placa, configurar la placa como se indica a continuación la Figura 3.
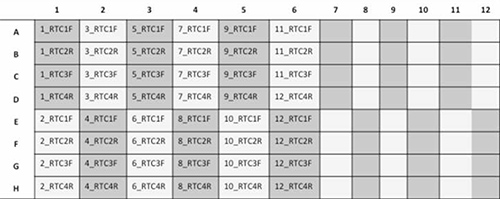
Figura 3. Representación Esquema de una placa de 96 pocillos con 12 muestras de pacientes se secuenciaron con cebadores 4 cada uno (RTC1F, RTC2R, RTC3F, y RTC4R). Haga clic aquí para ver la imagen más grande.
- Añadir 1,0 l de la muestra de ADN (~ 20 a 40 ng), cubrir la placa con unn adhesiva cubierta de aluminio y luego mezclar con cuidado.
- Se centrifuga a 3.000 g durante 1 min. Quite la cubierta de aluminio y añadir una estera de goma del lacre.
- Colocar la placa en el termociclador y ejecute el siguiente programa de ciclos se muestra en la Figura 4.
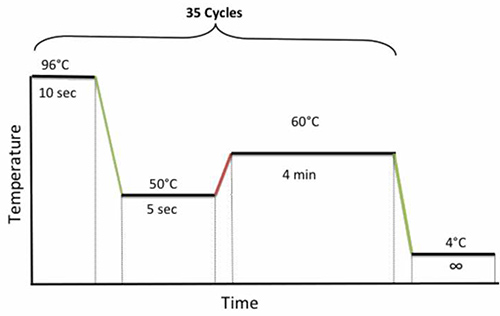
Figura 4. PCR ciclismo condiciones para la secuenciación. Haz click aquí para ver la imagen más grande.
- Cuando el PCR termina, limpiar el producto secuencial inmediatamente.
10. Limpieza de Secuenciación
- Para cada reacción de secuenciación, mezclar 50 l de etanol absoluto y 5 l 3 M de acetato de sodio.
- Con una pipeta multicanal, añada 55 &# 956; l de la solución de acetato de sodio / EtOH para cada pocillo.
- Selle los pocillos con tapa de aluminio adhesivo, asegurándose de que cada pozo está sellado correctamente.
- Centrifugar a 3000 xg durante 20 min.
- Después de 20 minutos, retire la tapa e invierta la placa, con un movimiento suave, en un pañuelo doblado laboratorio (NO golpear a deshacerse del sobrenadante ya que esto desalojar el sedimento!).
- Centrifugar la placa invertida sobre el mismo tejido en 150 xg durante 2 min.
- Inmediatamente después, añadir 150 l frío EtOH al 70%. NO retrasar la adición de etanol en este paso.
- Sello con la misma tapa de aluminio adhesiva y de vórtice.
- Se centrifuga a 3000 × g durante 5 min.
- Invertir la placa en un nuevo tejido y centrifugar plegado invertido a 150 xg durante 1 min.
- Después de la centrifugación, colocar al descubierto en el termociclador y se seca a 50 ° C durante 2 min.
- Una vez que las placas se seca, sellarlo con adhesivas cubiertas de papel de aluminio, envuelva en papel de aluminio y se almacenan a -20 ˚ C hasta que esté listo para proceder wiª La electroforesis de secuenciación.
- Cuando esté listo para la secuencia, disuelva los productos de secuenciación limpiados en 10 ml de formamida Hi-Di, desnaturalizar y la carga para la electroforesis.
11. Bioinformática
- Asamblea de secuencia
- El lanzamiento del programa Geneious.
- Crear una carpeta de trabajo para almacenar las secuencias.
- Importe los archivos ABI generados por la máquina de secuenciación para la carpeta de trabajo utilizando la herramienta de importación. Geneious asignará porcentaje puntuación de calidad para cada secuencia importada.
- Secuencias abiertas con puntuaciones de calidad> 70% haciendo doble clic en ellos.
- Cada archivo se abrirá en una nueva ventana. El software indicará la calidad en cada posición de nucleótidos del cromatograma de la calidad de la secuencia utilizando barras de color azul claro. La más alta la barra, mejor es la calidad de la llamada base.
- Utilizando el cursor, seleccione la sección media de la secuencia dejando de lado los extremos, que suelen ser de mala calidad.
- Haga clic en el botón de extracción para extraer la región con una secuencia buena calidad.
- Seleccionar todos los cuatro secuencias extraídos para cada muestra y ensamblarlos en contra de una secuencia de referencia.
- Inspeccione la secuencia de ensamblado para asegurarse de que usted está en el marco de lectura correcto. Si se encuentra en la fase de lectura correcta, el comienzo de la proteasa debe comenzar con los siguientes aminoácidos: PQITLW. El comienzo de la RT se iniciará con PISPIE.
- Extracto de la región contig cubre el inicio de relaciones públicas para el 300 RT codón. Durante este proceso, también para comprobar inserciones o deleciones.
- Ir a través de la secuencia de consenso de la contig extraído, la identificación de las ambigüedades y verificar las posiciones con bases mixtas mediante la inspección de la calidad (simetría, la altura, de fondo y los hombros de acompañamiento regiones) de las llamadas bases.
- Seleccione la secuencia de consenso y haga clic en el botón de extracción para crear un archivo independiente de la secuencia de consenso de los cuatro cebadores y Label adecuadamente.
- Exportar la secuencia a una carpeta de almacenamiento de copia de seguridad en el ordenador o una carpeta de red.
- Evaluación de la Calidad de la secuencia (HIVDB)
- Analizar la secuencia utilizando el programa HIVDB en http://hivdb.stanford.edu .
- Comprobar si las supresiones y las inserciones en los datos de resumen y asegúrese de que la secuencia cubre todos los 99 de la proteasa (PR) codones y los codones de primera 300 RT.
- Verifique que no haya resaltado la garantía de calidad (QA) cuestiones tanto en las regiones de PR y RT, tales como codones de parada, cambios de marco, posiciones ambiguas y residuos no habituales.
- Control de calidad de Secuenciación
- Explosión de la nueva secuencia contra una base de datos local secuencia de ejecución anterior.
- Si la nueva secuencia es> 97% similar a cualquier secuencia en la base de datos, todas las etapas del protocolo se deben revisar, a partir del análisis de la secuencia y volver a la extracción de ARN para garantie que no había ups de mezcla (posible cambio de muestras, etiquetado incorrecto) o la contaminación.
- Si no se identifican problemas, repita el análisis tanto de las viejas y nuevas muestras de la etapa de extracción de RNA.
- Si las secuencias siguen siendo> 97% similar, revisar la historia clínica del paciente para valorar cualquier vínculo epidemiológico entre los individuos.
- Análisis filogenético
- Alinear todas las secuencias de la base de datos utilizando el programa ClustalW en Geneious.
- Compruebe manualmente la alineación de secuencias desalineados, supresiones e inserciones y editar en consecuencia.
- Construya un árbol filogenético utilizando PHYML, Geneious constructor de árboles u otros constructores de árboles en Geneious.
- Examine el árbol para las muestras con longitudes de rama corta.
- Revise las muestras con longitudes cortas rama para una posible contaminación.
12. REGA DB Informática
- Secuencia Subir
- Inicie sesión en el REGADB utilizando un nombre de usuario y una contraseña únicos.
- En el menú desplegable, debajo de identificación del paciente, seleccione "Comienza con".
- Agregue la identificación del paciente y seleccionar el individuo cuyo genotipo es para ser cargado.
- En el menú de la izquierda, seleccione "aislado viral".
- De las opciones que se muestra viral seleccionar "agregar".
- Entre la fecha de la muestra, ID de la muestra, ID de secuencia y la fecha de Secuencia.
- Seleccione "Elegir archivo" y navegue hasta el archivo FASTA de la secuencia que se va a cargar.
- Después de seleccionar el archivo fasta para ser cargado, haga clic en Cargar.
- Una vez que la secuencia de cargado aparece en el cuadro de nucleótidos bajo las identifica la secuencia y las fechas, haga clic en el botón Aceptar en la parte inferior derecha de la ventana.
- Compruebe PR y RT proteína alineación haciendo clic en el botón de la proteína y la selección de cualquiera de PR o RT.
- Compruebe si la mutación de resistencia de drogas haciendo clic en el botón de la resistencia. Esto daque los perfiles de resistencia de tres algoritmos: ANRS, Stanford HIVDB y RegaDB.
- La generación de informes utilizando REGA
- Inicie sesión en el RegaDB usando su nombre de usuario y una contraseña únicos.
- En el menú desplegable, debajo de identificación del paciente, seleccione "Comienza con".
- Agregue la identificación del paciente y seleccione la persona cuyo informe se va a generar.
- En el menú de la derecha, seleccione aislado viral.
- De las opciones que se muestra viral haga clic en "vista".
- Haga doble clic en el aislado viral para el que desea crear un informe.
- En la ventana de aislamiento viral, haga clic en la ficha Informe muestra viral.
- Seleccione los algoritmos para la interpretación del genotipo del menú desplegable y luego seleccione la plantilla de informes para usar.
- Una vez seleccionados el algoritmo y la plantilla, haga clic en el botón "generar".
- Descargue el documento rtf generado.
- Abra el rtf hacercumento como un documento de Word.
- Cambiar el tamaño de la tabla de historial de tratamiento.
- Después de la tabla, agregue la sección "carta clínica e interpretación de resistencia".
- Utilizando los datos en la tabla de la resistencia y de la historia clínica, añadir una descripción del perfil de resistencia del paciente a partir de la historia de tratamiento del paciente, y los medicamentos a los cuales el aislado viral es resistente. Añada también una descripción de la carga viral del paciente y CD4 + perfiles de recuento de células de la tabla.
- Enviar el informe a las Enfermedades Infecciosas (ID) del especialista para su revisión y recomendaciones sobre la futura gestión de los pacientes. Este proceso es también una etapa muy importante de garantía de calidad. Cualquier error en el genotipo o inconsistencias en la historia del tratamiento, perfiles virológicos e inmunológicos pueden ser identificados y revisados antes de enviar un informe final, con todas las recomendaciones, para el clínico el tratamiento del paciente.
Resultados
El método validado fue una modificación de un método descrito previamente 20. El método de genotipificación ViroSeq, que ha sido aprobado por la FDA, se utilizó como método de referencia en la validación. Un panel de muestras de ensayos de aptitud obtenidos de las agencias nacionales de francés para la Investigación sobre el SIDA y la hepatitis viral (ANRS) se utilizó en la comparación principal entre los dos métodos. Los dos métodos de genotipificación fueron 100% concordantes en la identificación de todas las mutaciones clínicamente importantes asociadas a la resistencia de la droga tal como fue interpretado por el programa HIVDB para las muestras que fueron amplificados con éxito por ambos métodos. Como se muestra en la Tabla 6, las secuencias de nucleótidos de los tres pares eran 99,5% idéntica. Las secuencias de aminoácidos predichas eran 100% idénticas. Una muestra de cada cinco no pudo ser amplificado con éxito por ViroSeq. Además de la muestra no amplificada por ViroSeq, el método de la casa no pudo amplificar una segunda muestra, que se muestraser un virus recombinante de circulación (CRF02_AG) por ViroSeq. Las tres muestras que amplificaron con ambas metodologías eran del subtipo B (dos muestras) y el subtipo A (una muestra).
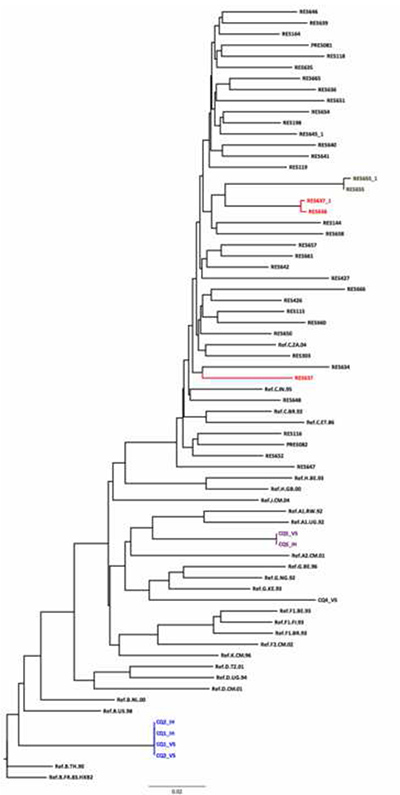
Figura 5. El uso de un Vecino Participar árbol HKY hecho como parte de la garantía de calidad de secuencias. Hay cuatro pares / grupos de secuencia con las distancias genéticas muy cortos. La distancia genética entre RES655 y RES655_1 (mismas muestras en secuencia en diferentes días) es 0,003. El es un posible error con el par RES637_1/RES638 como su distancia genética es demasiado corto (0.075) para muestras de diferentes individuos no vinculadas epidemiológicamente. Hay otro RES637 en el árbol con una distancia de 0,075 cuando se compara con RES638_1. El clúster CQ01/CQ02 sugiere que las dos muestrasdesde el panel son duplicados de la misma muestra. Se agrupan junto con la secuencia de referencia subtipo B que confirma el subtipo asignado por la herramienta REGA Subtipificación. CQ05 y CQ04 agrupan con los subtipos A y G, respectivamente, mientras que la herramienta de subtipificación REGA los clasificó como A y CRF02_AG respectivamente. Otra herramienta útil para los subtipos y la recombinación del VIH es SCUEL, que está disponible en http://www.datamonkey.org. Haga clic aquí para ver la imagen más grande.
Se utilizó un panel de cinco muestras para evaluar la precisión del método en-casa. Diez genotipos replicados se generaron para cada una de las cinco muestras. Usando el analizador genético 16 Capilar 3130xl, 48 de los 50 genotipos fueron generados a partir de las 24 carreras, preparados en el mismo día. Para todas las cinco muestras, las secuencias de aminoácidos predichas fueron 100% concordantes entre repeticiones. Para la secuencias de ácido nucleico, THere era> 99% de similitud por pares.
Durante los dos primeros años de la utilización de este método, sesenta muestras se repiten al azar de la extracción de RNA a secuenciación. No hubo diferencias estadísticamente significativas entre la puntuación de calidad de secuencia y el número de bases mixtas entre las repeticiones. Tanto el de nucleótidos y de aminoácidos comparaciones por pares para las sesenta pares fueron mayores que 99% idéntica. Por lo tanto las mutaciones de resistencia a fármacos para todos los pares fueron 100% concordantes.
Reducción de costes
Los volúmenes de reacción para RT, PCR y secuenciación se redujeron en al menos la mitad, en relación con el método original 20, 32, sin comprometer la calidad de las secuencias generadas. Esto permitió una reducción en el costo de 50% para las etapas de RT y PCR.
El nuevo método fue diseñado originalmente para trabajar con seis cebador de secuenciación para secuenciar todos los 99 codones del gen de la proteasa y los primeros 300 codones del gen de la transcriptasa inversa 20, 32. Métodos similares también utilizan seis a ocho cebadores 33, 34. Algunos métodos recientemente publicados han utilizado menos de seis cebadores, aunque a veces la secuenciación de la proteasa y los genes RT seprately 35, 36. Hemos tratado de reducir el número de cebadores de secuenciación de seis a cuatro, (Figura 6)
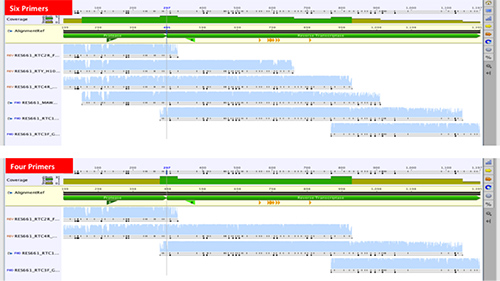
Figura 6. Comparación de secuencias contiguas de seis vs cuatro cebadores de secuenciación para la generación de la secuencia de Pol 1197 pb que cubre todos los 99 VIH-1 codones de la proteasa y los primeros 300 codones del gen de la transcriptasa inversa.242/51242fig6highres.jpg "target =" _blank "> Haga clic aquí para ver la imagen más grande.
Secuencias de un conjunto de 17 muestras generadas a partir de seis cebadores se compararon con las secuencias generadas después de la exclusión de dos cebadores (MAW46 y RTY). Los subtipos fueron 14 el subtipo C, dos subtipo B, y un subtipo A. No hubo diferencias significativas en las puntuaciones de calidad de secuencias. Una vez más, la identidad promedio pairwise entre los 17 pares de ácido nucleico fue de 99% y 100% en el nivel de aminoácidos. Por lo tanto, la reducción de los cebadores de secuenciación de seis a cuatro resultó en una reducción en el costo de secuenciación en casi un tercio.
La única herramienta de software propio que se utiliza en este protocolo fue Geneious para la secuencia de montaje. Las herramientas de interpretación resistencia a los medicamentos, así como el informe de la generación de herramientas son todo, herramientas de acceso abierto y gratuito. Esto reduce el coste aún más mediante la eliminación de los costos asociados con el uso de software propietario. Además, collective la negociación permitió a los reactivos para este protocolo para ser empaquetados en un estuche para facilitar el acceso de Life Technologies y está disponible como las Tecnologías de Saturno / Life método de genotipificación de 37. Por otra parte, los miembros de Saturno puede acceder a los reactivos a un precio con descuento.
Ámbito clínico
El protocolo descrito ha sido implementado en el control y la vigilancia de la resistencia a las drogas en una comunidad rural de KwaZulu-Natal. Un total de 604 genotipos fueron generados a partir de muestras clínicas, entre diciembre de 2010 y mayo de 2013, a una tasa de amplificación de 95% para las muestras con una carga viral> 1000 ARN copias / ml. Este estudio clínico de resistencia del VIH fue aprobado por el Comité de Ética de la Investigación Biomédica de la Universidad de KwaZulu-Natal (BF052/10 ref.) y el Comité de Investigación de la Salud del Departamento de Salud de KwaZulu-Natal (ref. HRKM 176/10). Los informes individuales de los pacientes fueron generados y enviados de vuelta a las clínicaspara el manejo del paciente.
Setenta y dos (72) genotipos también fueron generados como parte de un estudio de vigilancia de la resistencia a los medicamentos de transmisión, anidan dentro de un amplio estudio de vigilancia del VIH de base poblacional prospectivo. Las muestras primarias fueron toda pinchazo sangre recogida en microtubos EDTA. En genotipado hubo una tasa de amplificación de 79% 19. Ética aprobación para el genotipado de las muestras del estudio de vigilancia se obtuvo de la Universidad de Comité de Ética de la Investigación Biomédica de KwaZulu-Natal (BE066107 ref.).
| Nombre Primer | Secuencia | Longitud | Dirección | Posición HXB2 | |
| MAW-26 | TTGGAAATGTGGAAA GGAAGGAC | 23 | Adelante | 2028-2050 | Primera ronda de PCR |
| RT-21 | CTGTATTTCAGCTATC AAGTCCTTTGATGGG | 31 | Reverse | 3539-3509 | Primera ronda de PCR |
| Pro-1 | TAGAGCCAACAGCCC cacca | 20 | Adelante | 2147-2166 | Segunda ronda de PCR |
| RT-20 | CTGCCAATTCTAATTC TGCTTC | 22 | Reverse | 3462-3441 | Segunda ronda de PCR |
| RTC1F | ACCTACACCTGTCAA CATAATTG | 23 | Adelante | 2486-2508 | Secuenciación |
| RTC2R | TGTCAATGGCCATTG TTTAACCTTTGG | 27 | Reverse | 2630-2604 | Secuenciación |
| RTC3F | CACCAGGGATTAGAT ATCAATATAATGTGC | 30 | Adelante | 2956-2994 | Secuenciación |
| RTC4R | CTAAATCAGATCCTAC ATACAAGTCATCC | 29 | Reverse | 3129-3101 | Secuenciación |
| RT-Y | GTGTCTCATTGTTTAT ACTAGG | 22 | Reverse | 2967-2946 | Secuenciación |
| MAW-46 | TCCCTCAGATCACTC TTTGGCAACGAC | 27 | Adelante | 2251-2277 | Secuenciación |
Tabla 1. La transcripción, PCR, y cebadores de secuenciación personalizados utilizados en la generación de un fragmento de 1197 pb Pol cubriendo todos los 99 VIH-1 codones de la proteasa y los primeros 300 codones del gen trascriptase inversa inversa.
| RT21 (5pmol/ml) | 0.5 | 0.2 |
| dNTP (10 mM) | 0.5 | 0.4 |
| Total | 1 |
Tabla 2. dNTP / Primer mezcla para la reacción de transcripción inversa.
| Reactivo | Volumen (ml) / reacción | Concentración / reacción |
| Primera Strand Buffer (10x) | 1 | 1 |
| MgCl2 (25 mM) | 2 | 4 |
| DTT (0,1 M) | 1 | 0.008 |
| RNaseOUT (40 U / ml) | 0.5 | 16 |
| Superíndice III transcriptasa inversa (200U/ml) | 0.5 | 8 |
| Total | 5 |
Tabla 3. Mezcla de enzimas para la reacción de transcripción inversa.
| Reactivo | Volumen (ml) / reacción | Concentración final / Reacción |
| DEPC agua tratada | 18.4 | - |
| Tampón de PCR (10x) | 2.5 | 1 |
| MgCl2 (50 mM) | 1 | 2 |
| mezcla de dNTP (10 mM) | 0.5 | 0.2 |
| Cebador Froward (5 pmol / ml) | 0.25 | 0.05 |
| Cebador inverso (5 pmol / ml) | 0.25 | 0.05 |
| Platinum Taq polimerasa (5 U / ml) | 0.1 | 0.02 |
| Total parcial | 23 | - |
Tabla 4. Master mezcla para la PCR anidada.
| Reactivo | Volumen (ml) / reacción | Concentración / reacción |
| DEPC agua tratada | 6.1 | |
| Secuenciación de búfer (5x) | 2 | 1 |
| Primer (3,2 pmol / ml) | 0.5 | 0.16 |
| Grandes terminador mezcla de secuenciación del tinte | 0.4 | - |
| Total | 9 |
Tabla 5. Mezcla maestra para las reacciones de secuenciación.
| ViroSeq | Inhouse | % NA Similitud | |||||||
| ID de la muestra | Subtipo | El nivel de calidad | PR mutaciones | Mutaciones en RT | Subtipo | El nivel de calidad | Mutaciones PR | RT mutaciones | |
| CQ01 | B | 99.9 | M46L, I54L, V82A, L90M | D67N, T69D, K70R, M184V, T215V, K219Q | B | 99.2 | M46L, I54L, V82A, L90M | D67N, T69D, K70R, M184V, T215V, K219Q | 100 |
| CQ02 | B | 99.5 | M46L, I54L, V82A, L90M | D67N, T69D, K70R, M184V, T215V, K219Q | B | 99.5 | M46L, I54L, V82A, L90M | D67N, T69D, K70R, M184V, T215V, K219Q | 100 |
| CQ03 | NA | NA | NA | NA | NA | NA | |||
| CQ04 | CRF02_AG | 98.4 | I54V, V82F, I84V | M41L, L74I, L210W, T215Y, V108I, Y181C | NA | NA | NA | NA | NA |
| CQ05 | La | 99.7 | K103N | La | 93 | K103N | 100 | ||
Tabla 6. Resul comparativots de un análisis paralelo entre el método de genotipificación ViroSeq y el método de la casa usando un panel de muestras proporcionada por la ANRS.
Discusión
Varios de bajo costo métodos internos se han descrito en los esfuerzos para tratar de hacer el genotipado de resistencia del VIH más asequible 33, 34, 36. No hay duda de la necesidad de integrar las pruebas de resistencia a los medicamentos en el continuo de la atención a las personas en tratamiento antirretroviral en entornos con recursos limitados. Sin embargo, la mayoría de los métodos denunciados se centran en la aplicación de genotipado resistencia a los medicamentos en la vigilancia de la resistencia a los medicamentos a nivel de población. El método de genotipificación SATURN / Life Technologies es un protocolo totalmente integrado para la vigilancia y el seguimiento de la farmacorresistencia. Este método fue diseñado para ser un protocolo asequible implementar fuente mayoría abiertos y abiertas recursos bioinformáticos de acceso para la interpretación de la resistencia a los medicamentos y la generación de informes para la gestión clínica.
Se ha demostrado a través de la comparación con el método de genotipificación ViroSeq aprobado por la FDA para serprecisa en la identificación de mutaciones de resistencia a medicamentos de un panel de muestras de ensayos de aptitud ANRS, en el 100% de las muestras del panel de laboratorio que fueron amplificados con éxito. La precisión también se evaluó en muestras clínicas de los virus del subtipo C, el subtipo más dominante en el sur de África. El método era tan precisa sobre muestras del subtipo C, ya que era el subtipo A y B. Sin embargo, si el método se utiliza en otras partes del mundo donde CRF02_AG es frecuente, hay una necesidad para la modificación de los cebadores ya que el método fallado para amplificar una de las muestras de panel que ha demostrado tener CRF02_AG. Alternativamente, un conjunto degenerado de cebadores sensibles a todos los virus del grupo M 33, 36 podría ser utilizado en regiones en las que la distribución subtipo es más heterogéneo 38.
La sensibilidad de la transcripción inversa y la PCR se puede aumentar mediante la extracción de ARN a partir de mayores volúmenes de plasma, tales como 500 ml. El plasma se puede Centrifuged a 21.000 xg durante 90 minutos para concentrar las partículas virales antes de proceder con el protocolo descrito por el mini-kit de extracción de ARN viral QIAamp.
Como se muestra, el nuevo método tiene una ventaja adicional que produce informes completos para la gestión individual del paciente. Estos informes son una consolidación del genotipo, el inmunológico y los datos de vigilancia virológica, así como la historia clínica y el tratamiento de RegaDB. Esto va acompañado de una interpretación de laboratorio detallada del perfil de resistencia seguida de un examen igualmente detallada de la historia clínica del paciente, así como recomendaciones de tratamiento. La utilización de un médico especialista para revisar los informes y proporcionar recomendaciones de tratamiento para los pacientes ofrece la tutoría muy necesaria para los profesionales de enfermería, así como a los médicos sin experiencia, que están proporcionando cada vez ART en Sudáfrica como parte de las recomendaciones de la OMS para el cambio de tareas. Estos clínicainformes han demostrado ser medios de enseñanza eficaces para los médicos con poca o ninguna experiencia en la gestión de resistencia a los medicamentos. Desde la perspectiva del paciente, nuestro método reduce la necesidad de viajar a sitios centralizados para acceder a los servicios de VIH especializados.
Por lo tanto, el protocolo descrito considerado en su conjunto ofrece una buena plataforma a través del cual la gestión de la resistencia del VIH se puede integrar, a un costo accesible, en la continuidad de la atención a las personas infectadas por el VIH no ART. Los datos generados se pueden usar para fines epidemiológicos para evaluar la evolución y la transmisión de la resistencia a los medicamentos en la comunidad. El tamaño del fragmento pol generado es lo suficientemente bueno para el análisis filogenético más complejo que producirá mejor comprensión de la epidemia a nivel de la población.
Divulgaciones
Este trabajo fue financiado por el Wellcome Trust (082384/Z/07/Z), Unión Europea (SANTE 2007 147-790), el Centro de EE.UU. para el control de enfermedades a través de CAPRISA (Título del proyecto: Fortalecimiento de Sistemas de Salud y el fracaso del tratamiento del VIH (VIH- TFC)), y el Programa Común de Investigación suizo africano del sur (SSJRP) beca de investigación titulado "Swiss Prot / Sudáfrica: Proteína Bioinformática Desarrollo de Recursos de patógenos relacionados con la salud importantes". RL es apoyada por el Wellcome Trust (número de concesión 090999 / Z / 09 / Z). Los financiadores no tenía papel en el diseño del estudio, recogida y análisis de datos, decisión a publicar, o la preparación del manuscrito. Los autores declaran no tener intereses financieros en competencia.
Agradecimientos
Los autores desean dar las gracias a todos los colegas que hicieron posible este trabajo, especialmente Maya Balamane, Elizabeth Johnston Blanco, Sharon Sjoblom, Greg Ording Zakhona Gumede, Xolile Kineri, Phindile Mabaso, Lungisa Ndwandwe, James Garvey, Gavin Cobb, Senzo Maphanga, Terusha Chetty , Kevi Naidoo, Andrew Skingsley, Katharine Stott y Lungani Ndwandwe. Los autores también desean agradecer a todo el personal del Departamento de Salud y personal de África Centro que trabajan el Tratamiento del VIH Hlabisa y el Programa de Atención.
Materiales
| Name | Company | Catalog Number | Comments |
| Superscript III 1st strand Synthesis kit | Life Technologies | 18080051 | Reverse Transcription |
| SATURN/LiFE Technologies Custom Primers | Life Technologies | 4473517 | PCR |
| Platinum Taq | Life Technologies | 10966026 | PCR |
| PureLink QUICK PCR Purification Kit | Life Technologies | K310002 | PCR |
| Viroseq | ABBOTT | 4J94-20 | Reverse Transcription and PCR |
| Agarose Tablets (Dnase/Rnase free) | BIOLINE | BIO-41027 | PCR |
| TBE Buffer | MERCK | 1.06177.2500 | PCR |
| O'Range Ruler 200 bp DNA Ladder | Fermentas | FE SM0633 | PCR |
| Novel Juice | GeneDireX | LD001-1000 | PCR |
| MiniBis Bioimaging System | DNR Bioimaging Systems Ltd | Gel Documentation | |
| Power Pac 300 | BIORAD | Gel Electrophoresis | |
| Big Dye Terminator Kit version 3.1 | Life Technologies | 4337456 | Sequencing |
| Arrays | Life Technolgies | 4319899 | Sequencing |
| PoP | Life Technologies | 4363785 | Sequencing |
| 10x EDTA Buffer | Life Technologies | 402824 | Sequencing |
| Formamide | Life Technologies | 4311320 | Sequencing |
| 5x Sequencing Buffer | Life Tecgnologies | 4336697 | Sequencing |
| 3130 xl Genetic Analyzer | Life Technologies | Sequencing | |
| GeneAmp PCR System 9700 | Life Technologies | RT/PCR/Sequencing | |
| Centrifuge 5804 | EPPENDORF | Sample Processing | |
| Centrifuge 5415R | EPPENDORF | RNA Extraction | |
| Centrifuge 5415R | EPPENDORF | RT and PCR | |
| Centrifuge 5415D | EPPENDORF | PCR Product Clean up | |
| Centrifuge 5810 | EPPENDORF | Sequencing Clean up | |
| Picofuge | BIORAD | C1301-230V | RT and PCR |
| Vortex Genius 3 | IKA | RNA extraction and reagent preparation | |
| Vortex mixer | IKA | Sequencing Cleanup | |
| NanoDrop 2000 UV/VIS spectrophotometer | ThermoScientific | DNA quantification | |
| 3 M Sodium Acetate | MERCK | 567422 | Sequencing Clean up |
| Absolute Ethanol | MERCK | SAAR2233540LP | Sequencing Cleanup |
| 1.5 ml SARSTEDT Tubes | BIODEX | 72.692.005 | RNA Extraction |
| 2 ml SARSTEDT Tubes | BIODEX | 72.693.005 | RNA Extraction |
| 2 ml Collection tubes | SCIENTIFIC GROUP | MCT-200-NC/S | RNA Extraction |
| Optical MicroAmp 96-well reaction plates | Life Technologies | N8010560 | Sequencing |
| 200 µl 8 Strip StarPCR Tubes with attached flat caps | STAR Lab - supplied by CELTIC | A1402-3700 | RT and PCR |
| 200 µl PCR individual tubes | Scientific Group | CR/3745 | RT and PCR |
| Geneious | Biomatters | Sequence analysis | |
| Internet Access | Preferrably high speed | ||
| Web resources | |||
| hivdb.stanford.edu | Stanford University | Drug reistance analysis | |
| http://bioafrica.mrc.ac.za:8080/regadb-ui/RegaDB | SATuRN | database | |
| http://bioafrica.mrc.ac.za/tools/pppweb.html | SATuRN | Sequence quality tool | |
Referencias
- Shao, Y., Williamson, C. The HIV-1 epidemic: low- to middle-income countries. Cold Spring Harbor Persp. Med. 2, (2012).
- Mutevedzi, P. C., et al. Scale-up of a decentralized HIV treatment programme in rural KwaZulu-Natal, South Africa: does rapid expansion affect patient outcomes. Bull. World Health Organ. 88, 593-600 (2010).
- Houlihan, C. F., et al. Cohort profile: Hlabisa HIV treatment and care programme. Int. J. Epidemiol. 40, 318-326 (2011).
- Tanser, F., Barnighausen, T., Grapsa, E., Zaidi, J., Newell, M. L. High coverage of ART associated with decline in risk of HIV acquisition in rural KwaZulu-Natal. South Africa. Science. 339, 966-971 (2013).
- Bor, J., Herbst, A. J., Newell, M. L., Barnighausen, T. Increases in adult life expectancy in rural South Africa: valuing the scale-up of HIV treatment. Science. 339, 961-965 (2013).
- Montaner, J. S., et al. The case for expanding access to highly active antiretroviral therapy to curb the growth of the HIV epidemic. Lancet. 368, 531-536 (2006).
- Granich, R. M., Gilks, C. F., Dye, C., De Cock, K. M., Williams, B. G. Universal voluntary HIV testing with immediate antiretroviral therapy as a strategy for elimination of HIV transmission: a mathematical model. Lancet. 373, 48-57 (2009).
- Johnson, L. F., et al. Life expectancies of South african adults starting antiretroviral treatment: collaborative analysis of cohort studies. PLoS Med. 10, (2013).
- Blower, S., Ma, L., Farmer, P., Koenig, S. Predicting the impact of antiretrovirals in resource-poor settings: preventing HIV infections whilst controlling drug resistance. Curr. Drug Targets. 3, 345-353 (2003).
- Geretti, A. M. Epidemiology of antiretroviral drug resistance in drug-naive persons. Curr. Opin. Infect. Dis. 20, 22-32 (2007).
- Larder, B. A., Darby, G., Richman, D. D. HIV with reduced sensitivity to zidovudine (AZT) isolated during prolonged therapy. Science. 243, 1731-1734 (1989).
- Erice, A., et al. Brief report: primary infection with zidovudine-resistant human immunodeficiency virus type 1. N. Engl. J. Med. 328, 1163-1165 (1993).
- Williams, I., et al. British HIV Association guidelines for the treatment of HIV-1-positive adults with antiretroviral therapy. HIV Med. 13 Suppl 2, 1-85 (2012).
- DHHS, . US Panel on Antiretroviral Guidelines for Adults and Adolescents. Guidelines for the use of antiretroviral agents in HIV-1-infected adults and adolescents. , (2012).
- Vandamme, A. M., et al. European recommendations for the clinical use of HIV drug resistance testing: 2011 update. AIDS Rev. 13, 77-108 (2011).
- DHHS, . US Panel on Antiretroviral Therapy and Medical Management of HIV-Infected Children. Guidelines for the Use of Antiretroviral Agents in Pediatric HIV Infection. , (2012).
- . Department of Health. Clinical guidelines for the management of HIV & AIDS in adults and adolescents. , (2010).
- Conradie, F., et al. The 2012 southern African ARV drug resistance testing guidelines. S. Afr. J.HIV Med. 13, 162-167 (2012).
- Manasa, J., et al. Primary Drug Resistance in South Africa: Data from 10 Years of Surveys. AIDS Res. Hum. Retroviruses. 28, 558-565 (2012).
- Tshabalala, M., et al. Surveillance of transmitted antiretroviral drug resistance among HIV-1 infected women attending antenatal clinics in Chitungwiza, Zimbabwe. PLoS ONE. 6, (2011).
- Bartolo, I., et al. Antiretroviral drug resistance surveillance among treatment-naive human immunodeficiency virus type 1-infected individuals in Angola: evidence for low level of transmitted drug resistance. Antimicrob. Agents Chemother. 53, 3156-3158 (2009).
- Bartolo, I., et al. HIV-1 genetic diversity and transmitted drug resistance in health care settings in Maputo, Mozambique. J. Acquir. Immune Defic. Syndr. 51, 323-331 (2009).
- Hamers, R. L., et al. HIV-1 drug resistance in antiretroviral-naive individuals in sub-Saharan Africa after rollout of antiretroviral therapy: a multicentre observational study. Lancet Infect. Dis. 11, 750-759 (2011).
- Hamers, R. L., et al. HIV-1 Drug Resistance Mutations Are Present in Six Percent of Persons Initiating Antiretroviral Therapy in Lusaka, Zambia. J. Acquir. Immune Defic. Syndr. , (2010).
- Nwobegahay, J., Selabe, G., Ndjeka, N. O., Manhaeve, C., Bessong, P. O. Low prevalence of transmitted genetic drug resistance in a cohort of HIV infected naive patients entering antiretroviral treatment programs at two sites in northern South Africa. J. Med. Virol. 84, 1839-1843 (2012).
- Iweriebor, B. C., et al. Molecular epidemiology of HIV in two highly endemic areas of northeastern South Africa. Arch. Virol. 157, 455-465 (2012).
- Parboosing, R., Naidoo, A., Gordon, M., Taylor, M., Vella, V. Resistance to antiretroviral drugs in newly diagnosed, young treatment-naive HIV-positive pregnant women in the province of KwaZulu-Natal South Africa. J. Med. Virol. 83, 1508-1513 (2011).
- Nwobegahay, J. M., et al. Prevalence of antiretroviral drug resistance mutations and HIV-I subtypes among newly-diagnosed drug-naive persons visiting a voluntary testing and counselling centre in northeastern South Africa. J. Health Popul. Nutr. 29, 303-309 (2011).
- Nwobegahay, J., et al. Prevalence of drug-resistant mutations in newly diagnosed drug-naive HIV-1-infected individuals in a treatment site in the waterberg district, limpopo province). S. Afr. Med. J. 101 (2011), 335-337 (2011).
- Rosen, S., Long, L., Sanne, I., Stevens, W. S., Fox, M. P. The net cost of incorporating resistance testing into HIV/AIDS treatment in South Africa: a Markov model with primary data. J. Int. AIDS Soc. 14, 24 (2011).
- Dalai, S. C., et al. Evolution and molecular epidemiology of subtype C HIV-1 in Zimbabwe. AIDS. 23, 2523-2532 (2009).
- Chen, J. H., et al. In-house human immunodeficiency virus-1 genotype resistance testing to determine highly active antiretroviral therapy resistance mutations in Hong Kong. Hong Kong Med. 18, 20-24 (2012).
- Lee, C. K., et al. An in-house HIV genotyping assay for the detection of drug resistance mutations in Southeast Asian patients infected with HIV-1. J. Med. Virol. 84, 394-401 (2012).
- Aitken, S. C., et al. A Pragmatic Approach to HIV-1 Drug Resistance Determination in Resource-Limited Settings by Use of a Novel Genotyping Assay Targeting the Reverse Transcriptase-Encoding Region Only. J. Clin. Microbiol. 51, 1757-1761 (2013).
- Zhou, Z., et al. Optimization of a low cost and broadly sensitive genotyping assay for HIV-1 drug resistance surveillance and monitoring in resource-limited settings. PLoS One. 6, (2011).
- , Life Technologies. Life Technologies and SATuRN Collaborate to Increase Access to HIV Testing in Africa. , (2012).
- Lihana, R. W., Ssemwanga, D., Abimiku, A., Ndembi, N. Update on HIV-1 diversity in Africa: a decade in review. AIDS Rev. 14, 83-100 (2012).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados