Method Article
Un metodo di monitoraggio Affordable HIV-1 resistenza ai farmaci per le impostazioni limitate risorse
In questo articolo
Riepilogo
Test di resistenza ai farmaci per l'HIV-1 individui infetti in mancanza di terapia antiretrovirale (ART) in grado di guidare le terapie future e migliorare i risultati del trattamento. Ottimizzazione dei singoli e della popolazione esiti di salute in alta prevalenza di HIV, ma a risorse limitate in ultima analisi richiederà conveniente e accessibile genotipizzazione resistenza ai farmaci e metodi di interpretazione.
Abstract
HIV-1 farmacoresistenza ha il potenziale di compromettere seriamente l'efficacia e l'impatto della terapia antiretrovirale (ART). Poiché i programmi ART nell'Africa sub-sahariana continuano ad espandersi, gli individui su ART devono essere attentamente monitorati per la comparsa di resistenza ai farmaci. Sorveglianza della resistenza trasmessa farmaco per monitorare la trasmissione di ceppi virali resistenti ai già ART è anche critico. Purtroppo, test di resistenza ai farmaci non è ancora facilmente accessibile nelle impostazioni limitate risorse, perché la genotipizzazione è costosa e richiede sofisticate di laboratorio e infrastruttura di gestione dei dati. Un genotipica metodo di monitoraggio farmacoresistenza accesso aperto per gestire gli individui e valutare la resistenza ai farmaci trasmesso e descritta. Il metodo utilizza il software libero e open source per l'interpretazione dei modelli di resistenza ai farmaci e la generazione di report dei singoli pazienti. Il protocollo genotipizzazione ha un indice di amplificazione superiore al 95% per i campioni di plasma con avcarico Iral> 1,000 HIV-1 RNA copie / ml. La sensibilità diminuisce in modo significativo per carica virale <1.000 copie di HIV-1 RNA / ml. Il metodo qui descritto è stato validato nei confronti di un metodo di HIV-1 resistenza Drug Testing approvata dagli Stati Uniti Food and Drug Administration (FDA), il metodo di genotipizzazione Viroseq. Limiti del metodo qui descritto comprendono il fatto che non è automatizzato e che essa non anche per amplificare la forma circolante CRF02_AG ricombinante da un gruppo di validazione di campioni, anche se amplificato sottotipi A e B dello stesso pannello.
Introduzione
L'epidemia di HIV in Africa australe si è evoluto rapidamente 1 con un aumento esponenziale concomitante persone in terapia antiretrovirale (ART), soprattutto in Sud Africa 2, 3. Come prova sull'impatto epidemiologico dei programmi di trattamento su larga scala nel ridurre l'incidenza 4 e aumentando l'aspettativa di vita in contesti a risorse limitate (RLS) 5 continua ad accumularsi, saranno intensificati gli sforzi per aumentare la copertura ART. L'evoluzione di orientamenti verso l'uso del trattamento come strumento di prevenzione 6, 7 sotto test e programmi di trattamento significa che il numero assoluto di individui in trattamento aumenterà ulteriormente. Un gran numero di persone saranno in ART per periodi di tempo più lunghi come l'aspettativa di vita media degli individui su ART si avvicina a quella della popolazione infetta da HIV 8. Lo sviluppo e la trasmissione di resistenza ai farmaci HIV ha Always stato considerato una minaccia per le conquiste della ART 9-12. Pertanto, vi è la necessità per la sorveglianza ed il monitoraggio della resistenza ai farmaci più rigorosa come più individui sono avviate su ART.
Test di resistenza ai farmaci genotipica (TSL) è stato utilizzato con successo nei paesi sviluppati, sia per la sorveglianza e il monitoraggio del virus HIV-1 farmaco-resistenza nei soggetti trattati con ART. In queste impostazioni, TSL è stato integrato nel continuum di cura per l'HIV-1 individui infetti. La maggior parte delle linee guida internazionali raccomandano tsl per i pazienti pediatrici mancanza ART (di prima linea e di seconda linea) 13-15 adulto o, pazienti pediatrici esposti alla prevenzione della trasmissione da madre a figlio (PMTCT) regimi, ma successivamente infettati 16, e in ambienti con alti livelli di resistenza trasmessa droga tra gli individui con infezione acuta 13-15. Tuttavia, il costo, la tecnologia e infrastruttura, hanno limitato l'attuazionezione di approcci simili alla droga monitoraggio della resistenza in RLS.
Il trattamento South African HIV e il monitoraggio attualmente non raccomandano l'uso di tsl nel guidare la scelta di ART per gli individui in mancanza di prima linea regimi 17. Gli individui sono accesi basata principalmente su parametri (carico virale di HIV-1 RNA) virologica. Tuttavia, nel 2012, il Southern African HIV clinici Society ha pubblicato le prime linee guida del Sud ARV africana test resistenza ai farmaci 18. Queste linee guida raccomandano test tsl per tutti gli adulti in mancanza di prima linea e di seconda linea ART e per i neonati infetti e bambini esposti a PMTCT 18. Tuttavia, TSL non è raccomandato 18 per gli individui con infezione acuta perché non ci sono evidenze attuali livelli elevati di resistenza ai farmaci trasmessa in Sud Africa 19-29. Si prevede che alcune di queste raccomandazioni saranno integrati nel tempo in tre nazionaliatment e monitoraggio linee guida dei vari paesi della regione. Già, in 2013 linee guida di trattamento sudafricani ora c'è la raccomandazione di tsl al momento del guasto di seconda linea per gli adulti e al momento del guasto regime basato-PI-prima o seconda linea per i bambini 30.
E 'stato dimostrato che l'integrazione GRT nelle linee guida di trattamento in Sud Africa sarebbe potenzialmente costo-neutrale. Considerando il costo dei farmaci di seconda linea di regime che sono relativamente più costosi rispetto ai farmaci di prima linea, con TSL per identificare i pazienti che hanno veramente bisogno di essere passato a terapia di seconda linea non comporterà alcun costo aggiuntivo per il programma. Inoltre, GRT può anche identificare altre ragioni per il fallimento, conservare le opzioni di trattamento e generare informazioni sui modelli di resistenza emergenti 31. Pertanto, è necessario ridurre il costo di metodi di monitoraggio della farmaco resistenza ulteriormente per migliorare l'accesso, la qualità delle cure und esiti.
Qui, vi presentiamo un metodo GRT progettato per utilizzare generici (open source) primer per la trascrizione inversa, la reazione a catena della polimerasi (PCR) e sequenziamento (Tabella 1), così come il software open source per lo più per l'interpretazione resistenza ai farmaci. Per la gestione clinica, il protocollo è accompagnato da una revisione e reporting metodo completo con l'interpretazione specialista del farmaco rapporto resistenza laboratorio con stretta aderenza alle linee guida nazionali di trattamento. Il protocollo è diviso in quattro componenti differenti; 1) HIV Acido ribonucleico (RNA) Estrazione, 2) trascrizione inversa e Polymerase Chain Reaction (PCR) amplificazione di bersagli virali, 3) sequenziali e 4) metodi bioinformatici per l'analisi dei cromatogrammi, allineamento, curation e l'interpretazione dei dati di sequenza.
Protocollo
1. Etilendiammintetraacetato (EDTA) intero trattamento del sangue
Nota: Il sangue può essere elaborato subito dopo la raccolta di può essere conservato a 4 ° C per non più di 24 ore.
- Lavorare in un armadio biosicurezza, proibisce l'intero campione di sangue EDTA a temperatura ambiente.
- Per ogni campione, etichettare abbastanza cryovials con l'identificazione del campione (ID), materiale di accumulo (plasma), e la data.
- Centrifugare i campioni per 10 min a 1000 x g. Non usare i freni per arrestare centrifuga. Questo produrrà tre strati (dall'alto verso il basso): plasma, leucociti (buffy coat) - uno strato molto sottile - ed eritrociti, comprese le piastrine.
- Con attenzione aspirare il surnatante (plasma) e un'aliquota di 500 ml in ogni esageratamente. Fare attenzione a non disturbare lo strato di cellule (buffy coat) o il trasferimento di tutte le cellule.
- Conservare a -80 ° C fino al momento della estrazione di RNA o procedere alla estrazione di RNA immediatamente.
- Preparare un foglio di estrazione con gli ID dei campioni da estrarre compresi i controlli plasma positivi e negativi.
- Per ogni campione da estrarre, etichettare un 1,5 ml sterile provetta con l'ID del campione, la data di estrazione e "RNA". Etichettare anche una colonna e raccolta tubo assemblato così come una provetta contenente 2 ml di soluzione di lisi lavorare con i numeri corrispondenti del foglio di estrazione.
- Lavorare nel Gabinetto Bio-sicurezza, aggiungere 200 microlitri del campione al corrispondente provetta 2 ml di soluzione di lisi di lavoro.
- Vortex bene e incubare per 10 minuti a temperatura ambiente.
- Dopo 10 minuti, centrifugare brevemente la provetta.
- Aggiungere 800 ml di etanolo assoluto a ciascuno dei tubi.
- Mescolare nel vortex impulso e centrifugare brevemente.
- Trasferire 600 ml di questa soluzione per il gruppo tubo della colonna / collezione corrispondente. Centrifugare a 6.000 xg per 1 min.
- Trasferire colonna a un nuovo tubo di raccolta e gettare il vecchio tubo di raccolta contenente il filtrato. Ripetere il passaggio precedente 2.8 (sopra) due volte di più.
- Aggiungere 500 microlitri di tampone di lavaggio AW1 per ogni colonna e centrifugare a 6000 xg per 1 min.
- Scartare il tubo filtrato e la raccolta e trasferire la colonna su un nuovo tubo di raccolta.
- Aggiungere 500 microlitri tampone era AW2 e centrifugare a 20.000 xg per 3 min. Ripetere passo 2.11.
- Centrifugare in un nuovo tubo di raccolta a 20.000 xg per altri 2 min.
- Eliminare colonna filtrato e posto in 1,5 ml provetta.
- Aggiungere 60 microlitri tampone AVE (RNasi acqua libera), al centro della colonna garantire che non erogare il liquido sul lato della colonna.
- Incubare a temperatura ambiente per 1 min.
- Centrifugare a 6000 xg per 2 minuti.
- Eliminare la colonna e tappare i tubi microcentrifuga 1,5 ml.
- I campioni sono ora pronti per reverSE trascrizione.
- Se il test deve essere eseguito immediatamente, conservare a 4 ° C per un massimo di 6 ore. Tuttavia, se il test deve essere ritardato poi posto a -80 ° C immediatamente. NB: non congelare / scongelare i campioni più di 3x.
3. Preparazione del reagente per la trascrizione inversa
- Prima di iniziare, calcolare il volume di ciascuno dei reagenti necessari per il numero di campioni in lavorazione compresi, i controlli plasma positivi e negativi. Anche aggiungere un controllo reattivo.
- Utilizzando i volumi calcolati dal punto 3.1 (sopra), preparare il trifosfato deossiribonucleotide mix (dNTP) primer in un ambiente pulito, sterile 200 ml provetta PCR seguita da una breve impulso vortex. Ogni campione deve avere 0,5 ml di rovescio fondo RT21 e 0,5 ml di dNTP, vedi Tabella 2.
- Aliquotare 1,0 microlitri della miscela dNTP-primer a 200 microlitri tubi PCR.
- Preparare trascrittasi inversa (RT) miscela enzimatica aggiungendo 1 μ, L del tampone trascrizione inversa 10x, 1 ml di 0,1 M DTT e 2 ml di 25 mM MgCl 2 in una provetta sterile seguito da vortex e centrifugare brevemente, vedere tabella 3.
- Aggiungere 0,5 microlitri ciascuno degli enzimi RNAseOUT e Apice III trascrittasi inversa al tubo mix enzima quindi toccare la provetta delicatamente per miscelare.
- Tenere le provette con i dNTP mix-primer e mix enzimatico su un blocco freddo e passare alla stazione di RNA.
4. Trascrizione inversa
- Aggiungere 6 ml di campione di RNA al tubo dNTP mix-primer seguito da brevemente vortex per miscelare.
- Dopo l'aggiunta del RNA, passare alla camera PCR sia con dNTP / Primer / mix RNA e RT Enzyme mescolare tubi su un blocco fredda o ghiaccio.
- Centrifugare brevemente le provette dNTP / Primer / RNA mix (dal punto 4.2) e metterli in un termociclatore.
- Riscaldare a 65 ° C per 5 minuti per denaturare l'RNA.
- Rapidamente raffreddare a 4 ° C, tenere premuto per 2min.
- Pausa termociclatore mentre ancora a 4 ° C; estrarre i tubi.
- Aggiungere rapidamente 5 ml di miscela enzimatica mantenendo i tubi su un blocco di raffreddamento.
- Mescolare delicatamente toccando il tubo quindi centrifugare brevemente le provette e tornare al termociclatore.
- Tenere le provette a 50 ° C per 60 min per invertire trascrivere RNA seguita da denaturazione enzimatica a 85 ° C per 5 minuti per fermare la trascrizione inversa.
- Raffreddare a 37 ° C. Non appena la temperatura arriva a 37 ° C, mettere in pausa e prendere il tubo dal termociclatore.
- Aggiungere rapidamente 0,5 ml di RNAsi H ai tubi e tornare al termociclatore.
- Tenere a 37 ° C per 20 minuti e poi raffreddare a 4 ° C.
- Il DNA complementare (cDNA) può essere utilizzato immediatamente o può essere conservato a -20 ° C o inferiore fino a quando necessario. Tuttavia, la conservazione a lungo termine di cDNA dovrebbe essere a -80 ° C.
5. Preparazione dei reagenti per la PCR
- Before partendo, calcolare il volume di ciascuno dei reagenti necessari per il numero di campioni in lavorazione ed i controlli. Oltre ai tre controlli (positivo, negativo, e reagenti), è anche possibile aggiungere un controllo PCR (DNA HIV). Il primo e secondo miscele rotondi PCR possono essere preparati e contemporaneamente il secondo master mix conservati a -20 ° C fino al momento dell'uso. Miscele possono essere conservate per circa 8 ore.
- Aggiungere 18,4 microlitri di acqua, 2,5 microlitri tampone 10x, 1,0 microlitri MgCl 2, 0,5 microlitri dNTP, e 0,25 ml di ciascuno dei primer come indicato nella Tabella 4 e vortice.
- Aggiungere 0.1 ml di Platinum Taq polimerasi (5U/μl) e mescolare delicatamente il tubo toccandola.
- Aliquota 23 ml di master mix a 200 provette microlitri PCR.
- Con i tubi master mix su un blocco di raffreddamento o spostare ghiaccio per la camera PCR.
6. Nested PCR
- Aggiungere 2 ml di cDNA a 23 ml di 1 ° round PCR Master mescolare.
- Chiudere i cilindri, mettere i campioni nel termociclatore e utilizzare le seguenti condizioni di ciclo di PCR: 94 ° C per 2 min, 30 cicli di 95 ° C per 30 sec, 58 ° C per 20 sec, e 72 ° C per 2 min, seguita da una estensione finale a 72 ° C per 10 min, come mostrato in Figura 1.
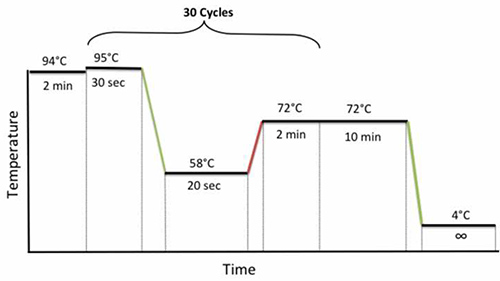
Figura 1. Condizioni di ciclismo nested PCR. Clicca qui per vedere l'immagine ingrandita.
- Proseguire fino alla 2 ° turno fase PCR o memorizzare i prodotti di PCR primo turno a -20 ° C o inferiore fino al momento in una fase successiva.
- Per rotondo PCR secondo, aggiungere 2 ml di 1 ° turno prodotto di PCR a 23 ml di 2 ° turno di PCR master mixd utilizzare lo stesso programma PCR in Figura 1.
7. Elettroforesi su gel
- Preparazione Gel
- Aggiungere 0,5 g di agarosio compressa in un pallone di vetro da 250 ml e aggiungere 50 ml di 1x tampone TBE al pallone.
- Calore nel forno a microonde per ebollizione; agitare frequentemente (circa ogni 30 secondi) fino a completa solubilizzazione. Utilizzare una presa di silicone o silicone forno guanto per afferrare il pallone caldo. La soluzione di agarosio può far bollire fuori del pallone molto facilmente in modo da seguire da vicino questo processo.
- Raffreddare a temperatura ambiente per circa 10 minuti.
- Versare agarosio in un cassetto del gel contenente appropriato pettine dimensione; gel è pronto per l'uso in circa 20-30 min.
- Mettere gel in camera di elettroforesi e gestito come raccomandato dal costruttore.
- Elettroforesi su gel e visualizzazione.
- Vortex Juice Novel per 10 secondi prima dell'uso.
- Diluire 1 ml di succo di Novel con 5 ml di DCampione di NA e mix.
- Diluire 3 ml di succo di Novel con 3 ml di marcatore di peso molecolare e mescolare.
- Caricare i mix dalle sezioni 7.2.2 e 7.2.3 (sopra) ed eseguire il gel a 100 V e 400 mA per 40 min per valutare l'amplificazione PCR.
- Amplificazione positivo può essere visualizzati sotto luce UV come frammento 1.315 bp, Figura 2.
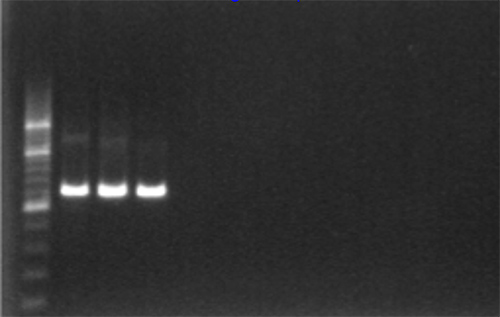
Figura 2. Conferma Gel di amplificazione PCR utilizzando 1% gel elettroforesi e una scala di 200 bp. Clicca qui per vedere l'immagine ingrandita.- Non ci dovrebbe essere nessuna amplificazione nei controlli negativi e reagenti, indicando così l'assenza di contaminazione.
8. PCR Cleanup prodotto
- In preparazione per la reazione di sequenziamento, i secondi prodotti PCR rotondi positivi vengono eliminati utilizzando il kit di purificazione PCR PureLink.
- Aggiungere 80 ml di lavoro binding buffer ad alta Cutoff (B3) a 20 ml di prodotto di PCR e mix pipetta.
- Aggiungere il campione miscelato con il tampone di legame a una colonna di spin in un tubo di raccolta.
- Centrifugare la colonna a 10.000 xg per 1 min. Trasferire la colonna in un nuovo tubo di raccolta.
- Lavare la colonna con 650 ml di tampone di lavaggio con etanolo.
- Centrifugare la colonna a 10.000 xg per 1 min. Trasferire la colonna in un nuovo tubo di raccolta.
- Centrifugare la colonna alla massima velocità per 2-3 minuti per rimuovere qualsiasi residuo di tampone di lavaggio.
- Porre la colonna di spin in un ambiente pulito provetta 1,7 ml di eluizione fornito con il kit.
- Aggiungere 40 ml di tampone di eluizione al centro della colonna e incubare la colonna in camera temperature per 1 min.
- Centrifugare la colonna alla massima velocità per 2 min (> 10.000 xg).
- Il tubo di eluizione contiene il prodotto purificato PCR pronto per il sequenziamento. Eliminare la colonna.
- Determinare la concentrazione e la qualità del DNA utilizzando un Nanodrop.
- Se non ci sono strutture di sequenziamento in-house sono disponibili, i prodotti di PCR purificati possono essere inviati a un laboratorio di sequenziamento imprese in questa fase.
9. Reazioni di sequenziamento
- I prodotti di PCR sono sequenziati utilizzando il kit big dye terminator versione 3.1 e 4 iniettori per ogni campione (due avanti e due retromarce). Le sequenze dei primer sono mostrati nella Tabella 2. Pertanto, dopo la corsa di sequenziamento, ogni campione avrà quattro sequenze essere assemblati in un contig.
- Impostare le reazioni di sequenziamento come indicato nella tabella 5 per ciascuna delle quattro primer.
- Mescolare il tampone di sequenziamento e primer da vortex prima dell'uso.
- Mescolare ilacqua, tampone e primer prima della aggiunta del grande sequenziamento colorante. Mescolare nel vortex.
- Mescolare delicatamente il master mix dopo aver aggiunto il grande mix di sequenziamento colorante invertendo il tubo o picchiettando delicatamente.
- Aliquotare 9 microlitri della master mix in una lamina ottica a 96 pozzetti.
- Al fine di eseguire 24 campioni / piastra, impostare la piastra come indicato di seguito Figura 3.
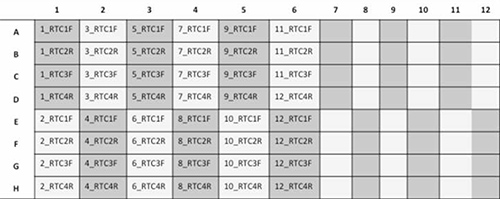
Figura 3. Rappresentazione Schema di una piastra a 96 pozzetti con 12 campioni di pazienti in fase sequenziati con 4 iniettori ciascuno (RTC1F, RTC2R, RTC3F, e RTC4R). Clicca qui per vedere l'immagine ingrandita.
- Aggiungere 1,0 ml di campione di DNA (~ 20-40 ng), coprire la piastra con unn adesivo coperchio in alluminio e poi mescolare delicatamente.
- Centrifugare a 3000 xg per 1 min. Togliere il coperchio di alluminio e aggiungere un tappetino di tenuta in gomma.
- Posizionare la piastra sul termociclatore ed eseguire il seguente programma ciclismo illustrato nella Figura 4.
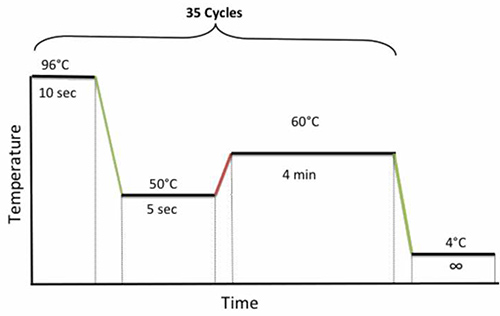
Figura 4. PCR condizioni di ciclo per il sequenziamento. Clicca qui per vedere l'immagine ingrandita.
- Al termine del PCR, ripulire il prodotto sequenziamento immediatamente.
10. Sequencing Cleanup
- Per ogni reazione di sequenziamento, miscelare 50 ml di etanolo assoluto e 5 microlitri 3 M acetato di sodio.
- Usando una pipetta multicanale, aggiungere 55 &# 956; l di soluzione di acetato di sodio / EtOH a ciascun pozzetto.
- Sigillare i pozzetti con coperchio in alluminio adesivo, in modo che ogni pozzetto è sigillato correttamente.
- Centrifugare a 3000 xg per 20 min.
- Dopo 20 minuti, togliere il coperchio e capovolgere la piastra, in un unico movimento fluido, su un tessuto di laboratorio piegato (NON battere per sbarazzarsi di surnatante in quanto ciò staccare il pellet!).
- Centrifugare la piastra capovolta sullo stesso tessuto a 150 xg per 2 minuti.
- Aggiungere immediatamente 150 ml freddo il 70% EtOH. NON ritardare aggiunta di etanolo in questa fase.
- Sigillare con la stessa copertina di alluminio adesivo e vortex.
- Centrifugare a 3000 xg per 5 min.
- Invertire piastra su un nuovo tessuto piegato e centrifugare invertita a 150 xg per 1 min.
- Dopo la centrifugazione, collocare scoperto nel termociclatore e asciugare a 50 ° C per 2 min.
- Una volta che i piatti siano asciutti, sigillarlo con adesivo foglio adesivo, avvolgere nella pellicola e conservare a -20 ˚ C fino al momento di procedere with elettroforesi sequenziamento.
- Quando si è pronti a sequenza, sciogliere i prodotti di sequenziamento puliti in 10 ml Hi-Di formammide, denaturare e carico per elettroforesi.
11. Bioinformatica
- Assemblea Sequence
- Avviare il programma Geneious.
- Creare una cartella di lavoro per memorizzare le sequenze.
- Importare i file ABI generati dalla macchina sequenziamento alla cartella di lavoro utilizzando lo strumento di importazione. Geneious assegnerà percentuale di punteggio di qualità per ogni sequenza importata.
- Sequenze aperti con i punteggi di qualità> 70% con un doppio clic su di essi.
- Ogni file dovrebbe aprire in una nuova finestra. Il software indicherà la qualità in ogni posizione nucleotidica del cromatogramma della qualità sequenza usando luce barre blu. Il livello della barra, migliore è la qualità della chiamata base.
- Con il cursore, selezionare la sezione centrale della sequenza lasciando fuori le estremità, che di solito sono di scarsa qualità.
- Fare clic sul pulsante Estrai per estrarre la regione con sequenza di buona qualità.
- Seleziona tutti i quattro sequenze estratte per ogni campione e montarli contro una sequenza di riferimento.
- Ispezionare la sequenza assemblata per garantire che si trovi in fase di lettura corretta. Se siete in fase di lettura corretta, l'inizio della proteasi dovrebbe iniziare con i seguenti aminoacidi: PQITLW. L'inizio della RT inizierà con PISPIE.
- Estrarre la regione contiguo che copre l'inizio del PR a 300 ° RT codone. Durante questo processo, controllare anche per le inserzioni o delezioni.
- Passare attraverso la sequenza di consenso del contig estratto, individuare eventuali ambiguità e verificare le posizioni con basi misti ispezionando qualità (simmetria, altezza, sfondo e le spalle delle regioni fiancheggianti) delle chiamate di base.
- Selezionare la sequenza consenso e fare clic sul pulsante Estrai per creare un file separato della sequenza di consenso dai quattro primer e Label modo appropriato.
- Esportare la sequenza di una cartella di archiviazione di backup sul computer o una cartella di rete.
- Sequenza Quality Assessment (HIVDB)
- Analizzare la sequenza utilizzando il programma HIVDB a http://hivdb.stanford.edu .
- Verificare la presenza di delezioni e inserzioni nei dati di sintesi e accertarsi che la sequenza copre tutte le 99 della proteasi (PR) codoni e le prime 300 RT codoni.
- Verificare la presenza evidenziata la garanzia della qualità problemi (QA) in entrambe le regioni PR e RT, come codoni di stop, telaio turni, le posizioni ambigue e residui insoliti.
- Controllo di qualità Sequencing
- Blast la nuova sequenza in un database di sequenze locale dalla corsa precedente.
- Se la nuova sequenza è> 97% simile a qualsiasi sequenza del database, tutte le fasi del protocollo devono essere riesaminati, iniziando con l'analisi di sequenza e tornare alla estrazione di RNA per assicue che non ci sono inghippi (switching campione, mislabeling) o di contaminazione.
- Se vengono individuati problemi, ripetere l'analisi sia dei vecchi e nuovi campioni della fase di estrazione di RNA.
- Se le sequenze sono ancora simile> 97%, rivedere la storia del paziente per valutare qualsiasi legame epidemiologico tra gli individui.
- L'analisi filogenetica
- Allineare tutte le sequenze del database utilizzando il programma ClustalW in Geneious.
- Controllare manualmente l'allineamento di sequenze allineati, eliminazioni e inserimenti e modificare di conseguenza.
- Costruire un albero filogenetico con PHYML, Geneious albero costruttore o di altri costruttori albero in Geneious.
- Esaminare l'albero per campioni con lunghezze breve filiali.
- Rivedere i campioni con lunghezze di diramazione per possibile contaminazione.
12. REGA DB Informatica
- Sequenza Carica
- Accedere al RegadB utilizzando un nome utente e una password univoci.
- Dal menu a discesa, sotto ID paziente, selezionare "Inizia con".
- Aggiungere l'ID del paziente e seleziona la persona il cui genotipo è da caricare.
- Dal menu a sinistra, selezionare "isolato virale".
- Dalle opzioni sotto l'isolato virale selezionare "add".
- Immettere la data Campione, ID campione, ID Sequenza e data sequenza.
- Seleziona "Scegli file" e quindi individuare il file FASTA della sequenza da caricare.
- Dopo aver selezionato il file FASTA da caricare, fare clic su Upload.
- Una volta caricata la sequenza viene visualizzato nella casella nucleotide sotto le identifica e le date di sequenza, fare clic sul pulsante OK in basso a destra della finestra.
- Verificare la presenza di PR e RT allineamento proteina facendo clic sul pulsante di proteine e selezionando PR o RT.
- Controllare la mutazione resistenza ai farmaci cliccando sul pulsante resistenza. Questo dài profili di resistenza da tre algoritmi: ANRS, Stanford HIVDB e RegaDB.
- Generazione di report utilizzando REGA
- Accedere al RegaDB utilizzando il tuo username e password.
- Dal menu a discesa, sotto ID paziente, selezionare "Inizia con".
- Aggiungere l'ID del paziente e seleziona la persona il cui rapporto deve essere generato.
- Dal menu di destra, selezionare l'isolato virale.
- Dalle opzioni sotto l'isolato virale clicca su "view".
- Fare doppio clic sul isolato virale per il quale si desidera creare un report.
- Nella finestra isolato virale, fare clic sulla scheda rapporto isolato virale.
- Selezionare gli algoritmi per l'interpretazione del genotipo dal menu a discesa e quindi selezionare modello di rapporto da utilizzare.
- Una volta selezionati l'algoritmo e il modello, fare clic sul pulsante "generate".
- Scarica il documento rtf generato.
- Aprire il rtf farecumento come documento word.
- Ridimensionare il grafico cronologico trattamento.
- Dopo il grafico, aggiungere la sezione "cartella clinica e l'interpretazione resistenza".
- Utilizzando i dati sulla tabella di resistenza e cartella clinica, aggiungere una descrizione del profilo di resistenza del paziente iniziando con la storia di trattamento del paziente, e farmaci per cui il ceppo virale è resistente. Aggiungere anche una descrizione della carica virale del paziente e CD4 + profili conta delle cellule dal grafico.
- Inviare il report per le Malattie Infettive (ID) specializzati per la revisione e raccomandazioni sulla futura gestione del paziente. Questo processo è anche una fase molto importante garanzia di qualità. Eventuali errori nel genotipo o incongruenze nella storia del trattamento, profili virologici e immunologici possono essere identificati e recensione prima di un rapporto finale è inviato, con tutte le raccomandazioni per la gestione clinico del paziente.
Risultati
Il metodo convalidato era una modifica di un metodo riportato in precedenza 20. Il metodo di genotipizzazione Viroseq, che è stato approvato dalla FDA, è stato utilizzato come metodo di riferimento per la convalida. Un gruppo di campioni di prova di abilità ottenuti dalle Agenzie nazionali francesi per la ricerca sull'AIDS e l'epatite virale (ANRS) è stato utilizzato nel confronto principale tra i due metodi. I due metodi di genotipizzazione sono state del 100% concorde nell'individuare tutti clinicamente importanti mutazioni associate alla resistenza droga come interpretato dal programma HIVDB per i campioni che sono stati amplificati con successo con entrambi i metodi. Come mostrato in Tabella 6, le sequenze nucleotidiche dei tre coppie erano 99,5% identico. Le sequenze amminoacidiche predette erano identici al 100%. Un campione su cinque non poteva essere amplificato con successo da Viroseq. In aggiunta al campione non amplificato dal Viroseq, il metodo in-house omesso di amplificare un secondo campione che è stato mostratoessere un virus ricombinante circolante (CRF02_AG) da Viroseq. I tre campioni amplificati con entrambe le metodologie erano sottotipo B (due campioni) e sottotipo A (un campione).
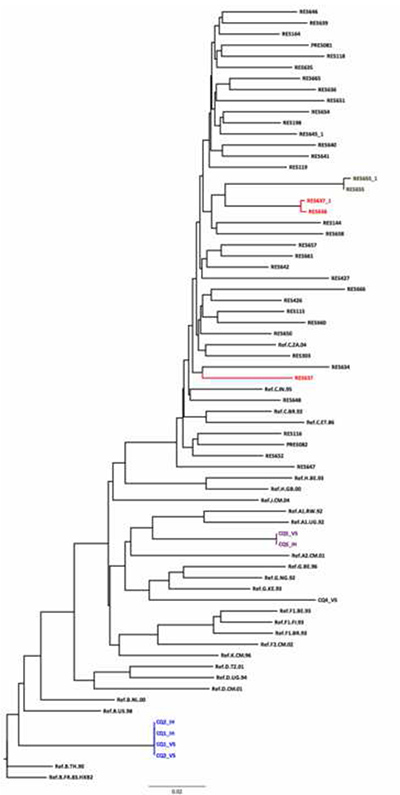
Figura 5. L'uso di un vicino di giunzione albero HKY fatto come parte della garanzia della qualità sequenza. Ci sono quattro coppie / gruppi di sequenza con distanze molto brevi genetiche. La distanza genetica tra RES655 e RES655_1 (stessi campioni sequenziati in giorni diversi) è 0.003. Il è un potenziale errore con la coppia RES637_1/RES638 come la loro distanza genetica è troppo breve (0.075) per i campioni provenienti da diversi individui epidemiologico scollegate. C'è un altro RES637 sull'albero con una distanza di 0,075 rispetto al RES638_1. Il cluster CQ01/CQ02 suggerisce che i due campionidal pannello sono duplicati dello stesso campione. Essi raggruppano insieme alla sequenza di riferimento sottotipo B confermando il sottotipo assegnato dallo strumento REGA Subtyping. CQ05 e CQ04 raggruppati con sottotipi A e G, rispettivamente, considerando che lo strumento REGA subtyping li ha classificati come A e CRF02_AG rispettivamente. Un altro strumento utile per subtyping HIV e ricombinazione è SCUEL, che è disponibile a http://www.datamonkey.org. Clicca qui per vedere l'immagine ingrandita.
Un gruppo di cinque campioni è stato utilizzato per valutare la precisione del metodo in house. Dieci genotipi replicati sono stati generati per ciascuno dei cinque campioni. Utilizzando l'analizzatore genetico 16 Capillare 3130xl, 48 dei 50 genotipi sono stati generati da 24 piste, preparate lo stesso giorno. Per tutti i cinque campioni, le sequenze amminoacidiche predette erano 100% concorde tra replicati. Per le sequenze di acido nucleico, there era> 99% di somiglianza a coppie.
Durante i primi due anni di utilizzo di questo metodo, sessanta campioni sono stati ripetuti in modo casuale da estrazione di RNA a sequenziamento. Non ci sono state differenze statisticamente significative tra il punteggio di qualità sequenza e il numero di basi misti tra le repliche. Sia il nucleotidiche e amminoacidiche confronti a coppie per i sessanta coppie erano superiore al 99% identico. Così le mutazioni di farmacoresistenza per tutte le coppie erano 100% concorde.
Riduzione dei costi
I volumi di reazione di RT, PCR e sequenziamento sono stati ridotti di almeno la metà, rispetto al metodo originale 20, 32, senza compromettere la qualità delle sequenze generate. Ciò ha consentito una riduzione dei costi del 50% per le fasi RT e PCR.
Il nuovo metodo è stato originariamente progettato per funzionare con sei sequenziamento primer sequenza tutti i 99 codoni del gene della proteasi e le prime 300 codoni della trascrittasi inversa gene 20, 32. Metodi simili usano anche sei a otto iniettori 33, 34. Alcuni metodi recentemente pubblicati hanno utilizzato meno di sei primer, anche se a volte il sequenziamento dei geni della proteasi e RT seprately 35, 36. Si è cercato di ridurre il numero di primer di sequenziamento da sei a quattro, (figura 6)
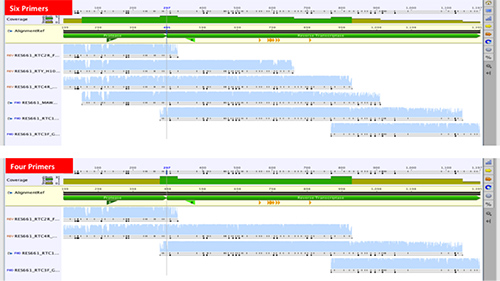
Figura 6. Confronto di sequenze contigue sei confrontarli quattro primer di sequenziamento per la generazione della sequenza pol 1197 bp che copre tutti i 99 HIV-1 proteasi codoni e le prime 300 codoni del gene trascrittasi inversa.242/51242fig6highres.jpg "target =" _blank "> Clicca qui per vedere l'immagine ingrandita.
Sequenze di una serie di 17 campioni generati da sei inneschi sono stati confrontati con sequenze generate dopo l'esclusione di due primer (MAW46 e RTY). I sottotipi erano 14 sottotipo C, due sottotipo B, e un sottotipo A. Non ci sono state differenze significative nei punteggi di qualità sequenza. Ancora una volta, l'identità pairwise media tra le 17 coppie di acido nucleico è stata del 99% e il 100% sul livello amminoacidico. Così, riducendo i primer di sequenziamento da sei a quattro determinato una riduzione del costo sequenziamento di quasi un terzo.
L'unico strumento software proprietario utilizzato in questo protocollo è stato Geneious per l'assemblaggio sequenza. Gli strumenti di interpretazione farmaco-resistenza, nonché la relazione di generare strumenti sono tutti gratuiti, strumenti di accesso aperto. Questo riduce il costo ulteriore eliminando i costi associati con l'uso di software proprietario. Inoltre, collective negoziazione ha consentito i reagenti per questo protocollo da confezionare in un kit per un facile accesso da Life Technologies ed è disponibile come Saturn / Life Technologies metodo 37 genotipizzazione. Inoltre, i membri Saturno può accedere ai reagenti ad un prezzo scontato.
Ambito Clinico
Il protocollo descritto è stato implementato il monitoraggio e la sorveglianza della resistenza ai farmaci in una comunità rurale in KwaZulu-Natal. Un totale di 604 genotipi sono stati generati da campioni clinici tra dicembre 2010 e maggio 2013 con un tasso di amplificazione del 95% per i campioni con carica virale> 1.000 RNA copie / ml. Questo studio clinico di HIV farmaco-resistenza è stato approvato dal Comitato Etico di Ricerca Biomedica dell'Università di KwaZulu-Natal (rif. BF052/10) e il Comitato Health Research del Dipartimento di KwaZulu-Natal della Salute (rif. HRKM 176/10). Relazioni dei singoli pazienti sono stati generati e rispediti alle clinicheper la gestione del paziente.
Settantadue (72) genotipi sono stati generati come parte di una sorveglianza della resistenza ai farmaci trasmessa studio, annidati all'interno di un ampio studio prospettico di sorveglianza HIV basato sulla popolazione. I campioni primari erano l'ago puntura sangue intero raccolto in microtubi EDTA. Al genotipizzazione c'era un tasso di amplificazione di 79% 19. Approvazione etica per la genotipizzazione di campioni dallo studio di sorveglianza è stato ottenuto presso l'Università di KwaZulu-Natal Ricerca Biomedica Comitato Etico (rif. BE066107).
| Nome Primer | Sequenza | Lunghezza | Direzione | Posizione HXB2 | |
| MAW-26 | TTGGAAATGTGGAAA GGAAGGAC | 23 | Avanti | 2028-2050 | 1 ° Turno PCR |
| RT-21 | CTGTATTTCAGCTATC AAGTCCTTTGATGGG | 31 | Reverse | 3539-3509 | 1 ° Turno PCR |
| Pro-1 | TAGAGCCAACAGCCC cacca | 20 | Avanti | 2147-2166 | Turno PCR seconda |
| RT-20 | CTGCCAATTCTAATTC TGCTTC | 22 | Reverse | 3462-3441 | Turno PCR seconda |
| RTC1F | ACCTACACCTGTCAA CATAATTG | 23 | Avanti | 2486-2508 | Sequencing |
| RTC2R | TGTCAATGGCCATTG TTTAACCTTTGG | 27 | Reverse | 2630-2604 | Sequencing |
| RTC3F | CACCAGGGATTAGAT ATCAATATAATGTGC | 30 | Avanti | 2956-2994 | Sequencing |
| RTC4R | CTAAATCAGATCCTAC ATACAAGTCATCC | 29 | Reverse | 3129-3101 | Sequencing |
| RT-y | GTGTCTCATTGTTTAT ACTAGG | 22 | Reverse | 2967-2946 | Sequencing |
| MAW-46 | TCCCTCAGATCACTC TTTGGCAACGAC | 27 | Avanti | 2251-2277 | Sequencing |
Tabella 1. Trascrizione, PCR e sequenziamento primer personalizzati utilizzati nella generazione di un frammento di 1197 bp pol copre tutti i 99 HIV-1 proteasi codoni e le prime 300 codoni del gene trascriptase inversa inverse.
| RT21 (5pmol/ml) | 0.5 | 0.2 |
| dNTP (10 mM) | 0.5 | 0.4 |
| Totale | 1 |
Tabella 2. dNTP / Primer miscela per la reazione di trascrizione inversa.
| Reagente | Volume (ml) / reazione | Concentrazione / reazione |
| First Strand Buffer (10x) | 1 | 1 |
| MgCl 2 (25 mm) | 2 | 4 |
| DTT (0,1 M) | 1 | 0.008 |
| RNaseOUT (40 U / ml) | 0.5 | 16 |
| Apice III trascrittasi inversa (200U/ml) | 0.5 | 8 |
| Totale | 5 |
Tabella 3. Enzima miscela per la reazione di trascrizione inversa.
| Reagente | Volume (ml) / reazione | Concentrazione Finale / Reazione |
| DEPC acqua trattata | 18.4 | - |
| PCR Buffer (10x) | 2.5 | 1 |
| MGCI 2 (50 mm) | 1 | 2 |
| miscela dNTP (10 mM) | 0.5 | 0.2 |
| Primer Froward (5 pmol / ml) | 0.25 | 0.05 |
| Primer reverse (5 pmol / ml) | 0.25 | 0.05 |
| Platinum Taq Polymerase (5 U / ml) | 0.1 | 0.02 |
| Totale parziale | 23 | - |
Tabella 4. Master mix per la nested PCR.
| Reagente | Volume (ml) / reazione | Concentrazione / reazione |
| DEPC acqua trattata | 6.1 | |
| Sequencing Buffer (5x) | 2 | 1 |
| Primer (3.2 pmol / ml) | 0.5 | 0.16 |
| Big Dye terminator mix Sequencing | 0.4 | - |
| Totale | 9 |
Tabella 5. Master mix per le reazioni di sequenziamento.
| Viroseq | Inhouse | % NA somiglianza | |||||||
| ID campione | Sottotipo | Punteggio di qualità | PR Mutazioni | Mutazioni RT | Sottotipo | Punteggio di qualità | Mutazioni PR | RT Mutazioni | |
| CQ01 | B | 99.9 | M46L, I54L, V82A, L90M | D67N, T69D, K70R, M184V, T215V, K219Q | B | 99.2 | M46L, I54L, V82A, L90M | D67N, T69D, K70R, M184V, T215V, K219Q | 100 |
| CQ02 | B | 99.5 | M46L, I54L, V82A, L90M | D67N, T69D, K70R, M184V, T215V, K219Q | B | 99.5 | M46L, I54L, V82A, L90M | D67N, T69D, K70R, M184V, T215V, K219Q | 100 |
| CQ03 | NA | NA | NA | NA | NA | NA | |||
| CQ04 | CRF02_AG | 98.4 | I54V, V82F, I84V | M41L, L74I, L210W, T215Y, V108I, Y181C | NA | NA | NA | NA | NA |
| CQ05 | La | 99,7 | K103N | La | 93 | K103N | 100 | ||
Tabella 6. Resul comparativats da una analisi parallela tra il metodo di genotipizzazione Viroseq e il metodo internamente con un pannello di campioni forniti dal ANRS.
Discussione
Diversi low cost metodi in-house sono state descritte negli sforzi per cercare di rendere l'HIV farmaco-resistenza genotipizzazione più abbordabile 33, 34, 36. Non vi è alcun dubbio sulla necessità di integrare test di resistenza ai farmaci nel continuum di cura per le persone in terapia antiretrovirale in paesi a risorse limitate. Tuttavia, la maggior parte dei metodi riportati riguardare l'applicazione di farmacoresistenza genotipizzazione nella sorveglianza della resistenza ai farmaci a livello di popolazione. Il metodo di genotipizzazione Saturno / Life Technologies è un protocollo completamente integrato per la sorveglianza e il monitoraggio della resistenza ai farmaci. Questo metodo è stato progettato per essere un protocollo di attuazione conveniente soprattutto open source e aperto le risorse di bioinformatica di accesso per l'interpretazione della resistenza ai farmaci e la generazione di report per la gestione clinica.
E 'stato dimostrato attraverso il confronto con il metodo di genotipizzazione Viroseq approvato dalla FDA per essereaccurate per identificare mutazioni di resistenza droga da un panel di ANRS campioni di prova di abilità, nel 100% dei campioni di pannelli di laboratorio che sono stati amplificati con successo. La precisione è stata valutata anche su campioni clinici di virus sottotipo C, il sottotipo più dominante in Africa australe. Il metodo era esatto sul sottotipo campioni C come era il sottotipo A e B. Tuttavia, se il metodo potrebbe essere utilizzato in altre parti del mondo dove CRF02_AG è prevalente, vi è la necessità per la modifica dei primer in quanto il metodo fallito per amplificare uno dei campioni del pannello che è stato dimostrato di avere CRF02_AG. In alternativa, una serie di primer degenerato sensibili a tutto gruppo M virus 33, 36 potrebbe essere utilizzata in regioni in cui la distribuzione sottotipo è più eterogenea 38.
La sensibilità della trascrizione inversa e PCR può essere aumentata con l'estrazione di RNA da maggiori volumi di plasma, come 500 ml. Il plasma può essere centrifuged a 21.000 xg per 90 min a concentrare le particelle virali prima di procedere con il protocollo come descritto dal mini kit di estrazione di RNA virale QIAamp.
Come mostrato, il nuovo metodo ha un ulteriore vantaggio che produce rapporti completi per la gestione del singolo paziente. Queste relazioni sono un consolidamento del genotipo, il immunologici e virologici dati di monitoraggio così come la storia clinica e il trattamento da RegaDB. Questo è accompagnato da una interpretazione laboratorio dettagliata del profilo di resistenza seguita da una revisione altrettanto dettagliata della storia clinica del paziente e raccomandazioni di trattamento. L'utilizzo di un medico specialista per esaminare i rapporti e fornire raccomandazioni per il trattamento per i pazienti fornisce mentorship tanto necessaria per infermieri e medici inesperti, che sono sempre forniscono ART in Sud Africa come parte delle raccomandazioni dell'OMS per le attività di spostamento. Questi clinicarelazioni hanno dimostrato di essere strumenti didattici efficaci per i medici con poca o nessuna esperienza nella gestione della resistenza ai farmaci. Dal punto di vista del paziente, il nostro metodo riduce la necessità di recarsi in luoghi centralizzati per accedere ai servizi di HIV specialistici.
Pertanto, il protocollo descritto nel suo complesso offre una buona piattaforma attraverso la quale l'HIV gestione farmacoresistenza può essere integrato, ad un costo accessibile, nel continuum di cure per individui infetti da HIV in mancanza ART. I dati generati possono essere usate per scopi epidemiologici per valutare l'evoluzione e trasmissione di farmacoresistenza nella comunità. La dimensione del frammento pol generato è sufficiente per più complessa analisi filogenetica che produrrà una migliore comprensione della epidemia a livello di popolazione.
Divulgazioni
Questo lavoro è stato supportato dal Wellcome Trust (082384/Z/07/Z), Unione europea (SANTE 2007 147-790), il Centro statunitense per il controllo delle malattie tramite CAPRISA (titolo del progetto: rafforzamento dei sistemi sanitari e di fallimento del trattamento HIV (HIV- TFC)), e il programma Swiss South African comune di ricerca (SSJRP) assegno di ricerca dal titolo "Swiss Prot / Sudafrica: Proteine Bioinformatica Sviluppo delle risorse per importanti patogeni relative alla salute". RL è supportato dal Wellcome Trust (codice di autorizzazione 090.999 / Z / 09 / Z). i finanziatori hanno avuto alcun ruolo nel disegno dello studio, la raccolta e l'analisi dei dati, la decisione di pubblicare, o preparazione del manoscritto. Gli autori dichiarano interessi finanziari concorrenti.
Riconoscimenti
Gli autori desiderano ringraziare tutti i colleghi che hanno reso possibile questo lavoro, in particolare Maya Balamane, Elizabeth Johnston Bianco, Sharon Sjoblom, Greg Ording Zakhona Gumede, Xolile Kineri, Phindile Mabaso, Lungisa Ndwandwe, James Garvey, Gavin Cobb, Senzo Maphanga, Terusha Chetty , Kevi Naidoo, Andrew Skingsley, Katharine Stott, e Lungani Ndwandwe. Gli autori desiderano inoltre ringraziare tutto il personale del Dipartimento di Salute e personale Africa Centro che lavorano la Hlabisa trattamento di HIV e il programma Care.
Materiali
| Name | Company | Catalog Number | Comments |
| Superscript III 1st strand Synthesis kit | Life Technologies | 18080051 | Reverse Transcription |
| SATURN/LiFE Technologies Custom Primers | Life Technologies | 4473517 | PCR |
| Platinum Taq | Life Technologies | 10966026 | PCR |
| PureLink QUICK PCR Purification Kit | Life Technologies | K310002 | PCR |
| Viroseq | ABBOTT | 4J94-20 | Reverse Transcription and PCR |
| Agarose Tablets (Dnase/Rnase free) | BIOLINE | BIO-41027 | PCR |
| TBE Buffer | MERCK | 1.06177.2500 | PCR |
| O'Range Ruler 200 bp DNA Ladder | Fermentas | FE SM0633 | PCR |
| Novel Juice | GeneDireX | LD001-1000 | PCR |
| MiniBis Bioimaging System | DNR Bioimaging Systems Ltd | Gel Documentation | |
| Power Pac 300 | BIORAD | Gel Electrophoresis | |
| Big Dye Terminator Kit version 3.1 | Life Technologies | 4337456 | Sequencing |
| Arrays | Life Technolgies | 4319899 | Sequencing |
| PoP | Life Technologies | 4363785 | Sequencing |
| 10x EDTA Buffer | Life Technologies | 402824 | Sequencing |
| Formamide | Life Technologies | 4311320 | Sequencing |
| 5x Sequencing Buffer | Life Tecgnologies | 4336697 | Sequencing |
| 3130 xl Genetic Analyzer | Life Technologies | Sequencing | |
| GeneAmp PCR System 9700 | Life Technologies | RT/PCR/Sequencing | |
| Centrifuge 5804 | EPPENDORF | Sample Processing | |
| Centrifuge 5415R | EPPENDORF | RNA Extraction | |
| Centrifuge 5415R | EPPENDORF | RT and PCR | |
| Centrifuge 5415D | EPPENDORF | PCR Product Clean up | |
| Centrifuge 5810 | EPPENDORF | Sequencing Clean up | |
| Picofuge | BIORAD | C1301-230V | RT and PCR |
| Vortex Genius 3 | IKA | RNA extraction and reagent preparation | |
| Vortex mixer | IKA | Sequencing Cleanup | |
| NanoDrop 2000 UV/VIS spectrophotometer | ThermoScientific | DNA quantification | |
| 3 M Sodium Acetate | MERCK | 567422 | Sequencing Clean up |
| Absolute Ethanol | MERCK | SAAR2233540LP | Sequencing Cleanup |
| 1.5 ml SARSTEDT Tubes | BIODEX | 72.692.005 | RNA Extraction |
| 2 ml SARSTEDT Tubes | BIODEX | 72.693.005 | RNA Extraction |
| 2 ml Collection tubes | SCIENTIFIC GROUP | MCT-200-NC/S | RNA Extraction |
| Optical MicroAmp 96-well reaction plates | Life Technologies | N8010560 | Sequencing |
| 200 µl 8 Strip StarPCR Tubes with attached flat caps | STAR Lab - supplied by CELTIC | A1402-3700 | RT and PCR |
| 200 µl PCR individual tubes | Scientific Group | CR/3745 | RT and PCR |
| Geneious | Biomatters | Sequence analysis | |
| Internet Access | Preferrably high speed | ||
| Web resources | |||
| hivdb.stanford.edu | Stanford University | Drug reistance analysis | |
| http://bioafrica.mrc.ac.za:8080/regadb-ui/RegaDB | SATuRN | database | |
| http://bioafrica.mrc.ac.za/tools/pppweb.html | SATuRN | Sequence quality tool | |
Riferimenti
- Shao, Y., Williamson, C. The HIV-1 epidemic: low- to middle-income countries. Cold Spring Harbor Persp. Med. 2, (2012).
- Mutevedzi, P. C., et al. Scale-up of a decentralized HIV treatment programme in rural KwaZulu-Natal, South Africa: does rapid expansion affect patient outcomes. Bull. World Health Organ. 88, 593-600 (2010).
- Houlihan, C. F., et al. Cohort profile: Hlabisa HIV treatment and care programme. Int. J. Epidemiol. 40, 318-326 (2011).
- Tanser, F., Barnighausen, T., Grapsa, E., Zaidi, J., Newell, M. L. High coverage of ART associated with decline in risk of HIV acquisition in rural KwaZulu-Natal. South Africa. Science. 339, 966-971 (2013).
- Bor, J., Herbst, A. J., Newell, M. L., Barnighausen, T. Increases in adult life expectancy in rural South Africa: valuing the scale-up of HIV treatment. Science. 339, 961-965 (2013).
- Montaner, J. S., et al. The case for expanding access to highly active antiretroviral therapy to curb the growth of the HIV epidemic. Lancet. 368, 531-536 (2006).
- Granich, R. M., Gilks, C. F., Dye, C., De Cock, K. M., Williams, B. G. Universal voluntary HIV testing with immediate antiretroviral therapy as a strategy for elimination of HIV transmission: a mathematical model. Lancet. 373, 48-57 (2009).
- Johnson, L. F., et al. Life expectancies of South african adults starting antiretroviral treatment: collaborative analysis of cohort studies. PLoS Med. 10, (2013).
- Blower, S., Ma, L., Farmer, P., Koenig, S. Predicting the impact of antiretrovirals in resource-poor settings: preventing HIV infections whilst controlling drug resistance. Curr. Drug Targets. 3, 345-353 (2003).
- Geretti, A. M. Epidemiology of antiretroviral drug resistance in drug-naive persons. Curr. Opin. Infect. Dis. 20, 22-32 (2007).
- Larder, B. A., Darby, G., Richman, D. D. HIV with reduced sensitivity to zidovudine (AZT) isolated during prolonged therapy. Science. 243, 1731-1734 (1989).
- Erice, A., et al. Brief report: primary infection with zidovudine-resistant human immunodeficiency virus type 1. N. Engl. J. Med. 328, 1163-1165 (1993).
- Williams, I., et al. British HIV Association guidelines for the treatment of HIV-1-positive adults with antiretroviral therapy. HIV Med. 13 Suppl 2, 1-85 (2012).
- DHHS, . US Panel on Antiretroviral Guidelines for Adults and Adolescents. Guidelines for the use of antiretroviral agents in HIV-1-infected adults and adolescents. , (2012).
- Vandamme, A. M., et al. European recommendations for the clinical use of HIV drug resistance testing: 2011 update. AIDS Rev. 13, 77-108 (2011).
- DHHS, . US Panel on Antiretroviral Therapy and Medical Management of HIV-Infected Children. Guidelines for the Use of Antiretroviral Agents in Pediatric HIV Infection. , (2012).
- . Department of Health. Clinical guidelines for the management of HIV & AIDS in adults and adolescents. , (2010).
- Conradie, F., et al. The 2012 southern African ARV drug resistance testing guidelines. S. Afr. J.HIV Med. 13, 162-167 (2012).
- Manasa, J., et al. Primary Drug Resistance in South Africa: Data from 10 Years of Surveys. AIDS Res. Hum. Retroviruses. 28, 558-565 (2012).
- Tshabalala, M., et al. Surveillance of transmitted antiretroviral drug resistance among HIV-1 infected women attending antenatal clinics in Chitungwiza, Zimbabwe. PLoS ONE. 6, (2011).
- Bartolo, I., et al. Antiretroviral drug resistance surveillance among treatment-naive human immunodeficiency virus type 1-infected individuals in Angola: evidence for low level of transmitted drug resistance. Antimicrob. Agents Chemother. 53, 3156-3158 (2009).
- Bartolo, I., et al. HIV-1 genetic diversity and transmitted drug resistance in health care settings in Maputo, Mozambique. J. Acquir. Immune Defic. Syndr. 51, 323-331 (2009).
- Hamers, R. L., et al. HIV-1 drug resistance in antiretroviral-naive individuals in sub-Saharan Africa after rollout of antiretroviral therapy: a multicentre observational study. Lancet Infect. Dis. 11, 750-759 (2011).
- Hamers, R. L., et al. HIV-1 Drug Resistance Mutations Are Present in Six Percent of Persons Initiating Antiretroviral Therapy in Lusaka, Zambia. J. Acquir. Immune Defic. Syndr. , (2010).
- Nwobegahay, J., Selabe, G., Ndjeka, N. O., Manhaeve, C., Bessong, P. O. Low prevalence of transmitted genetic drug resistance in a cohort of HIV infected naive patients entering antiretroviral treatment programs at two sites in northern South Africa. J. Med. Virol. 84, 1839-1843 (2012).
- Iweriebor, B. C., et al. Molecular epidemiology of HIV in two highly endemic areas of northeastern South Africa. Arch. Virol. 157, 455-465 (2012).
- Parboosing, R., Naidoo, A., Gordon, M., Taylor, M., Vella, V. Resistance to antiretroviral drugs in newly diagnosed, young treatment-naive HIV-positive pregnant women in the province of KwaZulu-Natal South Africa. J. Med. Virol. 83, 1508-1513 (2011).
- Nwobegahay, J. M., et al. Prevalence of antiretroviral drug resistance mutations and HIV-I subtypes among newly-diagnosed drug-naive persons visiting a voluntary testing and counselling centre in northeastern South Africa. J. Health Popul. Nutr. 29, 303-309 (2011).
- Nwobegahay, J., et al. Prevalence of drug-resistant mutations in newly diagnosed drug-naive HIV-1-infected individuals in a treatment site in the waterberg district, limpopo province). S. Afr. Med. J. 101 (2011), 335-337 (2011).
- Rosen, S., Long, L., Sanne, I., Stevens, W. S., Fox, M. P. The net cost of incorporating resistance testing into HIV/AIDS treatment in South Africa: a Markov model with primary data. J. Int. AIDS Soc. 14, 24 (2011).
- Dalai, S. C., et al. Evolution and molecular epidemiology of subtype C HIV-1 in Zimbabwe. AIDS. 23, 2523-2532 (2009).
- Chen, J. H., et al. In-house human immunodeficiency virus-1 genotype resistance testing to determine highly active antiretroviral therapy resistance mutations in Hong Kong. Hong Kong Med. 18, 20-24 (2012).
- Lee, C. K., et al. An in-house HIV genotyping assay for the detection of drug resistance mutations in Southeast Asian patients infected with HIV-1. J. Med. Virol. 84, 394-401 (2012).
- Aitken, S. C., et al. A Pragmatic Approach to HIV-1 Drug Resistance Determination in Resource-Limited Settings by Use of a Novel Genotyping Assay Targeting the Reverse Transcriptase-Encoding Region Only. J. Clin. Microbiol. 51, 1757-1761 (2013).
- Zhou, Z., et al. Optimization of a low cost and broadly sensitive genotyping assay for HIV-1 drug resistance surveillance and monitoring in resource-limited settings. PLoS One. 6, (2011).
- , Life Technologies. Life Technologies and SATuRN Collaborate to Increase Access to HIV Testing in Africa. , (2012).
- Lihana, R. W., Ssemwanga, D., Abimiku, A., Ndembi, N. Update on HIV-1 diversity in Africa: a decade in review. AIDS Rev. 14, 83-100 (2012).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon