Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
De Fluorescencia Fast Imaging a la Ley de Difusión Molecular en membranas de células vivas en un microscopio Comercial
En este artículo
Resumen
Spatial distribution and temporal dynamics of plasma membrane proteins and lipids is a hot topic in biology. Here this issue is addressed by a spatio-temporal image fluctuation analysis that provides conceptually the same physical quantities of single particle tracking, but it uses small molecular labels and standard microscopy setups.
Resumen
Es cada vez más evidente que la distribución espacial y el movimiento de los componentes de la membrana, como lípidos y proteínas son factores clave en la regulación de muchas funciones celulares. Sin embargo, debido a la dinámica rápida y las diminutas estructuras involucradas, se requiere una resolución espacio-temporal muy alta para capturar el comportamiento real de las moléculas. Aquí presentamos el protocolo experimental para el estudio de la dinámica de las proteínas y los lípidos del plasma membrana fluorescencia marcado en células vivas con alta resolución espacial y temporal. En particular, este enfoque no es necesario realizar un seguimiento de cada molécula, pero calcula comportamiento de la población utilizando todas las moléculas en una región dada de la membrana. El punto de partida es una imagen rápida de una determinada región de la membrana. Después, una función de autocorrelación espacio-temporal completa se calcula la correlación de imágenes adquiridas a aumentar los retardos de tiempo, por ejemplo cada 2, 3, n repeticiones. Es posible demostrar que el anchodel pico de los incrementos espaciales función de autocorrelación en el aumento de retardo de tiempo como una función del movimiento de las partículas debido a la difusión. Por lo tanto, encajando de la serie de funciones de autocorrelación permite extraer la proteína real significa desplazamiento cuadrático de formación de imágenes (IMSD), que aquí se presenta en forma de difusividad aparente vs desplazamiento medio. Esto produce una vista cuantitativo de la dinámica promedio de moléculas individuales con una precisión nanométrica. Mediante el uso de una variante de GFP-etiquetados del receptor de transferrina (TfR) y un ATTO488 el nombre 1-palmitoil-2-hidroxi-sn-glicero-3 fosfoetanolamina (PPE) es posible observar la regulación espaciotemporal de la difusión de proteínas y lípidos en regiones de membrana-micras de tamaño en el rango de tiempo-micro-para mili-segundos.
Introducción
A partir del modelo original "mosaico fluido" de Singer y Nicolson, la imagen de la membrana plasmática celular se ha actualizado continuamente durante las últimas décadas con el fin de incluir el papel emergente de citoesqueleto y lípidos dominios 1,2.
Las primeras observaciones fueron obtenidos por la recuperación de fluorescencia después de photobleaching (FRAP) descubrimiento de que una fracción significativa de proteínas de la membrana es inmóvil 3-5. Estos estudios pioneros, aunque muy informativo, sufrieron de la relativamente pobre resolución en el espacio (micras) y el tiempo (segundos) de las configuraciones del FRAP. Además, al ser una medida promedio de conjunto, FRAP le falta en dar información sobre el comportamiento de una sola molécula.
En este contexto, la posibilidad de etiquetar específicamente una única molécula con etiquetas muy brillantes (lo que permite el estudio del proceso de difusión de una molécula a la vez) ha sido muy exitosa. Particularmente, empujando elresolución en el tiempo del enfoque único de rastreo de partículas (SPT) de la escala de tiempo de microsegundos, Kusumi, et al. obtenido acceso a características desconocidas de la dinámica de lípidos y proteínas que contribuyeron en gran medida al reconocimiento del papel de esqueleto de la membrana actina basada en la fisiología de la membrana 6 , 7. Estos resultados generan la llamada el "piquete y la valla" modelo, en el que los lípidos y la proteína está regulada por la difusión esqueleto basado en actina. Sin embargo, con el fin de tener acceso a la enorme cantidad de información proporcionada por SPT muchos temas experimentales que abordar. En particular, el procedimiento de etiquetado se compone típicamente por muchos pasos como la producción, la purificación y la introducción de las especies marcadas en el sistema. Por otra parte, las grandes etiquetas, al igual que los puntos cuánticos o nanopartículas metálicas, a menudo se requieren para llegar a la escala de tiempo sub-milisegundo y la reticulación de las moléculas diana por la etiqueta no se podría evitar en muchos casos. Por último, muchas trayectoriastienen que ser registrado para adaptarse a criterios estadísticos y concomitantemente se requiere una baja densidad de la etiqueta para permitir el seguimiento.
En comparación con SPT, espectroscopia de correlación de fluorescencia (FCS), la superación de muchos de estos inconvenientes, representa un enfoque muy prometedor para estudiar la dinámica molecular. De hecho, FCS también funciona bien con etiquetas tenues y densas, lo que permite estudiar la dinámica de moléculas de proteínas etiquetadas fluorescentes en las células transfectadas transitoriamente. Además, permite alcanzar altas estadísticas en una cantidad limitada de tiempo. Por último, a pesar de la densidad de "alta" de etiquetas FCS proporciona información moléculas individuales. Gracias a todas estas propiedades, FCS representa un enfoque muy sencillo y se ha aplicado ampliamente para estudiar la dinámica de lípidos y proteínas tanto en membranas modelo y en las células vivas 8-10. Se han propuesto muchos enfoques diferentes para aumentar la capacidad de FCS para revelar los detalles de la difusión molecular. Por ejemplo, era shpropia que mediante la realización de FCS en áreas de observación de diferentes tamaños se puede definir una "ley de difusión FCS" características esclarecedoras ocultos de movimiento molecular 11,12. Además de ser variado en tamaño, la esfera de actividad también se duplicó 13, se trasladó en el espacio a lo largo de las líneas 14-20 o conjugados con cámaras rápidas 21,22. El uso de estos correlación 'espacio-temporal' enfoques, parámetros biológicos relevantes de varios componentes de la membrana se designarán cuantitativamente por ambas membranas modelo y las biológicas reales, por tanto, una visión que dé en la membrana organización espacial.
Sin embargo, en toda la FRAP y aplicaciones FCS descrito hasta ahora el tamaño de la esfera de actividad representa un límite de la resolución espacial que no puede ser superada. Varios métodos de imagen super-resolución se han desarrollado recientemente para eludir este límite. Algunos se basan en la precisión de localización, como la microscopía óptica reconstrucción estocástico (TORMENTA) 23,24, microscopía de localización de fotoactivación (Palm) 25, PALM fluorescencia (FPALM) 26, y de una sola partícula de seguimiento de palma (sptPALM) 27: la cantidad relativamente grande de fotones requeridas en cada instantánea, sin embargo, limita la resolución de tiempo de estos métodos a por lo menos varios milisegundos, lo cual dificulta su aplicabilidad en vivo.
En contraste, una alternativa prometedora para formación de imágenes de súper resolución haber sido abierto por modular espacialmente la emisión de fluorescencia con los métodos de agotamiento de la emisión estimulada (STED o transiciones reversibles saturables ópticos de fluorescencia (RESOLFT)) 28,29. Estos enfoques se combinan la conformación del volumen de observación muy por debajo del límite de difracción con la posibilidad de usar microscopios de barrido rápido y sistemas de detección. En combinación con el análisis de fluctuación de fluorescencia, microscopía STED permitió a la sonda directamente la dinámica espacio-temporales a nanoescala de lípidos y proteins en las membranas celulares en vivo 30,31.
Las mismas cantidades físicas de microscopia basado en STED se pueden obtener por una espectroscopia de correlación de imagen espacio-temporal método modificado (STICS 32,33) que es adecuado para el estudio de la dinámica de las proteínas etiquetadas con fluorescencia de membrana y / o lípidos en células vivas y por un microscopio comercial. El protocolo experimental que se presenta aquí está compuesto por dos pasos. La primera de ellas requiere una imagen rápida de la región de interés en la membrana. Entonces, la pila resultante de imágenes se utiliza para calcular las funciones de correlación promedio espacio-temporales. Mediante el ajuste de la serie de funciones de correlación, la "ley de difusión 'molecular puede ser obtenida directamente de formación de imágenes en forma de una difusividad aparente (D app) - vs -Promedio trazado de desplazamiento. Esta parcela depende de manera crítica sobre el medio ambiente explorado por las moléculas y permite reconocer directamente los modos de difusión realesdel lípido / proteína de interés.
En más detalles, como se indica anteriormente 34, la función de auto-correlación espacio-temporal de la serie de imagen adquirida depende fundamentalmente de la dinámica de las moléculas que se mueven en la serie de imágenes recogido (por favor, tenga en cuenta que el mismo razonamiento se puede aplicar en una adquisición de la línea donde se considera sólo una dimensión en el espacio). En particular, definimos la función de correlación como:
 (1)
(1)
donde  representa la intensidad de fluorescencia medido en la posición x, y y en el tiempo t,
representa la intensidad de fluorescencia medido en la posición x, y y en el tiempo t, ![]() y
y ![]() representa la distancia en la x yY. respectivamente,
representa la distancia en la x yY. respectivamente, ![]() representa el intervalo de tiempo, y
representa el intervalo de tiempo, y ![]() representa la media. Esta función puede expresarse como:
representa la media. Esta función puede expresarse como:
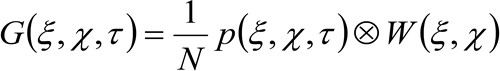 (2)
(2)
donde 'N' representa el número medio de moléculas en el área de observación, ![]() representa la operación de convolución en el espacio, y
representa la operación de convolución en el espacio, y 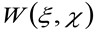 representa la autocorrelación de la cintura instrumental. Este último puede interpretarse como una medida de cómo están distribuidos los fotones de un solo emisor en el espacio debido a la configuración óptica / grabación (el llamado Point Spread Function, PSF, genRally bien aproximada por una función de Gauss). Finalmente,
representa la autocorrelación de la cintura instrumental. Este último puede interpretarse como una medida de cómo están distribuidos los fotones de un solo emisor en el espacio debido a la configuración óptica / grabación (el llamado Point Spread Function, PSF, genRally bien aproximada por una función de Gauss). Finalmente, 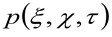 representa la probabilidad de encontrar una partícula a una distancia
representa la probabilidad de encontrar una partícula a una distancia ![]() y
y ![]() después de un tiempo de retardo
después de un tiempo de retardo ![]() . Si tenemos en cuenta una dinámica de difusión, en el que las partículas se mueven al azar en todas las direcciones y los flujos netos no están presentes, esta función también está bien aproximada por una función gaussiana donde la varianza puede ser identificado como el Mean Square Desplazamiento (MSD) de la partícula en movimiento . Por lo tanto, la cintura de la función de correlación (también referido como
. Si tenemos en cuenta una dinámica de difusión, en el que las partículas se mueven al azar en todas las direcciones y los flujos netos no están presentes, esta función también está bien aproximada por una función gaussiana donde la varianza puede ser identificado como el Mean Square Desplazamiento (MSD) de la partícula en movimiento . Por lo tanto, la cintura de la función de correlación (también referido como ![]() ), Se puede definir como la suma de los trastornos musculoesqueléticos de partículas y la cintura instrumental y puede ser medida por un ajuste de GaussTing de la función de correlación para cada retardo de tiempo. La medida i MSD se puede utilizar para calcular una aparente difusividad de las moléculas que se mueven
), Se puede definir como la suma de los trastornos musculoesqueléticos de partículas y la cintura instrumental y puede ser medida por un ajuste de GaussTing de la función de correlación para cada retardo de tiempo. La medida i MSD se puede utilizar para calcular una aparente difusividad de las moléculas que se mueven ![]() y un desplazamiento medio
y un desplazamiento medio ![]() como:
como:
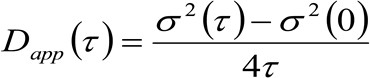 (3)
(3)
 (4)
(4)
Algunas consideraciones sobre la configuración experimental utilizado puede guiar al lector a través de las siguientes secciones. Con el fin de excitar selectivamente los fluoróforos en la membrana basal de las células que utilizaremos una iluminación reflexión interna total (TIR) que viven, utilizando una fluorescencia TIR comercial (TIRF) microscopio (los detalles se pueden encontrar en la sección de materiales). Por otra parte, con el fin de recoger ªe fluorescencia vamos a utilizar un objetivo de gran aumento (100X NA 1,47, se requiere una alta apertura numérica para iluminación TIRF) y una cámara de EMCCD (tamaño físico del píxel en el chip 16 micras). Para llegar a un tamaño de píxel de 100 nm aplicamos una lente de aumento adicional de 1.6X. Como se discute más adelante, una resolución de tiempo por debajo de 1 mseg sería necesario para describir adecuadamente la dinámica de los lípidos de membrana rápido por debajo de 100 nm. Para llegar a esta resolución temporal que necesitamos para seleccionar una región de interés (ROI) más pequeño que todo el chip de la cámara (512 x 512). De esta manera, la cámara leer un número reducido de líneas de aumento de la resolución en el tiempo. Sin embargo, en este régimen de lectura de la hora marco estaría limitado por el tiempo requerido para cambiar los cargos de la exposición a la lectura del chip de la cámara y está normalmente en el orden de milisegundos para EMCCD 512 x 512 píxel. Para superar este límite, una tecnología emergente permite desplazar las líneas de retorno de la inversión sólo en lugar de todo el marco, won una reducción práctica efectiva del tamaño del chip expuesto (llamado Modo Sensor recortada en nuestra EMCCD). Para que esta configuración sea eficaz, el chip fuera de la ROI debe estar cubierto por un par de hendiduras montados en el camino óptico. Gracias a esta configuración una resolución de tiempo hasta 10 -4 segundo se puede lograr. Tenga en cuenta, sin embargo, que este enfoque puede combinarse con diversos montajes experimentales, como se explica en la sección de "discusión".
Demostración del método que se proporcionará en células vivas, usando tanto un marcado ATTO488 1-palmitoil-2-hidroxi-sn-glicero-3 fosfoetanolamina (ATTO488-PPE) y una variante de GFP marcado del receptor de transferrina (GFP TfR). En el caso de ATTO488-PPE este enfoque puede recuperar con éxito una aplicación casi constante D como una función del desplazamiento medio indica una difusión sobre todo libre, como se informó anteriormente 30,35. Por el contrario, TfR-GFP muestra una disminución D aplicación como una función del desplazamiento promedio, lo que sugiere la difusión parcialmente confinado-6. Además, en el último caso, es posible cuantificar la constante de difusión local y el área promedio de confinamiento durante muchos micras en el plano de la membrana.
Protocolo
Calibración 1. Sistema
- Spread Function Point (PSF) de calibración
- Diluir 10 l de 30 nm solución de bolas fluorescentes (aproximadamente 5 mM) en 90 l de agua destilada y luego someter a ultrasonidos la solución durante 20 min. Cortar un cuadrado (1 cm x 1 cm) pieza de gel de agarosa (3%) y depósito de 10 l de la solución en la parte superior del gel. Volcar la pieza de gel en el cristal de fondo de un plato Petri 2 cm y exprimir la caída en el cristal.
- A su vez en la configuración de la adquisición, se coloca la muestra en el soporte, ajuste la exposición de la cámara y EMgain (100 ms y 1000 son buenos parámetros pero optimizan según el sistema) y esperar a que la cámara se enfríe.
- Ajuste la exposición de la cámara a 100 ms, cámara EMgain a 1000, el modo de adquisición al fotograma de transferencia, 100 repeticiones y auto ajuste de ahorro.
- Usando el ocular y enfoque la luz transmitida en la frontera del gel y luego mover el objetivo hacia el centro del gel, ajustar el enfoque y STAta el procedimiento de alineación láser (en LAS AF, seleccione "Configuración TIRF 'y siga el procedimiento de alineación automática).
- Encuentra un campo de visión con manchas individuales aisladas, precisa centrarse en el punto más brillante (que por lo general representa los granos agregado) como referencia, adquirir 100 cuadros y repita el paso 5-6 veces con el fin de adquirir varios puntos individuales.
- Importar la serie adquirida a un programa de procesamiento de datos y un promedio de la pila en el tiempo (Figura 1A) y seleccionar un único glóbulo aislado. Tenga cuidado de seleccionar los más pequeños para evitar agregados de partículas.
- Montar la distribución seleccionada intensidad (un ejemplo de perfil único cuentas se presenta en la Figura 1B) con una función de Gauss con el comando "gaussfit" (en las herramientas ICS-Matlab en los Materiales en Matlab). Verificar la bondad del ajuste mediante la inspección de los residuos obtenidos (un ejemplo de perfil gaussiano equipada con los residuos correspondientes i se presentan Figura 1B).
- Calibración de la cámara
- Encienda la cámara y espere a que la cámara se enfríe. AJUSTE adquisición de la cámara, (es decir, para la cámara utilizada fijamos la exposición a 0,5 ms, cámara EMgain a 1000, el modo de adquisición de modo recortada, el tamaño ROI de 32 x 128, 10.000 repeticiones) y comenzar la adquisición del fondo de la cámara señal.
- Importación adquirió serie del marco de un programa de procesamiento de datos. Calcular e inspeccionar la intensidad media en cada píxel con el fin de verificar que el fondo de la cámara es aproximadamente plana en la región seleccionada del chip. En el modo recortada, retire la primera y las últimas líneas horizontales (3 a 10 dependiendo del tamaño de la ROI) para cada trama porque el fondo de cámara está normalmente sesgado en las líneas fronterizas.
- Crear un histograma de los valores (también definido de nivel digital, DL) en imágenes adquiridas Stack (con el comando 'hist' en Matlab) y trazar el logaritmode resultar frecuencia (con el comando semilogy en Matlab). Un ejemplo de la distribución de DL para el fondo de la cámara se presenta en la Figura 2.
NOTA: Si la cámara está funcionando bien, el gráfico mostrará un pico de aproximadamente gaussiana (un perfil parabólico en el diario de escala) que representa la distribución de los valores asociados al fotón cero seguido de un decaimiento exponencial (una recta con pendiente negativa en el diario de escala ) que representa la distribución de los valores asociados a 1 de fotones (Figura 2). En particular, el centro y la varianza de la función gaussiana representan el desplazamiento de la cámara y de error, respectivamente, mientras que la constante de desintegración de la parte exponencial representa una estimación de la DL asignado por la cámara para cada fotón único. En Matlab utilizar la sección "CalibrateCamera" de la escritura en los materiales de apoyo. - Repita la operación para toda la cámara seleccionada EMGain y Ganancia.
2. EtiquetadaPreparación de células
- Para preparar los liposomas requeridas para la incorporación de lípidos 36, se disuelven por separado 1 mg de DOPE (1,2-dioleoyl- sn-glicero-3 fosfoetanolamina), 1 mg de DOTAP (1,2-dioleoil-3-trimetilamonio-propano), y 1 mg de PPE-ATTO488 en 1 ml de cloroformo. Mezclar 0,5 ml de solución de DOPE, 0,5 ml de solución de DOTAP, y 25 l de solución de PPE-ATTO488 y secar a vacío durante 24 horas. Añadir 0,5 ml de tampón HEPES 20 mM, vórtice durante 15 minutos y se somete a ultrasonidos durante 15 min a 40 ° C.
- Para preparar la celda, lavar 3 veces con PBS un plato p100 de confluente CHO-K1 (ovario de hámster chino), añadir 1 ml de tripsina y almacenar en incubadora durante 5 min. Suspender células desprendidas adición de 9 ml de medio DMEM / F12 suplementado con 10% de FBS y SEED 150 l de solución de células en una placa de Petri que contiene 800 l del mismo medio.
- Almacenar en incubadora durante 24 horas a 37 ° C y 5% de CO 2. Para la incorporación de lípidos, reemplace medio celular con 500l de medio libre de suero; después de 30 minutos, añadir 2 l de solución de liposomas; después de 15 minutos de lavado con PSB y añadir nuevo medio DMEM / F12 para imágenes.
- Para la transfección, transfectar células según el protocolo Lipofectamine (las instrucciones del fabricante) utilizando el plásmido TfR-GFP y tienda de 24 horas en la incubadora antes de exponer.
3. Adquisición de Datos
- Preparación de instalación
- Con el fin de termostato el microscopio, 24 h antes del experimento encender la incubadora.
- Con el fin de aplicar el tiempo de adquisición más rápido posible, el trabajo en el modo de sensor recortada (véase la introducción) y el uso de una primera cámara para obtener imágenes (cámara 1) y la segunda cámara para seleccionar la celda (cámara 2). Un esquema de la configuración de la instalación se presenta en la Figura complementario S1. Entonces, para alinear el turno dos cámaras en el microscopio y esperar a que las cámaras se enfríen.
- Ubicado en ambas cámaras los parámetros para la imagen de luz transmitida (es decir, , 20 ms para el tiempo de exposición, 1 para EM Gain) y poner el microscopio en el modo Campo Claro.
- Poner las muestras en el soporte y enfocar con el ocular, enviar la luz a la cámara 1 y empuje suavemente hacia las ranuras permitiendo que la luz sólo en el retorno de la inversión se utiliza para obtener imágenes de células (en este caso un ROI de 32 x 32 píxeles).
- Mover una celda de la región seleccionada y enviar la luz a la cámara 2, a continuación, dibuje un retorno de la inversión en el software que controla la cámara 2 con el fin de tener una referencia.
- Imaging (Figura 3A)
- En primer lugar, alinear el láser TIRF acuerdo con el procedimiento de su configuración. En nuestra configuración, seleccione el 'setup TIRF' e iniciar el procedimiento de alineación automática. Cuando está alineado el láser establecer 70 nm de profundidad de penetración (aproximadamente 70 °).
- Establecer el tiempo de exposición a 70 ms y EMGain a 100 tanto en las cámaras 1 y 2; a continuación, seleccione una celda usando la cámara 1, a continuación, enviar la luz en la cámara 2 y enfocar con precisión la membrana celular. Ajuste la exposición mínima en caMera 2, 1000 EMGain, modo de sensor recortada, 10 5 repeticiones y autoguardado establecer como archivos FITS (Flexible Image Transport System, un formato que puede ser fácilmente administrado).
- Iniciar la adquisición de grabar la serie de imágenes. Suelte la ganancia y el modo recortada para permitir la estabilización de temperatura antes de adquirir un nuevo celular, a continuación, repita los dos últimos pasos para la adquisición de 8-10 células.
4. Cálculo del Mean Square Desplazamiento de imágenes (i MSD)
NOTA: El siguiente protocolo se puede aplicar directamente a los datos en bruto. Al mismo tiempo, todo el protocolo es válido para adquisiciones de datos simulados tanto en Matlab y en SimFCS. El enlace a los tutoriales correspondientes se pueden encontrar en la sección "Materiales".
- Cálculo por Matlab
- Importar la serie adquirida a Matlab usando la escritura ImportImageSeries. Calcular la intensidad media de cada imagen en el tiempo mediante el command significar en los primeros 2 dimensiones y el uso de parcela para ver el vector resultante.
- Si más del 10% de photobleaching está presente, desechar la serie o eliminar la primera parte de ellos. Si es inferior, tratar de corregir el efecto sobre la función de correlación restando a cada imagen su intensidad media, como se muestra antes de 37.
- Calcular el promedio de intensidad de cada píxel mediante el uso de media en la tercera dimensión y ver la imagen resultante.
Se requiere una atención especial para evitar correlaciones de artefactos: NOTA. De hecho, como se muestra anteriormente para técnicas similares 38, los bordes de celda, así como de las vesículas de enfoque podría introducir una fuerte correlación. Si la inspección de la imagen promedio revela los bordes de celda o de vesículas de enfoque, tratar de excluir la región que participan de lo contrario descartar la adquisición. Para corregir el efecto de estas estructuras inmóviles restar la intensidad media temporal de cada píxel 39. - Calcular tél correlación espacio-temporal (G (ξ, χ, τ)) mediante el uso de la función CalculateSTICScorrfunc. Eliminar G (ξ, χ, 0) porque la correlación debido al ruido de un disparo en régimen de poca luz domina G (0,0,0); la correlación debido al domina el detector G (± 1,0,0), y el movimiento de las partículas durante el tiempo de exposición podría deformar G (ξ, χ, τ) para τ = 0 mediante el aumento de la cintura medido (este efecto desaparece para τ > 0) 34.
- Promedio de G (ξ, χ, τ> 0) usando un tiempo de-bin logarítmica para reducir el ruido mediante el uso de la función "LogBinStack" en el material de apoyo y luego se ajusta el G resultante (ξ, χ, τ) usando la función "gaussfit" de las herramientas ICS-Matlab en los Materiales para recuperar el i MSD (la segunda columna de la matriz resultante).
- Trazar la σ obtenido de la cintura (τ) 2 (i MSD) como una función del tiempo. Si los datos son demasiado ruidosos, tratar de aumentar el número de acqumarcos IRED, aumentan la potencia del láser, la media más G (ξ, χ, τ) juntos.
- Cálculo por SimFCS
- Abra los archivos adquiridos con ImageJ usando BIOFORMAT importador plugin y guardar serie adquirida como secuencia Tiff.
- Abrir herramienta SimFCS y seleccione RICS y seleccione Archivo> Importar imágenes múltiples (Figura suplementaria S2).
- Seleccione Fit, introduzca los parámetros de adquisición correctos y cerrar la ventana de ajuste (Figura complementario S3).
- Seleccione Pantalla> Intensidad Media> CH1 y verificar la presencia de photobleaching (que complementa la figura S4).
- Si más del 10% de photobleaching está presente descarte la serie o si es posible la carga de nuevo la secuencia de imágenes de retirar la primera parte de la serie.
- Si blanquear es inferior al 10% seleccione Herramientas> i MSD> Ajustar parámetros, reviso 'Uso de media móvil', situado en el panel de retorno de la inversión de la izquierda unúmero de marco para la atención de pagar de media móvil que el tiempo corresponsal es mayor que el tiempo de difusión característica (por partícula que se mueve a 1 m 2 s-1 un tiempo de 10 segundos es una buena media móvil)
- Seleccione Herramientas> IMSD> Calcular i MSD (Figura S5) y en forma y exportar el i MSD del cojín de nota (Figura S6).
5. Cálculo de la Ley de Difusión de la i MSD
- Montar los primeros puntos para extrapolar el intercepto (σ 0 2) (5 puntos suelen ser suficiente, pero más puntos se pueden instalar si muestran un comportamiento lineal) y comparar este valor con el PSF medido previamente 2. Si son comparables, se están siguiendo la dinámica de fluoróforos aisladas. Por el contrario, si σ 0 2 >> PSF 2 tratar de adquirir más rápido para asegurar quedinámica ocultos están presentes 34.
- Calcular la difusividad aparente (app D) y el desplazamiento medio (R) utilizando las ecuaciones 3 y 4 (véase la introducción).
- Parcela aplicación D como una función de R para obtener una ley de difusión comparable con lo que se mide con la variación lugar basado FCS 12 (Figura 3D).
Resultados
Con el fin de calibrar la cintura instrumental, la imagen de un solo cordón de nano-fluorescente puede ser medida como se describe en el Protocolo paso 1.1. Una imagen fluorescente típica de estas cuentas se presenta en la Figura 1. La instalación de distribución de intensidad mediante una función gaussiana 2D devuelve buenas residuos y permite la medición de la cintura instrumentales a 270 nm. Este valor está en buen acuerdo con el límite de difracción esperado estimado por la ecuación de Ray...
Discusión
Rastreo de partículas Individual (SPT) representa una de las estrategias más comunes para estudiar la dinámica molecular y tiene la gran ventaja de la medición de trayectorias de las partículas. Esto a su vez permite sondear el comportamiento de incluso pocas partículas marcadas en un sistema complejo. Sin embargo, para alcanzar esta ventaja SPT típicamente necesita una baja densidad de la sonda y etiquetas muy brillantes. Particularmente, para obtener alta resolución temporal (rango mu s) sonda inorgánico norm...
Divulgaciones
The authors have nothing to disclose.
Agradecimientos
This work is supported in part by NIH-P41 P41-RRO3155 and NIH P50-GM076516 (grant to EG), and Fondazione Monte dei Paschi di Siena (grant to FB).
Materiales
| Name | Company | Catalog Number | Comments |
| iXon Ultra 897 | Andor | DU-897U-CS0 | |
| Solis | Andor | ||
| CHO-K1 | ATCC | CCL-61 | |
| ATTO 488 labeled PPE | ATTO-TEC GmbH | AD 488-151 | |
| DOPE | Avanti Polar Lipids, Inc. | 850725 | |
| DOTAP | Avanti Polar Lipids, Inc. | 890890 | |
| 100x Penicillin-Streptomycin-Glutamine | Gibco | 10378-016 | |
| DMEM/F-12 | Gibco | 21331 | |
| FBS | Gibco | 10082147 | |
| HEPES | Gibco | 15630-106 | |
| PBS | Gibco | 10010-023 | |
| SimFCS 3.0 | Globals Software | the software can be downloaded here: http://www.lfd.uci.edu/globals/ | |
| DMI6000 with TIRF modulus | Leica | ||
| LAS AF | Leica | ||
| Lipofectamine 2000 | Lipofectamine | 11668019 | |
| Matlab | MathWork | ||
| ImageJ | NIH | ||
| [header] | |||
| C-terminal GFP tagged Tranferrin Receptor | OriGene | RG200980 | |
| Agar | Sigma Aldrich | A5306 | |
| Chloroform | Sigma Aldrich | 528730 | |
| Latex beads, fluorescent yellow-green, 30 nm | Sigma Aldrich | L5155 | |
| SONICA Ultrasonic Cleaners | SOLTEC | ETH S3 | |
| Petri Dishes | Willco | GWSt-3522 | |
| Bio-Format importer for Matlab | http://www.openmicroscopy.org/site/support/bio-formats5/users/matlab/ | ||
| ICS-MatLab Tools | https://www.cellmigration.org/resource/imaging/software/ICSMATLAB_28-02-06.zip | ||
| Simulation by Matlab Tutorial | https://www.cellmigration.org/resource/imaging/icsmatlab/ICSTutorial.html | ||
| Simulation by SimFCS Tutorial | https://www.cellmigration.org/resource/imaging/ppt-pdf/RICS%20Simulations.ppt | ||
Referencias
- Engelman, D. M. Membranes are more mosaic than fluid. Nature. 438 (7068), 578-580 (2005).
- Vereb, G., et al. yet structured: The cell membrane three decades after the Singer-Nicolson model. Proc. Natl. Acad. Sci. U. S. A. 100 (14), 8053-8058 (1073).
- Ishihara, A., Hou, Y., Jacobson, K. The Thy-1 antigen exhibits rapid lateral diffusion in the plasma membrane of rodent lymphoid cells and fibroblasts. 84 (5), 1290-1293 (1987).
- Axelrod, D., et al. Lateral motion of fluorescently labeled acetylcholine receptors in membranes of developing muscle fibers. Proc. Natl. Acad. Sci. U. S. A. 73 (12), 4594-4598 (1976).
- Jacobson, K., Derzko, Z., Wu, E. S., Hou, Y., Poste, G. Measurement of the lateral mobility of cell surface components in single, living cells by fluorescence recovery after photobleaching. J. Supramol. Struct. 5 (4), 10-1002 (1976).
- Kusumi, A., et al. Paradigm shift of the plasma membrane concept from the two-dimensional continuum fluid to the partitioned fluid: high-speed single-molecule tracking of membrane molecules. Annu. Rev. Biophys. Biomol. Struct. 34, 351-378 (2005).
- Kusumi, A., Ike, H., Nakada, C., Murase, K., Fujiwara, T. Single-molecule tracking of membrane molecules: plasma membrane compartmentalization and dynamic assembly of raft-philic signaling molecules. Semin. Immunol. 17 (1), 3-21 (2005).
- Schwille, P., Korlach, J., Webb, W. W. Fluorescence correlation spectroscopy with single-molecule sensitivity on cell and model membranes. Cytometry. 36, 176-182 (1999).
- Gielen, E., et al. Diffusion of sphingomyelin and myelin oligodendrocyte glycoprotein in the membrane of OLN-93 oligodendroglial cells studied by fluorescence correlation spectroscopy. C. R. Biol. 328 (12), 1057-1064 (2005).
- Weiss, M., Hashimoto, H., Nilsson, T. Anomalous protein diffusion in living cells as seen by fluorescence correlation spectroscopy. Biophys. J. 84, 4043-4052 (2003).
- Wawrezinieck, L., Rigneault, H., Marguet, D., Lenne, P. F. Fluorescence correlation spectroscopy diffusion laws to probe the submicron cell membrane organization. Biophys. J. 89 (6), 4029-4042 (2005).
- Lenne, P. F., et al. Dynamic molecular confinement in the plasma membrane by microdomains and the cytoskeleton meshwork. EMBO J. 25 (14), 3245-3256 (2006).
- Ries, J., Schwille, P. Studying slow membrane dynamics with continuous wave scanning fluorescence correlation spectroscopy. Biophys. J. 91 (5), 1915-1924 (2006).
- Ruan, Q., Cheng, M. A., Levi, M., Gratton, E., Mantulin, W. W. Spatial-temporal studies of membrane dynamics: scanning fluorescence correlation spectroscopy (SFCS). Biophys. J. 87 (2), 1260-1267 (2004).
- Berland, K. M., So, P. T., Chen, Y., Mantulin, W. W., Gratton, E. Scanning two-photon fluctuation correlation spectroscopy: particle counting measurements for detection of molecular aggregation. Biophys. J. 71, 410-420 (1996).
- Heinemann, F., Betaneli, V., Thomas, F. A., Schwille, P. Quantifying lipid diffusion by fluorescence correlation spectroscopy: a critical treatise. Langmuir. 28 (37), 13395-13404 (2012).
- Cardarelli, F., Lanzano, L., Gratton, E. Capturing directed molecular motion in the nuclear pore complex of live cells. Proc. Natl. Acad. Sci. U. S. A. 109 (25), 9863-9868 (2012).
- Sanchez, S. A., Tricerri, M. A., Gratton, E. Laurdan generalized polarization fluctuations measures membrane packing micro-heterogeneity in vivo. Proc. Natl. Acad. Sci. U. S. A. 109 (19), 7314-7319 (2012).
- Cardarelli, F., Lanzano, L., Gratton, E. Fluorescence correlation spectroscopy of intact nuclear pore complexes. Biophys. J. 101 (4), 27-29 (2012).
- Di Rienzo, C., et al. Unveiling LOX-1 receptor interplay with nanotopography: mechanotransduction and atherosclerosis onset. Sci. Rep. 3, 10-1038 (2013).
- Unruh, J. R., Gratton, E. Analysis of molecular concentration and brightness from fluorescence fluctuation data with an electron multiplied CCD camera. Biophys. J. 95 (11), 5385-5398 (2008).
- Kannan, B., et al. Electron multiplying charge-coupled device camera based fluorescence correlation spectroscopy. Anal. Chem. 78 (10), 3444-3451 (2006).
- Jones, S. A., Shim, S. H., He, J., Fast Zhuang, X. three-dimensional super-resolution imaging of live cells. Nat. Methods. 8 (6), 499-508 (2011).
- Rust, M. J., Bates, M., Zhuang, X. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy. 3 (10), 793-795 (2006).
- Betzig, E., et al. Imaging intracellular fluorescent proteins at nanometer resolution. Science. 313 (5793), 1642-1645 (2006).
- Hess, S. T., Girirajan, T. P., Mason, M. D. Ultra-high resolution imaging by fluorescence photoactivation localization microscopy. Biophys. J. 91 (11), 4258-4272 (2006).
- Manley, S., et al. High-density mapping of single-molecule trajectories with photoactivated localization microscopy. Nat. Methods. 5 (2), 155-157 (2008).
- Hell, S. W. Far-field optical nanoscopy. Science. 316 (5828), 1153-1158 (2007).
- Klar, T. A., Hell, S. W. Subdiffraction resolution in far-field fluorescence microscopy. Opt. Lett. 24 (14), 954-956 (1999).
- Eggeling, C., et al. Direct observation of the nanoscale dynamics of membrane lipids in a living cell. Nature. 457 (7233), 1159-1162 (2009).
- Hedde, P. N., et al. Stimulated emission depletion-based raster image correlation spectroscopy reveals biomolecular dynamics in live cells. Nat. Commun. 4, .
- Hebert, B., Costantino, S., Wiseman, P. W. Spatiotemporal image correlation spectroscopy (STICS) theory, verification, and application to protein velocity mapping in living CHO cells. Biophys. J. 88 (5), 3601-3614 (2005).
- Brown, C. M., et al. Probing the integrin-actin linkage using high-resolution protein velocity mapping. J. Cell Sci. 119, 5204-5214 (2006).
- Di Rienzo, C., Gratton, E., Beltram, F., Cardarelli, F. Fast spatiotemporal correlation spectroscopy to determine protein lateral diffusion laws in live cell membranes. Proc. Natl. Acad. Sci. U. S. A. 110 (30), 12307-12312 (2013).
- Mueller, V., et al. STED nanoscopy reveals molecular details of cholesterol- and cytoskeleton-modulated lipid interactions in living cells. Biophys. J. 101 (7), 1651-1660 (2011).
- Kleusch, C., Hersch, N., Hoffmann, B., Merkel, R., Csiszar, A. Fluorescent lipids: functional parts of fusogenic liposomes and tools for cell membrane labeling and visualization. Molecules. 17 (1), 1055-1073 (2012).
- Ries, J., Chiantia, S., Schwille, P. Accurate determination of membrane dynamics with line-scan FCS. Biophys. J. 96 (5), 1999-2008 (2009).
- Kolin, D. L., Wiseman, P. W. Advances in image correlation spectroscopy: measuring number densities, aggregation states, and dynamics of fluorescently labeled macromolecules in cells. Cell Biochem. Biophys. 49 (3), 141-164 (2007).
- Digman, M. A., et al. Measuring fast dynamics in solutions and cells with a laser scanning microscope. Biophys. J. 89 (2), 1317-1327 (2005).
- Ritchie, K., et al. Detection of non-Brownian diffusion in the cell membrane in single molecule tracking. Biophys. J. 88 (3), 2266-2277 (2005).
- Voie, A. H., Burns, D. H., Spelman, F. A. Orthogonal-plane fluorescence optical sectioning: three-dimensional imaging of macroscopic biological specimens. J. Microsc. 170, 229-236 (1993).
- Huisken, J., Swoger, J., Del Bene, ., Wittbrodt, F., J, E. H., Stelzer, Optical sectioning deep inside live embryos by selective plane illumination microscopy. Science. 305 (5686), 1007-1009 (2004).
- Wohland, T., Shi, X., Sankaran, J., Stelzer, E. H. Single plane illumination fluorescence correlation spectroscopy (SPIM-FCS) probes inhomogeneous three-dimensional environments. Opt. Express. 18 (10), 10627-10641 (2010).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados