É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
De fast imagens de fluorescência com a Lei difusão molecular em membranas de células vivas em um microscópio comercial
Neste Artigo
Resumo
Spatial distribution and temporal dynamics of plasma membrane proteins and lipids is a hot topic in biology. Here this issue is addressed by a spatio-temporal image fluctuation analysis that provides conceptually the same physical quantities of single particle tracking, but it uses small molecular labels and standard microscopy setups.
Resumo
Tornou-se cada vez mais evidente que a distribuição espacial eo movimento de componentes da membrana, como lipídios e proteínas são fatores-chave na regulação de muitas funções celulares. No entanto, devido à dinâmica rápida e as pequenas estruturas envolvidas, uma resolução espaço-temporal muito elevada é necessário para captar o comportamento real do moléculas. Aqui apresentamos o protocolo experimental para o estudo da dinâmica de proteínas e lipídios plasmáticos à membrana marcados com fluorescência em células vivas, com alta resolução espaço-temporal. Notavelmente, esta abordagem não é necessário para controlar cada molécula, mas calcula o comportamento população usando todas as moléculas numa dada região da membrana. O ponto de partida é uma imagem rápida de uma determinada região na membrana. Em seguida, uma função completa autocorrelação espaço-temporal é calculada correlacionando imagens adquiridas em aumentar os atrasos, por exemplo, a cada 2, 3, n repetições. É possível demonstrar que a largurado pico dos aumentos espaciais da função de autocorrelação em aumentar o tempo de atraso em função do movimento de partículas devido à difusão. Portanto, ajuste da série de funções de autocorrelação permite extrair a proteína real deslocamento médio quadrado de imagem (iMSD), aqui apresentada na forma de aparente diffusivity vs deslocamento médio. Isso gera uma visão quantitativa da dinâmica média de moléculas individuais com precisão nanométrica. Ao utilizar uma variante de GFP marcada com o Receptor de Transferrina (TfR) e uma ATTO488 identificada como 1-palmitoil-2-hidroxi-sn-glicero-3-fosfoetanolamina (PPE), é possível observar a regulação espaço-temporal de proteína e lípido de difusão em regiões de membrana de tamanho micrometros de tempo na gama de micro-a-mili-segundo.
Introdução
A partir do "mosaico fluido" modelo original por Singer e Nicolson, a imagem da membrana plasmática celular tem sido continuamente atualizado durante as últimas décadas, a fim de incluir o papel emergente do citoesqueleto e lipídios domínios 1,2.
As primeiras observações foram obtidos por recuperação de fluorescência após a fotodegradação (FRAP) revelação de que uma fração significativa de proteínas de membrana é imóvel 3-5. Estes estudos pioneiros, embora muito informativo, sofreu com a relativamente baixa resolução no espaço (microns) e de tempo (segundos) de setups FRAP. Além disso, sendo uma medição de média conjunto, FRAP falta em dar informações sobre o comportamento única molécula.
Neste contexto, a possibilidade de marcar especificamente uma única molécula com marcações muito brilhantes (permitindo o estudo do processo de difusão de uma molécula de cada vez), tem sido muito bem sucedida. Particularmente, empurrando otempo de resolução da abordagem única partícula Tracking (SPT) para a escala de tempo microssegundos, Kusumi, et al. acesso ganhou a recursos desconhecidos de lipídeos e proteínas dinâmica que muito contribuíram para o reconhecimento do papel do esqueleto da membrana à base de actina na fisiologia da membrana 6 , 7. Essas descobertas geraram a chamada do "piquete e cerca 'modelo, em que lipídios e proteínas difusão é regulada por esqueleto à base de actina. No entanto, a fim de ter acesso à enorme quantidade de informações fornecidas pelo SPT muitas questões experimentais têm de ser abordadas. Particularmente, o processo de marcação é tipicamente composta por várias etapas, como produção, purificação e introdução das espécies marcadas no sistema. Além disso, grandes gravadoras, como pontos quânticos ou nanopartículas de metal, muitas vezes são necessárias para alcançar a escala de tempo sub-milissegundo ea reticulação das moléculas alvo pelo rótulo não pode ser evitado em muitos casos. Finalmente, muitas trajetóriaster de ser de forma a caber critérios estatísticos e concomitantemente uma baixa densidade da etiqueta é necessária para permitir o rastreamento.
Comparado a SPT, espectroscopia de correlação de fluorescência (FCS), superando muitos destes inconvenientes, representa uma abordagem muito promissora para o estudo de dinâmica molecular. Na verdade, FCS funciona bem também com rótulos escuros e densos, permitindo estudar a dinâmica das moléculas marcadas em proteínas fluorescentes em células transfectadas transitoriamente. Além disso, permite alcançar altas estatísticas em uma quantidade limitada de tempo. Por fim, apesar da "alta" densidade de rótulos FCS fornece moléculas únicas informações. Graças a todas essas propriedades, FCS representa uma abordagem muito simples e tem sido amplamente utilizada no estudo da dinâmica de lipídeos e proteínas, tanto em membranas modelo e em células vivas 8-10. Muitas abordagens diferentes têm sido propostos para aumentar a capacidade de FCS para revelar os detalhes de difusão molecular. Por exemplo, foi shprópria, que através da realização FCS em áreas de observação de diferentes tamanhos pode-se definir uma "lei de difusão FCS" recursos escondidos esclarecedoras do movimento molecular 11,12. Além de ser variaram em tamanho, a área focal também foi duplicada 13, mudou-se no espaço ao longo das linhas 14-20 ou conjugadas com câmeras rápidas 21,22. Usando estes correlação 'espaço-temporal' abordagens, os parâmetros biológicos relevantes para vários componentes da membrana foram quantitativamente descrito em ambas as membranas modelo e as biológicas reais, portanto, uma visão rendimento em membrana de organização espacial.
No entanto, em todas as aplicações e FRAP FCS descrito até agora, a dimensão da área focal representa um limite de resolução espacial, em que não podem ser ultrapassados. Vários métodos de imagem de super-resolução foram recentemente desenvolvidos para contornar este limite. Alguns são baseados em precisão de localização, tais como microscopia óptica reconstrução estocástica (STORM) 23,24, microscopia fotoativação localização (PALM) 25, PALM fluorescência (FPALM) 26 e de uma única partícula de rastreamento PALM (sptPALM) 27: a quantidade relativamente grande de fótons necessários em cada snapshot, no entanto, limita o tempo de resolução de estes métodos para, pelo menos, vários milissegundos, prejudicando assim a sua aplicabilidade in vivo.
Em contraste, uma alternativa promissora para a resolução de imagem de super ter sido aberto por espacialmente modulando a emissão de fluorescência com os métodos de depleção de emissão estimulada (STED ou reversíveis transições saturáveis óptica de fluorescência (RESOLFT)) 28,29. Estas abordagens combinar a formação de um volume de observação bem abaixo do limite de difracção, com a possibilidade de utilização de microscópios varrimento rápido e sistemas de detecção. Em combinação com a análise de variação de fluorescência, microscopia STED permissão para investigar diretamente a dinâmica espaço-temporais em nanoescala de lipídeos e proteins em membranas de células vivas 30,31.
As mesmas quantidades físicas de microscopia-STED base pode ser obtida por uma espectroscopia modificado espaço-temporal correlação imagem (STICS 32,33) método que é adequado para o estudo da dinâmica das proteínas marcadas com fluorescência de membrana e / ou lípidos em células vivas e por um microscópio comercial. O protocolo experimental aqui apresentada é composta por poucos passos. O primeiro exige uma imagem rápida da região de interesse na membrana. Em seguida, a pilha resultante de imagens é usada para calcular as funções de correlação espaço-temporais médios. Ajustando-se a série de funções de correlação, a molecular "lei difusão" pode ser obtida directamente a partir de imagens na forma de uma difusividade aparente (D app) - vs -Média plotagem de deslocamento. Este lote depende criticamente sobre o meio ambiente explorado pelas moléculas e permite reconhecer diretamente os modos de difusão reaisdo lípido / proteína de interesse.
Em mais detalhes, como demonstrado anteriormente 34, a função de auto-correlação espaço-temporal da série imagem adquirida de forma crítica depende da dinâmica das moléculas em movimento na série de imagens coletadas (por favor, note que o mesmo raciocínio pode ser aplicado em uma aquisição de linha onde apenas uma dimensão do espaço é considerado). Em particular, podemos definir a função de correlação como:
 (1)
(1)
onde  representa a intensidade da fluorescência medida, na posição x, y e no momento t,
representa a intensidade da fluorescência medida, na posição x, y e no momento t, ![]() e
e ![]() representa a distância em x edireções y respectivamente,
representa a distância em x edireções y respectivamente, ![]() representa o intervalo de tempo, e
representa o intervalo de tempo, e ![]() representa a média. Esta função pode ser expressa como:
representa a média. Esta função pode ser expressa como:
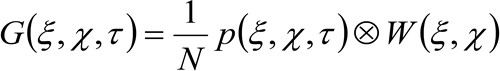 (2)
(2)
onde "N" representa o número médio de moléculas na área de observação, ![]() representa a operação de convolução no espaço, e
representa a operação de convolução no espaço, e 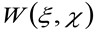 representa a autocorrelação da cintura instrumental. Este último pode ser interpretada como uma medida de como os fotões de um único emissor estão espalhados no espaço, devido à configuração óptica / gravação (o chamado ponto de propagação função, PSF, generali bem aproximada por uma função de Gauss). Finalmente,
representa a autocorrelação da cintura instrumental. Este último pode ser interpretada como uma medida de como os fotões de um único emissor estão espalhados no espaço, devido à configuração óptica / gravação (o chamado ponto de propagação função, PSF, generali bem aproximada por uma função de Gauss). Finalmente, 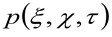 representa a probabilidade de encontrar uma partícula a uma distância
representa a probabilidade de encontrar uma partícula a uma distância ![]() e
e ![]() depois de um tempo de atraso
depois de um tempo de atraso ![]() . Se considerarmos uma dinâmica difusos, em que as partículas se movem aleatoriamente em todas as direções e fluxos líquidos não estão presentes, esta função também é bem aproximada por uma função de Gauss, onde a variância pode ser identificado como o Mean Square Deslocamento (MSD) da partícula em movimento . Assim, a cintura da função de correlação (também referida como
. Se considerarmos uma dinâmica difusos, em que as partículas se movem aleatoriamente em todas as direções e fluxos líquidos não estão presentes, esta função também é bem aproximada por uma função de Gauss, onde a variância pode ser identificado como o Mean Square Deslocamento (MSD) da partícula em movimento . Assim, a cintura da função de correlação (também referida como ![]() ), Pode ser definido como a soma das perturbações músculo-esqueléticas e de partículas da cintura instrumental e pode ser medido por um ajuste de Gaussção da função de correlação, para cada intervalo de tempo. A medida MSD i pode ser utilizada para calcular uma difusividade aparente das moléculas que se deslocam
), Pode ser definido como a soma das perturbações músculo-esqueléticas e de partículas da cintura instrumental e pode ser medido por um ajuste de Gaussção da função de correlação, para cada intervalo de tempo. A medida MSD i pode ser utilizada para calcular uma difusividade aparente das moléculas que se deslocam ![]() e uma média de deslocamento
e uma média de deslocamento ![]() como:
como:
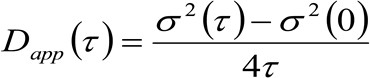 (3)
(3)
 (4)
(4)
Algumas considerações sobre a configuração experimental utilizada pode guiar o leitor ao longo das próximas seções. A fim de estimular selectivamente os fluoróforos na membrana basal de células que irão utilizar uma iluminação de reflexão interna total (TIR), vivo, usando uma fluorescência TIR comercial (TIRF) microscópio (detalhes podem ser encontrados na secção de material). Além disso, a fim de recolher the fluorescência usaremos um objectivo ampliação elevada (100X NA 1,47, abertura numérica elevada é necessária para a iluminação TIRF) e uma câmara EMCCD (tamanho físico do pixel no chip 16 mm). Para chegar a um tamanho de pixel de 100 nm, aplicamos uma lente de ampliação adicional de 1.6X. Como discutido abaixo, uma resolução temporal inferior a 1 ms seria necessário para descrever adequadamente a dinâmica dos lípidos da membrana rápidos abaixo de 100 nm. Para chegar a esta resolução temporal, precisamos selecionar uma região de interesse (ROI) menor do que todo o chip da câmera (512 x 512). Desta forma, a câmara irá ler um número reduzido de linhas de aumentar a resolução de tempo. No entanto, neste regime de tempo de leitura do quadro seria limitada pelo tempo necessário para mudar as taxas de exposição ao chip de leitura na câmara e é geralmente da ordem de milissegundos para 512 x 512 pixels EMCCD. Para vencer esse limite, uma tecnologia emergente permite deslocar as linhas de ROI só que ao invés de todo o quadro, wom uma redução efetiva prática do tamanho do chip exposto (chamado Modo Sensor recortada em nosso EMCCD). Para esta configuração para ser eficaz, o chip de fora do ROI deve ser coberta por um par de fendas montadas no caminho óptico. Graças a esta configuração de uma resolução de tempo até 10 segundos -4 pode ser alcançado. Observe, porém, que esta abordagem pode ser acoplado com muitas montagens experimentais diferentes, como explicado na seção "discussão".
Demonstração do método irá ser fornecida em células vivas, utilizando tanto uma ATTO488 identificada como 1-palmitoil-2-hidroxi-sn-glicero-3-fosfoetanolamina (ATTO488-PPE) e uma variante marcada com GFP do Receptor de Transferrina (GFP TFT). No caso de ATTO488-PPE esta abordagem pode recuperar com sucesso um aplicativo D quase constante como uma função da média de deslocamento que indica uma difusão maior parte livre, como relatado anteriormente 30,35. Por contraste, o TfR-GFP demonstra uma diminuição D app em função do deslocamento da média, sugerindo difusão parcialmente confinado 6. Além disso, neste último caso, é possível quantificar a constante de difusão local, e a média da área ao longo de muitos micra confinamento no plano da membrana.
Protocolo
Calibração do sistema 1.
- Espalhe Ponto de Função (PSF) de calibração
- Dilui-se 10 ul de solução de 30 nm talão fluorescente (cerca de 5 mM) em 90 mL de água destilada e, em seguida, sonicar a solução durante 20 min. Corte um quadrado (1 cm x 1 cm) de gel de agarose a peça (3%) e de depósito de 10 ul da solução sobre a superfície do gel. Derrubar o pedaço de gel no vidro de fundo de uma placa de Petri de dois centímetros e espremer a gota sobre o vidro.
- Ligue a configuração de aquisição, colocar a amostra no suporte, definir a exposição da câmera e EMgain (100 ms e 1000 são bons parâmetros, mas otimizar acordo com o sistema) e esperar que a câmara arrefecer.
- Defina a exposição da câmera para 100 ms, câmera EMgain para 1000, o modo de aquisição para o Quadro de Transferência, 100 repetição e auto definição de poupança.
- Usando a ocular e transmitida foco de luz, na fronteira com o gel e depois passar o objetivo para o centro do gel, ajustar o foco e start o procedimento de alinhamento a laser (em LAS AF, selecione 'setup TIRF "e siga o procedimento de alinhamento automático).
- Encontre um campo de visão com pontos isolados simples, precisa se concentrar no local mais brilhante (que geralmente representa contas agregado) como referência, adquirir 100 quadros e repita o passo 5-6 vezes, a fim de adquirir vários pontos individuais.
- Importe a série adquiriu a um programa de processamento de dados e, em média, a pilha no tempo (Figura 1A) e selecionar um único grânulo isolado. Tome o cuidado de selecionar os mais pequenos para evitar agregados de partículas.
- Coloque a distribuição selecionada intensidade (um exemplo único perfil contas é apresentado na Figura 1B) com uma função de Gauss usando o comando "gaussfit" (nas ferramentas ICS-Matlab nos Materiais em Matlab). Verificar a qualidade do ajuste, inspecionando os resíduos obtidos (um exemplo de perfil Gaussian equipada com os resíduos correspondentes é apresentado in Figura 1B).
- Calibração da câmara
- Ligue a câmera e espere que a câmara arrefecer. Defina a configuração de aquisição da câmera (ou seja, para a câmera usada montamos a exposição a 0,5 ms, câmera EMgain de 1000, o modo de aquisição de modo recortada, o tamanho do ROI de 32 x 128, 10.000 repetições) e começar a aquisição do fundo da câmara sinal.
- Importação adquirido série de quadros para um programa de processamento de dados. Calcule e verifique a intensidade média em cada pixel, a fim de verificar se o fundo da câmara é de aproximadamente plana na região selecionada do chip. No modo recortado, remover a primeira e as últimas linhas horizontais (3 a 10, dependendo do tamanho do ROI) para cada quadro, pois o fundo da câmara é normalmente enviesados nas linhas de fronteira.
- Criar um histograma dos valores (também definido Nível Digital, DL) em imagens adquiridas pilha (usando o comando 'hist' em Matlab) e traçar o logaritmoresultando de frequência (usando o comando semilogy em Matlab). Um exemplo de distribuição DL para o fundo da câmara é apresentado na Figura 2.
NOTA: Se a câmara está a funcionar bem, a trama vai mostrar um pico de cerca de Gauss (um perfil parabólico no diário de escala) representa a distribuição dos valores associados a zero fóton seguido de um decaimento exponencial (uma linha com inclinação negativa no log escala ) que representa a distribuição de valores associados a um fotão (Figura 2). Em particular, o centro e a variância da função de Gauss representam o deslocamento da câmara e erro, respectivamente, enquanto que a constante de decaimento exponencial da parte representa uma estimativa do DL atribuído pela câmara para cada um único fotão. Em Matlab use a seção "CalibrateCamera" do Script em materiais de apoio. - Repita a operação para toda a câmera selecionada EMGain e Gain.
2. RotuladoPreparação celular
- Para preparar os lipossomas necessários para a incorporação de lípido 36, dissolver separadamente 1 mg de DOPE (1,2-dioleoyl- sn-glicero-3-fosfoetanolamino), 1 mg de DOTAP (1,2-dioleoil-3-trimetilamónio-propano), e 1 mg de EPI-ATTO488 em 1 ml de clorofórmio. Misture 0,5 ml de solução de DOPE, 0,5 ml de solução de DOTAP, e 25 ul de solução de EPI-ATTO488 e seco sob vácuo durante 24 horas. Adicionar 0,5 ml de tampão HEPES 20 mM, vortex durante 15 min e sonicado durante 15 minutos a 40 ° C.
- Para preparar a célula, lavar 3 vezes com PBS, um prato p100 confluente de células CHO-K1 (ovário de hamster chinês), adicionar 1 ml de tripsina e armazenar na incubadora durante 5 min. Suspender as células isoladas adição de 9 ml de meio DMEM / F12 suplementado com 10% de FBS e semear 150 mL de solução de células em uma placa de Petri contendo 800 ul do mesmo meio.
- Loja na incubadora durante 24 horas a 37 ° C e 5% de CO 2. Para a incorporação de lípidos, substitua com meio de células 500ul de meio isento de soro; depois de 30 min, adicionar 2 ml de solução de lipossomas; após 15 minutos de lavagem com PSB e adicionar novo meio DMEM / F12 para geração de imagens.
- Para a transfecção, as células transfectar acordo com o protocolo Lipofectamina (as instruções do fabricante), utilizando TfR-GFP plasmídeo e loja 24 horas na incubadora antes de imagem.
3 Aquisição de Dados
- Preparação Setup
- A fim de o termostato microscópio, 24 h antes do experimento de ligar a incubadora.
- A fim de aplicar o tempo de aquisição mais rápido possível, trabalhar em modo Sensor recortada (ver introdução) e usar uma primeira câmera para imagens (câmera 1) e segunda câmera para selecionar a célula (câmera 2). Um esquema da configuração de instalação é apresentado na Figura Suplementar S1. Então, para alinhar a vez duas câmeras no microscópio e esperar que as câmeras para esfriar.
- Situado em ambas as câmaras os parâmetros para transmissão de imagens de luz (ou seja, , 20 ms para o tempo de exposição, 1 para EM Gain) e colocar o microscópio no modo brilhante Field.
- Coloque as amostras no suporte e focar através da ocular, envie a luz para a câmera 1 e empurre as fendas permitindo que a luz apenas no ROI usado para imagens de células (aqui um ROI de 32 x 32 pixels).
- Mover uma célula na região seleccionada e enviar a luz para a câmara 2, então desenhar um ROI no software que controla câmara 2, a fim de ter uma referência.
- Imagem (Figura 3A)
- Primeiro de tudo, alinhe o laser TIRF de acordo com o procedimento de sua configuração. Em nossa configuração, selecione o 'setup TIRF' e iniciar o procedimento de alinhamento automático. Quando o laser é alinhado configurado 70 nm de profundidade de penetração (cerca de 70 °).
- Definir o tempo de exposição a 70 ms e EMGain a 100 em ambos câmera 1 e câmera de 2; em seguida, selecione uma célula usando a câmera 1, em seguida, enviar a luz na câmera 2 e focar com precisão a membrana celular. Defina a exposição mínima em caMera 2, 1000 EMGain, Modo Sensor Curta, 10 5 repetições e definir autosave como arquivos FITS (Flexible Image Transport System, um formato que pode ser facilmente gerenciado).
- Comece a aquisição de gravar a série imagem. Solte o ganho eo Modo cortada para permitir a estabilização da temperatura antes de adquirir um novo celular, em seguida, repita as duas últimas etapas, a fim de adquirir 8-10 células.
4 Cálculo do Mean Square Deslocamento de Imagem (i MSD)
NOTA: O protocolo a seguir podem ser aplicados diretamente aos dados brutos. Ao mesmo tempo, todo o protocolo é válido para aquisição de dados simulados tanto em Matlab e em SimFCS. O link para os tutoriais correspondentes podem ser encontradas na seção "Materiais".
- Cálculo pelo Matlab
- Importe a série adquiriu ao Matlab utilizando roteiro ImportImageSeries. Calcula-se a média de intensidade de cada imagem em tempo utilizando o command dizer sobre as duas primeiras dimensões e usar complô para ver o vetor resultante.
- Se mais do que 10% de foto-branqueamento se encontra presente, a série descartar ou remover a primeira parte deles. Se for inferior, tentar corrigir o efeito sobre a função de correlação, subtraindo a cada imagem sua intensidade média, como mostrado antes de 37.
- Calcule a intensidade média de cada pixel, utilizando média na terceira dimensão e ver imagem resultante.
É necessária uma atenção especial para evitar correlações de artefatos: NOTA. De fato, como demonstrado anteriormente por técnicas semelhantes 38, os contornos das células, bem como de vesículas foco poderia introduzir uma correlação forte. Se a inspecção da imagem média revela os contornos das células ou de vesículas de foco, tente excluir a região envolvida caso contrário descartar a aquisição. Para corrigir o efeito destas estruturas imóveis subtrair a intensidade média temporal de cada pixel 39. - Calcule tele correlação espaço-temporal (G (ξ, χ, τ)) usando a função CalculateSTICScorrfunc. Remover G (ξ, χ, 0), porque a correlação devido ao ruído tiro no regime de pouca luz domina G (0,0,0); a correlação devido ao detector domina o L (± 1,0,0), e o movimento das partículas durante o tempo de exposição pode deformar-se L (ξ, χ, τ) para τ = 0 através do aumento da cintura medida (este efeito desaparece para τ > 0) 34.
- Média G (ξ, χ, τ> 0), utilizando um time-bin logarítmica para reduzir o ruído usando a função "LogBinStack" no material de apoio e, em seguida, encaixar o G resultante (ξ, χ, τ), utilizando a função "gaussfit" de as ferramentas ICS-Matlab em Materiais para recuperar o i MSD (a segunda coluna da matriz resultante).
- Traça-se a σ obtido cintura (τ) 2 (i RIA) como uma função do tempo. Se os dados são muito barulhento, tentar aumentar o número de acququadros IRED, aumentar a potência do laser, a média mais G (ξ, χ, τ) juntos.
- Cálculo por SimFCS
- Abra os arquivos adquiridos com ImageJ usando BioFormat importador plugin e salvar séries adquiridas como Tiff sequência.
- Abrir ferramenta SimFCS e selecionar RICS e selecione Arquivo> Importar várias imagens (Figura S2 Suplementar).
- Selecione Fit, inserir os parâmetros de aquisição corretos e fechar a janela fit (Figura S3 Suplementar).
- Selecione Tela> Intensidade Média> CH1 e verificar a presença de fotodegradação (Figura S4 Suplementar).
- Se mais do que 10% de fotobranqueamento está presente descarte da série ou, se é possível a carga de novo a sequência de imagens de remover a primeira parte da série.
- Se o clareamento é inferior a 10% selecione Ferramentas> i MSD> Definir parâmetros, marque "Use média móvel ', situado no painel de ROI na esquerda umúmero de moldura para o pagamento de média móvel atenção que o tempo correspondente for maior do que o tempo de difusão característica (para partículas movendo-se a 1 mM 2 seg -1 um tempo de 10 segundos é um bom média móvel)
- Selecione Ferramentas> iMSD> Calcule i MSD (Figura Suplementar S5) e apto e exportar o i MSD a partir do bloco de notas (Figura Suplementar S6).
5 Cálculo da Lei de Difusão do i MSD
- Coloque os primeiros pontos para extrapolar a interceptação (σ 0 2) (5 pontos são geralmente suficiente, mas mais pontos podem ser montados se mostram um comportamento linear) e comparar esse valor com o PSF medido anteriormente 2. Se eles são comparáveis, a dinâmica de fluoróforos isolados estão sendo seguidas. Por outro lado, se σ 0 2 2 >> PSF tentar adquirir mais rápido para garantir quedinâmica não ocultas estão presentes 34.
- Calcule a difusividade aparente (app D) eo deslocamento médio (R), utilizando equações 3 e 4 (ver Introdução).
- App Lote D como uma função de R para se obter uma lei de difusão comparável com o que é medido com variação local baseada FCS 12 (Figura 3D).
Resultados
A fim de calibrar o instrumento de cintura, a imagem de um único nano-grânulo fluorescente pode ser medida como descrito no protocolo passo 1.1. A imagem de fluorescência típica destes grânulos é apresentada na Figura 1. O encaixe de distribuição de intensidade por uma função de Gauss 2D devolve boas resíduos e permite a medição da cintura instrumental a 270 nm. Este valor está em boa concordância com o limite de difracção esperado estimada pela equação de Rayleigh. Esta calibração ...
Discussão
Rastreamento de partículas Individual (SPT) representa uma das estratégias mais comuns para o estudo da dinâmica molecular e tem a grande vantagem de medir as trajetórias das partículas. Isto por sua vez permite que o comportamento de sondagem até poucas partículas marcadas em um sistema complexo. No entanto, para chegar a esta vantagem SPT normalmente precisa de uma baixa densidade da sonda e rótulos muito brilhantes. Em particular, para obter alta resolução temporal (gama ms) sonda inorgânico são normalmen...
Divulgações
The authors have nothing to disclose.
Agradecimentos
This work is supported in part by NIH-P41 P41-RRO3155 and NIH P50-GM076516 (grant to EG), and Fondazione Monte dei Paschi di Siena (grant to FB).
Materiais
| Name | Company | Catalog Number | Comments |
| iXon Ultra 897 | Andor | DU-897U-CS0 | |
| Solis | Andor | ||
| CHO-K1 | ATCC | CCL-61 | |
| ATTO 488 labeled PPE | ATTO-TEC GmbH | AD 488-151 | |
| DOPE | Avanti Polar Lipids, Inc. | 850725 | |
| DOTAP | Avanti Polar Lipids, Inc. | 890890 | |
| 100x Penicillin-Streptomycin-Glutamine | Gibco | 10378-016 | |
| DMEM/F-12 | Gibco | 21331 | |
| FBS | Gibco | 10082147 | |
| HEPES | Gibco | 15630-106 | |
| PBS | Gibco | 10010-023 | |
| SimFCS 3.0 | Globals Software | the software can be downloaded here: http://www.lfd.uci.edu/globals/ | |
| DMI6000 with TIRF modulus | Leica | ||
| LAS AF | Leica | ||
| Lipofectamine 2000 | Lipofectamine | 11668019 | |
| Matlab | MathWork | ||
| ImageJ | NIH | ||
| [header] | |||
| C-terminal GFP tagged Tranferrin Receptor | OriGene | RG200980 | |
| Agar | Sigma Aldrich | A5306 | |
| Chloroform | Sigma Aldrich | 528730 | |
| Latex beads, fluorescent yellow-green, 30 nm | Sigma Aldrich | L5155 | |
| SONICA Ultrasonic Cleaners | SOLTEC | ETH S3 | |
| Petri Dishes | Willco | GWSt-3522 | |
| Bio-Format importer for Matlab | http://www.openmicroscopy.org/site/support/bio-formats5/users/matlab/ | ||
| ICS-MatLab Tools | https://www.cellmigration.org/resource/imaging/software/ICSMATLAB_28-02-06.zip | ||
| Simulation by Matlab Tutorial | https://www.cellmigration.org/resource/imaging/icsmatlab/ICSTutorial.html | ||
| Simulation by SimFCS Tutorial | https://www.cellmigration.org/resource/imaging/ppt-pdf/RICS%20Simulations.ppt | ||
Referências
- Engelman, D. M. Membranes are more mosaic than fluid. Nature. 438 (7068), 578-580 (2005).
- Vereb, G., et al. yet structured: The cell membrane three decades after the Singer-Nicolson model. Proc. Natl. Acad. Sci. U. S. A. 100 (14), 8053-8058 (1073).
- Ishihara, A., Hou, Y., Jacobson, K. The Thy-1 antigen exhibits rapid lateral diffusion in the plasma membrane of rodent lymphoid cells and fibroblasts. 84 (5), 1290-1293 (1987).
- Axelrod, D., et al. Lateral motion of fluorescently labeled acetylcholine receptors in membranes of developing muscle fibers. Proc. Natl. Acad. Sci. U. S. A. 73 (12), 4594-4598 (1976).
- Jacobson, K., Derzko, Z., Wu, E. S., Hou, Y., Poste, G. Measurement of the lateral mobility of cell surface components in single, living cells by fluorescence recovery after photobleaching. J. Supramol. Struct. 5 (4), 10-1002 (1976).
- Kusumi, A., et al. Paradigm shift of the plasma membrane concept from the two-dimensional continuum fluid to the partitioned fluid: high-speed single-molecule tracking of membrane molecules. Annu. Rev. Biophys. Biomol. Struct. 34, 351-378 (2005).
- Kusumi, A., Ike, H., Nakada, C., Murase, K., Fujiwara, T. Single-molecule tracking of membrane molecules: plasma membrane compartmentalization and dynamic assembly of raft-philic signaling molecules. Semin. Immunol. 17 (1), 3-21 (2005).
- Schwille, P., Korlach, J., Webb, W. W. Fluorescence correlation spectroscopy with single-molecule sensitivity on cell and model membranes. Cytometry. 36, 176-182 (1999).
- Gielen, E., et al. Diffusion of sphingomyelin and myelin oligodendrocyte glycoprotein in the membrane of OLN-93 oligodendroglial cells studied by fluorescence correlation spectroscopy. C. R. Biol. 328 (12), 1057-1064 (2005).
- Weiss, M., Hashimoto, H., Nilsson, T. Anomalous protein diffusion in living cells as seen by fluorescence correlation spectroscopy. Biophys. J. 84, 4043-4052 (2003).
- Wawrezinieck, L., Rigneault, H., Marguet, D., Lenne, P. F. Fluorescence correlation spectroscopy diffusion laws to probe the submicron cell membrane organization. Biophys. J. 89 (6), 4029-4042 (2005).
- Lenne, P. F., et al. Dynamic molecular confinement in the plasma membrane by microdomains and the cytoskeleton meshwork. EMBO J. 25 (14), 3245-3256 (2006).
- Ries, J., Schwille, P. Studying slow membrane dynamics with continuous wave scanning fluorescence correlation spectroscopy. Biophys. J. 91 (5), 1915-1924 (2006).
- Ruan, Q., Cheng, M. A., Levi, M., Gratton, E., Mantulin, W. W. Spatial-temporal studies of membrane dynamics: scanning fluorescence correlation spectroscopy (SFCS). Biophys. J. 87 (2), 1260-1267 (2004).
- Berland, K. M., So, P. T., Chen, Y., Mantulin, W. W., Gratton, E. Scanning two-photon fluctuation correlation spectroscopy: particle counting measurements for detection of molecular aggregation. Biophys. J. 71, 410-420 (1996).
- Heinemann, F., Betaneli, V., Thomas, F. A., Schwille, P. Quantifying lipid diffusion by fluorescence correlation spectroscopy: a critical treatise. Langmuir. 28 (37), 13395-13404 (2012).
- Cardarelli, F., Lanzano, L., Gratton, E. Capturing directed molecular motion in the nuclear pore complex of live cells. Proc. Natl. Acad. Sci. U. S. A. 109 (25), 9863-9868 (2012).
- Sanchez, S. A., Tricerri, M. A., Gratton, E. Laurdan generalized polarization fluctuations measures membrane packing micro-heterogeneity in vivo. Proc. Natl. Acad. Sci. U. S. A. 109 (19), 7314-7319 (2012).
- Cardarelli, F., Lanzano, L., Gratton, E. Fluorescence correlation spectroscopy of intact nuclear pore complexes. Biophys. J. 101 (4), 27-29 (2012).
- Di Rienzo, C., et al. Unveiling LOX-1 receptor interplay with nanotopography: mechanotransduction and atherosclerosis onset. Sci. Rep. 3, 10-1038 (2013).
- Unruh, J. R., Gratton, E. Analysis of molecular concentration and brightness from fluorescence fluctuation data with an electron multiplied CCD camera. Biophys. J. 95 (11), 5385-5398 (2008).
- Kannan, B., et al. Electron multiplying charge-coupled device camera based fluorescence correlation spectroscopy. Anal. Chem. 78 (10), 3444-3451 (2006).
- Jones, S. A., Shim, S. H., He, J., Fast Zhuang, X. three-dimensional super-resolution imaging of live cells. Nat. Methods. 8 (6), 499-508 (2011).
- Rust, M. J., Bates, M., Zhuang, X. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy. 3 (10), 793-795 (2006).
- Betzig, E., et al. Imaging intracellular fluorescent proteins at nanometer resolution. Science. 313 (5793), 1642-1645 (2006).
- Hess, S. T., Girirajan, T. P., Mason, M. D. Ultra-high resolution imaging by fluorescence photoactivation localization microscopy. Biophys. J. 91 (11), 4258-4272 (2006).
- Manley, S., et al. High-density mapping of single-molecule trajectories with photoactivated localization microscopy. Nat. Methods. 5 (2), 155-157 (2008).
- Hell, S. W. Far-field optical nanoscopy. Science. 316 (5828), 1153-1158 (2007).
- Klar, T. A., Hell, S. W. Subdiffraction resolution in far-field fluorescence microscopy. Opt. Lett. 24 (14), 954-956 (1999).
- Eggeling, C., et al. Direct observation of the nanoscale dynamics of membrane lipids in a living cell. Nature. 457 (7233), 1159-1162 (2009).
- Hedde, P. N., et al. Stimulated emission depletion-based raster image correlation spectroscopy reveals biomolecular dynamics in live cells. Nat. Commun. 4, .
- Hebert, B., Costantino, S., Wiseman, P. W. Spatiotemporal image correlation spectroscopy (STICS) theory, verification, and application to protein velocity mapping in living CHO cells. Biophys. J. 88 (5), 3601-3614 (2005).
- Brown, C. M., et al. Probing the integrin-actin linkage using high-resolution protein velocity mapping. J. Cell Sci. 119, 5204-5214 (2006).
- Di Rienzo, C., Gratton, E., Beltram, F., Cardarelli, F. Fast spatiotemporal correlation spectroscopy to determine protein lateral diffusion laws in live cell membranes. Proc. Natl. Acad. Sci. U. S. A. 110 (30), 12307-12312 (2013).
- Mueller, V., et al. STED nanoscopy reveals molecular details of cholesterol- and cytoskeleton-modulated lipid interactions in living cells. Biophys. J. 101 (7), 1651-1660 (2011).
- Kleusch, C., Hersch, N., Hoffmann, B., Merkel, R., Csiszar, A. Fluorescent lipids: functional parts of fusogenic liposomes and tools for cell membrane labeling and visualization. Molecules. 17 (1), 1055-1073 (2012).
- Ries, J., Chiantia, S., Schwille, P. Accurate determination of membrane dynamics with line-scan FCS. Biophys. J. 96 (5), 1999-2008 (2009).
- Kolin, D. L., Wiseman, P. W. Advances in image correlation spectroscopy: measuring number densities, aggregation states, and dynamics of fluorescently labeled macromolecules in cells. Cell Biochem. Biophys. 49 (3), 141-164 (2007).
- Digman, M. A., et al. Measuring fast dynamics in solutions and cells with a laser scanning microscope. Biophys. J. 89 (2), 1317-1327 (2005).
- Ritchie, K., et al. Detection of non-Brownian diffusion in the cell membrane in single molecule tracking. Biophys. J. 88 (3), 2266-2277 (2005).
- Voie, A. H., Burns, D. H., Spelman, F. A. Orthogonal-plane fluorescence optical sectioning: three-dimensional imaging of macroscopic biological specimens. J. Microsc. 170, 229-236 (1993).
- Huisken, J., Swoger, J., Del Bene, ., Wittbrodt, F., J, E. H., Stelzer, Optical sectioning deep inside live embryos by selective plane illumination microscopy. Science. 305 (5686), 1007-1009 (2004).
- Wohland, T., Shi, X., Sankaran, J., Stelzer, E. H. Single plane illumination fluorescence correlation spectroscopy (SPIM-FCS) probes inhomogeneous three-dimensional environments. Opt. Express. 18 (10), 10627-10641 (2010).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados