Method Article
Visualización de las células Multiciliated en el pez cebra a través de un protocolo combinado de todo Monte fluorescente In Situ hibridación e inmunofluorescencia
En este artículo
Resumen
Desarrollo de cilios es vital para la correcta organogénesis. Este protocolo describe un método optimizado para la etiqueta y visualizar las células ciliadas del pez cebra.
Resumen
En los últimos años, el embrión del pez cebra se ha convertido en un modelo popular para estudiar la biología del desarrollo debido a rasgos como ex útero desarrollo del embrión y la transparencia óptica. En particular, el embrión del pez cebra se ha convertido en un organismo importante para el estudio de la organogénesis de vertebrados del riñón así como desarrollo de las células multiciliated (MCC). Para visualizar el MCC en el riñón de embrión de pez cebra, han desarrollado un protocolo combinado de montaje conjunto fluorescente en situ del hibridación (pescado) y todo Monte inmunofluorescencia (IF) que permite la proyección de imagen de alta resolución. Este manuscrito describe la técnica para localizar Co las transcripciones del RNA y la proteína como una herramienta para entender mejor la regulación de los programas de desarrollo a través de la expresión de diversos factores de linaje.
Introducción
En las últimas décadas, el pez cebra (Danio rerio) ha surgido como un organismo modelo principal para el estudio de la biología del desarrollo. Los embriones desarrollan fuera de la madre y son ópticamente transparentes. Además, la formación de órganos vitales como el ojo, el riñón y el cerebro anterior se produce rápidamente, con estructuras formadas por sólo 24 h post fertilización (hpf). Lo importante, el genoma del pez cebra se conserva altamente con mamíferos1,2,3. Además, pez cebra y órganos mamíferos tienen similar fisiología y anatomía. El riñón embrionario del pez cebra, o Pronefros, demuestra el valor del sistema modelo de examen de funciones de los genes durante nephrogenesis precoz y determinación del destino de las poblaciones de la célula epitelial conservado de la nefrona vertebrados4, 5 , 6 , 7 , 8 , 9 , 10. del mismo modo, el embrión del pez cebra se ha vuelto cada vez más importante en el examen de la ontogenia de MCC11,12,13,14,15, 16 , 17.
Como su nombre lo indica, MCC es las células epiteliales se caracterizan por un haz de cilios móviles ubicados en la superficie apical17. En el pez cebra, MCC funciona en flujo de fluidos y se dispersa en un "canosos" como la moda a lo largo de la mitad de cada Nefrona de los Pronefros 24 hpf11,12,13,14, 15 , 16 , 17. aunque sólo se han observado en un puñado de riñón humano enfermedad casos18,19,20,21, MCC es frecuente en otros tejidos mamíferos tales como el cerebro y tráquea22,23,24, que plantea una serie de retos para el diseño experimental. Estudios elegante en varios modelos vertebrados incluyendo peces cebra han demostrado un camino conservado del destino MCC, con la muesca que se vía de señalización como un inhibidor de la MCC desarrollo12,17,25 ,26,27. Por lo tanto, el Pronefros de pez cebra proporciona un modelo de fácil acceso para estudiar los mecanismos genéticos de MCC desarrollo en vivo11,12,13,14, 15 , 16 , 17.
Transparencia y fácil manipulación genética de embriones de pez cebra han demostrado para ser inestimable rasgos al estudiar las vías genéticas y moleculares que regulan el destino celular, crecimiento y desarrollo del temprano embrión1,2 ,3. Como tales, las técnicas tradicionales a visualizar transcripciones proteína y gen, como hibridación en situ y en caso de montar todo, han sido aplicados a y optimizado para el pez cebra16,28,29, 30,31,32,33,34,35,36,37,38. Mediante la combinación de protocolos populares de pescado y si, es posible etiquetar y analizar MCC en vivo16,28,37,38.
Protocolo
El siguiente protocolo utiliza pez cebra adultos mantenidos y atendidos por el centro para la investigación del pez cebra en la Universidad de Notre Dame. Todos los métodos para trabajar con embriones y pez cebra adultos fueron aprobados por el cuidado institucional de Animal y uso.
1. embrión fijación
- Recolectar embriones de pez cebra con métodos previamente descritos39,40. A las 24 hpf, Añadir 500 μl de solución de mezcla de proteasas no específicas (50 mg/mL) para el E3 en un embrión de plato incubar a temperatura ambiente (RT).
- Una vez que los embriones comienzan rompiendo hacia fuera del corión, quitar todo el líquido del plato embrión.
- Lavar los embriones 2 - 3 veces con E3 fresco, añadir aproximadamente 20 mL de E3, girando suavemente el E3 en el plato y luego decantar el líquido en un recipiente de desechos líquido.
- Para eutanasia a los embriones antes de fijar, añadir 2 mL de metanosulfonato de 0.2% para el E3.
- Transferencia de los embriones en un frasco de vidrio de 5 mL con una pipeta Pasteur de plástico. Eliminar la mayor parte de la solución de Metanosulfonato/E3 utilizando la pipeta después de que los embriones se han asentado en el fondo del frasco.
- Solución 24 embriones de hpf con 5 mL de paraformaldehído al 4% (PFA) para 2-4 h a temperatura ambiente, sin agitación.
PRECAUCIÓN: El PFA es tóxico y soluciones PFA deben manejarse bajo una campana química mientras el investigador está usando protección para los ojos, guantes y una bata de laboratorio. Preparación del fijador de PFA del polvo granulado de PFA debe realizarse con cuidado excepcional, como la PFA granulado tiende a dispersarse a través de la carga estática.
Nota: Los embriones se pueden fijar en 4% PFA durante la noche a 4 ° C. Preparar 4% PFA calentando 1 x de tampón fosfato salino (PBS) a ebullición agitando constantemente la solución para disolver completamente la PFA granulado. Una vez que la solución se ha enfriado a RT, el 4% del PFA es filtro esterilizado pasando a través de un sistema desechable de 0.45 μm para la filtración de vacío, entonces y alícuotas en porciones de 15 mL o 50 mL para el almacenamiento a-20 ° C. - Quitar los 4% PFA y embriones de lavado dos veces con tampón de fosfato X 1 salina con 0,1% Tween-20 (SAFT) a TA. lavar los embriones una vez con 5 mL de metanol (MeOH) de 100% en RT. Añadir 5 mL de dulce 100% MeOH a los embriones e incubar a-20 ° C durante al menos 20 minutos.
Nota: Los embriones se pueden almacenar en un 100% MeOH a-20 ° C hasta que esté listo para la preparación del embrión.
2. hibridación y la preparación de embriones
- Rehidratar los embriones por lavado una vez en 5 mL de 50% MeOH 1 x PBST 5 min a TA. continuar rehidratación por lavar los embriones en 5 mL de 30% MeOH en 1 x PBST por 5 min a TA. Lave los embriones en 5 mL de 1 x PBST dos veces por 5 min a RT.
- Permeabilizar los embriones por incubación en 5 mL de una solución de proteinasa K de 1:1,000 (pK) (10 mg/mL) en 1 x PBST por 2 min a TA.
Nota: Embriones mayores pueden incubarse más de 2 minutos en la concentración de pK arriba indicada, sin embargo una concentración de 5 mg/mL e incubación tiempo de menos de 2 min se sugiere para embriones menores de 24 hpf. - Quitar pK la solución y lavar inmediatamente en 5 mL de 1 x SAFT dos veces en RT. fijar los embriones en 5 mL de 4% PFA a temperatura ambiente durante al menos 20 minutos.
Nota: Los embriones se pueden fijar en 4% PFA durante la noche a 4 ° C. - Quitar PFA y lavado dos veces en 5 mL de 1 x PBST a TA.
- Transferencia de embriones en 5 mL de 1 x PBST en tubos de microcentrífuga de fondo plano posición vertical en un rack de microcentrífuga a temperatura ambiente para facilitar las interacciones entre cada embrión y la solución de hibridación posterior.
- Lavar los embriones dos veces en 1,5 mL de solución de hibridación (Hyb +) RT. Añadir 1.5 ml de Hyb fresco + e incubar embriones a 70 ° C en un horno de hibridación durante 4-6 h.
- Reemplazar los 1,5 mL de Hyb + con un volumen de solución de la sonda (10 μl RNA sonda/500 μl Hyb +) suficiente para cubrir completamente los embriones.
Nota: Síntesis antisentido sonda RNA usando anteriormente descrito métodos39. - Hibridar la sonda durante la noche en el horno de hibridación a 70 ° C con el vertical de tubos individuales colocados en el estante de microcentrífuga, sin agitar.
3. caliente lavados y bloqueo
Nota: Se realizaron lavados caliente poniendo la solución adecuada en los embriones y luego incubar en el horno de hibridación a 70 ° C. Para mantener las soluciones de lavado a 70 º C, coloque tubos de 50 mL de cada solución en el horno de hibridación cuando la sonda / Hyb + mezcla es añadido.
- Extraiga la sonda. Lavar los embriones dos veces a 70 ° C en 1,5 mL de formamida 50% / 2 x buffer citrato salino-sódico (SSC) para 20-30 minutos cada uno.
Nota: La sonda se puede almacenar en Hyb + a-20 ° C y volver a utilizarse en una fecha posterior. - Lavar los embriones una vez a 70 ° C en 1,5 mL de 2 x SSC por 15 minutos lavar los embriones dos veces a 70 ° C en 1,5 mL de 0,2 X SSC de 20-30 minutos cada uno. X SSC reemplazar 0.2 con 1,5 mL de reactivo de bloqueo e incubar durante la noche a 4 ° C.
Nota: Es posible incubar bloqueo reactivo a temperatura ambiente durante 4 horas en lugar de la noche a la mañana.
4. anticuerpo incubación y lavados de ácido Maleic amortiguamiento
Nota: Mantener embriones protegidos de la luz ambiental.
- Reemplazar el bloqueo reactivo con 0,2 mL de anti-DIG-POD en el bloqueo reactivo a razón de 1:1,000 e incubar durante 3 h en papel de aluminio o tapa de bloqueo de luz otra a TA.
Nota: Como alternativa, el anticuerpo puede incubar durante una noche a 4 ° C. - Después de 3 horas, lave los embriones en 1,5 mL de tampón ácido maleico 2 - 4 veces por 10-15 minutos cada uno en RT. incubar los embriones durante la noche a 4 ° C en 1,5 mL de tampón ácido maleico fresco.
Nota: No es necesario incubar durante una noche en ácido maleic amortiguamiento, pero hacer disminuye tan fondo sonda.
5. sonda de detección y eliminación de la peroxidasa
Nota: Mantener embriones protegidos de la luz ambiental.
- Elimine el tampón de ácido maleico y reemplazar con 1,5 mL de PBS 1 x. Lavar los embriones dos veces en 1,5 mL de PBS 1 x por 5 min en RT. Incubar embriones en 0,2 mL de Cy3 fluorescente manchas solución durante 60 min a TA.
Nota: El tiempo de incubación puede variar de diversas puntas de prueba. - Lave los embriones una vez con 1,5 mL de lo siguiente porcentaje MeOH 1 X PBS durante 10 minutos a temperatura ambiente: 30% MeOH, 50% MeOH, 75% MeOH y 100% MeOH.
- Incubar los embriones con 1,5 mL de 1% H2O2 en MeOH durante 30 min a RT. Wash los embriones una vez con 1,5 mL de las siguientes soluciones de MeOH en PBS 1 x durante 10 min a temperatura ambiente: 75% MeOH, 50% MeOH y 30% MeOH. Lavar los embriones dos veces por 5 minutos a temperatura ambiente en 1,5 mL de PBS 1 x.
Nota: Los embriones pueden almacenarse en PBS durante la noche a 4° C.
6. inmunofluorescencia
Nota: Mantener embriones protegidos de la luz ambiental.
- Lave los embriones en 1,5 mL de ddH2O 5 minutos en RT. Wash los embriones en 1,5 mL de acetona enfriada previamente (mantenido a-20 ° C) de 7 min a TA. lavar los embriones una vez en 1,5 mL de ddH2O 5 minutos en RT. Wash los embriones una vez en 1.5 mL de 1 x PBST con DMSO 1% (PBDT) por 5 min a TA.
- Incubar los embriones en 1,5 mL de PBDT + 10% suero bovino fetal (FBS) en un eje de balancín a temperatura ambiente por 2 h.
- Quite PBDT + FBS y reemplácelo con 1,5 mL de anticuerpos primarios, tubulina acetilada anti (producido de ratón) y anti-γ-tubulina (producido de conejo), juntos diluido al 1: 400 en PBDT + 1% FBS. Incubar los embriones durante la noche a 4 ° C.
Nota: Tiempos de incubación pueden variar dependiendo del anticuerpo primario. Pruebas de diferentes intervalos de tiempo pueden ser necesario para optimizar el etiquetado.
7. secundaria anticuerpo
Nota: Mantener embriones protegidos de la luz ambiental.
- Para retirar el anticuerpo no Unido exceso de los embriones, lavar en 1,5 mL de PBDT + 1% FBS + 0,1 M de NaCl durante 1 min a temperatura ambiente en un eje de balancín.
- Lavar los embriones 5 veces en 1,5 mL de PBDT + 1% FBS + 0,1 M de NaCl por 30 min en un eje de balancín en RT. Lave los embriones en 1,5 mL de PBDT + 1% FBS por 30 min a temperatura ambiente en un eje de balancín.
- Incubar los embriones durante la noche a 4 ° C en 200 μL de los anticuerpos secundarios, Alexa 488 cabra anti-ratón IgG y Alexa 647 cabra anti-IgG de conejo, diluido juntos en PBDT al 1: 500.
8. tinción DAPI
Nota: Mantener embriones protegidos de la luz ambiental.
- Lavar los embriones rápidamente a temperatura ambiente en 1,5 mL de PBDT dos veces. Reemplazar PBDT con 1,5 mL de DAPI, diluido 1:15000 en 1 X Saft e Incubar 15 min a temperatura ambiente en un eje de balancín.
- Lave los embriones en 1,5 mL de PDBT + 1% FBS + 0.1 M NaCl tres veces a TA durante 15-20 min cada uno. Almacenar embriones en 1,5 mL de PBDT a 4 ° C hasta que esté listo de montar y de imagen.
9. montaje y proyección de imagen de pez cebra Pronefros
- Endecha del embrión lateral sobre portaobjetos de cristal en un pequeño volumen, aproximadamente 10 μl, de PBDT.
- Apriete el embrión detrás de los ojos con un par de Pinzas finas para quitar la cabeza y algunos de la bola de yema de huevo.
- Utilizar la pinza fina para raspar suavemente la bola de yema de huevo restante del cuerpo del embrión. Quitar la yema disociado y el líquido extra de la diapositiva con un pañuelo fino.
- Añadir una gota de montaje de los medios de comunicación a la cola restante del embrión y coloque de manera que la cola se coloca lateralmente. Coloque un cubreobjetos encima de la cola para aplanar la muestra, luego coloque en una caja de tobogán.
- Cilios renales con 60 aumentos en un microscopio confocal utilizando los siguientes canales de imagen: DAPI en azul (láser ajustado a 408.0 nm), FITC en verde (laser set en 488.0 nm), Cy3 teñir-etiquetados en rojo (laser set en 561.0 nm) y Alexa 680 en blanco (láser conjunto en 637.0 nm).
Resultados
Embriones de pez cebra de tipo salvaje se fijaban en 24 hpf e inmediatamente preparado como se describió anteriormente. La figura 1 muestra un flujo de trabajo experimental junto con las etapas ilustradas. El flujo de trabajo descrito en los pasos 1-8 abarca los procesos de obtención de muestras, fijación y manipulación de los tejidos fijos etiquetar transcritos endógenos con riboprobes antisentido seguido por inmunofluorescencia a etiqueta proteínas de interés. Paso 9 en el flujo de trabajo se refiere a técnicas diseñadas específicamente para optimizar la visualización del tronco embrionario pez cebra de imagen y el montaje. En los dibujos que acompañan el paso 9, ilustramos el método de manipulación para colocar el tejido en un portaobjetos de vidrio. En este paso, el balón de cabeza y yema de embrión se retiran, dejando la cola lateralmente se coloca entre el cubreobjetos y portaobjetos de vidrio. Extracción de la bola de la yema y se sugiere para la mejor proyección de imagen de la población residente de MCC en el Pronefros, como la bola de yema auto fluorescencia y obstruye el proceso de montaje.
Obtenidos mediante un microscopio confocal de imágenes representativas se encuentran en la figura 2, que muestra el mismo embrión de tipo salvaje a 60 aumentos y un zoom digital en el mismo aumento. Los paneles superiores proporcionan imágenes de cada canal individual, mientras que los dos paneles de la parte inferior son el recubrimiento compuesto de estos datos de imágenes. La caja blanca (en la imagen de la columna de la izquierda) describe la zona que es el foco del zoom digital (la imagen de la columna de la derecha). Resaltado en el panel final del zoom digital, los núcleos fueron etiquetados en DAPI, y MCC se detectaron basado en un riboprobe antisentido diseñado para reconocer odf3b, donde γ-tubulina se une a los anticuerpos indican los cuerpos básicos y α-tubulina denota los cilios. En el zoom digital del recubrimiento compuesto, el círculo amarillo discontinuo indica el perímetro de un MCC, que fue identificado por la posesión de certificados de odf3b , múltiples cuerpos basales y cilios. La célula ciliada mono adyacente, marcada con un círculo punteado amarillo, identificó basado en el fenotipo de poseer un solo cuerpo basal y un cilio único.
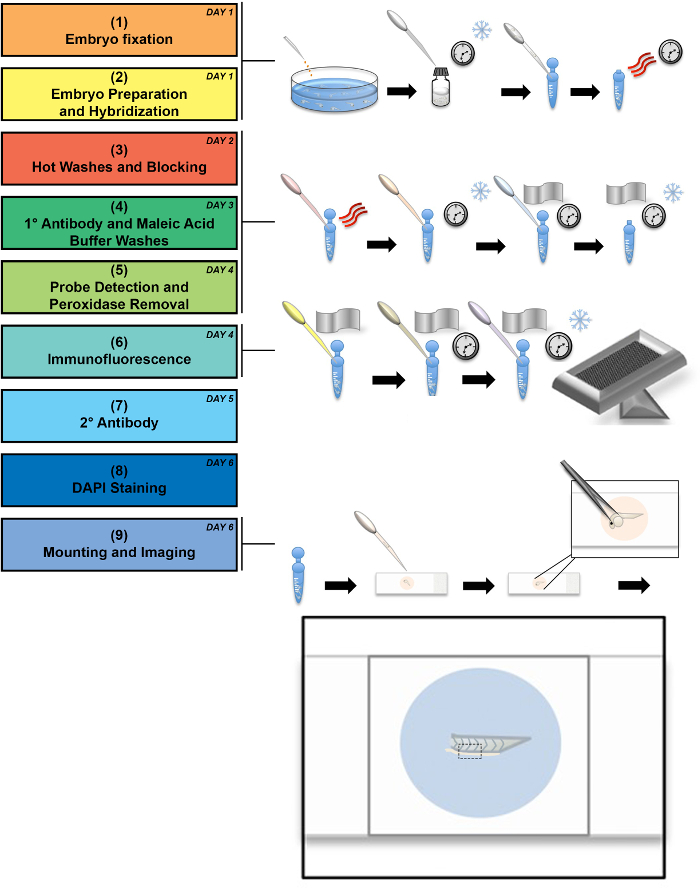
Figura 1: esquema de diagrama de flujo experimental. Este diagrama muestra un flujo de trabajo experimental, acompañado por las ilustraciones de las etapas críticas en que el copo de nieve indica frío incubación, las tres líneas curvas representan el calor y el reloj representa tiempos de incubación prolongados. El flujo de trabajo puede realizarse en un mínimo de 6 días (véase número de día en la esquina superior derecha de cada etapa del proceso), aunque algunos pasos se pueden realizar sobre períodos más largos, como se señala en el protocolo. Una descripción detallada del proceso de montaje se dibuja hacia fuera, mostrando la cola final embrión montado entre el vidrio portaobjetos y cubreobjetos. Una caja negra punteada indica la zona que es reflejada en un aumento de 60 X. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: resultados de representante para la visualización de MCCs del pez cebra Pronefros. Proyecciones de imagen máxima de un embrión de pez cebra tipo hpf 24 a 60 X aumentos y un zoom digital en el confocal con 60 aumentos del embrión mismo. Las cajas blancas indican el área enfocado para el zoom. Manchas individuales de DAPI (núcleos), odf3b (MCC), γ-tubulina (cuerpos basales) y α-tubulina (cilios) son etiquetadas y luego fusionarse en los paneles de la parte inferior dos. En el zoom digital, brindamos aproximaciones de los lugares de la célula como sigue: un MCC está delineado por el círculo amarillo punteado, y el círculo punteado amarillo describe una célula ciliada de mono. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
El protocolo detallado arriba está optimizado para el etiquetado de MCC en el Pronefros con transcripciones de odf3b y α-tubulina en embriones de pez cebra de 24 hpf. Para obtener los mejores resultados se recomienda utilizar embriones recién arreglados y preparados. Embriones que han sido fijados y almacenados en MeOH o Hyb + a-20 ° C por más de una semana se pueden utilizar, pero la probabilidad de fondo no deseados manchas aumenta con el tiempo que los embriones se mantienen en el almacenamiento.
Modificaciones y la resolución de problemas de esta técnica están necesarios adaptar este método para otras suites de marcadores particulares, por ejemplo otros riboprobes antisentido y otros anticuerpos. Muchos métodos para ajustar los parámetros de pasos en el proceso de montaje todo en situ hibridación han sido documentado previamente por nuestro grupo y otros31,34,36,38, 39. Asimismo, cada anticuerpo proteína requerirá algunos problemas en cuanto a tiempos de tinción y rangos aproximados de diluciones y tiempos de incubación para cada anticuerpo primario se estiman mejor mediante la evaluación de resultados documentados28, 35. embriones menores de 24 hpf, tratamiento pK debe ser menor que 2 min, donde embriones mayores de 24 hpf debe tratarse con pK por mas de 2 minutos. También, los embriones mayores de 24 hpf debe ser blanqueada de pigmento antes del tratamiento de pK.
Después de la tinción con la solución de tinción fluorescente, es fundamental para quitar cualquier mancha, anticuerpo y peroxidasas restantes mediante la realización de la serie de lavados de metanol y agua oxigenada. Los lavados de PBS inmediatamente después son vitales para eliminar el metanol sobrante de los embriones. Lo importante, antes de proceder con los anticuerpos IF, hemos encontrado que la acetona y agua desionizada lavados hacen los embriones menos propensos a adherirse a uno con el otro o los tubos de la centrífuga. En nuestra experiencia, anticuerpos para factores de transcripción específicos y otros genes, aunque puede trabajar eficientemente Western blots, no funcionan bien pues si en el pez cebra. Sin embargo, altamente conservadas y abundantes proteínas, como la α-tubulina y β-catenina, funcionan bien en el pez cebra para IF16,37.
Con peces, es posible visualizar las transcripciones del RNA de los genes que aún no tienen anticuerpos específicos en el pez cebra. Mediante la combinación de pescado con IF, según lo demostrado por este protocolo, co-localización de las transcripciones del RNA y la proteína puede ser visto en vivo (figura 2). La naturaleza flexible del protocolo permite la rápida solución de problemas para la visualización de distintos transcritos de RNA y proteínas en numerosos tejidos y puntos del tiempo. Cuando se combina con los rasgos ya respetados de embriones de pez cebra, este protocolo de pescado + si proporciona otra herramienta para explorar la expresión de genes y proteínas importantes a la regulación de la vía del desarrollo.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este trabajo fue apoyado en parte por la beca R01DK100237 R.A.W. y el nacional ciencia Fundación postgrado investigación beca no. DGE-1313583 a A.N.M. También agradecemos a la Universidad de ciencia verano pregrado investigación programa de becas para apoyo a M.U. Nos gustaría agradecer el centro para la investigación de pez cebra en la Universidad de Notre Dame para su cuidado dedicado de nuestro pez cebra. También nos gustaría dar las gracias al Departamento de ciencias biológicas así como los miembros de nuestro laboratorio por todo su apoyo y valiosa información.
Materiales
| Name | Company | Catalog Number | Comments |
| non-specific protease mixture solution | Roche | 11459643001, pronase from Streptomyces griseus | Dilute in E3 without methylene blue to make 50mg/mL stock solution; store at -20°C |
| E3 solution | Dilute 50X E3 (250 mM NaCl, 8.5 mM KCl, 16.5 mM CaCl2, 16.5 mM MgSO4 in distilled water) in distilled water; add 1 x 10-5 M methylene blue (sigma M9140) to inhibit contamination | ||
| tricaine (ethyl 3-aminobenzoate methanesulfonate) | Fluka | A5040-250G | To make a 0.2% stock solution, dissolve 1 g of tricaine powder in 10 mL Tris, pH 9.5 and distilled water up to 500 mL |
| embryo dishes | VWR | 351029 | |
| 5 mL glass vials | Wheaton | 225012 | |
| disposable plastic Pasteur pippettes | VWR | 414004-004 | |
| 4% paraformaldehyde (PFA) solution | Electron Microscopy Services | 19210 | Dissolve 4% PFA in 1X PBS and bring to a boil under a fume hood. Cool and aliquot, then store at -20°C. Do not refreeze once thawed for use. |
| 10X PBS | American Bioanalytical | AB11072 | Dilute in distilled water to make a 1X stock |
| Tween-20 stock | American Bioanalytical | AB02038 | Make a 0.1% Tween-20 stock by diluting in distilled water. |
| 1X phosphate buffered saline with 0.1% Tween 20 (PBST) | Sigma | P9416 | 0.1% Tween-20 in 1X PBS |
| methanol (MeOH) | Sigma | 34860-4L | |
| proteinase K | Roche | 3115879001 | Dissolve in distilled water to make a 10 mg/mL stock; aliquot and store at -20°C |
| flat bottom microcentrifuge tubes | VWR | 87003-300; 87003-298 | |
| formamide | American Bioanalytical | AB00600 | store at -20°C |
| hybridization solution (HYB+) | 50% formamide, 5X SSC, 0.1% Tween-20, 5 mg/mL yeast torula RNA, 50 μg/μL heparin; store at -20°C | ||
| hybridization oven | Fisher Scientific | 13-247-10Q | |
| 20X saline-sodium citrate (SSC) buffer solution | American Bioanalytical | AB13156 | dilute in distilled water to make 2X and 0.2X stocks |
| blocking reagent solution | Roche | 11096176001 | dilute in maleic acid buffer to make a 10% stock solution; store at 4°C |
| maleic acid buffer solution | Sigma | M0375 and S7653 | 150mM maleic acid, 100mM NaCl (pH 7.5) |
| anti-Digoxigenin-POD, Fab fragments | Roche | 11207739910 | store at 4°C |
| Cy3 fluorescent staining solution | PerkinElmer, Inc. | NEL744001KT, TSA Plus Cyanin3 system | store at 4°C; prepare staining solution fresh by making a 1:50 dilution of TSA reagent (dissolved in 60 uL of DMSO) in the kit buffer |
| Hydrogen peroxide (H2O2) | Sigma | H1009-500mL | store at 4°C |
| molecular grade distilled water (ddH2O) | Mediatech | 25-055-CM | |
| acetone | American Bioanalytical | AB00636-01000 | store an aliquot at -20°C |
| DMSO | American Bioanalytical | AB00435-01000 | |
| fetal bovine serum (FBS) | Gibco | 10438-034 | aliquot and store at -20°C |
| monoclonal anti-acetylated tubulin clone 6-11B-1 | Sigma-Aldrich | T6793 | aliquot and store at -20°C |
| anti-γ-tubulin anitbody produced in rabbit | Sigma-Aldrich | T5192 | aliquot and store at -20°C |
| odf3b cDNA clone MGC:63985 | OpenBiosystems | IMAGE:6792478 | store bacterial glycerol stock at -80°C |
| NaCL | American Bioanalytical | AB01915-05000 | |
| Alexa flour 647 goat anti-rabbit IgG | Life Technologies | A21245 | store at 4°C; protect from light |
| Alexa fluor 488 goat anti-mouse IgG | Life Technologies | A11029 | store at 4°C; protect from light |
| DAPI | Life Technologies | D1306 | aliquot and store at -20°C |
| glass slide | Thermo-Fisher | 4445 | |
| glass coverslip | Thermo-Fisher | 12-540A | 18 x 18 mm |
| fine forceps | Roboz | RS-1050 | Dumont Tweezers Pattern #55 |
| mounting media | Polysciences, Inc. | 18606, Aqua-Poly/Mount | store at 4°C |
| confocal microscope and associated software | We use a Nikon C2plus Confocal Microscope with NIS-elements AR software | ||
| rocker | Bio-Rad | 1660710EDU |
Referencias
- Lieschke, G. J., Currie, P. D. Animal models of human disease: zebrafish swim into view. Nat Rev Genet. 8, 353-367 (2007).
- Goldsmith, J. R., Jobin, C. Think small: zebrafish as a model system of human pathology. J Biomed Biotechnol. 2012, 817341(2012).
- Howe, K., et al. The zebrafish reference genome sequence and its relationship to the human genome. Nature. 496, 498-503 (2013).
- Drummond, I. A. Kidney development and disease in the zebrafish. J Am Soc Nephrol. 16, 299-304 (2005).
- Wingert, R. A., et al. The cdx genes and retinoic acid control the positioning and segmentation of the zebrafish pronephros. PLoS Genet. 3, 1922-1938 (2007).
- Wingert, R. A., Davidson, A. J. The zebrafish pronephros: a model to study segmentation. Kindey Int. 73 (10), 1120-1127 (2008).
- Wingert, R. A., Davidson, A. J. Zebrafish nephrogenesis involves dynamic spatiotemporal expression changes in renal progenitors and essential signals from retinoic acid and irx3b. Dev Dyn. 240, 2011-2027 (2011).
- Chambers, B. E., Wingert, R. A. Renal progenitors: roles in kidney disease and regeneration. World J Stem Cells. 8 (11), 367-375 (2016).
- Drummond, B. E., Wingert, R. A. Insights into kidney stem cell development and regeneration using zebrafish. World J Stem Cells. 8 (2), 22-31 (2016).
- Poureetezadi, S. J., Wingert, R. A. Little fish, big catch: zebrafish as a model for kidney disease. Kidney Int. 89 (6), 1204-1210 (2016).
- Kramer-Zucker, A. G., Olale, F., Haycraft, C. J., Yoder, B. K., Schier, A. F., Drummond, I. A. Cilia-driven fluid flow in the zebrafish pronephros, brain and Kupffer's vesicle is required for normal organogenesis. Development. 132, 1907-1921 (2005).
- Liu, Y., Narendra, P., Kramer-Zucker, A., Drummond, I. A. Notch signaling controls the differentiation of transporting epithlelia and multiciliated cells in the zebrafish pronephros. Development. 134, 1111-1122 (2007).
- Ma, M., Jiang, Y. J. Jagged2a-Notch signaling mediates cell fate choice in the zebrafish pronephric duct. PLoS Genetics. 3, e18(2007).
- Li, Y., Cheng, C. N., Verdun, V. A., Wingert, R. A. Zebrafish nephrogenesis is regulated by interactions between retinoic acid, mecom, and Notch signaling. Dev Biol. 386 (1), 111-122 (2014).
- Wang, L., et al. miR-34b regulates multiciliogenesis during organ formation in zebrafish. Development. 140, 2755-2764 (2013).
- Marra, A. N., Wingert, R. A. Epithelial cell fate in the nephron tubule is mediated by the ETS transcription factors etv5a and etv4 during zebrafish development. Dev Biol. 411 (2), 231-245 (2016).
- Marra, A. N., Li, Y., Wingert, R. A. Antennas of organ morphogenesis: the roles of cilia in vertebrate kidney development. Genesis. 54 (9), 457-469 (2016).
- Duffy, J. L., Suzuki, Y. Ciliated human renal proximal tubular cells. Observations in three cases of hypercalcemia. Am J Pathol. 53, 609-616 (1968).
- Katz, S. M., Morgan, J. J. Cilia in the human kidney. Ultrastruct Pathol. 6, 285-294 (1984).
- Hassan, M. O., Subramanyan, S. Ciliated renal tubular cells in crescentic glomerulonephritis. Ultrastruct Pathol. 19, 201-203 (1995).
- Ong, A. C., Wagner, B. Detection of proximal tubular motile cilia in a patient with renal sarcoidosis associated with hypercalcemia. Am J Kidney Dis. 45, 1096-1099 (2005).
- Worthington, W. C., Cathcart, R. S. III Ependymal cilia: distribution and activity in the adult human brain. Science. 139, 221-222 (1963).
- Cowan, M. J., Galdwin, M. T., Shelhamer, J. H. Disorders of ciliary motility. Am J Med Sci. 321, 3-10 (2001).
- Jain, R., et al. Temporal relationship between primary and motile ciliogenesis in airway epithelial cells. Am J Respir Cell Mol Biol. 43, 731-739 (2010).
- Stubbs, J. L., Vladar, E. K., Axlerod, J. D., Kitner, C. Multicilin promotes centriole assembly and ciliogenesis during multiciliate cell differentiation. Nat Cell Biol. 14, 140-147 (2012).
- Tan, F. E., et al. Myb promotes centriole amplification and later steps of the multiciliogenesis program. Development. 140, 4277-4286 (2013).
- Zhou, F., Narasimhan, V., Shboul, M., Chong, Y. L., Reversade, B., Roy, S. Gmnc is a master regulator of the multiciliated cell differentiation program. Curr Biol. 25, 3267-3273 (2015).
- Jowett, T. Analysis of Protein and gene expression. Methods Cell Biol. 59, 63-85 (1999).
- Schulte-Merker, S. Chapter 2: Looking at Embryos. Zebrafish: A practical approach. 261, 39-58 (2002).
- Brend, T., Holley, S. A. Zebrafish whole mount high-resolution double fluorescent in situ hybridization. J Vis Exp. (25), (2009).
- Lauter, G., Soll, I., Hauptmann, G. Two-color fluorescent in situ hybridization in the embryonic zebrafish brain using differential detection systems. BMC Dev Biol. 11, (2011).
- Drummond, B. E., Li, Y., Marra, A. N., Cheng, C. N., Wingert, R. A. The tbx2a/b transcription factors direct pronephros segmentation and corpuscle of Stannius formation in zebrafish. Dev Biol. 421 (1), 52-66 (2017).
- Jaffe, K. M., Thiberge, S. Y., Bisher, M. E., Burdine, R. D. Imaging cilia in zebrafish. Methods Cell Biol. 97, 415-435 (2010).
- Lauter, G., Soll, I., Hauptmann, G. Multicolor fluorescent in situ hybridization to define abutting and overlapping gene expression in the embryonic zebrafish brain. Neural Dev. 6, (2011).
- Sorrells, S., Toruno, C., Stewart, R. A., Jette, C. Analysis of apoptosis in zebrafish embryos by whole-mount immunofluorescence to detect activated caspase 3. J Vis Exp. (82), e51060(2013).
- Schumacher, J. A., Zhao, E. J., Kofron, M. J., Sumanas, S. Two-color fluorescent in situ hybridization using chromogenic substrates in zebrafish. Bio Techniques. 57, 254-256 (2014).
- Julich, D., et al. beamter/deltaC and the role of Notch ligands in the zebrafish somite segmentation, hindbrain neurogenesis and hypochord differentiation. Dev Biol. 286, 391-404 (2005).
- Gross-Thebing, T., Paksa, A., Raz, E. Simultaneous high-resolution detection of multiple transcripts combined with localization of proteins in whole-mount embryos. BMC Biology. 12, (2014).
- Cheng, C. N., Li, Y., Marra, A. N., Verdun, V., Wingert, R. A. Flat Mount Preparation for Observation and Analysis of Zebrafish Embryo Specimens Stained by Whole Mount In situ Hybridization. J Vis Exp. (89), e51604(2014).
- Poureetezadi, S. J., Donahue, E. K., Wingert, R. A. A Manual Small Molecule Screen Approaching High-throughput Using Zebrafish Embryos. J Vis Exp. (93), e52063(2014).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados