Method Article
Sondas de detección óptica de novela bacterias específicas en el tejido de pulmón humano Ex Vivo por láser Confocal Endomicroscopy
En este artículo
Resumen
Esta técnica describe un proceso de selección eficiente para la evaluación de agentes ópticos de imagen específicos de bacterias dentro del tejido de pulmón humano ex vivo , por microscopía de fluorescencia confocal con fibras para la identificación rápida de pequeñas moléculas químicas sonda-candidatos con potencial traducible.
Resumen
Mejorar la velocidad y la precisión en la detección bacteriana es importante para la estratificación de los pacientes y para asegurar el uso apropiado de antimicrobianos. Para lograr este objetivo, el desarrollo de técnicas de diagnóstico para reconocer la presencia bacteriana en tiempo real en el punto de cuidado es necesario. Imágenes ópticas para la identificación directa de bacterias dentro del host son un enfoque atractivo. Se han investigado varias tentativas en el diseño de la sonda química y validación, sin embargo ninguno están aún correctamente traducido en la clínica. Aquí se describe un método para la validación de ex vivo de las bacterias específicas sondas para la identificación de bacterias en el pulmón distal, reflejada por microscopía de fluorescencia confocal fibrado (FCFM). Nuestro modelo utiliza ex vivo el tejido de pulmón humano y una plataforma de endomicroscopy (CLE) de láser confocal clínicamente aprobado para pantalla novela bacterias específicas de compuestos, cerca mímico condiciones imagen esperadas a encontrarse con los pacientes. Por lo tanto, compuestos de detección mediante esta técnica proporciona confianza de maleabilidad clínica potencial.
Introducción
Esta técnica describe un proceso de cribado rápido para evaluar agentes ópticos de imagen específicos de bacterias, ex vivo el tejido de pulmón humano por CLE utilizando FCFM para la identificación rápida de compuestos con potencial utilidad clínica para la visualización de bacterias en el pulmón distal in situ.
Hay un requisito global urgente para racionar la prescripción de antimicrobianos en la época de creciente resistencia a los antimicrobianos1. Para ello, el desarrollo de métodos de diagnóstico que actúan para identificar infección bacteriana con alta especificidad, sensibilidad y en tiempo real son altamente buscado2. Las técnicas actuales para confirmar el diagnóstico de neumonía en pacientes críticamente enfermos, tales como ésos dentro de unidades de cuidados intensivos (UCI), a menudo se basan en la interpretación no específica características clínicas o radiológicas junto a técnicas de cultivo bacteriano de aspiración líquidos/tejidos, que puede tardar hasta 3 días para producir resultados. Además, cultivo bacteriano del líquido instilado en el pulmón distal y obtenido es propenso a la contaminación de las vías aéreas más proximales3 es a menudo la cultura negativa debido a la terapia antimicrobiana concomitante o técnicas de muestreo pobre. Además, las técnicas moleculares como la reacción en cadena de polimerasa son demasiado sensibles cuando se usa en los fluidos aspirados, correr el riesgo de sobretratamiento de los pacientes. Un nuevo enfoque diagnóstico es imagen óptica molecular, lo que en situ la patología molecular de los tejidos una posibilidad; sin embargo, el desarrollo y la validación de compuestos de proyección de imagen ópticos se requiere. Sin embargo, la visualización directa de las bacterias, a través de sondas ópticas activatable es potencialmente un método muy de gran alcance para permitir el estudio de la presencia y evolución de la neumonía en el paciente y lo que es importante, podría utilizarse para estudiar las interacciones huésped-patógeno en respuesta a los tratamientos en tiempo real en situ.
CLE es un procedimiento de investigación establecido en múltiples enfermedades4, incluyendo dentro de los campos de Gastroenterología5, Oncología6,7y para interrogar a las vías respiratorias y los sacos alveolares8, 9. permite punto de atención la proyección de imagen estructural de tejidos enfermos utilizando una fibra paquete, que pasa a través del canal de trabajo de un endoscopio de clínico de imágenes y formas de contacto dirigen con la superficie del tejido a ser reflejada por microscopia confocal. Sin embargo, una limitación que permanece es la necesidad de agentes de contraste genérico. Por lo tanto, el uso de sondas específicas de la enfermedad, tales como agentes bacterianos específicos, podría ampliar enormemente la utilidad de esta modalidad al visualizar directamente las bacterias en el sitio de sospecha de infección. Agentes ópticos ofrecen muchas ventajas sobre otras técnicas que permiten proyección de imagen de alta resolución en tiempo real con un potencial diagnóstico. Por otra parte, sondas ópticas ofrecen la posibilidad de multiplexación para interrogar a varios destinos, todo logrados a un costo relativamente bajo. Un número de agentes ópticos está bajo desarrollo para tal fin, sin embargo ningunos todavía han sido implementados con éxito dentro de la clínica10. Hemos sintetizado una biblioteca de sondas químicas de molécula pequeña con especificidad hacia bacterias y desarrollado una tubería rápida y eficaz para la evaluación de la función de la sonda para la detección de neumonía bacteriana en situ11.
Para identificar a los candidatos de la sonda adecuado, los siguientes requisitos previos tenían que cumplirse antes de la interrogación de la sonda sobre el ex vivo el tejido de pulmón humano por FCFM: i) acuoso solubilidad, ii) especificidad y selectividad para rápidamente etiquetado clínicamente bacterias relevantes, iii) un alto cociente signal-to-noise y iv) resistencia a la degradación en el entorno de pulmón. Este último fue evaluado mediante lavado broncoalveolar (BALF) de pacientes con síndrome de dificultad respiratoria agudo (SDRA), que es una condición que se caracteriza por entornos proteolíticas e inflamatorias en el pulmón en la UCI. Por otra parte, las sondas debían tener un fluoróforo adecuado para la detección por un clínico óptica CLE imagen dispositivo aprobado dentro del tejido alveolar de pulmón humano.
La tubería para interrogar a cada uno de estos requisitos previos era como sigue (en cada etapa, sólo las puntas de prueba que se llevaban adelante a la siguiente): (1) una biblioteca de sondas para investigar fue sintetizada; (2) cada punta de prueba se agregó a un grupo de bacterias vivas por láser confocal de barrido (CLSM) la microscopia para asegurar el etiquetado bacteriana; (3) selectividad de etiquetado bacterianas sobre las células mamíferas en co-cultivos con neutrófilos humanos primarios fue establecido por CLSM; (4) estabilidad y éxito de etiquetado de las bacterias en presencia de la paciente SDRA BALF se determinó por CLSM y matriz asistida por láser desorción/ionización tiempo de vuelo (MALDI-TOF) espectrometría de masas; (5) óptima concentración de candidatos se determinó por CLSM, asegurar la selectividad de bacterias sobre las células de mamíferas se mantuvo; (6) candidatos fueron reflejados por la FCFM en suspensión y en ex vivo humano alveolar tejido pulmonar para asegurar la estabilidad y la signal to noise era adecuada para la detección de. Paso 6 se describe en detalle dentro de este protocolo. Metodología para los pasos 1-5 ha sido previamente reportado11.
Protocolo
Todo tejido de pulmón humano se obtuvo tras el consentimiento informado y el estudio fue aprobado por el Comité Regional de ética.
1. preparación de muestras biológicas
-
Preparación de sondas
- Conforman una solución stock de 1 mM de cada punta de prueba (e.g., calceína AM, UBI-3, UBI-10, etc.) en el estéril dH2O usando un fino equilibrio para pesar el liofilizado sonda compuesto11. Calcular el volumen de dH2O añadir basado en la masa pesada y peso molecular de la sonda.
-
Preparación de cultivos bacterianos
Nota: Para este método, estafilococo áureo fue utilizado como la cepa del ejemplar. Puede seleccionarse cualquier cepa bacteriana adecuada. Cualquier tinte comercial que etiquetas bacterias con una excitación adecuada y espectros de emisión puede seleccionarse positivamente etiquetar las bacterias.- Seleccione una sola Colonia de la cepa bacteriana deseada en una placa de agar caldo de lisogenia (LB) fresca utilizando un asa estéril. Inocular la Colonia en 10 mL de LB en un tubo de centrífuga de 50 mL sumergiendo el extremo del lazo en los medios de cultivo. Incubar a 37 ° C, 250 rpm durante 16 horas (o toda la noche).
- Determinar el OD595 de la cultura durante la noche añadiendo 100 μl de la cultura durante la noche a 900 μl de LB en una cubeta de 1 mL. Medir la do a 595 nm (utilizando una cubeta con 1 mL LB como un espacio en blanco) en un espectrofotómetro. El OD obtenido se multiplican por 10 para obtener la do para la cultura durante la noche.
- Subcultura de la cultura durante la noche. Para ello, ajustar la densidad óptica a 595 nm (OD595) a 0.1 en 10 mL de lb fresco calcular el volumen requerido de la cultura durante la noche a 10 mL fresco LB para ajustar OD595 a 0.1. Incubar el cultivo a 37 ° C, 250 rpm hasta que la cultura llegue a fase de registro medio (OD595 0.6 - 0.8), aproximadamente 4 horas.
- Medir la densidad óptica de la cultura (paso 1.2.2) y cosecha 1 x 108 Colonia formando unidades (UFC) (OD595 ~ 1-1 x 108 UFC/mL) del cultivo bacteriano en un microtubo de 1.5 mL (por ejemplo, si el cultivo bacteriano es OD595 0.6, recoger mL 1,67). Centrifugar el cultivo a 10.000 x g durante 1 min para que sedimenten las bacterias. Lavar el pellet dos veces en fosfato tampón salino (PBS) transfiriendo resuspender (arriba y abajo con cuidado) el sedimento en 1 mL de PBS, centrifugar como arriba, descartando el sobrenadante y repetir. Tenga cuidado de no para desalojar el pellet bacteriano al retirar el sobrenadante. Resuspender el precipitado final en 1 mL de PBS.
Nota: Preparar tantas muestras como sea necesario para cada procedimiento de etiquetado. El protocolo puede hacer una pausa aquí para hasta 1 h, seguido de paso 1.2.5.1 y 1.2.5.2 1.2.5.3. - Tinción de bacterias
- Para etiquetar las bacterias con calceína AM, añadir el colorante a una concentración final de 1 μm en el cultivo bacteriano lavado. Incubar el cultivo durante 30 min a 37 ° C con agitación a 300 rpm. Lavar la suspensión bacteriana counterstained en PBS 3 veces por centrifugación como en el paso 1.2.4 para eliminar el exceso de tinte. Resuspender en 1 mL de PBS, diluir 100 μL 1:1 en PBS para obtener 200 μL de OD595 calceína 0,5 AM bacterias teñidas. El protocolo puede hacer una pausa aquí para un máximo de 1 h.
- Para etiquetar las bacterias con puntas de prueba (p. ej., UBI-10 o UBI-3), diluir 100 μl de cultivo bacteriano 1:1 en PBS para obtener OD595 0.5 en 200 μL PBS. Añadir que cualquiera de las puntas de prueba a una concentración final de 10 μm. invertir los microtubos varias veces para asegurar una mezcla completa de las bacterias y la sonda.
Nota: la proyección de imagen debe realizarse inmediatamente después de la adición de la sonda para imitar el escenario clínico. - Para preparar el control de muestras bacterianas sin manchas, diluir 100 μl de la cultura 1:1 con PBS para obtener 200 μL de sin mancha OD595 0.5 muestra.
-
Preparación del ex vivo el tejido de pulmón humano
Nota: Las muestras de tejido de pulmón humano se obtuvieron de pacientes sometidos a resección quirúrgica para el carcinoma de pulmón. Todo el tejido utilizado para la proyección de imagen se obtuvo de muestras de tejido del pulmón normal de crecimiento canceroso. Se toma fresco de la sala de operaciones y almacenados en microtubos o centrifugar los tubos a-80 ° C hasta su uso.- Inmediatamente antes de la proyección de imagen, retire la muestra de tejido de pulmón humano del congelador de hielo seco. A temperatura ambiente, permita que el tejido se descongele un poco; lo suficiente como para ser cortado con un bisturí en secciones de 1 x 4 mm2 .
Nota: El nivel de deshielo es importante; también congelado y el tejido no se corte sin saltar, demasiado descongelado y el tejido es demasiado blando para cortar. - Utilizando pinzas, coloque el tejido pulmonar humano en rodajas en pocillos de una placa de cultivo de tejidos claro 96 pozos de fondo plano. Retomar cualquier tejido de pulmón humano sin usar inmediatamente el recipiente y el lugar en hielo seco para el transporte hacia el congelador de-80 ° C.
- Añadir 100 μl PBS a cada muestra de tejido de pulmón con una pipeta. Utilizar la pipeta para asegurarse todo el tejido es cubierto en el PBS (y no pegado a las paredes del pozo). El tejido se hinche ligeramente y puede flotar. Deja el PBS en la muestra por unos minutos permitir que cualquier sangre del tejido en la solución de lixiviación. Eliminar gran parte del PBS como sea posible. El tejido puede bloquear el extremo de la punta de la pipeta; trate de la colocación de la placa o la punta del ángulo para prevenir esto.
- Pipetear 100 μl de sin mancha, calceína estoy etiquetado, prueba indicados o bacterias para cada bien que contenga tejido pulmonar. También configurar los controles con tejido pulmonar y 100 μl PBS. A estos pozos de control, sondas sin bacterias pueden agregarse para medir cualquier aumento en fluorescencia del fondo o en la activación inespecífica de la sonda por tejido pulmonar sola. También debe incluir un pozo con tejido pulmonar y PBS.
- Inmediatamente antes de la proyección de imagen, retire la muestra de tejido de pulmón humano del congelador de hielo seco. A temperatura ambiente, permita que el tejido se descongele un poco; lo suficiente como para ser cortado con un bisturí en secciones de 1 x 4 mm2 .
2. la proyección de imagen con el dispositivo CLE con FCFM
-
Puesta en marcha de dispositivos CLE
Nota: Prepare el sistema CLE 20 min antes de la calibración para permitir que el láser caliente.- Presione el interruptor de encendido en la parte posterior del transformador del sistema y encienda la computadora designada. Presione el botón de encendido/apagado en la parte delantera de la unidad (LSU) de escaneo láser. Confirman la aparición de la luz verde, indicando que la unidad está encendida.
- Haga doble clic en el icono del software CLE. Introduce los datos de inicio de sesión y esperar la LSU inicializar (10-30 s).
- Para el uso de la FCFM nuevo fibras de la proyección de imagen, instalación con el CD suministrado es necesario. Inserte el CD de instalación en el equipo unidad de CD y siga las instrucciones en pantalla.
- Limpie la unidad de FCFM imagen fibra conector con una fibra más limpia. Frote el conector de la cinta de limpieza en un movimiento hacia adelante para quitar cualquier polvo o la suciedad. Retire la tapa amarilla de la parte delantera de la LSU.
- Preparar el centro LSU girando suavemente el centro de plata hacia la izquierda hasta que se detenga. Inserte el conector de fibra imagen FCFM en el cubo con el lado plano del conector hacia arriba. Mantener la fibra en su lugar y gire el cubo de plata hacia la derecha hasta que haga clic dos veces. Completar la conexión girando el cubo de plata hacia la izquierda hasta 45° más.
Nota: Si la fibra no es reconocida, verifique que la FCFM de la fibra ha sido instalada y conectado en la orientación correcta. - Siga las instrucciones que aparecerán para completar la FCFM calibración fibra la proyección de imagen. Hay 3 pasos: (1) FCFM fibra imagenológico (pasos 2.1.7 - 2.1.8), adquisición (2) Fondo (paso 2.1.9), detección (3) fibra (paso 2.1.10).
- Pulse el botón 'start laser' en la pantalla. El láser se centrará.
- Seleccione frascos frescos (amarillo: calibrar; rojo: clean; azul: enjuague) de la calibración de kit y siga las instrucciones en pantalla: Coloque el extremo distal de la FCFM de la fibra en el frasco amarillo y ver para el aumento de fluorescencia en el monitor, luego coloque el punta de fibra en el frasco rojo (sin revolver). Espere a que la fluorescencia (como se muestra en la pantalla del ordenador) a desaparecer. Finalmente, enjuague la punta de la fibra en el frasco azul.
Nota: Si la fluorescencia desaparece, limpiar la fibra imagen FCFM con 8% H2O2 y los tejidos de limpieza y empezar de nuevo. Repita el proceso hasta que se obtienen resultados satisfactorios (calidad de la imagen es clara, y marcas de suciedad no son evidentes). - Lugar de la FCFM de la fibra en el frasco azul. Pulse 'start láser' seguido de 'calcular' cuando esto se convierte en una opción.
- Lugar de la FCFM de la fibra en el frasco amarillo. Pulse 'start láser' seguido de 'calcular' cuando esto se convierte en una opción.
- Durante la calibración automática, limpie el extremo distal de la FCFM de fibra colocando en el frasco rojo para > 10 s, seguido por el frasco azul para > 4 s, según lo indicado por el software.
-
Recopilación de datos con CLE
- Tras la instalación, se abrirá una ventana para seleccionar la ubicación de almacenamiento y prefijo de archivo. Seleccione la carpeta de almacenamiento de datos y por consiguiente el nombre el prefijo.
- Coloque los pedales por lo que se puede acceder fácilmente por el operador. Pedal izquierdo: láser de encendido/apagado; pedal del centro: una pausa; pedal derecho: grabar/detener.
Nota: Los controles de láser también se pueden acceder a través de controles en pantalla. - Haga clic en 'Inicio' en la pantalla o pulse el pedal izquierdo para encender el láser. Esto comenzará la adquisición y obtener imágenes usando 100% láser de potencia y una velocidad de 12 fotogramas/s (por defecto).
Nota: Para otras aplicaciones, estas configuraciones pueden modificarse en la pantalla si es necesario, dependiendo del tipo de muestra. - Imagen de cada una de las muestras de la suspensión bacteriana.
- Inserte el extremo distal de la fibra de proyección de imagen de FCFM y mover la fibra lentamente a través de la suspensión a interrogar a la muestra.
- Grabar videos de cualquier longitud (hasta 10 min) pulsando el pedal de pie derecho o selección de los controles en pantalla de registro, mientras que la fibra se mueve lentamente alrededor de la muestra.
- Limpie el extremo distal de la fibra de proyección de imagen de FCFM con 8% H2O2 y los tejidos entre las muestras de limpieza.
Nota: Longitudes videos típico de 10-30 s son suficientes para proyección de imagen en vitro .
- Imagen de cada una de las muestras de tejido pulmonar.
- Inserte el extremo distal de la FCFM de la fibra en la muestra, asegurándose que el contacto directo entre el extremo de la fibra y el tejido se hace. Suavemente hacia el extremo de la fibra de alrededor de interrogar a la muestra.
Nota: Levantar el extremo de la fibra del tejido eliminará el tejido desde el plano focal; sin embargo, esto puede usarse para bacterias etiquetadas que no se adhieren al tejido de la imagen. - Grabar videos de cualquier longitud (hasta 10 min) pulsando el pedal de pie derecho o selección de los controles en pantalla de registro, mientras que la fibra se mueve lentamente alrededor de la muestra.
Nota: Normalmente, videos largos de 30 s es suficiente para ex vivo la proyección de imagen sobre el tejido. - Limpie el extremo distal de la fibra de proyección de imagen de FCFM con tejidos y 8% H2O2 entre muestras para limpiar lentes.
- Inserte el extremo distal de la FCFM de la fibra en la muestra, asegurándose que el contacto directo entre el extremo de la fibra y el tejido se hace. Suavemente hacia el extremo de la fibra de alrededor de interrogar a la muestra.
-
Apagar el sistema
- Apague el láser presionando el pedal izquierdo o pulsando el botón en pantalla.
- Desconecte la fibra imagen FCFM el dispositivo CLE girando el plata centro LSU en sentido antihorario hasta que se detenga. Retire la FCFM de fibra desde el hub LSU tirando suavemente del conector de la fibra de la LSU.
- Limpie y desinfecte la fibra imagen FCFM con 8% H2O2 y limpieza de los tejidos. Retomar las tapas protectoras del extremo proximal de la fibra de proyección de imagen de FCFM y el frente de la unidad LSU. Coloque suavemente la fibra en la caja.
- Cierre los datos de software de captura y copiar los archivos guardados en un dispositivo USB externo. Apague el equipo y apague el dispositivo LSU presionando el panel frontal I/O para 3 s hasta que desaparezca la luz verde.
- Disponer de tejido pulmonar humano y bacterias según las normas locales.
3. Análisis de los datos
- Abra el software y los archivos para el análisis de importación seleccionando el directorio apropiado en el equipo mediante el icono 'Ir a' en el tablero de instrumentos de software. Alternativamente, los archivos se pueden arrastra y coloca en el tablero de instrumentos de software.
- Haga doble clic en cada archivo de vídeo para abrirlos. Los videos se reproducirá con la tabla de color optimizado lookup (LUT) y ajustar la tabla de colores. Desactivar la intensidad automática de escala, haciendo clic en el botón del tubo encima de la barra de escala de intensidad. La función se desactiva cuando no hay ninguna sombra negra alrededor del botón.
Nota: Escala de intensidad automática debe desactivarse para evitar que el realce del contraste continuo a lo largo de cada video, lo que hace imposible comparar y analizar los videos del mismo conjunto de datos. - Seleccionar la intensidad deseada escala moviendo las barras mínimas y máximas para dar el mejor contraste. Utilizar la herramienta Histograma cuando seleccionando la intensidad escala para garantizar el más amplio rango dinámico es capturado.
Nota: Asegúrese de que el rango dinámico es tal que las imágenes no son saturadas (es decir, limitar las regiones blancas de la imagen, que indican saturación), por lo que no le falten características de baja intensidad. - Una vez que se ha logrado la expansión deseada, haga clic derecho sobre el botón de menú desplegable aparece como 'Default (verde)'. Seleccione la opción de guardar el LUT. Guardar el LUT a una ubicación deseada.
- Uno para el otro video dentro del conjunto de datos, aplicar el LUT misma haciendo clic derecho sobre el menú desplegable 'Por defecto (verde)' y seleccione 'Carga LUT'. Seleccione el archivo apropiado aplicar intensidad constante a todos los videos dentro de un conjunto de datos.
- Exportar vídeos procesados haciendo clic en el botón 'carrete de película'. Seleccione el formato de vídeo deseado, por ejemplo, 'para fines de presentación', que producirá un archivo .mpg. Pulse 'Exportación' y eligió la ubicación del archivo para guardar el archivo de vídeo. Fotos de marcos individuales pueden exportarse haciendo clic en el botón de 'cámara'. Es posible guardar un archivo .png, .bmp o .jpg. Elija el destino del archivo y oprima guardar.
Nota: Videos luego pueden ser importados en cualquier software para preparación de presentaciones o más cuantificación. Etiquetado bacterias se visualizan como puntos verdes 'parpadear' en el video. La estructura del tejido pulmonar será evidente como filamentos fluorescentes ordenados, con espacio alveolar parece negro.
Resultados
En este estudio, hemos demostrado un método para el cribado rápido de novela sondas específicas de bacterias en un ex vivo pulmón alveolar humano tejido modelo de infección mediante un dispositivo clínico aprobado del CLE.
CLE de FCFM es idóneo para la obtención de información estructural en el pulmón distal, ya que esta región (debido a una alta abundancia de elastina y colágeno) naturalmente es altamente fluorescente cuando emocionado con un 488 nm láser8. Por el contrario, el espacio alveolar no es fluorescente, y como tal permite alto contraste entre la estructura del tejido y espacio aereo para ser visualizados (figura 1).
La adición de enfermedades relacionadas con las sondas o agentes de contraste, tales como sondas específicas de bacterias deben permitir información funcional acerca de los procesos de enfermedad que se obtendrán en tiempo real. Anteriormente hemos descrito la síntesis y proyección inicial en vitro de una biblioteca de bacterias específicas sondas11; donde especificidad bacteriana, se determinó la estabilidad proteolítica y la retención dentro de la membrana bacteriana con el tiempo. Una sonda prometedor de bacterias específicas (UBI-10) fue identificada en el estudio, así como uno que mostraba retención pobre dentro de la membrana celular bacteriana (UBI-3). Estos fueron comparados con un control de comercial counterstaining (calceína AM) que estaba etiquetado como S. aureus.
Mancha, calceína AM, UBI-3 y UBI-10 con la etiqueta S. aureus fueron reflejada en suspensión por la FCFM con 100% 488 nm láser de potencia y una velocidad de 12 fotogramas/s (figura 2). Donde las bacterias sin etiqueta en PBS se desliza señal fluorescente no era perceptible. Esto está en contraste con cuando las bacterias marcadas fueron reflejadas. Cuando suspensiones bacterianas con UBI-3 o 10 de UBI se desliza por la FCFM fue aparente que la fluorescencia del fondo general de la solución fue elevado en comparación con los controles solos PBS, esto es porque al siguiente día laborable (sonda fluoróforo) emiten una pequeña cantidad de señal de fluorescencia en solución acuosa, sin embargo, brillantes punteadas puntos son claramente visibles a lo largo de la solución, sin necesidad de un paso de lavado. Esto es debido a un aumento en la señal de fluorescencia emitida por NDB en un entorno polar , es decir, la membrana bacteriana. Calceína AM no es una sonda activatable, así un paso de lavado después de la tinción bacteriana fue necesaria para eliminar el fondo fluorescente alta de sonda independiente en la solución. Como UBI-3 y UBI-10 etiquetadas bacterias, bacterias con calceína AM fueron detectadas en solución por FCFM como puntos punteadas verdes brillantes. Como los datos se recopilan en formato de vídeo, estos puntos parecen 'twinkle' mientras se mueven entre corazones y entrada y salida del foco, un rasgo característico de la imagen las bacterias marcadas por este método.
Las bacterias marcadas fueron posteriormente agregadas a pequeños sectores de ex vivo el tejido de pulmón humano y reflejadas otra vez por FCFM (figura 3). Donde el tejido pulmonar se agrega sólo PBS o sin etiqueta S. aureus , se detectó sólo el pulmón autofluorescent estructura del tejido (visto como brillantes verdes filamentos de colágeno y elastina y las zonas oscuras del espacio alveolar). Se detectaron ningunos puntos punteados para estas condiciones de control. Del mismo modo, se visualizan la estructura de tejido de pulmón único (y ningunos puntos punteados) para la condición del tejido pulmonar con S. aureus más UBI-3; indicando que este sondeo no se mantuvo estable en el bacteriano de la célula membrana , es decir, era eliminada o se degrada en presencia de enzimas proteolíticas nativas dentro del tejido pulmonar (como anteriormente demostrado11).
Sin embargo, brillantes punteadas puntos eran visibles en ambos la calceína AM marcado muestra de control positivo S. aureus y con la sonda más prometedor de bacterias específicas (UBI-10) S. aureus muestra. Los puntos 'centelleo' eran visibles a pesar de la autofluorescencia de fuerte tejido (figura 3). Así, los resultados obtenidos por la FCFM en concurrencia con la proyección previa en vitro del panel de sondas específicas de bacterias por CLSM y demostraron un método de detección clínicamente relevante para las infecciones en tiempo real la proyección de imagen.
Los resultados presentados aquí demuestran que el pulmón es un sistema del órgano apropiado para la proyección de imagen de FCFM debido a su distintivo autofluorescencia. Las estructuras distintivas brillantes permiten al operador CLE determinar que se encuentran en el espacio alveolar. Estas regiones, juntadas con los sacos de aire alveolares oscuros proporcionan el escenario perfecto para fluorescencia etiquetadas bacterias con alto contraste de imagen.
Aunque la detección de bacterias presentada en este estudio se determina cualitativamente por visualización brillantes punteadas puntos, podría ser posible cuantificar el número de puntos punteadas cuadro por cuadro usando un software de secundario con el fin de caracterizar bibliotecas de la sonda.
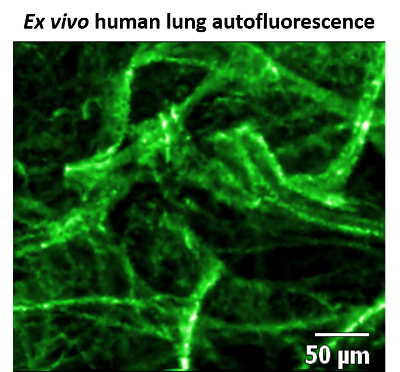
Figura 1: Imagen estática de Autofluorescencia de tejido de pulmón humano. Imagen endomicroscopy (CLE) de láser confocal ex vivo el tejido de pulmón humano utilizando microscopía de fluorescencia confocal fibrado (FCFM), en el 488 de nm de excitación, energía del laser de 100% y 12 frames/s. elastina y el colágeno son altamente fluorescentes (falso color verde), Considerando que el espacio alveolar no es y aparece como regiones negras. Esta figura ha sido modificada de Akram et al. 11 Haga clic aquí para ver una versión más grande de esta figura.
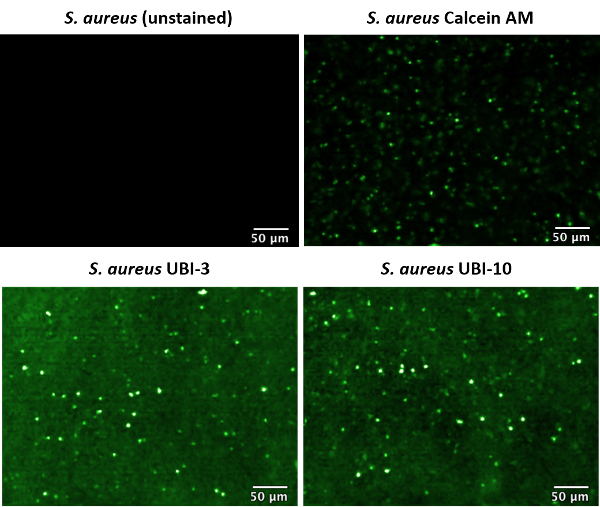
Figura 2: Imagen de endomicroscopy (CLE) de láser confocal de etiquetado S. aureus suspensión. Microscopía de fluorescencia confocal fibrado (FCFM) se utilizó para imagen bacterias previamente etiquetadas, en 488 de nm de excitación, energía del laser de 100% y 12 frames/s. etiquetadas las bacterias muestran como altamente fluorescentes puntos punteadas (falso color verde). Esta figura ha sido modificada de Akram et al. 11 Haga clic aquí para ver una versión más grande de esta figura.

Figura 3: Imagen de endomicroscopy (CLE) de láser confocal ex vivo tejido de pulmón humano con etiqueta S. aureus. Microscopía de fluorescencia confocal fibrado (FCFM) se utilizó para imagen ex vivo humano tejido pulmonar y la etiqueta de S. aureus, en 488 de nm de excitación, energía del laser 100% y 12 frames/s. etiquetadas bacterias mostrar puntos punteadas altamente fluorescentes (falso color verde) dentro de la muestra de tejido del pulmón cuando etiquetados con calceína AM o UBI-10. El mayor contraste se observa donde las bacterias se reflejada en el espacio alveolar. Esta figura ha sido modificada de Akram et al. 11 Haga clic aquí para ver una versión más grande de esta figura.
Discusión
Las infecciones del tracto respiratorio inferior representan la segunda mayor carga de enfermedad a nivel mundial12,13y un substancial aumentan en el número de infecciones atribuidas a bacterias resistentes a los antimicrobianos ha sido reportado14. La neumonía sigue siendo una causa frecuente de hospitalización. En la UCI, el desarrollo de una neumonía agravado por la incertidumbre de diagnóstico y se asocia con una mortalidad extremadamente alta tasa15. Durante el inicio de la neumonía, las bacterias proliferan en el espacio alveolar del pulmón distal, un área que es relativamente estéril, con un mínimo microbiota en la salud.
Este método describe relativamente última etapa ex vivo validación de bacterias específicas óptico imagen sondas11, pero el diseño, síntesis y evaluación de la sonda antes de comenzar con este paso de validación es imprescindible, como se muestra previamente11 .
CLE es una técnica clínica emergente para interrogar a enfermedad en situ en tiempo real. Ofrece muchas ventajas sobre las técnicas tradicionales para investigar la presunta patología pulmonar, que puede implicar una colección del líquido de lavado y biopsia. Las biopsias son invasivas y pueden causar morbilidad y mortalidad en pacientes ventilados, y líquido del lavado recolectadas a menudo está contaminado con bacterias de las vías aéreas superiores. El uso del CLE en la detección de neumonía es sin embargo algo limitado debido a la pobre disponibilidad de imágenes compatible con sondas que pueden proporcionar información funcional de la enfermedad, a pesar de muchos esfuerzos10. CLE la combinación con agentes ópticos ofrece la posibilidad de diagnosticar neumonía más rápida y menos invasiva en comparación con la práctica estándar actual.
Los pasos críticos de este protocolo son la preparación de la muestra y la instalación de la plataforma CLE. Obtención de tejido humano relevante para la aplicación clínica final también es importante, como el tejido de pulmón humano según lo demostrado en este estudio. Es necesario utilizar tejido humano porque el grado de tejido autofluorescence muestra gran variación entre las especies y por lo tanto puede inducir la sensibilidad de la bacteria-sonda que se va a examinar. Además, es esencial obtener ética para la recuperación y uso del tejido de pulmón humano. De un nivel, correcta limpieza técnica, accesorio de la fibra de proyección de imagen para la proyección de imagen plataforma LSU y calibración es esencial para la buena resolución e imagen consistente, es garantizar un número equivalente de bacterias se agrega a cada muestra de tejido pulmonar. Para ampliar aún más la utilidad de este método para paneles de sondas de detección, es necesario repetir el procedimiento con una amplia gama de patógenos, como los que parecen ser agentes causales de neumonía.
La mayor limitación de esta técnica es que el dispositivo CLE clínicamente aprobado tiene un único láser (488 nm). Por lo tanto, actualmente, la selección de fluoróforo para diseño de la sonda es limitada para el uso con este sistema, aunque clínicamente aprobado solo color existen dispositivos con longitudes de onda de excitación de 660 nm y el infrarrojo cercano. Es deseable tener una segunda línea de láser en el mismo dispositivo para habilitar un sondeo a desarrollarse con un fluoróforo espectralmente distinto para mejorar la sensibilidad de las bacterias-sonda sobre el nivel del tejido autofluorescencia. Mientras que los dispositivos de color dual CLE están en desarrollo, son o no clínicamente aprobados o su costo es significativo16.
CLE en vitro con bacterias patógenas y ex vivo tejido pulmonar humano a las sondas de potencial pantalla llena el vacío entre convencional en vitro técnicas como la citometría de flujo y CLSM y utilidad clínica. Este paso ofrece confianza al seleccionar compuestos prometedores para llevar a cabo para acoplarse con clínica CLE la proyección de imagen; y darán indicación acerca de si la sonda de prueba mantiene la especificidad de la blanco, o muestra cualquier objetivo de etiquetado, como atar directamente al tejido o muestra inestabilidad con gran cantidad de enzimas proteolíticas. También sería pertinente añadir cada una de las sondas activables directamente a muestras de tejido pulmonar humano más bacterias, para caracterizar completamente la velocidad de la prueba enlace y la activación en tiempo real.
Creemos que nuestra tubería de detección rápidamente nuevas sondas de bacterias específicas para evaluar su potencial para la proyección de imagen en el pulmón distal de pacientes tendrá como resultado una traducción mucho más rápida a la clínica. Esto es en gran parte porque la sonda específica de bacterias podría ser entregada localmente en el pulmón a través de un catéter insertado por el canal de trabajo de un broncoscopio, lo que significa que microdose (< 100 μg) cantidades podrían ser entregadas. Por lo tanto, entrega sistémica y biodistribución del compuesto no es una preocupación, como es el caso para muchos otros objetivos de infección en el cuerpo, o con la proyección de imagen nuclear. Además, entrega la sonda de imagen en una pequeña dosis reduce el riesgo de toxicidad relacionada con complicaciones (aunque el tamizaje de la toxicidad sería necesario para la traducción). Después de la instilación de la sonda, el catéter luego podría ser sustituido por la fibra de la FCFM y la misma región del pulmón interrogado por CLE, del mismo modo hemos realizado dentro de este método. La proyección de imagen debe realizarse rápidamente después de la instalación de la sonda antes de que la sonda se remueve a concentraciones indetectables.
También es importante tener en cuenta esa proyección de identificar enfermedad sondas mediante esta técnica no debe limitarse a los agentes de la proyección de imagen de bacterias, pero también podría extenderse a la validación de las sondas con objetivos alternativos, tales como inflamación. Este enfoque también debe ser adaptable a otras localizaciones de la enfermedad dentro del cuerpo donde está permitida la proyección de imagen a través de la FCFM.
Divulgaciones
KD: Director fundador de la proyección de imagen Molecular de Edimburgo. Asesoramiento recibido de Mauna Kea Technologies como asesor.
MB: Director fundador de la proyección de imagen Molecular de Edimburgo.
Agradecimientos
Nos gustaría agradecer la beca ingeniería y colaboración interdisciplinaria de investigación Physical Sciences Research Council (EPSRC, Reino Unido) EP/K03197X/1, del Departamento de salud y el Wellcome Trust a través de la salud innovación Challenge Fund (HICF) . Número de referencia de financiación: 0510-069.
Materiales
| Name | Company | Catalog Number | Comments |
| 1-14 Microfuge | SciQuip | 90616 | Small benchtop microcentrifuge |
| 96-well plate | Corning | 3370 | Assay plate |
| Calcein AM | Sigma Aldrich | 17783 | Commercial fluorescent dye |
| Cellvizio 488 nm Research CLE | Mauna Kea Technologies | LC-0001-488 | Confocal laser endomicroscopy device |
| Cletop-S | Cletop | 14110601 | Fibre cleaner |
| Eppendorf (1.5 mL) | Eppendorf | 30120086 | 1.5 mL microfuge tube |
| Eppendorf Research Plus Pipettes | Fisher Scientific | 11568663 | Micro pipettes |
| Falcon tube (50 mL) | Scientific Lab Supplies | 352070 | 50 ml centrifugation tube |
| Gibco Phosphate Buffered Saline | Thermo Fisher Scientific | 10010023 | PBS - wash media |
| IC-Viewer | Mauna Kea Technologies | LW-0001 | Data collection and processing software for the research CellVizio 488 nm system |
| Incu-Shake midi | Sciquip | SQ-4020 | Floor standing shaking incubator |
| Lysogeny Broth | Sigma Aldrich (Miller) | L3522 | LB Growth media for S. aureus |
| non-standard research AlveoFlex 488 nm | Mauna Kea Technologies | MP-0002-AF3 | Fibred confocal fluorescence microscopy fibre |
| Quanti Kit 488 nm | Mauna Kea Technologies | LQ-0005 | Calibration kit for the CellVizio 488 system |
| S. aureus ATCC 25923 | ATCC | 25923 | Bacterial strain used in this study |
| Semi-micro spectrophotometry cuvette | Sigma Aldrich | C5416-100EA | For spectrophotometry |
| Thermomixer comfort | Eppendorf | 41102422 | Benchtop heater with shaking |
| UV 1101 Biotech photometer | Biochrom WPA | Spectrophotometer |
Referencias
- Tackling drug-resistant infections globally: final report and recommendations. London: H M Government/Wellcome Trust Available from: https://amr-review.org/sites/default/files/160518_Final%20paper_with%20cover.pdf (2016)
- Caliendo, A. M., et al. Better Tests, Better Care: Improved Diagnostics for Infectious Diseases. Clin. Infect. Dis. 57, 139-170 (2013).
- Zumla, A., et al. Rapid point of care diagnostic tests for viral and bacterial respiratory tract infections-needs, advances, and future prospects. Lancet Infect. Dis. 14 (11), 1123-1135 (2014).
- Neumann, H., Kiesslich, R. . Yamada's Textbook of Gastroent. , 2944-2949 (2015).
- Chauhan, S. S., et al. Confocal laser endomicroscopy. Gastrointest Endosc. 80 (6), 928-938 (2014).
- Goetz, M., Malek, N. P., Kiesslich, R. Microscopic imaging in endoscopy: endomicroscopy and endocytoscopy. Nat Rev Gastroenterol Hepatol. 11 (1), 11-18 (2014).
- Abbaci, M., et al. Confocal laser endomicroscopy for non-invasive head and neck cancer imaging: A comprehensive review. Oral Oncol. 50 (8), 711-716 (2014).
- Thiberville, L., et al. Human in vivo fluorescence microimaging of the alveolar ducts and sacs during bronchoscopy. Eur Respir J. 33, (2009).
- Thiberville, L., et al. Confocal fluorescence endomicroscopy of the human airways. Proc Am Thorac Soc. 6 (5), 444-449 (2009).
- Mills, B., Bradley, M., Dhaliwal, K. Optical imaging of bacterial infections. Clin. Transl. Imaging. 4 (3), 163-174 (2016).
- Akram, A. R., et al. A labelled-ubiquicidin antimicrobial peptide for immediate in situ optical detection of live bacteria in human alveolar lung tissue. Chem. Sci. 6 (12), 6971-6979 (2015).
- Dickson, R. P., Erb-Downward, J. R., Huffnagle, G. B. Towards an ecology of the lung: new conceptual models of pulmonary microbiology and pneumonia pathogenesis. Lancet Respir Med. 2 (3), 238-246 (2014).
- Lozano, R., et al. Global and regional mortality from 235 causes of death for 20 age groups in 1990 and 2010: a systematic analysis for the Global Burden of Disease Study 2010. Lancet. 380 (9859), 2095-2128 (2012).
- Magiorakos, A. P., et al. The rise of carbapenem resistance in Europe: just the tip of the iceberg. Antimicrob Resist Infect Control. 2 (1), 6 (2013).
- Chastre, J., Fagon, J. -. Y. Ventilator-associated Pneumonia. Am. J. Respir Crit Care Med. 165 (7), 867-903 (2002).
- Krstajić, N., et al. Two-color widefield fluorescence microendoscopy enables multiplexed molecular imaging in the alveolar space of human lung tissue. J Biomed Opt. 21 (4), 046009-046009 (2016).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados