Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Valoración de ELISA como método para determinar la constante de disociación de la interacción del ligando del Receptor
En este artículo
Resumen
Se describe un protocolo detallado para llevar a cabo una valoración de ELISA. Por otra parte, se presenta un nuevo algoritmo para evaluar valoración ELISAs y obtener una constante de disociación de la Unión de un ligando soluble a un receptor inmovilizado en la placa de microtitulación.
Resumen
La constante de disociación describe la interacción entre dos socios en el equilibrio de la Unión y es una medida de su afinidad. Es un parámetro crucial para comparar diferentes ligandos, por ejemplo, los inhibidores competitivos, isoformas de la proteína y mutantes, por su fuerza vinculante a un socio de la Unión. Constantes de disociación se determinan trazando las concentraciones de consolidados versus gratis ligando como curvas de enlace. En contraste, curvas de titulación, en el cual una señal que es proporcional a la concentración del ligand encuadernado se enfrenta a la concentración total de ligando añadida, son más fáciles de grabar. La señal puede ser detectada espectroscópico y por el análisis enzima-ligado del inmunosorbente (ELISA). Esto se ejemplifica en un protocolo para una valoración de ELISA que mide la Unión de la rhodocetin derivado de veneno de serpiente su dominio destino inmovilizados de la integrina α2β1. Valoración ELISA es versátiles y ampliamente utilizado. Cualquier par de interacción de proteínas puede utilizarse como inmovilizado receptor y ligando soluble, siempre que ambas proteínas son puros, y sus concentraciones son conocidas. Hasta ahora la dificultad ha sido determinar la constante de disociación de una curva de titulación. En este estudio, se introduce una función matemática subyacente a las curvas de titulación. Sin ninguna estimación de errores gráfico de un rendimiento de saturación, este algoritmo permite el procesamiento de los datos en bruto (intensidades de la señal a diferentes concentraciones de ligando agregado) directamente por la evaluación matemática mediante regresión no lineal. Así, varias curvas de valoración se pueden grabar simultáneamente y transformadas en un conjunto de parámetros característicos, entre ellos la constante de disociación y la concentración de receptores de enlace activo, y pueden ser evaluados estadísticamente. Cuando se combina con este algoritmo, titulación ELISA obtener la ventaja de presentar directamente la constante de disociación. Por lo tanto, se puede utilizar más eficientemente en el futuro.
Introducción
La constante de disociación K es un parámetro clave para describir la afinidad de un receptor (R) por su ligando (L). Basado en la ley de acción de masas, K se define el equilibrio, en el que el RL complejos ligando-receptor se disocia en el receptor R y el ligando L:
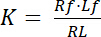 Ecuación 1
Ecuación 1
con los índices f que indica el estado libre de/sin consolidar del receptor y ligando. La concentración del complejo receptor-ligando, RL, es idéntica a la concentración del ligand del receptor-limita Lb. Como la concentración total de receptores Rt es la suma de la libre receptor Rf y enlazado a ligando receptor Rb = Lb, la constante de disociación también se puede escribir como:
 Ecuación 2
Ecuación 2
Por lo tanto, la saturación del rendimiento Y, definido como la fracción de ligando enlazado Lb en la relación de la concentración total de receptores Rt,
 Ecuación 3
Ecuación 3
depende de la concentración de ligando libre Lf:
 Ecuación 4
Ecuación 4
Esta relación hiperbólica describe la curva de unión de una interacción ligando-receptor y su muestra la concentración de ligando enlazado Lb como una función de la concentración de ligando libre Lf. De la curva de enlace, la constante de disociación K puede ser derivada como la concentración de ligando libre en el rendimiento máximo de media saturación. Por otra parte, se han establecido diferentes algoritmos para linealizar las curvas de enlace, tales como el diagrama recíproco doble por Klotz1,2, o transformaciones según Scatchard o Hanes (revisado por Bisswanger3). Sin embargo, todos los algoritmos sufren el problema de que el máximo valor de la producción de saturación, que se acerca asintóticamente a altas concentraciones de ligando libre en la curva de enlace, debe ser estimado en una evaluación de la gráfica y por lo tanto es propenso a errores.
Además, la determinación de una curva de enlace requiere la cuantificación de ligando libre y atado durante el equilibrio de la Unión. Con este fin, el ligando libre tiene que ser separado del ligand del receptor-limita y cuantificado. Por tanto, el ligando y el receptor que difieren en sus propiedades, tales como un ligando de la proteína no frente a un receptor de la proteína. Si ambos socios de la Unión son las proteínas, tienen que ser distinguibles en sus tamaños, cargos u otras características moleculares. Sin embargo, la cuantificación de las concentraciones de ligando en enfoques vinculantes en pequeña escala es una tarea difícil. Etiquetado radiactivo del ligand ha sido necesario para detectar la baja concentración de ligando enlazado, sobre todo si grandes cantidades de receptores no estaban disponible o asequible. Por otra parte, el ligando del receptor-limita puede disociar durante y después del aislamiento de manera no despreciable. Por lo tanto, métodos complejos, tales como equilibrio gel filtración4electroforesis capilar5y pulso proteólisis6, se requiere cuantificar ligando receptor-limita y separarlo del ligando libre.
En contraste con estos ensayos de Unión, experimentos de titulación no requieren la separación cuantitativa de ligand encuadernado y libre. Con este fin, se titula un receptor a una concentración constante con diferentes concentraciones de ligando añadido. Atando al receptor, el ligand encuadernado tiene una propiedad biofísica que distingue desde el ligando libre y es mensurable por, por ejemplo, fotometría, fluorometría o detección de anticuerpos. Así, una señal de S, que es proporcional a la saturación del rendimiento Y y por lo tanto también a la concentración del ligand del receptor-limita (Lb), se detecta como una función de la concentración total de agregado ligando (Lt). Ambos parámetros, la señal S y la concentración total de ligando añadido se cuantifican de manera directa y más fácil que las concentraciones de ligando dependiente y libre. Sobre todo, la detección del ligando del receptor-limita por análisis enzima-ligado del inmunosorbente (ELISA) permitida la reducción de volúmenes de muestra que por debajo de 100 μl, así como mediciones paralelas de varias concentraciones de ligando en placas de varios pocillos de microtitulación. En una titulación ELISA, un receptor es físicamente adsorbido sobre una placa de microtitulación en la misma concentración y tituló con ligando soluble. El receptor se inmoviliza a la superficie de plástico esencialmente por adsorción hidrofóbica. La concentración superficial de inmovilizados receptor se correlaciona con la concentración de la capa del receptor en una relación no lineal, probable acuerdo de isoterma de adsorción de Langmuir´s7. Además el número total de moléculas adsorbidas del receptor, su estado de actividad es otro parámetro importante para los ensayos de titulación. Sólo inmovilizó receptores que mantienen la actividad de unión del ligando, son relevantes para el ensayo de titulación y eventualmente contribuyan a la concentración total de receptores activos Rt de la prueba de valoración, que no puede determinarse directamente.
Sitios en la superficie de plástico, que no están cubiertos por el receptor inmovilizado son propensos a absorber otras proteínas, como el ligando. Adsorción física del ligand de tales sitios de superficie plásticas resultaría en una señal similar como el ligand del receptor-limita, sin embargo, de una manera no específica. Para reducir esta señal inespecífica, los sitios de superficie plásticos de las placas de microtitulación que no han sido cubiertos con proteínas aún se bloquearán con albúmina de suero bovino (BSA). Sin embargo, para algunos ensayos de titulación del receptor-ligando, se pueden observar señales de fondo no específicas. Entonces, otros agentes de bloqueo, como una solución de 0,2% de gelatina o de 0.04% Tween 20, se recomienda.
Después de atar al receptor, el ligando libre se retira por dos pasos de lavado. Ligand encuadernado se queda con el receptor, que es inmovilizado en la superficie del pocillo de microtitulación de plástico y opcionalmente reforzado por fijación química. Para la posterior covalente eslabón dependiente ligando y receptor inmovilizado con glutaraldehído, se sustituye la sustancia tampón TRIS de HEPES, sin ningún cambio en el atascamiento del ligand. HEPES, en contraste con TRIS, desactivar glutaraldehído. El Cruz-el acoplamiento covalente con glutaraldehído fija el ligando enlazado con su receptor y evita su disociación durante los pasos posteriores de lavado e incubación. Así, la interacción receptor-ligando se congela químicamente y garantiza una curva de titulación que es inafectada por los pasos posteriores de lavado e incubación. Sin embargo, fijación de glutaraldehído puede modificar químicamente el ligando y el receptor de tal manera que su interacción es reducida o suprimida. Por otra parte, modificación de epítopos en el ligand puede cambiar la afinidad del anticuerpo de detección, especialmente si se utiliza un anticuerpo monoclonal para cuantificar ligand encuadernado. Aunque ninguno de estos efectos adversos de la fijación de glutaraldehído se produce en esta titulación ELISA, la sensibilidad de la prueba hacia el glutaraldehído debe comprobarse para cada interacción receptor-ligando antes del experimento de titulación. Después de la fijación, se quita exceso glutaraldehído en tres pasos de lavado con tampón de TRIS que contiene. TRIS inactiva restantes grupos aldehído, que pueden reaccionar un con detección de anticuerpos en el paso posterior.
Se cuantifica la cantidad de ligando enlazado con los anticuerpos enzima-ligados, que proporcionan una señal fotométrica de ELISA S. Esto se grafica versus el total ligand concentración Lt agregadas a cada pozo. A pesar de su fácil adquisición, la curva de valoración no es una función hiperbólica en contraste con la curva de enlace. Además, ha sido claro cómo calcular la constante de disociación K de una curva de titulación. Aunque algoritmos para linealizar las curvas de titulación espectroscópico adquirida se han reportado independientemente por Stockell8 y Heyn y Weischet9, cayó debido a la incertidumbre de estimar la señal de máximo valor que el rendimiento de saturación se aproxima a altas concentraciones de ligando añadido.
Aquí, una titulación de ELISA y el algoritmo de regresión no lineal se describen para derivar la constante de disociación K para una interacción del ligand del receptor de una curva de titulación. Este protocolo se ejemplifica por la interacción del colágeno-enlace A-dominio de la integrina α2β1 con un inhibidor derivado de veneno de serpiente. Las integrinas son moléculas de adhesión celular, que median el anclaje de las células a la matriz extracelular circundante o a la membrana basal subyacente10,11. Además, las integrinas transmitan importantes señales entre las células y la matriz extracelular mediante la contratación de las moléculas de señalización adicional y la formación de nuevos orgánulos celulares, adhesomes, al celular-matriz de interacción de12,13, 14. colágeno, el ligando de la integrina α2β1, es la proteína más abundante del cuerpo humano y es un componente crucial del andamio del tejido conectivo15. La interacción entre el colágeno y la integrina α2β1 está mediada por el dominio A de la subunidad de integrina α2. El integrina α2A-dominio contiene un catión divalente, que se requiere para el atascamiento de colágeno y estabiliza su estructura. La forma salvaje como mutantes del dominio α2A, como la de que los residuos de superficie expuesta Y216 habían sido reemplazado por una glicina, fácilmente pueden producidos por vía recombinante en un sistema de expresión bacteriana y aislados por sus oligo-su-etiquetas con un NiNTA columna de Superflow con una diálisis posterior contra solución salina tamponada con TRIS (TBS; 50 mM TRIS/HCl, pH 7,4, 150 mM NaCl) con 2 mM de MgCl216. Sus concentraciones se determinaron con el ensayo ácido bicinchoninic (BCA) y sus purezas son probados por convencional SDS-PAGE y teñidos con Coomassie Brilliant Blue R250.
La interacción entre el colágeno y la integrina α2β1 está bloqueada por la Unión de la componente de veneno de serpiente, rhodocetin, de la víbora Malaya (Calloselasma rhodostoma)16,17. Utilizado como un ligando soluble en esta titulación ELISA, rhodocetin se purificó del crudo veneno como se describió anteriormente16. Se disuelve en solución salina tamponada con HEPES (HBS; 10 mM HEPES/NaOH, pH 7,4, 150 mM NaCl) y se almacena congelado a-20 ° C. Su concentración fue determinada por BCA y su pureza fue probada por SDS-PAGE. Como un antagonista, rhodocetin no sólo bloques de colágeno vinculante a la integrina α2β1 A dominio, pero también estabiliza la conformación inactiva de la integrina evitando cualquier señalización de colágeno en las células o las plaquetas18. Es de gran importancia biomédica para determinar la constante de disociación de rhodocetin con su target receptor y así desentrañar su mecanismo molecular y farmacéuticos potenciales p. ej., como agente antitrombótico. Para ello, una valoración de ELISA se describe incluyendo su evaluación, que es aplicable a casi cualquier interacción receptor-ligando con una estequiometría 1:1 interacción.
Protocolo
1. soluciones
- Para preparar 100 mL de 10 x TBS solución de pH 7.4, disolver 6,06 g de TRIS y 8,77 g de NaCl en 90 mL de agua desionizada, ajustar el pH a 7.4 con solución de HCl 37%, completar el volumen a 100 mL con agua desionizada y filtrar la solución.
- Para preparar 100 mL de 1 M HEPES/NaOH, solución pH 7,4, disolver 23,83 g de HEPES en 90 mL de agua desionizada, ajustar el pH a 7.4 con 1,5 M NaOH, completar el volumen a 100 mL con agua desionizada y filtrar la solución.
- Para preparar 100 mL de solución de NaCl de 5 M, se disuelven 29,2 g de NaCl en 90 mL de agua desionizada. Completar el volumen a 100 mL con agua desionizada y filtrar la solución.
- Para preparar 100 mL de solución de 1 M MgCl2 , disolver 20,33 g de MgCl2 · 6H2O 90 ml de agua desionizada. Completar el volumen a 100 mL con agua desionizada y filtrar la solución.
- Para preparar 5% BSA inactivadas por calor en el agua, pesar 2,5 g de BSA (fracción V, pH 7.0) en un tubo de 50 mL y disolver en 45 mL de agua desionizada. Llenar la solución a 50 mL con agua desionizada y calentar la solución en un baño de agua a 68 ° C por 45 min enfriar en un baño de hielo y almacenar a-20 ° C.
- Preparar solución acuosa de glutaraldehído al 25%.
PRECAUCIÓN: Glutaraldehído es dañino si se traga, tóxico por inhalación y causa quemaduras. Usar ropa protectora, guantes y protección ojos/cara. - Levante el antisuero de conejo como se describió anteriormente19. El título del antisuero se determinó según protocolos estándar20.
- Preparación de anticuerpos de inmunoglobulina de Anti-conejos de cabra conjugado con fosfatasa alcalina.
- Para preparar 100 mL de solución de glicina de 0.1 M, se disuelven 0.75 g de glicina en 90 mL de agua desionizada. Ajustar el pH a 10.4 con solución de NaOH de 1.5 M, completar el volumen a 100 mL con agua desionizada y filtrar la solución.
- Para preparar 100 mL de 0,5 M Zn (II)-solución de acetato de disolver 10,98 g de Zn (II)-acetato · 2H2O 90 ml de agua desionizada. Completar el volumen a 100 mL con agua desionizada y filtrar la solución.
- Para preparar 100 mL de solución de NaOH de 1.5 M, disolver 6,0 g de NaOH en 90 mL de agua desionizada. Completar el volumen a 100 mL con agua desionizada y filtrar la solución.
2. preparación de Buffers y soluciones
- Diluir 5 mL de 10 x TBS pH 7,4, con 45 mL de agua desionizada y añada 100 μl de una solución de 1 M MgCl2 . Mantener a temperatura ambiente. El TBS, pH 7.4, 2 mM de MgCl2 solución es para la inmovilización de los receptores y lavado de la placa de microtitulación.
- Diluir 10 mL de 5% de BSA inactivadas por calor en agua y 5 mL de 10 x TBS, pH 7,4 con agua desionizada a 50 mL. Añada 100 μl de solución stock de 1 M MgCl2 y mezcle bien la solución. Mantenerla en hielo y guardarla para otros experimentos a-20 ° C. Nota que 2,5 mL de 1% de BSA en TBS, pH 7.4, 2 mM de MgCl2 se requieren para cada curva de titulación.
- Diluir 2,5 mL de 1 M HEPES/NaOH, solución de NaCl de 5 M pH 7.4 y 1.5 mL a 50 mL con agua desionizada, añadir 100 μL 1 M solución de MgCl2 y mezclar la solución de fondo para preparar 50 mL de HBS, pH 7.4: 50 mM HEPES/NaOH , 150 mM NaCl.
- Añadir 5 μl de solución stock de 1M MgCl2 y 2 μl 0.5 M Zn (II)-solución madre de acetato de 5 mL 0,1 M glicina solución pH 10.4 preparar buffer de fosfatasa alcalina (AP) (solución de glicina de 0.1 M, pH 10,4, 1 mM MgCl2, 0,2 mM Zn(II)-acetate).
3. inmovilización de los receptores (integrina α2A-dominio) a una placa de microtitulación
- Diluir la solución madre de integrina α2A-dominio en TBS, pH 7.4, 2 mM de MgCl2 para una concentración final de 5 μg/mL.
Nota: El volumen de la solución de recubrimiento es 650 μL para una curva de valoración que incluye un 12 fila de pozo de una microplaca de zona media. - Llene cada pocillo de una fila en una microplaca de la zona media con 50 μL/pocillo de la solución de recubrimiento μg/mL 5 integrina α2A-dominio (tipo salvaje o mutante). Realizar cada fila de titulación al menos por duplicado (en este ejemplo como cuadrúpedos; ver diseño de placa de microtitulación en la figura 1).
- Sellar la placa con papel de aluminio o cerrarlo con una tapa. Dejar la placa a 4 ° C durante la noche.
4. lavado cubrió los pocillos de la placa dos veces con TBS, pH 7.4, 2 mM de MgCl2
- Retirar del receptor soluble las moléculas, que no han sido inmovilizadas a la superficie de plástico por adsorción física, retirar la solución de recubrimiento y llenan cada uno bien con 50 μl de TBS, pH 7.4, 2 mM MgCl2.
- Retire la solución de lavado. Asegurar que los pozos no se convierta en seco. Por lo tanto, no golpee la placa de microtitulación sobre un paño de tejido para eliminar el líquido residual. Utilice una pipeta de varias fases o una pipeta multicanal para llenar los pozos rápidamente.
- Repita este paso de lavado una vez.
5. bloquear sitios de Unión inespecíficos
- Añadir 50 μl de solución de BSA 1% en TBS, pH 7.4, 2 mM de MgCl2 a cada pocillo.
- Sellar los pozos con papel de aluminio o cerrar con una tapa.
- Incubar los pocillos durante 1 h a temperatura ambiente.
Nota: Cualquiera de los pasos de incubación de este protocolo se puede realizar en una plataforma oscilante o agitación. Sin embargo, esto no es necesario y no altera el resultado del experimento.
6. preparación de la dilución Serial consecutivo del ligando, Rhodocetin
- Variar la concentración de inicio de rhodocetin y el factor de dilución de la dilución serial, para obtener un rango de concentraciones de ligando apropiado y registrar una curva de titulación completa con una señal de mínima y máxima. En este experimento, emplear una concentración de comienzo de 243 nM rhodocetin y un factor de dilución de 2.3. Diluir la solución madre rhodocetin a la mayor concentración de ligando de la fila de dilución seriada. Para cada curva de titulación con un factor de dilución de 2.3, preparar 115 μl de solución de rhodocetin de 243 nM (es decir, 15.2 μg/mL) en el 1% de BSA/TBS, pH 7.4, 2 mM de MgCl2 en tubo de ensayo #1.
- Llenar 65 μl de solución de BSA 1% en TBS, pH 7.4, 2 mM de MgCl2 en 10 tubos de ensayo, con la etiqueta #2 al #11.
- Transferir 50 μl de la dilución de rhodocetin de tubo de ensayo #1 para tubo de ensayo #2, mezclar ambas soluciones (volumen total: 115 μl; factor de dilución: 1: 2. 3) por trituración y, a continuación, transferir 50 μl de esta mezcla a la probeta #3, etc.
- Continuar esta dilución seriada hasta el tubo de prueba #11.
Nota: Los volúmenes en pasos 6.1-6.4 son suficientes para una curva de titulación. Estos volúmenes se multiplican por el número de repeticiones. En este caso, realizar ocho curvas de titulación (cuatrillizos de dos formas de dominio α2A) y preparar los siguientes volúmenes: 920 μl de solución de rhodocetin en la mayor concentración en la probeta #1; 520 μl de 1% de BSA en TBS, pH 7.4, 2 mM de MgCl2 llenar en cada uno de los tubos de prueba #2 y #11; y 400 μL de volumen de transferencia de un tubo a otro.
7. atascamiento de Ligand (Rhodocetin) a diferentes concentraciones de inmovilizado del Receptor (integrina α2A-dominio)
- Retire la solución de bloqueo de los pocillos de la placa por una línea de vacío.
- Inmediatamente añada 50 μl de la solución de rhodocetin en 1% BSA/TBS, solución de pH 7.4/MgCl2 de tubo de ensayo #1 en los pocillos de la columna 1, solución de probeta #2 en los pocillos de la columna 2,etc. añadir 50 μl de 1% de BSA en TBS, pH 7.4 , 2 mM de MgCl2 (tampón de dilución y el bloqueo) como un control libre de ligando en los pocillos de la columna 12 (ver diseño de placa de microtitulación, figura 1).
- Sellar los pozos con papel de aluminio o cerrar con una tapa.
- Incubar los pocillos durante 1,5 h a temperatura ambiente (alrededor de 20-22 ° C).
8. lavado de pozos de la placa dos veces con HBS, pH 7.4, 2 mM de MgCl2
- Para eliminar moléculas de ligando sin limite, retire la solución de enlace y rellenar cada una con 50 μl de HBS, pH 7.4, 2 mM de MgCl2. A continuación, retire la solución de lavado.
- Tenga cuidado de que los pozos no se queden secos. Por lo tanto, no golpee la placa de microtitulación sobre un paño de tejido para eliminar el líquido residual. Utilice una pipeta de varias fases o una pipeta multicanal para llenar los pozos rápidamente.
- Repita este paso de lavado una vez.
9. Fije el ligando del Receptor-limita con 2.5% glutaraldehído en HBS, pH 7.4, 2 mM de MgCl2
- Preparar una solución de glutaraldehído al 2.5% fresco mezclando 1 parte de solución de glutaraldehído al 25% y 9 partes de HBS, pH 7.4, 2 mM de MgCl2.
- Llene cada pocillo de la placa de microtitulación con 50 μl de la solución de glutaraldehído al 2.5% de HBS, pH 7.4, 2 mM de MgCl2. Incubar la placa de microtitulación durante 10 min a temperatura ambiente.
10. lavar los pocillos de la placa tres veces con 50 μL/pocillo de TBS, pH 7.4, 2 mM de MgCl2
- Para quitar y desactivar exceso glutaraldehído, retirar la solución de fijación y rellenar cada una con 50 μl de TBS, pH 7.4, 2 mM de MgCl2. Retire la solución de lavado.
Nota: Verter la solución que contiene glutaraldehído de fijación de la placa de microtitulación en un plato y deseche la solución de fijación después de que se ha desactivado por un volumen igual de TBS, pH 7,4. - Tenga cuidado de que los pozos no se queden secos. Por lo tanto, no golpee la placa de microtitulación sobre un paño de tejido para eliminar el líquido residual. Utilice una pipeta de varias fases o una pipeta multicanal para llenar los pozos rápidamente.
- Repita este paso de lavado dos veces.
11. cuantificación del Ligand del Receptor-limita por ELISA
- Añadir 50 μL/pocillo de la solución de anticuerpo primario de 1% de BSA en TBS, pH 7.4, 2 mM de MgCl2. La solución de anticuerpo primario es un antisuero de conejo contra rhodocetin19, diluido 1:2,000 en 1% de BSA en TBS, pH 7.4, 2 mM de MgCl2.
- Incubar la placa de 75-90 min a temperatura ambiente. Lavar todos los pocillos de la placa tres veces con 50 μL/pocillo de TBS, pH 7.4, 2 mM de MgCl2. Durante los tres pasos de lavado, golpecitos de la placa de microtitulación sobre un tejido, la tela no es necesaria.
- Añadir 50 μL/pocillo de la solución de anticuerpo secundario en 1% de BSA en TBS, pH 7.4, 2 mM de MgCl2. Para ello, diluir el anticuerpo secundario, dirigidas a la inmunoglobulina cabra anticuerpos de conejo conjugados con fosfatasa alcalina, a 1:2,000 en 1% de BSA en TBS, pH 7.4, 2 mM de MgCl2. Incubar la placa de 75-90 min a temperatura ambiente.
- Preparar la solución de detección de AP disolviendo una tableta de 5 mg que contiene 4-nitrofenil fosfato disodio sal hexahidrato (sustrato de la fosfatasa) en 5 mL de tampón de AP (solución de glicina de 0.1 M, pH 10,4, con 1 mM de MgCl2 y 0,2 mM Zn(II)-acetate).
- Lavar todos los pocillos de la placa tres veces con 50 μL/pocillo de TBS, pH 7.4, 2 mM de MgCl2, inmediatamente antes de realizar el siguiente paso. Grifo de la microplaca sobre un paño de tejido después del último paso de lavado para eliminar todo rastro de líquido.
- Añadir 50 μL/pocillo de la solución de detección de AP en los pocillos de la microplaca. Añadir solución de detección de AP puntualmente a todos los pozos para iniciar la conversión enzimática de simultáneamente como sea posible. Por lo tanto, utilice una pipeta multicanal.
- Incubar la placa a temperatura ambiente hasta que la solución en los pozos con mayor concentración de ligando se ponen amarillas.
Nota: El tiempo de incubación puede variar entre 5 minutos y 1 hora dependiendo de la intensidad de la señal. - Detener la conversión del substrato fosfatasa por añadiendo 50 μL/pocillo de solución de NaOH de 1.5 M. Deje la placa vertical durante varios minutos asegurar sin rayar una mezcla de ambas soluciones. Para justificar el mismo período de incubación en todos los pocillos, utilice una pipeta de varios canales y añadir el NaOH M 1,5 en el mismo orden que se añade a los pocillos que del sustrato detección de AP en paso 11.8.
- Medir la densidad óptica (do) a 405 nm de cada pozo por un lector de ELISA.
12. evaluación de las señales de valoración
- Abra la tabla de datos brutos, los valores de OD405 nm , con Excel. Como estos valores de la señal de las curvas de titulación se leen en filas, transposición de los valores de una columna y la etiqueta con las concentraciones de ligando añadido en otra columna.
- Abra el Graphpad Prism 5 (versión 5.0). Abra un nuevo archivo de proyecto en el menú principal. Elegir el formato XY gráficoy nuevos datos. Elija la opción Enter y la trama de un punto único para cada valor del eje y.
- Copie las dos columnas, concentración de ligando agregado y valores (OD405 nm ) desde el excel archivan y pegarlos en la hoja de datos de GraphPad prisma como la señal X y Y de valores, respectivamente.
- Abra el subprograma análisis de GraphPad Prism 5 y elija la opción regresión no-lineal de XY-análisis. Seleccionar ecuación definida por el usuario y presionar el botón nuevo para crear una nueva ecuación.
- Escriba la ecuación de la curva de titulación en la forma: Y =(Smax-Smin)*((X+R+K)-sqrt((X+R+K)^2-4*R*X)) /(2*R) Smin + B * X en la hoja plantilla recién inaugurado, y siendo el valor de la señal S, X siendo la concentración de ligando agregado L , R es la concentración de inmovilizado del receptor, siendo la constante de disociación, K y B siendo el fondo de la cuesta. Definir las restricciones adecuadas, como K > 0 y R > 0.
Nota: Esta ecuación es la misma ecuación de la ecuación 9 en forma diferente. - Analizar los valores de la hoja de datos, escogiendo la ecuación definida por el usuario recién creado. Abra la tabla con los valores de la aproximación calculada (K, Rt, Smax, Sminy el B) que se muestran en la sección de resultado en el lado izquierdo de la pantalla del software.
Nota: El software determina los 5 parámetros ajuste iterativo de la regresión no lineal sólo si la curva de titulación consta de al menos 5 puntos. - Evaluar estadísticamente los parámetros K, Rt, Smax, Sminy B para cada grupo de curvas de titulación y los parámetros se correlacionan con la función específica del grupo (transformado o modificado químicamente ligando o receptores).
Resultados
Después de la ELISA se ha desarrollado, el color amarillo del sustrato de la fosfatasa alcalina se puede convertir, para- nitrofenolato, indica que la cantidad de ligando enlazado rhodocetin disminuye con la disminución de las concentraciones de mayor rhodocetin de columnas 1 a 11 (figura 1). Los pozos descoloridos en los pozos libres de rhodocetin en la columna 12 muestran una señal de bajo fondo.
Discusión
La valoración de ELISA es un sistema versátil de la prueba para determinar la disociación de una interacción ligando-receptor. Como la valoración ELISA evita la necesidad de separar eficazmente ligandos libres y encuadernados y analizar cuantitativamente, sus concentraciones substancialmente más estudios y publicaciones han empleado titulación ELISA en vez de curvas de enlace de la grabación . Por otra parte, titulación ELISAs son fáciles de realizar y requieren cantidades razonablemente bajas de receptor y lig...
Divulgaciones
El autor no tiene nada que revelar.
Agradecimientos
El protocolo y el algoritmo se desarrollaron dentro de un proyecto financiado por la Deutsche Forschungsgemeinschaft (DFG concesión SFB1009 A09 y EB177/13-1). El autor agradece a Barbara Schedding y Félix Schmalbein para soporte técnico y Dr. Niland para leer críticamente el manuscrito.
Materiales
| Name | Company | Catalog Number | Comments |
| TRIS | neoFrox | 1125KG001 | |
| HEPES | Sigma-Aldrich | H4034 | |
| NaCl | Applichem | 1,316,591,214 | |
| MgCl2 | Merck | 172571 | |
| integrin a2A, wild-type and mutant, recombinant | isolated in author's lab | ||
| NiNTA superflow column | Qiagen, Germany | 30821 | |
| Coomassie-Brilliant Blue R250 | Serva | 35050 | |
| bicinchoninic acid assay (BCA), protein concentration determination kit | Fisher Scientific | 23225 | |
| bovine serum albumine (BSA), fraction V | Applichem | A1391 | |
| 25 % solution of glutaraldehyde | Merck | 354400 | |
| anti-rabbit immunglobulin-antibodies from goat, conjugated with alkaline phosphatase | Sigma-Aldrich | A9919 | |
| Glycine | Applichem | A1377 | |
| Zn(II)-acetate | Applichem | A4324 | |
| NaOH | Applichem | A1551 | |
| Alkaline phosphatase substrate tablet (5 mg) | Sigma-Aldrich | S0942 | |
| Costar half-area microtiter plate | Thermo Scientific | Corning 3690 | |
| micro reaction tubes | Eppendorf | 30120086 | |
| Microplate ELISA reader | BioTek | Synergy HT |
Referencias
- Klotz, I. M. The application of the law of mass action to binding by proteins; interactions with calcium. Arch Biochem. 9, 109-117 (1946).
- Klotz, I. M. Ligand-receptor complexes: origin and development of the concept. J Biol Chem. 279 (1), 1-12 (2004).
- Bisswanger, H. Ch. 1: Multiple Equilibria, Principles and Derivations. Enzyme Kinetics: Principles and Methods. , 1-26 (2017).
- Shimura, K., Kasai, K. Affinity gel titration: quantitative analysis of the binding equilibrium between immobilized protein and free ligand by a continuous titration procedure. Anal Biochem. 149 (2), 369-378 (1985).
- Gong, M., Nikcevic, I., Wehmeyer, K. R., Limbach, P. A., Heineman, W. R. Protein-aptamer binding studies using microchip affinity capillary electrophoresis. Electrophoresis. 29 (7), 1415-1422 (2008).
- Hanes, M. S., Ratcliff, K., Marqusee, S., Handel, T. M. Protein-protein binding affinities by pulse proteolysis: application to TEM-1/BLIP protein complexes. Protein Sci. 19 (10), 1996-2000 (2010).
- Latour, R. A. The Langmuir isotherm: a commonly applied but misleading approach for the analysis of protein adsorption behavior. J Biomed Mater Res A. 103 (3), 949-958 (2015).
- Stockell, A. The binding of diphosphopyridine nucleotide by yeast glyceraldehyde-3-phosphate dehydrogenase. J Biol Chem. 234 (5), 1286-1292 (1959).
- Heyn, M. P., Weischet, W. O. Circular dichroism and fluorescence studies on the binding of ligands to the α subunit of tryptophan synthase. Biochem. 14 (13), 2962-2968 (1975).
- Campbell, I. D., Humphries, M. J. Integrin structure, activation, and interactions. Cold Spring Harb Perspect Biol. 3 (3), (2011).
- Zeltz, C., Gullberg, D. The integrin-collagen connection--a glue for tissue repair?. J Cell Sci. 129 (4), 653-664 (2016).
- Kanchanawong, P., et al. Nanoscale architecture of integrin-based cell adhesions. Nature. 468 (7323), 580-584 (2010).
- Luo, B. H., Carman, C. V., Springer, T. A. Structural basis of integrin regulation and signaling. Annu Rev Immunol. 25, 619-647 (2007).
- Luo, B. H., Springer, T. A. Integrin structures and conformational signaling. Curr Opin Cell Biol. 18 (5), 579-586 (2006).
- Ricard-Blum, S. The collagen family. Cold Spring Harb Perspect Biol. 3 (1), a004978 (2011).
- Eble, J. A., Tuckwell, D. S. The α2β1 integrin inhibitor rhodocetin binds to the A-domain of the integrin α2 subunit proximal to the collagen-binding site. Biochem J. 376 (Pt 1), 77-85 (2003).
- Eble, J. A., et al. The α2β1 integrin-specific antagonist rhodocetin is a cruciform, heterotetrameric molecule. FASEB J. 23 (9), 2917-2927 (2009).
- Eble, J. A., et al. Dramatic and concerted conformational changes enable rhodocetin to block α2β1 integrin selectively. PLoS Biol. 15 (7), e2001492 (2017).
- Bracht, T., Figueiredo de Rezende, F., Stetefeld, J., Sorokin, L. M., Eble, J. A. Monoclonal antibodies reveal the alteration of the rhodocetin structure upon α2β1 integrin binding. Biochem J. 440 (1), 1-11 (2011).
- Harlow, E., Lane, D. Chapter 5: Immunizations. Antibodies, a laboratory manual. 5, 53-138 (1988).
- Lu, D. Analyzing interactions between SSB and proteins by the use of fluorescence anisotropy. Methods Mol Biol. 922, 155-159 (2012).
- Fielding, L. NMR methods for the determination of protein-ligand dissociation constants. Curr Top Med Chem. 3 (1), 39-53 (2003).
- Leavitt, S., Freire, E. Direct measurement of protein binding energetics by isothermal titration calorimetry. Curr Opin Struct Biol. 11 (5), 560-566 (2001).
- McDonnell, J. M. Surface plasmon resonance: towards an understanding of the mechanisms of biological molecular recognition. Curr Opin Chem Biol. 5 (5), 572-577 (2001).
- Rich, R. L., Myszka, D. G. Advances in surface plasmon resonance biosensor analysis. Curr Opin Biotechnol. 11 (1), 54-61 (2000).
- Pesquero, N. C., et al. Real-time monitoring and kinetic parameter estimation of the affinity interaction of jArtinM and rArtinM with peroxidase glycoprotein by the electrogravimetric technique. Biosens Bioelectron. 26 (1), 36-42 (2010).
- Scheuermann, T. H., Padrick, S. B., Gardner, K. H., Brautigam, C. A. On the acquisition and analysis of microscale thermophoresis data. Anal Biochem. 496, 79-93 (2016).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados