Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Un gasoducto de bioinformática para analizar de forma precisa y eficiente los transcriptomes de microRNA en las plantas
* Estos autores han contribuido por igual
En este artículo
Resumen
Una canalización bioinformática, a saber, miRDeep-P2 (miRDP2 para abreviar), con criterios de miRNA de plantas actualizados y un algoritmo revisado, podría analizar con precisión y eficiencia transcriptometrías de microARN en plantas, especialmente para especies con genomas complejos y grandes.
Resumen
Los MicroRNAs (miRNAs) son pequeños ARN endógenos (sARN) de 20 a 24 nucleótidos (nt) que existen ampliamente en plantas y animales que desempeñan un papel potente en la regulación de la expresión génica a nivel post-transcripción. La secuenciación de bibliotecas de ARNS mediante métodos de secuenciación de próxima generación (NGS) se ha empleado ampliamente para identificar y analizar transcriptomías de miRNA en la última década, lo que ha dado lugar a un rápido aumento del descubrimiento de miRNA. Sin embargo, surgen dos grandes desafíos en la anotación de miRNA vegetal debido a la creciente profundidad de las bibliotecas de ARNS secuenciados, así como al tamaño y la complejidad de los genomas de las plantas. En primer lugar, muchos otros tipos de ARN, en particular, ARN de interferencia corta (siRNAs) de bibliotecas de SRNA, son erróneamente anotados como miRNAs por muchas herramientas computacionales. En segundo lugar, se convierte en un proceso extremadamente lento para analizar transcriptomes de miRNA en especies vegetales con genomas grandes y complejos. Para superar estos desafíos, recientemente actualizamos miRDeep-P (una herramienta popular para los análisis de transcriptoma de miRNA) a miRDeep-P2 (miRDP2 para abreviar) empleando una nueva estrategia de filtrado, revisando el algoritmo de puntuación e incorporando miRNA de planta recién actualizado criterios de anotación. Probamos miRDP2 contra poblaciones de ARNS secuenciados en cinco plantas representativas con creciente complejidad genómica, incluyendo Arabidopsis, arroz, tomate, maíz y trigo. Los resultados indican que miRDP2 procesó estas tareas con muy alta eficiencia. Además, miRDP2 superó a otras herramientas de predicción con respecto a la sensibilidad y precisión. En conjunto, nuestros resultados demuestran miRDP2 como una herramienta rápida y precisa para analizar los transcriptomes de miRNA de plantas, por lo tanto, una herramienta útil para ayudar a la comunidad a anotar mejor los miRNAs en las plantas.
Introducción
Uno de los descubrimientos más emocionantes de las últimas dos décadas en biología es el papel proliferante de las especies de ARNS en la regulación de diversas funciones del genoma1. En particular, los miRNAs constituyen una clase importante de ARN de 20 a 24 nt en eucariotas, y funcionan principalmente a nivel post-transcripcional como reguladores genéticos prominentes a lo largo de las etapas de desarrollo del ciclo de vida, así como en respuestas de estímulo y estrés2,3. En las plantas, los miRNAs surgen de transcripciones primarias llamadas pri-miRNAs, que generalmente son transcritas por ARN polimerasa II como unidades de transcripción individuales4,5. Procesados por maquinaria celular evolutivamente conservada (Drosha RNase III en animales, como DICER en plantas), los pri-miRNAs se extirpan en los precursores inmediatos del miRNA, pre-miRNAs, que contienen secuencias que forman estructuras de bucle de tallo intramolecular6,7. Los pre-miRNA se procesan en intermedios de doble cadena, a saber, dúplex de miRNA, que consisten en la hebra funcional, miRNA maduro y el socio menos frecuentemente funcional, miRNA*2,8. Después de cargarlos en el complejo de silenciamiento inducido por ARN (RISC), los miRNAs maduros podrían reconocer sus objetivos de ARNm en función de la complementariedad de la secuencia, lo que dio lugar a una función reguladora negativa2,8. los miRNAs podrían desestabilizar sus transcripciones objetivo o impedir la traducción de objetivos, pero la forma anterior está dominada en las plantas8,9.
Desde el descubrimiento fortuito del primer miRNA en el nematodo Caenorhabditis elegans10,11, se ha investigado mucho con la identificación de miRNA y su análisis funcional, especialmente después de la disponibilidad del método NGS. La amplia aplicación del método NGS ha promovido en gran medida la utilización de herramientas computacionales que fueron diseñadas para capturar la característica única de los miRNAs, como la estructura de bucle de vástago de precursores y su acumulación preferencial de lecturas de secuencia en miRNA maduro y miRNA*. Como resultado, los investigadores han logrado un éxito notable en la identificación de miRNAs en diversas especies. Basándonos en un modelo de probabilidad12descrito anteriormente, desarrollamos miRDeep-P13, que fue la primera herramienta computacional para descubrir miRNAs de plantas a partir de datos NGS. miRDeep-P estaba específicamente dirigido a la conquista de los desafíos de la decodificación de miRNAs vegetales con longitud precursora más variable y grandes familias paralocitas13,14,15. Después de su lanzamiento, este programa ha sido descargado miles de veces y utilizado para anotar transcriptomes de miRNA en más de 40 especies de plantas16. Propulsado por herramientas basadas en NGS como miRDeep-P, se ha producido un aumento espectacular en el número de miRNAs registrados en el repositorio público miRNAmiRBase 17,donde actualmente se alojan más de 38.000 elementos de miRNA (versión 22.1) en comparación con sólo 500 elementos de miRNA (versión 2.0) en 200818.
Sin embargo, han surgido dos nuevos desafíos a partir de la anotación del miRNA vegetal. En primer lugar, las altas proporciones de falsos positivos han afectado en gran medida la calidad de las anotaciones de miRNA vegetal16,19 por las siguientes razones: 1) un diluvio de ARN (ARN) de interferencia corta endógena (SIRNAs) de las bibliotecas de ARNS de NGS fueron erróneamente anotados como miRNAs debido a la falta de un criterio de anotación de miRNA estricto; 2) para las especies sin información de miRNA a priori, los falsos positivos previstos sobre la base de los datos de NGS son difíciles de eliminar. Utilizando miRBase como ejemplo, Taylor yotros 20 encontraron que un tercio de las entradas de miRNA de plantas en el repositorio público21 (versión 21) carecían de pruebas de apoyo convincentes e incluso tres cuartas partes de las familias de miRNA de plantas eran cuestionables. En segundo lugar, se convierte en un proceso extremadamente lento para predecir miRNAs vegetales con genomas grandes y complejos16. Para superar estos desafíos, actualizamos miRDeep-P agregando una nueva estrategia de filtrado, revisando el algoritmo de puntuación e integrando nuevos criterios para la anotación de miRNA de planta, y lanzamos la nueva versión miRDP2. Además, probamos miRDP2 utilizando conjuntos de datos de ARN NGS con tamaños de genoma sin aumento gradual: Arabidopsis, arroz, tomate, maíz y trigo. En comparación con otras cinco herramientas ampliamente utilizadas y su versión antigua, miRDP2 analizó estos datos de ARNS y analizó los transcriptomas de miRNA más rápido con una precisión y sensibilidad mejoradas.
Contenido del paquete miRDP2
El paquete miRDP2 consta de seis scripts Perl documentados que el script bash preparado debe ejecutar secuencialmente. De los seis scripts, tres (convert_bowtie_to_blast.pl, filter_alignments.ply excise_candidate.pl) se heredan de miRDeep-P. Los otros scripts se modifican de la versión original. Las funciones de los seis scripts se describen a continuación:
preprocess_reads.pl filtra las lecturas de entrada, incluidas las lecturas demasiado largas o demasiado cortas (<19 nt o >25 nt), y lee correlacionadas con secuencias de Rfam ncRNA, así como lecturas con RPM (lecturas por millón) inferiores a 5. A continuación, el script recupera las lecturas correlacionadas con secuencias maduras de miRNA conocidas. Los archivos de entrada son lecturas originales en formato FASTA/FASTQ y salida bowtie2 de asignaciones de lecturas a secuencias miRNA y ncRNA.
La fórmula para calcular RPM es la siguiente:
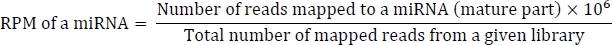
convert_bowtie_to_blast.pl cambia el formato bowtie al formato BLAST-parsed. El formato analizado por BLAST es un formato separado por tabular personalizado derivado del formato NCBI BLASToutput estándar.
filter_alignments.pl filtra las alineaciones de las lecturas de secuenciación profunda a un genoma. Filtra las alineaciones parciales, así como las lecturas multialineadas (corte de frecuencia especificado por el usuario). La entrada básica es un archivo en formato BLAST-parsed.
excise_candidate.pl elimina las posibles secuencias precursoras de una secuencia de referencia utilizando lecturas alineadas como directrices. La entrada básica es un archivo en formato BLAST-parsed y un archivo FASTA. La salida es todas las secuencias precursoras potenciales en formato FASTA.
mod-miRDP.pl necesita dos archivos de entrada, archivo de firma y archivo de estructura, que se modifica desde el algoritmo de miRDeep-P principal cambiando el sistema de puntuación con parámetros específicos de la planta. Los archivos de entrada son un archivo de estructura precursor de corchete de punto y lee el archivo de firma de distribución.
mod-rm_redundant_meet_plant.pl necesita tres archivos de entrada: chromosome_length, precursores y original_prediction generados por mod-miRDP.pl. Genera dos archivos de salida, el archivo predicho no redundante y el archivo predicho filtrado por los criterios de miRNA de planta recién actualizados. Los detalles sobre el formato del archivo de salida se describen en la sección 1.4.
Protocolo
1. Instalación y pruebas
- Descargue las dependencias requeridas: Bowtie222 y RNAfold23. Se recomiendan los paquetes compilados.
- Descarga Bowtie2, una herramienta de mapeo de lectura, desde su sitio de origen (http://bowtie-bio.sourceforge.net/bowtie2/index.shtml).
- Descargar RNAfold, una herramienta del paquete de Viena utilizada para predecir la estructura secundaria del ARN, desde http://www.tbi.univie.ac.at/~ivo/RNA/.
- Antes de instalar miRDP2, asegúrese de que estas dos dependencias están correctamente instaladas y personalice el archivo de entorno bash (por ejemplo, .bashrc) para establecer una ruta de acceso correcta para estas dos dependencias.
NOTA: Otras herramientas de mapeo como Bowtie24 también son adecuadas para miRDP2; Bowtie o Bowtie2 se pueden utilizar después de la versión 1.1.3.
- Para descargar el paquete miRDP2, vaya a https://sourceforge.net/projects/mirdp2/files/latest_version/ y obtenga los archivos tarball.
- Antes de instalar miRDP2, asegúrese de que Perl está en PATH. Para instalar miRDP2, extraiga todo el contenido del archivo tarball descargado en una carpeta (líneas de comandos como en 1.4.2) y, a continuación, establezca la ruta de la carpeta en PATH.
NOTA: Se recomienda un ordenador o nodo informático con al menos 8 GB de RAM y 100 GB de almacenamiento para ejecutar miRDP2. - Pruebe la canalización MiRDP2.
- Para comprobar si miRDP2 se ha instalado correctamente, utilice los datos de prueba y la salida esperada que se encuentra en https://sourceforge.net/projects/mirdp2/files/TestData/. Los datos de prueba contienen un archivo de secuenciación GSM con formato y un archivo de genoma de Arabidopsis thaliana.
- Mueva todos los archivos descargados al directorio de trabajo actual:
mv miRDP2-v*.tar.gz TestData.tar.gz ncRNA_rfam.tar.gz
cd - Extraiga los archivos de tarball comprimidos:
tar –xvzf miRDP2-v*.tar.gz
tar –xvzf TestData.tar.gz
tar –xvzf ncRNA_rfam.tar.gz - Construir el índice de referencia del genoma de Arabidopsis:
bowtie2-build -f ./TestData/TAIR10_genome.fa ./TestData/TAIR10_genome - Cree el índice de referencia ncRNA:
bowtie2-build -f ./ncRNA_rfam.fa ./1.1.3/script/index/rfam_index - Ejecute la canalización miRDP2:
bash ./1.1.3/miRDP2-v1.1.3_pipeline.bash –g ./TestData/TAIR10_genome.fa -i ./ TestData/TAIR 10_genome –f ./TestData/GSM2094927.fa –o .
NOTA: Los comandos de Linux utilizados están en negrita y cursiva, con opciones de línea de comandos en cursiva. *indica la versión de miRDP2 (la versión actual es 1.1.3). El comando bowtie2-build debe tardar aproximadamente 10 minutos, y la canalización miRDP2 debe terminar en cuestión de minutos
- Compruebe las salidas de prueba.
- Tenga en cuenta que una carpeta denominada 'GSM2094927-15-0-10' se genera automáticamente en , que contiene todos los archivos y resultados intermedios.
- Compruebe que el archivo de salida delimitado por tabulaciones GSM2094927-15-0-10_filter_P_prediction, la salida final de los miRNAs pronosticados, contiene columnas que indican el identificador del cromosoma, la dirección del hilo, las lecturas representativas id, precursor id, ubicación madura del miRNA, precursor ubicación, secuencia madura y secuencia precursora. Tenga en cuenta el archivo de cama adicional derivado de este archivo para facilitar el análisis posterior.
- Compruebe el archivo "progress_log", que proporciona información sobre los pasos terminados, y los archivos "script_log" y "script_err", que contienen la salida del programa y las advertencias.
NOTA: Actualmente, hemos probado miRDP2 en dos plataformas Linux, incluyendo CentOS versión 6.5 en un servidor de clúster, y Cygwin 2.6.0 en el sistema PC Windows, y miRDP2 debe funcionar en sistemas similares que admitan Perl.
2. Identificación de nuevos miRNAs
- Antes de ejecutar la canalización, asegúrese de que las lecturas de entrada se preprocesan en el formato adecuado.
NOTA: La nueva versión 1.1.3 de miRDP2 puede aceptar archivos de formato FASTQ originales como entradas, aunque el proceso de formateo de lecturas se lleva a cabo como en versiones anteriores.- En primer lugar, retire los adaptadores de los extremos de 5' y 3' de las lecturas de secuenciación profunda (si están presentes).
- En segundo lugar, analice las lecturas de secuenciación profunda en formato FASTA.
- En tercer lugar, quite la redundancia tal que las lecturas con la secuencia idéntica se representen con una entrada FASTA única y única.
- Por último, asegúrese de que todos los identificadores FASTA son únicos. Cada identificador de secuencia debe terminar con un '_x' y un entero, que indica el número de copia de la secuencia exacta que se recuperó en los conjuntos de datos de secuenciación profunda. Una manera de garantizar un identificador FASTA único es incluir un número en ejecución en el identificador. Para obtener referencia, consulte el archivo GSM2094927.fa en los datos de prueba (https://sourceforge.net/projects/mirdp2/files/TestData/).
- Consulte los siguientes ejemplos de lecturas con formato correcto:
>read0_x29909
TTTGGGATTGAAGGGAGCTCTA
>read1_x36974
TTCCACAGCTTCTTGAACTG
>read2_x32635
TTCCACAGCTTCTTGAACTT
- Cree índices de referencia.
- Para la referencia del genoma, para ahorrar tiempo, descargue los archivos de índice Bowtie2 desde el sitio web de iGenomes (https://support.illumina.com/sequencing/sequencing_software/igenome.html) si se han indexado las secuencias del genoma de la especie de interés. De lo contrario, los usuarios indexan secuencias de referencia y mantienen el archivo de índice durante un tiempo hasta que el proyecto haya terminado, ya que es posible que sea necesario volver a indizar la secuencia del genoma. Los detalles sobre cómo indexar una referencia del genoma se incluyen en el manual bowtie2 (http://bowtie-bio.sourceforge.net/bowtie2/manual.shtml).
- También se necesita otro índice ncRNA no miRNA para filtrar las secuencias ruidosos de otros fragmentos de ARN que no codifican. El archivo es una colección de secuencias ncRNA principales de Rfam, incluyendo rRNA, tRNA, snRNA y snoRNA. Para crear este índice, consulte la parte 1.4, ya que el índice debe colocarse y nombrarse correctamente, es decir, /script/index/rfam_index.
- Ejecute miRDP2.
- Para utilizar miRDP2 para detectar nuevos miRNAs a partir de datos de secuenciación profunda, ejecute el script bash en el paquete para iniciar la canalización de análisis (se puede encontrar un ejemplo en el paso 1.4):
/miRDP2-v*.*_pipeline.bash –g -i -f -o
donde * indica la versión del script bash de canalización. Hay tres parámetros que se pueden modificar: 1) el número de ubicaciones diferentes a las que se podría asignar una lectura, 2) el número de discordancia para ejecutar bowtie2 y 3) el umbral de RPM (lecturas por millón). Modifique estas opciones mediante las opciones –L, -M y –R, respectivamente. Una explicación detallada se encuentra en la sección 3.1.
- Para utilizar miRDP2 para detectar nuevos miRNAs a partir de datos de secuenciación profunda, ejecute el script bash en el paquete para iniciar la canalización de análisis (se puede encontrar un ejemplo en el paso 1.4):
- Compruebe las salidas miRDP2.
- Tenga en cuenta que la carpeta de salida se generará automáticamente en y se denominará '-15-0-10'; los últimos 3 números indican los valores (predeterminado en este caso) para los parámetros 1, 2 y 3, respectivamente. El archivo _filter_P_prediction contiene información de los miRNAs finales predichos que satisfacen los criterios de anotación de miRNA de planta recién actualizados. Los detalles sobre el formato del archivo de salida se describen en la parte 1.4.
3. Modificaciones y precaución usando miRDP2
- Parámetros que se pueden modificar
- Utilice la opción '-L' para establecer el límite de cuántas ubicaciones podría asignarse una lectura (parámetro 1). La asignación de lectura a demasiados sitios posiblemente están asociadas con secuencias de repetición y no es probable que los miRNAs. El valor predeterminado es 15. Para especies específicas, si hay familias de miRNA con muchos miembros, el primer parámetro puede aumentarse manualmente para adaptarse al paisaje del genoma.
- Utilice la opción '-M' para establecer las discrepancias permitidas para bowtie (parámetro 2). El valor predeterminado es 0.
- Utilice la opción '-R' para establecer el umbral para las lecturas potencialmente correspondientes a los miRNAs maduros (parámetro 3). Para reducir el consumo de tiempo y los falsos positivos, filtre las lecturas por RPM. Sólo las lecturas que superen un determinado umbral de RPM pueden representar secuencias maduras de miRNAs en lugar de ruido de fondo, y se conservarían para un análisis posterior. El valor predeterminado es 10 RPM.
- Tenga en cuenta que cambiar estos parámetros puede afectar potencialmente al rendimiento y al consumo de tiempo. En general, un aumento de los parámetros 1 y 2 y una disminución del parámetro 3 generarían un resultado menos estricto y un tiempo de ejecución más largo y viceversa.
- Redundancia y miRNA*
- Tenga en cuenta que los miRNAs de salida de miRDP2 pueden diferir de los miRNAs conocidos. Encontramos que esto se debe principalmente a una de dos razones: heterogeneidad de los miRNAs maduros o la abundancia relativa de miRNA y miRNA*. Encontramos que esto no afecta a la selección de longitud óptima de los precursores y el perfilado de genes miRNA conocidos.
Resultados
El oleoducto de anotación miRNA, miRDP2, descrito en el presente documento se aplica a 10 bibliotecas públicas de SRNA-seq de 5 especies de plantas con aumento gradual de la longitud del genoma, incluyendo Arabidopsis thaliana, Oryza sativa (arroz), Solanum lycopersicum (tomate), Zea mays (maíz) y Triticum aestivum (trigo)(Figura 1A). En general, para cada especie, 2 bibliotecas de ARNs representativos de diferentes tejidos (co...
Discusión
Con la llegada de NGS, se ha identificado un gran número de loci de miRNA a partir de una cantidad cada vez mayor de datos de secuenciación de ARNS en diversas especies29,30. En la base de datos de la comunidad centralizada miRBase21, los elementos de miRNA depositados han aumentado casi 100 veces en la última década. Sin embargo, en comparación con los miRNAs en animales, los miRNAs vegetales tienen muchas características únicas qu...
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este trabajo ha sido apoyado por beijing Academy of Agriculture and Forestry Sciences (KJCX201917, KJCX20180425 y KJCX20180204) a XY y National Natural Science Foundation of China (31621001) a LL.
Materiales
| Name | Company | Catalog Number | Comments |
| Computer/computing node | N/A | N/A | Perl is required; at least 8 GB RAM and 100 GB storage are recommended |
Referencias
- Ghildiyal, M., Zamore, P. D. Small silencing RNAs: an expanding universe. Nature Reviews Genetics. 10 (2), 94-108 (2009).
- Bartel, D. P. MicroRNAs: target recognition and regulatory functions. Cell. 136 (2), 215-233 (2009).
- Moran, Y., Agron, M., Praher, D., Technau, U. The evolutionary origin of plant and animal microRNAs. Nature Ecology Evolution. 1 (3), 27 (2017).
- Xie, Z., et al. Expression of Arabidopsis MIRNA genes. Plant Physiology. 138 (4), 2145-2154 (2005).
- Zhao, X., Zhang, H., Li, L. Identification and analysis of the proximal promoters of microRNA genes in Arabidopsis. Genomics. 101 (3), 187-194 (2013).
- Bologna, N. G., Mateos, J. L., Bresso, E. G., Palatnik, J. F. A loop-to-base processing mechanism underlies the biogenesis of plant microRNAs miR319 and miR159. EMBO JOURNAL. 28 (23), 3646-3656 (2009).
- Rogers, K., Chen, X. Biogenesis, turnover, and mode of action of plant microRNAs. Plant Cell. 25 (7), 2383-2399 (2013).
- Voinnet, O. Origin, biogenesis, and activity of plant microRNAs. Cell. 136 (4), 669-687 (2009).
- Iwakawa, H. O., Tomari, Y. The Functions of MicroRNAs: mRNA Decay and Translational Repression. Trends in Cell Biology. 25 (11), 651-665 (2015).
- Lee, R. C., Feinbaum, R. L., Ambros, V. The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14. Cell. 75 (5), 843-854 (1993).
- Wightman, B., Ha, I., Ruvkun, G. Posttranscriptional regulation of the heterochronic gene lin-14 by lin-4 mediates temporal pattern formation in C. elegans. Cell. 75 (5), 855-862 (1993).
- Friedlander, M. R., et al. Discovering microRNAs from deep sequencing data using miRDeep. Nature Biotechnology. 26 (4), 407-415 (2008).
- Yang, X., Li, L. miRDeep-P: a computational tool for analyzing the microRNA transcriptome in plants. Bioinformatics. 27 (18), 2614-2615 (2011).
- Meyers, B. C., et al. Criteria for annotation of plant MicroRNAs. Plant Cell. 20 (12), 3186-3190 (2008).
- Yang, X., Zhang, H., Li, L. Global analysis of gene-level microRNA expression in Arabidopsis using deep sequencing data. Genomics. 98 (1), 40-46 (2011).
- Kuang, Z., Wang, Y., Li, L., Yang, X. miRDeep-P2: accurate and fast analysis of the microRNA transcriptome in plants. Bioinformatics. , (2018).
- Kozomara, A., Birgaoanu, M., Griffiths-Jones, S. miRBase: from microRNA sequences to function. Nucleic Acids Research. 47 (1), 155-162 (2019).
- Griffiths-Jones, S., Saini, H. K., van Dongen, S., Enright, A. J. miRBase: tools for microRNA genomics. Nucleic Acids Research. 36, 154-158 (2008).
- Axtell, M. J., Meyers, B. C. Revisiting Criteria for Plant MicroRNA Annotation in the Era of Big Data. Plant Cell. 30 (2), 272-284 (2018).
- Taylor, R. S., Tarver, J. E., Hiscock, S. J., Donoghue, P. C. Evolutionary history of plant microRNAs. Trends in Plant Science. 19 (3), 175-182 (2014).
- Kozomara, A., Griffiths-Jones, S. miRBase: annotating high confidence microRNAs using deep sequencing data. Nucleic Acids Research. 42, 68-73 (2014).
- Langmead, B., Salzberg, S. L. Fast gapped-read alignment with Bowtie 2. Nature Methods. 9 (4), 357-359 (2012).
- Lorenz, R., et al. ViennaRNA Package 2.0. Algorithms for Molecular Biology. 6, 26 (2011).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biology. 10 (3), 25 (2009).
- An, J., Lai, J., Sajjanhar, A., Lehman, M. L., Nelson, C. C. miRPlant: an integrated tool for identification of plant miRNA from RNA sequencing data. BMC Bioinformatics. 15, 275 (2014).
- Lei, J., Sun, Y. miR-PREFeR: an accurate, fast and easy-to-use plant miRNA prediction tool using small RNA-Seq data. Bioinformatics. 30 (19), 2837-2839 (2014).
- Evers, M., Huttner, M., Dueck, A., Meister, G., Engelmann, J. C. miRA: adaptable novel miRNA identification in plants using small RNA sequencing data. BMC Bioinformatics. 16, 370 (2015).
- Mathelier, A., Carbone, A. MIReNA: finding microRNAs with high accuracy and no learning at genome scale and from deep sequencing data. Bioinformatics. 26 (18), 2226-2234 (2010).
- Zhu, Q. H., et al. A diverse set of microRNAs and microRNA-like small RNAs in developing rice grains. Genome Research. 18 (9), 1456-1465 (2008).
- Fahlgren, N., et al. MicroRNA gene evolution in Arabidopsis lyrata and Arabidopsis thaliana. Plant Cell. 22 (4), 1074-1089 (2010).
- Fromm, B., et al. A Uniform System for the Annotation of Vertebrate microRNA Genes and the Evolution of the Human microRNAome. Annual Review of Genetics. 49, 213-242 (2015).
- Blevins, T., et al. Identification of Pol IV and RDR2-dependent precursors of 24 nt siRNAs guiding de novo DNA methylation in Arabidopsis. Elife. 4, 09591 (2015).
- Zhai, J., et al. A One Precursor One siRNA Model for Pol IV-Dependent siRNA Biogenesis. Cell. 163 (2), 445-455 (2015).
- Werner, S., Wollmann, H., Schneeberger, K., Weigel, D. Structure determinants for accurate processing of miR172a in Arabidopsis thaliana. Current Biology. 20 (1), 42-48 (2010).
- Mateos, J. L., Bologna, N. G., Chorostecki, U., Palatnik, J. F. Identification of microRNA processing determinants by random mutagenesis of Arabidopsis MIR172a precursor. Current Biology. 20 (1), 49-54 (2010).
- Vitsios, D. M., et al. Mirnovo: genome-free prediction of microRNAs from small RNA sequencing data and single-cells using decision forests. Nucleic Acids Research. 45 (21), 177 (2017).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados